Grundlagen der Apoptose
Werbung

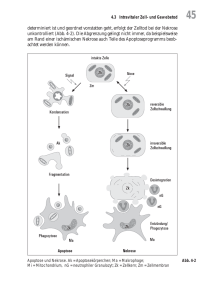
Grundlagen der Apoptose Agenda • Was ist Apoptose? (Morphologische u. biochemische Charakteristika) • Molekulare Grundlagen (pro- u. anti-apoptotische Mechanismen) • Apoptose-Signalkaskade intrinsisch / extrinsisch • Physiologische u. pathologische Funktionen • Apoptose in Tumor- und anderen Erkrankungen Zwei Arten von Zelltod Zwei Arten von Zelltod Nekrose Apoptose • Zellen schwellen an • Kern schwillt an • Vakuolen • Zellen schrumpfen • Chromatin kondensiert • „Membrane blebbing“ • Immunantwort (Entzündungsreaktion) • „reguläre“ Phagozytose • keine Immunantwort Charakteristika der Apoptose Morphologische: • • • • • • „Membrane Blebbing“ Chromatin-Kondensation Nukleäre Fragmentierung Verlust der Adhäsion, Abrundung Zelle schrumpft Apoptotische Körperchen („apoptotic bodies“) • Phagozytose (Absorption) durch Macrophagen oder benachbarte Zellen Biochemische: • Spaltung genomischer DNA -> „DNA-Leiter“ • Externalisierung von Phophatidylserin • Proteolytische Spaltung intrazellulärer Substrate • Aktivierung distinkter Proteasen (Caspasen: Aspartat spezifische Cysteinyl-Proteasen) 2.2 µm Charakteristika der Apoptose Morphologische: • • • • • • „Blebbing“ Chromatin-Kondensation Nukleäre Fragmentierung Verlust der Adhäsion, Abrundung Zelle schrumpft Apoptotische Körperchen („apoptotisch bodies“) • Phagozytose (Absorption) durch Macrophagen oder benachbarte Zellen Biochemische: • Spaltung genomischer DNA -> „DNA-Leiter“ • Externalisierung von Phophatidylserin • Proteolytische Spaltung intrazellulärer Substrate • Aktivierung distinkter Proteasen (Caspasen: Aspartat spezifische Cysteinyl-Proteasen) Charakteristika der Apoptose Morphologische: • • • • • • „Blebbing“ Chromatin-Kondensation Nukleäre Fragmentierung Verlust der Adhäsion, Abrundung Zelle schrumpft Apoptotische Körperchen („apoptotisch bodies“) • Phagozytose (Absorption) durch Macrophagen oder benachbarte Zellen Biochemische: • Spaltung genomischer DNA -> „DNA-Leiter“ • Externalisierung von Phophatidylserin • Proteolytische Spaltung intrazellulärer Substrate • Aktivierung distinkter Proteasen (Caspasen: Aspartat spezifische Cysteinyl-Proteasen) !Agarosegel Internukleosomale DNA-Spaltung Chromatin-Organisation DNA Doppelhelix Nukleosomales Filament Chromatin Filament Übergeordnete Strukturen Internukleosomale DNA-Spaltung Chromatin-Organisation DNA Doppelhelix Nukleosomales Filament Chromatin Filament Internukleosomale DNA-Spaltung !DNA-Spaltstellen von Endonukleasen !Agarosegel Physiologische Rolle der Apoptose A. B. C. D. E. F. B Formgebung Deletion überflüssiger Strukturen Kontrolle der Zellzahl (cell turnover) Eliminierung autoreaktiver Thymozyten Regulation der Immunantwort Eliminierung von Tumorzellen und virusinfizierter Zellen Müllerian duct Wolffian duct A C apoptotisch cells 3 - 5 days Deletion unerwünschter Zellen: Stem cells Rolle der Apoptose Physiologische Rolle der Apoptose ... 1. 2. 3. ... während der Embryonalentwicklung ... (in Vertebraten) bei der Homöostase (Erhalt der Gewebsstruktur) ... im Immunsystem negative Selektion Regulation der Immunantwort Deletion infizierter und abnormer Zellen Pathophysiologische Rolle der Apoptose... ... bei unzureichender Apoptose ... 1. 2. 3. ... Autoimmunerkrankungen ... Krebs ... Virusinfektion ... bei übermäßiger Apoptose ... 1. 2. 3. ... AIDS ... Schlaganfall ... bei neurodengenerativen Erkrankungen - Alzheimer - Parkinson - Retinitis Pigmentosa Homöostase Leben Differenzieren Teilen Sterben Teile Sterb en n Homöostase Leben Differenzieren Teilen Sterben Sterben Teilen Gleichgewicht Physiologische Rolle: Embryonalentwicklung Fuß eines Mäuseembryos. Die Ausbidung der Extremitäten beginnt bei den meisten Säugetieren als “Flosse” ohne Finger oder Zehen.. Kurz darauf entstehen Bereiche apoptotischer Zellen (interdigitale apoptotische Regionen) - zwischen den Bereichen, welche die Finger oder Zehen bilden werden. Diese sterbenden Zellen werden von Makrophagen aufgenommen, die zu diesen Bereichen wandern (grün). Zurück bleiben die zukünftigen Zehen bzw. Finger. Physiologische Rolle: Phagozytose Apoptose-Signale Intrinsische Signale Extrinsische Signale (Die Zelle detektiert selbst, daß sie überflüssig oder schädlich ist.) (Die Zelle erhält ein Aktivierungssignal für Apoptose.) • Stress (Reaktive Sauerstoffverbindungen “ROS”, Ca2+, pH, ATP-Level, Glukose-Level) • Chemotherapeutische Agentien • UV-Strahlen > DNA-Schaden • Entzug von Wachstumsfaktoren • Entzug von Überlebensfaktoren (Anoikis = Verlust der Adhäsion an die extrazelluläre Matrix) • Todesliganden (Death Ligands) Apoptose-Signale Apoptosemechanismen Wurm (Nematode) Fruchtfliege C. elegans Caenorhabditis elegans Drosophila Drosophila melanogaster Invertebraten C. elegans besteht aus 1090 somatischen Zellen, 131 Zellen sterben durch Apoptose, so daß 959 Zellen den voll entwickelten Wurm bilden. Mensch Homo sapiens Vertebraten Apoptosemechanismen Wurm (Nematode) Fruchtfliege C. elegans Caenorhabditis elegans Drosophila Drosophila melanogaster Invertebraten Einfaches Modell - evolutionär einfacher, prototypischer Organismus oder - stark komprimierte oder vereinfachte Version eines komplexeren Organismus. Mensch Homo sapiens Vertebraten Apoptosemechanismen Wurm (Nematode) Fruchtfliege C. elegans Caenorhabditis elegans Drosophila Drosophila melanogaster Invertebraten Einfaches Modell - evolutionär einfacher, prototypischer Organismus oder - stark komprimierte oder vereinfachte Version eines komplexeren Organismus. Komplexes Modell Mensch Homo sapiens Vertebraten Apoptosemechanismen Wurm (Nematode) Fruchtfliege C. elegans Caenorhabditis elegans Drosophila Drosophila melanogaster Invertebraten Einfaches Modell - evolutionär einfacher, prototypischer Organismus oder - stark komprimierte oder vereinfachte Version eines komplexeren Organismus. Komplexes Modell Mensch Homo sapiens Vertebraten Komplexeres Modell Apoptose: Evolutionäre Konservierung Caenorhabditis elegans Regulator EGL-1 CED-9 Adapter CED-4 Effektor CED-3 Apoptose Apoptose: Evolutionäre Konservierung Vertebraten EGL-1 CED-9 Intrazelluläre Membran Caenorhabditis elegans Mitochondrium Bik Bcl-2 Bad Adapter CED-4 Effektor CED-3 Caspase-9 Apoptose Apoptose Apaf-1 intrinsisch Apoptose: Evolutionäre Konservierung Vertebraten EGL-1 CED-9 Adapter CED-4 Effektor Intrazelluläre Membran Caenorhabditis elegans Rezeptor Mitochondrium Plasmamembran Bik Bcl-2 CD95 Bad Apaf-1 FADD CED-3 Caspase-9 Caspase-8 Apoptose Apoptose Apoptose intrinsisch extrinsisch Caspasen • • • • Erstmals 1992 beschrieben (ICE: Interleukin-1β Converting Enzyme) Identifizierung von CED-3 in C. elegans - notwendig für Apoptose ist identisch mit ICE Familie der Cystein-Proteasen, spalten nach Aspartat (Cysteinyl-Aspartasen) 1994 erste Struktur veröffentlicht S Caspase-4 CARD L S Caspase-1 CARD L S Caspase-7 L S Caspase-3 L S Caspase-6 L S L S L S L S L S L S Caspase-8 Caspase-10 Caspase-9 Caspase-2 Caspase-14 DED DED DED DED CARD CARD Differenzierung von Keratinozyten ? CARD: Caspase recruiting domain DED: death effector domain L: large subunit S: small subunit Exekution L Induktion CARD ? Caspase-5 Inflammation Humane Caspasen Caspasen - in anderen Organismen Mensch und Maus Caspase-1 Caspase-2 Caspase-3 Caspase-4 Caspase-5 Caspase-6 Caspase-7 Caspase-8 Caspase-9 C. elegans CED-3 CSP-1 CSP-2Drosophila Caspases Drosophila melanogaster DAMM DCP-1 DECAY DREDD DrICE DRONC STRICA Caspase-10 Zebrafisch Caspase-11 Caspy-1 Caspy-2 Casp-3 Casp-8 Caspase-12 Caspase-14 DED CARD Death effector domain Caspase recruitment domain Pyrin Pyrin Pyrin Pyrinreiche Domäne / DAPIN unbekanntes Motif Aktivierung von Caspasen Inaktives Zymogen (Pro-Caspase-1) Prodomäne His-Cys Diade SHG QACRG p20 p10 identische oder andere Caspase Autoproteolyse des Intermediates p10 p20 Prodomäne p10 Prodomäne Aktive Caspase-1 (p20/p10)2 Tetramer Caspase-9 Substratspezifität von Caspasen Tetrapeptid-Substratspezifität (P4-P3-P2-P1) Asp an P1 Apoptose-Signalkaskade Apoptosesignal InitiatorCaspase D D D D Caspase-8 Caspase-9 Caspase-3 Caspase-6 Caspase-7 EffektorCaspase Spaltung von Substraten D D D A) Auflösung B) Chromatinkondensation C) Inaktivierung von der Zellstruktur DNA-Fragmentierung DNA-Reparatur und Überlebensproteinen Caspase-Kaskade Caspase-9 Caspase-7 Caspase-3 Caspase-6 Caspase-8 Caspase-2 Caspase-10 Caspase-Substrate Inaktivierung von Apoptoseinhibitoren: CAD/ICAD CAD Degradation intrazellulärer Strukturen: Deregulation von Aktivitäten: + + Regulation Katalyse tion a l gu Re Ka tal yse Chromatinkondensation und DNA-Fragmentierung Casp3(-/-) WT Con ETO DOX TNF Con ETO 0 24 0 6 12 18 24 WT KO Con ETO 0 24 0 6 12 18 24 Chromatinkondensation und DNA-Fragmentierung Casp3(-/-) WT Con ETO DOX TNF Con ETO 0 24 0 6 12 18 24 WT KO Con ETO 0 24 0 6 12 18 24 Intrinsische Regulation Dimerisierung Regulation Porenbildung Membrananker BH1 BH2 TM Bcl-xL Bcl-w Mcl-1 A1/Bfl1 Boo/Diva Nrf3 BH1 BH2 TM Bak Bok/Mtd TM Bad Bik Blk Bmf Hrk/DP5 Bnip3 Nix Noxa Puma Anti-apoptotische Bcl-2 Proteine Bcl-2 BH4 BH3 Pro-apoptotische Bcl-2 Proteine Bax BH3 “BH3-only” Bim BH3 Bid BH3 Entscheidung am Mitochondrium Freisetzung von Cytochrom c Apoptose Cytochrom c als Signalprotein Pro-apoptotische Pro-apoptotische Bcl-2 Bcl-2 Proteine Anti-apoptotische Anti-apoptotische Bcl-2 Bcl-2 Proteine Bcl-2 Bcl-xL Bcl-w Mcl-1 Bax Bak Bax BH3-only Bik tBid Bim BIk Puma Bak Bcl-2 Bcl-xL Cyt C Mitochondrium Cytochrom c als Signalprotein Pro-apoptotische Pro-apoptotische Bcl-2 Bcl-2 Proteine Anti-apoptotische Anti-apoptotische Bcl-2 Bcl-2 Proteine Bcl-2 Bcl-xL Bcl-w Mcl-1 Bax Bak Bax BIM Bcl-xL BIK Bcl-2 BH3-only Bik tBid Bim BIk Bak Puma Cytochrom c als Signalprotein M. Sattler, Science 1997,275,983 Wechselwirkung der BH3-only Proteine Bax-Clustering QuickTime™ and a MPEG-4 Video decompressor are needed to see this picture. Cytochrom c-Freisetzung GFP-Cyt c Annexin-V DNA-dye Cytochrom c-Freisetzung GFP-Cyt c Annexin-V DNA-dye Das Apoptosom Smac/DIABLO Smac/DIABLO Stress IAPs Anti-apoptotische Anti-apoptotische Bcl-2 Bcl-2 Proteine Cytochrom c pro-caspase-9 Mitochondrium Apaf-1 Apaf-1 Pro-apoptotische Pro-apoptotische Bcl-2 Bcl-2 Proteine Apoptose Apoptose Caspase-3 Caspase-7 Caspase-6 APOPTOSOM Aktivierung von Apaf-1 Das Apoptosom Zusammenfassung • • • • • • • Effektor-Caspasen werden durch limitierte Proteolyse von InitiatorCaspasen aktiviert. Das Mitochondrium ist zentrale Regulationsstelle des intrinsischen Signalweges. Bcl-2 Familien Proteine regulieren die Freisetzung von Cytochrom c. Die Freisetzung von Cytochrom c initiiert die Bildung des Apoptosoms. Das Apoptosom wird von Apaf-1/Cyt c/Caspase-9 unter Mitwirkung von dATP gebildet. Caspasen werden durch IAPs inhibiert (xIAP inhibiert Caspase-9 und -3). Smac/DIABLO inaktivert (x)IAP und aktiviert somit Caspase-9 und -3. Rezeptor-vermittelte Apoptose TNF und die TNF-Rezeptor TNF-Rezeptor--Familie CD95L FasL APO-1L Apo2L TRAIL LIGHT HVEML Apo3L TWEAK TNFα LTα LTα/LTβ BLyS BAFF THANK OPGL TALL-1 TRANCE gp39 CD27L GITRL 4-1BBL APRIL RANKL CD40LCD30L CD70 gp34 OX-40L NGF ? CD95 Fas APO-1 DR6 OPG DR3 TNF-RII TR1 APO-3 CD120b wsl-1 TRAMP LT-βR DR5 DcR2 LARD FN-14 TNF-RI TRAIL-RII CD120a AIR TRICK2 Killer DR4 TRAIL-RI APO-2 DcR3 TR6 DcR1 TRID HVEM ATAR TR2 DcR3 TR6 BCMA TACI RANK OPG CD40 CD30 CD27 TR1 GITR 4-1BB Viral: OX-40 NGF-R SFV-T2 Va53 G4RG crmB Rezeptor-vermittelte Apoptose Der Todesligand und die TodesrezeptorTodesrezeptor-Familie CD95L (FasL/APO-1L) Apo2L/TRAIL Apo3L TNFα LTα OPG CD95 DcR3 DR4 DcR1 DR5 DcR2 DR3 FN-14 TNF-RI TNF-RII Rezeptor-vermittelte Apoptose Ligand DD DD Todesrezeptor DD: Death Domain Rezeptor-Trimerisierung Rezeptor-vermittelte Apoptose Ligand Rezeptor-Trimerisierung DISC Death Inducing Signalling Complex FADD Caspase-8 DD: Death Domain DD FADD: Fas Associated protein with Death Domain DED DED: Death Effector Domain Pro-caspase-8 DED DED „Auto-Aktivierung“ von Caspase-8 Ligand DED Rezeptor-Trimerisierung DED DED FADD DED Cys Cys „Induced proximity“ Modell Caspase-8 Rezeptor-vermittelte Apoptose Ligand Rezeptor-Trimerisierung DISC Death Inducing Signalling Complex FADD Caspase-8 aktive Caspase-8 Rezeptor-vermittelte Apoptose Ligand Rezeptor-Trimerisierung DISC FADD Death Inducing Signalling Complex Caspase-8 aktive Caspase-8 Aktivierung von Caspase-3 / -6 / -7 Apoptose Rezeptor-vermittelte Apoptose TNF FasR TNF-R1 TRADD FADD Caspase-8/-10 Caspase-10 Caspase-8 FADD Kinase Domäne TRADD RIP RAIDD Caspase-2 FasL CD95L-induzierte Apoptose Intrinsisch vs. Extrinsisch Apaf-1 Caspase-9 Adapter FADD Initiator Caspase-8 Apoptosom DISC Effektor Caspase-3 / -6 / -7 Verknüpfung der Signalwege: BID tBid Apaf-1 Caspase-9 Bid Apoptosom DISC Effektor Caspase-3 / -6 / -7 Caspase-Kaskade Caspase-9 Caspase-7 Caspase-8 Caspase-3 Mitochondrium Caspase-6 Caspase-2 tBid Bid Caspase-8 Caspase-10 Zusammenfassung • • • • • Rezeptor-vermittelte Apoptose wird durch den entsprechenden Liganden induziert (CD95/Fas/Apo1 - CD95L/FasL/Apo1L; TNF-R - TNF; TRAIL-R - TRAIL) CD95 trimerisiert und ermöglicht die Bindung von FADD an die intrazelluläre Todesdomäne (DD) Caspase-8 bindet über die DED an den FADD/Rezeptor-Komplex und bildet den DISC Caspase-8 wird autoproteolytisch gespalten und aktiviert EffektorCaspasen Bid wird von Caspase-8 zu tBid gespalten, das den mitochondrialen Apoptose-Signalweg aktivert (Amplifikation des Rezeptor-Signals) Apoptose-Signalweg Post-translationaler Signalweg der überwiegend auf spezifischer limitierter Proteolyse beruht! Physiologische Rolle des CD95 Rezeptor-Ligand Systems Immunsystem - Herunterregulation der Immunantwort - Erhalt der peripheren Toleranz - Aktivität zytotoxischer T-Zellen - Mechanismen der Immunprivilegierung (Auge, Hoden, Plazenta) Nicht-Immunsystem - Gewebshomöostase - Erneuerung des Epithelgewebes - Keratinocyten-Erneuerung - Tumor-Immunüberwachung Aktivierungsinduzierter Zelltod von T-Lymphozyten CD95 Klonale Expansion CD95L Zellzahl Deletion MHC TCR autokrin AG CD95L 3 6 9 parakrin 12 CD95L Tage Caspasen Rolle von CD95 für immunprivilegiertes Gewebe Normales Gewebe Immunprivilegierte Seite Zytotoxische T-Zelle Zytotoxische T-Zelle CD95 CD95L Apoptose CD95L Gen CD95L T-Zell-Rezeptor CD95 CD95L CD95 MHC/Antigen Apoptose Zielzelle CD95L Gen Zielzelle Rolle von CD95 Tumor-Gegenangriff Normales Gewebe Immunprivilegierte Seite Zytotoxische T-Zelle Zytotoxische T-Zelle CD95 CD95L Apoptose CD95L Gen CD95L T-Zell-Rezeptor CD95 CD95L CD95 MHC/Antigen Apoptose Zielzelle CD95L Gen Tumorzelle Apoptosemodulation durch intrazelluläre Bakterien Salmonella Shigella Yersinia YopJ/P SipB IpaB TRAF-2 RIP Procaspase-1, -2 Aktive Caspase-1, -2 IκB Kinase NF-B Apoptose Zytokine Apoptose Überlebensgene Virale Strategien zur Apoptosemodulation Viren nutzen anti-apoptotische Moleküle, um die Apoptose der Wirtszelle zu blockieren. • • Virale Caspase-Inhibitoren CrmA (Kuh-Pockenvirus) p35 (Baculovirus) Virales Flip (v-Flip) analog zu zellulärem (c-Flip) Virales Flip Ligand DED Rezeptor-Trimerisierung DED DED FADD DED Caspase-8 Cys Cys FADD Virales Flip Ligand DED v-Flip DED DED Rezeptor-Trimerisierung FADD DED v-Flip c-Flip (long) FADD c-Flip (short) Caspase-8 Cys X Cys CrmA (Cytokine Response Modifier A) Virale Strategien zur Apoptosemodulation Viren nutzen anti-apoptotische Moleküle, um die Apoptose der Wirtszelle zu blockieren. • • • • Virale Caspase-Inhibitoren CrmA (Kuh-Pockenvirus) p35 (Baculovirus) Virales Flip (v-Flip) analog zu zellulärem (c-Flip) Virale IAPs (erstes gefundenes IAP) Virale Bcl-2 Homologe Apoptoseresistenz von Tumoren Expression anti-apoptotischer Proteine Modulation des p53-Signalweges Bcl-2 Familien Proteine Bcl-2 Bcl-xL Mcl-1 p53 Lösliche Todesrezeptoren Soluble CD95 DcR3 Modulation des PI3K/Akt-Signalweges IAPs Survivin cIAP2 ML-IAP Flip INK4A/ARF ASPP PI3K PTEN AKT PI-9/SPI-6 Unterdrückung pro-apoptotischer Proteine Weitere Mechanismen Bcl-2 Familien Proteine Expression von Transportern: Bax, Nbk Apaf-1 Änderung der NF-κB Aktivität Caspasen Caspase-3 Caspase-8 Todesrezeptoren CD95 TRAIL-1 TRAIL-R2 Extrazelluläre Matrix MDR MRPs Tumorresistenz gegen Chemotherapie Anthracycline MDRÜberexpression Bcl-2Überexpression Methotrexate Topoisomeraseinhibitoren Akkumulation Primärziele Apoptose Cytarabin Nukleus γ-Bestrahlung Chemotherapeutika Caspasen DNA Schaden Mitochondrium PUMA Pro-apoptotische Bcl-2-Proteine Cytochrom cFreisetzung