Skript zur Vorlesung
Werbung

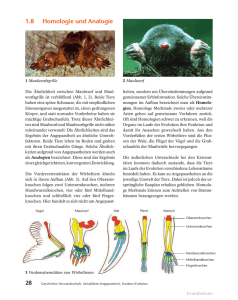
Skript zur Vorlesung „Allgemeine Evolutionsbiologie“ | WS 2015/2016 | A. Franzke & M. Koch Vorlesung 12 ♦ Korrektur! Im Skript zur zweiten Vorlesung hat sich leider ein Fehler eingeschlichen. Im Kapitel „Moderne Genetik“ hatte Mendel die Lebensdaten von Darwin bekommen. Das ist nun korrigiert. ♦ Polyploidisierung im Pflanzenreich − Bedeutung. Man schätzt, dass etwa 35 % aller rezenten Blütenpflanzenarten polyploid sind. In älteren Lehrbüchern bzw. Arbeiten zum Thema liest man häufig weiterhin, dass man davon ausgeht, dass etwa 75% der höheren Pflanzen in ihrer Entwicklungsgeschichte polyploide Vorfahren hatten. Heute müsste man eigentlich „100%“ schreiben: Genomanalysen der letzten Jahre haben erbracht, dass wohl an der Basis aller Samenpflanzen und auch an der Basis der Blütenpflanzen (Angiospermen) jeweils eine „whole genome duplication“ („WGD“) stattgefunden hat. „WGD“ ist eine heute immer üblicher werdende Bezeichnung für Polyploidie. Diese Genomduplikationen werden in Zusammenhang mit der großen Radiation und Dominanz der Samen-, bzw. insbesondere der Blütenpflanzen gebracht. Auch nach der Genomduplikation an der Basis der Angiospermen hat es weitere „WGDs“ innerhalb der Blütenpflanzen gegeben. „Auf dem Weg zur“ bedeutendsten Modellpflanze für botanische Forschungen, Arabidopsis thaliana (Brassicaceae), z.B. drei weitere. Grundsätzlich erfolgt nach einer „WGD“ eine (evolutionäre) Phase der Diploidisierung mit Genverlusten und Genomverkleinerung. Früher ging man eher davon aus, dass Speziation durch Allopolyploidie deutlich häufiger vorkommt als Artbildung durch Autopolyploidie. In den letzten 20 Jahren geht man immer mehr dazu über, auch der Autopolyploidie eine größere Bedeutung beizumessen. Ein Grund für die frühere Unterschätzung der Bedeutung der Autopolyploidie liegt auch wohl darin, dass es für Botaniker traditionell „kein Problem“ darstellt(e) einen einzigen Artnamen für gleich aussehende Pflanzen zu vergeben (taxonomisches Artkonzept), auch wenn es sich eigentlich um Sippen mit unterschiedlichen Ploidiestufen („Chromosomenrassen“) handelt. Möglicherweise stehen in vielen Fällen hinter diesen Cytotypen aber biologische Arten und damit wären die Artenzahlen bei Angiospermen möglicherweise stark unterschätzt: Soltis et al. 2007. Autopolyploidy in angiosperms: have we grossly underestimated the number of species? Taxon 56: 13–30. Generell kann man bei Artbildung durch Polyploidisierung davon ausgehen, dass solche Arten auch mehrfach entstanden sein können. Evolutionäre „Vorteile“ der Polyploidie: Es kann zu Heterosiseffekten (s.o.) kommen. Durch Redundanz von Genen können schädliche rezessive Allele − auch in der gametophytischen Phase − maskiert sein. Eine besondere Bedeutung der Genredundanz liegt darin, dass so ein „evolutionäres Experimentieren“ möglich ist. Ein Genduplikat kann zu neuen Funktionen evolvieren („neofunctionalization“) oder die beiden, durch Verdopplung entstandenen Gene, übernehmen jeweils Teilaufgaben des ursprünglichen Gens („subfunctionalization“). Häufiger ist Polyploidie auch mit einem Verlust von Selbstinkompatibilität und damit resultierender Möglichkeit zur Selbstbefruchtung verbunden und/oder die asexuelle Reproduktion wird gefördert. Beide Phänomene können im Rahmen einer Kolonisierung förderlich sein. Die arktische Flora ist durch einen hohen Anteil von Polyploiden gekennzeichnet und generell nimmt deren Häufigkeit und auch die Höhe der Ploidielevel hier in nördlicher Richtung zu. Offenbar konnten die (Allo)polyploiden durch Heterosis und/oder das Abpuffern von Inzuchtdepressionen sehr effizient die nach den Eiszeiten wieder eisfreien Gebiete erobern. Mögliche Nachteile von Auto- aber auch Allopolyploiden: Gestörte Meiosen und/oder epigenetisch instabile Genome. ♦ Polyploidisierung im Tierreich − Bedeutung. Polyploidie kommt bei Tieren generell sehr viel seltener vor als bei Pflanzen, gehäufter bei Fischen und Amphibien. Evtl. ist Polyploidie bei Tieren aber auch bisher „oft übersehen“ worden, da hier einfach, im Vergleich zu Pflanzen, weniger Chromosomenzählungen in Populationen durchgeführt wurden. In der Evolution der Tiere spielten aber auch „WGDs“ eine Rolle: In der frühen Wirbeltierevolution gab es zwei „WGDs“ und eine weitere ist spezifisch für die Echten Knochenfische (Teleostei). ♦ Artbildungs- & Aussterberaten. Es ist sicher nicht möglich, allgemeingültige konstante natürliche Artbildungs- und Aussterberaten, die für das gesamte Organismenreich gelten, anzugeben. Dennoch gibt es Versuche – trotz aller großen methodischen Probleme − solche Abschätzungen vorzunehmen. Hier, über viele Organismengruppen, „brutal“ gemittelte (!) Zahlen, um eine Vorstellung von den Größenordnungen zu bekommen. Zeitdauer einer Artbildung: Etwa 7 Mio. Jahre. Nochmal: Das ist ein Mittelwert und es gibt auch viele rezente (Pflanzen)Arten, die sicher deutlich jünger sind, die Artbildung hier also viel schneller passierte, evtl. nur wenige 100.000 Jahre. (Natürliche) Lebensdauer von Arten: Etwa 2 bis 10 Mio. Jahre. Die Aussterberate ist aktuell durch den Einfluss des Menschen wohl mindestens 100-mal höher („Anthropozän“). ♦ 1 Skript zur Vorlesung „Allgemeine Evolutionsbiologie“ | WS 2015/2016 | A. Franzke & M. Koch Systematik. Die Systematik ist das Teilgebiet der Biologie, welches sich mit der organismischen Vielfalt beschäftigt und dabei vier große Aufgabengebiete bearbeitet: 1.) Beschreibung und Benennung aller Arten; 2.) Schaffung eines phylogenetischen Klassifizierungssystems; 3.) Dokumentation evolutionären Wandels und 4.) Rekonstruktion des Stammbaum des Lebens („tree of life“). Über den „tree of life“ („TOL“) informieren z.B. http://tolweb.org/tree und die „angiosperm phylogeny website“ http://www.mobot.org/mobot/research/apweb. Die ersten beiden Punkte kann man im engeren Sinne als Taxonomie bezeichnen. Sehr häufig werden die Begriffe „Systematik“ und „Taxonomie“ aber auch synonym verwendet. Eine völlig strikte Trennung der genannten vier Aufgabenbereiche ist ohnehin nicht wirklich möglich bzw. sinnvoll. Im Zusammenhang mit dem Stammbaum des Lebens hier noch das erwähnte, schöne Zitat aus dem Artenbuch: “… the great Tree of Life, which fills with its dead and broken branches the crust of the earth, and covers the surface with its ever branching and beautiful ramifications.”. ♦ Hierarchisch-enkaptische Klassifikation. Die binomiale Nomenklatur zur Benennung von Arten geht auf den „Vater der Taxonomie“, den schwedischen Botaniker und Zoologen Carl Nilsson Linnæus (1707–1778, seit 1756 Carl von Linné) und seine Werke „Species Plantarum“ (1753) und „Systema Naturæ“ (10. Auflage, 1758) zurück: Ein (latinisierter) Artname besteht aus zwei Teilen, dem (großgeschriebenen) Gattungsnamen und dem (kleingeschriebenen) Artepithet, z.B. Homo sapiens. (Es geht hier um wissenschaftliche, latinisierte Namen und nicht – wie man fälschlicherweise oft liest bzw. hört, um lateinische Namen.) In der Regel werden Artnamen in wissenschaftlichen Publikationen dabei kursiv gesetzt (oder wurden früher unterstrichen). Diese Binomen ersetzten vorher verwendete längere beschreibende Phrasen. Arten werden in Gattungen, Gattungen in Familien, diese in Ordnungen und diese in Abteilungen (hierarchisch-enkaptisch) zusammengefasst, wobei es jeweils auch Unterkategorien geben kann, z.B. Unterfamilien („Campbell“: Abb. 26.3). In der Botanik und Zoologie kennzeichnen jeweils bestimmte Endungen die jeweiligen taxonomischen Kategorien (Botanik: Familie = -aceae, Ordnung: -ales, Abteilung = -phyta). ♦ Klassifikation & Phylogenie. Gleichsam eine Ikone der Evolutionstheorie stellt ein 1837 erster, von Darwin in einem Notizbuch gezeichneter − bzw. eher gekritzelter − Stammbaum dar, der mit „I think“ überschrieben war. Ein Teil seiner Evolutionstheorie war eben auch die gemeinsame Abstammung und Phylogenie (Stammesgeschichte) aller Arten, die man stammbaummäßig darstellen kann. (Die einzige Abbildung im Artenbuch ist übrigens ein Stammbaum.) In einem Brief von 1857 (an Thomas Henry Huxley) zeigt Darwin sich überzeugt, dass „ The time will come I believe, though I shall not live to see it, when we shall have very fairly true genealogical trees of each great kingdom of nature.“ und im Artenbuch (1859) “fordert” er schließlich: "all true classification is genealogical". Ein solches phylogenetisches Klassifizerungssystem („Campbell“: Abb. 26.3), das die wahre Verwandtschaft widerspiegelt, wird (heute) auch als ein „natürliches System“ bezeichnet. Künstliche Systeme demgegenüber ordnen nach anderen Kriterien als „wahrer“ Verwandtschaft. ♦ Phylogenetische Stammbäume. Es gibt verschiedene bildliche Darstellungsweisen von Phylogenien: Ein Kladogramm („Campbell“: Abb. 26.4) zeigt „lediglich“ die phylogenetischen Verzweigungsmuster, also die (vermuteten bzw. hypothetischen) Verwandtschaftsbeziehungen zwischen den Taxa an. Von einem Verzweigungspunkt ausgehende Gruppen („Äste“) werden auch als Kladen bezeichnet. Bei einem Phylogramm korrelieren die Astlängen mit der Unterschiedlichkeit in den untersuchten Merkmalen, bzw. in einem Chronogramm mit der evolutionären Zeit. Jeder Knoten in einem Stammbaum repräsentiert einen ausgestorbenen, gemeinsamen Vorfahren. Ein Stammbaum kann nur aus dichotomen Verzweigungen bestehen oder aber auch Polytomien aufweisen, die bedeuten, dass hier die Verwandtschaftsverhältnisse (noch) nicht klar sind („Campbell“: Abb. 26.5). Man sagt dann auch, dass der Stammbaum hier nicht aufgelöst ist. Zumeist sind Stammbäume bewurzelt dargestellt. Ein unbewurzelter Stammbaum („unrooted tree“) ist ein so genanntes Netzwerk. ♦ „Tree Thinking“. Ein Bezug auf phylogenetische Stammbäume ist mittlerweile in sehr vielen Bereichen der Biologie − auch außerhalb der Systematik − vorhanden. Es ist daher von Vorteil, Stammbäume richtig lesen zu können. Wichtig ist bei solchen Stammbäumen, dass es wirklich nur auf das Verzweigungsmuster ankommt. Man kann die Zweige an den Knoten drehen, ohne dass sich die Aussage ändert: Die Schwestertaxa (Adelphotaxa) Taxon B und C („Campbell“: Abb. 26.5) können z.B. ihre Positionen in der Darstellung tauschen, ohne dass sich eine andere Aussage ergibt. Populäre falsche (!!!) Auffassungen in Bezug auf Stammbäume sind: 1.) Dass ein rezentes Taxon aus einem anderen rezenten Taxon entstanden ist: Taxon B und C in Abb. 26.5 sind nicht (!) aus Taxon A hervorgegangen, sondern alle drei Taxa hatten einen gemeinsamen, inzwischen ausgestorbenen Vorfahren. 2.) „Taxa basaler Linien sind uralt bzw. unverändert ursprünglich“: Im Zusammenhang mit 2 Skript zur Vorlesung „Allgemeine Evolutionsbiologie“ | WS 2015/2016 | A. Franzke & M. Koch Verzweigungen nahe der Wurzel eines Stammbaums spricht man oft von „basalen Linien“. Die untersuchten Arten dieser Linie sind deshalb aber (fast) nie genauso alt wie die Linie an sich. Es handelt sich lediglich um rezente Vertreter einer älteren Linie, die möglicherweise aber selbst auch vergleichsweise junge Arten sein können. Die Taxa basaler Linien sind auch nicht (!) „komplett unverändert ursprünglich“, wie der Begriff „basal“ evtl. suggeriert. Jedes rezente Taxon hat insgesamt eine gleich lange Evolutionszeit hinter sich und hat daher auch immer abgeleitete Merkmale. Von einer einzigen Linie zu sprechen, die basal zu anderen steht (z.B. die Aussage „Taxon A steht basal zu einer Klade aus B und D“) − ist eigentlich auch nicht wirklich sinnvoll, obwohl solche Formulierungen oft verwendet werden. Besser ist hier eigentlich (neutral) von einem Schwesterverhältnis zu sprechen. Ein kürzlich erschienenes Buch in dem es um Stammbäume und deren Rekonstruktion geht (s.u.) scheint – wenn man nach seinen Rezensionen urteilt − wohl sehr empfehlenswert zu sein: Baum & Smith 2012. Tree Thinking: An Introduction to Phylogenetic Biology. ♦ Exkurs: “progenitor–derivative species pairs”. Hinter der Aussage, dass keine rezente Art von einer anderen rezenten Art abstammt (s.o.) steht letztlich die Annahme, dass man es immer mit allopatrischen Speziationen durch Vikarianz zu tun hat: Eine Population wird geographisch getrennt und beide Teile evolvieren (langsam) simultan getrennt voneinander. Bei einer peripatrischen Speziation (s.o.), wo eine isolierte periphere Population in vergleichsweiser kurzer Zeit stark evolviert und die Ursprungspopulation dagegen fast unverändert bleibt, spricht man in der Botanik − etwa 30 Fälle sind derzeit genauer untersucht − dann aber doch von einem „progenitor–derivative species pair“. Mehr oder weniger impliziert solch ein Modell, dass es sich um vergleichsweise kürzlich abgeschlossene Speziationen handelt; insgesamt sicher eher „Sonderfälle“. ♦ Stammbaumrekonstruktionen. Stammbäume werden im wahrsten Sinne des Wortes re(!)konstruiert, zumeist „nur“ auf der Basis von Merkmalsausprägungen von rezenten Arten. Wichtig ist, sich klarzumachen, dass man dabei letztlich immer „nur“ Merkmalsphylogenien betrachtet, von denen man dann auf die Phylogenie der Arten schließt. Der im Englischen in diesem Zusammenhang verwendete Begriff „marker“ für zur Rekonstruktion verwendete Merkmale macht deutlich, dass hier etwas „nur“ als Markierung verwendet wird. Ein – wie auch immer rekonstruierter Stammbaum − ist daher immer nur eine Hypothese (!) zur Phylogenie der untersuchten Gruppe. In der „Praxis“ verwendet man dann aber dennoch oft sprachlich vereinfachte, bequeme „Sprachfiguren“ wie: „Das hier ist der Stammbaum der Rosengewächse!“. Die vornehmste Aufgabe des Systematikers, der einen Stammbaum rekonstruieren möchte, besteht darin, diejenigen Merkmale zu finden und zu verwenden, die geeignet sind, die wahre Phylogenie der Arten widerzuspiegeln. Die Abb. 26.2 im „alten Campbell“ verdeutlicht, dass bei einer Verwendung des Merkmals „allgemeine Gestalt“ ein Stammbaum resultieren würde, bei dem Pflanzen und Pilze als näher verwandt erscheinen, wohingegen die DNASequenzdaten für eine nähere Verwandtschaft von Pilz und Mensch sprechen. Die Abb. 26.2 im „neuen Campbell“ veranschaulicht ein anderes Beispiel. ♦ Homologie & Analogie. Für phylogenetische Stammbaumrekonstruktionen muss man also Merkmale (vergleichend) verwenden, die in der Lage sind die Phylogenie widerzuspiegeln (s.o.). In diesem Zusammenhang spielen die Begriffe Homologie und Analogie eine entscheidende Rolle, die zunächst (1848) − also vor dem Artenbuch − vom britischen Zoologen Richard Owen (1804−1892) nicht in einem phylogenetischen, sondern rein morphologischen Zusammenhang definiert wurden und auch nicht zwingend immer einen Gegensatz bilden: Homologie als "the same organ in different animals under every variety of form and function." Und Analogie als "a part or organ in one animal that has the same function as another part or organ in a different animal". Heute sind beide Begriffe deutlich enger gefasst und werden nun in einem phylogenetischen Kontext verwendet. Homologien sind Merkmale, die mit oder ohne Veränderung direkt von einem gemeinsamen Vorfahren abgeleitet sind. Solche homologen Merkmale bzw. deren Merkmalszustände sind daher (potentiell) für phylogenetische Stammbaumrekonstruktionen geeignet. Die Vorderextremitäten bei verschiedenen Landwirbeltieren haben trotz unterschiedlicher Ausprägungen (z.B. Vogelflügel versus Pferdebein) einen gemeinsamen „geerbten Bauplan“ und sind homologisierbar. Analogien hingegen sind strukturell ähnliche Merkmale bzw. Ähnlichkeiten, die nicht (!) aus der Übernahme von einem gemeinsamen Vorfahren stammen, sondern auf Anpassungen an gleiche Funktion zurückgehen, z.B. das Merkmal „Flügel“ bei Vögeln und Fledermäusen. Die Abb. 26.7 im „Campbell“ zeigt analoge Merkmale bei im Boden lebenden Tieren: Nordamerikanischer Maulfwurf (Plazentatier) versus Australischer Beutelmull (Beuteltier). Analogie weist also immer eine Beziehung zur Funktion bzw. Lebensweise, steht immer im Gegensatz zur Homologie und entsteht durch konvergente bzw. parallele Evolution (s.u.). ♦ Konvergenz versus Parallelismus. Konvergente bzw. parallele Evolution führt zu analogen Merkmalen. Oft wird zwischen diesen beiden Begriffen nicht unterschieden und dann nur der Begriff 3 Skript zur Vorlesung „Allgemeine Evolutionsbiologie“ | WS 2015/2016 | A. Franzke & M. Koch Konvergenz verwendet. Von Konvergenz (im engeren Sinne) spricht man, wenn die Analogie Organe betrifft, die nicht homolog sind, z.B. Spross- versus Blattdornen bei Pflanzen, Insekten- versus Vogelflügel. Parallelismus bedeutet eine Analogie, die homologe Strukturen bzw. Organe betrifft: Kakteenwuchs mit verdicktem Spross und zu Dornen abgewandelten Blättern bei (den meisten) Kakteen (Cactaceae) und z.B. auch bei einigen Wolfsmilcharten (Euphorbia, Euphorbiaceae). (Konvergente Zustände evolvieren also gleichsam unabhängig von „verschiedenen Startpunkten“ aus; parallele Zustände vom „gleichen Startpunkt“.) Beide Formen von analogen Merkmalszuständen werden in der Kladistik (s.u.) als Homoplasien bezeichnet, Merkmale, die nicht die (wahre) Phylogenie widerspiegeln (können), da sie nicht gemeinsam von einem Vorläufer geerbt wurden. (Achtung, der Begriff „Homoplasie“ ist im „alten Campbell“ falsch erklärt! Die Erklärung im „neuen Campbell“ ist in Ordnung.) ♦ Homologiekriterien nach Remane. Homologe von analogen Merkmalen zu unterscheiden ist naturgemäß nicht einfach, ein Walflosse sieht zunächst einmal „verdammt nach einer Fischflosse aus“. Der deutsche Zoologe Adolf Remane (1898−1976) hat 1952 so genannte Homologiekriterien formuliert, die es erlauben sollten a priori homologe anatomische Strukturen zu erkennen. 1) Kriterium der Lage: Der Vergleich von Lageähnlichkeit in einem Gefügesystem kann homologe Strukturen detektieren, z.B. Blattdornen sind von ihrer Lage homolog zu „normalen“ Blättern. 2) Kriterium der spezifischen Qualität und Struktur: Strukturen sind homolog, obwohl sie das Kriterium der Lage nicht erfüllen, wenn sie spezifische Sondermerkmale aufweisen, z.B. Zahn der Vertebraten und HaiHautschuppe (beide außen mit Zahnschmelz und innen mit Dentin). 3) Kriterium der Verknüpfung durch Zwischenformen (Stetigkeitskriterium): Extreme Ausbildungsformen von Organen können durch ontogenetische Zwischenformen oder Formenreihen verbunden werden. Die Schwimmblase von Knochenfischen und die Lungen der Landwirbeltiere gelten auf der Basis ihrer ähnlichen embryonalen Entwicklungsprozesse als homolog. Grundsätzlich sind Aussagen zu Homologien von Merkmalen natürlich immer „nur“ als Hypothesen zu verstehen (vergleiche Stammbaumrekonstruktion als Hypothese, s.o.). ♦ Ortho- & paraloge Gene. Bei DNA-Sequenzen (Genen) gibt es zwei Typen der Homologie: Orthologe Gene entstehen durch Kladogenese, wohingegen paraloge Gene durch Duplikation und Divergenz entstehen. Orthologe Gene sind also für phylogenetische Analysen von DNA-Sequenzen (Molekulare Systematik) geeignet. Eine Stammbaumanalyse, in der orthologe und paraloge Sequenzen gemischt vorkommen, wird die Kladogenese also nicht korrekt widerspiegeln. Das wäre so – ein sehr schräger Vergleich – als ob man für eine Vogelphylogenie z.T. Flügel- mit Beinmerkmalen vergleicht. Durch Stammbaumanalysen lassen sich Genduplikationen nachweisen: Wenn alle Kopien enthalten sind, ergibt sich ein spiegelbildlicher Stammbaum, in dem sich beide „orthologen Gruppen“ gegenüberstehen. In der Molekularbiologie bzw. Bioinformatik wird der Begriff Homologie häufig − „weniger streng“ – einfach nur für ähnliche DNA-Sequenzen verwendet. ♦ 4