Genomreplikation von DNA
Werbung

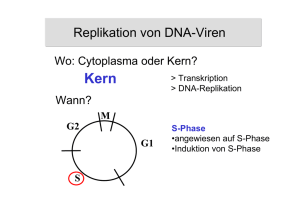
Sommersemester 2006 Molekularbiologie IV: Strategien der Virusreplikation Genomreplikation von DNA-Viren 20. Juni 2006 Elisabeth Schwarz DKFZ Genexpression und DNA-Replikation Early-late switch früh (early, E) spät (late, L) Replikation von DNA-Viren Wo: Cytoplasma oder Kern? Kern > Transkription > DNA-Replikation Wann? M G2 G1 S S-Phase •angewiesen auf S-Phase •Induktion von S-Phase DNA-Replikation DNA-abhängige DNA-Polymerasen semikonservative Replikation + Initiation: origin of replication = ori (Replikationsursprung) Elongation: bidirektionale Replikation Replikationsgabeln bidirektionale Wanderungsrichtung der Replikationsgabel Replikationsblasen DNA-Replikation • keine de-novo-Synthese! RNA-Primer • DNA-Synthese durch Anheftung von Nucleotiden an das 3'-OH-Ende der RNA-Primer • Template (Matrize) und Primer (Starter) • Direktionalität: Kettenverlängerung in 5'-> 3'-Richtung © Molecular Cell Biology, 2004 • dsDNA: Stränge komplementär und antiparallel Polymerisationsrichtung = Wanderungsrichtung der Replikationsgabel >kontinuierliche Synthese (leading strand, Leitstrang, Vorwärtsstrang) Polymerisationsrichtung gegenläufig zur Wanderungsrichtung >diskontinuierliche Synthese (lagging strand, Folgestrang, Rückwärtsstrang) Initiation der DNA-Replikation SV40 Zelle 1. T-Ag ORC 2. T-Ag Cdc6/ Cdt1/ ORC 3. T-Ag MCM 4. T-Ag Cdc6/ Cdt1/ ORC DNA-Replikation SV40 Chromosom • Ziel: viele neue Virusgenome • • • • • T-Antigen Ziel: Verdopplung! origin „licensing“ + origin-Aktivierung ORC (origin recognition complex, 6 Proteine) • CDC6, CDT1 • MCM2-7 • CDC7, S-CDKs, CDC45 1 1 DNAReplikation von SV40 2 2 3 •ori •bidirektionale Replikation 3 4 4 Fareed et al., 1972 © Molecular Cell Biology, 1999 DNA-Replikation von SV40 • Bindung von T-Antigen (doppeltes Hexamer) an ORI • lokales Entwinden der Doppelhelix (Helicase-Aktivität von T-Antigen) • Öffnen des Doppelstrangs (benötigt ATP + RFA als ssDBP) • Bindung von Primase-Polymerasealpha • Synthese der Primer durch Primase • Bindung von RFC stimuliert die Leitstrang-Synthese © Molecular Cell Biology, 1999 DNA-Replikation von SV40 © Molecular Cell Biology, 1999 • Bindung von PCNA > Inhibition und Verdrängung von Polα > Unterbrechung der LeitstrangSynthese • Bindung von Polδ an PCNA/RFC > erhöhte Prozessivität > Synthese des Leitstrangs • Bindung von Polα-Primase (+ RFC) an Folgestrang-Template • Synthese des Folgestrangs > Okazaki-Fragmente (100-200 Nucl.) • Entfernen der RNA-Primer durch 5’>3’-Exonuclease-Aktivität von Polα • Auffüllen und Ligation der Fragmente • Wanderung der Replikationsgabel: durch T-Antigen erleichtert • Trennung der beiden Tochtermoleküle: Topoisomerase II © Molecular Cell Biology, 2004 Struktur des SV40 ori minimaler ori: ca. 65 bp, 3 essentielle Elemente – T-Ag-Bindungsstelle II • inverted repeat, 23-34 bp, GC-reich • T-Bindungsstellen G-A/G-G-G-C – AT-reiche Sequenz (15-20 bp) – early imperfect palindrome © Principles of Virology, 1999, 2004 SV40: T-Ag und origin 1. Hexamer-Bildung von T-Ag Bindung an LT-BS II 2. Strukturveränderung von early palindrome (EP) 3. Bindung von RFA (RpA) Entwinden der DNA-Stränge Stabilisierung durch RFA © Principles of Virology, 1999, 2004 SV40 DNA-Replikation • Präinitiationskomplex: ori + T-Ag + RFA • Initiationskomplex: (ori + T-Ag + RFA) + Primase-Polα © Principles of Virology, 1999, 2004 • Bildung des Initiationskomplexes bestimmt den engen Wirtsbereich von Polyomaviren SV40 DNA-Replikation Topoisomerasen Decatenation © Principles of Virology, 2004 Papillomaviruses Different modes of DNA replication 1. Establishment replication Limited amplification: 50-100 copies/cell 2. Maintenance replication (1+2: bidirectional replication) 3. Productive replication (rolling circle replication) © Principles of Virology, 2004 Rolling-circle replication © Principles of Virology, 1999 Initiation der DNA-Replikation bei Papillomviren 1. Kooperative Bindung von E1 und E2 an ori. 2. ATP-abhängige Bindung von weiteren E1-Molekülen und Verdrängung von E2 Das Enden-Replikationsproblem bei linearen DNA-Genomen © Principles of Virology, 1999 Problem: Replikation der Genomenden Problemlösungen • eukaryote Chromosomen ¾ Telomere / Telomerase • • • • ¾ ¾ ¾ ¾ Bakteriophage T7 Bakteriophage T4 Adenoviren Herpesviren Bakteriophage Lambda • Pockenviren • Parvoviren terminale Redundanz (tR) tR, zirkuläre Permutation Protein-Primer Zirkularisierung ¾ self priming, hairpin loops ¾ self priming Bakteriophage T7 Virion: polyedrischer Kopf (Durchmesser 30 nm) kurzer, nicht kontraktiler Schwanz Familie Podoviridae Genom: ds-DNA, linear, ca. 40 kb Sequenzwiederholungen an den Enden (160 bp): > terminale Redundanz © Bakterienviren, 1992 Bakteriophage T7: DNA-Replikation • origin bei ca. 15% am linken Genomende • bidirektionale Replikation (RNA-Primer) • unvollständige Replikation des Folgestrangs > einzelsträngige 3’-Enden • wegen terminaler Repeats: überstehende 3’-Enden komplementär • Zusammenlagerung zu Dimeren • Auffüllen der Lücken durch DNA-Ligase • sequenzspezifische versetzte Spaltung durch Endonuclease gp3 > überstehende 5’-Enden • Auffüllen der Einzelstrang-Enden > vollständige Genome mit terminalen Repeats • bei weiterer Replikation: Bildung von Konkatemeren (bis ca. 100 Kopien) durch Stranginvasion (strand invasion) Bakteriophage T7: DNA-Replikation ori 5‘-ATTCAT 3‘-TAAGTA ATTCAT-3‘ TAAGTA-5‘ bid. Replikation 5‘-ATTCAT 3‘-TAAGTA + 5‘3‘-TAAGTA ATTCAT-3‘ -5‘ a ATTCAT-3‘ TAAGTA-5‘ b Zusammenlagerung Ligation 5‘-ATTCAT 3‘-TAAGTA a ATTCAT TAAGTA b ATTCAT-3‘ TAAGTA-5‘ Bakteriophage T7: DNA-Replikation 5‘-ATTCAT 3‘-TAAGTA a b ATTCATTAAGTA- ATTCAT-3‘ TAAGTA-5‘ Spaltung durch gp3 5‘-ATTCAT 3‘-TAAGTA a 5‘-ATTCAT 5‘-ATTCAT 3‘-TAAGTA + TAAGTA-5‘ ATTCAT-3‘ TAAGTA-5‘ Auffüllen der Enden a 5‘-ATTCAT 3‘-TAAGTA b ATTCAT-3‘ TAAGTA-5‘ + b ATTCAT-3‘ TAAGTA-5‘ Bakteriophage T4 Virion: elongierter polyedrischer Kopf (111 nm x 78 nm) langer kontraktiler Schwanz Familie Myoviridae, geradzahlige T-Phagen Genom: ds-DNA, linear, ca. 166 kb Genom in Virionen ca. 171 kb, wegen: Sequenzwiederholungen an den Enden (ca 5 kb): terminale Redundanz zirkuläre Permutation © Viren, 1993 Bakteriophage T4: DNA-Replikation • mehrere origins • frühe Replikation: bidirektionale Replikation > unvollständige Enden • späte Replikation: Konkatemerbildung durch Rekombination (recombination-dependent DNA replication) Invasion der 3’-Enden in homologe ds-DNA (strand invasion) > komplexes Netzwerk von replizierender Virus-DNA • Verpackung des Genoms in Phagenkopf: “head full”-Mechanismus Verpackungslänge größer als Genomlänge > terminale Redundanz, zirkuläre Permutation Bakteriophage T4: DNA-Replikation Terminale Redundanz und zirkuläre Permutation A Z A Z A Z A Z A Z A Z A Z A ZA ABCD...........YZA BCDE..........YZAB CDEF.........YZABC ZA Z A Z A T4 + T7: Virale Replikationsfaktoren T4 T7 • DNA-Polymerase (gp43) • Helikase, Primase (gp41 + gp61) • Einzelstrang-bindendes Protein gp32 • ca. 30 virale Genprodukte insgesamt • DNA-Polymerase (gp5) • Helikase, Primase (gp4) • Endonuclease (gp3) • DNA-Ligase >> autonomer viraler Replikationsapparat mit allen notwendigen Komponenten Adenoviren • 1953 aus Rachenmandeln (adenoids) isoliert • Erkrankungen der Atemwege, des Gastrointestinalbereichs, der Augenbindehaut • >100 Adenovirus-Typen bei Wirbeltieren Viruspartikel: • Ikosaeder (Durchmesser 80-110 nm) • 252 Capsomere (240 Hexone, 12 Pentone) © Viren, 1993 Adenoviren als Modellsysteme: • effiziente Vermehrung in Zellkultur • Genexpression: Splicing • DNA-Replikation • Onkogenese, Zelltransformation • Vektoren Adenoviren: Genom • lineare doppelsträngige DNA, 36 – 38 kb • inverted terminal repeats (ITRs, 54-166 bp) © Molekulare Virologie, 2003 • terminales Protein TP kovalent an 5‘-Enden • Interaktion der TPs > quasizirkulärer Zustand der DNA • Strangtrennung: ITRs > Hybridisierung der komplementären Sequenzen > Pfannenstiel-Strukturen Adenoviren: Initiation TP 55 kD © Molekulare Virologie, 2003 TP 80 kD • Bindung von pTP + Pol an terminalen ORI • Bindung von NF-1 und Oct-1 an ORI • kovalente Bindung von dCMP an Serin-OH: TP-C (Protein-Primer) • TP-C komplementär zu 3‘-terminalem G • Initiation der DNA-Synthese an TP-C • Bindung von ssDBP an verdrängten Einzelstrang Adenoviren: Elongation Phase I: • kontinuierliche Leitstrang -synthese • Strangverdrängung (strand displacement) Phase II: • verdrängter Strang: ITR >Pfannenstielstruktur • Gegenstrangsynthese Virale Replikationsproteine: • Pol, pTP, ssDBP © Molekulare Virologie, 2003 Herpesviren • große umhüllte Viren (Durchmesser 100-200 nm) • Envelope durch Knospung an innerer Kernmembran • lytische Infektion • Etablierung von latenten Infektionen (> Viruspersistenz) • Reaktivierung Genom: • lineare ds-DNA (130-240 kb) • im Virus-Core © Viren, 1993 spulenförmig um © Molekulare Virologie, 2003 Proteinmatrix gewickelt • Enden werden zusammengehalten • unique (ULund US) und repetitive Sequenzen Herpesviren beim Menschen Herpes-simplex-Virus: Genom ca. 150 kb HSV1: Herpes labialis (Lippenbläschen) HSV2: Herpes genitalis Varizellen-Zoster-Virus (VZV): Genom ca. 125 kb Varizellen = Windpocken: Primärinfektion Zoster = Gürtelrose: Rezidiv Cytomegalievirus (CMV): Genom ca. 230 kb inapparente Infektionen bei Immunsupprimierten schwere Verläufe Epstein-Barr-Virus (EBV): Genom ca. 172 kb infektiöse Mononucleose (Pfeiffer’sches Drüsenfieber) Burkitt-Lymphom (B-Zell-Lymphom), Afrika Nasopharynx-Karzinom, Südchina Herpesviren beim Menschen Humanes Herpesvirus 6 (HHV-6): 1986 entdeckt Exanthema subitum (Rubeola infantum, Dreitagefieber) Humanes Herpesvirus 7 1990 entdeckt, verwandt mit HHV-6 Humanes Herpesvirus 8: Genom ca. 270 kb 1994 entdeckt Kaposi-Sarkome bei AIDS-Patienten Herpesviren: DNA-Replikation • Zirkularisierung der DNA • rolling-circle-Mechanismus > head-to-tail-Konkatemere • Spaltung der Konkatemere und Verpackung: miteinander verbundene Prozesse (virales cleavage-packaging protein) •„head full“-Mechanismus Rolling-circle replication © Principles of Virology, 1999 Herpesviren HSV-kodierte Enzyme für DNA-Replikation Proteine für virale origin-abhängige DNA-Replikation DNA-Polymerase Helicase-Primase origin-bindendes Protein ssDNA-bindendes Protein Prozessivitätsfaktoren Enzyme für Nucleinsäuremetabolismus Bakteriophage Lambda • Genom: dsDNA, linear, 48502 bp • überstehende 5‘-Enden (m, m‘, cos) (12 Nucleotide, komplementär) • Infektion: Ringschluss • ori, O-Protein, P-Protein • 1. Bidirektionale Replikation • 2. „Rolling Circle“-Replikation • Concatemere > Encapsidierung > Spaltung durch Terminase (GenA+GenNu1) an cos(m/m‘) © Molekulare Genetik, 2001 Pockenviren große, komplex aufgebaute Viruspartikel (350 x 270 nm) im Lichtmikroskop sichtbar Virusreplikation: im Cytoplasma, in „factory areas“ (Virusfabriken) in kernlosen Zellen möglich Beginn der DNA-Replikation: ca 1-2 h p.i. Produktion: ca. 10.000 Genomkopien/Zelle ca. 50 % werden verpackt © Viren, 1993 © Molekulare Virologie, 2003 Pockenviren Variolavirus Erreger der Pocken (variola: scheckig) im 6. Jh. von Asien nach Europa eingeschleppt Höhepunkt der Pockenepidemie in Europa: 18. Jh. Variola major: schwarze Blattern (hämorrhagische Pocken), Letalität 5-40% Variola minor: weiße Blattern, Letalität ca. 1 % Ausrottung der Pocken 1977 durch weltweites Impfprogramm der WHO Pockenviren Virale Faktoren für DNA-Replikation DNA-Replikation: im Cytoplasma: keine Verwendung zellulärer Faktoren möglich alle Replikationsfaktoren vom Virusgenom codiert Enzyme für Nucleotidmetabolismus (um genügend große Mengen an dNTPs für DNA-Replikation herzustellen): Thymidinkinase Thymidylatkinase Ribonucleotidreductase dUTPase Pockenviren Genom: lineare ds-DNA 130-300 kb Kodierungskapazität: 150-200 Gene hairpin loops an den Enden + inverted terminal repeats (ITRs) ITR-Länge: 725 bp (Variola) bis 12 kb (Vaccinia) Vacciniavirus ITR © Molekulare Virologie, 2003 Pockenviren DNA-Replikation Priming: Setzen eines Nicks: self-priming (i.e. DNA-Primer) Rückfalten der ITRs kein definierter ori Elongation: Strand displacement (i.e. keine lagging strand-Synthese) Concatemer-Bildung Auflösung der Concatemere in unit-length-Moleküle (Monomere) erfordert Synthese von späten Proteinen Pockenviren nicks >self-priming nick inverted repeats >hairpin loops concatemers ©Fields Virology, 2001 Parvoviren kleine nicht umhüllte DNA-Viren (Durchmesser 18-26 nm) (parvus = klein) Genom: lineare ss-DNA, ca. 5000 Basen Parvovirinae (Vertebraten), Densovirinae (Insekten) felines Panleukopenievirus (Katzenseuche) Minute virus of mice (MVM) Aleutian mink disease virus (Aleuten-Nerzvirus) B19: Erythema infectiosum (Ringelröteln) Dependovirus (helferabhängige Viren): AAV 1-5 Helferfunktionen für AAV Adenoviren: frühe Genprodukte > Induktion von S-Phase keine direkte Interaktion von Ad-Proteinen mit AAV-Genom Herpesviren: HSV-Replikationsproteine Parvoviren DNA-Replikation Genom: • lineare einzelsträngige DNA, ca. 5000 Basen • Palindromsequenzen am 5'- und 3'-Ende: > hairpin-Strukturen • 3'-Palindrom: ca 100 Basen, 5'-Palindrom: 200-250 Basen DNA-Replikation: • im Kern, notwendig: S-Phase • Parvoviren können S-Phase nicht induzieren >Parvoviren sind die am stärksten von zellulären Funktionen abhängigen Viren • Primer: terminale 3'-Sequenzen der Genom-DNA • single strand displacement (model) • unidirektionale Leitstrangsynthese • „rolling hairpin“-Synthese Virales Protein für Replikation: NS-1 nukleäres Phosphoprotein, sequenz-spezifische DNA-Bindung, Helicase, an 5'-Ende von replikativer DNA (+ Virion-DNA)kovalent gebunden Parvoviren DNA-Replikation AAV-2 3‘-terminaler hairpin > primer-template ¾Initiation Nick (Rep) > Elongation replication intermediate genomic DNA self-priming, Rückfaltung der Termini strand displacement, „rolling hairpin“ © Principles of Virology, 1999 Genomreplikation von DNA-Viren Viren Genom Replikationsproteine Priming viral zellulär Elongation Polyoma T-Ag RNA 5 kb Primase bidirektionale Replikation Polymerase SSBP (RF-A) RF-C (Bel.faktor) PCNA (Proz.faktor) Topo II Papillom E1 wie Polyoma RNA 8 kb E2 bid. Replikation Genomreplikation von DNA-Viren Viren Genom Replikationsproteine Priming viral zellulär Elongation Adeno 35 kb pTP Initiation: Protein (pTP) Pol NFI strand displacement DBP NFIII/Oct1 Elongation: NFII (Topo) für Elongation >9000 bp Genomreplikation von DNA-Viren Viren Genom Replikationsproteine viral zellulär Herpes 150 kb DNA-Pol. UL30 ssDBP UL29 ori-bind. Protein UL9 Proz.faktor UL42 Primase/Helicase: UL5, 8, 52 Priming Elongation RNA rolling circle head-to-tailKonkatemere Spaltung in Monomere Genomreplikation von DNA-Viren Viren Genom Replikationsproteine viral zellulär Pocken DNA-Polymerase 130-300 kb ???? Resolvase Replikation im Cytoplasma! Parvo 5 kb NS1 DNA-Pol. Priming Elongation self-priming Rückfaltung der Termini strand displacement „rolling hairpin“ self-priming Rückf. der Termini strand displacement „rolling hairpin“