Genomreplikation von DNA
Werbung

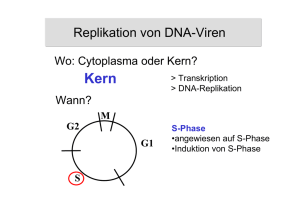
Sommersemester 2006 Molekularbiologie IV: Strategien der Virusreplikation Genexpression von DNA-Viren 13. Juni 2006 Elisabeth Schwarz DKFZ Erhalt eines Virus als infektiöse Einheit > Vermehrung (Replikation) > produktive Infektion dazu notwendig: Expression der viralen Gene Replikation des Virusgenoms Bildung neuer Viruspartikel Phasen einer produktiven Virusinfektion Attachment/Adsorption/Anheftung: zelluläre Rezeptoren virale Proteine (Envelope oder Capsid) Penetration: rezeptorvermittelte Endozytose Membranfusion Uncoating: Freisetzung der viralen Nucleinsäure Replikation: Expression der viralen Gene Replikation des Virusgenoms Synthese der Virionkomponenten Assembly/Morphogenese/Reifung Release/Freisetzung: Lyse Budding/Knospung Virusinfektion - verschiedene Möglichkeiten Wechselwirkungen zwischen Virus und Wirtszelle entscheiden über Verlauf einer Virusinfektion Grundvoraussetzung: Gegensatz: suszeptible Zellen (= infizierbare Zellen) nichtsuszeptible Zellen (nicht-infizierbar) permissive Zellen - produktive Infektion (>Lyse) nicht-permissive Zellen - abortive Infektion transient-permissive Z. - restriktive (restringierte) Infektion Persistente (persistierende) Infektion: Latente Infektion: Persistenz des viralen Genoms, aber keine Bildung infektiöser Partikel Chronische Infektion: Viruspersistenz mit Bildung infektiöser Partikel Zum Aufbau der Vorlesungsstunden · Genexpression und Genomreplikation sind voneinander abhängige und koordiniert ablaufende Prozesse · Trennung in zwei Vorlesungsteile: um jeweils spezifische Probleme und Problemlösungen deutlich zu machen (an Beispielen) · damit verbinden: Beschreibung der einzelnen Familien von DNA-Viren D N A V i r e n © Principles of Virology, 2004 Viren als obligate intrazelluläre Parasiten abhängig von Faktoren der Wirtszellen: Grundbausteine (Aminosäuren, Nucleotide) Translationsapparat Transkriptionsapparat Replikationsapparat nicht alle Faktoren sind zu jeder Zeit und an jeder Stelle verfügbar räumliche Aufteilung: WO? zeitliche Aufteilung: WANN? Genomreplikation und Genexpression Zeitlicher Verlauf: Alle DNA-Viren benötigen für Genomreplikation mindestens ein virales Protein ¾Genexpression vor DNA-Replikation Synthese von Capsidproteinen zur Verpackung der neu synthetisierten DNA ¾Genexpression während DNA-Replikation Proteinmengen: Regulatorproteine vs. Capsidproteine Stufen der viralen Genexpression Early-late switch früh (early, E) spät (late, L) Genexpression von DNA-Viren - Allgemeine Betrachtungen • Regulation von früher und später Phase erfolgt hauptsächlich auf Ebene der Transkription • Synthese der viralen mRNAs erfolgt durch zelluläre RNA-Polymerase II • Ausnahme: Pockenviren Voraussetzungen: ¾virale DNA trägt Signalelemente, die von zellulärer Transkriptionsmaschinerie und Regulationsfaktoren (Aktivatoren, Repressoren) erkannt werden ¾Studium von Viren führt zu Erkenntnissen über die Wirtszellen ¾Virusgenom im Zellkern ¾Vorhandensein der benötigten zellulären Transkriptionsfaktoren in den infizierten Zellen Eukaryote Transkription: Initiation Transkriptionsaktivatoren und Repressoren TFIID: TBP + TAFs (TBP-assoziierte Faktoren) Allgemeine Transkriptionsfaktoren (TFIIs) TFIIH: Helikase + Kinase (CTD Phosphorylierung) Genexpression von DNA-Viren: Virale Transkriptionsfaktoren • Transkription der viralen Gene erfolgt in festgelegter zeitlicher Reihenfolge • Regulation durch virale Transkriptionsfaktoren • verstärkte Synthese viraler mRNAs • early-late switch • Autoregulation (Aktivierung, Repression) • Kooperation mit zellulären Faktoren • DNA-bindend / nicht DNA-bindend Späte Genexpression - allgemeine Betrachtungen • Späte Proteine (Capsidproteine) werden in großen Mengen benötigt zur Bildung neuer Virionen • große Mengen fremder Proteine sind meist toxisch für Zellen • zu frühe Synthese würde Effizienz der Virusvermehrung vermindern • Problemlösung: Kopplung der späten Genexpression an die virale DNA-Replikation – Inhibition der viralen DNA-Replikation verhindert auch späte Genexpression • Replizierte virale DNA: – Matrize für weitere Replikation – Matrize für Transkription – Genom zum Verpacken • Mechanismen für DNA-replikationsgekoppelte Aktivierung der späten Transkription noch wenig bekannt Genexpression von DNA-Viren Beispiele in Vorlesung: Regulation der frühen viralen Genexpression – Polyomaviren: SV40 T-Antigen – Adenoviren: Adenovirus E1A Early-late-switch und späte virale Genexpression – SV40 – Adenovirus – Papillomviren Stufenweise Genexpression bei SV40, Adenovirus und Herpesvirus SV40 early > late Adeno immediate early > early > late HSV Virion protein > immediate early > early > late © Principles of Virology, 1999, 2004 Polyomaviren • 12 Viren bekannt, enger Wirtsbereich • produktive Infektion nur in Zellen des natürlichen Wirts • Transformation nicht-permissiver Zellen • Genom: zirkuläre dsDNA, ca. 5000 bp • Virionen: Ikosaeder, Durchmesser ca. 40 nm Polyomavirus • zuerst entdecktes Virus der Polyomaviren (1953, Ludwik Gross) • Arbeiten mit Mäuse-Leukämievirus: einige Mäuse entwickelten Tumoren der Speicheldrüsen (und keine Leukämien) • Zwei-Viren-Hypothese, Isolierung des zweiten Virus • Virus ruft viele andere Tumoren hervor: Name Polyomavirus (1958) • Tumorbildung nur bei Inokulation in neugeborene Labormäuse, bei erwachsenen Mäusen keine Poloymavirus-Erkrankungen • Infektion von embryonalen Mauszellen (permissiv): lytische Infektion > Analyse des produktiven Infektionszyklus • Infektion von Maus- und Hamsterzellen (semipermissiv): Zelltransformation > Analyse der Zelltransformation Polyomaviren: SV40 SV40 = Simian virus 40 • Entdeckung von SV40: Nebenprodukt bei Arbeiten mit Poliovirus (1960; Sweet and Hilleman) • Polio-Impfstoff (Salk-Vakzine) aus Rhesus-Affennierenzellen (Verwendung von menschlichen Zellen war verboten wg. möglicher Gefahr eines nicht bekannten Tumorvirus) • Inaktivierung der Polioviren mit Formalin • Test des Impfstoffs: Zellkulturen: kein cytopathischer Effekt Injektion in neugeborene Hamster: Tumoren (Latenzzeit 4-10 Monaten) • neues Virus: Infektion führt in Affennierenzellen zu Bläschenbildung > Simian vacuolating virus 40 = SV40 • Transformation von Zellen • keine Erkrankungen und Tumoren im natürlichen Wirt (Affe) • keine erhöhte Tumorrate bei Polio-Geimpften Beiträge von Polyomaviren zur Molekularbiologie • Struktur von superhelikaler DNA • SV40 DNA als Minichromosom: DNA + Histone, Nucleosomenstruktur • eukaryote DNA-Replikation • eukaryote Replikationsursprünge • Mechanismen der negativen und positiven Regulation der Genexpression • Enhancer und Promotor • alternatives Spleißen • Mechanismen der Onkogenese • Onkogene und Tumorsuppressorgene • Regulation des Zellzyklus SV40 in permissiven (A) und nichtpermissiven (B) Zellen A A B © Molecular Cell Biology, 1995 B ori/Transkriptionskontrollregion SV40 s-TAg frühe Region VP2 späte Region VP3 L-TAg frühe mRNAs VP1 späte mRNAs SV40 frühe Genexpression • Frühe Transkription zunächst ausschließlich durch zelluläre Faktoren • alternatives Spleißen der frühen mRNAs: • L-T (708 AS) und s-T (174 AS) • N-terminale 82 AS gemeinsam • s-T (small t-Ag, kleines t-Antigen): Wachstumsstimulation, Akkumulation von Virus-DNA im Kern, nicht notwendig für produktive Infektion in Zellkultur • L-T (large T-Antigen, großes T-Antigen): multifunktionales Protein, DNA-Replikation, Regulation der frühen und späten Transkription, early-late switch SV40 L-T intrazelluläre Lokalisation: im Kern (95 %); NLS (bei L-T erstmals identifiziert) an Plasmamembran (5 %) 5-10 x 105 Moleküle pro lytisch infizierter Zelle DNA-Bindung an GAGGC Bindungsstelle I: Autoregulation der frühen viralen Transkription Bindungsstelle II (ori): DNA-Replikation Komplexbildung mit zellulären Proteinen •DNA-Polα/Primase: DNA-Replikation •pRB, p53, Coaktivator p300, TBP, TEF-1, AP-2 Regulation des frühen Promotors durch SV40 L-T in früher Phase (niedrige L-T-Konzentration): Aktivierung unabhängig von DNA-Bindung von L-T unabhängig von Kernlokalisation von L-T in später Phase (höhere L-T-Konzentration): Repression •Bindung an Bindungsstelle I •stört Zusammenbau des Transkriptionsinitiations-Komplexes SV40 Enhancer TATA-Box (Chambon, 1980) 21-bp repeat: GC-reich, Sp1-Bindungsstellen (CCGCCC) 72-bp repeat: •Enhancer für frühen Promotor (Enhancer-Konzept, 1980) •Bindungsstellen für zelluläre Transkriptionsfaktoren: TEF, AP-1, NF-κB •ubiquitär aktiv (>Expressionsvektoren) Early-late switch und späte Genexpression bei Polyomaviren Änderung der frühen Genexpression: • Startstellen verschieben sich von early-early-Position (EE) zu late-early (LE) • längere 5‘-Enden • vor T-Antigen-ORF ein kleiner ORF für early-leader protein (ELP, 23 aa) • ELP-ORF vermindert Translationsstart an T-Ag-Startcodon Transkription der späten Gene: • Start am späten Promotor (keine TATA-Box): heterogene 5‘-Enden Aktivierung: • Amplifikation der Genom-DNA durch DNA-Replikation • T-Antigen: •unabhängig von DNA-Bindung von T-Ag •wahrscheinlich durch Modifikation zellulärer Faktoren •kein T-Ag in späten Transkriptionskomplexen vorhanden • „Verdünnen“ von zellulären Repressoren Regulation des späten SV40-Promotors durch zellulären Repressor (Ibp-Protein) Ibp bindet im Bereich des späten Promotors (Ibp: initiator-binding protein, Steroid-Thyroidhormon-Rezeptor Familie) Frühe Phase: [Ibp] > [Genom] wenige DNA-Moleküle Ibp blockiert späten Promotor © Principles of Virology Späte Phase: [Ibp] << [Genom] ansteigende Genom-Menge zu wenig Ibp Blockade entfällt DNA-Viren: Stufenweise Genexpression SV40 early > late Adeno immediate early > early > late HSV Virion protein > immediate early > early > late © Principles of Virology, 1999, 2004 Adenoviren: Genexpression • Genom ca. 35 kb • 5 frühe Transkriptionseinheiten mit jeweils mehreren alternativ gespleißten mRNAs: E1A, E1B, E2, E3, E4 • eine späte Transkriptionseinheit (MLTU: major late transcription unit) • Strang und Gegenstrang enthalten kodierende Informationen • Erhöhen der Kodierungskapazität durch differentielles Spleißen (Spleißen entdeckt bei Studium von Adenovirus-mRNAs) Adenoviren: gespleißte mRNA © Molecular Cell Biology 2004 Adenoviren frühe und späte mRNAs E1 E3 E2 E4 Adenoviren Ziele der frühen Genexpression • Herstellung einer optimalen Umgebung für Virusreplikation: Induktion der S-Phase: E1A x pRB Apoptoseblock: E1B-55K: Bindung an p53 E1B-19 kd (wirkt ähnlich wie Bcl-2, verhindert Freisetzung von Cytochrom C aus Mitochondrien) • Synthese viraler Genprodukte für virale DNA-Replikation (E2) • Synthese viraler Genprodukte, die antivirale Verteidigungsstrategien des Organismus blockieren (E3 und E4) Adeno E1A-Einheit • als erste exprimiert: immediate early • konstitutiv aktiver Promotor • mehr als 20 Bindungsstellen für Transkriptionsfaktoren • Transkription zunächst unter Kontrolle zellulärer Transkriptionsfaktoren • später: Autoaktivierung durch E1A (289 aa) • in späteren Phasen: Autorepression durch E1A (289 aa und 243 aa) E1A-mRNAs und -Proteine: • 13S-RNA (ca. 1000 b): 289 aa (CR1 + CR2 + CR3) • 12S-RNA (ca. 900 b): 243 aa (CR1 + CR2) zusätzlich: • 11S (217 aa), 10S (171 aa), 9S (55 aa) Adeno E1A Aktivierung der Transkription aller viraler Gene • E1B, E2, E3, E4: delayed early • ML (major late) Induktion von S-Phase: E1A x pRB Transkriptionsregulation durch E1A: • keine DNA-Bindung • Interaktion mit anderen Proteinen: • TBP: Aktivierung über TATA-Box • Coaktivator p300: Repression von Enhancern • YY1: Aufhebung der YY1-vermittelten Repression • pRB: Freisetzung von Transkriptionsfaktor E2F (Adeno E2-Promotor: Name für E2F) Adeno-negativ Kontrolle: Adeno-E1-transformiert ProteinInteraktionen von E1A • radioaktive Markierung der Proteine • Immunpräzipitation mit Antikörpern gg. SV40 T (PAb416) oder E1A (αE1A) • Gelelektrophorese (publ. 1986) Adenoviren: early-late switch Early-late switch • Reduktion der frühen Genexpression • Aktivierung der Transkription von: • IVa2, pIX • major late transcription unit (MLP) E1A IVa2 DNA-Replikation IVa2: • Aktivierung wahrscheinlich durch „Verdünnen“ eines zellulären Repressors • sequenzspezifischer Transkriptionsaktivator für späten Promotor pIX: • pIX-Gen überlagert von E1B-Transkriptionseinheit • pIX-Promotor blockiert, solange E1B transkribiert wird (promoter occlusion) • pIX-Promotor wird zugänglich, wenn [DNA] und E1B . • pIX-Protein: assoziiert mit Hexoncapsomeren. Adenoviren: späte Genexpression Major late transcription unit: • Transkriptionsstart an major late promoter (MLP) • Primärtranskript ca. 30.000 Basen (Genomlänge ca. 36 kb) RNA-Prozessierung: diff. Spleißen diff. poly(A)-Stellen dreiteilige nichtcodierende „leader“-Sequenz • 18 verschiedene mRNAs • 5 RNA-Familien L1-L5 • RNAs einer Familie terminieren an gleichem poly(A)-Signal Adenoviren Termination der späten Transkripte Frühe Phase: • unvollständige Elongation und frühzeitige Termination Späte Phase: • vollständige Elongation und Termination, abhängig von replizierter viraler DNA • selektiver Transport der späten viralen mRNAs ins Cytoplasma (E1B-55K + E4-34K) • selektive Translation der späten viralen mRNAs (Inaktivierung von eIF-4F, cap binding complex; noch vorhandenes aktives eIF-4F wird von dreiteiligem leader gebunden) Papillomaviruses • DNA viruses • icosahedral capsids (diameter 55 nm) • circular dsDNA (6800-8400 bp) • host species-specificity • replication in squamous epithelial cells Animal papillomaviruses: • bovine papillomavirus (BPV) • cottontail rabbit papillomavirus (CRPV) Human papillomaviruses (> 100 genotypes): • cutaneous HPV types: warts (papillomas) • mucosal HPV types: condylomata, carcinomas Human Papillomaviruses Cutaneous HPV types • HPV1, 2, u.a. • Infections of keratinized epithelia = skin • Skin warts (papillomas) = benign tumors A B C A: Verrucae vulgares (common warts) B: Verrucae plantares (plantar warts) C: Verrucae planae (flat warts) HPV-infizierte Plattenepithelien Verruca plana (links) Condyloma acuminatum (rechts) HPV-Virionen in infizierten Zellen N C Papillomviren und Krebs Zervixkarzinom (Gebärmutterhalskrebs, cervical carcinoma) Mehrstufenprozess der Krebsentstehung: • persistierende „high-risk“ HPV-Infektion •„high risk“-HPV-Typen: HPV16, 18, 31, 33, 35, 39, 45, 51, 52, 56, 58, 59, 66, 68, 73, u.a. [„low risk“-HPV-Typen: HPV6, HPV11 (Genitalwarzen)] • Nachweis von „high-risk“ HPV-DNA in fast 100 % aller Gebärmutterhalstumoren und in Tumorzell-Linien •„high risk“ HPV-Onkogene E6 und E7: – Expression in Krebszellen – Inaktivierung von Tumorsuppressorproteinen (pRB, p53) – Immortalisierung und Transformation von Zellen • Aktivierung von Onkogenen • Integration der HPV-DNA in das Zellgenom • Genominstabilität, Aneuploidie Zytologische Untersuchung von Zellabstrichen (Papanicolau) und Epithelveränderungen bei HPV-bedingter Karzinogenese CIN III normale Zellen Koilozyten Karzinom Papillomviren: Abhängigkeit der viralen Genexpression und DNA-Replikation vom Differenzierungszustand der Wirtszellen Fehrmann and Laimins, Oncogene, 22, 5201 (2003) Papillomaviruses early and late genes and gene functions E7 E6 E1 L2 E2 L1 L1 URR E6 o p E7 E1 pearly early transcripts pAe pAl L2 late transcripts E2 E6: ~150 aa, transactivator, oncoprotein, p53 degradation E7: ~100 aa, oncoprotein, inactivation of pRB and p21 CDKI E1: ~650 aa, replication factor (ori binding) E2: ~360 aa, viral activator/repressor, replication factor (ori binding) L1 : ~530 aa, major capsid protein L2 : ~470 aa, minor capsid protein Papillomviren (HPV31) alternatives Spleißen! Frühe Proteine E6: Hemmung der Apoptose, Onkoprotein, p53 E7: Induktion S-Phase, Onkoprotein, pRB E1: DNA-Replikation E2: Transkriptionsfaktor, DNA-Replikation E5: Wachstumsfaktor Späte Proteine L1: Capsidprotein L2: Capsidprotein HPV16-Genexpression in infizierten Gebärmutterhalsepithelien (LSIL: low grade squamous intraepithelial lesion) Middleton et al., J. Virol, 77, 10186 (2003) • Access to basal layer • Establishment as a low-copy-number episome • E7 expression (red circles) • uninfected/nonpermissive cells (blue circles) • E4 expression (green cells) • L1+L2 expression (orange nuclei) S-phase competent E4 (E2, E1, E5) E6, E7 (E1, E2, E4, E5) Productive HPV Infection Middleton et al. (2003) J. Virol., 77, 10186-10201, Figure 2 HPV-Genexpression bei produktiver HPV-Infektion (alle HPV-Typen) HPV-Genexpression in Krebsvorstufen („high-risk“ HPV-Typen)