Fachinformation 50 (02/14) | Molekulare Onkologie
Werbung

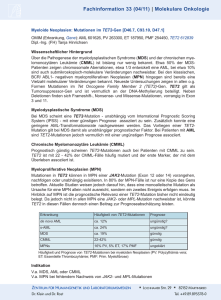
Fachinfo-Onkologie-20140211_zweispaltig_4b.qxd 11.02.14 16:12 Seite 335 Fachinformation 50 (02/14) | Molekulare Onkologie Update: Molekulare Marker für die Diagnostik von Myeloproliferativen Neoplasien (MPN) [D45, D47.1, D47.3, D47.4] OMIM-Nummer: 608232 (CML), 151410 (BCR-ABL1), 263300 (PV), 147796 (JAK2), 614521 (ET), 147796 (JAK2), 159530 (MPL), 109091 (CALR), 254450 (PMF), 147796 (JAK2), 159530 (MPL), 109091 (CALR) Dipl.-Ing. (FH) Tanja Hinrichsen Wissenschaftlicher Hintergrund Die häufigsten Erscheinungsformen von MPN sind - Chronische myeloische Leukämie (CML) - Polyzythämia vera (PV) - Essentielle Thrombozythämie (ET) und - Primäre Myelofibrose (PMF), deren Gemeinsamkeit eine gesteigerte klonale und autonome Proliferation hämatopoetischer Stammzellen, insbesondere im Knochenmark ist. Dabei werden funktionstüchtige, reife Blutzellen einer oder mehrerer myeloider Zellreihen gebildet. Während des Krankheitsverlaufs ist nicht selten ein phänotypischer Wandel der Erkrankung zu beobachten, was letztlich auch in einer akuten Leukämie enden kann. Für die sichere Abgrenzung der CML zu anderen MPN wird seit vielen Jahren der zytogenetische Nachweis der Philadelphia-Translokation t(9;22)(q34;q11.2) bzw. der molekulargenetische Nachweis des BCRABL1-Fusionstranskripts eingesetzt. Die BCR-ABL1negativen MPNs können wiederum durch folgende molekulare Marker von anderen Neoplasien abgegrenzt werden: Janus-Kinase 2 (JAK2)-V617F-Mutation, JAK2-Exon 12-Mutationen Laut Klassifikation der WHO (2008) für BCR-ABL1negative MPN gehört das Vorliegen einer JAK2-V617FMutation bzw. einer Mutation im JAK2-Exon 12 zu den Hauptkriterien für die Diagnose einer MPN. Bei der JAK2-V617F-Mutation kommt es zu einer Substitution von Valin durch Phenylalanin an Aminosäureposition 617 des JAK2-Polypeptids, was zu einer dysregulierten Kinaseaktiviät führt. Bei den JAK2-Exon 12-Mutationen handelt es sich um verschiedene somatische Missense-, Deletions- oder Insertionsmutationen (z.B. K539L, N542-E543del, F537-K539delinsL, H538Q, K539L, R541-E543delinsK, H538-K539delinsL, E543D544del), bei denen die Aminosäuren in Position 538543 involviert sind. Es wird vermutet, dass Mutationen im Exon 12 des JAK2-Gens zu einer veränderten JAK2-Signaltransduktion führen. Die JAK2-V617FMutation kann bei nahezu allen Patienten mit PV nachgewiesen werden und ist zudem bei ca. 50-60% der Patienten mit ET bzw. mit PMF detektierbar. Bei JAK2-V617F-negativen Patienten mit PV oder idiopathischer Erythrozytose ist meist eine JAK2-Exon 12Mutation nachzuweisen [2 - 6]. Mutationen im Myeloproliferative Leukemia Virus Onkogen (MPL) Die Mutationen W515L, W515K und W515A im MPLGen (codiert für den Thrombopoetin-Rezeptor) kommen etwa in 5-10% der JAK2-V617F-negativen Fälle mit ET bzw. PMF vor. Bisher bekannte Mutationen im MPL-Gen führen zu einem Aminosäureaustausch von Tryptophan nach Leucin (W515L), Lysin (W515K) oder Alanin (W515A) und somit zu einer konstitutiven Aktivierung des JAK-STAT-Signalweges. Eine weitere Mutation (MPL-S505N) ist im Zusammenhang mit familiärer ET beschrieben. Es können auch andere, seltenere MPL-Varianten (A506T, A519T, L510P) gefunden werden [7 - 9]. Mutationen im Calreticulin-Gen (CALR) CALR codiert für ein endoplasmatisches Retikulum Ca2+-bindendes Chaperon. Mutationen im CALR-Gen finden sich in ca. 70-80% der Patienten mit ET oder PMF, die keine Mutationen im JAK2- bzw. MPL-Gen aufweisen. Dabei handelt es sich um verschiedene somatische Deletions- bzw. Insertionsmutationen (häufigste: L367fs*46 und K385fs*47) in Exon 9 des CALR-Gens, die zu einem Frameshift und somit zu einem spezifischen, alternativen Leserahmen führen. Vermutlich kommt es so zu einer Aktivierung des JAKSTAT-Signalweges. CALR-Mutationen finden sich nicht bei Patienten mit PV [10,11]. ET-Patienten mit CALRMutationen sind im Vergleich zu JAK2-V617F- bzw. MPL-mutationspositiven ET-Patienten jünger, haben eine deutlich erhöhte Thrombozytenzahl, niedrigeres Hämoglobin, verminderte Leukozytenzahlen sowie ein geringes Thromboserisiko [12,13]. Expression des Polycythämia rubra vera-1 (PRV-1)Gens Eine PRV-1-Überexpression wird in ca. 70-80% der Patienten mit PV, in ca. 20-50% der Patienten mit ET und in ca. 50% der Patienten mit PMF beobachtet. Die PRV-1-Überexpression korreliert hierbei mit dem Auftreten von EPO-unabhängigen, erythroiden Kolonien und ist daher langfristig mit der Ausbildung einer PV assoziiert [14]. Hinweis Im Zusammenhang mit MPN sind ebenfalls Mutationen in den Genen TET2, EZH2, ASXL1 und CBL beschrieben, die in Zusammenhang mit Mutationen in JAK2, MPL und CALR auftreten können. Bei Erstdiagnose einer PV, ET oder PMF kann ein veränderter Karyotyp nachgewiesen werden. Spezifische zytogenetische Veränderungen sind in diesem Zusammenhang jedoch nicht beschrieben. Zu den häufigsten zytogenetischen Veränderungen zählen numerische Aberrationen der Chromsomen 1, 8 und 9, Deletionen der Chromsomen 5q, 13q und 20q sowie Zugewinne von Chromsom 9p15 Aus diesem MVZ Martinsried, Lochhamer Str. 29, 82152 Martinsried, www.medizinische-genetik.de, Tel. +49.89.895578-0 Fachinfo-Onkologie-20140211_zweispaltig_4b.qxd 11.02.14 16:12 Seite 336 50. Fachinformation - Molekulare Onkologie Grund erscheint eine zytogenetische bzw. molekularzytogenetische Analyse ebenfalls sinnvoll. Indikation Verdacht auf Myeloproliferative Neoplasie (PV, ET, PMF) Anforderung/Versand Ü-Schein Muster 10 mit folgenden Angaben Ausnahmekennziffer: 32012 Diagnose: MPN (ICD-10 Code: [D47.1]), Polyzythämia vera (ICD-10 Code: [D45]), Essentielle Thrombozythämie (ICD-10 Code: [D47.3]), Primäre Myelofibrose (ICD-10 Code: [D47.4]) Auftrag: JAK2-V617F-Mutation, JAK2-Exon12Mutationen, MPL-W515-Mutation, CALR-Exon9 – Mutationen, PRV1-Expression humangenetisches Gutachten Material 5-10 ml EDTA-Blut Methode Mutationanalysen: Aus dem eingesandten Probenmaterial werden mittels Magnet-Beads Granulozyten angereichert, DNA extrahiert und Exon 12 und 14 des JAK2-Gens, Exon 10 des MPL-Gens sowie Exon 9 des CALR-Gens mittels PCR amplifiziert und sequenziert. Der Sequenzabgleich und die Verifikation von Polymorphismen und Mutationen erfolgt mit Hilfe der EMBL- und NCBI-Datenbanken (GenBank, dbSNP). Expression des PRV-1-Gens: Aus dem eingesandten Probenmaterial werden mittels Magnet-Beads Granulozyten angereichert, Gesamt-RNA extrahiert und die RNA durch quantitative RT-PCR auf die Expression des PRV-1-Gens untersucht. Als Referenz für die relative Quantifizierung der PRV-1-Transkripte dient die Expression von Glycerinaldehyd-3-Phosphatdehydrogenase (GAPDH). Dauer der Untersuchung 5-7 Werktage Literatur [1] WHO Classification of Tumours of Haematopoietic and Lymphoid Tissues (2008) [2] Tefferi et al, Leukemia, 22:14 (2008) [3] Pietra et al, Blood 111:1686 (2008) [4] Li et al, Blood 111:3863 (2008) [5] Scott et al, NEJM 356:459 (2007) [6] Williams et al, Exp Hematol. 35:1641 (2007) [7] Koppikar et al, Acta Haematol, 119:218 (2008) [8] Kilpivaara et al, Leukemia, 22:P106 (2008) [9] Chaligne et al, Leukemia 22: 1557 (2008) [10] Klampfl et al, NEJM 369(25):2379 (2013) [11] Nangalia et al, NEJM 369(25):2391 (2013) [12] Rumi et al, Blood 121(21):4388 (2013) [13] Rotunno et al, Blood, Epub ahead of print (2013) [14] Sirhan et al, Haematologica, 90: 406 (2005) [15] Kralovics, Leukemia, 22:1841 (2008) BCR-ABL1-negave MPN ET PV PMF 50% 5% 60% JAK2-Exon 12-Mutaon 10% 35% 95% 5% JAK2-V617F 25% MPL-W515 CALR-Exon 9-Mutaon JAK-STAT-Pathway MVZ Martinsried, Lochhamer Str. 29, 82152 Martinsried, www.medizinische-genetik.de, Tel. +49.89.895578-0