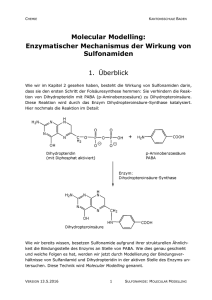
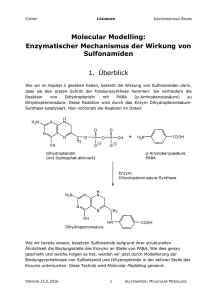
Molecular Modelling Einführung und Visualisierung
Werbung

MM 02.10.2012 Molecular Modelling Einführung und Visualisierung Michael Meyer Vorlesung I Michael Meyer Molecular Modelling Überblick • klassisches Molekular Modeling, Computational Chemistry Kraftfelder, Moleküldynamik, Quantenchemie, Konformationsanalyse • Chemoinformatik Molekulare Deskriptoren, Regression und Klassifikation, QSAR- und QSPR-Modelle • Biopolymere Sequenz und Struktur, Homologiemodeling, Docking • Wirkstoffdesign und -entwickung virtuelle Bibliotheken, ADMET-Modelle, Leitstrukturoptimierung • Randgebiete Bioinformatik, Biostatistik Michael Meyer Einführung Molecular Modelling 1 MM 02.10.2012 Molecular Modelling - warum? • Ressourcen und Zeitersparnis Substanzen müssen nicht gekauft, synthetisiert, gelagert oder entsorgt werden • Planung und Reduzierung der Anzahl von Experimenten Welche Substanz soll als nächste synthetisiert werden? Welche Substanz hat die größte Aussicht auf Erfolg? • Untersuchung von schwer zugänglichen Substanzen Daten von nicht synthetisierte Verbindungen • Mikroskopische Eigenschaften Wie läuft eine Reaktion ab? Wie „sieht“ ein Molekül genau aus? Michael Meyer Molecular Modelling Anwendungsgebiete • Life Science Wirkstoffdesign, ADMET Reaktionsmechanismus von Enzymen Simulationen • Werkstoffwissenschaften Materialeigenschaften • Mechanismen der (an)organischen Chemie • Spektroskopische Eigenschaften • Physikochemische Eigenschaften Michael Meyer Einführung Molecular Modelling 2 MM 02.10.2012 Drug Design Zyklus Synthesis Acquire Published Data Screening /Bioassays Library Design Knowledgebase Drug Candidate Profiles/ Priorization Virtual Screening Model Building Michael Meyer Virtual Library Molecular Modelling Visualisierung von Molekülen Van der Waals Radien von Atomen (Å) H C N O F P S Cl Michael Meyer Einführung 1.20 1.70 1.55 1.52 1.47 1.80 1.80 1.89 Solvent accessible surface Die van der Waals Oberfläche wird mit einer Kugel von der Größe eines Lösungsmittelmoleküls abgefahren (contact surface). Lücken in der Oberfläche werden mit Zwischenstücken (reentrant surface) geschlossen, die dem Radius des Lösungsmittelmoleküls entsprechen. Molecular Modelling 3 MM 02.10.2012 Visualisierung von Moleküloberflächen und elektrostatischen Eigenschaften Elektrostatisches Potenzial (ESP) φ (r ) = φ nucl (r ) + φ el (r ) N φ nucl (r ) = ∑ A=1 φ el (r ) = − ∫ Connolly Oberfläche mit elektrostatischem Potenzial Michael Meyer Φ(r) ρ ZA RA ZA r − RA ρ (r )` dr r´−r ESP am Ort r Ladungsdichte der Elektronen Kernladung des Atoms A Ort des Atomkerns A Molecular Modelling Visualisierung von Orbitalen H H N H H H Pyrrol HOMO LUMO Highest Occupied Molecular Orbital Lowest Unoccupied Molecular Orbital Michael Meyer Einführung Molecular Modelling 4 MM 02.10.2012 Visualisierung von Proteinstrukturen I Coronavirus Main Proteinase Sekundärstruktur Oberfläche K. Anand et al., Science 300, 1763 (2003). (PDB ID: 1p9u) Michael Meyer Molecular Modelling Visualisierung von Proteinstrukturen II Ferrichrome FhuA Rezeptor mit Ferrichrome K. P. Locher et al., Cell 95, 771 (1998). (PDB ID: 1by5) Michael Meyer Einführung contact preferences Molecular Modelling 5 MM 02.10.2012 Visualisierung von Nukleinsäuren Elektrostatisches Potential Na+-Ionen G-Tetraden und in einem Tetraplex Michael Meyer Literatur K. Phillips et al., J. Mol. Biol. 273, 171 (1997). Kristallstruktur PDB: http://www.rcsb.org (ID: 352d) NDB: http://ndbserver.rutgers.edu (ID udf062) Molecular Modelling Visualisierung von Daten Modell zur Löslichkeit organischer Substanzen in Wasser Observations: 1290 Descriptors : 6 RMSE: 0.74 R 2: 0.87 RMSEcv: 0.75 Q2: 0.86 Daten: T. J. Hou et al., J. Chem. Inf. Comput. Sci. 44, 266 (2004) Michael Meyer Einführung Molecular Modelling 6 MM 02.10.2012 4 Klassifizierung von Molekülen 15 3 2 3 2 Einteilung von Liganden des P-Glykoproteins (PGP) in unterschiedliche Gruppen 1 calc$x 1 6 0 9 16 17 5 4 11 7 10 12 13 -1 8 18 19 20 21 -2 14 5 10 15 20 Index Michael Meyer Einführung Molecular Modelling 7