Enzyme - Universität Salzburg
Werbung

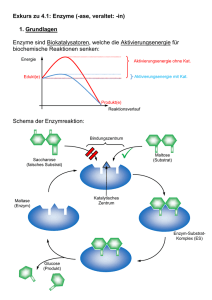
Enzyme Prof. Dr. Albert Duschl Katalyse Reaktionen laufen normalerweise nicht spontan ab, auch wenn insgesamt dabei Energie gewonnen werden sollte. Es muß zunächst eine Aktivierungsenergie aufgebracht werden. Katalysatoren verringern die benötigte Aktivierungsenergie, erhöhen also die Wahrscheinlichkeit daß die Reaktion abläuft. Aktivierungsenergie wird normalerweise durch energiereiche Zusammenstösse mit anderen Molekülen zugeführt. Im Verlauf der Reaktion entstehen Zwischenzustände, die energetisch ungünstiger sind als der Ausgangszustand. Aktivierungsenergie wird benötigt um diese energieungünstigen Zwischenzustände herzustellen. Aktivierungsenergie kann durch Erhitzen zugeführt werden (energiereiche Zusammenstösse!). Alternativen sind technische Katalysatoren oder, im biologischen Bereich, Enzyme. © Alberts et al.: Molecular Biology of the Cell Enzyme Fast alle Enzyme sind Proteine (Ausnahme: Einige katalytisch wirkende RNAs). Sie sind spezifisch für bestimmte Substrate und katalysieren definierte Reaktionen. Die Aktivität von Enzymen wird normalerweise reguliert. Wie alle Katalysatoren nehmen auch Enzyme an einer Reaktion teil, gehen am Ende aber wieder im Ausgangszustand daraus hervor. Ein Enzym wird also nicht verbraucht wie ein Substrat. Enzyme können verschiedene Formen von Energie nutzbar machen, beim Sehvorgang z.B. Licht. Meistens wird jedoch chemische Energie in Form energiereicher Bindungen verwendet. Das Enzym beschleunigt die Spaltung der Bindungen, deren Energie dadurch frei wird. Ein häufig dafür verwendetes Molekül ist Adenosintriphosphat (ATP). © Stryer: Biochemistry Reaktionsgleichgewicht Enzyme verschieben das Reaktionsgleichgewicht nicht. Sie senken die Aktivierungsenergie für Hin- und Rückreaktion, treiben also keineswegs die Reaktion in eine bestimmte Richtung. Sie verändern nur die Geschwindigkeit, mit der sich das Gleichgewicht einstellt. Die Schnelligkeit der Gleichgewichtseinstellung hängt von der Wechselzahl eines Enzyms ab. Die Wechselzahl gibt die Zahl der Reaktionen an, die pro Sekunde katalysiert werden. Sie kann sehr unterschiedlich sein - Lysozym hat 0.5, DNAPolymerase hat 15, Carboanhydrase hat 600.000, und Katalase hat 40.000.000. In biologischen Systemen werden in der Regel die entstehenden Produkte sofort wieder als Substrate weiterer Reaktionen genützt. Es stellt sich also nicht wie im Reagenzglas ein einfaches Gleichgewicht ein, sondern viele Substanzen sind über vernetzte Reaktionen miteinander verbunden. Sie stehen in einem Fließgleichgewicht. Für die Bestimmung der Eigenschaften einzelner Enzyme verwendet man am besten Reaktionen in vitro. in vitro "im Reagenzglas" – in vivo "im Leben" © Löffler/Petrides: Biochemie und Pathobiochemie Definitionen Coenzyme sind kleine organische Moleküle die für die Funktion der Enzyme notwendig sind. Cofaktoren ist eine allgemeine Bezeichnung für solche assistierende Elemente in Enzymen, die auch die häufig vorkommenden Metallionen umfasst. Metalle wie Fe3+ oder Zn2+ sind z.B. oft für Redoxreaktionen erforderlich. Prosthetische Gruppen sind Cofaktoren die dauerhaft an das Enzym gebunden sind und für die Funktion wichtig sind. Cosubstrat wäre dagegen angemessen wenn ein Coenzym nur vorübergehend an das Enzym bindet. Holoenzym ist ein katalytisch aktiver Komplex aus Proteinanteil und Cofaktor. Apoenzym ist nur der Proteinanteil dem der Cofaktor fehlt, und der deswegen auch nicht funktionsfähig ist. Aktives Zentrum der Carboanhydrase © Voet/Voet/Pratt: Biochemie [A] + [B] → [AB] Eine Reaktion [A] + [B] → [AB] hängt von den Konzentrationen von A und B ab: Je höher die Konzentrationen, umso häufiger finden sich die Partner. Die Wahrscheinlichkeit eines Zerfalls [AB] → [A] + [B] ist für ein einzelnes Molekül zwar von der Konzentration von AB unabhängig, aber da in einer Molekülpopulation Hin- und Rückreaktion im Gleichgewicht stehen, gehen die Konzentrationen doch wieder ein. Für die Analyse von Einzelmolekülen gelten andere Formeln. Die Geschwindigkeit v einer Reaktion hängt ab von der Konzentration der Moleküle (oder des Moleküls) und einer Proportionalitätskonstanten k, die von den Eigenschaften der spezifischen Reaktion bestimmt wird. Für die Hinreaktion gilt also v+1 = k+1 ∙ [A] ∙ [B] Und für die Rückreaktion v-1 = k-1 ∙ [AB] Stehen beide Reaktionen im Gleichgewicht, ist v der Hinreaktion (v+1) genau so gross wie v der Rückreaktion (v-1). Die beiden Konstanten (k+1, k-1) ändern sich dabei nicht. Es ist dann k+1 ∙ [A] ∙ [B] = k-1 ∙ [AB] Oder anders ausgedrückt K = k+1 ∕ k-1 = [AB] ∕ [A] ∙ [B] K bezeichnet man als Gleichgewichtskonstante der Reaktion. Enzymkatalysierte Reaktionen Denken wir an eine enzymkatalysierte Reaktion, dann muß im einfachsten Fall zunächst das Substrat S an das Enzym E binden. Im so gebildeten Enzym-SubstratKomplex laufen dann die Umformungen ab, aus denen am Ende das Produkt P und das (unveränderte!) Enzym E hervorgehen. Die Rückreaktion von E + P zurück in den Komplex ist so unwahrscheinlich, daß wir sie vernachlässigen können. In einer solchen Reaktion gilt die Michaelis-Menten-Gleichung. Sie beschreibt die Beziehung zwischen der Geschwindigkeit einer Reaktion und der Substratkonzentration. Michaelis-Menten-Gleichung Leonor Michaelis und Maud Menten postulierten diese berühmte Gleichung 1913. V ist die Reaktionsgeschwindigkeit. Vmax ist die Maximalgeschwindigkeit. Sie wird dann erreicht, wenn alle vorhandenen Enzymmoleküle in einem Enzym-Substrat-Komplex vorliegen. Km ist die Michaelis-Konstante, ein Maß für die Bindungsstärke (oder Affinität) zwischen Enzym und Substrat. Km ist abhängig on Ionenstärke, pH-Wert, Temperatur und anderen Umweltbedingungen. [S] ist die Substratkonzentration. Bei graphischer Auftragung von V gegen [S] ergibt sich eine Hyperbel, deren Anfangssteigung Vo ist (die Anfangsgeschwindigkeit der Reaktion). Die Michaeliskonstate Km ist die Substratkonzentration bei der halben Maximalgeschwindigkeit, ½ Vmax. © Nelson/Cox: Lehninger Principles of Biochemistry Lineweaver-Burk-Diagramm Man möchte zur Charakterisierung eines Enzyms gern Vmax und Km kennen. Bei der Hyperbel aus dem Michaelis-MentenDiagramm lässt sich jedoch Vmax kaum exakt ablesen; dadurch ist dann auch Km nicht genau zu bestimmen. Wenn man die selben Daten nach Lineweaver und Burk aufträgt, erhält man eine Gerade Der X-Achsenabschnitt ist in dieser Auftragung -1/Km, der Y-Achsenabschnitt ist 1/Vmax und die Steigung ist Km/Vmax. © Nelson/Cox: Lehninger Principles of Biochemistry Reaktionsablauf Während einer Reaktion ändern sich die Konzentrationen von Substrat, Produkt, freiem Enzym und Enzym-Substrat-Komplex. Die Abbildung rechts stellt eine in vitro Reaktion dar in der ein Substrat komplett umgesetzt wird. In vivo werden die Verhältnisse anders sein, z.B. kann Substrat nachgeliefert und Produkt verbraucht oder in ein anderes Kompartiment verschoben werden (Fließgleichgewicht!). Es können Inhibitoren oder Aktivatoren auftauchen, Parameter mit Einfluss auf Proteineigenschaften können sich ändern, ein Substrat kann gleichzeitig in mehrere unterschiedliche Reaktionen eintreten so dass verschiedene Enzyme kompetieren, und durch Enzymsynthese und -abbau kann sich sogar [E] ändern. © Wikipedia Enzymhemmung Enyzme können durch kompetitive Inhibitoren gehemmt werden. Diese binden ebenso wie das Substrat an das aktive Zentrum des Enzyms und haben eine ähnliche Struktur wie das echte Substrat. Malonat ist zum Beispiel ein kompetitiver Inhibitor der Succinatdehydrogenase. Kompetitive Hemmung lässt sich duch Zugabe von mehr Substrat aufheben. Nicht-kompetitive Hemmstoffe binden an anderen Stellen des Enzyms und ändern dadurch dessen Struktur und damit auch die Funktion. Zugabe von mehr Substrat hilft in diesem Fall nichts. Allosterische Enzyme, die bei Bindung von Hemmstoffen oder Substraten ihre Strukutur ändern, unterliegen nicht der Michaelis-MentenKinetik, sondern anderen, komplizierteren Beziehungen. © both figures Stryer: Biochemistry Beispiel: Chymotrypsin Chymotrypsin ist eine Protease. Proteasen spalten Peptidbindungen. Chymotrypsin ist ein Verdauungsenzym das im Pankreas produziert wird. Es besteht im aktiven Zustand aus zwei Ketten (Quartärstruktur!). Es gehört zur grossen Gruppe der Serinproteasen. Die Serinproteasen tragen diesen Namen, weil für ihren Reaktionsmechanismus ein Serinrest der Protease eine entscheidende Rolle spielt. © Anthony J. Frisby, Thomas Jefferson University Aktivierung von Chymotrypsin Chymotrypsin entsteht durch enzymatische Spaltung von Chymotrypsinogen, einem Proenzym (Zymogen). Die Synthese eines inaktiven Vorläufermoleküls schützt den Pankreas vor Selbstverdauung. Chymotrypsinogen wird von Trypsin gespalten, einer weiteren Serinprotease. Trypsin selber entsteht durch Spaltung des Proenzyms Trypsinogen. Trypsinogen wird durch Enteropeptidase gespalten, einer dritten Serinprotease, die so sequenzspezifisch spaltet, daß Trypsinogen praktisch ihr einziges Substrat ist. © Physiologische Chemie, Universität Würzburg Substratspezifität Proteasen spalten nicht beliebige Peptidbindungen. Bei Serinproteasen richtet sich die Spezifität nach der Aminosäure des Substrats, die N-terminal von der Spaltstelle liegt. Chymotrypsin: Aromatische oder grosse unpolare Seitenkette. Trypsin: Lysin oder Arginin. Elastase: Kleine, ungeladene Seitenkette. Diese Selektivität wird durch eine Tasche in der Proteasestruktur festgelegt, in der die Seitenkette der N-terminalen Aminosäure während der Spaltung fixiert wird. Die Struktur des aktiven Zentrums bestimmt damit, welche Substrate verwendet werden. © Stryer: Biochemistry Mechanismus der Serinproteasen Durch die Wasserstoffbrückenkette die von Asp 102 über His 57 nach Ser 195 verläuft, wird das Proton der OHGruppe von Ser 195 zum His 57 verschoben. Die resultierende -CH2OGruppe kann die Carbonylgruppe einer Peptidkette nukleophil angreifen. Es entsteht ein H2N-Produkt und ein AcylEnzym-Intermediat. Danach wird die Carboxylgruppe auf Wasser als Akzeptor übertragen. Dieser Reaktionsmechanismus findet sich in ähnlicher Form z.B. bei der nicht verwandten bakteriellen Protease Subtilisin, manchen Esterasen (etwa Acetylcholin-Esterase) und manchen Lipasen. © Stryer: Biochemistry Katalytische Triade Die drei Aminosäuren der katalytischen Triade liegen in der Struktur von Chymotrypsin sehr eng beisammen, obwohl sie in der Sequenz weit voneinander entfernt sind. Es gibt nur vier bekannte Mechanismen für Proteasen: Serinproteasen (Trypsin, Chymotrypsin, Elastase, Thrombin) Saure Proteasen (Pepsin, HIV-1 Protease) Zinkproteasen (Carboxypeptidase A und B) (Achtung: Zinkproteasen sind nicht das gleiche wie Zinkfingerproteine) Thiol-Proteasen (Papain, Cathepsin B) © both pictures Anthony J. Frisby, Thomas Jefferson University Enzyme ... Enzyme sind eine Grundlage des Lebens, weil ohne Enzyme biologische Reaktionen wegen der fehlenden hohen Aktivierungsenergie nicht ablaufen könnten und Energiezufuhr durch Erhitzen komplexe Strukturen zerstört, da die Wärmebewegung (Brown'sche Molekularbewegung) zu gross wird. Enzyme waren daher von Anfang an Teil des Lebens. Enzyme sind nützlich beim Kochen. © Waverley Root: Food. An authoritative and visual history and dictionary of the foods of the world