Plasmidpräparation - molekulargenetsiche Experimente IV
Werbung

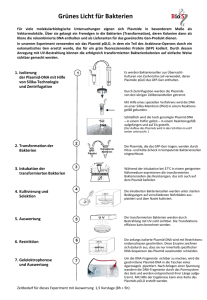
Molekulargenetische Experimente IV: Plasmidpräparation Plasmide sind kleine, ringförmige DNA-Moleküle in Bakterien, die in der Lage sind, sich selbst mit Hilfe von Enzymen zu replizieren. Gene, die auf Plasmiden lokalisiert sind, besitzen eine große Mobilität, das heißt, in der Natur tauschen Bakterien in parasexuellen Vorgängen Erbinformationen aus, die auf Plasmid-DNA sitzt. Diese Informationen stellen häufig Resistenzgene gegen Antibiotika dar, sind also für die Bakterien extrem wichtig im Überlebenskampf, allerdings sehr zum Nachteil bei der Bekämpfung von Infektionskrankheiten. Plasmide haben eine große Bedeutung in der Gentechnologie erlangt. Da sie sich so leicht isolieren und einschleusen lassen, kann man sie als Vektoren verwenden, um bestimmte Erbanlagen in andere Organismen künstlich einzubauen und ihnen damit Fähigkeiten zu verleihen, die sie von Natur aus nicht hätten. Paradebeispiel sind Bakterien, die durch Einbau menschlicher Gene in der Lage sind, das Hormon Insulin zu produzieren, das an Diabetes erkrankte Personen injiziert werden kann. Diesen Prozess bezeichnet man als Transformation. Im Rahmen unseres Praktikums im Februar 2007 führten wir im Genlabor der EFS von Frau Dr. Hausmann auch eine Plasmid-Präparation durch. Wir experimentierten mit den für Menschen harmlosen Escherichia coli K-12 Stämmen DH-5α α und HB101. Die Plasmid-Präparation besteht im Wesentlichen aus folgenden Schritten: 1. Alkalische Lyse der Bakterienzellen und Reinigung der Plasmid-DNA Hierzu werden die Bakterienzellwände mit einer alkalischen Lösung aufgeschlossen und denaturiert. Durch den TENS-Puffer werden die Wasserstoffbrückenbindungen der gesamten DNA aufgehoben. Durch anschließende Neutralisation mit Natriumacetat finden die Einzelstränge des Plasmids wieder zusammen, sie bleiben in Lösung. Die zelluläre DNA des Bakteriums ist zu groß, um basengenau zusammen zu finden, es kommt zu Fehlbasenpaarungen, deshalb kann man sie als Knäuel durch Zentrifugation vom Plasmid abtrennen. Reinigungsprozesse, Abzentrifugieren und Verdau durch eine RNase sollen für einen hohen Reinheitsgrad der Plasmid-DNA sorgen. 2. Restriktionsanalyse Die Restriktionsanalyse liefert in erster Näherung, ob ein Plasmid, das man in Bakterien eines anderen Stammes einschleusen wollte, dort auch tatsächlich repliziert wurde, also ob die Transformation auch gelungen ist. Dazu wird in einem Restriktionsverdau Plasmid-DNA mit Hilfe eines spezifischen Enzyms, das wie eine molekulare Schere arbeitet, in Fragmente geschnitten. Wir verwendeten die Nuclease EcoRI. Durch einen Probenpuffer färbten wir die Reaktionslösung blau. 3. Gelelektrophorese Die Proben werden zusammen mit einer DNA-Leiter als Maßstab in die Taschen eins Agarose-Gels gefüllt. Dabei muss man aufpassen, dass man mit der Pipettenspitze nicht das Gel durchstößt. Die Fragmente werden nun im elektrischen Feld der Größe nach aufgetrennt. Dabei kann man übrigens auch sehen, wer sauber gearbeitet hat! 4. Transformation Es gibt verschiedene Methoden der Transformation, aber in jedem Fall möchte man ein ausgetestetes Plasmid mit einem bestimmten Gen (oder mehreren) in einen anderen Organismus schleusen. Unser Plasmid enthielt zusätzlich ein Resistenzgen gegen das Antibiotikum Ampicillin (ampr). Damit die Bakterien die Plasmide aufnehmen, „stressten“ wir sie in der Kälte, so dass die Zellwände perforiert wurden. Nach Inkubation und Schütteln plattierten wir die Bakterien auf Ampicillin-Platten. Das Antibiotikum selektierte also alle Bakterien aus, die kein Plasmid aufgenommen haben. das Verlaufsschema Frau Dr. Hausmann gießt die Agarose-Gele in den vorbereiteten Kammern. Vanessa zentrifugiert bei 13000 rpm feste Bestandteile und denaturierte DNA ab. Aufgepasst Mädels: Nichts ist kribbliger als Vortexen! Julia, Mikela, Tamara und Angela Höchste Konzentration beim Umgang Eppendorf-Pipetten: Patrick und Eduard mit den sensiblen UV-Licht bringt es an den Tag! Hier kann man auch sehen, wer sauber gearbeitet hat!