Charakterisierung von Bakterien
Werbung
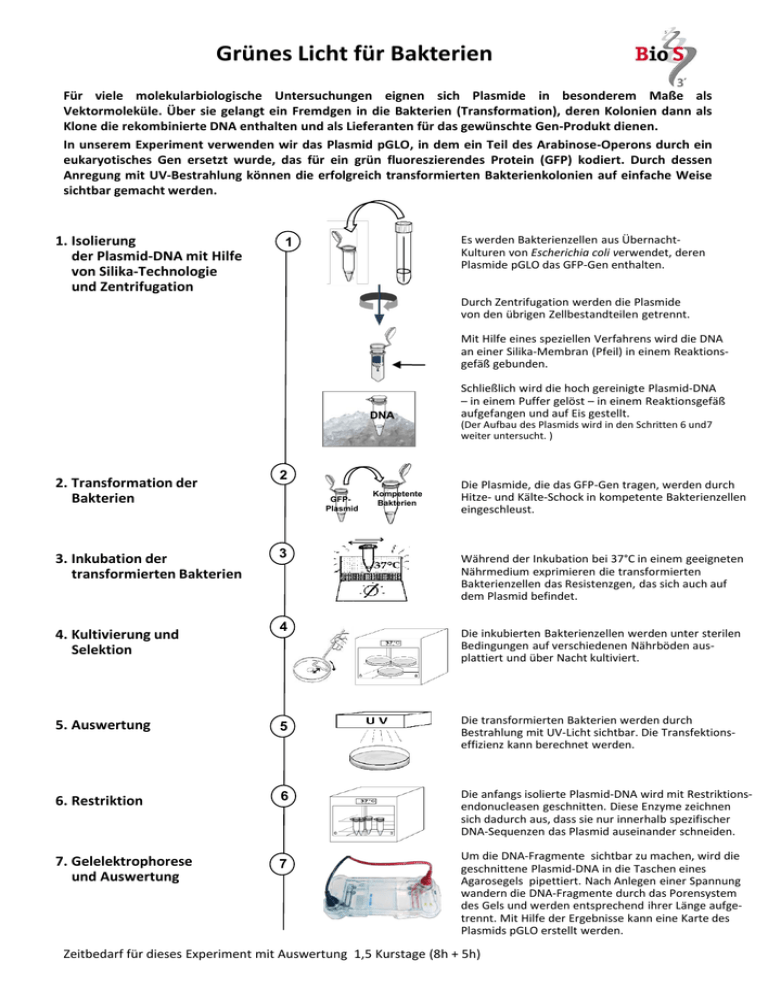
Grünes Licht für Bakterien Für viele molekularbiologische Untersuchungen eignen sich Plasmide in besonderem Maße als Vektormoleküle. Über sie gelangt ein Fremdgen in die Bakterien (Transformation), deren Kolonien dann als Klone die rekombinierte DNA enthalten und als Lieferanten für das gewünschte Gen-Produkt dienen. In unserem Experiment verwenden wir das Plasmid pGLO, in dem ein Teil des Arabinose-Operons durch ein eukaryotisches Gen ersetzt wurde, das für ein grün fluoreszierendes Protein (GFP) kodiert. Durch dessen Anregung mit UV-Bestrahlung können die erfolgreich transformierten Bakterienkolonien auf einfache Weise sichtbar gemacht werden. 1. Isolierung der Plasmid-DNA mit Hilfe von Silika-Technologie und Zentrifugation Es werden Bakterienzellen aus ÜbernachtKulturen von Escherichia coli verwendet, deren Plasmide pGLO das GFP-Gen enthalten. 1 Durch Zentrifugation werden die Plasmide von den übrigen Zellbestandteilen getrennt. Mit Hilfe eines speziellen Verfahrens wird die DNA an einer Silika-Membran (Pfeil) in einem Reaktionsgefäß gebunden. DNA 2. Transformation der Bakterien 3. Inkubation der transformierten Bakterien 4. Kultivierung und Selektion 2 GFPPlasmid 3 4 5. Auswertung 5 6. Restriktion 6 7. Gelelektrophorese und Auswertung 7 Kompetente Bakterien Schließlich wird die hoch gereinigte Plasmid-DNA – in einem Puffer gelöst – in einem Reaktionsgefäß aufgefangen und auf Eis gestellt. (Der Aufbau des Plasmids wird in den Schritten 6 und7 weiter untersucht. ) Die Plasmide, die das GFP-Gen tragen, werden durch Hitze- und Kälte-Schock in kompetente Bakterienzellen eingeschleust. Während der Inkubation bei 37°C in einem geeigneten Nährmedium exprimieren die transformierten Bakterienzellen das Resistenzgen, das sich auch auf dem Plasmid befindet. Die inkubierten Bakterienzellen werden unter sterilen Bedingungen auf verschiedenen Nährböden ausplattiert und über Nacht kultiviert. Die transformierten Bakterien werden durch Bestrahlung mit UV-Licht sichtbar. Die Transfektionseffizienz kann berechnet werden. Die anfangs isolierte Plasmid-DNA wird mit Restriktionsendonucleasen geschnitten. Diese Enzyme zeichnen sich dadurch aus, dass sie nur innerhalb spezifischer DNA-Sequenzen das Plasmid auseinander schneiden. Um die DNA-Fragmente sichtbar zu machen, wird die geschnittene Plasmid-DNA in die Taschen eines Agarosegels pipettiert. Nach Anlegen einer Spannung wandern die DNA-Fragmente durch das Porensystem des Gels und werden entsprechend ihrer Länge aufgetrennt. Mit Hilfe der Ergebnisse kann eine Karte des Plasmids pGLO erstellt werden. Zeitbedarf für dieses Experiment mit Auswertung 1,5 Kurstage (8h + 5h)











