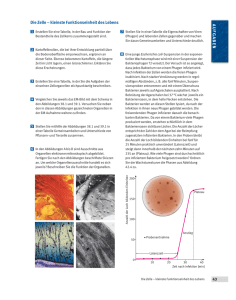
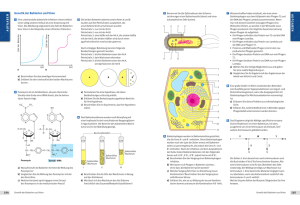
Fortgeschrittenen Kurs Genetik und Molekularbiologie, WS 2005/2006
Werbung

Fortgeschrittenen Kurs Gentechnik und Molekularbiologie, WS 2006/2007 Die Technik der Phagenpräsentation 1. Woche: Mo- Do: Phage Display 2. Woche: Mo: Phagen SDS Gel; Di: Sequenzierung; Do: Proteinstruktur; Fr: Sequenzauswertung Leitung: Kristian Müller, Institut für Biologie III; Schänzlestr. 1, 79104 Freiburg Tel. 203-2748; e-mail [email protected] Technische Assistentin: Susanne Knall, Karin Schmidtkunz Kursassistenz: Janina Speck, Dinah Mattay, Christina Räuber Skriptversion 1.1/07 Ziel des Kurses In dem Kurs soll die Technik der Phagenpräsentation (Phage Display) anhand eines anwendungsorientierten Beispiels erlernt werden. Der Trick des Phage Display besteht darin, daß ein Protein und das dazugehörige kodierende Gen durch den Phagen in einer sehr gut handhabbaren Form, d.h. dem Phagen, zusammengehalten werden. Erzeugt man beispielsweise durch verzufallte Gensynthese hundert Millionen bis eine Milliarde verschiedene Gene, lassen sich mit Hilfe des Phage Display hundert Millionen bis eine Milliarde verschiedener Phagen erzeugen, in denen jeweils das präsentierte Peptid mit dem Gen verbunden ist. Aus diesem hoch komplexen Gemisch lassen sich dann Phagen anreichern, die beispielsweise ein bestimmtes Protein binden. In dem Kurs wird mit einer vorgefertigten Phagenbibliothek (New England Biolabs Ph.D-7 Phage Display Peptide Library Kit) gearbeitet, aus der Streptavidin bindende Peptide gewonnen werden sollen. Hierzu werden insgesamt drei Anreicherungsrunden (Panning-Runden) durchgeführt, zwei davon im Praktikum. Hierbei wird die Technik des “Panning” erlernt. Die Phagen werden hierfür amplifiziert und durch Fällung gereinigt. Die Anreicherung bindender Phagen soll durch TiterBestimmung mit Plaques, photometrische Phagenbestimmung und Phagen-ELISA verfolgt werden. In einem Nebenexperiment werden besonders reine Phagen durch eine Ultrazentrifugation im CsClGradienten gewonnen. Am Ende wird durch DNA-Sequenzierung die Aminosäure Sequenz eines binden Peptids abgeleitet, das von jeder Gruppe im Praktikum gefunden werden soll. Ergänzt werden die experimentellen Arbeiten durch Computer-gestützte Auswertungen. An einem halben Tag sollen vorhandene Strukturdaten eines Peptids im Komplex mit Streptavidin am Rechner dargestellt werden. An einem weiteren Tag wird eine Rechner-gestützte Klonierung durchgeführt, wobei der Phagen Vektor mit Bibliothek am Rechner zusammengesetzt wird. Die am Computer erzeugte Sequenz dient dann dazu, die Sequenzierung des gefundenen Klons auszuwerten. Streptavidin bindende Peptide, die über Phage Display gewonnen wurden, werden in der Praxis für die Proteinreinigung und -detektion genutzt. Gentechnik Kurs: Phagen Präsentation -2- Hinweis zum Ablauf Das Praktikum besteht aus einem einzigen Versuch mit mehreren Teilen, der so wie im richtigen Labor durchgeführt wird. Auch Profis benötigen vom Start bis zur DNA Sequenz ca. sieben Tage. Um diesen Versuch in einem einwöchigen Praktikum durchzuführen, wurde die erste von drei Panning-Runden bereits durchgeführt. Das Praktikum beginnt also mit Panning-Runde zwei (P2). Während des Praktikums muß jeder Teilnehmer ein “Labor-Buch” führen. Hierzu wird ein Heft ausgegeben, in dem jeder Versuch und das Ergebnis mit Datum und Uhrzeit einzutragen ist. Das Heft wird während des Kurses von den Assistenten eingesehen, und die Hefte einer Gruppe müssen mit dem Protokoll abgegeben werden. Hinweis zur Einteilung Das Praktikum wird aus didaktischen und finanziellen Gründen in zweier Gruppen absolviert. Damit jedoch jeder Teilnehmer ausreichend experimentelle Erfahrung sammelt, werden Teile der Versuche von jedem Teilnehmer durchgeführt. D.h. jeder führt zwei “Panning-Runden” durch, gerätetechnisch oder finanziell limitierte Schritte wie die Phagenbestimmung auf Platten, die Ultrazentrifugation oder die Sequenzierung werden jedoch nur mit einer Probe pro Gruppe durchgeführt. Um den Fortschritt der Phagenpräsentation von Runde zu Runde zu beobachten, sollte sich jede Gruppe von Anfang an auf eine Probe als die “Hauptprobe” festlegen. Übersicht Methode Phagenselektion Immunoröhrchen belegen Phagen binden Phagenproduktion Zellen in Flüssigkultur Phagen waschen Phagen eluieren Titerbestimmung Plaques / infizierte Zellen auf Agar-Platte PEG/NaCl-Fällung Abb. 1 Übersicht Phagenpräsentation Zellen infizieren Gentechnik Kurs: Phagen Präsentation -3- Übersicht Versuch Phage Display Versuch Mo Zusätzliche Arbeiten Bindung der ausgegebenen Phagen an ausgegebene Röhrchen, d.h. Panning Runde P2 das Neutralisationstest; Belegen eines Immunoröhrchens und der ELISA Platten; X-Gal-Platten gießen Elution der Phagen (pro Gruppe) Amplifikation der Phagen mit ausgegebener Kultur Fällung der amplifizierten Phagen Über-Nacht Kultur von ER2738 Zellen (pro Gruppe) Di Aufarbeitung der Phagenfällung P2 Panning Runde P3 mit selbst belegtem Röhrchen Blocken der und Platten Immunoröhrchen Titerbestimmung durch Plattieren (pro Gruppe) und Absorptionsmessung von Eluat P2, Amplifikation P2 und Eluat P3 Phagenamplifikation P3 mit eigener Kultur Phagenfällung P3 Mi Aufarbeitung der Phagenfällung P3 Phagenkultur eines Klons von der Platte der Titerbestimmung P3 Präparation einzelsträngiger DNA Do Phagen ELISA Phagenreinigung mit Gradient (pro Gruppe) Fr Doktorandenvorträge Mo SDS Gel mit Phagen Di Sequenzierreaktion (pro Gruppe) Do Fr CsCl Proteinstruktur am Computer Sequenzauswertung des Klons (pro Gruppe) Computer gestütztes Klonieren Gentechnik Kurs: Phagen Präsentation -4- Einführung Selektion und die Kopplung von Genotyp und Phänotyp Abb. 2 Übersicht über mögliche Kopplungen von Genotyp und Phänotyp auf molekularer Ebene. A) Fusion an das Phagenprotein VIII, B) Fusion an das Phagenprotein III im Phagensystem, C) Fusion an das Phagenprotein III im Phagmidsystem, D) Fusion an ein Zelloberflächenprotein, E) Kopplung des Proteins mit der mRNA. Erklärungen zum Phagendisplay siehe Text. Die Entstehung der M13 Phagenvektoren Bakteriophagen, auch bakterielle Viren oder kurz Phagen genannt, infizieren, wie der Name besagt, Bakterien. Phagen wurden unabhängig voneinander 1915 von Frederick W. Twort und 1917 von Félix d'Hérelle beschrieben. d'Hérelle prägte dem damaligen Verständnis folgend den Begriff “Bakteriophage” (Bakterienesser). Für die Phagenpräsentation werden filamentöse, nicht lytische Bakteriophagen als Vehikel verwendet. Filamentöse Phagen sind zum Verpacken manipulierter DNA besonders geeignet, da die Phagenlänge automatisch der Genomgröße angepaßt wird. Diese Phagen wurden ursprünglich aufgrund ihrer auf “männliche” E. coli-Stämme beschränkten Infektiösität isoliert. Diese Bakteriophagen werden wie folgt klassifiziert: Viren; ssDNA Viren; Inoviridae; Inovirus. Es gibt drei sehr nah verwandte filamentöse Phage “f1“, “M13“ und “fd” deren Genome zu etwa 98 % identisch ist. Der Phage M13 (für München 13) wurde 1963 das erste Mal beschrieben (Hofschneider, 1963). Das Genom dieses wild-Typ Phagen umfaßt 6407 Nukleotide. Um diesen Phagen als Klonierungsvektor zur Produktion einzelsträngiger DNA, die ursprünglich zur Sanger Sequenzierung und später für die Mutagenese benötigt wurde, einsetzen zu können, wurde ein 789 Bp großes Fragment in das Genom eingesetzt, das das lac Operon und die ersten 146 Aminosäuren der ß-Galactosidase enthielt. Dies führte zu dem Klon M13mp1, dem ersten Klon der sehr erfolgreichen Serie der M13mp Phagenvektoren (mp für Max-Planck-Institut) (Messing et al. 1977). Durch chemische Mutagenese mit Nitrosomethylharnstoff und Selektion und später durch Klonierungen wurden zusätzliche Restriktionsschnittstellen vor allem als multiple Klonierungsstelle in das Phagengenom eingeführt. Aus diesen Phagenvektoren leitet sich die ebenfalls sehr weit verbreitete pUC-Phagmidserie ab (Vieira & Messing 1982). Das in den M13 Phagen kodierte ß-Galactosidase Fragment, das historisch bedingt auch α-Fragment genannt wird, ermöglicht die sogenannte α-Komplementation im Zusammenspiel mit einem E. coliStamm, der eine Galactosidase ohne die N-terminalen Aminosäuren (auch ω-Akzeptor genannt) exprimiert (Ullmann et al. 1967). In der Praxis wird häufig die lacZΔM15 Deletion verwendet, bei der die Aminosäuren 11-41 der Galactosidase fehlen. Die α-Komplementation läßt sich durch die Gentechnik Kurs: Phagen Präsentation -5- Zugabe des Farbstoffs X-Gal sichtbar machen, wodurch die sogenannte blau-weiß Selektion ermöglicht wird. Literatur: Hofschneider HP, 1963, “Untersuchung über kleine E. coli K-12 Bacteriophagen M12, M13 und M20“ Z. Naturforsch. 18, 203-205. Messing J, Gronenborn B, Müller-Hill B, Hofschneider PH, 1977, “Filamentous coliphage M13 as a cloning vehicle: Insertion of a HindII fragment of the lac regulatory region in M13 replicative form in vitro”, PNAS 74, 3642-6. Vieira J, Messing J, 1982, “The pUC plasmids, an M13mp7-derived system for insertion mutagenesis and sequencing with synthetic universal primers”, Gene, 19, 259-68. Yanisch-Perron C, Vieira J, Messing J., 1985, “Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors”, Gene, 33, 103-19. Ullmann A, Jacob F, Monod J., 1967, “Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the beta-galactosidase structural gene of Escherichia coli”, J Mol Biol., 24(2):339-43. Die Erfindung der Phagenpräsentation Georg Smith publizierte die Idee der Phagenpräsentation 1985 in einer wegweisenden Publikation mit dem Titel: “Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface.” Science 1985 228(4705):1315-7. In seiner Zusammenfassung beschreibt er diese Entwicklung wie folgt: “Foreign DNA fragments can be inserted into filamentous phage gene III to create a fusion protein with the foreign sequence in the middle. The fusion protein is incorporated into the virion, which retains infectivity and displays the foreign amino acids in immunologically accessible form. These "fusion phage" can be enriched more than 1000-fold over ordinary phage by affinity for antibody directed against the foreign sequence. Fusion phage may provide a simple way of cloning a gene when an antibody against the product of that gene is available.” Diese Technik findet seit Anfang der 90er Jahre weite Verbreitung vor allem seit man merkte, daß sich so sehr einfach aus verzufallten Sequenzbibliotheken Peptide aber auch Proteine mit bestimmten Bindungseigenschaften isolieren lassen. Hierin begründet sich auch die große kommerzielle Bedeutung, da sich so einfach Antikörperfragment-Bibliotheken auf spezifische Bindung oder Peptide auf Bindung oder Inhibition selektionieren lassen. Die Idee der Phagenpräsentation legte auch den Grundstein für zahlreiche weitere Präsentationstechniken, wie zelluläre Präsentation, Viren-Präsentation und Ribosomen-Präsentation. Gentechnik Kurs: Phagen Präsentation -6- Abb. 3 Vermehrung eines M13 Phagen. 1) Infektion; 2) Herstellung der doppelsträngigen Form = relikative Form (RF) der DNA und Vermehrung zu ca. 100 Kopien; 3) Herstellung der einzelsträngigen Form; 4) Herstellung der Phagenproteine; 5) nicht lytische Ausschleusung von ca. 200 Phagenpartikeln. Prinzip und Varianten der Phagenpräsentation Abstrakt betrachtet ermöglicht die Phagenpräsentation die Kopplung eines Phänotyps mit einem stabil verpackten Genotyp, da die Proteine, die den Phagen bilden, in seinem Genom kodiert werden. Bei einer Transformation mit einem Phagemid oder einem Phagengenom oder bei einer Phageninfektion gelangt in der Regel jeweils nur ein Genom in die Zelle und es werden in dieser Zelle nur zusammengehörige Protein-Genom Einheiten verpackt. Das zu selektionierende Protein kann zur Präsentation mit verschiedenen Hüllproteinen des Phagen fusioniert werden. Das GenIII Protein bietet sich hierzu besonders an, da es nur in 3 bis 5 Kopien an der Spitze des Phagen vorkommt und gut zugänglich ist. Das GenIII-Protein gliedert sich in drei Domänen, wobei die zwei N-terminalen Domänen nach außen weisen und für die Bindung des Phagen an Zellen und somit für die Infektiösität des Phagen wichtig sind. Die C-terminale Domäne verankert das Protein in der Phagenhülle. Kurze Peptide bis zu 20 Aminosäuren interferieren nur wenig mit der Zellbindung und können genetisch direkt an das 5'-Ende des genIII fusioniert werden. Durch die mehrfache Präsentation solcher Fusionen an der Oberfläche erhält man einen Aviditätseffekt, d.h. die beobachtete Affinität eines Phagenpartikels ist deutlich höher als die intrinsische Affinität eines einzelnen Peptids. Diese Art der Phagenpräsentation wird im Praktikum angewendet. Ist kein Aviditätseffekt erwünscht oder sollen Proteine präsentiert werden, die aufgrund ihrer Größe mit der Infektiösität interferieren, greift man auf ein sogenanntes Phagmid (engl. Phagemid) zurück. Phagmide sind Plasmide, die einen Plasmid Replikationsursprung enthalten und daher wie Plasmide Gentechnik Kurs: Phagen Präsentation -7- propagiert werden können. Zudem enthalten sie einen Phagen Replikationsursprung, der in Gegenwart entsprechender Proteine die Verpackung einzelsträngiger DNA in Phagen ermöglicht. Um die Verpackung einzelsträngiger DNA zu ermöglichen superinfiziert man Phagmid tragende Zellen mit einem Helfer-Phagen. Dieser Helfer-Phage enthält alle Gene, die benötigt werden, um Phagenpartikel zu produzieren. In seinem Genom ist jedoch eine Mutation, die dazu führt, daß sein eigenes Genom nur sehr schlecht verpackt wird. Daher wird nach einer Superinfektion mit HelferPhagen fast ausschließlich das Phagmid in Phagenpartikel verpackt. Phagmide für die genIII Phagenpräsentation enthalten eine genetische Fusion mit dem vollständigen genIII oder nur mit dem für die C-terminal kodierenden Bereich. Die Expression dieser Fusion erfolgt durch einen schwachen Promotor, so daß weniger Fusionsproteine gebildet werden als vollständiges GenIIIProteine durch den Helfer-Phagen. Im Mittel werden daher nur ein Fusionsprotein und zwei bis vier Wild-Typ GenIII-Proteine auf dem Phagen präsentiert, der so seine Infektiösität erhält. Ist ein besonders hoher Aviditätseffekt erwünscht, kann auch das GenVIII-Protein, das in mehreren tausend Kopien die Phagenhülle bildet, als Fusionspartner verwendet werden. Hierbei kann wiederum auf Phagen oder Phagemid zurückgegriffen werden. Da Fusionen mit dem genVIII den Phagenaufbau beeinflussen können, sind Phagen mit mutiertem genVIII entwickelt worden. Weitere Hüllproteine des Phagen sind für Fusionen getestet worden, diese Konstrukte finden jedoch keine weite Verbreitung. Der Praktikumsversuch Für das Praktikum wird eine kommerziell erhältliche Phagenbibliothek eingesetzt, bei der sieben vollständig verzufallte Kodone (nnk) zwischen die Signalsequenz und das GenIII-Protein fusioniert wurden. Um dies zu erreichen wurde zuerst aus dem Phagen M13mp18 eine Kpn I Schnittstelle durch eine Deletion aus der multiplen Klonierungstelle entfernt und dann die Bibliothek über KpnI und EagI kloniert. Hieraus resultiert die unten ausschnittsweise gezeigte Genstruktur. Im Praktikum sollen aus dieser Phagenbibliothek, die ca. 1,2×109 verschiedene Phagen enthält, Sequenzen gefischt werden, die Streptavidin binden. Hierzu werden insgesamt 3 Panning-Runden benötigt. Gentechnik Kurs: Phagen Präsentation -8- Genstruktur der Bibliothek |-> genIIIp Signal Sequenz ATTCACCTCGAAAGCAAGCTGATAAACCGATACAATTAAAGGCTCCTTTTGGAGCCTTTTTTTTTGGAGATTTTCAACGTGAAAAAATTATTATTCGCAA 1501 ---------+---------+---------+---------+---------+---------+---------+---------+---------+---------+ 1600 TAAGTGGAGCTTTCGTTCGACTATTTGGCTATGTTAATTTCCGAGGAAAACCTCGGAAAAAAAAACCTCTAAAAGTTGCACTTTTTTAATAATAAGCGTT a V K K L L F A I |->reifes genIIIp KpnI/Acc65I |-> Bibliothek <-| EagI | | | TTCCTTTAGTGGTACCtttctattctcactctnnknnknnknnknnknnknnkggtggaggttCGGCCGAAACTGTTGAAAGTTGTTTAGCAAAATCCCA 1601 ---------+---------+---------+---------+---------+---------+---------+---------+---------+---------+ 1700 AAGGAAATCACCATGGaaagataagagtgagannmnnmnnmnnmnnmnnmnnmccacctccaaGCCGGCTTTGACAACTTTCAACAAATCGTTTTAGGGT a P L V V P F Y S H S ? ? ? ? ? ? ? G G G S A E T V E S C L A K S H - TACAGAAAATTCATTTACTAACGTCTGGAAAGACGACAAAACTTTAGATCGTTACGCTAACTATGAGGGTTGTCTGTGGAATGCTACAGGCGTTGTAGTT 1701 ---------+---------+---------+---------+---------+---------+---------+---------+---------+---------+ 1800 ATGTCTTTTAAGTAAATGATTGCAGACCTTTCTGCTGTTTTGAAATCTAGCAATGCGATTGATACTCCCAACAGACACCTTACGATGTCCGCAACATCAA |- -96 primer ------| Abb. 4 Elektronenmikroskopische Aufnahme eines filamentösen Phagen. Diese Phagen messen 6-8 nm im Durchmesser und können zwischen 760 und 1900 nm lang sein. Abb. 5 Schema der Phagenbindung undpräsentation Gentechnik Kurs: Phagen Präsentation -9- Technische Information Phagengenom in der Datenbank Die Phagengenome sind beispielsweise in der EMBL Gendatenbank abgelegt und können mit den Identifikationsnummer V00604 für M13 (6407 Bp) und X02513 für M13mp18 (7249 Bp) abgerufen werden. Der Genetische Code mit JCBN / IUBMB Abkürzungen Joint Commission on Biochemical Nomenclature (JCBN). The JCBN is jointly responsible to the International Union of Pure and Applied Chemistry (IUPAC) and the International Union of Biochemistry and Molecular Biology (IUBMB). Symbol Bedeutung Begründung der Namensgebung G G Guanine A A Adenine T T Thymine C C Cytosine R G or A puRine Y T or C pYrimidine M A or C aMino K G or T Keto S G or C Strong interaction (3 H bonds) W A or T Weak interaction (2 H bonds) H A or C or T not-G, H follows G in the alphabet B G or T or C not-A, B follows A V G or C or A not-T (not-U), V follows U D G or A or T not-C, D follows C N G or A or T or C aNy Definition der komplementären Symbole Symbol A B Complement T C D G H K V G H C M S D M K * In certain cases the symbol and its complement are identical. S* T V W A B W* N N* Gentechnik Kurs: Phagen Präsentation - 10 - Der bakterielle Übersetzungsschlüssel: AAs Starts Base1 Base2 Base3 = = = = = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG ---M---------------M------------MMMM---------------M-----------TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG Der Einbuchstabenschlüssel für Aminosäuren: (http://www.chem.qmul.ac.uk/iupac/AminoAcid/AA1n2.html) Trivial name Symbols Systematic name Alanine Arginine Ala A 2-Aminopropanoic acid CH3-CH(NH2)-COOH Arg R 2-Amino-5-guanidinopentanoic acid H2N-C(=NH)-NH-[CH2]3-CH(NH2)COOH Asnd N d 2-Amino-3-carbamoylpropanoic acid H2N-CO-CH2-CH(NH2)-COOH Aspd D d 2-Aminobutanedioic acid HOOC-CH2-CH(NH2)-COOH Cys C 2-Amino-3-mercaptopropanoic acid HS-CH2-CH(NH2)-COOH Glnd Qd 2-Amino-4-carbamoylbutanoic acid H2N-CO-[CH2]2-CH(NH2)-COOH Glud E d 2-Aminopentanedioic acid HOOC-[CH2]2-CH(NH2)-COOH Gly G Aminoethanoic acid CH2(NH2)-COOH His H 2-Amino-3-(1H-imidazol-4-yl)propanoic acid Asparagine Aspartic acid Cysteine Glutamine Glutamic acid Glycine Histidine Isoleucine Leucine Lysine Methionine Phenylalanine Proline Ile Leu Lys Met Phe Pro I L K M F P 2-Amino-3-methylpentanoic acide 2-Amino-4-methylpentanoic acid 2,6-Diaminohexanoic acid 2-Amino-4-(methylthio)butanoic acid 2-Amino-3-phenylpropanoic acid Pyrrolidine-2-carboxylic acid Serine Threonine Tryptophan Ser S 2-Amino-3-hydroxypropanoic acid Thr T 2-Amino-3-hydroxybutanoic acid e Trp W 2-Amino-3-(lH-indol-3-yl)propanoic acid Tyrosine Tyr Y 2-Amino-3-(4-hydroxyphenyl)propanoic acid Valine Val V 2-Amino-3-methylbutanoic acid Unspecified amino acid Xaa Xf Formula C2H5-CH(CH3)-CH(NH2)-COOH (CH3)2CH-CH2-CH(NH2)-COOH H2N-[CH2]4-CH(NH2)-COOH CH3-S-[CH2]2-CH(NH2)-COOH C6H5-CH2-CH(NH2)-COOH HO-CH2-CH(NH2)-COOH CH3-CH(OH)-CH(NH2)-COOH (CH3)2CH-CH(NH2)-COOH Gentechnik Kurs: Phagen Präsentation - 11 - Montag Das Praktikum beginnt aus Zeitgründen mit der zweiten Runde (P2) Materialien Phagen Stock amplifiziert nach der ersten Runde (P1-Ampl) 2 Immunoröhrchen belegt mit Streptavidin 2 neue Immunoröhrchen 2 96-well Flachboden ELISA Platten ca. 6.5 ml Streptavidin 30 μg/ml in 0.1 M NaHCO3-Puffer pH 9.0 50 mM Tris, 150 mM NaCl, 0.1% (v/v) Tween 20, pH 7,5 (TBST Puffer) Abgeschnitte Spitzen für Tween 0.2 M Glycin/HCl Puffer pH 2,2 Rollentaumelmischer / Radmischer Zentrifuge für 50 ml Gefäße Photometer bei 600 nm Über-Nacht Kultur von ER2738 Zellen ( F́ proA+B+ lacIq Δ(lacZ)M15 zzf:: Tn(TetR)/ fhuA2 glnV Δ(lac-proAB) thi-1Δ(hsdS-mcrB)5 ) 1 l Kolben LB Medium Tetrazyklin Stammlösung 20 mg/ml BSA Stammlösung 100 mg/ml 1 M Tris-Stammlösung pH 9.1 Stift, Klebeetiketten, Klebepunkte, Heft 1.5 % Agar 150 ml IPTG/ X-gal 150 μl (50 mg/ml Isopropyl-β-D-thiogalactosid, 40 mg/ml 5-Bromo-4-chloro-3indolyl-β-D-galactosid in Dimethylformamid) pH Stäbchen 20% PEG 6000 / 2.5 M NaCl Lösung Feuerzeug 50 ml Plastikgefäße Plastikküvetten P20, P200, P1000, Eppiständer, Spitzenboxen, Eiskübel (aus Praktikumsbestand) Eis, Eispott Kultur für die Phagenamplifikation Nach der Selektion auf Bindung müssen die bindenden Phagen wieder vermehrt werden. Die optische Dichte bei 600 nm der Über-Nacht-Kultur (OD 600) wird mit einem Photometer gegen das reine Medium als Referenz bestimmt. Von der Über-Nacht-Kultur wird eine 100 ml Hauptkultur angesetzt. Hierzu wird mit einem 50 ml PP Gefäß das Volumen an LB Medium abgemessen, in ein 1 l Kolben gegeben, mit Tet (20 μg/ml final) versetzt und Start OD von 0,1 angesetzt und bei 37 °C geschüttelt. Da sich der Schüttler im 4. Stock befindet, bitte die Probe auf den bereitgestellten Wagen stellen. Gentechnik Kurs: Phagen Präsentation - 12 - Im weiteren Verlauf werden Zellen bei einer OD von ca. 0.5 für die Phageninfektion benötigt. Das Hochwachsen der Zellen dauert in Abhängigkeit der Vitalität der Zellen 1 bis 3 Stunden. Die optische Dichte der Zellen wird daher mindestens stündlich kontrolliert. Sollten die Zellen zu schnell wachsen, können sie bis zur weiteren Anwendung bei 4°C gelagert werden. Anmerkung: Phagen infizieren über Pili, die sich am besten in der frühen log-Phase und unter günstigen Wachstumsbedingungen (ausreichend Sauerstoffzufuhr, reiches Medium) ausbilden; zu heftiges Schütteln von Schikanekolben sollte vermieden werden. Die für die Bildung der Pili erforderliche Information befindet sich auf dem F Episom, das eine Tet Resistenz trägt. Selektion bindender Phagen Dieser Schritt ist das Kernstück der Phagenpräsentation. 200 ml TBST-Puffer mit TBS (Tris Buffered Saline) und 0,1% (v/v) Tween 20 in einer kleinen Flasche ansetzen. Zum Waschen Immunoröhrchen 2 mal mit TBST füllen und in Abfallgefäß entleeren. Immunoröhrchen mit 1 ml TBST füllen, BSA Stammlösung auf final 1 mg/ml zusetzen und 30 μl Phagen Stock von einer ersten Panning-Runde “P1” zugeben. Röhrchen 1 h auf einem Rollentaumelmischer inkubieren. Anmerkung: Häufig wird zum Blockieren unspezifischer Bindung fettarmes Trockenmilchpulver verwendet, dieses ist billiger als BSA und blockiert in der Regel besser. Milch enthält jedoch Spuren von Biotin, das das Streptavidin blockieren würde. Mikro-Titration der Neutralisation Die Bindung der Peptide an Streptavidin wird durch Absenkung des pH-Wertes gebrochen. Da die Phagen bei niedrigem pH schneller denaturieren, wird nach der Elution neutralisiert und diese Neutralisation wird vorab einmal pro Gruppe getestet. 1 ml Gly/HCl pH 2,2 mit 100 μl 1M Tris pH >= 9 versetzen und 10 μl auf ein pH Stäbchen geben. Wenn der pH Wert unter 7 liegt, nochmals 50 μl zugeben und nochmals auf einem neuen pH-Stäbchen testen. Wenn sich der pH in etwa einstellt, in 10 μl Schritten weiterarbeiten. Abschließend wird empfohlen, das getestete Volumen nochmals mit 1 ml der Gly/HCl-Lsg zu testen. Anmerkung: Diese Art der Mikrotitration soll hier exemplarisch gezeigt werden, sie eignet sich zur schnellen Bestimmung sehr kleiner Probenvolumen, auch mit 5 μl Tropfen kann der pH Wert in vielen Fällen noch genau genug bestimmt werden. Gegen Ende der Phagen-Inkubation sollte die optische Dichte der Zellen kontrolliert werden. Phagen Waschen, Elution und Neutralisation Nach der Inkubation wird die Phagenlösung in den biologischen Abfall gegeben. Das Röhrchen wird 6 mal mit TBST bis zum Rand gefüllt und ausgeleert und ausgeklopft. Zur Elution wird 1 ml Glycin/HCl pH 2.2 mit final 1mg/ml BSA versetzt und in das Röhrchen gegeben und 10 min auf dem Taumelrollenmischer gedreht. Ein 1,5 ml Eppi wird mit einem kleinen Haftetikett markiert. Hierauf wird der Name, das Datum und der einzufüllende Inhalt festgehalten (Phagen Eluat 2. Panning Runde anti Streptavidin, z.B. Gentechnik Kurs: Phagen Präsentation - 13 - kurz P2EL Strept). Zusätzlich kommt ein Aufkleber auf den Deckel, der nur die Abkürzung trägt. Mit einem rundherum geklebten Tesafilm wird das Etikett geschützt. Die Phagenelution wird in das beschriftete 1,5 ml Eppi überführt und mit der in der Mikrotitration bestimmten Menge an Tris-Puffer (ca. 150 μl) neutralisiert. Phagen Amplifikation 1 ml des Phageneluats wird zu 40 ml ER 2738 Zellen in der frühen log Phase bei einer OD600 von ca. 0,5 gegeben. 20 ml der 100 ml Kultur werden verworfen und zum Autoklavieren gegeben. Nach der Zugabe der Phagen die Kultur schwenken und 10 min auf dem Tisch stehen lassen, dann 4 (- 5 h) bei 37 °C schütteln. Belegen eines Immunoröhrchens und einer Mikrotiterplatte Für eine weitere Panning-Runde müssen ein Immunoröhrchen und für den Test der Phagenbindung eine Mikrotiterplatte mit Streptavidin belegt werden (siehe Schema vom Donnerstag). Jeweils 1,5 ml der bereits angesetzten Beleglösung (30 μg/ml Streptavidin in NaHCO3-Puffer) in ein Immunoröhrchen geben. Auf zwei Mikrotiterplatten in der Mitte ein Raster von 3 × 5 markieren und jeweils 75 μl der Beleglösung hineinpipettieren. Zwei Vertiefungen werden zusätzlich mit 75 μl als Referenz befüllt und der Rest auf die zwei bereits befüllten Immunoröhrchen verteilen. Die Röhrchen und Platten werden im Kühlraum über Nacht bei 4 °C gedreht bzw. geschüttelt. Mittagspause Herstellung von Platten für die Phagentiter Bestimmung 150 ml 1.5 % Agar in LB werden im Wasserbad flüssig gehalten. Eine 1:1000 Verdünnung der IPTG / X-Gal Stammlösung zugeben. 10 dünne Platten gießen, bei denen der Boden gerade eben bedeckt ist. Während des Gießens die Platte bewegen, so daß sich die Flüssigkeit trotz der Oberflächenspannung verteilt. Nach dem Abkühlen werden die Platten lichtgeschützt (z.B. in Aluminiumfolie eingeschlagen) und bei 4 °C gelagert. Phagen Fällung Die Phagen werden durch Zentrifugation und anschließende Fällung aus dem Kulturmedium gereinigt und angereichert. 40 ml Zellsuspension werden in ein 50 ml Schraubdeckel Plastikgefäß gegeben; bei 5000 × g, 4 °C, 10 min werden die Zellen in der Heraeus Variofuge abzentrifugiert. Achtung: Die gewünschten Phagen befinden sich im Überstand. Anmerkung: Zum Abzentrifugieren von Zellen wäre eine höhere g-Zahl besser, die Variofuge leistet jedoch nicht mehr und die Rotoren anderer Zentrifugen fassen nicht genug Proben für den gesamten Kurs. Im Labor verwenden wir 9000 × g. Der Überstand wird in ein neues Gefäß überführt und es wird 1/6 Volumen der Phagensuspension an PEG/NaCl-Lösung zugegeben. Gentechnik Kurs: Phagen Präsentation - 14 - Die Phagen fallen bei einer Inkubation bei 4°C aus; diese erfolgt über Nacht. Hierzu werden die Proben in den Kühlraum gebracht. (Anmerkung: wenn man es eilig hat und eine schlechtere Fällung in Kauf nimmt reichen zwei Stunden.) Die Phagen werden am nächsten Tag aufgearbeitet. Dienstag Materialien siehe auch erster Kurstag Zellen, Phagen, Platten und Immunoröhrchen aus erstem Kurstag Photometer, das Spektren aufzeichnen kann Einwegküvetten, Quarzküvette Wasserbad 45 °C Reagenzgläser, Ständer Brutschrank Kultur für Phagenamplifikation Kultur für Phagentiter Wischtücher Spritzflasche Kultur für die Phagenamplifikation Wie am ersten Kurstag. Die Zellen werden während der 2. Phagenfällung ausgegeben. Immunoröhrchen und Platten blocken Die Streptavidin-Lösung in den belegten Immunoröhrchen und in den Platten wird verworfen. Die belegten Immunoröhrchen (ca. 4 ml) und Mikrotiterplatten Vertiefungen (ca. 400 μl) werden randvoll mit 5 mg/ml BSA in NaHCO3-Puffer gefüllt. Das benötigte Volumen abschätzen und ausreichend Blocklösung mit der BSA Stammlösung und dem NaHCO3 Puffer in einem 50 ml Plastikschraubgefäß ansetzen. Mindestens 1 h durch Schwenken bzw. Rollen bei Raumtemperatur die Oberflächen blockieren. Die Blocklösung durch Ausgießen bzw. Abschlagen verwerfen und die Platten bzw. Röhrechen bei 4 °C verschlossen bzw. abgedeckt lagern. Die Oberflächen vor Gebrauch 3 mal mit TBST-Puffer waschen. Phagenfällung aufarbeiten Gefällte Phagensuspension 15 min bei 5000 × g, 4 °C zentrifugieren. Achtung: die gewünschten Phagen befinden sich im Zentrifugat (=Pellet). Überstand vorsichtig dekantieren und verwerfen, restliche Flüssigkeit vorsichtig mir einer Pipette abnehmen. (Anmerkung: man kann das Plastikgefäß zum Auslaufen auch auf einem Papier auf den Kopf stellen, dies kann jedoch eher zu Kontaminationen führen) Zentrifugat in 1 ml TBS aufnehmen und in ein 1.5 ml Eppi überführen. Gentechnik Kurs: Phagen Präsentation - 15 - 5 min bei 13000 ×g zentrifugieren Überstand mit den gelösten Phagen in eine neues Eppi geben. Für eine zweite Fällung 1/6 Volumen PEG/NaCl zugeben (ca. 167 μl). 20 min - 60 min auf Eis inkubieren. (Anmerkung: Eine längere Lagerung der zweiten PEG/NaCl Fällung verbessert die Ausbeute; 20 min passen jedoch besser zum Praktikum). In der Wartezeit werden die Zellen ausgegeben. 8 min bei maximaler Geschwindigkeit zentrifugieren. Überstand vorsichtig mit einer Pipette abnehmen und verwerfen. Zentrifugat in 300 μl TBS aufnehmen. 1 min bei maximaler Geschwindigkeit zentrifugieren. Überstand in ein neues Eppi geben. Die durch PEG-Fällung gereinigten Phagen auf Eis lagern. Anmerkung: Für eine längere Lagerung der gereinigten Phagen können diese aufgrund ihrer geringen Größe noch sterilfiltriert werden. Panning Runde P3 wie am ersten Kurstag Titerbestimmung Amplifikation P2 durch Absorptionsmessung Filamentöse Phagen besitzen bezogen auf die Masse ca. sechsmal mehr Protein als DNA. Das Absorptionsspektrum der Phagen ist daher durch ein breites Plateau von 260 - 280 nm gekennzeichnet mit einem flachen Maximum bei 269 nm. (Andere Phagen wie λ oder T4 besitzen etwa gleiche Massen an Protein und DNA). Nach Day und Wiseman kann die Phagenkonzentration aus der Differenz der Absorption bei 269 und 320 nm berechnet werden. ( A269 A320 ) 6 1016 Phagen / ml Verdünnungsfaktor Anzahl _ der _ Basen _ Phagengenom Gemessen wird gegen eine Referenz mit reinem TBS Puffer. Der Abzug der Absorption bei 320 nm, einer Wellenlänge bei der Proteine und DNA fast nicht absorbieren, dient der Korrektur des Streulichtes durch Phagenpartikel und partikuläre Verunreinigungen. Noch genauer kann das Streulicht durch eine lineare Extrapolation der Absorption zwischen 350 und 320 nm auf 269 nm approximiert werden. Der verwendete Phage M13kelib besitzt 7265 Basen. Zur Messung wird ein Spektrum von 350 bis 220 nm aufgenommen. Um das eingesetzte Volumen klein zu halten, wird in einer geschwärzten und gesockelten Quarzmikroküvette gemessen. Hierzu werden 10 μl der Phagenamplifikation in 290 μl TBS aufgenommen. Sollte bei der Titerbestimmung ein ungewöhnlich niedriger Wert bestimt werden, sollte der Ansatz zur Panning Runde P3 nachgebessert werden. Gentechnik Kurs: Phagen Präsentation - 16 - Phagenamplifikation P3 wie am ersten Kurstag (eine frische Kultur zur Infektion wird ausgegeben). Mittagspause Titerbestimmung Eluat P2, Amplifikation P2 und Eluat P3 durch Plattieren 9 LB X-gal/IPTG Platten im Brutschrank auf 37 °C aufwärmen. 9 sterile Reagenzgläser in einem Wasserbad auf 45 °C vorwärmen. Top-Agar (LB + 0,7% Agar) in der Mikrowelle im 4. Stock aufkochen, jeweils 3 ml in die erwärmten Reagenzgläser geben und weiterhin bei 45 °C flüssig halten. Durch serielles Verdünnen eine 1:102, 1:103 und eine 1:104 Verdünnung des Phageneluats der Runde P2 und P3 herstellen (z.B. 10 μl zu 990 μl LB Medium ergibt eine 1:102 Verdünnung, hiervon 100 μl zu 900 μl LB Medium ergibt eine 1:103 Verdünnung usw., die Pipettenspitze dabei mehrmals füllen und leeren, für jede Verdünnung eine neue Pipettenspitze verwenden, nach jeder Verdünnung gut mischen). Durch serielles Verdünnen eine 1:108, 1:109 und 1:1010 Verdünnung des Phagenamplifikats der Runde P2 herstellen (z.B. 10 μl zu 990 μl ergibt eine 1:102 Verdünnung, hiervon 10 μl zu 990 μl eine 1:104 Verdünnung, usw. ergibt 1:106 und 1:108, hiervon 100 μl zu 900 μl eine 1:109 Verdünnung. Bei seriellen Verdünnung ergeben sich größere Pipettierfehler und die Adsorption der Phagen an die Gefäßwände kann sich bemerkbar machen. Wieviel Liter würde man benötigen, wenn man 10 μl direkt 1:1010 verdünnen wollte?). Für jede Verdünnung 190 μl der Zellen der Infektionskultur bei OD 0,5 in ein Eppi geben. 10 μl der jeweiligen Phagenverdünnung zu den vorbereiteten Zellen geben, mischen und 5 min für die Infektion bei Raumtemperatur stehen lassen. Top-Agar aus dem Wasserbad nehmen, kurz auf ca. 42 °C abkühlen lassen, die infizierten Zellen zugeben, durch mehrmaliges Schwenken gut durchmischen, auf die warmen LB-Platten gießen und durch Schwenken und Kippen gleichmäßig verteilen. Nach dem Härten die Platten über Nacht im Dunkeln bei 37 °C bebrüten. Anmerkung: vor dem Bebrüten können die Platten einige Zeit im Dunkeln bei Raumtemperatur gelagert werden. ( Die Platten können auch ohne Lichtschutz gehandhabt werden, die Farbintensität nimmt jedoch im Laufe der Zeit erheblich ab. Um den Luftzutritt zu den Platten zu erhalten, dürfen die Platten nicht eingewickelt, sondern nur abgedeckt werden) Gentechnik Kurs: Phagen Präsentation - 17 - Mittwoch Materialien Zahnstocher DNAprep-Säulchen Heizblock 50°C Aufarbeitung der Phagenfällung wie bereits beschrieben Titerbestimmung Amplifikation P3 wie bereits beschrieben durch Absortionsmessung Präparation Einzelsträngiger DNA (wird zuert angesetzt) Jeder Kursteilnehmer pickt mit Hilfe eines Zahnstochers einen Klon der Titerbestimmung seiner 3. panning Runde und infiziert eine 4 ml Kultur von ER2738 in der frühen log-Phase. Danach wachsen die Zellen ca. 4 h bei 37 °C. Die DNA Präparation erfolgt mit Hilfe einer Silicamembran-Chromatographie. Im Folgenden ist die Anleitung des Herstellers für die Verwendung dieses “Kits” wiedergegeben. Während des Zellwachstums werden die anderen Versuche des Tages angesetzt. QIAprep Spin M13 Protocol, QIAprep M13 Handbook 02/99 This protocol is designed for preparation of single-stranded M13 DNA from 1– 3 ml E. coli culture grown in 2x YT or LB medium, using spin columns in a microcentrifuge. Up to 3 μg of ssDNA can be expected per 1 ml phage supernatant depending on the particular phage clone. E. coli strains used for infection must contain the F’episome that drives pilus biosynthesis (e.g., JM101, JM109, TG1). 1. Grow an infected M13 culture. Cultivation of M13 infected cultures should be performed at 37°C with constant agitation. Do not grow cultures infected with recombinant M13 bacteriophages for longer than 5–6 hours. Longer growth results in selection of deletion mutants and contamination of cultures with M13 RF, chromosomal DNA, and nucleases from lysed cells. For general information about M13 propagation, refer to the Appendix on page 28 or to molecular biology manuals such as “Current Protocols in Molecular Biology”, Ausubel, F.M. et al., eds (1991), Chapter 1.14. Mittagspause 2. Spin down bacterial cells at 5000 rpm for 15 min at room temperature. 3. Transfer supernatant containing M13 bacteriophage to a fresh reaction tube. Be careful not to disturb the bacterial pellet. Any carryover of bacterial cells will result in contamination of the M13 precipitation with bacterial chromosomal DNA or double-stranded bacteriophage RF DNA. 4. Optional: Repeat the centrifugation step. A second centrifugation step may be necessary to ensure a clean supernatant fraction, and no bacterial cell carryover. Gentechnik Kurs: Phagen Präsentation - 18 - 5. Add 1/100 volume of Buffer MP for M13 precipitation. (i.e., 10 μl per 1 ml of phage supernatant.) Mix by vortexing and incubate at room temperature for at least 2 min. During this step, bacteriophage particles are precipitated from the culture medium. 6. Place a QIAprep spin column in a 2-ml microcentrifuge tube and apply 0.7 ml of the sample to the QIAprep spin column. The bacteriophage supernatant must be loaded in successive 0.7 ml fractions due to the capacity of the QIAprep spin column. 7. Centrifuge for 15 sec at 8000 rpm and discard flow-through from collection tube. During this step intact bacteriophage are retained on the QIAprep silica-gel membrane. 8. Repeat the loading and centrifugation (steps 6 and 7) until all of the sample has been loaded onto the QIAprep spin column. 9. Add 0.7 ml Buffer MLB, for M13 lysis and binding, to the QIAprep spin column and centrifuge for 15 sec at 8000 rpm. This step creates appropriate conditions for binding of the M13 DNA to the QIAprep silica-gel membrane. Bacteriophage lysis begins. 10. Add another 0.7 ml Buffer MLB to the QIAprep spin column, and incubate for 1 min at room temperature to lyse bacteriophages completely. Centrifuge for 15 sec at 8000 rpm. M13 singlestranded DNA is released from bacteriophage particles and adsorbed to the QIAprep silica-gel membrane. 11. Add 0.7 ml Wash Buffer PE and centrifuge for 15 sec at 8000 rpm. Residual salt is removed in this step. 12. Discard Wash Buffer PE from collection tube. Centrifuge QIAprep spin column for 15 sec at 8000 rpm to remove residual Buffer PE. It is important to dry the QIAprep membrane with a quick microcentrifugation step. This prevents residual ethanol from being carried over into subsequent reactions. 13. Place QIAprep spin column in a clean 1.5-ml microcentrifuge tube. To elute DNA add 100 μl of Buffer EB (10 mM Tris·Cl, pH 8.5) to the center of the column membrane, incubate for 10 min, and centrifuge for 30 sec at 8000 rpm. Incubation of elution buffer in the QIAprep spin column significantly increases the recovery of single-stranded M13 DNA molecules, which adsorb tightly to the silica membrane. If yields are low or variable, recovery may be enhanced by preheating the elution buffer to 50°C. The DNA can also be eluted with H2O. When using water for elution, make sure that the pH is in the range 7.0–8.5. Elution efficiency is dependent on pH and the maximum elution efficiency is achieved within this range. Konzentrationsbestimmung der DNA 20 μl der DNA-Lösung werden mit 280 μl EB-Puffer gemischt und das Absorptionsspektrum von 340 bis 220 nm aufgenommen. Als Referenz wird das Spektrum von reinem EB-Puffer verwendet bzw. abgezogen. Die DNA Konzentration wird aus der Absorption bei 260 nm abzüglich des Streulichtes bei 320 nm berechnet. Kursteil 2: gerichtete Evolution (siehe Skript) Gentechnik Kurs: Phagen Präsentation - 19 - Donnerstag Materialien Ultrazentrifuge, Schweißgerät, Waage, Pinzette, Bechergläser CsCl Lsg 0,4 g/ml Quickseal Röhrchen, Kanülen, 5ml Spritzen Stativ, Hintergrund, Taschenlampe Photometer Aliquotiertes ABTS ELISA Reader Phagenreinigung in einem Cäsium Chlorid Gradienten und Ultrazentrifugation Etwa 80% des Volumens der PEG-Fällung der 3. Runde (Achtung: es werden noch Phagen für den Phagen ELISA benötigt) werden zu 5 ml einer CsCl/TBS-Lösung gegeben, die 2 g CsCl enthält. Durch mehrmaliges Umdrehen des Plastikgefäßes wird gemischt. Mit Hilfe einer Spritze und Kanüle werden 5 ml in ein beschriftetes 13 × 38 mm Polyallomer Quick-Seal Zentrifugenröhrchen geben. Nach dem Einfüllen durch Absaugen sicherstellen, daß der Hals des Röhrchens trocken ist. An einer elektronischen Waage werden die Röhrchen mit Hilfe der Platzhalter und Deckel paarweise auf 10 mg genau tariert. Mit einem speziellen Schweißgerät werden die Röhrchen verschlossen und durch Drücken auf Dichtigkeit geprüft. Die Zentrifugation erfolgt in einer Ultrazentrifuge bei 60 000 rpm (ca. 400 000 × g) für 4 h in einem Beckman VTi 65.2 Rotor, die Beschleunigung wird “schnell” und die Bremse auf “langsam” eingestellt. Anmerkung: Die Zentrifuge darf nur nach Einweisung bedient werden. Bei dem Rotor handelt es sich um einen Vertikalrotor bei dem sich die Schichten im Gleichgewicht vertikal ausbilden. Während des langsamen Abbremsens (15 bis 20 min) lagern sich die Schichten in eine horizontale Lage um. Ein Vertikalrotor wird verwendet, da sich bei diesem aufgrund der kurzen Strecke der Dichtegradient schnell ausbildet. Nach Beendigung der Zentrifugation die Röhrchen sehr vorsichtig herausnehmen um eine Durchmischung der Schichten zu vermeiden. Gewinnung der Phagen: Das Zentrifugenröhrchen wird mit einer Stativklemme an einem Stativ befestigt und vor einem schwarzen Hintergrund von oben mit einer ebenfalls am Stativ befestigten Taschenlampe durchleuchtet. Aufgrund des Streulichts erscheint die Phagenbande milchig weiß. Zur Belüftung des Röhrchens eine Kanüle an der Spitze des Röhrchens einstechen. Eine zweite Kanüle unterhalb der Phagenbande einstechen und nach schräg oben führen bis die Öffnung an der Phagenbande anliegt. Bei der Verwendung der Kanülen nicht auf die eigene Hand zielen!! Die Phagenbande von etwa 400 bis 500 μl vorsichtig absaugen. Anmerkung: Üblicherweise wird das CsCl durch Verdünnung (z.B. 2,5 ml TBS) und nochmalige pelletierende Zentrifugation (z.B. 50 000 rpm / 135 000 × g) oder Dialyse entfernt. Von den gereinigten Phagen wird ein Spektrum gegen CsCl/TBS-Puffer aufgenommen. Gentechnik Kurs: Phagen Präsentation - 20 - Phagen ELISA Die belegte und geblockte Mikrotiter-Platte wird 5 x mit TBST gewaschen. Für den Phagen ELISA werden die Phagenamplifikate eingesetzt. Für jede Panning-Runde werden drei serielle Verdünnungen angesetzt, wobei die erste und zweite Verdünnung als Doppelbestimmung durchgeführt werden. Die Vertiefungen werden mit jeweils 75 μl gefüllt. Die Phagenkonzentration sollte dabei anhand der Absorptionsspektren so gut wie möglich bestimmt werden. Bei der geringsten Verdünnung, also der höchsten Phagenkonzentration, sollen 5×1012 Phagen in jede Vertiefung pipettiert werden. Hierzu werden die Phagen mit TBST / 3 mg/ml BSA verdünnt. Bei sehr geringen Ausbeuten werden die Phagen unverdünnt eingesetzt. Die Phagen der Runde P0 werden mit Konzentrationsangabe ausgegeben. Das folgende Pipettierschema sollte angewendet werden: Zeile Spalte 3 P0 4 P2 5 P3 6 B P0 5*10^12 P2 5*10^12 P3 5*10^12 2. Ak C P0 5*10^12 P2 5*10^12 P3 5*10^12 1.+ 2. Ak D P0 5*10^11 P2 5*10^11 P3 5*10^11 nur ABTS E P0 5*10^11 P2 5*10^11 P3 5*10^11 F P0 5*10^10 P2 5*10^10 P3 5*10^10 Die Phagen werden 45 - 60 min bei RT auf einer Plattform geschüttelt. Die Platten werden abgeschlagen und 5 mal mit TBST gewaschen. Der Flüssigabfall wird in ein Becherglas gegeben. Jeweils 75 μl eines murinen monoklonalen anti-M13genVIII Antikörpers, der mit Peroxidase gekoppelt ist, werden in einer Verdünnung von etwa 1:3000 zugesetzt. Anmerkung: Häufig verwendet man einen primären monoklonalen Antikörper und einen generell einsetzbaren sekundären Antikörper der mit einem Enzym gekoppelt ist. Dies ist in der Regel sensitiver und billiger, erfordert jedoch einen weiteren Arbeitsschritt und das Signal ist weniger linear zur Antigendichte. 45 - 60 min schütteln. 5 mal waschen. Jeweils 75 μl einer ABTS Substratlösung zusetzen, die aliquotiert ausgegeben wird (1 g/l ABTS, 3.25 mM Na-Perborat, 40 mM Zitronensäure, 60 mM Di-Na-Hydrogenphosphat, pH 4,5). Nach ca. 20 min Farbveränderung beobachten und qualitativ festhalten. Die Platten werden genauer mit einem Mikrotiterplattenlesegerät bei 405 nm ausgewertet (im Labor 4. OG Ost). Freitag Kursteil 2 Vorträge Doktoranden Gentechnik Kurs: Phagen Präsentation - 21 - Montag (zweite Woche) Materialien Glasplatte (80x100 mm), weiße Porzellanplatte mit Ohren (80x100 mm) Zwei Abstandshalter, Kamm (Dicke muss zusammenpassen) Klebeband, Klammern zum Abdichten 10 ml Spritze mit Kanüle (zum Ausspülen der Taschen) Vertikal Gelelektrophorese Apparaturen Stromnetzgeräte, Anschlusskabel Heizblock 95°C; Eis 70% EtOH, 20% Isopropanol 2 Bechergläser zum Pipettieren Glaspipetten, Pipettierhilfe Handschuhe, Schutzbrille Je zwei Gruppen stellen zusammen ein SDS Gel her und lassen es gemeinsam laufen. Jeder Teilnehmer trägt seine Phagenprobe (Gereinigtes Amplifikat P2 oder P3) auf wertet diese aus. SDS Polyacrylamid Gelelektrophorese (SDS PAGE), Gel gießen Glasplatte, Porzellanplatte, Abstandshalter und Kamm mit 70% Ethanol reinigen und zum Trockenen auf eine saubere Unterlage legen. Gelkammer zusammenstapeln: Glasplatte unten, im Querformat links und rechts Abstandshalter an der Kante anlegen, Porzellanplatte auflegen. Gelkammer abkleben: vorsichtig an der Tischkante der mit der Unterseite (ohne Ohren) überstehen lassen und mit sanftem Druck zusammenhalten, Klebebeand mittig auf den Rand setzen und an beiden Seiten straff umfalzen und faltenfrei auf die Platten kleben, an den Enden Klebeband knapp überstehen lassen und zusammenkleben; Gelkammer an Tischkante drehen und an den beiden Seiten ebenso verfahren, jedoch die Oberseite frei lassen. Gelkammer mit je 2 Klammern an den geklebten Seiten zusammenhalten. Trenngellösung im Becherglas zusammenpipettieren: Lösung Trenngel Sammelgel H2O 4,8 ml 6,8 ml Acrylamid/Bisacrylamid 6,3 ml 1,7 ml 1,5 M Tris/HCl pH 8,8 3,5 ml 1 M Tris/HCl pH 6,8 1,3 ml 20% (w/v) SDS 75 µl 50 µl (Sodium dodecyl sulfate) 10% (w/v) APS 150 µl 100 µl (Ammonium Peroxydisulfate) TEMED 10 µl 10 µl Gentechnik Kurs: Phagen Präsentation - 22 - Mit Pipettenspitze durch Rühren blasenfrei mischen. Kammer mit der Aussparung nach oben leicht schräg halten und Trenngellösung mit dem Becherglas Blasenfrei hineingießen bis zu einer Höhe von ca. 5 cm. Trenngellösung vorsichtig mit 500 µl Wasser gesättigtem Isopropanol überschichten. Auf Papiertuch abstellen und waagrecht justieren. (Wenn das Gel ausläuft, Prozedur von vorne wiederholen) 20 - 30 min polymerisieren lassen. Isopropanol durch Strürzen auf Papiertuch entfernen. Sammelgellösung pipettieren (siehe oben) und auf das Trenngel gießen. Über einem Papiertuch Handschuhe tragend Kamm zwischen die Glasplatten in das Trenngel schieben. Gel 20 – 30 min polymerisieren lassen. Klebeband entfernen. Kamm langsam nach oben abziehen und Taschen mit Laufpuffer mit Hilfe von Spritze und Kanüle vorsichtig ausspülen. Gel in Gelkammer mit Klammern einspannen und Laufpuffer in obere und untere Kammer füllen, so dass der Puffer über den Taschen steht. Probe vorbereiten und Elektrophorese starten 11 2×10 Phagen mit dem 5-fachen Volumen an Protein-Auftragspuffer (Ladepuffer) 250 mM Tris/HCl pH 8,0, 500 mM DTT, 50% (v/v) Glycerin, 10% SDS (w/v) 0.5% (w/v) Bromphenolblau Na-Salz, mischen. 10 min bei 95°C erhitzen und dann 1 min bei max Geschwindigkeit abzentrifugieren. Mit 20 µl Pipette ca. je 15 µl Proben und Molekulargewichtsmaerker vorsichtig in Tasche auftragen. Proben am besten links und rechts des Markers assymetrisch auftragen, z.B. ein und zwei Taschen entfernt. Apperatur mit richtiger Polung an Stromversorgung anschließen (SDS ist negativ geladen und läuft zum Plus-Pol/Anode) und Gel spannungskontrolliert bei 120 V laufen lassen. Der Lauf ist beendet, wenn der Farbmarker den unteren Rand des Gels erreicht. Gel Färben und Auswerten Stromversorgung abklemmen und Gel aus der Kammer nehmen. Platten mit Hilfe der Abstandshalter langsam und vorsichtig trennen, so dass das Gel auf einer Platte liegt. Gel vorsichtig in eine Dose fallen lassen und mit 0,05% Coomassie R-250 Lösung ca. 30 min unter Schwenken auf dem Schüttler färben. Färbelösung in Sammelgefäß geben und Gel mit 20% Essigsäure unter Schwenken entfärben, bis Banden gut zu erkennen sind. Ggf. Entfärbelösung 1- 2 mal ersetzen. Laufstecke der Banden mit Lineal ausmessen und auf eine Eichgerade des Markers in eine x,y Grafik mit logarithmischer Molekulargewichtsachse eintragen. Aufgrund der Molekulargewichtsbestimmung sollen die Banden den bekannten Phagenproteinen zugeordnet werden. Gentechnik Kurs: Phagen Präsentation - 23 - Dienstag (zweite Woche) Materialien Sequenzier Pre-Mix PCR-Maschine Sequenziergerät / Sequenzierservice Sequenzierreaktion Protokoll für Applied Biosystems Gerät Reaktionsansatz: Für die Sequenzierreaktion werden die folgenden Reagenzien in ein dünnwandiges 500 μl PCR Gefäß pipettiert. Premix+Primer 2.0 μl (wird im Eppi ausgegeben) ssDNA-Template ca. 0.1 μg (typisch 2-3 μl, die DNA-Konzentration wurde durch Absorptionsmessung bestimmt) H2O auf 10 μl Anmerkungen: Das “Premix” enthält Taq Polymerase, Puffer, dNTP und fluoreszenzmarkierte ddNTP in jeweils 4 unterschiedlichen Fluoreszenzfarben. Es werden 10 pmol Primer mit dem “Premix” ausgegeben. Größere DNA Mengen liefern in der Regel stärkere Signale. Thermocycler Protokoll ( Eppendorf Mastercycler PCR Maschine): Bei diesem Schritt erfolgt die eigentliche Sequenzierreaktion. Gerätegrundeinstellung: Standardgeschwindigkeit 3 °C/s (gilt, wenn nicht im Programm anders definiert) 500 μl Gefäß (wird vor dem Start des Programms abgefragt) 10 μl Füllvolumen (wird vor dem Start des Programms abgefragt) 105 °C geheizter Deckel Auto-Abkühlung des Deckels nach Ende des Programs Programm: Schritt 1) 96 °C bis Eingabe (dient dem Aufheizen der Maschine und des Deckels; hier erfolgt die Zugabe der vorbereiteten Probengefäße und Bestätigung der Fortführung des Programms) Schritt 2) 96 °C für 30 s (dient der Denaturierung der DNA) Schritt 3) 50 °C für 15 s (diese Temperatur soll knapp unterhalb der Schmelztemperatur des Primers liegen) Schritt 4) 60 °C für 4 min (bei diesem Schritt werden die Primer verlängert bis zu einem Abbruch mit ddNTP) Schritt 5) Wiederholung ab einschließlich Schritt 2) 14 mal Schritt 5) 4 °C halten, bis Eingabe Schritt 6) Ende Nach Beendigung des Programms kurz zentrifugieren, um die Probe am Gefäßboden zu sammeln. Hierzu wird das kleine Gefäß in ein 1.5 ml Eppdorfgefäß gestellt. Probenreinigung: Die Sequenzierreaktion enthält nicht inkorporierte fluoreszenzmarkierte ddNTP, die mit dem Auslesen von bis zu 40 Basenpaaren interferieren würden. Diese müssen Gentechnik Kurs: Phagen Präsentation - 24 - daher durch z.B. Größenausschlußchromatographie abgetrennt werden. Hierzu werden Zentrifugiersäulchen gepackt und eingesetzt. Das Säulenmaterial darf während der einzelnen Arbeitsschritte nicht austrocknen. Das Sephadex G50 Material für die Größenausschlußchromatographie ist mindestens 30 min vorgequollen und wird gebrauchsfertig ausgegeben, eventuell überschüssiges Wasser vor Gebrauch dekantieren. o Die Vorratsflasche wird durch zweimaliges Schwenken durchmischt. Mit einer abgeschnittenen 1 ml Spitze werden 700 μl auf eine Spin-Säule mit Fritte geben und diese in ein 2 ml Zentrifugiergefäß geben. o 2 × 2 min bei 1000 ×g zentrifugieren (es bleibt genau so viel H2O zurück wie das Säulenmaterial aufnehmen kann), das Zentrifugat verwerfen, Zentrifugiergefäß aufheben. Bei der Zentrifugation auf die Orientierung des Eppi achten. o Den Sequenzieransatz mit H2O auf 20 μl auffüllen. o Säule in ein beschriftetes 1.5 ml Reaktionsgefäß geben und den 20 μl Reaktionsansatz mittig auf das schräg liegende Säulenmaterial geben o 2 min bei 1000 × g zentrifugieren (falls weniger als 12 μl eluieren, nochmals zentrifugieren) o Das Eluat in ein 200 μl Eppi überführen und auf Eis aufbewahren. Probenauftrag: Die Proben werden gesammelt und von der Arbeitsgruppe Igloi in die Sequenziermaschiene geladen. Die Sequenzprofile werden am nächsten Tag verteilt. Bei der Abgabe der Proben wird das Sequenziergerät vorgestellt. Mittwoch (2. Woche) nur 2. Kurteil Donnerstag (2. Woche) Material CIP pool, 10 Computer mit Internetanschluß, Internet browser, swiss pdb viewer, acrobat reader, Grafikprogramm, LCD Projektor, Leinwand, Internetanschluss für Laptop Proteinstruktur Darstellung und Manipulation am Computer, DNA und Proteindatenbanken Freitag (2. Woche) Sequenzierauswertung, Klonierung am Computer