Kapillarelektrophorese DNA-Sequenzierung Kapillarelektrophorese
Werbung
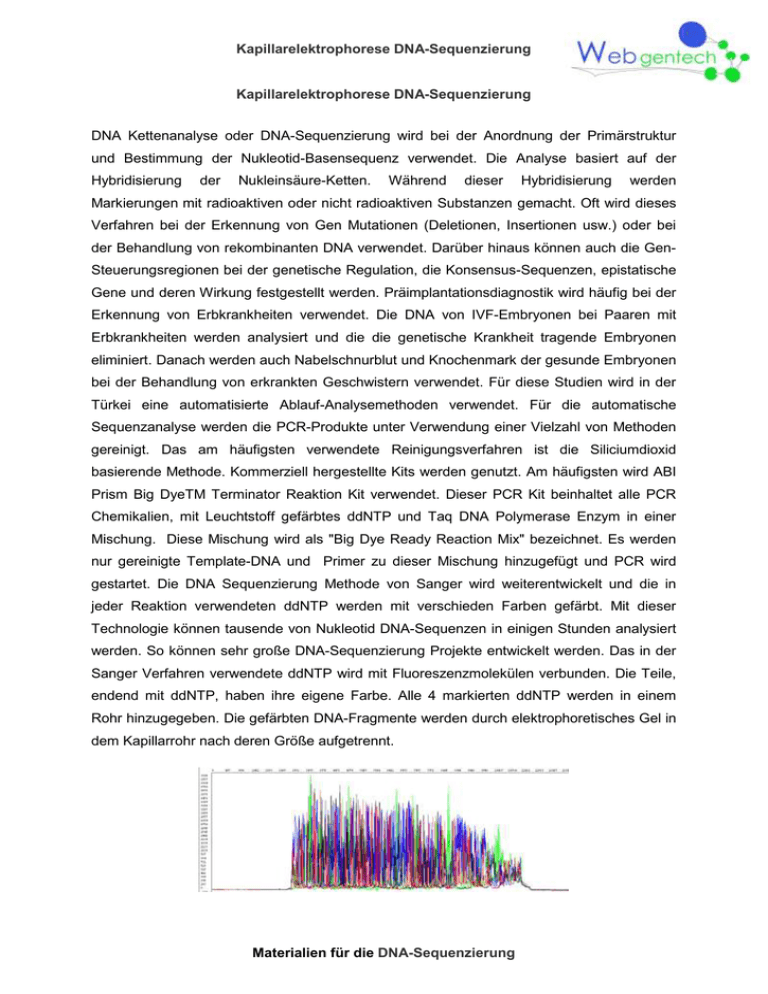
Kapillarelektrophorese DNA-Sequenzierung Kapillarelektrophorese DNA-Sequenzierung DNA Kettenanalyse oder DNA-Sequenzierung wird bei der Anordnung der Primärstruktur und Bestimmung der Nukleotid-Basensequenz verwendet. Die Analyse basiert auf der Hybridisierung der Nukleinsäure-Ketten. Während dieser Hybridisierung werden Markierungen mit radioaktiven oder nicht radioaktiven Substanzen gemacht. Oft wird dieses Verfahren bei der Erkennung von Gen Mutationen (Deletionen, Insertionen usw.) oder bei der Behandlung von rekombinanten DNA verwendet. Darüber hinaus können auch die GenSteuerungsregionen bei der genetische Regulation, die Konsensus-Sequenzen, epistatische Gene und deren Wirkung festgestellt werden. Präimplantationsdiagnostik wird häufig bei der Erkennung von Erbkrankheiten verwendet. Die DNA von IVF-Embryonen bei Paaren mit Erbkrankheiten werden analysiert und die die genetische Krankheit tragende Embryonen eliminiert. Danach werden auch Nabelschnurblut und Knochenmark der gesunde Embryonen bei der Behandlung von erkrankten Geschwistern verwendet. Für diese Studien wird in der Türkei eine automatisierte Ablauf-Analysemethoden verwendet. Für die automatische Sequenzanalyse werden die PCR-Produkte unter Verwendung einer Vielzahl von Methoden gereinigt. Das am häufigsten verwendete Reinigungsverfahren ist die Siliciumdioxid basierende Methode. Kommerziell hergestellte Kits werden genutzt. Am häufigsten wird ABI Prism Big DyeTM Terminator Reaktion Kit verwendet. Dieser PCR Kit beinhaltet alle PCR Chemikalien, mit Leuchtstoff gefärbtes ddNTP und Taq DNA Polymerase Enzym in einer Mischung. Diese Mischung wird als "Big Dye Ready Reaction Mix" bezeichnet. Es werden nur gereinigte Template-DNA und Primer zu dieser Mischung hinzugefügt und PCR wird gestartet. Die DNA Sequenzierung Methode von Sanger wird weiterentwickelt und die in jeder Reaktion verwendeten ddNTP werden mit verschieden Farben gefärbt. Mit dieser Technologie können tausende von Nukleotid DNA-Sequenzen in einigen Stunden analysiert werden. So können sehr große DNA-Sequenzierung Projekte entwickelt werden. Das in der Sanger Verfahren verwendete ddNTP wird mit Fluoreszenzmolekülen verbunden. Die Teile, endend mit ddNTP, haben ihre eigene Farbe. Alle 4 markierten ddNTP werden in einem Rohr hinzugegeben. Die gefärbten DNA-Fragmente werden durch elektrophoretisches Gel in dem Kapillarrohr nach deren Größe aufgetrennt. Materialien für die DNA-Sequenzierung Kapillarelektrophorese DNA-Sequenzierung DANN-Templat -Mix Wasser Vorwärtsprimer Rückwärtsprimer PCR-Mischung -Polymerase -SAP Purifikation Enzym Purifikation Kolonne Dye-Terminator-Mix Sephadex Laufpuffer Polymersequenz Sequenzen Platte Methode der DNA-Sequenzierung A. Vorbereitung des Genomischen DNA-Templats a. Genomische DNA wird nach Standard-Protokolle zur Isolierung hochmolekulare DNA hergestellt. Kapillarelektrophorese DNA-Sequenzierung b. Genomische DNA-Konzentration wird nach spektrophotometrische Verfahren bestimmt. c. Genomische DNA wird nach der gemessenen Konzentration mit sterilem, deionisiertem Wasser oder 10 mM Tris, pH 8,0 verdünnt. i. Für Humanes Gen soll die DNA bis zwischen 30-60 ng / ul verdünnt. d. Verdünnte DNA portioniert und bei -15 ° C bis -25 ° C aufbewahrt. 1. Vermeiden Sie mehrfachtes Enteisen und Einfrieren. e. DNA-Qualität wird durch Berechnung des Verhältnisses A260/A280 bestimmt. 1. Anmerkung: Bei der hochwertigen DNA wird das Verhältnis A260/A280 dazwischen 1,71,9 sein. B. PCR Amplifikation a. Vorbereitung der PCR-Reaktion: i. Das Reaktionsgemisch wird vorbereitet. ii. Wenn die zu erarbeitende Anzahl der Proben zur Herstellung der Reaktionsmischung hoch ist, können die gemeinsamen Chemikalien als Master-Mischung –wie PCR-Master-Mix G / C Enhancer- vorbereitet werden. Die benötigte Menge den Master-Mix wird nach der Anzahl der Proben berechnet und in den PCR Rohre gleichmäßig verteilt. b. Reaktionsmischung Kapillarelektrophorese DNA-Sequenzierung a. PCR Master Mix ............................... 12,5 µl 1. In PCR-Mastermix sind für die PCR Reaktion benötigte MgCl2, dNTPs, Taq-Polymerase, und der Puffer-Lösung zu finden. b. Forward (Vorwärts) Primer ..................... 2,5 µl 1. In dieser Studie wird speziell für genomische DNA-Bereiche konzipierter vorwärts (forward) Primer verwendet. c. Rückwärts (Reverse) Primäre ...................... 2,5 µl 1. In dieser Studie wird speziell für genomische DNA-Bereiche konzipierter Rückwärts (Reverse) Primer verwendet. d. G / C Enhancer ................................... 5 µl 1. Die Verbindung der Primer zur Genomische DNA-Sequenz wird erleichtert. e. Genomische DNA (30-60 ml) ................ 2,5 µl a. Nachdem Eingeben aller Chemikalien werden die PCR-Röhrchen kurz verwirbelt. Das Gesamtvolumen nach der Zugabe aller Chemikalien sollte 25 µl betragen. Kapillarelektrophorese DNA-Sequenzierung f. Die PCR Röhrchen werden im PCR Thermikzyklus- Gerät platziert. g. Die Ergebnisse der Aktivierung, Amplifikation und Dehnung sowie Zeiteinstellungen werden je nach Experiment gespeichert. Beispielseinträge sind wie folgt: Aktivierung: 1 Schleife 95°C ..................... 10‘ 95°C..................... 40‘' Verstärkung: 40 Zyklen 62°C....................... 1 ' 72°C....................... 50'' Dehnung: 1 Zyklus 72°C ....................... 7 ' Lagerung 4°C...........................∞ C. Bestimmung der Menge der DNA a. Nach der PCR-Reaktion wird das 5µl PCR-Produkt - um die Menge der DNA zu bestimmen - von der DNA entfernt, mit Ladepuffer vermischt und in bereits vorbereitetes 2%iges Agarosegel gegeben. DNA-Marker wird in die leer Vertiefungen/Brunnen gefüllt. Die Reaktion wird mittels Illumination UV Bildgebungsvorrichtung betrachtet. gelassenen. Kapillarelektrophorese DNA-Sequenzierung b. Reinigung von PCR-Produkten Nach der PCR Reaktion werden die PCR-Gefäße kurz zentrifugiert. I. 5 µl wird in ein anderes PCR-Röhrchen als das, in dem das PCR-Produkts enthalten ist, gegeben. II. Um die PCR-Reaktion-Rückstände zu entfernen werden 2 µl Transaktion-spezifisches Enzym zugegeben. III. Gesamtvolumen soll 7 µl betragen. IV. Die vorbereiteten Röhrchen werden in dem Thermikzyklus- Gerät platziert und je nach verwendetem Enzym die notwendigen Voraussetzungen geschaffen, um das Gerät starten zu können. Repräsentative Voraussetzungen sind wie folgt: Enzyme Inkubation: 37 ° C ..................... 30 Enzymdeaktivierung: 80 ° C..................... 15 ' Lagerung: 4 ° C ..................... ∞ Kapillarelektrophorese DNA-Sequenzierung D. Vorbereitung der Sequenzreaktion a. Die nach der PCR-Reaktion gereinigten Produkte werden für die Sequenz-Reaktion vorbereitet. Die benötigte Materialien für die Sequenz-Reaktion sind wie folgt: Farbe Terminator Mischung (Dye Terminator Miks )......................... 2 µL Sequenzpuffer ......................... 2 µL Forward (F) / Rückwärts-Primer (R) ......................... 2 µL PCR-Produkt ......................... 2 µL destilliertes Wasser ......................... 2 µL b. Das Gesamtvolumen wird 10 µL betragen. Die vorbereiteten Röhrchen werden kurz verwirbelt , zentrifugiert und in dem Thermikzyklus- Gerät platziert. Kapillarelektrophorese DNA-Sequenzierung Beispiel: Thermikzyklus Bedingungen sind wie folgt: Aktivierung: 96 ° C ..................... 1 ' Cycle: 25 Wiederholung 96 ° C ..................... 10'' 50 ° C ....................., 5'' 60 ° C ..................... 4 ' Lagerung: 4 ° C ..................... ∞ E. Purifikation der Extensor-Produkte a. Die Produkte werden unter Verwendung von Sephadex-Spalten/ Säulen purifiziert. Vorbereitung der Spalten: a. Es werden zu Sephadex-Spalten/ Säulen 750 µL ultrareinem Wasser gegeben. b. Sie Spalten/Säulen werden bis sich das Sephadex Pulver auflöst verwirbelt. c. Nachdem die Spalten 30 Minuten bei Raumtemperatur geruht haben, werden sie 2 Minuten bei 4600 prm zentrifugiert. In dem Spalte entsteht eine zylindrischen geformte Matix. d. Es wird sicherstellt dass das gesammelte Wasser in der Spalte vollständig abgelassen wurde. e. Alle Sequenzprodukte werden sorgfältig auf der Mitte der Sephadex Matrix der Sequenz mit einer Pipette platziert. f. Die Spalte/Säule wird erneut 2 Minuten bei 4600 rpm zentrifugiert. Kapillarelektrophorese DNA-Sequenzierung F. Installation des Purifizierten Produkts zum Kapillarelektrophorese Serie Analyzer a. Alle in dem Spalte-Container gesammelten Produkte werden auf einer Platte mit 96 Vertiefungen/Brunnen übertragen. b. Die Platte wird mit Septa verschlossen. c. Die Platte wird im Plattenhalter platziert d. Die mittels Kapillarelektrophorese erhalten Daten werden ausgewertet.











