Vortrage 14.
Werbung

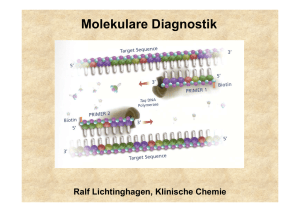
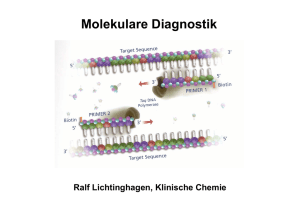
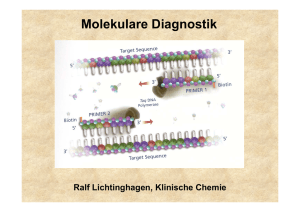
1. Molekulare Diagnostik 1.1. Methodische Grundlagen der DNA/RNA- Analytik 1.2. Anwendungen der molekularen Diagnostik: Assoziation von DNA-Polymorphismen mit Krankheiten oder Krankheitsrisiken Pharmakogenetik Tumordiagnostik auf Grundlage von molekularbiologischen Methoden 2. Humorale Tumormarker Untersuchung von DNA • • • • • • • Southern-Blot Polymerase-Kettenreaktion (PCR) RestriktionsfragmentlängenpolymorphismusPCR Allel-spezifische Oligonukleotid-Hybridisierung Sreeningmethoden für Punktmutationen Single-Strand Conformation Polymorphism PCR DNA-Sequenzierung Polymerase Chain Reaction (PCR) cycle 1 cycle 2 94°C ∼1min 94°C ∼1min 72°C ∼1min 72°C ∼1min 55°C ∼1min 55°C ∼1min 1 1 1 1 2 1 2 1 2 denaturation annealing 2 2 1 2 2 1 2 elongation DNA-Template, Primer (sense, antisense) Nukleotide, Puffer, Mg2+, thermostabile DNA-Polymerase Ermittlung eines Genotyps durch die Analyse einer bekannten Mutation •RFLP (Restriktionsfragmentlängenpolymorphismus)-PCR (Häufigkeit des Einsatzes etwa 25%) •Allel-spezifische Oligonukleotid-Hybridisierung (Häufigkeit des Einsatzes etwa 50%) Genchip-Analyse zur gleichzeitigen Erfassung vieler unterschiedlicher Mutationen kommerziell bedingt erhältlich (z.B. CYP450-Array mit 33 versch. Mutationen) RFLP-PCR Ermittlung der Punktmutation (C/T) bei Nukleotid 677 im MTHFR-Gen (Methylentetrahydrofolsäure-Reduktase) Bedeutung für den VitB12/Folsäure-Stoffwechsel (Artheroskleroserisiko über Einfluss auf Homocystein-Konzentration im Serum) HE N N N N N HO HE N M bp 300 200 100 •Amplifikation des DNA Bereiches •DNA-Fragment von 198 bp •Verdauung der DNA mit Hinf I •Wildtyp: 198 bp •Homozyg. Mutation: 175 bp •Heterozyg. Mutation: 175+198 bp Real-Time-PCR Fluoreszenz-Resonanz-Energietransfer (FRET) zwischen Hybridisierungssonden •Methode geeignet zur Quantifizierung von PCR-Produkten Messung eines zunehmenden Fluoreszenzsignals während der laufenden PCR. Beginn des Signalanstiegs ist das Maß für die Ausgangskonzentration des betreffenden DNA-Bereiches. •Methode geeignet zur Mutationsanalyse Nach der abgeschlossenen PCR wird das Schmelzverhalten der FRETSonden geprüft. Mutation und Wildtyp haben aufgrund von BasenFehlpaarungen verschiedene Schmelzpunkte. Genotypisierung mittels Schmelzkurvenanalyse Schmelzkurve 45 –75 °C WT HE HO WT HO HE Im Beispiel: FRET-Sonden haben eine 100%ige Übereinstimmung mit der Wildtyp-Sequenz Ableitung -d[Fluoreszenz]/dT Aufspüren einer bislang unbekannten Mutation •SSCP-PCR (single strand conformation polymorphism) •DNA-Sequenzierung Increasing role of environment and stochastic factors Assoziation von DNA-Polymorphismen mit Krankheiten oder Krankheitsrisiken •G6PD deficiency •Hemochromatosis •Acute intermittent porphyria •Type I diabetes mellitus • α1-Antitrypsin ZZ •Apo E-2/E-2 hyperlipoproteinämie •Phenylketonuria •Sickle-cell anemia •Cystic fibrosis •Huntington disease •Duchenne dystrophy •Tay Sachs Increasing role of genotype at other loci Automatisierte DNA-Sequenzierung Diodenlaser zur Detektion eines Fluoreszenz-Labels einer automatisierten DNA-Sequenzierung mit Online-Übertragung der Sequenzdaten Hochdurchsatzsequenzierung Basenbestimmung durch Analyse einer Serie von Bildern Source: 454 Gegenüberstellung Sanger – Next Gen Erste Generation (Sanger Sequenzierung): 100kb/Lauf, mittlere Read Länge 1000bp, Preis: 500$/Mb Zweite Generation: Roche(454): 450Mb/Lauf, 400bp, 20$/Mb Illumina: 35Gb/Lauf, 100bp, 0.50$/Mb SOLiD: 50Gb/Lauf, 50bp, 0.50$/Mb Heliscope: 37Gb/Lauf, 32bp, <0.50$/Mb Dritte Generation: PacBio SMRT: 25Gb/Lauf, >1000bp, ?$/Mb Ganz neu: Ion Torrent: 100bp/Lauf, 10x billiger als Roche(454) Häufig untersuchte Gene • Alpha-1-Antitrypsin • Angiotensin converting enzyme (ACE) • Apolipoprotein B100 • Apolipoprotein E • Faktor II-Leiden (Prothrombin) • Faktor V-Leiden • HFE (HLA-H) • HLA (Klasse I und Klasse II, zur Typisierung) • Laktase (Laktosetoleranzstörung) • MTHFR • Mukoviszidose • UDP-Glucuronosyltransferase (Gilbert-Meulengracht) Nachweis (und Quantifizierung) von RNA •Hybridisierungsverfahren (semiquantitativ) Northern-Blot •Amplifikationsverfahren (qualitatitv-quantitativ) Reverse Transkription (RT)-PCR Isolierung von RNA Synthese einer cDNA (enzymatische Erststrangsynthese mittels reverser Transkriptase) Durchführung einer (real time) PCR Methoden zur Aufdeckung monogener Erkrankungen Netzwerk aus bekannten und neuen Genen (z.B. für geistige Behinderung) Epigenomik Die Epigenomik ist ein neues Wissenschaftsgebiet aus der Fusion der Epigenetik und der Genomik, dessen Ziel es ist die genetische Regelung auf Genomebene kenenzulernen. Die Epigenetik untersucht die Chromatin-modifikationen auf der Ebene der einzelnen Gene. 1. DNA Methylierung (5-Methyl-Cytosin) 2. Histonmodifikationen: • • • • Methylierung, Acetylierung, Phosphorilierung, SUMOylierung (Small Ubiquitinrelated MOdifier‚ kleiner Ubiquitinverwandter Modifikator) P Ac Met Pharmakogenomik Untersucht, wie das genetische Profil die Reaktionen des Organismus’ auf Medikamente beeinflusst. • • • • • • Wirksamere Medikamente Bessere und sicherere Medikamente Genauere Methoden zum Feststellen der notwendigen Dosis Entwickeltere Untersuchung auf Erkrankungen Besserer Impfstoff Verbesserung in der Fortentwicklung der Medikamente und dem Genehmigungsprotokoll Senkung der durchschnittlichen Kosten der Heilkunde Viele stellen die Pharmakogenomik mit der individualisierten Medizin gleich Pharmakogenetik Enzym Häufigkeit Medikamente Probleme Cytochrom P450 CYP2D6 CYP2C19 CYP1A2 3-10% 2-5% 12% viele (Antidepressiva Neuroleptika, Beta-Blocker.....) Meist verstärkte Wirkung N-Acetyltransferase NAT2 50% Isoniazid, Sulfapyridin Dapson, Koffein, Hydralazin Überempfindlichkeitsreaktionen Thiopurinmethyltransf. TPMT 0,3% Azathioprin, Mercaptopurin Leukopenie Dihydropyrimidin-Dehydr. 5% (Het.) (DPD, DPYD) 5-Fluoruracil verstärkte Wirkung Glukose-6-Phos.-Dehydr. 1% Sulfonamide, Primaquin Hämolyse Serum-Cholinesterase 0,04% Succinylcholin Apnoe Ca-Transport im ER 0,005% Inhalationsanästhetika Maligne Hyperthermie Mutationsnachweise in der molekulargenetischen Tumordiagnostik •Keimbahnmutationen •somatische Mutationen Tumordiagnostik: Keimbahn-Mutationen (Familiäre Disposition: 2% aller Krebserkrankungen) Erkrankung Gen Fam. Retinoblastom Fam adenomatöse Polyposis (FAP) Heredit. kolorektale Karzinome o. Polyposis Fam. Brust-/Ovarialkrebs Fam. Brustkrebs Li Fraumeni Syndrom Multiple endokrine Neoplasie (MEN) Typ1 Fam. Medulläres Schilddrüsenkarzinom MEN Typ 2A und 2B Fam. Melanom Neurofibromatose Typ 1 Neurofibromatose Typ 2 Gorlin Sydrom, Basalzellkarzinom Von Hippel-Lindau Syndrom Fam. Wilms-Tumor rb apc mlh 1 brca 1 brca 2 p53 men 1 (menin) ret p16 nfl 1 nfl 2 ptch vhl wt 1 Tumordiagnostik: somatische Mutationen Genetische Instabilität in malignen Tumoren: Entweder kausal für Tumorgenese verantwortlich, und/oder für Tumorprogression verantwortlich! Wichtige Gene: p53, k-ras Mutationsanalyse zur Früherkennung von Tumoren. Tumorzellnachweise in Körperflüssigkeiten und Stuhl. Problem: Vielzahl möglicher Mutationen, Nachweis eines mutierten Allels unter einem Überschuss von Wildtyp-Allelen. Tumoren: z.B. Kolon-Karzinom, Pankreas-Karzinom Tumordiagnostik: somatische Translokationen Nachweis von Chromosomenanomalien in Tumoren hämatopoetischer Zellen leichter. Große Mehrzahl bisher gesicherter Chromosomenanomalien bei Leukämien und Lymphomen. Gen Rearrangement Erkrankung Rearrangements, kodierende Sequenz exprimiert und nicht verändert BCL2 t(14;18)(q32;q21) follikuläres Lymphom Rearrangements, Hybridgene BCR-ABL t(9;22) (q34;q11) CML, B-ALL Philadelphia-Chromosom zytogenetisch bei bis zu 90% der CML nachweisbar!!! Nachweis mittels PCR /RT-PCR mit Primern von beiden Seiten der chromosomalen Bruchstelle. Tumordiagnostik: zirkulierende Tumorzellen Nachweis residualer Tumorzellen im peripheren Blut, Knochenmark oder Blutprodukten über Marker (Transkripte, Methylierungsgrad der DNA), welche nicht in hämatopoetischen Zellen vorhanden sind. Target: meist Gene klassischer Tumormarker: z.B. AFP PSA CK19 alpha-Fetoprotein Prostataspezifisches Antigen Zytokeratin 19 Nachweis mittels RT-PCR o. quantitativer RT-PCR Nachweisgrenzen: ca. 1 Tumorzelle unter 106 Zellen Methylierungsgrad der DNA: (neuerer Trend). DNA von Tumoren ist häufig 5mC). (spez. in Promorbereichen) hypermethyliert (C Nachweis des Methylierungsgrades von ausgewählten Indikatorgenen zum sensitiven Nachweis einer Metastasenbildung. Nachweis von Tumorzellen Klinische Diagnose Langes, „unsichtbares“ Vorstadium Monate bis viele Jahre Tumormasse Tumorzellen 1µg 103 1 mg 106 1g 109 10 - 100 g 1011 Immunologische Methoden Biophysikalische Methoden Röntgen CT Ultraschall Diagnostische Schwelle früh spät 1 kg 1012 Der ideale Tumormarker ... Tumor/Tumorrezidiv-Früherkennung (hohe Sensitivität >50%) Unterscheidung - maligne vs. gesund/benigne (hohe Spezifität >95%) Gute positive und negative Prädiktion Unterscheidung distinkter Tumorentitäten Unterstützung bei der Therapieentscheidung Korrelation mit Tumormasse Prognostische Aussage Einteilung der humoralen Tumormarker Onkofetale und onkoplazentare Antigene (CEA, AFP, hCG) Mit monoklonalen Antikörpern erkennbare Kohlehydratepitope (CA 19-9, CA 125, CA 15-3) Differenzierungs-und Proliferationsantigene: (NSE, PSA, TPA, 2-Mikroglobulin) Von Tumorzellen gebildete Hormone: Calcitonin beim medullären SD-Karzinom, Insulin beim Insulinom, ektop gebildetes ACTH oder Calcitonin beim SCLC Von Tumorzellen gebildete Proteine: (monoklonales Immunglobulin oder Bence Jones-Protein bei monoklonalen Gammopathien) Von Tumorzellen gebildete Enzyme: (z.B. NSE beim SCLC, PAP beim Prostatakarzinom) TM-Konzentration abhängig von ... Tumormasse Tumorzelluläre Syntheserate Freisetzungsrate aus Tumor Iatrogene Maßnahmen (digitale Untersuchung, antineoplastische Therapie) Tumordurchblutung Leberfunktion Nierenfunktion TM-Diagnostik zur …. Kontrolle des Therapieerfolgs Verlaufskontrolle von Tumorerkrankungen Rezidiv-Früherkennung Therapeutische Konsequenzen? Lebensqualität? Diagnosesicherung: in Einzelfällen! Patientin (53 J) mit Mammakarzinom 700 Cut-off: 5 µg/L Exitus 500 Lebermetastasen 400 300 Pleuraerguss 16 Chemotherapie 12 Knochenmetastasen 8 200 4 Remission 100 0 0 2 4 6 8 Monate 10 12 14 Bilirubin (mg/dL) CEA (µg/L) 600 Wann sollten die Marker bestimmt werden? vor der ersten Therapiemaßnahme: Chirurgie, Chemotherapie, Hormontherapie, Radiotherapie nach der Therapiemaßnahme abhängig von der Höhe des Ausgangswertes und der Halbwertszeit nach etwa 2 bis 14 Tagen anfänglich alle drei Monate, später alle 6 Monate in der Verlaufskontrolle vor jedem Therapiewechsel bei Verdacht auf ein Rezidiv bei Verdacht auf Metastasierung bei neuem Staging bei deutlichem Werteanstieg: 2 – 4 Wochen später wiederholen Signifikanter Werteanstieg, wenn Anstieg um 25 – 50 % jährlich beim PSA-Screening: ab dem 50. Lebensjahr