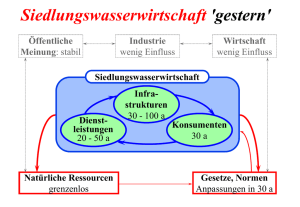
Identifizierung von Regulationsmechanismen - oparu
Werbung

Universitätsklinikum Ulm Zentrum für Chirurgie Klinik für Allgemein-, Viszeral- und Transplantationschirurgie (Ärztliche Direktorin: Prof. Dr. med. Doris Henne-Bruns) Die Casein-Kinase-1δ (CK1δ): Identifizierung von Regulationsmechanismen Dissertation zur Erlangung des Doktorgrades der Medizin der Medizinischen Fakultät der Universität Ulm vorgelegt von Sven-Jannis Randoll geboren in Heidelberg Ulm 2013 Amtierender Dekan: Prof. Dr. Thomas Wirth 1. Berichterstatter: Prof. Dr. Uwe Knippschild 2. Berichterstatter: Prof. Dr. Marion Schneider Tag der Promotion: 20.12.2013 Für Emma Inhaltsverzeichnis I Inhaltsverzeichnis Abkürzungsverzeichnis ................................................................................................... III 1 Einleitung ........................................................................................................................... 1 1.1 Signaltransduktion ....................................................................................................... 1 1.2 Die Casein-Kinase 1 Familie ....................................................................................... 2 1.2.1 Substratspezifität von CK1 und Konsensussequenz ................................................. 4 1.2.2 Regulationsmechanismen der CK1-Expression und -Aktivität ................................ 6 1.2.3 Funktionen von CK1 ................................................................................................ 8 1.2.4 CK1 und das Tumorsuppressorprotein p53 .............................................................. 8 1.2.5 Wnt-Signalweg ......................................................................................................... 9 1.2.6 Antiapoptotische Effekte von CK1......................................................................... 10 1.2.7 Beteiligung von CK1 an der Entstehung von Krankheiten .................................... 11 1.2.8 CK1-spezifische Inhibitoren ................................................................................... 12 1.3 Problemstellung ......................................................................................................... 13 2 Material und Methoden .................................................................................................... 14 2.1 Material ...................................................................................................................... 14 2.1.1 Chemikalien und Reagenzien ................................................................................. 14 2.1.2 Radiochemikalien ................................................................................................... 15 2.1.3 Größenstandards ..................................................................................................... 15 2.1.4 Primer ..................................................................................................................... 16 2.1.5 Bakterienstämme .................................................................................................... 16 2.1.6 Enzyme und Proteine .............................................................................................. 16 2.1.7 Kits und Verbrauchsmaterialien ............................................................................. 16 2.1.8 Geräte...................................................................................................................... 17 Inhaltsverzeichnis II 2.1.9 Software .................................................................................................................. 19 2.2 Methoden ................................................................................................................... 20 2.2.1 Proteinbiochemische Methoden ............................................................................. 20 2.2.2 Bakterien ................................................................................................................. 26 2.2.3 DNA ....................................................................................................................... 29 2.2.4 Agarosegelelektrophorese und Herstellung von Agarosegelen .............................. 32 2.2.5 Statistische Methoden ............................................................................................. 34 3 Ergebnisse......................................................................................................................... 35 3.1 Beeinflussung der Aktivität von CK1 durch Phosphorylierung .............................. 35 3.1.1 Phosphorylierung des C-Terminus von CK1 durch Clk2 ..................................... 36 3.1.2 Chk2 phosphoryliert CK1 in der C-terminalen Domäne ...................................... 37 3.1.3 Phosphorylierung des C-Terminus von CK1 durch Chk1 .................................... 38 3.2 Zweidimensionale Phosphopeptidanalyse von GST-CK1 - Fusionsproteinen nach Phosphorylierung durch Chk1 in vitro ............................................................................ 39 3.3 Generierung einer GST-CK1 T397A-Mutante............................................................ 44 3.4 Charakterisierung der in vitro Kinaseaktivität von GST- CK1 T397A....................... 44 4 Diskussion ........................................................................................................................ 47 5 Zusammenfassung ............................................................................................................ 54 6 Literaturverzeichnis .......................................................................................................... 55 7 Abbildungsverzeichnis ..................................................................................................... 66 8 Tabellenverzeichnis .......................................................................................................... 68 Anhang ................................................................................................................................ 69 Danksagung ......................................................................................................................... 70 Lebenslauf ........................................................................................................................... 71 III Abkürzungsverzeichnis Abkürzungsverzeichnis α alpha A Alanin Abb. Abbildung Akt andere Bezeichnung für Proteinkinase B APS Ammoniumpersulfat AS Aminosäure ATP Adenosintriphosphat ATM Ataxia-telangiectasia mutated AutoID autoinhibitorische Domäne β beta BCA 2,2'-Bichinolon-4,4'-dicarbonsäure Bid BH3 Domäne - only death agonist protein bp Basenpaare BSA Rinderserumalbumin bzw. beziehungsweise ϒ gamma °C Grad Celsius C- Carboxy ca. circa CAMK Calcium/Calmodulin abhängige Proteinkinase cAMP zyklisches Adenosinmonophosphat Cdc cell division cycle IV Abkürzungsverzeichnis Cdk Cyclin-abhängige Kinase cDNA komplementäre DNA Chk Checkpoint-Kinase Ci Curie CK Casein Kinase CLK2 CDC-ähnliche Kinase 2 cm Zentimeter cpm Impulse (counts) pro Minute δ delta DC Dünnschichtchromatographie dH2O destilliertes Wasser ddH2O doppelt destilliertes Wasser d.h. das heißt DNA Desoxyribonukleinsäure DNA-PK DNA-dependent protein kinase DNP Dinitrophenole dNTP Desoxynukleotidtriphosphat DTT Dithiothreitol ε epsilon EDTA Ethylendiamintetraessigsäure EGTA Ethylenglykol-bis2(2- aminoetyhlether)-tetraessigsäure ePK eukaryontische Proteinkinase V Abkürzungsverzeichnis ER Endoplasmatisches Retikulum Erk extracellular signal-regulated kinases g Gramm Gal-3 Galectin 3 GSK3 Glykogen Synthase Kinase 3 GST Glutathion-S-transferase GTP Guanosintriphosphat H2O Wasser H2O2 Wasserstoffperoxid h Stunde His Histidin IC50 mittlere inhibitorische Konzentration IPTG Isopropyl-β-D-thiogalaktopyranosid k Kilo kb Kilobasenpaare kDa Kilodalton KHD Kinesin homologe Domäne Km Michaeliskonstante (Maß für die Enzymaffinität) l Liter LB Luria-Bertani-Medium LC-MS Liquid-Chromatographie-Massenspektometrie LRP6 low density lipoprotein receptor-related protein 6 VI Abkürzungsverzeichnis μ mikro m milli m Meter M molar (mol/l) MBP myelin basic protein Mdm murin double minute-Gen min Minute mPER Mammalian period circadian protein mRNA messenger Ribonukleinsäure mut Mutante n nano N Normalität N- Amino NF-AT nuclear factor of activated T-cells NLS Kerntransportsignal NP40 Nonidet P40 OD optische Dichte 32 radioaktive Phosphorisotop P Phosphat, Phosphorylierungsstelle p38 MAPK p38-mitogenaktivierte Proteinkinasen PAA Phosphoaminosäurebestimmung PAGE Polyacrylamid-Gelelektrophorese P VII Abkürzungsverzeichnis PBS Phosphat-gepufferte Salzlösung PCR Polymerase-Ketten-Reaktion pH negativer dekadischer Logarithmus der Konzentration der Wasserstoffionen PIP2 Phosphatidylinositol 4,5-bisphosphat PK Proteinkinase PP2A Proteinphosphatase 2A PVP Polyvinylpyrrolidon RhoA Ras homolog gene family, member A RNA Ribonukleinsäure RNase Ribonuklease rpm Umdrehungen pro Minute RT Raumtemperatur RXR retinoid X receptor S Serin SCS Sulfatid und Cholesterol-3-Sulfate sek, s Sekunde SDS Natriumdodecylsulfat STE Kinasen mit Ähnlichkeit zu SterilKinasen aus Hefe T Threonin TAE Tris-Acetat-EDTA-Puffer VIII Abkürzungsverzeichnis TBS Tris-gepufferte Salzlösung TEMED N,N,N‘,N‘-Tetramethylethylendiamin TK Tyrosinkinase TKL Tyrosinkinasen-ähnliche Kinasen TRAIL TNF-related apoptosis-inducing ligand Tris Tris (hydroxymethyl)-aminomethan TTBK Tau-Tubulin-Kinase U unit u.a. unter anderem UV Ultraviolett V Volt v Geschwindigkeit Vmax maximale Reaktionsgeschwindigkeit VRK Vaccinia related kinase Wnt wingless wt Wildtyp X beliebige Aminosäure 1 Einleitung 1 1 Einleitung 1.1 Signaltransduktion Über den Mechanismus der zellulären Signaltransduktion werden die Funktionen von Effektormolekülen an die vorherrschenden äußeren Bedingungen und Erfordernisse angepasst. Als initiierender Faktor dient hierbei entweder eine Änderung des Membranpotenzials oder die Bindung eines Neurotransmitters, Hormons oder eines anderen Signalstoffes an Zellrezeptoren. Die dadurch in Gang gesetzten Signalkaskaden regulieren die Funktion des jeweiligen Effektormoleküls (z.B. Ionenkanäle, Enzyme) (Hunter 1995; Cohen 2002). Die Kommunikation der Zellen untereinander kann juxtakrin (über direkten Kontakt), parakrin (über kurze Entfernungen) und endokrin (durch Botenstoffe) erfolgen (Sporn und Todaro 1980; Bosenberg und Massague 1993). In dem komplexen System der Signaltransduktion spielen viele verschiedene Komponenten eine bedeutende Rolle. In besonderem Maße sind dies Proteine, wie Zelloberflächenrezeptoren, intrazelluläre Rezeptoren, Proteinkinasen und -phosphatasen, GTP - bindende Proteine und eine Vielzahl intrazellulärer Proteine, welche mit diesen Komponenten in Wechselwirkung stehen. Häufig sind die in den Zielzellen aktivierten Signalübertragungskaskaden einander sehr ähnlich, wenn nicht sogar identisch. Die Bindung eines Botenstoffes an ein spezifisches Rezeptorprotein der Zielzelle löst eine Kaskade molekularer Veränderungen aus und bewirkt so schließlich eine spezifische Antwort in der Zielzelle (Hunter 1995; Cohen 2002). In den letzten Jahrzehnten konnte eine Vielzahl von Signaltransduktionswegen, sowie die daran beteiligten Signalkomponenten identifiziert werden. Um eine ungestörte Funktion des komplexen Signalnetzwerks zu gewährleisten, müssen Regulationsmechanismen bestehen, welche in die Signaltransduktionskaskaden über deren Komponenten rasch eingreifen können. Von großer Bedeutung sind in diesem Kontext die Proteinkinasen und Proteinphosphatasen, welche die Fähigkeit besitzen, Proteine durch reversible Phosphorylierung bzw. durch Dephosphorylierung kurzfristig in ihrer Aktivität, Stabilität, Interaktionsfähigkeit und subzellulären Lokalisation zu verändern, wodurch eine äußerst schnelle intrazelluläre Signaltransduktion ermöglicht wird (Hunter 1995; Cohen 2002). 1 Einleitung 2 Proteinkinasen sind in vielen wichtigen zellulären Prozessen von großer Bedeutung und stellen mit 518 Mitgliedern eine der größten eukaryontischen Genfamilien dar (Hunter et al. 1997; Rubin et al. 2000; Lander et al. 2001). Hiervon werden 478 Kinasen zur Gruppe der typischen eukaryotischen Proteinkinasen (ePK) zusammengefasst und sind durch eine stark konservierte katalytische Domäne charakterisiert, welche 250-300 Aminosäuren umfasst. Davon abzugrenzen sind 40 atypische Proteinkinasen (aPK), welche zwar eine enzymatische Aktivität besitzen, jedoch keine konservierte katalytische Domäne aufweisen (Manning et al. 2002). Bei den meisten der eukaryontischen Proteinkinasen konnte eine konservierte katalytische Domäne nachgewiesen werden. Diese Proteinkinasen lassen sich in sieben Hauptgruppen untergliedern (AGC: beinhaltet die PKA-, PKG-, PKC-Familien; CAMK: Calcium/Calmodulin-abhängige Proteinkinase, CMGC: schließt die CDK-, MAPK-, GSK3-, CLK-Familien ein, CK1: Casein-Kinase 1, STE: Kinasen mit Ähnlichkeit zu Steril-7-, Steril-11-, Steril-20-Kinasen aus Hefe, TKL: Tyrosinkinasen-ähnliche Kinasen („Tyrosin kinase like“), TK: Tyrosinkinase). Diese Hauptgruppen können in zusätzliche Familien und Unterfamilien gegliedert werden (Hanks et al. 1995). 1.2 Die Casein-Kinase 1 Familie Die in allen eukaryotischen Zellen, von Hefen bis zum Menschen, vorkommenden Mitglieder der Casein-Kinase-1 (CK1)-Familie sind evolutionär hoch konserviert (Gross und Anderson 1998; Knippschild 2005). In Säugetieren sind sieben verschiedene CK1-Isoformen bekannt, deren Hauptunterschied in der Länge und der Primärstruktur der regulatorischen, nicht-katalytischen N- und Cterminalen Domänen besteht. Bei den sieben Isoformen handelt es sich um: α, β, γ1, γ2, γ3, δ, ε (Rowles et al. 1991; Fish et al. 1995; Graves und Roach 1995; Zhai et al. 1995; Wang et al. 1996; Gross und Anderson 1998). Für CK1α, CK1γ2, CK1γ3, CK1 und CK1ε wurden mehrere Splicevarianten beschrieben (Rowles et al. 1991; Tapia et al. 1994; Wang et al. 1996; Zhang et al. 1996; Green und Bennett 1998; Ma et al. 2004; Takano et al. 2004), welche durch alternatives Spleißen entstehen. Allen CK1-Isoformen ist gemein, dass sie Second-messenger-unabhängige, monomere Serin/Threonin-spezifische Proteinkinasen sind, die das Phosphat von ATP (oder seltener von GTP) übertragen (Flotow et al. 1990; Tuazon et al. 1991) und bevorzugt Casein als in vitro Substrat verwenden. Zwischen CK1 und CK1 besteht mit einer Homologie von 98% in der 1 Einleitung 3 katalytischen Domäne die engste Verwandtschaft (Graves et al. 1993; Zhai et al. 1995; Gross und Anderson 1998). Alle CK1-Isoformen gleichen sich darin, dass sie eine zentral liegende, in ihrer Aminosäuresequenz hoch konservierte Kinasedomäne enthalten, welche von variablen N- und C-terminalen Abschnitten eingerahmt wird. Der N-Terminus besteht aus 9-76 Aminosäuren, der C-Terminus kann jedoch zwischen 24 und 200 Aminosäuren lang sein. Das Molekulargewicht reicht von 37 kDa bei CK1α bis hin zu 51 kDa bei CK1γ3. Die Kinasedomäne stellt allerdings mit 293 Aminosäuren den größten Teil des Proteins dar. Diese enthält nicht nur die ATP-Bindungsstelle, welche für den Phosphattransfer wichtig ist, sondern auch ein nukleäres Lokalisationssignal (NLS). Für einen Transport in den Zellkern sind jedoch nach neusten Erkenntnissen weitere NLS notwendig. Ein zusätzliches NLS im L-Exon mancher CK1a-Spleißvarianten ermöglicht den Transport in den Zellkern (Fu et al. 2001). Ebenfalls gibt es Hinweise darauf, dass im Fall von CK1 die Kinaseaktivität für deren subzelluläre Lokalisation notwendig ist (Milne et al. 2001). A B N - Terminus Kinase - Domäne CK1a C - Terminus CK1g1 CK1g2 CK1g3 CK1 CK1 konservierte Aminosäuren Abbildung 1: Evolutionsbedingte Verwandtschaft der Casein Kinase 1 (CK1)-Isoformen von Hefen zu Menschen und deren Verwandtschaft zu anderen Kinase Gattungen des Menschen A: Phylogenetische Verwandtschaft humaner CK1 Isoformen (CK1a, g1-3, und ) und anderer Mitglieder der CK1 Familie (TTBK1, 2: Tau-Tubulin-Kinase 1 und 2; VRK1-3: Vaccinia-related kinase 1-3). B: Schematische Dargestellung der humanen CK1-Isoformen. Die N- und C-terminalen Bereiche sind in blau und die Kinasedomäne in grau dargestellt. 1 Einleitung 4 Abbildung 2: Domänenstruktur von CK1 (schematische Darstellung) Die jeweilige Position von Aminosäureresten ist anhand der Nummerierung erkennbar. Der gelbe Bereich kennzeichnet den N-Terminus, die Kinasedomäne ist in blau dargestellt und Bereiche des C-Terminus in grün. (ATP BS: ATP-Bindungstelle, KHD: Kinesin-homologe Domäne, NLS: putatives Kernlokalisationssignal, AutoID: autoinhibitorische Domäne, P: potenzielle Autophosphorylierungsstellen) Auch wenn auf Ebene der Primärstruktur keine nennenswerten Ähnlichkeiten zu anderen Proteinkinasefamilien auftreten, so besteht jedoch eine hohe Ähnlichkeit zwischen der Tertiärstruktur ihrer Kinasedomäne mit der dreidimensionalen Struktur der Kinasedomäne anderer Serin/Threonin spezifischer Kinasen. Die Tertiärstruktur der CK1 Kinasedomäne konnte dank Röntgenstrukturanalyse der C-Terminal trunkierten CK1 Isoformen CK1 und CKi1 entschlüsselt werden (Carmel et al. 1994; Xu et al. 1995; Longenecker et al. 1996; Longenecker et al. 1998; Long et al. 2012). Die Kinasedomäne weist eine zweilappige Struktur auf, welche sich aus einer kleineren N-terminalen -Domäne und einer großen C-Terminal gelegenen a-Domäne zusammensetzt. Diese a- und -Domänen bilden zusammen mit einer kurzen, sie verbindenden Scharnierregion, welche für die Flexibilität beider Domänen verantwortlich ist, das katalytische Zentrum. Zudem wird angenommen, dass an der Substratbindung zwei Loops beteiligt sind, von denen einer das putative Kernlokalisationssignal und der zweite die Kinesin-homologe Sequenz enthält (Xu et al. 1995; Longenecker et al. 1996). 1.2.1 Substratspezifität von CK1 und Konsensussequenz Die ursprüngliche Definition klassifizierte die Proteinkinasen der CK1-Familie als solche Kinasen, die das Phosphoprotein Casein als in vitro Substrat bevorzugen. 1 Einleitung 5 Casein ist jedoch kein physiologisches Substrat dieser Kinasen (Tuazon und Traugh 1991). Es steht vielmehr für die generelle Bevorzugung von Substraten, die saure oder bereits phosphorylierte Aminosäuren („hierarchische Phosphorylierung“) in der Umgebung von Serin- bzw. Threoninresten besitzen. Man bezeichnet diese Art der Konsensussequenz als „kanonisch“. Die kanonische Konsensussequenz für CK1 lautet S/T(P)-X3-S/T, welche durch die erfolgreiche Charakterisierung der Substratspezifität der CK1-Isoformen identifiziert werden konnte. Hierbei beschreibt S/T(P) ein phosphoryliertes Serin bzw. Threonin und X steht für eine beliebige Aminosäure. Hieraus wird ersichtlich, dass für die Modifizierung von Serin- sowie Threoninresten durch CK1 eine vorausgehende Phosphorylierung von Aminosäureresten N-Terminal der Zielphosphorylierungsstelle erforderlich ist (Flotow et al. 1990; Meggio et al. 1991; Meggio et al. 1992; siehe auch Abbildung 3). Abbildung 3: Kanonische Konsensussequenz für CK1 Das sich fortlaufend wiederholende S/T(P)-X-X-S/T Motiv führt zur Generierung der kanonischen Konsensussequenzen für CK1. Diese Abbildung zeigt dies beispielhaft anhand der Konsensussequenz von mPER1 und mPER2, welche in die Regulation des zirkadianen Rhythmus involviert sind (Okamura et al. 2004b). (mPER: Mammalian period circadian protein) CK1-vermittelte Phosphorylierung kann auch nach Bindung an das FXXXF Motiv erfolgen, welches ein in vielen CK1-Substraten vorhandenes Bindungsmotiv darstellt (Okamura et al. 2004b; Winter et al. 2004). Des Weiteren wurde nachgewiesen, dass ein aus der Sequenz S*LS bestehendes nichtkanonisches Motiv mit dem entsprechenden Ziel-Serin (S*) in Kombination mit einer Ansammlung saurer Aminosäurereste C-Terminal der Ziel-Phosphorylierungsstelle von CK1 erkannt wird (Marin et al. 2003). Solche Motive weisen beispielsweise die CK1-in 1 Einleitung 6 vivo-Substrate NF-AT sowie -Catenin auf. Die Phosphorylierung eines solchen nichtkanonischen Motivs ist jedoch im Vergleich zu einem von einer Phosphoaminosäure eingeleiteten Motivs 15 bis 25 Mal weniger effizient (Marin et al. 2003). Es ist allerdings anzunehmen, dass eine Substratphosphorylierung durch CK1 nicht allein vollkommen von der Konsensussequenz sondern auch von der Tertiärstruktur des Substrats abhängig ist (Cegielska et al. 1998). Im Jahr 2008 wurde eine neue Konsensussequenz für CK1 nachgewiesen: K/R-X-K/R-XX-S/T (Kawakami et al. 2008). Zelluläre SCS-(Sulfatid und Cholesterol-3-Sulfate)Bindeproteine, welche diese Konsensussequenz enthalten, können von den CK1-Isoformen CK1 a, und phosphoryliert werden. SCS-Bindeproteine treten gehäuft im Gehirn auf. Zu dieser Gruppe gehören das „myelin basic protein“ (MBP), das Tau Protein und die kleine GTPase RhoA (Kawakami et al. 2008). In neuesten Studien mit LRP6 im Wnt/β-Catenin-Signalweg wurde beobachtet, dass bei der intrazellulären Domäne von LRP6 eine verlängerte Variante dominiert. Diese Verlängerung hebt vermutlich eine konformationsbedingte Auto-Inhibierung der PPP(S/T)PX(S/T) Phosphorylierungsstelle auf, bzw. präsentiert CK1 die Phosphorylierungsstellen mit S/T-Häufungen und erlaubt GSK3 und CK1 ihre Phosphorylierungsstellen zu erreichen, um somit LRP6 und den Signalweg in downstream Richtung zu aktivieren (Liu et al. 2011). Bindungsmotive, welche saure Restgruppen im N-oder C-Terminus von Phosphoserin bzw. Phosphothreonin enthalten, werden häufiger durch CK1 phosphoryliert. In aktuellen Studien fiel insbesondere auf, dass gehäuft Motive mit einem Prolin an Position +1 durch CK1 phosphoryliert wurden. Allerdings konnte nicht beobachtet werden, dass alle Peptide mit einem gleichen bestimmten Motiv auch gleichermaßen phosphoryliert werden (Amanchy et al. 2011). 1.2.2 Regulationsmechanismen der CK1-Expression und -Aktivität Die CK1-Isoformen können als konstitutiv aktive Enzyme aus den unterschiedlichsten Organismen, Geweben und Zelllinien isoliert werden (Tuazon und Traugh 1991). Da die Kinasen konstitutiv aktiv sind, müssen sie folglich durch verschiedene Mechanismen 1 Einleitung 7 reguliert werden. Bislang konnten vielzählige Regulationsmechanismen der Aktivität von CK1 in vitro und in vivo ermittelt werden. Als Effektoren, die zu einer gesteigerten CK1-Aktivität führen, sind zu nennen: Stimulation von Zellen durch Insulin (Cobb und Rosen 1983) oder durch virale Transformation (Elias et al. 1981), Topoisomerase-Inhibitoren (Knippschild et al. 1997), sowie die Bestrahlung von Zellen mit g-Strahlen (Santos et al. 1996). Phosphatidylinositol 4,5-bisphosphat (PIP2) in der Membran wirkt hingegen inhibitorisch auf CK1a (Bazenet et al. 1990; Brockman und Anderson 1991; Gross et al. 1995). Die subzelluläre Lokalisation und Kompartimentierung der CK1-Isoformen ist von zentraler Bedeutung für die Regulation der Interaktion zwischen Kinase und Substrat. Durch Sequestrierung der Kinasen an intrazelluläre Strukturen oder Proteinkomplexe kann CK1 mit Modulatoren oder Substraten in Verbindung gebracht werden (Silibourne et al. 2002). Des Weiteren konnte nachgewiesen werden, dass die nukleare Lokalisation von CK1 und CK1 in Abhängigkeit von der Tageszeit reguliert wird, wie Studien zum zirkadianen Rhythmus in Mäusen zeigten (Lee et al. 2001). Die Regulation der Aktivität von CK1 durch posttranslationale Modifikation wird vorrangig durch reversible Phosphorylierung durchgeführt. Im Allgemeinen werden die CK1-Isoformen als aktive, jedoch phosphorylierte Kinasen isoliert. Eine in vitro Dephosporylierung durch ProteinPhosphatase 1 oder 2 kann zu einer kurzfristigen Aktivitätserhöhung führen. Die Autophosphorylierung des C-Terminus der Kinasen hat eine Inhibition der Substratphosphorylierung zur Folge und neutralisiert auf diese Weise den Effekt der Aktivitätssteigerung (Graves und Roach 1995; Cegielska et al. 1998; Gietzen und Virshup 1999). Zelluläre Kinasen phosphorylieren ebenfalls CK1, wodurch deren Aktivität reguliert werden kann. So wird CK1 neben Akt (Proteinkinase B), CLK2 (CDC-ähnliche Kinase 2) und PKCa (Proteinkinase C) vor allem durch PKA (cAMP abhängige Protein Kinase) in vitro und in vivo an Serin 370 phosphoryliert, was zu einer Abschwächung der Substratphosphorylierung dieser Kinase führt. Eine Mutation von Serin 370 zu Alanin erhöht bei CK1 im Vergleich zum Wildtyp die Phosphorylierungsaktivität der Kinase in vitro (Giamas et al. 2007). 1 Einleitung 8 Eine gesteigerte Aktivität der Casein Kinasen 1 resultiert u.a. aus einer Abspaltung des CTerminus und damit der autoinhibitorischen Domäne, als auch speziell bei CK1 aus einer durch Wnt-Signale (am ehesten durch PP2A) herbeigeführte in vivo Dephosphorylierung der Autophosphorylierungsstellen. Auch Interaktionen mit zellulären Proteinen, z.B. CK1BP, können die Regulation der Aktivität beeinflussen. Werden die Proteine Tau und α-Synuklein als Substrate eingesetzt, so zeigt sich, dass deren Phosphorylierung durch CK1 in Anwesenheit von CK1BP reduziert ist (Yin et al. 2006), während die Assoziation von CK1 Isoformen mit der RNA Helikase DDX3 deren Aktivität erhöht (Cruciat et al. 2013). 1.2.3 Funktionen von CK1 Die Mitglieder der CK1 Familie sind in zahlreichen Zellorganellen wie dem Zellkern, dem Zytoplasma und dem Zytoskelett, sowie in Vesikeln und in der Plasmamembran lokalisiert und spielen dort eine große Rolle bei vielen intrazellulären Prozessen. Diesbezüglich sind insbesondere zu nennen: Vesikeltransport, Apoptose, DNA-Reparatur, Chromosomenverteilung, Zellzyklusprogresseion, Transkription, zirkadianer Rhythmus und Mikrotubulidynamik (Zur Übersicht: Knippschild et al. 2005). 1.2.4 CK1 und das Tumorsuppressorprotein p53 CK1 spielt eine wichtige Rolle in der Krebsentstehung. Durch DNA-Schädigung und anderen zellulären Stress wird p53 aktiviert und führt sodann, je nach ankommendem Signal und Zelltyp, über Aktivierung entsprechender Signalwege entweder zu Zellzyklusarrest (Kuerbitz et al. 1992) oder Apoptose (Oren 1994; Schuler und Green 2001). Das Tumorsuppressorprotein p53 kann sowohl in vivo, als auch in vitro durch die CK1 Isoformen CK1a, CK1 und CK1 N-terminal (Ser 6, Ser 9 und Thr 18) phosphoryliert werden (Knippschild et al. 1997; Dumaz et al. 1999; Huart et al. 2009; Venerando et al. 2010). Nach einer DNA-Schädigung wird angenommen, dass p53 an das „p53 responsible Element“ (p53RE) im Bereich der Promotorregion von CK1 bindet, wodurch es zur Induktion von CK1 kommt. Es handelt sich somit um einen autoregulatorischen Loop zwischen diesen beiden Proteinen, was dafür spricht, dass die 1 Einleitung 9 Kinase von großer Bedeutung bei der Regulation von p53 in Stress-Situationen ist (Knippschild et al. 1997; Dumaz et al. 1999). Mdm2 und Mdm4 repräsentieren zelluläre Gegenspieler von p53 (Marine et al. 2006). Die Phosphorylierung von Threonin 18 über CK1 bzw. scheint die Interaktion von p53 mit dessen inhibitorischen Gegenspieler Mdm2 abzuschwächen, was die Aktivierung und Stabilisierung von p53 zur Folge hat. Mdm2 ist an der Ubiquitinierung und Degradation von p53 beteiligt und beeinflusst dessen Regulation somit wesentlich. Die Interaktion von CK1 und p53 wird jedoch nicht nur vom Phosphorylierungsstatus von p53, sondern auch vom Phosphorylierungsstatus von Mdm2 beeinflusst. Die Phosphorylierung von Mdm2 durch CK1 erfolgt an mehreren Serinresten (S240, S242 und S246), wodurch es die Degradation von p53 unterstützt (Winter et al. 2004). 1.2.5 Wnt-Signalweg CK1-Isoformen sind an der Regulation diverser Signalwege beteiligt, u.a. sind sie auch in den Wnt-Signalweg involviert und können diesen sowohl positiv als auch negativ beeinflussen. Eine Hyperaktivierung des Wnt/β-Catenin-Signalwegs kann zu einer Vielzahl von Tumoren führen, wohingegen eine Hypoaktivierung Knochenmissbildungen und neurodegenerative Erkrankungen begünstigt (Verkaar und Zaman 2011). Der WntSignalweg spielt eine wichtige Rolle bei der Regulation zentraler zellulärer Prozesse, wie Wachstum, Differenzierung embryonaler Zellen oder Proliferations- und Differenzierungsprozesse im adulten Organismus (Peifer und Polakis 2000; Huelsken und Birchmeier 2001; McKay et al. 2001; Tanaka et al. 2011). In neuesten Studien konnte nachgewiesen werden, dass CK1ε Mutanten, wie sie in Mamma-Carcinom-Zellen gefunden werden können, ein loss of function im kanonischen Wnt/β-Catenin Signalweg bewirken, jedoch zu einem gain of function im nichtkanonischen Wnt/Rac1/JNK und Wnt/Ca2+ Signalweg führen. Diese neuen Erkenntnisse zeigen, dass CK1ε aufgrund seines negativen Einflusses auf den Wnt/Rac1/JNK und NFAT-Signalweg möglicherweise als Tumorsuppressor bei Brustkrebs eingesetzt werden könnte (Foldynová-Trantírková et al. 2010). 1 Einleitung 11 1.2.7 Beteiligung von CK1 an der Entstehung von Krankheiten Die Kenntnis über eine Beteiligung von CK1 bei diesen Prozessen führte auch zum Nachweis einer Verknüpfung zwischen diversen Krankheitsbildern und CK1-Mutationen bzw. Änderungen in Expression oder Aktivität von CK1. So scheinen Fehlregulationen von CK1 bei neurodegenerativen Erkrankungen wie M. Alzheimer oder M. Parkinson und der Entstehung von Schlafrhythmusstörungen, sowie in der Tumorgenese und Tumorprogression eine Rolle zu spielen (Green et al. 2000; Li et al. 2000; Perez et al. 2011). Eine verstärkte Expression verschiedener CK1-Isoformen ist assoziiert mit dem Auftreten unterschiedlichster Tumorentitäten. So konnte in Nierenzellkarzinomen eine erhöhte CK1g3-Expression nachgewiesen werden (Masuda et al. 2003). In Chorionkarzinomen hingegen war CK1 nachweislich vermehrt exprimiert (Stöter et al. 2005). Aktuellen Studien zufolge konnte in duktalen Adenokarzinomen des Pankreas eine Expressionsverstärkung von CK1 und CK1 gefunden werden (Brockschmidt et al. 2008). Wie immunhistochemische Untersuchungen zeigten war die Expression von CK1 sowohl in schlecht differenzierten invasiven Mammakarzinomen, als auch in in situ Karzinomen herabgesetzt (Knippschild et al. 2005). Ebenso wurde für CK1 eine verminderte Expression festgestellt und eine Verknüpfung von verminderter Proteinreaktivität, Verlust der Heterozygotie und somatischen Mutationen nachgewiesen (Fuja et al. 2004, Knippschild et al. 2005). In tierexperimentellen Arbeiten konnte gezeigt werden, dass die Reaktion auf epithelialen DNA-Schaden von einer beträchtlichen p53-Aktivierung begleitet wird. Dies deutet darauf hin, dass der p53-Signalweg möglicherweise dem positiv kanzerogenen Potenzial einer Wnt-Hyperaktivierung entgegenwirkt. Des Weiteren verursacht ein Verlust der Heterozygotie des CK1α Gens die Entstehung von hochinvasiven Karzinomen in p53defizienten Darmzellen, was darauf hindeutet, dass CK1α als Tumorsuppressor fungiert, wenn p53 inaktiviert wurde (Elyada et al. 2011). Aktuelle Studien haben zudem gezeigt, dass CK1 in Ovarkarzinomen überexprimiert ist und mit einem schlechteren Überleben assoziiert ist (Rodriguez et al. 2012). 1 Einleitung 12 1.2.8 CK1-spezifische Inhibitoren Bisher wurden bereits mehrere CK1 spezifische „small molecule“ Inhibitoren charakterisiert. Kompetitve Inhibitoren, wie CKI-7, IC261 oder D4476 (Chijiwa et al. 1989; Mashhoon et al. 2000; Rena et al. 2004), treten mit ATP in Konkurrenz um seine Bindungsstelle. Ihre Wirkungsweise konnte aus Analysen der Kristallstruktur des bei einer IC50 von 1M für die Isoformen CK1 und CK1 spezifischen Inhibitors IC261 (3[(2,4,6-Trimethoxyphenyl)methylindenyl]indolin-2-on) im Komplex mit C-Terminal trunkierter CK1 abgeleitet werden (Mashhoon et al. 2000). Obwohl IC261 in vitro die Aktivität von CK1 Isoformen spezifisch inhibiert, wird inzwischen jedoch davon ausgegangen, dass die proliferationsinhibierenden Eigenschaften von IC261 auf die Mikrotubuli-destabilisierenden Eigenschaften zurückzuführen ist und nicht primär auf eine Inhibition von CK1/ (Cheong 2011). Im Jahr 2007 wurde mit Imidazol PF-670462 von Pfizer Global Research und Development ein spezifischer Inhibitor gegen CK1 und entwickelt. Der IC50-Wert für CK1 ist hierbei in vitro kleiner als 10 nM (Badura et al. 2007). Aufgrund der hohen Sequenzhomologie von CK1 und entwickelten Pfizer Global Research and Development PF-4800567 (3-(3-chloro-phenoxymethyl)-1-(tetrahydro-pyran-4-yl)-1H- pyrazolo[3,4-yl]-pyrimidin-4-ylamine), einen Inhibitor, welcher CK1δ im Vergleich zu CK1ε 22-fach schlechter inhibiert. Somit ist der Inhibitor PF-4800567 von großer Bedeutung bei der Untersuchung der unterschiedlichen Rollen von CK1 und in verschiedenen Signalwegen. Zudem führten in vitro Kinase Assays zur Identifizierung von einem dualen Inhibitor gegen p38a (IC50 = 0,45 µM) und CK1 (IC50 = 0,23 µM). Des Weiteren wurden zusätzliche Inhibitoren mit höherer Affinität zu CK1 als zu p38a entdeckt. Von diesen erwies sich CKP138 mit einem IC50-Wert von 5nM als der potenteste Inhibitor gegen CK1 Peifer et al. 2009). Von den dual-spezifischen Inhibitoren kommen bereits einige Roscovitin Derivate beim M. Alzheimer und diversen Krebserkrankungen zum Einsatz. Diese Inhibitoren können sowohl Cyklin-abhängige Kinasen als auch Mitglieder der CK1-Familie inhibieren (Oumata et al. 2008). 1 Einleitung 13 Im Jahr 2012 wurden zudem 2-Benzamido-N-(1H-benzo[d]imidazol-2-yl)thiazole-4carboxamide Derivative als potente Inhibitoren von CK1δ/ε beschrieben. Hiervon kristallisierten sich vor allem zwei heterocyclische Derivate 5 und 6 als potente und spezifische Inhibitoren von CK1δ (IC50 = 0.040 bzw. 0.042 μM) heraus. Während Derivat 5 eine 5-fach höhere Affinität zu CK1δ als zu CK1ε (IC50 CK1ε = 0.199 μM) zeigte, hemmte Derivat 6 zudem CK1ε (IC50 = 0.0326 μM) in gleichem Ausmaß wie CK1δ (Bischof et al. 2012). 1.3 Problemstellung CK1 ist an der Regulation einer Vielzahl physiologischer Prozesse beteiligt. Daher kann eine Deregulation von CK1 zur Entwicklung neurodegenerativer Erkrankungen, Schlafrhythmusstörungen und malignen Tumorerkrankungen beitragen. Mutationen sowie Veränderungen der CK1 Expression/Aktivität konnten in verschiedenen Tumorentitäten nachgewiesen werden und tragen zur Tumorprogression und Apoptoseresistenz aggressiver Tumore bei. Auf Proteinebene erfolgt die Regulation von CK1 durch Interaktionen mit zellulären Proteinen, durch proteolytische Abspaltung des regulatorischen C-Terminus sowie durch reversible Phosphorylierung. Erste Ergebnisse weisen zudem darauf hin, dass die Aktivität von CK1 durch zelluläre Kinasen (u.a. von PKA), die CK1 im C-Terminus phosphorylieren, moduliert wird. Ziel des Projekts war es daher, weitere zelluläre Kinasen, die CK1 spezifisch in der C-terminalen Domäne phosphorylieren, zu identifizieren und deren Phosphorylierungsstellen auf dem CK1 Molekül einzugrenzen. 2 Material und Methoden 14 2 Material und Methoden 2.1 Material 2.1.1 Chemikalien und Reagenzien Alle verwendeten Chemikalien wurden von folgenden Firmen in Analysenqualität bezogen: Aceton AppliChem GmbH, Darmstadt, Deutschland Acrylamid Carl Roth GmbH + Co. KG, Karlsruhe, Deutschland Ammoniumpersulfat (APS) Sigma-Aldrich, München, Deutschland Aprotinin Bayer, Leverkusen, Deutschland Benzamidin Sigma-Aldrich, München, Deutschland β-Mercaptoethanol Sigma-Aldrich, München, Deutschland Bromphenolblau Sigma-Aldrich, München, Deutschland BSA NEB, Frankfurt a.M., Deutschland Coomassie Brilliant Blue R-250 Waldeck/ Chroma, Münster, Deutschland Dithiothreitol (DTT) Invitrogen, Karlsruhe, Deutschland dNTPs Eppendorf, Hamburg, Deutschland Ethylendiamin-N,N,N',N'- Sigma-Aldrich, München, Deutschland tetraessigsäure (EDTA) Ethylenglykol-bis2(2- Sigma-Aldrich, München, Deutschland aminoethylether)-N,N,N',N'tetraessigsäure (EGTA) Essigsäure Sigma-Aldrich, Seelze, Deutschland Ethanol absolut puriss. Sigma-Aldrich, Seelze, Deutschland Ethidiumbromid Roche Applied Sciences, Mannheim, Deutschland Formaldehyd J.T. Baker, Deventer, Holland Glycerol Carl Roth GmbH + Co. KG, Karlsruhe, Deutschland 2 Material und Methoden 15 Glycin AppliChem, Darmstadt, Deutschland Glutathion-Sepharose Amersham Biosciences, Freiburg, Deutschland Hefeextrakt Sigma-Aldrich, Seelze, Deutschland IC261 (CK1 Inhibitor) ICOS Corp., USA IPTG Biomol, Hamburg, Deutschland Isopropanol Sigma-Aldrich, München, Deutschland Leupeptin Sigma-Aldrich, München, Deutschland Luminol (3-Aminophthalhydrazid) Sigma-Aldrich, Seelze, Deutschland Methanol Sigma-Aldrich, Seelze, Deutschland MgCl2 Sigma-Aldrich, Seelze, Deutschland Milchpulver Carl Roth GmbH + Co. KG, Karlsruhe, Deutschland Natriumchlorid (NaCl) AppliChem, Darmstadt, Deutschland Nonidet P-40 (NP-40) Sigma-Aldrich, Seelze, Deutschland Natriumdodecylsulfat (SDS) Carl Roth GmbH + Co. KG, Karlsruhe, Deutschland N,N,N',N'- Carl Roth GmbH + Co. KG, Karlsruhe, Tetramethylethylendiamin Deutschland (TEMED) Salzsäure (HCl) Merck, Darmstadt, Deutschland Stickstoff (N2), flüssig mti®, Neu-Ulm, Deutschland Tris(hydroxymethyl)-aminomethan USB, Cleveland, USA (Tris) Triton X-100 Sigma-Aldrich, München, Deutschland Wasserstoffperoxid (H2O2) J.T. Baker, Deventer, Holland 2.1.2 Radiochemikalien [γ-³²P] ATP Hartmann, Braunschweig, Deutschland 2.1.3 Größenstandards 1 Kb DNA-Marker Fermentas, St.-Leon-Roth; Deutschland 2 Material und Methoden 16 6x DNA-Ladepuffer Fermentas, St.-Leon-Roth; Deutschland Precision Plus Protein Biorad, München, Deutschland Dual Color Standards 2.1.4 Primer Tabelle 1: Auflistung aller verwendeten Primer SDM: Site-directed mutagenesis, T397A: CK1-Mutante mit Threonin zu Alanin Mutation an Position 397, A: Adenin, C: Cytosin, G: Guanin, T:Thymin 374 5’SDMT397ACK1drat 5’-ATA CCT CTC GCA TGT CCG CCT CAC AGA GGA GCA GGG ACA T-3’ 375 3’SDMT397ACK1rat 5’-TAT GGA GAG CGT ACA GGC GGA GTG TCT CCT CGT CCC TGT A-3’ 2.1.5 Bakterienstämme E.coli DH5α fhuA2Δ(argF-lacZ)U169 phoA glnV44 Φ80 Δ(lacZ)M15 2.1.6 Enzyme und Proteine DpnI NEB, Frankfurt a.M., Deutschland Lysozym Sigma, München, Deutschland Restriktionsendonukleasen NEB, Frankfurt a.M., Deutschland RNaseA Sigma, München, Deutschland Taq-DNA-Polymerase Eppendorf, Hamburg, Deutschland TPCK-Trypsin Pierce Biotechnology, Rockford, USA 2.1.7 Kits und Verbrauchsmaterialien 96-well Mikrotiterplatte Thermo Fisher Scientific, Roskilde, Dänemark BCA Protein Assay Kit Pierce Biotechnology, Rockford, USA DermaClean® Non-Sterile Ansell, Kulim, Malaysia Examination Gloves Dialyseschläuche ZelluTrans Carl Roth GmbH, Karlsruhe, Deutschland 2 Material und Methoden Dünnschicht-Chromatographieplatten 17 Merck Inc., USA aus Zellulose Effectene Transfection Reagent Qiagen, Hilden, Deutschland epT.I.P.S. Standard 0,1-10 μl, 2- Eppendorf, Hamburg, Deutschland 200 μl, 50-1 000 μl Filterpapier Whatman 3MM Schleier und Schüll, Dassel, Deutschland Gel Extraction-Kit QIAEX II Qiagen, Hilden, Deutschland Gewebekulturschalen Nunc, Wiesbaden, Deutschland Laborwischtücher Kimtech Science™ Kimberly-Clark®, Dallas, Texas, USA PVDF Membran GE Healthcare, München, Deutschland QIAfilter Plasmid Maxi Kit Qiagen, Hilden, Deutschland QuikChange® Site-Directed Stratagene, Heidelberg, Deutschland Mutagenesis Kit Rapid Dephos and Ligation Kit Roche, Mannheim, Deutschland Resource Q Anionenaustauschersäule Amersham Biosciences, Freiburg, Deutschland Röntgenfilm Agfa Cronex 5 Agfa-Gevaert N. V., Mortsel, Belgien Safe-Lock Tubes 0,5 ml, 1,5 ml, 2 ml Eppendorf®, Hamburg, Deutschland Skalpelle Aesculap, Tuttlingen, Deutschland Sterilfilter 0.22, 0.45 µm Millipore, Schwalbach, Deutschland Zentrifugenröhrchen Falcon (15 ml, Becton Dickinson, Franklin Lakes, USA 50 ml) 2.1.8 Geräte Pipetten Research® (Eppendorf®, Hamburg, Deutschland) 0,1-2,5 μl, 0,5-10 μl, 2-20 μl, 10-100 μl, 20-200 μl, 1001000 μl Oxford-Pipette Oxford-Instrument Co Ltd, UK Zentrifugen Zentrifuge 5415C Eppendorf®, Hamburg, Deutschland Zentrifuge 5417 R, Eppendorf®, Hamburg, Deutschland 2 Material und Methoden 18 Rotor FA-45-30-11 Zentrifuge 5430, Eppendorf®, Hamburg, Deutschland Rotor FA-45-24-11-HS Zentrifuge 5810R Eppendorf®, Hamburg, Deutschland Zentrifuge Megafuge 1.0 R, Heraeus GmbH, Hanau, Deutschland Rotor 2705 Zentrifuge Sorvall RC-5B Sorvall, Bad Homburg, Deutschland Zentrifuge Universal 32R Hettich, Tuttlingen, Deutschland Kühl-/ Gefrierschränke Comfort Liebherr GmbH, Biberach an der Riss, Deutschland Hera freeze HFU 586 Basic Heraeus GmbH, Hanau, Deutschland Öko Super Liebherr GmbH, Biberach an der Riss, Deutschland Profiline Liebherr GmbH, Biberach an der Riss, Deutschland Mikrowellengeräte MWU512WS Bauknecht Hausgeräte GmbH, Stuttgart, Deutschland Waagen Analysenwaage H54AR Mettler, Giessen, Deutschland Analysenwaage BA21009 Sartorius GmbH, Göttingen, Deutschland R180-D Sartorius GmbH, Göttingen, Deutschland 440-35A Kern & Sohn GmbH, Balingen, Deutschland Sonstige Geräte Autoklav 32-2-3 KSG Sterilisatoren GmbH, Olching, Deutschland BioPhotometer Eppendorf®, Hamburg, Deutschland 2 Material und Methoden DPU-414 Thermal Printer 19 Seiko Instruments Inc., Chiba, Japan Geldokumentationsapparatur TFL-20M Föbel, Frankreich Gelkammer Modell 1068BD Life Technologies, USA Glasgefäße VWR® International, Darmstadt, Deutschland Heizblock Techne Dry-block DB2A Thermo-Dux, Werheim, Deutschland Hoch-Volt Elektrophoresekammer für T. Patschinsky, CCS GmbH, Hamburg Dünnschichtchromatographie-Platten Inkubator T 5042 E Heraeus, Hamburg, Deutschland Inkubationsschüttler Certomat BS-1 B. Braun Biotech, Melsungen, Deutschland Mikrotiterplatten-Lesegerät Tecan TECAN München GmbH, Kirchheim, SPECTRA Classic Deutschland Mini-Trans Blot Kammer Biorad, München, Deutschland Mischgerät Reax top Heidolph, Schwabach, Deutschland Model 1 000/500 Power Supply Bio-Rad, Hercules, California, USA Mörser VWR® International, Darmstadt, Deutschland pH-Meter PH 525 WTW, Weilheim, Deutschland Power-Supply PAC 300 Biorad, München, Deutschland Schwenktisch KS250 basic IKA Labortechnik, Staufen, Deutschland Spatel VWR® International, Darmstadt, Deutschland Speedvac Savant Bachofer, Reutlingen, Deutschland Sterilwerkbank HLB2472GS Nunc Wiesbaden, Deutschland Thermocycler Primus 25/96 MWG Biotech, Ebersberg, Deutschland Thermomixer comfort Eppendorf®, Hamburg, Deutschland Ultraschall Sonifier 250 Branson, USA Video Graphic Printer UP-895CE Sony, Tokio, Japan 2.1.9 Software Microsoft Office 2010 Microsoft GmbH, Unterschleißheim, (Excel, Powerpoint, Word) Deutschland EndNote X 6.0.1 Thomson Reuters, Philadelphia, USA CorelDRAW 12 Corel, München, Deutschland 2 Material und Methoden 20 2.2 Methoden 2.2.1 Proteinbiochemische Methoden 2.2.1.1 SDS-PAGE (sodium dodecylsulfate polyacrylamide gel electrophoresis) Zelllysate wurden in SDS-Probenpuffer aufgenommen, kurz (~5 min) auf 99°C erhitzt und auf 12,5% SDS Gelen aufgetrennt. Als Proteingrößenmarker wurde der Precision Plus Protein Dual Color Standard (BioRad) benutzt. SDS-Sammelgel: 0,1% (w/v) SDS, 4% (w/v) Acrylamid, 130 mM Tris-HCl (pH 6,8); kurz vor dem Gießen der Gele wurden 0,09% (w/v) APS und 0,3% (v/v) TEMED zugegeben SDS-Trenngel: 0,1% (w/v) SDS, 12,5% (w/v) Acrylamid, 130 mM Tris-HCl (pH 8,8); kurz vor dem Gießen der Gele wurden 0,05% (w/v) APS und 0,12% (v/v) TEMED zugegeben SDS-PAGE-Laufpuffer: 250 mM Tris-HCl, 1,9 M Glycin, 1% (w/v) SDS 5x SDS-Probenpuffer: 250 mM Tris-HCl (pH 6,8), 25% (v/v) ß-Mercaptoethanol, 50% (v/v) Glycerol, 10% (w/v) SDS, 0,5% (w/v) Bromphenolblau 2.2.1.2 Färbung von Proteinen in SDS-Gelen mit Coomassie Brilliant Blue Aufgrund seiner Eigenschaft sich an die basischen Seitenketten der Aminosäuren anzulagern, kann Coomassie Brilliant Blue zum unspezifischen Färben aller Proteine verwendet werden. Die Gele wurden zum Färben der Proteinbanden für 15 Minuten in die Coomassie-Färbelösung gegeben, danach erfolgte die Entfärbung des Hintergrundes durch Schwenken in Coomassie-Entfärber und anschließend in dH2O. Coomassie-Färbelösung: 1,5 g Coomassie Brilliant Blue R-250 in 1 l Methanol/ ddH2O/ Eisessig (5:5:1) Coomassie-Entfärber: 10% (v/v) Isopropanol, 10% (v/v) Essigsäure 2 Material und Methoden 21 2.2.1.3 Quantitative Proteinbestimmung Die zu bestimmenden Proteinproben wurden auf eine 96-well Mikrotiterplatte aufgetragen, mit der Messlösung versetzt und anschließend gegen eine im Vorfeld hergestellte BSAVerdünnungsreihe über ein Spektral-Photometer bei der Wellenlänge 595 nm gemessen. Zu Beginn wurden Verdünnungen der Lysate (5 µl Probe + 35 µl H2O), sowie BSAVerdünnungen (BSA Stocklösung 1 mg/ml, BSA-Verdünnungen: 0,1 mg/ml, 0,3 mg/ml, 0,5 mg/ml, 07 mg/ml) mit ddH2O hergestellt. Anschließend wurden die Spalten der Mikrotiterplatte der Reihe nach mit je 10 µl Wasser, der BSA-Verdünnungen und der Proben befüllt. Zu jeder Probe (auch Wasser und BSA-Verdünnungen) wurden je 200 µl der angesetzten Assay-Lösung (2,2'-Bichinolon-4,4'-dicarbonsäure (BCA) Reagenz) dazugegeben. Abschließend wurde die Mikrotiterplatte abgedeckt und 30 Minuten bei 37°C inkubiert, bevor die Proteinkonzentrationen im Spektral-Photometer bestimmt werden konnten. BCA Reagenz: BCA Protein Assay Kit (Pierce Biotechnology) 2.2.1.4 In vitro Kinase Assays Für die Durchführung der in vitro Kinasereaktionen wurde pro Ansatz, bei einem Gesamtvolumen von 20 µl, folgende Zusammensetzung gewählt: Kinasepuffer bestehend aus 25 mM Tris-HCl (pH 7,5), 10 mM MgCl2, 100 µM ATP, 100 mM EDTA, sowie jeweils 0,2 µCi [γ-32P]-ATP, zusätzlich das jeweilige Substrat und die angegebenen Proteinkinase. Als Substrate wurden vergleichbare Mengen von affinitätsgereinigten GSTCK1δ Fusionsproteinen verwendet. Als Enzymquellen dienten folgende Proteinkinasen: Chk1, Chk2 und Clk2. Die Kinasereaktionen wurden unter Standardbedingungen durchgeführt (30 Minuten bei 30°C). Durch Zugabe von 5 µl 5xSDS-Probenpuffer wurden die Reaktionen beendet. Zur Quantifizierung des Phosphateinbaus in die GST-Fusionsproteine wurden die entsprechenden radioaktiv markierten Proteinbanden nach elektrophoretischer Auftrennung und Autoradiographie aus getrockneten Gelen ausgeschnitten und die Radioaktivität durch Cherenkov-Zählung im Szintillationszähler bestimmt. Der entsprechende Wert erlaubte 2 Material und Methoden 22 eine erste Aussage über die Anzahlt der phosphorylierten Aminosäurereste gemacht pro Substratmolekül. Ansatz (20 µl): Kinase 3 µl (vorgelegt) Substrat 3-7 µl (~ 0,5 µg) 10x Kinasepuffer mit 10 µM ATP 2 µl [g-³²P]-ATP 0,2 µl (= 2 µCi) H2O ad 20 µl 10x Kinasepuffer: 1 mM ATP, 250 mM Tris-HCl [pH 7,5], 100 mM MgCl2, 1 mM EDTA 2.2.1.5 Probenvorbereitung für die zweidimensionale Phosphopeptidanalyse Zunächst wurden mit [γ-³²P]ATP markierte Proteine in der SDS-Page aufgetrennt und auf eine PVDF Membran transferiert. Anschließend wurden die radioaktiv markierten Proteine auf dem entwickelten Röngtenfilm identifiziert und nachfolgend aus der Membran ausgeschnitten. Anschließend folgte die Absättigung der Membranstücke für 30 Minuten bei 37°C in 5% Polyvinylpyrrolidon (PVP) in ddH2O. Im Anschluss daran wurden die Membranstücke mit 50 mM NH4HCO3 gewaschen. Nun erfolgte die tryptische Spaltung und Elution der Phospholipide aus Membranstücken in 150 µl 50 mM NH4HCO3 mit 10 µg TCPK-Trypsin und 10 µg BSA für 4 Stunden bei RT. Danach wurden erneut 10 µg Trypsin für zusätzliche 3 Stunden dazugegeben. Um Verunreinigungen von den Eluaten abzutrennen, folgte nun ein Zentrifugationsschritt für 10 Minuten bei 10000 rpm. Der dadurch erhaltene Überstand wurde in flüssigem Stickstoff schockgefroren und in der Speedvac lyophyllisiert. Danach wurden die Proben für 2 Stunden auf Eis in 50 µl Perameisensäure oxidiert. Die Oxidation durch Zugabe von 250 µl H2O gestoppt. Die Proben wurden lyophyllisiert und in 100 µl pH 3,6-Puffer resuspendiert und erneut lyophyllisiert. Die Bestimmung des radioaktiven Phosphateinbaus der getrockneten Phosphoeptide erfolgte mittels Cherenkov-Zählung. Abschließend wurden die Peptide in pH 3,6-Puffer aufgenommen. Perameisensäure: 10% H2O2, 90% Ameisensäure, 1 h bei RT im Dunkeln vorinkubiert 2 Material und Methoden pH 3,6-Puffer: 23 Essigsäure/Pyridin/H2O (10:1:289) 2.2.1.6 Zweidimensionale Phosphopeptidanalyse Die Chromatographie der Phosphopeptide erfolgte auf einer Dünnschichtchromatographie(DC) Platte (Zellulose, 20x20 cm) in einer Elektrophoresekammer. Die radioaktiv markierten, in pH 3,6-Puffer aufgenommenen, Peptide wurden mithilfe einer OxfordPipette auf den Startpunkt aufgetragen. Zuvor war die aufzutragende Menge der einzelnen Peptide anhand der cpm nach Cherenkov-Zählung angeglichen worden. Zudem wurde 1 µl Farbmarker (grün) am oberen Rand zur Kontrolle aufgetragen. Die Elektrophorese erfolgte bei 1300 V und unter 1,5-2 bar Druck für 30 Minuten. Im Anschluss daran folgte das Trocknen der DC-Platte. Als Kontrolle für eine erfolgreiche Durchführung diente die Auftrennung des Markers in blau und gelb. Im nächsten Schritt erfolgte die Auftrennung in zweiter Dimension chromatographisch in Chromatographiepuffer in etwa 12 Stunden. Die Platte wurde hierzu mit der Probe nach unten in eine Glaskammer gestellt, deren Boden mit Chromatographiepuffer bedeckt war. Anschließend folgten das Trocknen der Platte und die Exposition auf einen Röntgenfilm. Je nach Anzahl der aufgetragenen cpm schloss sich bis zur Entwicklung des Films eine unterschiedliche lange Expositionszeit (mehrere Tage) bei -80°C an. pH 1,9-Puffer: Ameisensäure/Essigsäure/Pyridin/ddH2O (60/12,5/2,5/925 ml) Chromatographiepuffer: n-Butanol/Essigsäure/Pyridin/ddH2O (75/15/50/60 ml) [pH5,2] Farbmarker: 5 mg/ml DNP-Lysin, 1 mg/ml Xylencyanol FF 2.2.1.7 Phosphoaminosäurebestimmung (PAA) Die PAA wurde im Anschluss an die zweidimensionale Phosphopeptidanalyse durchgeführt. Sie diente der Indentifikation der Aminosäure (Serin, Threonin oder Tyrosin), welche durch die, Phosphorylierungsstellen entsprechenden, Signalen auf dem Film sichtbar gemacht worden war. 2 Material und Methoden Hierzu wurden zunächst 24 die zu bestimmenden Spots der zweidimensionalen Phosphopeptidanalyse auf der DC-Platte markiert, daraufhin aus der Zellulose-Seite der Platte ausgekratzt und anschließend in ein Eppendorf-Tube überführt. Die abgekratzte Zellulose wurde in pH 3,6-Puffer aufgenommen und 1 h bei RT geschüttelt. Nach dem anschließend folgenden Zentrifugationsschritt bei maximaler Geschwindigkeit für 1 min wurde der Überstand in ein neues Tube überführt. Nach erneutem Zentrifugieren und Überführung des Überstandes in Schraubdeckelröhrchen wurden die Proben lyophyllisiert. Anschließend erfolgte die saure Hydrolyse in 50 µl 6 N HCl für 2 h bei 120°C. Nach Zugabe von 500 µl H2O wurde erneut lyophyllisiert. Im Anschluss daran wurden die Proben in einen Mix aus 1 µl Phospho-Serin (1 µg/µl), 1 µl Phospho-Threonin (1 µg/µl), 1 µl Phospho-Tyrosin (1 µg/µl) und 2 µl Puffer pH 3,6 aufgenommen und unter kaltem Fönen auf Cellulosedünnschichtplatten aufgetragen. Nach Befeuchtung der Platte mit Hilfe einer Filterpapiermaske mit Puffer pH 1,9 folgte die elektrophoretische Auftrennung in erster Dimension bei 1,5 kV in pH 1,9 Puffer für 15 min. Nachdem die DC-Platte danach wieder vollständig getrocknet war, wurde sie mit Puffer pH 3,6 befeuchtet. Für die Auftrennung in der zweiten Dimension wurde die Platte jetzt um 90°C gegen den Uhrzeigersinn gedreht in die Elektrophoresekammer eingelegt, sodass die Auftragepunkte zur Kathode zeigten. Die Auftrennung in 2. Dimension erfolgte nun bei 1,5 kV in Puffer pH 3,6 für 15 min. Nach vollständigem Trocknen der Platte, wurden die Phosphoaminosäuremarker durch Besprühen mit Ninhydrin-Färbelösung sichtbar gemacht und die Platte bei 60°C für 6 min in den Wärmeschrank gestellt. In Abhängigkeit von der Intensität des erwarteten Signals wurde anschließend ein Röntgenfilm sowie ein Verstärker-Screen aufgelegt und die Platte bei -80°C gelagert. Schließlich wurden die Positionen der Phosphoaminosäuremarker mit dem Ergebnis des Autoradiogramms abgeglichen, um die phosphorylierte Aminosäure identifizieren zu können. Puffer pH 1,9: Essigsäure/Ameisensäure/H20 78:25:857 (v/v) Puffer pH 3,6: Essigsäure/Pyridin/H2O Ninhydrin-Färbelösung: 0,2% Ninhydrin (w/v) in Aceton, 4% Essigsäure 50:5:945 (v/v) 2 Material und Methoden 25 2.2.1.8 Silberfärbung Die Silberfärbung diente der Anfärbung von Proteinen nach erfolgter Elektrophorese. Zu Beginn wurden die Gele 10 min in der Fixierlösung I (FIX I) geschüttelt, dadurch wurden die Proteine im Gel denaturiert und fielen aus. Daraufhin wurde das Gel 2-mal für je 5 min in Fixierlösung II (FIX II) und anschließend 1 min in 1x Oxidationslösung geschüttelt. Auf 3 Waschschritte á je 30 min in H2O folgte das Schütteln der Gele in 1x Silberlösung für 15 min. Dadurch lagerten sich Silberionen an die Proteine an. Im Anschluss an weitere Waschschritte erfolgte die Zugabe von Entwicklerlösung und das Wechseln derselben, nachdem sich die Lösung gelb gefärbt hatte. Durch die Zugabe von alkalischem Formaldehyd im Rahmen des Entwicklungsschrittes wurden die Silberionen zu elementarem Silber reduziert, was zu einer Schwarzfärbung der Proteine führte. Als der gewünschte Färbegrad erreicht war, wurde die Reaktion mit Stopplösung beendet. FIX I: FIX II: 20x Oxidationslösung: 10x Silberlösung: Entwicklerlösung (frisch!): Stopplösung: 40% MeOH 120 ml 10% Essigsäure 30 ml H2O 150 ml 10% EtOH 30 ml 5% Essigsäure 15 ml H2O 255 ml K2Cr2O7 0,5 g (lichtgeschützt bei 4°C) rauchende HNO3 75 μl H2O 500 ml AgNO3 1 g (lichtgeschützt bei 4°C) H2O 500 ml Na2CO3 2,89 g Formaldehyd 150 μl H2O 100 ml 5% Essigsäure 15 ml H2O 285 ml 2 Material und Methoden 26 2.2.2 Bakterien 2.2.2.1 Aufreinigung von GST-Fusionsproteinen Jeweils 50 ml LB-Medium mit 50 g/l Ampicillin wurde mit geringen Mengen entsprechender rekombinanter E.coli-Bakterien, Stamm DH5a, angeimpft und über Nacht bei 37°C und 180 rpm inkubiert. Nach Zugabe von weiteren 450 ml LB-Medium (mit 50 g/l Ampicillin) am folgenden Morgen und Fortsetzung der Inkubation bei 37°C und 180 rpm, wuchs die Bakterienkultur weiter bis sie eine OD595 von 0,700-0,900 erreicht hatte. Die Zugabe von 0,1 mM IPTG initiierte die Expression der gewünschten rekombinanten Proteine. Nachdem die Bakterienkultur für weitere zwei Stunden bei 37°C und 180 rpm inkubiert worden war, wurden die Bakterien durch Zentrifugation bei 5000 rpm und 4°C für 10 Minuten sedimentiert. Anschließend wurde das Bakterienpellet in 10 ml Lysispuffer resuspendiert und in ein 30 ml Sorvall-Röhrchen überführt. Auf die Zugabe von 2,5 mg Lysozym folgte die Inkubation der Bakterienlösung für 30 Minuten auf Eis und anschließend die Zugabe von weiteren 10 ml Lysispuffer. Im nächsten Schritt wurde die Lösung für zwei Sekunden Ultraschall ausgesetzt, wobei die bakterielle DNA fragmentiert wurde. Anschließend wurden die unlöslichen Bakterienbestandteile bei 10000 rpm und 4°C für 30 Minuten abzentrifugiert. Der Überstand wurde, nach Zufügen von 300 l aufgeschwemmter Glutathion-Sepharose (1:1 in PBS, v/v), bei 4°C zwei Stunden rotiert und dann für zwei Minuten bei 2000 rpm und 4°C zentifugiert. Nach zweimaligem Waschen des Pellets mit Lysispuffer folgten drei weitere Waschschritte mit Waschpuffer und daraufhin die Resuspension in 600 l Elutionspuffer und ein Rotationsschritt für 20 Minuten bei 4°C. Daraufhin wurde die Lösung bei 14000 rpm und 4°C für eine Minute zentrifugiert und der Überstand bei 4°C gelagert. Dieser Schritt wurde noch mit 300 l und mit 150 l Elutionspuffer wiederholt, wobei die jeweiligen Überstände zusammengeführt wurden. Im Anschluss wurde die Lösung für drei Stunden bei 4°C gegen die Dialyselösung dialysiert und dann 10% Glycerol zugegeben. Die Proteine wurden danach bei -80°C gelagert. Lysispuffer: 20 mM Tris HCl (pH 7,6), 150 mM NaCl, 10% Glycerol, 0,5% NP-40, 30 g/ml Aprotinin, 1 mM DTT, 1 mM EDTA, 1 mM EGTA, 1 mM Benzamidin, 50 M Leupeptin 2 Material und Methoden Waschpuffer: 27 20 mM Tris HCl (pH 7,6), 50 mM NaCl, 10% Glycerol, 1% Aprotinin, 1 mM EDTA Elutionspuffer: 50 mM Tris (pH 7,0), 10 mg reduziertes Glutathion, 1 mM EDTA Dialyselösung: 50 mM Tris (pH 7,0), 1 mM EDTA 2.2.2.2 Aufreinigung von Plasmid-DNA (Mini-Präp) Einzelne Bakterienkolonien von einer Agarplatte wurden zusammen mit 3 ml LB-Medium sowie 50 µg/µl Ampicillin angeimpft und über Nacht bei 37°C und 180 rpm inkubiert. Am folgenden Tag wurden 2 ml dieser Kultur für 5 Minuten bei 4°C und 5000 rpm abzentrifugiert. Anschließend folgte die Resuspension des Bakterienpellets in 200 µl Puffer S1. Dann wurden 200 µl S2-Puffer dazugegeben. Das darin enthaltene SDS zerstört die Zellen. Nun wurden 200 µl Puffer S3, welcher bei 4°C vorgekühlt worden war, zur Neutralisation dazugegeben. Durch mehrmaliges Wenden der Eppendorf-Tubes entstand eine homogene Suspension, welche ein weißes Flockulat enthielt. Nach Lagerung für 10 min auf Eis und Zentrifugieren folgte nach Überführung des Überstandes in ein neues Tube und Zugabe von 600 µl Isopropanol (100%) ein weiterer Zentrifugationsschritt für 10 min bei 4°C und 14000 rpm. Nach Verwerfen des Überstandes wurde das Pellet mit 200 µl Ethanol (70%) gewaschen. Nachdem abermals zentrifugiert worden war (10 min bei RT und 14000 rpm) wurde der Überstand vorsichtig abgenommen und das Pellet bei RT getrocknet. Anschließend wurde die DNA in 20 µl Wasser aufgenommen und auf -20°C eingefroren. LB-Medium: 1% Bacto Trypton, 0,5% Bacto Hefe-Extrakt, 1% NaCl Macherey-Nagel Kit Buffer S1: 500 mM Tris-HCl, 10 mM EDTA, 100 µg/ml Rnase, pH 8,0 Buffer S2: 200 mM NaOH, 1% SDS Buffer S3: 2,8 M KAc, pH 5,1 2 Material und Methoden 28 2.2.2.3 Aufreinigung von Plasmid DNA im größeren Maßstab (Maxi-Präp, High-copy plasmid DNA purification) Die Maxi-Präp Methode ermöglicht die Extraktion von Plasmid-DNA aus Bakterienzellen. Hierzu wurde das QIAfilter Plasmid Maxi Kit von Qiagen benutzt. Zu Beginn wurden 200 ml LB-Medium (mit 50 g/l Ampicillin) mit 50 l transformierten E.coli DH5a angeimpft und über Nacht bei 37°C und 180 rpm inkubiert. Am folgenden Tag erfolgte die Zentrifugation der Bakteriensuspension für 10 Minuten bei 4000 rpm und 4°C, der Überstand wurde verworfen. Nach Lösung des Pellets in 12 ml Puffer S1, wurden 12 ml Puffer S2 dazu gegeben und die Lösung durch mehrmaliges Wenden durchmischt. Weitere 12 ml 4°C-kalter Puffer S3 wurden nach einer Inkubationszeit von maximal 5 Minuten zugegeben und die Lösung wurde nach erneutem Durchmischen durch Wenden für 5 Minuten auf Eis gestellt. In der Zwischenzeit wurden Säulen mit 6 ml Puffer N2 äquilibriert, um sodann das gefilterte Bakterienlysat auf die Säule zu geben, die danach mit 32 ml Puffer N3 gewaschen wurde. Daraufhin folgte die Eluierung der in der Säule verbliebenen Plasmid-DNA mit 15 ml Puffer N5, daraufhin wurde die Plasmid-DNA mit 11 ml Isopropanol (100%) präzipitiert und für 30 Minuten bei 14000 rpm und 4°C abzentrifugiert. Schließlich wurde das Pellet mit 1 ml Ethanol (70%) gewaschen und für 10 Minuten bei 14000 rpm sowie Raumtemperatur abzentrifugiert. Nachdem das Pellet für etwa 20 Minuten an der Luft getrocknet worden war, wurde es in 50 l H2O gelöst und bei -20°C gelagert. Buffer S 1: 50 mM Tris-HCl, 10 mM EDTA, 100 µg/ml RNase A, pH 8.0 Buffer S 2: 200 mM NaOH, 1% SDS Buffer S 3: 2.8 M KAc, pH 5.1 Buffer N 2: 100 mM Tris, 15% Ethanol, 900 mM KCl, 0.15% Triton X-100, eingestellt auf pH 6.3 mit H3PO4 Buffer N 3: 100 mM Tris, 15% Ethanol, 1,15 M KCl, eingestellt auf pH 6.3 mit H3PO4 Buffer N 5: 100 mM Tris, 15% Ethanol, 1 M KCl, eingestellt auf pH 8.5 mit H3PO4 2 Material und Methoden 29 2.2.2.4 Transformation Zunächst wurden 50 µl kompetente Bakterien langsam auf Eis aufgetaut und mit 2 µl des Ligationsansatzes versetzt. Danach folgten die Inkubation auf Eis für etwa 30 min und anschließend ein Hitzeschock bei 42°C für 2 min. Nach einer weiteren Inkubation auf Eis für 3 min wurden 200 µl LB-Medium (ohne Antibiotikum) dazugegeben. Auf einen weiteren Inkubationsschritt bei 37°C für 45 min (oder länger) auf dem Thermomixer (schütteln bei etwa 750 rpm) folgte das Ausplattieren auf Agarplatten (mit Ampicillin zur Selektion). Daraufhin wurden die Platten über Nacht bei 37°C inkubiert. Bei erfolgreicher Transformation zeigte sich ein Wachstum auf der Übernacht-Kultur. 2.2.3 DNA 2.2.3.1 Polymerasekettenreaktion (PCR) Zu Beginn erfolgte zunächst die Denaturierung der zu amplifizierenden DNA durch Erhitzen bei 95°C, wodurch die Sekundärstrukturen der DNA aufgelöst und die Doppelstränge in zwei Einzelstränge aufgetrennt wurden. Anschließend musste die Temperatur auf die zuvor bestimmte ideale Anlagerungstemperatur der verwendeten Oligonukleotide gesenkt werden, sodass sich diese an die Einzelstränge anlagern und dadurch einen Startpunkt für die DNA-Polymerase bilden konnten. Die Taq-DNAPolymerase katalysierte nun bei 72°C die Synthese eines komplementären Strangs. Die Reihenfolge dieser drei Schritte wurde nun je nach PCR-Reaktion bis zu 35mal wiederholt, um die exponentielle Vermehrung der DNA-Stränge um das 2n-fache zu ermöglichen. Bei allen PCR-Reaktionen kam folgender Ansatz in einem Volumen von entweder 25 oder 50 µl zum Einsatz. 2 Material und Methoden 30 Tabelle 2: Ansatz für PCR-Reaktionen PCR: Polymerase-Ketten-Reaktion, Mg: Magnesium, Cl: Chlor, M: molar (mol/l), m: milli, µ: mikro, l: Liter, U: unit, DNA: Desoxyribonukleinsäure, cDNA: komplementäre DNA, dNTP: Desoxynukleotidtriphosphat PCR-Mix Stocklösung PCR-Reaktion PCR-Puffer 10x 1x dNTPs 2,5 mM 250 µM 3‘-Primer 10 µM 0,2 µM 5‘-Primer 10 µM 0,2 µM mit 25 mM MgCl2 genom. DNA; cDNA 1 µl bzw. Plasmid DNA Taq-Polymerase 5 U/µl H20 1 U (0,04 U/µl) Auf 25 bzw. 50 µl PCR-Reaktionen zur Site-spezifischen Mutagenese Die site-spezifische Mutagenese diente der Einführung einer Punktmutation an der Aminosäurenposition 397 (T→A) in die codierende Sequenz von CK1δ. Ein sich daran anschließender Verdau des Ursprungsvektors mit dem Restriktionsenzym DpnI führt zur Selektion des mutierten Vektors. Als Template für die PCR-Reaktion diente der pGEXCK1δ Vektor, nach Amplifikation mit den Primern 374 und 375 (siehe 2.1.4). Verwendet wurde der QuikChange® Site-Directed Mutagenesis Kit von Stratagene (Heidelberg, Deutschland), vorgegangen wurde genau nach Angaben des Herstellers. 2 Material und Methoden 31 Abbildung 5: Prinzip der Site-directed Mutagenesis. Mittels PCR kommt es zur Vermehrung des Zielgen tragenden Plasmids zusammen mit den mutationstragenden Primern, im gleichen Schritt wird hierbei die entsprechende Mutation eingebaut. Im Anschluss findet der Verdau der Template-DNA statt, letztendlich bleibt nur noch das mutationstragende Plasmid übrig. Dieses gelangt daraufhin durch Transformation in kompetente E.coli-Bakterien, welche anschließend für die Expression des mutierten Proteins verantwortlich sind (modifiziert nach Stratagene, Heidelberg, Deutschland). (PCR: Polymerase-Ketten-Reaktion, DNA: Desoxyribonukleinsäure) 2.2.3.2 Schneiden von DNA mit Restriktionsenzymen Hierbei werden entweder blunt-end oder sticky-end Schnittstellen generiert, in welche später ein gewünschtes Gen eingefügt werden kann. Bei einem gewünschten Endvolumen von 100 µl werden etwa 3 µg DNA eingesetzt (z.B. aus Mini- oder Maxi-Präps gewonnen). In Abhängigkeit von dem verwendeten Restriktionsenzym kommen etwa 5 U des Enzyms, sowie ein entsprechender Puffer 2 Material und Methoden 32 (orientiert sich am verwendeten Enzym, evtl. muss zudem zusätzlich BSA zugegeben werden) für die Herstellung des Ansatzes zum Einsatz (Def. U: Enzymaktivität, die pro min unter definierten Bedingungen die Umwandlung von 1 mmol Substrat katalysiert). Anschließend wurde der Ansatz mit H2O auf das Endvolumen aufgefüllt. Schließlich folgte der Restriktionsverdau bei 37°C für 1 h. 2.2.3.3 Ligation von DNA Hierzu wurden die Komponenten des Ansatzes in folgender Reihenfolge zueinander gegeben. Zum Wasser wurde zuerst der Puffer, dann aufeinander folgend die DNA und das Enzym gegeben, um evtl. auftretende Probleme, dadurch verursacht, dass das Enzym den pH-Wert des Wassers oder den unverdünnten Puffer nicht verträgt, zu vermeiden. Verwendet wurde der Rapid Dephos and Ligation Kit (Roche). Anschließend wurde der Ansatz über Nacht bei 15°C gelagert. T4-Ligase 1 µl 5x Ligase-Puffer 3 µl Plasmid-DNA 0,5 - 1 µl mit H2O auf 15 µl auffüllen 2.2.4 Agarosegelelektrophorese und Herstellung von Agarosegelen Es wurden 1%-ige Agarose-Gele hergestellt, welche dazu verwendet wurden, nach dem Auftragen von Proben, der Auftrennung von DNA-Fragmenten nach Größe und Ladung zu dienen. Zur Gelherstellung wurde 1g Agarose abgewogen, in einen Erlenmeyerkolben gegeben und 1x TAE (Tris-Acetat-Puffer ~pH 8,0 und EDTA) auf 100 ml aufgefüllt. Anschließend wurde die Agarose in der Mikrowelle aufgekocht, bis sich die gesamte Agarose aufgelöst hatte (Flüssigkeit muss glasklar werden). Daraufhin erfolgte die Abkühlung unter fließendem kaltem Wasser auf etwa 50°C. Nach Zugabe von 1 - 2 µl Ethidiumbromid (40 2 Material und Methoden 33 µg/l) wurde die Flüssigkeit durch leichtes Schwenken gemischt und dann in die zuvor zusammengebaute Gelform gegossen (Bläschenbildung sollte dabei vermieden werden). Nachdem das Gel nach 20 – 30 min ausgehärtet war, wurde das Gel in die Gelkammer, welche vollständig mit TAE bedeckt war, eingesetzt. TAE: 40 mM Tris 1 mM EDTA pH auf 8.0 einstellen mit konz. Essigsäure Für den Auftrag der Proben wurden 5 µl (6x) Lade-Puffer (Fermentas) zu den Proben (20 µl) dazugeben. Der Puffer enthielt die beiden Farbstoffe Xylene-Cyanol und Bromphenolblau. Außerdem wurden 5 µl Marker (1 kb-Ladder) auf das Gel aufgetragen, um die DNA-Fragmentlängen bestimmen zu können. Nachdem die Proben auftragen worden waren, wurde die Gelkammer geschlossen, an das Netzgerät angeschlossen und bei 120 V etwa 30 min laufen gelassen. Da die Proben zuvor mit dem Farbstoff Ethidiumbromid markiert worden waren, konnten die DNA-Banden nun unter UV-Licht betrachtet werden. Extraktion von DNA-Fragmenten aus Agarosegelen Hierzu wurde das QiaexII Kit von Qiagen verwendet. Durch Lösung der Agarose und der selektiven Bindung der DNA an QIAEXII Silica-Gel Partikeln in Anwesenheit von chaotropen Salzen konnte die Aufreinigung der DNA realisiert werden. QIAEXII ermöglicht es DNA von Agarose, Nukleotiden, Polyacrylamid, Proteinen und Salzen ohne Phenolextraktion oder Ethanolprezipitation zu trennen. 2 Material und Methoden 34 2.2.5 Statistische Methoden 2.2.5.1 Standardabweichung Die Versuche wurden pro Experiment je dreimal durchgeführt. In den Abbildungen sind die Mittelwerte mit der Standardabweichung dargestellt. Die Diagramme wurden mit Hilfe von Microsoft Excel erstellt. Signifikante Differenzen sind im Text erwähnt und in den Abbildungen gekennzeichnet 2.2.5.2 Michaelis-Menten-Kinetik Um zu untersuchen, wie sich die katalytische Aktivität eines bestimmten Enzyms in Bezug auf ein ausgewähltes Substrat verhält wurde mit Hilfe der Michaelis-Menten-Kinetik eine Sättigungsfunktion bestimmt. Die Sättigungsfunktion wurde durch Einsetzen der Parameter Km und Vmax folgendermaßen formuliert: v = (Vmax*[S])/(Km+[S]). 3 Ergebnisse 35 3 Ergebnisse 3.1 Beeinflussung der Aktivität von CK1 durch Phosphorylierung Die Regulation von CK1 erfolgt durch zahlreiche Mechanismen (Knippschild et al. 2005). So deuten frühere Ergebnisse darauf hin, dass auf Proteinebene auch durch Phosphorylierungstellen-spezifische Phosphorylierung durch zelluläre Kinasen die Aktivität von CK1 moduliert werden kann (Giamas et al. 2007). Eine durchgeführte Aminosäurensequenz-Analyse von CK1 mithilfe des „Scansite“Programms (http://scansite.mit.edu) ergab, dass Konsensussequenzen für mehrere zelluläre Kinasen in der Sequenz von CK1 vorkommen. Diejenigen, die CK1 in der regulatorischen C-terminalen Domäne phosphorylieren sind in Abbildung 6 dargestellt. Abbildung 6: Schematische Darstellung der Lage der Phosphorylierungsstellen potenzieller Kinasen, die CK1 C-Terminal phosphorylieren Dargestellt ist CK1 aus der Ratte Aminosäurensequenz 1-428). Zur Darstellung kommt hier vor allem die C-terminale Domäne von CK1 sowie die von „Scansite“ ermittelten Phosphorylierungsstellen. Außerdem die Proteinkinasen, die diese Serin/Threonin spezifischen Kinasen potenziell phosphorylieren können. Da Chk1-Konsensusmotive nicht in der Scansite-Software implementiert sind, wurden diese nach dem in O'Neill et al., 2002 beschriebenen Motiv im C-Terminus von CK1 gesucht. (CK1 Casein Kinase 1 delta, PKA: Proteinkinase A, PKC: Proteinkinase C, ATM Kinase: Ataxia-telangiectasia mutated Kinase, DNA-PK: DNA-dependent protein kinase, p38 MAPK: p38-mitogenaktivierte Proteinkinasen, Erk1 Kinase: extracellular signal-regulated kinases, Cdk5 Kinase: Cyclin-dependent kinase 5, Cdc2 Kinase: cell division cycle 2 (humanes Homolog zu Cdk1)-Kinase, GSK3: Glycogen synthase kinase 3, Chk1: Checkpoint-Kinase 1, Chk2: Checkpoint-Kinase 2, Akt: andere Bezeichnung für Proteinkinase B, Clk2: CDC-like Kinase 2, S: Serin, T:Threonin) 3 Ergebnisse 36 3.1.1 Phosphorylierung des C-Terminus von CK1 durch Clk2 Um diese Vermutung zu verifizieren, die von den durch das Scansite-Programm ermittelten potenziellen Kinasen phosphoryliert werden können, wurden zunächst Kinaseassays durchgeführt, in denen Chk1, Chk2 und Clk2 als Enzym und Glutathion-Stransferase oder verschiedene GST-CK1-Fusionsproteine (GST-CK1 305-343 , GST-CK1 305-428 und GST-CK1 375-428 305-357 , GST-CK1 ) als Substrat eingesetzt wurden. Hierfür wurden jeweils gleiche Mengen der verschiedenen GST-CK1 Fusionsproteine (s. Abbildung 7) in die Reaktionen eingesetzt. 1 428 1 305 375 428 305 357 428 305 343 428 1 353 375 428 T323A S370A T323A S370A T323A S328A S370A T323A S328A S370A 323 1 428 323 1 428 323 1 428 331 GST GST 305 428 1 305 GST 1 GST 428 S370A S331A T337A S370A 428 305 428 GST S370A 375 1 428 S331A 331 GST GST 1 1 GST GST 1 428 S318A GST GST 1 318 GST GST 1 1 GST GST 428 1 GST T397A 375 428 T397A Abbildung 7: Schematische Darstellung der verschiedenen GST-CK1 Fusionsproteine, die in Kinaseassays eingesetzt wurden Der GST-Anteil der Fusionsproteine ist in blau dargestellt, der CK1 spezifische Anteil in gelb. (GST: Glutathion-S-transferase, CK1: Casein Kinase 1, A: Alanin, S: Serin, T: Threonin) In Abbildung 8 sind die Ergebnisse der Kinaseassays dargestellt, in denen Clk2 als Kinase und gleiche Proteinmengen der verschiedenen GST-CK1 Fusionsproteine oder GST als Kontrolle eingesetzt wurden. Der radioaktive Phosphateinbau in die Substrate wurde durch ein Autoradiogramm sichtbar gemacht (Abb. 8A) und durch Cherenkov-Zählung quantifiziert (Abb. 8B). 3 Ergebnisse 37 Wie aus Abbildung 8B ersichtlich wurden nicht nur die verschiedenen GST-CK1 Fragmente durch Clk2 in vitro phosphoryliert, sondern auch GST selbst. Subtrahiert man die in GST eingebaute Phosphatmenge von der Phosphatmenge, die in die GST-CK1 Fusionsfragmente eingebaut wurde, so zeigen vier Fusionsproteine eine 1,8 – 2,5 fach erhöhte Phosphorylierung im Vergleich zu der Phosphorylierung von GST. Hierbei handelt es sich um GST-CK1 305-375, GST-CK1 305-357, GST-CK1 305-343 und GST-CK1 375-428. Schließt man den Phosphorylierungsgrad der GST-CK1 Substrate, die Mutationen an den Phosphorylierungsstellen aufweisen, mit in die Interpretation der Ergebnisse ein, so ergeben sich Hinweise darauf, dass Clk2 die Positionen S370, T397, sowie einen Serinoder Threoninrest im Bereich der Aminosäuren 305-343 phosphorylieren kann. A B Abbildung 8: Phosphateinbau in CK1-GST-Fusionsproteine durch Clk2 Es wurden Kinaseassays mit verschiedenen C-terminalen Fragmenten von CK1 oder GST als Substrate und Clk2 als Kinase durchgeführt. (A) Coomassie-gefärbtes SDS-Gel und entsprechendes Autoradiogramm (Expositionszeit: 17h). (B) Grafische Darstellung der Cherenkov-Zählung der Proben. Die Abbildung wurde mittels Microsoft Excel erstellt. Die Fehlerbalken beschreiben die Standardabweichung als Maß dafür, wie weit die jeweiligen Werte um den Mittelwert (Durchschnitt) aus den drei Stichproben streuen. Wo die Fehlerbalken fehlen unterschieden sich die einzelnen Werte kaum voneinander. (CK1 Casein Kinase 1 delta, Clk2: CDC-like Kinase 2, GST: Glutathion-S-transferase, SDS: sodium dodecylsulfate) 3.1.2 Chk2 phosphoryliert CK1 in der C-terminalen Domäne Basierend auf der Scansite-Analyse zeigte sich Chk2 als weitere potenzielle Kinase, die CK1 an zusätzlichen Stellen phosphorylieren könnte. In Abbildung 9 ist die Auswertung der Kinaseassays mit Chk2 dargestellt. Abbildung 9A zeigt das Coomassie-gefärbte SDSGel mit GST und den, in gleicher Menge eingesetzten GST-CK1-Fragmenten, sowie das dazugehörige Autoradiogramm. In Abbildung 9B sind die Ergebnisse der 3 Ergebnisse 38 Radioaktivitätsbestimmung der phosphorylierten Proteine mittels Cherenkov-Zählung grafisch dargestellt. Die Experimente mit Chk2 lieferten insgesamt wenig aussagekräftige Ergebnisse, erkennbar an den sehr schwachen Ergebnissen der Cherenkov-Zählung (s. Abbildung 9B). Daher bleiben nach Abzug der durch GST bedingten Background-Phosphorylierung lediglich zwei aussagekräftige Fragmente erhalten (GST-CK1 305-375 und GST-CK1 375428 ). Da das endständige GST-CK1 375-428 Fragment hierbei eine wesentlich stärkere Phosphorylierung aufweist, kann aus diesem Ergebnis die Schlussfolgerung gezogen werden, dass Chk2 CK1 zwischen den Aminosäuren 375-428 phosphoryliert. A B Abbildung 9: Chk2 phosphoryliert GST-CK1-Fusionsproteine Es wurden Kinaseassays mit verschiedenen GST-CK1 Fusionsproteinen oder GST als Substrate und Chk2 als Enzym durchgeführt. Das Coomassie-gefärbte SDS-Gel und das entsprechende Autoradiogramm (Expositionszeit: 17h) sind in A dargestellt. (B) Grafische Darstellung der Cherenkov-Zählung der Proben. Die Abbildung wurde mittels Microsoft Excel erstellt. Die Fehlerbalken beschreiben die Standardabweichung als Maß dafür, wie weit die jeweiligen Werte um den Mittelwert (Durchschnitt) aus den drei Stichproben streuen. Wo die Fehlerbalken fehlen unterschieden sich die einzelnen Werte kaum voneinander. (CK1 Casein Kinase 1 delta, Chk2: Checkpoint Kinase 2, GST: Glutathion-S-transferase, SDS: sodium dodecylsulfate) 3.1.3 Phosphorylierung des C-Terminus von CK1 durch Chk1 Neben Clk2 und Chk2 wurden auch Kinaseassays mit Chk1 durchgeführt, welche sich in der Scansite-Analyse als weitere potenzielle Kinase für die C-terminale Phosphorylierung von CK1 zeigte. In Abbildung 10A ist das mit Coomassie-Blue angefärbte SDS-Gel mit GST und den eingesetzten CK1-Fragmenten, sowie das entsprechende Autoradiogramm 3 Ergebnisse 39 zu sehen. Abbildung 10B zeigt außerdem die Ergebnisse der Radioaktivitätsbestimmung mittels Cherenkov-Zählung in Form eines Balkendiagramms. Das GST-CK1 375-428 Fusionsprotein wird durch Chk1 am stärksten phosphorliert. Die Fusionsproteine GST-CK1 305-375, GST-CK1 305-343 und GST-CK1 305-428 stellen zudem auch gute Substrate für Chk1 dar. Die Phosphorylierung der übrigen Fragmente ist hingegen schwach und liegt im Bereich derjenigen von GST, welches als Kontrollfragment verwendet wurde. Da v.a. GST-CK1 375-428, T397A keinen bedeutenden Phosphateinbau aufweist, deutet das Ergebnis darauf hin, dass vorwiegend Threonin an der Position 397 in CK1 durch Chk1 phosphoryliert wird. A B Abbildung 10: Phosphateinbau in CK1-GST-Fusionsproteine durch Chk1 Es wurden Kinaseassays mit verschiedenen C-terminalen Fragmenten von CK1 oder GST als Substrate und Chk1 als Kinase durchgeführt. Das Coomassie-gefärbte SDS-Gel und das entsprechende Autoradiogramm sind in (A) (Expositionszeit: 17 h) dargestellt, die grafische Auswertung nach Cherenkov-Zählung der Proben in (B). Threonin 397 wird bevorzugt von Chk1 phosphoryliert. Die Abbildung wurde mittels Microsoft Excel erstellt. Die Fehlerbalken beschreiben die Standardabweichung als Maß dafür, wie weit die jeweiligen Werte um den Mittelwert (Durchschnitt) aus den drei Stichproben streuen. Wo die Fehlerbalken fehlen unterschieden sich die einzelnen Werte kaum voneinander. (CK1 Casein Kinase 1 delta, Chk1: Checkpoint Kinase 1, GST: Glutathion-S-transferase, SDS: sodium dodecylsulfat) 3.2 Zweidimensionale Phosphopeptidanalyse von GST-CK1 - Fusionsproteinen nach Phosphorylierung durch Chk1 in vitro Die durchgeführten in vitro Kinaseassays mit Fragmenten unterschiedlicher Länge, zum Teil mit Mutationen in der Aminosäuresequenz, deuteten darauf hin, dass Chk1 Phosphorylierungsstellen von CK1 im Bereich der C-terminalen Aminosäuren 305-375, bzw. 375-428 phosphoryliert. 3 Ergebnisse 41 Zur Verifizierung der Annahme, dass es sich bei dem zusätzlich vorhandenen Peptid von GST-CK1 323-428, T323A, T329A, S370A (s. Pfeil in Abb. 11) um das Serin an Position 328 handelt, welches in der Phosphopeptidkartierung von GST-CK1 323-428, T323A, S328A, S370A fehlt, wurde eine Phosphoaminosäureanalyse dieses zusätzlichen Peptids durchgeführt (s. Material und Methoden 2.2.1.7). Zu diesem Zweck wurde dieses zusätzliche Peptid aus GST-CK1 323-428, T323A, T329A, S370A aus der Dünnschichtzellulose isoliert und wie in Material und Methoden beschrieben in 6 N HCl hydrolysiert. Die hydrolysierten Aminosäuren wurden zusammen mit Phosphoaminosäuremarkern zweidimensional elektrophoretisch aufgetrennt. Die Position der drei Phosphoaminosäuremarker wurde nach Ninhidrinfärbung sichtbar, die der 32 P-markierten Phosphoaminosäuren nach Entwicklung des Autoradiogramms. Als Ergebnis der durchgeführten Phosphoaminosäureanalyse mit dem ausgekratzten P-Peptid aus GST-CK1 323-428, T323A, T329A, S370A zeigt sich ein Signal auf der verwendeten Dünnschichtchromatographieplatte, welches anhand seiner Position nicht eindeutig zu identifizieren ist, jedoch am ehesten phosphoryliertem Serin entspricht (s. Abb. 12). Das Ergebnis dieser Phosphoaminosäureanalyse bestätigte somit die Vermutung, dass Serin 328 von CK1 durch Chk1 phosphoryliert wird. Abbildung 12: Phosphoaminosäureanalyse des Peptids aus der zweidimensionale Phosphopetidanalyse mit GST-CK1 323-428, T323A, T329A, S370A (s. Abb. 11, betreffendes Peptid mit Pfeil markiert) Die hydrolysierten Phosphopeptide wurden in 6 N HCl hydrolysiert und zusammen mit Phosphoaminosäuremarkern (Phosphoserin, -threonin, und tyrosin) zweidimensional bei pH 1,9 und pH 3,6 elektrophoretisch aufgetrennt. Die Phosphoaminosäuremarker wurden nach Anfärbung mit Ninhydrin sichtbar, die der 32P phosphorylierten Aminosäure nach Entwicklung des Autoradigramms. Hierbei zeigt 3 Ergebnisse 44 Abbildung 14: Zweidimensionale Phosphopetidanalyse des GST-CK1 375-428-C-terminalen Fragmentes nach in vitro-Phosphorylierung durch Chk1 Abgebildet sind die schematische Darstellung der Peptide (A) und die jeweiligen Autoradiogramme (B) nach zweidimensionaler Auftrennung der tryptisch gespaltenen Phosphopeptide von GST-CK1 375-428 und GSTCK1 375-428, T397A. Die Phosphopeptide wurden zunächst elektrophoretisch bei pH 1,9 und dann chromatographisch zweidimensional aufgetrennt. Die Auftrageorte sind durch Kreuze dargestellt, (-) und (+) kennzeichnen Kathode und Anode. (Chk1: Checkpoint Kinase 1, CK1 Casein Kinase 1 delta, GST: Glutathion-S-transferase) 3.3 Generierung einer GST-CK1 T397A-Mutante Wie die Vorversuche gezeigt haben, scheint die Phosphorylierung von Threonin 397 einen bedeutenden Einfluss auf die Substratphosphorylierung durch CK1 zu haben. Dies zeigte sich in einer erniedrigten Substratphosphorylierung von GST-p531-64 durch CK1 nachdem CK1 durch Chk1 phosphoryliert worden war. Ein Ziel dieser Arbeit war nun, mit Hilfe der Site-directed Mutagenesis eine GSTCK1 T397A-Mutante zu generieren, um deren kinetischen Eigenschaften mit denjenigen von GST-CK1 zu vergleichen (siehe Material und Methoden 2.2.3.1). Hierfür wurde eine PCR durchgeführt und die daraus resultierende, amplifizierte DNA nach Verdau mit DpnI in kompetente Bakterien transformiert. Anschließend wurde Plasmid-DNA aus mehreren Klonen isoliert und von GATC Biotech sequenziert. Bei den Sequenzierungen zeigte sich der erwünschte Basenaustausch, der zur Substitution von Threonin 397 zu Alanin (T397A) führt. In weiteren in vitro Experimenten soll CK1 T397A nun bezüglich seiner kinetischen Eigenschaften mit denjenigen von GST-CK1 verglichen werden. 3.4 Charakterisierung der in vitro Kinaseaktivität von GST- CK1 T397A Mit Hilfe der zwei Parameter Vmax und Km lässt sich bestimmen, wie sich die katalytische Aktivität eines bestimmten Enzyms in Bezug auf ein ausgewähltes Substrat verhält: 1.) Vmax beschreibt die maximale Reaktionsgeschwindigkeit im Verhältnis zu einer, den Sättigungsbereich anstrebenden, Substratkonzentration. 2.) Km beschreibt die Affinität eines Enzyms zu seinem Substrat, wobei ein höherer Km-Wert eine niedrigere Affinität bedeutet, sowie umgekehrt ein niedrigerer Km-Wert eine höhere Affinität. 3 Ergebnisse 45 Als Charakteristikum der Enzymkinetik gilt das Sättigungsverhalten. Dies bedeutet, dass im Falle sehr hoher Substratkonzentrationen die Umsatzgeschwindigkeit v nicht weiter gesteigert werden kann, also ein Wert Vmax erreicht wird. Die Sättigungsfunktion wird durch Einsetzen der Parameter Km und Vmax folgendermaßen formuliert: v = (Vmax*[S])/(Km+[S]). Km entspricht 0,5*Vmax. Um die Bedeutung von T397 für die katalytische Aktivität von CK1δ zu untersuchen, wurden in vitro Kinaseassays durchgeführt, in denen entweder gleiche Mengen von GSTCK1δ 1-428 (wtCK1δ) oder von GST-CK1δ verschiedene Konzentrationen von GST-p53 1-64 1-428, T397A (mutCK1δ) als Enzym und , welches die ersten 64 Aminosäuren von murinem p53 enthält, als Substrat eingesetzt wurden. Die Durchführung der Kinasierungsreaktionen wurde in Material und Methoden unter 2.2.1.4 beschrieben. Wie aus Abbildung 15 ersichtlich wird, besitzt das CK1δ Vergleich zum CK1δ 1-428 1-428, T397A Fusionsprotein im Fusionsprotein einen erhöhten Vmax-Wert (657,2 cpm), sowie einen erhöhten Km-Wert (0,3524 µM). In Bezug auf Vmax ist die Mutante 5,46-mal so aktiv wie wtCK1δ. Diese Unterschiede zwischen Wildtyp und der mutanten Kinase bedeuten, dass die Mutante (GST-CK1δ 1-428, T397A ) im Vergleich zum Wildtyp (GST-CK1δ 1-428 ) eine geringere Affinität besitzt CK1δ-Substrate zu phosphorylieren. Wie diese Ergebnisse belegen spielt T397 eine bedeutende Rolle bei der Regulation der CK1δ-Aktivität in vitro. 3 Ergebnisse 46 Abbildung 15: Michaelis-Menten-Kinetik CK1 wt vs. CK1 T397A Auf der y-Achse ist die Kinaseaktivität in Prozent dargestellt, die x-Achse zeigt die Substratkonzentration von GST-p53 1-64 in µM. Verglichen wurde die Kinaseaktivität vom Wildtyp (GST-CK1δwt), in schwarz dargestellt, und der Mutanten (GST-CK1 ), mit rot markiert. (Vmax: Maximale Enzymgeschwindigkeit, Km: Michaelis-Menten Konstante, S: Substrat, CK1 Casein Kinase 1 delta, GST: Glutathion-S-transferase) 4 Diskussion 47 4 Diskussion Da CK1 in die Regulation zahlreicher physiologischer Prozesse eingebunden ist (Gross und Anderson 1998; Knippschild et al. 2005), muss davon ausgegangen werden, dass die Aktivität von CK1 selbst strengen Kontrollmechanismen unterworfen ist. Diese Kontrolle geschieht auf unterschiedlichen Ebenen, von besonderer Bedeutung sind posttranslationale Modifikationen auf der Proteinebene. So konnte gezeigt werden, dass, neben der Autophosphorylierung, die Phosphorylierung von S370 durch die Proteinkinase A (PKA) die Aktivität von CK1 in vitro und in vivo beeinflusst (Giamas et al. 2007). Ziel dieser Arbeit war es daher, weitere zelluläre Kinasen zu identifizieren, die CK1 im C-Terminus phosphorylieren, und deren Phosphorylierungsstellen auf CK1 zu identifizieren. Die wichtigsten Ergebnisse dieser Arbeit sind: 1. Chk1 und Clk2 phosphorylieren GST-CK1 Fusionsproteine, allerdings werden auch Phosphorylierungsstellen im GST Anteil der Fusionsproteine phosphoryliert 2. Mit Hilfe von in vitro Kinaseassays und zweidimensionalen Phosphopeptidanalysen konnten Aminosäurereste im C-Terminus von CK1 identifiziert werden, die durch Chk1 phosphoryliert werden. Hierbei handelt es sich um Serin 328 und Threonin 397. Mehrere aktuelle Studien kamen zu dem Ergebnis, dass überwiegend eine Phosphorylierung von Serin (83%) beobachtet werden kann, gefolgt von Threonin (15%) und Tyrosin (2%) (Huttlin et al. 2010; ähnliche Beobachtungen machten auch Olsen et al. 2006). Diese Ergebnisse unterstreichen eine starke Bevorzugung von Serin bei der Phosphorylierung. Auch in dieser Arbeit konnte ein Serin an Position 328 im C-Terminus von CK1 als mögliche Phosphorylierungsstelle identifiziert werden, was die besondere Bedeutung von Serin als Phosphorylierungsstelle bestätigt. 4 Diskussion 48 Im Nachfolgenden werden nun die einzelnen Ergebnisse detailliert diskutiert. Mit Hilfe des „Scansite“-Programms (www.scansite.edu) wurden zunächst Kinasen ermittelt, die potenziell in der Lage sind, CK1 in der C-terminalen Domäne zu phosphorylieren. Das „Scansite“-Programm überprüft die Proteinsequenz von CK1 auf das Vorhandensein von bekannten Konsensusmotiven für zahlreiche Proteinkinasen aus Datenbanken (Obenauer et al. 2003). In diesem Zusammenhang ist es jedoch wichtig zu bedenken, dass die von diesem Programm ausgegebenen Phosphorylierungsstellen rein hypothetischen Charakter haben und nicht zwingend tatsächlich in vitro oder in vivo phosphoryliert werden müssen. Dennoch bedeuten diese Vorhersagen eine hilfreiche Unterstützung für die Planung neuer Experimente, um mögliche Kinase- Substratbeziehungen zu untersuchen. Mit Hilfe der durchgeführten in vitro Kinaseassays konnten wir zeigen, dass Chk1, Chk2 und Clk2 in der Lage sind, CK1 in vitro C-Terminal zu phosphorylieren. Kontrollexperimente, in denen GST als Substrat eingesetzt wurde, ergaben, dass alle drei Kinasen auch GST phosphorylieren können. Der Anteil der Phosphorylierung im GSTAnteil nach Phosphorylierung durch zelluläre Kinasen beträgt 10% bei Chk1, 40% bei Clk2 bzw. 50% bei Chk2. Nach Phosphorylierung von CK1 durch Clk2 wurden vier Fusionsproteine (GST-CK1 305-375 , GST-CK1 305-357 , GST-CK1 305-343 und GST-CK1 375-428 ) im Vergleich zu GST um das 1,8 - 2,5 fache stärker phosphoryliert als GST. Unter Beachtung des Phosphorylierungsgrades der GST-CK1 Phosphorylierungsstellen aufweisen, Substrate, ergeben sich die Mutationen Hinweise darauf, an dass den Clk2 möglicherweise Threonin 397, Serin 370, sowie einen Serin- oder Threoninrest im Bereich der Aminosäuren 305-343 phosphoryliert. Weitere Untersuchungen sind jedoch notwendig, um die durch Clk2 phosphorylierten Aminosäuren im C-terminalen Bereich von CK1 zu identifizieren. So müsste u.a. auf ein anderes Expressionssystem umgestellt werden, z.B. könnten die CK1δ-Fragmente nach vorhergehender Umklonierung als His-getaggte Proteine exprimiert und aufgereinigt werden. Alternativ könnte durch die Abspaltung des GST-Anteils das Problem der Phosphorylierung in GST umgangen werden. Unsere Untersuchungen zeigten zudem, dass CK1 durch Chk1 im C-terminalen Bereich phosphoryliert wird. Durch frühere Versuche war bereits bekannt, dass der C-terminale 4 Diskussion 49 Anteil von CK1 Phosphorylierungsstellen beinhaltet. So war das Serin an Position 370 schon in vorangegangenen Experimenten als Phoshorylierungsstelle von PKA identifiziert worden (Giamas et al. 2007). Durch den Einsatz verschiedener GST-CK Fusionsproteine konnte die Lage der durch Chk1 phosphorylierten Aminosäurereste auf bestimmte Bereiche eingegrenzt bzw. identifiziert werden. So phosphoryliert Chk1 CK1 zwischen den Aminosäuren 323 bis 428. Dies zeigten zweidimensionale Phosphopeptidanalysen mit den Fusionsproteinen GST-CK1 323-428, T323A, S328A, S370A und GST-CK1 323-428, T323A, T329A, S370A . Der Vergleich der Phosphopeptidkartierungen zeigt, dass bei der Phosphopeptidkartierung von GST-CK1 323-428, T323A, S328A, S370A ein positiv geladenes Peptid fehlt (Abb. 11). Aufgrund der unterschiedlichen Mutationen liegt nahe, dass es sich bei dem fehlenden Peptid um jenes handelt, welches Serin an Position 328 beinhaltet. Um diese Vermutung zu verifizieren, wurde eine Phosphoaminosäurenanalyse jenes Phosphopeptids des Fragments GST-CK1 323-428, T323A, T329A, S370A durchgeführt (Abb. 12). Das Ergebnis dieser Phosphoaminosäurenanalyse unterstützt die Vermutung, dass das fehlende Peptid in der Phosphopeptidkartei von GST-CK1 323-428, Chk1 Serin 328 enthält, welches in GST-CK1 323-428, T323A, S328A, S370A T323A, S328A, S370A und zur Phosphorylierung durch Chk1 zur Verfügung steht. Eine zusätzliche zweidimensionale Phosphopeptidkartierung mit den Fusionsproteinen GST-CK1 305-375 und GST-CK1 305-375, S328A und dem Enzym Chk1 wurde zur Bestätigung der Phosphorylierungsstelle an Position 328 erstellt. Die Phosphorylierung von GST-CK1 305-375, S328A fiel durch die Verkürzung im Vergleich zum vollen Cterminalen Bereich von CK1 (305-428) generell etwas schwächer aus. Auch hier ging ein Hauptpeptid verloren, wenn Serin 328 zu Alanin ausgetauscht wurde. Dies ist ein weiterer Hinweis dafür, dass Serin 328 eine bedeutende Phosphorylierungsstelle von Chk1 im CTerminus von CK1 darstellt. 4 Diskussion 50 Um die Rolle der Phosphorylierung in der Manifestierung physiologischer Unterschiede zwischen verschieden Geweben besser zu verstehen, wurden vor kurzer Zeit massenspektrometrische Untersuchungen von neun unterschiedlichen Gewebearten der Maus (Gehirn, braunes Fettgewebe, Herz, Leber, Lunge, Niere, Pankreas, Milz und Hoden) mittels LC-MS/MS durchgeführt. Hierbei konnten 12039 Proteine identifiziert werden, hierunter 6296 Phosphoproteine mit nahezu 36000 Phosphorylierungsstellen (Huttlin et al. 2010). Unter anderem konnte bei dieser in vivo Studie auch Serin 328 als potenzielle in vivo Phosphorylierungsstelle auf dem CK1 Molekül nachgewiesen werden, was die Bedeutung von Serin 328 für die Regulation der Funktionen von CK1 in der Zelle unterstreicht. In einer Studie von 2008 sollte zudem untersucht werden, ob hochauflösende massenspektrometrische Methoden (LC-MS/MS) auch bei soliden Tumoren angewendet werden können. Hierfür wurden TG3 mutierte Mäuse mit Melanomen der Haut untersucht. Die Experimente führten zur Identifizierung von mehr als 5600 Phosphorylierungsstellen auf 2250 Proteinen. Von allen Phosphorylierungsstellen entfielen 5120 (90%) auf Serin, 542 (9%) auf Threonin und 36 (1%) auf Tyrosin (Zanivan et al. 2008). Diese festgestellten Mengenverhältnisse sind ähnlich zu denen, welche erst kürzlich für eine humane Krebszelllinie veröffentlicht worden sind (Olsen et al. 2006). Viele davon spielen eine wichtige Rolle in typischen Melanom-Signalwegen. Auch in dieser in vivo Studie konnte Serin 328 als Phosphorylierungsstelle auf dem CK1 Molekül identifiziert werden. Es gelang außerdem mehrmals die Phosphorylierungsstelle Serin 328 bei in vivo Experimenten in humanem CK1 nachzuweisen (Daub et al. 2008, Dephoure et al. 2008, Oppermann et al. 2009). Inwieweit diese Erkenntnisse nützlich für die zukünftige Entwicklung neuer Therapieschemata für die Krebsbehandlung sind, wird sich erst noch herausstellen müssen. Jedoch unterstreichen diese in vivo Ergebnisse die Relevanz unserer Identifizierung von Serin 328 als in vitro Phosphorylierungsstelle. Um weitere Anhaltspunkte über die potenziellen Phosphorylierungsstellen von Chk1 auf dem CK1 Molekül im Bereich der Aminosäuren von 375 und 428 zu erhalten, wurden zweidimensionale Phosphopeptidanalysen mit den Fusionsproteinen GST-CK1 375-428 und 4 Diskussion 51 GST-CK1 375-428, T397A durchgeführt. Dass das GST-CK1 375-428, T397A Protein durch Chk1 deutlich schwächer phosphoryliert wird, lässt darauf schließen, dass in diesem Bereich vorwiegend Threonin an der Position 397 durch Chk1 phosphoryliert wird. Diese Annahme wurde auch durch die durchgeführten Phosphopeptidkartierungen unterstützt. So wies die Phosphorylierungskartierung von GST-CK1 375-428 nach Phosphorylierung mit Chk1 ein stark phosphoryliertes Peptid auf, welches bei dem Fusionsprotein GST-CK1 375-428, T397A fehlte. Inwiefern es sich hierbei im Vergleich mit dem, über dem Auftragungspunkt Phosphorylierungskartierung von befindlichen GST-CK1 375-428 Peptidmuster um der unterschiedliche Phosphorylierungsstellen handelt, oder ob lediglich eine durch die Mutation T397A bedingte Konformationsänderung zu einem veränderten Phosphorylierungsmuster führt, lässt sich anhand dieses Versuches nicht bestimmen. Das Ergebnis gibt jedoch Grund zur Annahme, dass es sich bei diesem Peptid um jenes Peptid handelt, das Threonin 397 enthält. Das Ergebnis einer Phosphoaminosäurebestimmung dieses Phosphopetids würde diese Vermutung unterstützen. Nach Generierung einer GST-CK1 T397A Mutante wurde die in vitro Kinaseaktivität von GST-CK1 T397A charakterisiert. Um die Bedeutung von Threonin 397 für die katalytische Aktivität von CK1δ zu untersuchen, wurden in vitro Kinaseassays durchgeführt, in denen gleiche Mengen von GST-CK1δ 1-428 (wtCK1δ) oder von GST- CK1δ 1-428, T397A (mutCK1δ) als Enzym und verschiedene Konzentrationen von GST-p53 164 als Substrat eingesetzt wurden. GST-p53 1-64 enthält die ersten 64 Aminosäuren von murinem p53, die potenzielle Phosphorylierungsstellen für CK1 (Serine 4, 6, 9, 15, Threonin 18 und Serine 33 und 37) aufweisen und GST-p53 1-64 als ideales in vitro Substrat für CK1 auszeichnen (Milne et al. 1992). Hierbei zeigte sich, dass das CK1δ 1-428, T397 Fusionsprotein im Vergleich zum CK1δ 1-428 Fusionsprotein sowohl einen erhöhten Vmax-Wert, als auch einen erhöhten Km-Wert besitzt. Hinsichtlich der maximalen Reaktionsgeschwindigkeit Vmax zeigte sich, dass die Mutante 5,46-mal so aktiv ist wie wtCK1δ. Bereits in früheren Studien der Arbeitsgruppe wurde die Bedeutung der Phosphorylierung von CK1, damals durch PKA, aufgezeigt. Die GST-CK1 S370A-Mutante besitzt allerdings im Gegensatz zur GST-CK1 T397A-Mutante in vitro niedrigere Km- und höhere Vmax-Werte 4 Diskussion 52 im Vergleich zu denen von Wildtyp-GST-CK1, ist also aktiver als die Wildtyp-Kinase (Giamas et al. 2007). Anhand dieser Unterschiede zwischen dem Wildtyp und der Mutante ist ersichtlich, dass die Mutante (GST-CK1δ 1-428, T397A ) im Vergleich zum Wildtyp (GST-CK1δ 1-428) eine geringere Affinität besitzt CK1δ-Substrate zu phosphorylieren. Diese Ergebnisse geben erste Hinweise darauf, dass Threonin 397 eine bedeutende Rolle bei der Regulation der CK1δ-Aktivität durch Chk1 in vitro einnehmen könnte. Im Jahr 2008 wurde mit Hilfe quantitativer Massenspektrometrie die Regulation des Zellzyklus bei Proteinkinasen untersucht. Dieses Vorgehen ermöglichte die Bestimmung von 219 Proteinkinasen von in S- und M-Phase des Zellzyklus angehaltenen humanen Krebszellen (HeLa S3). Es konnten mehr als 1000 Phosphorylierungsstellen auf diesen Proteinkinasen identifiziert warden. Auch Threonin 397 konnte in diesem in vivo Experiment als Phosphorylierungsstelle auf der humanen Isoform 2 des CK1-Proteins nachgewiesen werden (Daub et al. 2008). Diese in vivo Ergebnisse unterstreichen die Relevanz der von uns in vitro nachgewiesenen Phosphorylierungsstelle Threonin 397 für Vorgänge in der Zelle. Zusammenfassend konnte in dieser Arbeit somit bestätigt werden, dass CK1δ ein in vitro Substrat für Chk1 darstellt und durch Chk1 an Serin 328, sowie Threonin 397 phosphoryliert werden kann. Außerdem konnte nachgewiesen werden, dass der Phosphorylierungsstatus von Threonin 397 eine wichtige Rolle bei der Regulation der CK1δ-Aktivität spielt. Ferner konnten zusätzliche Kinasen, Chk2 und Clk2, identifiziert werden, welche ebenfalls in der Lage sind, CK1δ C-Terminal zu phosphorylieren. Die physiologische Bedeutung der Phosphorylierung von CK1δ durch Chk1 ist jedoch bislang noch ungeklärt und bedarf weiterer Nachforschung. Phosphorylierungs-spezifische Antikörper gegen CK1, das an Serin 328 bzw. Threonin 397 phosphoryliert ist, könnten für Nachforschungen bezüglich der Lokalisation und des Aktivitätsstatus in Abhängigkeit des Aktivitätsstatus von Chk1 eingesetzt werden. Dies würde weitere Analysen der Beeinflussung von CK1δ durch Chk1 in einem auf Zellkultur basierten Modell ermöglichen. 4 Diskussion 53 Für eine gezielte Modulation der CK1-Aktivität und ein tiefgreifendes Verständnis der Beteiligung von CK1 in Chk1-vermittelten Signalwegen wäre die Aufklärung des physiologischen Zusammenspiels von Chk1 und CK1 von großer Bedeutung. Da deregulierte Expression bzw. Aktivität von CK1 Isoformen an der Entstehung bestimmter Erkrankungen beteiligt sein kann, wären die gewonnenen Erkenntnisse ebenfalls bedeutend hinsichtlich der Entwicklung neuer Therapiekonzepte. 5 Zusammenfassung 54 5 Zusammenfassung Casein Kinase 1 delta (CK1 ist an der Regulation zahlreicher zellulärer Prozesse beteiligt. Dies bedingt, dass die CK1-Aktivität durch verschiedene Mechanismen reguliert werden muss. Auf Proteinebene nimmt die reversible Phosphorylierung eine wichtige Rolle für die Regulation der CK1 Aktivität ein, denn sowohl die Autophosphorylierung als auch zielgerichtete Phosphorylierung durch zelluläre Kinasen können die CK1Aktivität modulieren. Ziel dieser Arbeit war es daher, weitere zelluläre Kinasen zu identifizieren, die CK1 in der C-terminalen Domäne phosphorylieren. In vitro Kinasisierungsreaktionen, in denen GST-CK1-Fusionsproteine als Substrate eingesetzt wurden, ergaben, dass Chk1 und Clk2 CK1 in vitro phosphorylieren. Durch Verwendung von verschiedenen GST-Fusionsproteinen, die Mutationen an putativen Chk1 Phosphorylierungsstellen aufweisen, in in vitro Kinasierungen, zweidimensionalen Phosphopeptidanalysen und Phosphoaminosäurenbestimmungen konnten Serin 328, Serin 370 und Threonin 397 als Targets für Chk1 identifiziert werden. Die durchgeführten Michaelis-Menten Kinetiken zeigten zudem, dass GST-CK1δ 1-428, T397 eine höhere Affinität zu GST-p531-64 aufweist als GST-CK1δ 1-428. Die durchgeführten Arbeiten führten zur Identifizierung von drei zellulären Kinasen, die CK1 in der C-terminalen Domäne phosphorylieren. Für Chk1 konnten gezeigt werden, dass Serin 328, Serin 370 und Threonin 397 phosphoryliert werden. Weiterführende Untersuchungen sind jedoch notwendig, um die physiologische Relevanz der Chk1 vermittelten Phosphorylierung von CK1 zu charakterisieren. 6 Literaturverzeichnis 55 6 Literaturverzeichnis 1. Amanchy R, Kandasamy K, Mathivanan S, Periaswamy B, Reddy R, Yoon WH, Joore J, Beer MA, Cope L, Pandey A: Identification of Novel Phosphorylation Motifs Through an Integrative Computational and Experimental Analysis of the Human Phosphoproteome. J Proteomics Bioinform 4: 22-35 (2011) 2. Amit S, Hatzubai A, Birman Y, Andersen JS, Ben-Shushan E, Mann M, BenNeriah Y, Alkalay I: Axin-mediated CKI phosphorylation of beta-catenin at Ser 45: a molecular switch for the Wnt pathway. Genes Dev 16: 1066-76 (2002) 3. Badura L, Swanson T, Adamowicz W, Adams J, Cianfrogna J, Fisher K, Holland J, Kleiman R, Nelson F, Reynolds L, St Germain K, Schaeffer E, Tate B, Sprouse J: An inhibitor of casein kinase I epsilon induces phase delays in circadian rhythms under free-running and entrained conditions. J Pharmacol Exp Ther 322: 730-8 (2007) 4. Bazenet CE, Brockman JL, Lewis D, Chan C, Anderson RA: Erythroid membranebound protein kinase binds to a membrane component and is regulated by phosphatidylinositol 4,5-bisphosphate. J Biol Chem 265: 7369-76 (1990) 5. Bischof J, Leban J, Zaja M, Grothey A, Radunsky B, Othersen O, Strobl S, Vitt D, Knippschild U: 2-Benzamido-N-(1H-benzo[d]imidazol-2-yl)thiazole-4- carboxamide derivatives as potent inhibitors of CK1delta/epsilon. Amino Acids 43: 1577-91 (2012) 6. Bosenberg MW, Massague J: Juxtacrine cell signaling molecules. Curr Opin Cell Biol 5: 832-8 (1993) 7. Brockman JL, Anderson RA: Casein kinase I is regulated by phosphatidylinositol 4,5-bisphosphate in native membranes. J Biol Chem 266: 2508-12 (1991) 8. Brockschmidt C, Hirner H, Huber N, Eismann T, Hillenbrand A, Giamas G, Radunsky B, Ammerpohl O, Bohm B, Henne-Bruns D, Kalthoff H, Leithauser F, Trauzold A, Knippschild U: Anti-apoptotic and growth-stimulatory functions of CK1 delta and epsilon in ductal adenocarcinoma of the pancreas are inhibited by IC261 in vitro and in vivo. Gut 57: 799-806 (2008) 9. Carmel G, Leichus B, Cheng X, Patterson SD, Mirza U, Chait BT, Kuret J: Expression, purification, crystallization, and preliminary x-ray analysis of casein kinase-1 from Schizosaccharomyces pombe. J Biol Chem 269: 7304-9 (1994) 6 Literaturverzeichnis 10. 56 Cegielska A, Gietzen KF, Rivers A, Virshup DM: Autoinhibition of casein kinase I epsilon (CKI epsilon) is relieved by protein phosphatases and limited proteolysis. J Biol Chem 273: 1357-64 (1998) 11. Cheong JK, Nguyen TH, Wang H, Tan P, Voorhoeve PM, Lee SH, Virshup DM: IC261 induces cell cycle arrest and apoptosis of human cancer cells via CK1delta/varepsilon and Wnt/beta-catenin independent inhibition of mitotic spindle formation. Oncogene 30: 2558-69 (2011) 12. Chijiwa T, Hagiwara M, Hidaka H: A newly synthesized selective casein kinase I inhibitor, N-(2-aminoethyl)-5-chloroisoquinoline-8-sulfonamide, and affinity purification of casein kinase I from bovine testis. J Biol Chem 264: 4924-7 (1989) 13. Cobb MH, Rosen OM: Description of a protein kinase derived from insulin-treated 3T3-L1 cells that catalyzes the phosphorylation of ribosomal protein S6 and casein. J Biol Chem 258: 12472-81 (1983) 14. Cohen P: The origins of protein phosphorylation. Nat Cell Biol 4: E127-30 (2002) 15. Cruciat CM, Dolde C, de Groot RE, Ohkawara B, Reinhard C, Korswagen HC, Niehrs C: RNA Helicase DDX3 Is a Regulatory Subunit of Casein Kinase 1 in Wnt/beta-Catenin Signaling. Science;10.1126/science.1231499: epublished (2013) 16. Daub H, Olsen JV, Bairlein M, Gnad F, Oppermann FS, Korner R, Greff Z, Keri G, Stemmann O, Mann M: Kinase-selective enrichment enables quantitative phosphoproteomics of the kinome across the cell cycle. Mol Cell 31: 438-48 (2008) 17. Dephoure N, Zhou C, Villen J, Beausoleil SA, Bakalarski CE, Elledge SJ, Gygi SP: A quantitative atlas of mitotic phosphorylation. Proc Natl Acad Sci U S A 105: 10762-7 (2008) 18. Desagher S, Osen-Sand A, Montessuit S, Magnenat E, Vilbois F, Hochmann A, Journot L, Antonsson B, Martinou JC: Phosphorylation of bid by casein kinases I and II regulates its cleavage by caspase 8. Mol Cell 8: 601-11 (2001) 19. Dumaz N, Milne DM, Meek DW: Protein kinase CK1 is a p53-threonine 18 kinase which requires prior phosphorylation of serine 15. FEBS Lett 463: 312-6 (1999) 20. Elias L, Li AP, Longmire J: Cyclic adenosine 3':5'-monophosphate-dependent and independent protein kinase in acute myeloblastic leukemia. Cancer Res 41: 2182-8 (1981) 6 Literaturverzeichnis 21. 57 Elyada E, Pribluda A, Goldstein RE, Morgenstern Y, Brachya G, Cojocaru G, SnirAlkalay I, Burstain I, Haffner-Krausz R, Jung S, Wiener Z, Alitalo K, Oren M, Pikarsky E, Ben-Neriah Y: CKIalpha ablation highlights a critical role for p53 in invasiveness control. Nature 470: 409-13 (2011) 22. Fish KJ, Cegielska A, Getman ME, Landes GM, Virshup DM: Isolation and characterization of human casein kinase I epsilon (CKI), a novel member of the CKI gene family. J Biol Chem 270: 14875-83 (1995) 23. Flotow H, Graves PR, Wang AQ, Fiol CJ, Roeske RW, Roach PJ: Phosphate groups as substrate determinants for casein kinase I action. J Biol Chem 265: 14264-9 (1990) 24. Foldynova-Trantirkova S, Sekyrova P, Tmejova K, Brumovska E, Bernatik O, Blankenfeldt W, Krejci P, Kozubik A, Dolezal T, Trantirek L, Bryja V: Breast cancer-specific mutations in CK1epsilon inhibit Wnt/beta-catenin and activate the Wnt/Rac1/JNK and NFAT pathways to decrease cell adhesion and promote cell migration. Breast Cancer Res 12: R30 (2010) 25. Fu Z, Chakraborti T, Morse S, Bennett GS, Shaw G: Four casein kinase I isoforms are differentially partitioned between nucleus and cytoplasm. Exp Cell Res 269: 275-86 (2001) 26. Fuja TJ, Lin F, Osann KE, Bryant PJ: Somatic mutations and altered expression of the candidate tumor suppressors CSNK1 epsilon, DLG1, and EDD/hHYD in mammary ductal carcinoma. Cancer Res 64: 942-51 (2004) 27. Giamas G, Hirner H, Shoshiashvili L, Grothey A, Gessert S, Kuhl M, Henne-Bruns D, Vorgias CE, Knippschild U: Phosphorylation of CK1delta: identification of Ser370 as the major phosphorylation site targeted by PKA in vitro and in vivo. Biochem J 406: 389-98 (2007) 28. Gietzen KF, Virshup DM: Identification of inhibitory autophosphorylation sites in casein kinase I epsilon. J Biol Chem 274: 32063-70 (1999) 29. Graves PR, Haas DW, Hagedorn CH, DePaoli-Roach AA, Roach PJ: Molecular cloning, expression, and characterization of a 49-kilodalton casein kinase I isoform from rat testis. J Biol Chem 268: 6394-401 (1993) 30. Graves PR, Roach PJ: Role of COOH-terminal phosphorylation in the regulation of casein kinase I delta. J Biol Chem 270: 21689-94 (1995) 6 Literaturverzeichnis 31. 58 Green CL, Bennett GS: Identification of four alternatively spliced isoforms of chicken casein kinase I alpha that are all expressed in diverse cell types. Gene 216: 189-95 (1998) 32. Green JE, Shibata MA, Yoshidome K, Liu ML, Jorcyk C, Anver MR, Wigginton J, Wiltrout R, Shibata E, Kaczmarczyk S, Wang W, Liu ZY, Calvo A, Couldrey C: The C3(1)/SV40 T-antigen transgenic mouse model of mammary cancer: ductal epithelial cell targeting with multistage progression to carcinoma. Oncogene 19: 1020-7 (2000) 33. Gross SD, Anderson RA: Casein kinase I: spatial organization and positioning of a multifunctional protein kinase family. Cell Signal 10: 699-711 (1998) 34. Gross SD, Hoffman DP, Fisette PL, Baas P, Anderson RA: A phosphatidylinositol 4,5-bisphosphate-sensitive casein kinase I alpha associates with synaptic vesicles and phosphorylates a subset of vesicle proteins. J Cell Biol 130: 711-24 (1995) 35. Hanks SK, Hunter T: Protein kinases 6. The eukaryotic protein kinase superfamily: kinase (catalytic) domain structure and classification. FASEB J 9: 576-96 (1995) 36. http://scansite.mit.edu (2010) 37. Huart AS, MacLaine NJ, Meek DW, Hupp TR: CK1alpha plays a central role in mediating MDM2 control of p53 and E2F-1 protein stability. J Biol Chem 284: 32384-94 (2009) 38. Huelsken J, Birchmeier W: New aspects of Wnt signaling pathways in higher vertebrates. Curr Opin Genet Dev 11: 547-53 (2001) 39. Hunter T: Protein kinases and phosphatases: the yin and yang of protein phosphorylation and signaling. Cell 80: 225-36 (1995) 40. Hunter T, Plowman GD: The protein kinases of budding yeast: six score and more. Trends Biochem Sci 22: 18-22 (1997) 41. Huttlin EL, Jedrychowski MP, Elias JE, Goswami T, Rad R, Beausoleil SA, Villen J, Haas W, Sowa ME, Gygi SP: A tissue-specific atlas of mouse protein phosphorylation and expression. Cell 143: 1174-89 (2010) 42. Kawakami F, Suzuki K, Ohtsuki K: A novel consensus phosphorylation motif in sulfatide- and cholesterol-3-sulfate-binding protein substrates for CK1 in vitro. Biol Pharm Bull 31: 193-200 (2008) 6 Literaturverzeichnis 43. 59 Knippschild U, Gocht A, Wolff S, Huber N, Lohler J, Stoter M: The casein kinase 1 family: participation in multiple cellular processes in eukaryotes. Cell Signal 17: 675-89 (2005) 44. Knippschild U, Milne DM, Campbell LE, DeMaggio AJ, Christenson E, Hoekstra MF, Meek DW: p53 is phosphorylated in vitro and in vivo by the delta and epsilon isoforms of casein kinase 1 and enhances the level of casein kinase 1 delta in response to topoisomerase-directed drugs. Oncogene 15: 1727-36 (1997) 45. Kuerbitz SJ, Plunkett BS, Walsh WV, Kastan MB: Wild-type p53 is a cell cycle checkpoint determinant following irradiation. Proc Natl Acad Sci U S A 89: 7491-5 (1992) 46. Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, Devon K, Dewar K, Doyle M, FitzHugh W, Funke R, Gage D, Harris K, Heaford A, Howland J, Kann L, Lehoczky J, LeVine R, McEwan P, McKernan K, Meldrim J, Mesirov JP, Miranda C, Morris W, Naylor J, Raymond C, Rosetti M, Santos R, Sheridan A, Sougnez C, Stange-Thomann N, Stojanovic N, Subramanian A, Wyman D, Rogers J, Sulston J, Ainscough R, Beck S, Bentley D, Burton J, Clee C, Carter N, Coulson A, Deadman R, Deloukas P, Dunham A, Dunham I, Durbin R, French L, Grafham D, Gregory S, Hubbard T, Humphray S, Hunt A, Jones M, Lloyd C, McMurray A, Matthews L, Mercer S, Milne S, Mullikin JC, Mungall A, Plumb R, Ross M, Shownkeen R, Sims S, Waterston RH, Wilson RK, Hillier LW, McPherson JD, Marra MA, Mardis ER, Fulton LA, Chinwalla AT, Pepin KH, Gish WR, Chissoe SL, Wendl MC, Delehaunty KD, Miner TL, Delehaunty A, Kramer JB, Cook LL, Fulton RS, Johnson DL, Minx PJ, Clifton SW, Hawkins T, Branscomb E, Predki P, Richardson P, Wenning S, Slezak T, Doggett N, Cheng JF, Olsen A, Lucas S, Elkin C, Uberbacher E, Frazier M, Gibbs RA, Muzny DM, Scherer SE, Bouck JB, Sodergren EJ, Worley KC, Rives CM, Gorrell JH, Metzker ML, Naylor SL, Kucherlapati RS, Nelson DL, Weinstock GM, Sakaki Y, Fujiyama A, Hattori M, Yada T, Toyoda A, Itoh T, Kawagoe C, Watanabe H, Totoki Y, Taylor T, Weissenbach J, Heilig R, Saurin W, Artiguenave F, Brottier P, Bruls T, Pelletier E, Robert C, Wincker P, Smith DR, Doucette-Stamm L, Rubenfield M, Weinstock K, Lee HM, Dubois J, Rosenthal A, Platzer M, Nyakatura G, Taudien S, Rump A, Yang H, Yu J, Wang J, Huang G, Gu J, Hood L, Rowen L, Madan A, Qin S, Davis 6 Literaturverzeichnis 60 RW, Federspiel NA, Abola AP, Proctor MJ, Myers RM, Schmutz J, Dickson M, Grimwood J, Cox DR, Olson MV, Kaul R, Raymond C, Shimizu N, Kawasaki K, Minoshima S, Evans GA, Athanasiou M, Schultz R, Roe BA, Chen F, Pan H, Ramser J, Lehrach H, Reinhardt R, McCombie WR, de la Bastide M, Dedhia N, Blocker H, Hornischer K, Nordsiek G, Agarwala R, Aravind L, Bailey JA, Bateman A, Batzoglou S, Birney E, Bork P, Brown DG, Burge CB, Cerutti L, Chen HC, Church D, Clamp M, Copley RR, Doerks T, Eddy SR, Eichler EE, Furey TS, Galagan J, Gilbert JG, Harmon C, Hayashizaki Y, Haussler D, Hermjakob H, Hokamp K, Jang W, Johnson LS, Jones TA, Kasif S, Kaspryzk A, Kennedy S, Kent WJ, Kitts P, Koonin EV, Korf I, Kulp D, Lancet D, Lowe TM, McLysaght A, Mikkelsen T, Moran JV, Mulder N, Pollara VJ, Ponting CP, Schuler G, Schultz J, Slater G, Smit AF, Stupka E, Szustakowski J, Thierry-Mieg D, Thierry-Mieg J, Wagner L, Wallis J, Wheeler R, Williams A, Wolf YI, Wolfe KH, Yang SP, Yeh RF, Collins F, Guyer MS, Peterson J, Felsenfeld A, Wetterstrand KA, Patrinos A, Morgan MJ, de Jong P, Catanese JJ, Osoegawa K, Shizuya H, Choi S, Chen YJ: Initial sequencing and analysis of the human genome. Nature 409: 860-921 (2001) 47. Lee E, Salic A, Kirschner MW: Physiological regulation of [beta]-catenin stability by Tcf3 and CK1epsilon. J Cell Biol 154: 983-93 (2001) 48. Li M, Lewis B, Capuco AV, Laucirica R, Furth PA: WAP-TAg transgenic mice and the study of dysregulated cell survival, proliferation, and mutation during breast carcinogenesis. Oncogene 19: 1010-9 (2000) 49. Liu C, Li Y, Semenov M, Han C, Baeg GH, Tan Y, Zhang Z, Lin X, He X: Control of beta-catenin phosphorylation/degradation by a dual-kinase mechanism. Cell 108: 837-47 (2002) 50. Liu C, Yao M, Hogue CW: Near-membrane ensemble elongation in the proline-rich LRP6 intracellular domain may explain the mysterious initiation of the Wnt signaling pathway. BMC Bioinformatics 12 Suppl 13: S13 (2011) 51. Long AM, Zhao H, Huang X: Structural basis for the potent and selective inhibition of casein kinase 1 epsilon. J Med Chem 55: 10307-11 (2012) 52. Longenecker KL, Roach PJ, Hurley TD: Three-dimensional structure of mammalian casein kinase I: molecular basis for phosphate recognition. J Mol Biol 257: 618-31 (1996) 6 Literaturverzeichnis 53. 61 Longenecker KL, Roach PJ, Hurley TD: Crystallographic studies of casein kinase I delta toward a structural understanding of auto-inhibition. Acta Crystallogr D Biol Crystallogr 54 ( Pt 3): 473-5 (1998) 54. Longenecker KL, Roach PJ, Hurley TD: Crystallographic studies of casein kinase I delta toward a structural understanding of auto-inhibition. Acta Crystallogr D Biol Crystallogr 54: 473-5 (1998) 55. Ma YN, Hao L, Niu SL, Xu YF, Zhang Y, Pei P, Bu DF, Qi Y: [Five single nucleotide polymorphisms of casein kinase I gamma 2 gene in children with familial febrile convulsions]. Zhonghua Yi Xue Yi Chuan Xue Za Zhi 21: 347-50 (2004) 56. Manning G, Whyte DB, Martinez R, Hunter T, Sudarsanam S: The protein kinase complement of the human genome. Science 298: 1912-34 (2002) 57. Marin O, Bustos VH, Cesaro L, Meggio F, Pagano MA, Antonelli M, Allende CC, Pinna LA, Allende JE: A noncanonical sequence phosphorylated by casein kinase 1 in beta-catenin may play a role in casein kinase 1 targeting of important signaling proteins. Proc Natl Acad Sci U S A 100: 10193-200 (2003) 58. Marine JC, Francoz S, Maetens M, Wahl G, Toledo F, Lozano G: Keeping p53 in check: essential and synergistic functions of Mdm2 and Mdm4. Cell Death Differ 13: 927-34 (2006) 59. Mashhoon N, DeMaggio AJ, Tereshko V, Bergmeier SC, Egli M, Hoekstra MF, Kuret J: Crystal structure of a conformation-selective casein kinase-1 inhibitor. J Biol Chem 275: 20052-60 (2000) 60. Masuda K, Ono M, Okamoto M, Morikawa W, Otsubo M, Migita T, Tsuneyoshi M, Okuda H, Shuin T, Naito S, Kuwano M: Downregulation of Cap43 gene by von Hippel-Lindau tumor suppressor protein in human renal cancer cells. Int J Cancer 105: 803-10 (2003) 61. Matsubayashi H, Sese S, Lee JS, Shirakawa T, Iwatsubo T, Tomita T, Yanagawa S: Biochemical characterization of the Drosophila wingless signaling pathway based on RNA interference. Mol Cell Biol 24: 2012-24 (2004) 62. McKay RM, Peters JM, Graff JM: The casein kinase I family: roles in morphogenesis. Dev Biol 235: 378-87 (2001) 6 Literaturverzeichnis 63. 62 Meggio F, Perich JW, Marin O, Pinna LA: The comparative efficiencies of the Ser(P)-, Thr(P)- and Tyr(P)-residues as specificity determinants for casein kinase-1. Biochem Biophys Res Commun 182: 1460-5 (1992) 64. Meggio F, Perich JW, Reynolds EC, Pinna LA: A synthetic beta-casein phosphopeptide and analogues as model substrates for casein kinase-1, a ubiquitous, phosphate directed protein kinase. FEBS Lett 283: 303-6 (1991) 65. Milne DM, Looby P, Meek DW: Catalytic activity of protein kinase CK1 delta (casein kinase 1delta) is essential for its normal subcellular localization. Exp Cell Res 263: 43-54 (2001) 66. Obenauer JC, Cantley LC, Yaffe MB: Scansite 2.0: Proteome-wide prediction of cell signaling interactions using short sequence motifs. Nucleic Acids Res 31: 3635-41 (2003) 67. Okamura H, Garcia-Rodriguez C, Martinson H, Qin J, Virshup DM, Rao A: A conserved docking motif for CK1 binding controls the nuclear localization of NFAT1. Mol Cell Biol 24: 4184-95 (2004) 68. Olsen JV, Blagoev B, Gnad F, Macek B, Kumar C, Mortensen P, Mann M: Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell 127: 635-48 (2006) 69. Oppermann FS, Gnad F, Olsen JV, Hornberger R, Greff Z, Keri G, Mann M, Daub H: Large-scale proteomics analysis of the human kinome. Mol Cell Proteomics 8: 1751-64 (2009) 70. Oren M: Relationship of p53 to the control of apoptotic cell death. Semin Cancer Biol 5: 221-7 (1994) 71. Oumata N, Bettayeb K, Ferandin Y, Demange L, Lopez-Giral A, Goddard ML, Myrianthopoulos V, Mikros E, Flajolet M, Greengard P, Meijer L, Galons H: Roscovitine-derived, dual-specificity inhibitors of cyclin-dependent kinases and casein kinases 1. J Med Chem 51: 5229-42 (2008) 72. Peifer C, Abadleh M, Bischof J, Hauser D, Schattel V, Hirner H, Knippschild U, Laufer S: 3,4-Diaryl-isoxazoles and -imidazoles as potent dual inhibitors of p38alpha mitogen activated protein kinase and casein kinase 1delta. J Med Chem 52: 7618-30 (2009) 6 Literaturverzeichnis 73. 63 Peifer M, Polakis P: Wnt signaling in oncogenesis and embryogenesis--a look outside the nucleus. Science 287: 1606-9 (2000) 74. Perez DI, Gil C, Martinez A: Protein kinases CK1 and CK2 as new targets for neurodegenerative diseases. Med Res Rev 31: 924-54 (2011) 75. Rena G, Bain J, Elliott M, Cohen P: D4476, a cell-permeant inhibitor of CK1, suppresses the site-specific phosphorylation and nuclear exclusion of FOXO1a. EMBO Rep 5: 60-5 (2004) 76. Rodriguez N, Yang J, Hasselblatt K, Liu S, Zhou Y, Rauh-Hain JA, Ng SK, Choi PW, Fong WP, Agar NY, Welch WR, Berkowitz RS, Ng SW: Casein kinase I epsilon interacts with mitochondrial proteins for the growth and survival of human ovarian cancer cells. EMBO Mol Med 4: 952-63 (2012) 77. Rowles J, Slaughter C, Moomaw C, Hsu J, Cobb MH: Purification of casein kinase I and isolation of cDNAs encoding multiple casein kinase I-like enzymes. Proc Natl Acad Sci U S A 88: 9548-52 (1991) 78. Rubin GM, Yandell MD, Wortman JR, Gabor Miklos GL, Nelson CR, Hariharan IK, Fortini ME, Li PW, Apweiler R, Fleischmann W, Cherry JM, Henikoff S, Skupski MP, Misra S, Ashburner M, Birney E, Boguski MS, Brody T, Brokstein P, Celniker SE, Chervitz SA, Coates D, Cravchik A, Gabrielian A, Galle RF, Gelbart WM, George RA, Goldstein LS, Gong F, Guan P, Harris NL, Hay BA, Hoskins RA, Li J, Li Z, Hynes RO, Jones SJ, Kuehl PM, Lemaitre B, Littleton JT, Morrison DK, Mungall C, O'Farrell PH, Pickeral OK, Shue C, Vosshall LB, Zhang J, Zhao Q, Zheng XH, Lewis S: Comparative genomics of the eukaryotes. Science 287: 2204-15 (2000) 79. Sakanaka C: Phosphorylation and regulation of beta-catenin by casein kinase I epsilon. J Biochem 132: 697-703 (2002) 80. Santos JA, Logarinho E, Tapia C, Allende CC, Allende JE, Sunkel CE: The casein kinase 1 alpha gene of Drosophila melanogaster is developmentally regulated and the kinase activity of the protein induced by DNA damage. J Cell Sci 109 ( Pt 7): 1847-56 (1996) 81. Schuler M, Green DR: Mechanisms of p53-dependent apoptosis. Biochem Soc Trans 29: 684-8 (2001) 6 Literaturverzeichnis 82. 64 Sillibourne JE, Milne DM, Takahashi M, Ono Y, Meek DW: Centrosomal anchoring of the protein kinase CK1delta mediated by attachment to the large, coiled-coil scaffolding protein CG-NAP/AKAP450. J Mol Biol 322: 785-97 (2002) 83. Sporn MB, Todaro GJ: Autocrine secretion and malignant transformation of cells. N Engl J Med 303: 878-80 (1980) 84. Stoter M, Bamberger AM, Aslan B, Kurth M, Speidel D, Loning T, Frank HG, Kaufmann P, Lohler J, Henne-Bruns D, Deppert W, Knippschild U: Inhibition of casein kinase I delta alters mitotic spindle formation and induces apoptosis in trophoblast cells. Oncogene 24: 7964-75 (2005) 85. Stratagene: QuikChange II Site-Directed Mutagenesis Kit Instruction Manual Catalog #200523 and #200524 (2010) 86. Takano A, Uchiyama M, Kajimura N, Mishima K, Inoue Y, Kamei Y, Kitajima T, Shibui K, Katoh M, Watanabe T, Hashimotodani Y, Nakajima T, Ozeki Y, Hori T, Yamada N, Toyoshima R, Ozaki N, Okawa M, Nagai K, Takahashi K, Isojima Y, Yamauchi T, Ebisawa T: A missense variation in human casein kinase I epsilon gene that induces functional alteration and shows an inverse association with circadian rhythm sleep disorders. Neuropsychopharmacology 29: 1901-9 (2004) 87. Takenaka Y, Fukumori T, Yoshii T, Oka N, Inohara H, Kim HR, Bresalier RS, Raz A: Nuclear export of phosphorylated galectin-3 regulates its antiapoptotic activity in response to chemotherapeutic drugs. Mol Cell Biol 24: 4395-406 (2004) 88. Tanaka Y, Nakamura A, Morioka MS, Inoue S, Tamamori-Adachi M, Yamada K, Taketani K, Kawauchi J, Tanaka-Okamoto M, Miyoshi J, Tanaka H, Kitajima S: Systems analysis of ATF3 in stress response and cancer reveals opposing effects on pro-apoptotic genes in p53 pathway. PLoS One 6: e26848 (2011) 89. Tapia C, Featherstone T, Gomez C, Taillon-Miller P, Allende CC, Allende JE: Cloning and chromosomal localization of the gene coding for human protein kinase CK1. FEBS Lett 349: 307-12 (1994) 90. Tuazon PT, Traugh JA: Casein kinase I and II--multipotential serine protein kinases: structure, function, and regulation. Adv Second Messenger Phosphoprotein Res 23: 123-64 (1991) 6 Literaturverzeichnis 91. 65 Umar S, Wang Y, Morris AP, Sellin JH: Dual alterations in casein kinase I-epsilon and GSK-3beta modulate beta-catenin stability in hyperproliferating colonic epithelia. Am J Physiol Gastrointest Liver Physiol 292: G599-607 (2007) 92. Venerando A, Marin O, Cozza G, Bustos VH, Sarno S, Pinna LA: Isoform specific phosphorylation of p53 by protein kinase CK1. Cell Mol Life Sci 67: 1105-18 (2010) 93. Verkaar F, Zaman GJ: New avenues to target Wnt/beta-catenin signaling. Drug Discov Today 16: 35-41 (2011) 94. Wang X, Hoekstra MF, DeMaggio AJ, Dhillon N, Vancura A, Kuret J, Johnston GC, Singer RA: Prenylated isoforms of yeast casein kinase I, including the novel Yck3p, suppress the gcs1 blockage of cell proliferation from stationary phase. Mol Cell Biol 16: 5375-85 (1996) 95. Winter M, Milne D, Dias S, Kulikov R, Knippschild U, Blattner C, Meek D: Protein kinase CK1delta phosphorylates key sites in the acidic domain of murine double-minute clone 2 protein (MDM2) that regulate p53 turnover. Biochemistry 43: 16356-64 (2004) 96. Xu RM, Carmel G, Sweet RM, Kuret J, Cheng X: Crystal structure of casein kinase-1, a phosphate-directed protein kinase. EMBO J 14: 1015-23 (1995) 97. Yin H, Laguna KA, Li G, Kuret J: Dysbindin structural homologue CK1BP is an isoform-selective binding partner of human casein kinase-1. Biochemistry 45: 5297-308 (2006) 98. Zanivan S, Gnad F, Wickstrom SA, Geiger T, Macek B, Cox J, Fassler R, Mann M: Solid tumor proteome and phosphoproteome analysis by high resolution mass spectrometry. J Proteome Res 7: 5314-26 (2008) 99. Zhai L, Graves PR, Robinson LC, Italiano M, Culbertson MR, Rowles J, Cobb MH, DePaoli-Roach AA, Roach PJ: Casein kinase I gamma subfamily. Molecular cloning, expression, and characterization of three mammalian isoforms and complementation of defects in the Saccharomyces cerevisiae YCK genes. J Biol Chem 270: 12717-24 (1995) 100. Zhang J, Gross SD, Schroeder MD, Anderson RA: Casein kinase I alpha and alpha L: alternative splicing-generated kinases exhibit different catalytic properties. Biochemistry 35: 16319-27 (1996) 7 Abbildungsverzeichnis 66 7 Abbildungsverzeichnis Abbildung 1: Evolutionsbedingte Verwandtschaft der CK1-Isoformen von Hefen zu Menschen und deren Verwandtschaft zu anderen Kinase Gattungen des Menschen 3 Abbildung 2: Domänenstruktur von CK1 (schematische Darstellung) 4 Abbildung 3: Kanonische Konsensussequenz für CK1 5 Abbildung 4: Wnt-Signalweg und die Rolle der Casein Kinase 1 (CK1) 10 Abbildung 5: Prinzip der Site-directed Mutagenesis 31 Abbildung 6: Schematische Darstellung der Lage der Phosphorylierungsstellen potenzieller Kinasen, die CK1 C-Terminal phosphorylieren Abbildung 7: 35 Schematische Darstellung der verschiedenen GST-CK1 Fusionsproteine, die in Kinaseassays eingesetzt wurden 36 Abbildung 8: Phosphateinbau in CK1-GST-Fusionsproteine durch Clk2 37 Abbildung 9: Chk2 phosphoryliert GST-CK1-Fusionsproteine 38 Abbildung 10: Phosphateinbau in CK1-GST-Fusionsproteine durch Chk1 39 Abbildung 11: zweidimensionale Phosphopetidanalysen von GST-CK1 323-428-C-terminalen Fragmenten mit unterschiedlichen Mutationen an putativen Phosphorylierungsstellen nach in vitro-Phosphorylierung durch Chk1 40 Abbildung 12: Phosphoaminosäureanalyse des Peptids aus der zweidimensionale Phosphopetidanalyse mit GST-CK1 323-428, T323A, T329A, S370A 41 Abbildung 13: Zweidimensionale Phosphopetidanalyse von GST-CK1 Fusionsproteinen nach in vitro-Phosphorylierung durch Chk1 42 7 Abbildungsverzeichnis 67 Abbildung 14: zweidimensionale Phosphopetidanalyse des GST-CK1 375-428 -C-terminalen Fragmentes nach in vitro-Phosphorylierung durch Chk1 Abbildung 15: Michaelis-Menten-Kinetik CK1 wt vs. CK1 T397A 43 46 8 Tabellenverzeichnis 68 8 Tabellenverzeichnis Tabelle 1: Auflistung aller verwendeten Primer 16 Tabelle 2: Ansatz für PCR-Reaktionen 30 Anhang Anhang Eidesstattliche Erklärung Ich, Sven-Jannis Randoll, versichere hiermit, dass ich die Arbeit selbständig angefertigt habe und keine anderen als die angegebenen Quellen und Hilfsmittel benutzt sowie die wörtlich oder inhaltlich übernommenen Stellen als solche kenntlich gemacht habe. Ich habe keine Promotionsversuche an anderen Universitäten unternommen. Es laufen keine Strafverfahren gegen mich. Ulm, 15.03.2013............................................................................................ Datum, Unterschrift 69 Danksagung 70 Danksagung Mein Dank gilt allen voran Frau Prof. Dr. Doris Henne-Bruns, die es mir ermöglicht hat diese Arbeit am Universitätsklinikum Ulm in der Klinik für Allgemein-, Viszeral- und Transplantationschirurgie anzufertigen. Meinen ganz besonderen Dank möchte ich Herrn Prof. Dr. Uwe Knippschild für die Überlassung des interessanten Themas dieser Dissertation, die ausgezeichnete Betreuung während meiner Zeit in Ulm und die schnelle Durchsicht der Arbeit aussprechen. Zu großem Dank bin ich zudem Herrn Joachim Bischof verpflichtet, der mich immer bei allen praktischen Arbeiten einzigartig unterstützt hat und mich kompetent und verständlich mit jeder neuen Methodik vertraut machte. Vielen Dank auch fürs Korrekturlesen der Arbeit! Frau Annette Blatz danke ich für die vielen Hilfestellungen und die Tipps bezüglich der formalen Gestaltung der Doktorarbeit. Großen Dank auch an Nadine Süßner, die mir oft eine große Hilfe bei der Durchführung meiner Versuche war und immer für eine heitere Arbeitsatmosphäre sorgte. Mein Dank richtet sich außerdem an die restlichen Mitarbeiter des Forschungslabors der Chirurgie I, Arnhild Grothey, Helena Krist, Elena Yasovskaya, Kalim Ullah Khan und Pengfei Xu, die alle zu einem angenehmen Arbeitsumfeld beigetragen haben. Schließlich möchte ich noch ein ganz herzliches Dankeschön an meine Familie richten. Ein ganz großes Dankeschön gebührt hierbei meinen Eltern, die mir diesen Berufsweg erst ermöglicht haben und ohne deren Unterstützung mir das Verfassen meiner Doktorarbeit gar nicht möglich gewesen wäre. Außerordentlich dankbar bin ich auch Ingo Jirouschek für die große Hilfe bei der Formatierung der Arbeit. Ein ganz besonders liebevolles Dankeschön auch an meine Frau Carina und meine Tochter Emma, die mich immer aufmunterten und abschalten ließen, egal wie gestresst ich war. Ebenso möchte ich mich auch bei meinem Bruder Niklas ganz herzlich bedanken, der immer ein offenes Ohr für mich hatte. Lebenslauf 71 Lebenslauf Der Lebenslauf ist in der Online-Version aus Gründen des Datenschutzes nicht enthalten.