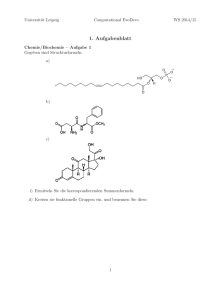
Untersuchungen zu den zytokinartigen Eigenschaften des Hormons
Werbung

Aus dem Institut für Immunologie und Transfusionsmedizin der Universität zu Lübeck Direktor: Prof. Dr. med. H. Kirchner Untersuchungen zu den zytokinartigen Eigenschaften des Hormons Prolaktin Inauguraldissertation zur Erlangung der Doktorwürde der Universität zu Lübeck - aus der medizinischen Fakultät vorgelegt von Katharina Cziupka aus Stade Lübeck, Mai 2007 1. Berichterstatter . . . . . . . . . . . . . . . . . . . . PD Dr. rer. nat. J. Luhm 2. Berichterstatter(in) . . . . . . . . . . . . . . . . . Prof. Dr. med. J. Steinhoff Tag der mündlichen Prüfung . . . . . . . . . . . 14.5.2007 Zum Druck genehmigt. Lübeck, den 14.5.2007 gez. Prof. Dr. med. Werner Solbach - Dekan der Medizinischen Fakultät - ¨ T HROUGHOUT THE UNIVERSE THERE REIGNS THE UNION OF OPPOSITES ... ¨ A. N. Whitehead, Adventures of Ideas, New York 196720th , S. 190 Abkürzungen APC bp cAMP CD cDNA cptcAMP CRE CREB DAR DEPC DNA EBNA ELISA EMSA GAPDH GAS GEArray Grb2 IFN-γ Ig IL IRF-1 Jak LPS MAPK MHC mRNA NA NF-κB antigen presenting cell base pairs cyclic adenosin monophosphate cluster of differentiation complementary DNA chlorophenylthio-cAMP cAMP responsive element CRE binding protein digital autoradiograph Diethyl pyrocarbonate Deoxyribonucleic acid Epstein-Barr Virus Nuclear Antigen-1 enzyme-linked immuno sorbent assay electrophoretic mobility shift assay Glycer aldehyde 3 phosphate dehydrogenase Interferon γ activation site Gene Expression Array Growth factor receptor-bound protein 2 Interferon γ immune globulin Interleukin Interferon-Regulatory-Factor 1 Janus kinase Lipopolysaccharide Mitogen activated protein kinase major histocompatibility complex messenger RNA norepinephrine Nuclear factor κB III Antigen-präsentierende Zelle Basenpaare zyklisches Adenosinmonophosphat Differenzierungsmuster komplementäre DNA Chlorophenylthio-cAMP auf cAMP reagierende Einheit CRE bindendes Protein digitaler Autoradiograph Diethylpyrocarbonat Desoxyribonukleinsäure Epstein-Barr Virus nukleäres Antigen-1 Enzym-Immunoassay Gel-Shift-Assay Glyceraldehyd-3phosphatdehydrogenase Interferon γ Aktivierungsstelle Genexpressionsarray Wachstumsfaktorrezeptorgebundenes Protein-2 Interferon γ Immunglobulin Interleukin Interferon-Regulations-Faktor-1 Januskinase Lipopolysaccharid Mitogen-aktivierte Proteinkinase Haupthistokompatibilitätskomlex Boten-RNA Noradrenalin nukleärer Faktor κB PBMC peripheral blood mnonuclear cells PBS PCR PHA Pit-1 Phosphate buffered solution Polymerase chain reaction Phythaemagglutinine Pituitary-specific transcription factor-1 Phorbol 12-myristate 13-acetate (humane) prolactin Prolactin receptor Ribonucleic acid reverse transcription-Polymerase Chain Reaction standard deviation Src homology 3 domain Src homology 2-containing protein tyrosine phosphatase-1 Systemic inflammatory response syndrome systemic lupus erythematosus Suppressor of cytokine signalling-1 PMA (h)PRL PRLR RNA RT-PCR SD SH3 SHP-1 SIRS SLE SOCS-1 Sp-1 STAT TH1 / TH2 TIVA TLR4 TNF TRH VIP Specificity protein-1 Signal transducer and activator of transcription T helper type 1 / 2 total intravenous anaesthesia Toll-like-receptor 4 Tumor-necrosis-faktor Thyreotropin-releasing-hormone vasoactive intestinal polypeptide IV mononukleäre Zellen des peripheren Blutes Phosphat-gepufferte Lösung Polymerase-Ketten-Reaktion Phythämagglutinin Hypophysen-spezifischer Transkriptionsfaktor-1 Phorbol-12-myristyl-13-acetat (humanes) Prolaktin Prolaktinrezeptor Ribonukleinsäure Reverse-Transkriptase-PolymeraseKettenreaktion Standardabweichung Src-Homologie-3-Domäne Src-Homologie-2-enthaltende Tyrosinphosphatase-1 Systemisches inflammatorisches Response-Syndrom systemischer Lupus erythematodes Suppressor der Zytokin-vermittelten Signaltransduktion-1 Spezifizitätsprotein-1 Signalübertrager und Transkriptionsaktivator T-Helfer-Zellen Typ 1 / 2 totale intravenöse Anästhesie Toll-like Rezeptor 4 Tumor-Nekrose-Faktor Thyreotropin-Releasing-Hormon vasoaktives intestinales Polypeptid INHALTSVERZEICHNIS INHALTSVERZEICHNIS Inhaltsverzeichnis 1 2 EINLEITUNG 1.1 Allgemeines . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1.2 Struktur und molekularbiologische Eigenschaften von Prolaktin . . . . . . 1.3 Bereits bekannte Orte der Prolaktinsynthese beim Menschen . . . . . . . . 1.3.1 Prolaktinsynthese in der menschlichen Plazenta, Dezidua und Endometrium . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1.3.2 Prolaktinsynthese in der menschlichen Glandula mammaria . . . . 1.3.3 Prolaktinsynthese im Immunsystem . . . . . . . . . . . . . . . . . 1.4 Prolaktinrezeptoren . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1.4.1 Struktur des Prolaktinrezeptors . . . . . . . . . . . . . . . . . . . . 1.4.2 Exprimierung des PRL-Rezeptors in unterschiedlichen Geweben . . 1.4.3 PRLR-Isoformen . . . . . . . . . . . . . . . . . . . . . . . . . . . 1.4.4 Aktivierung des Prolaktinrezeptors . . . . . . . . . . . . . . . . . 1.4.5 Mit dem Prolaktinrezeptor assoziierte Signaltransduktion . . . . . . 1.5 Immunmodulatorische Wirkung von Prolaktin . . . . . . . . . . . . . . . . 1.5.1 Spezifische und unspezifische Immunantwort . . . . . . . . . . . . 1.5.2 Immunologische Bedeutung der Zytokine TNF-α, IFN-γ, IL-12 und IL-10 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1.5.3 Wirkung von Prolaktin . . . . . . . . . . . . . . . . . . . . . . . . 1.5.4 PRL als Mitogen . . . . . . . . . . . . . . . . . . . . . . . . . . . 1.5.5 PRL als antiapoptotischer Faktor . . . . . . . . . . . . . . . . . . . 1.6 Zielsetzung der vorliegenden Arbeit . . . . . . . . . . . . . . . . . . . . . 5 5 8 10 MATERIAL UND METHODEN 2.1 Material . . . . . . . . . . . . . . . . . . . . . . . . . . 2.1.1 Geräte . . . . . . . . . . . . . . . . . . . . . . . 2.1.2 Laborbedarf . . . . . . . . . . . . . . . . . . . . 2.1.3 Zellkultur-Reagenzien, -Medien und -Zusätze . . 2.1.4 Reagenzien für molekularbiologische Methoden 2.1.5 Immunologische Reagenzien . . . . . . . . . . . 2.1.6 Testkits . . . . . . . . . . . . . . . . . . . . . . 2.1.7 Chemikalien und sonstige Reagenzien . . . . . . 2.2 Methoden . . . . . . . . . . . . . . . . . . . . . . . . . 2.2.1 Zellpräparationen . . . . . . . . . . . . . . . . . 25 25 25 27 29 29 30 30 31 32 32 1 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 10 11 11 11 11 12 14 15 15 19 19 21 21 22 22 24 INHALTSVERZEICHNIS 2.2.2 2.2.3 2.2.4 2.2.5 2.2.6 2.2.7 2.2.8 2.2.9 2.2.10 2.2.11 3 4 INHALTSVERZEICHNIS Zellkulturverfahren . . . . . . . . . . . . . . . Präparation von Kernextrakten . . . . . . . . . Überstände . . . . . . . . . . . . . . . . . . . RNA-Extraktion . . . . . . . . . . . . . . . . EMSA . . . . . . . . . . . . . . . . . . . . . Dotbloting . . . . . . . . . . . . . . . . . . . ELISA (enzyme-linked immuno sorbent assay) Elecsys . . . . . . . . . . . . . . . . . . . . . Nachweis der messenger RNA . . . . . . . . Auswertung der Ergebnisse / Statistik . . . . . . . . . . . . . . . 33 34 35 35 36 39 40 41 42 45 ERGEBNISSE 3.1 Wirkung von Prolaktin auf Immunzellen . . . . . . . . . . . . . . . . . . . 3.1.1 Einfluss von Prolaktin auf die Zytokinproduktion (IL-12, TNF-α, IL-10) im Vollblutansatz . . . . . . . . . . . . . . . . . . . . . . . 3.1.2 Einfluss von PRL auf die IFN-γ-Freisetzung durch Vollblutzellen . 3.1.3 Einfluss von PRL auf die Kerntranslokation von (NF-κB und IRF-1) 3.1.4 Einfluss von PRL auf die Aktivierung von STAT5b in PBMC . . . . 3.2 Prolaktinsynthese durch PBMC . . . . . . . . . . . . . . . . . . . . . . . . 3.2.1 Versuch des Nachweises einer möglichen PRL-Synthese auf Ebene der Transkriptionsfaktoren . . . . . . . . . . . . . . . . . . . . . . 3.2.2 Versuch des Nachweises einer möglichen PRL-Synthese auf Ebene der mRNA . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 3.2.3 Versuch des Nachweises auf Ebene des Proteins . . . . . . . . . . . 47 47 DISKUSSION 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem . . . . . . . . 4.1.1 Aktivierung von IRF-1 durch PRL . . . . . . . . . . . . . . . . . . 4.1.2 Aktivierung von NF-κB durch PRL . . . . . . . . . . . . . . . . . 4.1.3 Einordnung der eigenen Ergebnisse zur Beeinflussung von IRF-1 und NF-κB durch PRL . . . . . . . . . . . . . . . . . . . . . . . . 4.1.4 Beeinflussung der Zytokinsynthese durch PRL . . . . . . . . . . . 4.1.5 Zustände von Hyperprolaktinämie und mögliche klinische Bedeutungen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 4.1.6 Zusammenfassende Darstellung des Einflusses von PRL auf das Immunsystem . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 71 71 73 75 2 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 47 51 52 56 57 58 60 69 75 78 79 82 INHALTSVERZEICHNIS 4.2 4.3 4.4 5 INHALTSVERZEICHNIS Prolaktinsynthese durch PBMC . . . . . . . . . . . . . . . . . . . . . . . . 4.2.1 Aktivität der Transkriptionsfaktoren in Kernextrakten aus PBMC bezüglich der Bindung an den Prolaktin-Promotor . . . . . . . . . . . 4.2.2 Untersuchung von PBMC auf PRL-mRNA mittels GEArray und RTPCR . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 4.2.3 Untersuchung der Überstände von PBMC-Kulturen auf PRL mittels ELISA . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 4.2.4 Zusammenfassende Darstellung . . . . . . . . . . . . . . . . . . . 4.2.5 Diskussion von autokrinen und parakrinen Wirkungen von möglicherweise in PBMC synthetisiertem PRL . . . . . . . . . . . . . . Wirkung von hypophysärem PRL auf die Synthese von PRLR in PBMC . . Prolaktin - ein Zytokin ? . . . . . . . . . . . . . . . . . . . . . . . . . . . ZUSAMMENFASSUNG 83 85 86 87 88 88 89 90 92 APPENDIX 94 A Protokolle A.1 Isolierung von PBMC . . . . . . . . . . . . . . . . . . . . . . . . . A.1.1 Lösungen . . . . . . . . . . . . . . . . . . . . . . . . . . . A.1.2 Protokoll . . . . . . . . . . . . . . . . . . . . . . . . . . . A.2 Protokoll zur Herstellung von Kernextrakten . . . . . . . . . . . . . A.2.1 Erforderliche Reagenzien: . . . . . . . . . . . . . . . . . . A.2.2 Protokoll: . . . . . . . . . . . . . . . . . . . . . . . . . . . A.3 Bestimmung des RNA-Gehaltes einer Probe . . . . . . . . . . . . . A.4 Bandshift-Experimente . . . . . . . . . . . . . . . . . . . . . . . . A.4.1 5´-32 P-Oligonukleotidlabeling . . . . . . . . . . . . . . . . A.4.2 Bindereaktion . . . . . . . . . . . . . . . . . . . . . . . . . A.4.3 Bandshift-Gel und nachfolgende (digitale) Autoradiographie A.5 Genexpressions-Analyse . . . . . . . . . . . . . . . . . . . . . . . A.5.1 Protokoll zur Herstellung der benötigten Lösungen . . . . . A.5.2 GEArray ASSAY Protokoll . . . . . . . . . . . . . . . . . A.5.3 Hybridisierung . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 94 94 94 94 95 95 95 96 96 96 97 99 101 101 101 103 B Eigene Publikationen und Kongressbeiträge 104 C Danksagung 105 3 INHALTSVERZEICHNIS INHALTSVERZEICHNIS D Lebenslauf 106 ABBILDUNGSVERZEICHNIS 108 LITERATUR 110 4 1 1 1.1 EINLEITUNG EINLEITUNG Allgemeines Einen enormen Fortschritt in der Evolution stellte die Entwicklung von vielzelligen Lebewesen dar, deren Einzelkomponenten arbeitsteilig organisiert sind. Eine unabdingbare Voraussetzung für diesen Schritt der Entwicklung war das Entstehen eines diffizilen Systems der Signalübermittlung von Zelle zu Zelle aber auch von Organ zu Organ. Dieses System der interzellulären Signalübermittlung wird zum einen durch das Gefüge der Hormone und Zytokine sowie zum anderen bei höheren Lebewesen durch das Nervensystem repräsentiert. Das Wort “Hormon” kommt von griechisch “horman”, was “erregen” bedeutet. Der Begriff wurde in dem Vortrag “The Chemical Correlations of the Functions of the Body”, den der englische Physiologe Ernest Henry Starling 1905 hielt, eingeführt. Doch schon Claude Bernard prägte die Vorstellung von Hormonen als Signal des Körperinneren, indem er 1855 die innere Sekretion via Blut von der äußeren Sekretion über Schweiß und Tränen abgrenzte [43]. Hormone und Zytokine sind nach der heute vorherrschenden Lehrmeinung zumeist extrazelluläre Signalmoleküle, die mit einem Rezeptor auf einer Zielzelle, der meist ein integrales Membranprotein darstellt, in Wechselwirkung treten. Darauf folgt die Bildung eines intrazellulären Signalmoleküls, das über eventuelle Zwischenschritte zu dem Effekt des Hormons an der Zielzelle führt. Signalauslöschung geschieht meist durch intrazelluläre Inaktivierung des Signalmoleküls [46]. Klassischerweise unterscheidet man glanduläre Hormone, Gewebshormone und Zytokine. Dabei werden die glandulären Hormone von endokrinen Drüsen wie Hypophyse, Langerhans’sche Inseln des Pankreas, Schilddrüse, Nebenschilddrüse, Nebenniere, Keimdrüse und Plazenta gebildet, an das Blut abgegeben und gelangen so zu ihren Zielzellen. Auf diesem Weg vermitteln sie zwischen räumlich u.U. weit getrennten Organen. Hier regulieren sie verschiedenste Stoffwechselwege. Gewebshormone werden von spezialisierten Zellen synthetisiert, die in verschiedensten Geweben verstreut sind. Ihre Wirkung geschieht über den Blutweg, parakrin oder autokrin. Wichtige Vertreter sind Amine, Kinine, Eikosanoide und die gastrointestinalen Hormone [46]. 5 1.1 Allgemeines 1 EINLEITUNG Zytokine sind Polypeptide, die als Reaktion auf Mikroorganismen und andere Antigene produziert werden und Immun- und Entzündungsreaktionen vermitteln. Trotz ihrer strukturellen Verschiedenheit sind ihnen folgende Eigenschaften gemeinsam (nach [1]): 1. Die Zytokin-Sekretion ist ein kurzer, selbstlimitierender Prozess. Die Zytokine sind normalerweise nicht als präformierte Moleküle gespeichert und ihre Synthese ist gewöhnlich durch Transkription nach vorausgehender Zellaktivierung geregelt. Anschließend werden Zytokine schnell sezerniert und schlagartig freigesetzt. 2. Die Wirkungen der Zytokine sind pleiotrop, d.h., ein Zytokin wirkt auf verschiedene Zelltypen, und redundant, d.h., einen bestimmten Effekt können mehrere Zytokine losgelöst voneinander bewirken. 3. Zytokine beeinflussen oft die Synthese und die Eigenschaften anderer Zytokine. 4. Zytokine entfalten ihre Wirkung meist lokal eng begrenzt an einer immunologischen Synapse, aber bei massiver Freisetzung ist auch ein Übertritt in die Blutbahn mit Wirkung über längere Distanz im Sinne einer systemischen Wirkung wie im Rahmen einer Sepsis möglich. 5. Die Wirkung wird durch Bindung an spezifische Membranrezeptoren auf Zielzellen vermittelt, wobei die Dissoziationskonstanten zwischen 10−10 bis 10−12 M liegen. Durch diese starke Bindung reichen oft nur geringe Mengen eines Zytokins aus, um einen biologischen Effekt zu erzielen. 6. Äußere Signale regulieren die Expression von Zytokinrezeptoren und damit die Empfindlichkeit der Zielzelle für das Zytokin. Dies ist unabdingbar für die Spezifität des Immunsystems, da Zytokine nicht antigenspezifisch sind. 7. Die zelluläre Antwort auf die meisten Zytokine beruht auf einer Änderung der Genexpression von Zielgenen. Folgerichtig besteht eine scharfe Trennung zwischen endokrin wirkenden Hormonen und Zytokinen. Vielfach wurden jedoch in letzter Zeit endokrinen Hormonen zytokinartige Eigenschaften zugesprochen, sodass sich die Frage stellt, ob die Trennlinie wirklich existiert oder ob es Hormone gibt, die gleichzeitig alle Eigenschaften von Zytokinen erfüllen können. In der Literatur ist von einigen solchen Hormonen berichtet, wobei sich das Interesse v.a. auf das endokrine Hormon Prolaktin fokussiert. Die Untersuchung der möglicherweise zytokinartigen Eigenschaften dieses Hormons ist Thema dieser Arbeit. 6 1.1 Allgemeines 1 EINLEITUNG Prolaktin (PRL) ist ein Polypeptid, von dem zahlreiche verschiedene Funktionen im Laufe der Phylogenese bekannt sind. Diese beziehen sich vor allem auf die Reproduktion, so dass man die Wirkungen des Prolaktins zusammenfassend als Schaffung der Voraussetzung für die Brutpflege betrachten kann [70]. Im Rahmen der Ausübung dieser Funktion ist das Zielorgan bei den meisten Säugern und ganz besonders auch beim Menschen die Brustdrüse (Glandula mammaria), wo das Hormon - ein optimales hormonales Milieu vorausgesetzt Galaktogenese und Galaktopoese bewirkt. Diesen schon lange bekannten Funktionen verdankt Prolaktin seinen Namen. PRL übt viele weitere Effekte auf die unterschiedlichsten Gewebe und Organe aus. Bei Wirbeltieren sind über 300 verschiedene Funktionen von PRL beschrieben. Es ist in verschiedenen Organen wichtige Voraussetzung für die Differenzierung der organspezifischen Zellen, so z.B. in der Glandula mammaria, dem Ovar, der Prostata, den Tränen- und Speicheldrüsen, dem Pankreas und der Leber [33]. PRL ist zudem ein Regulator für das Ausmaß der Proliferation verschiedener Zelltypen u.a. der Mammaepithelien [66], Betazellen des Pankreas [13], Astrozyten [22], Zellen im Hypophysenvorderlappen [42], Adipozyten [59] und T-Lymphozyten [78]. Humanes Prolaktin (hPRL) wird von den laktotropen Zellen des Hypophysenvorderlappens produziert. Inhibitorisches Prinzip stellt Dopamin dar, das ständig von hypothalamischen dopaminergen Zellen ausgeschüttet wird. Daraus ergibt sich, dass Neuroleptika, die das dopaminerge System hemmen, erhöhte Prolaktinspiegel im Serum bewirken. Stimulierend auf die PRL-Ausschüttung können Thyreotropin-Releasing-Hormon (TRH), vasoaktives intestinales Polypeptid (VIP), Angiotensin und β-Endorphin wirken [70]. Zudem führt Reizung der Mechanorezeptoren der Mamille über Ausschüttung von PRLReleasing-Hormonen zu einer vermehrten hypophysären PRL-Sekretion. Dieser Prozess ist v.a. in der Stillzeit von immenser Bedeutung. Ein weiterer Trigger für die Freisetzung von PRL aus der Hypophyse ist Stress. Auch Narkotika - hier v.a. Opioide - können Zustände von Hyperprolaktinämie bedingen. Auch die Synthese und Sekretion von hPRL ist nicht wie lange Zeit angenommen auf die Adenohypophyse beschränkt, sondern viele Gewebe und Organe des menschlichen Organismus wie Plazenta, Glandula mammaria und eventuell auch das Immunsystem [26] scheinen diese Fähigkeit zu besitzen. So wurde schon der Name Omnipotin für dieses außerordentlich interessante Hormon vorgeschlagen [4]. 7 1.2 Struktur und molekularbiologische Eigenschaften von Prolaktin 1 EINLEITUNG Normalwerte für Prolaktin im Plasma liegen für Männer bei 0-11 ng/ml und für Frauen bei 2-16 ng/ml. Die Ausschüttung unterliegt einem zirkadianen Mechanismus mit hohen Spiegeln in der Nacht-Schlafphase. Die Halbwertszeit beträgt ca. 20 min [23]. Bei Patienten mit PRL-sezernierenden Hypophysentumoren werden nicht selten PRL-Serumkonzentrationen von 1000 ng/ml erreicht. 1.2 Struktur und molekularbiologische Eigenschaften von Prolaktin Basierend auf genetischen, strukturellen und funktionellen Eigenschaften gehört Prolaktin zur Gruppe der Prolaktin / Wachstumshormon / Plazentares Laktogen-Familie, deren kodierende Gene alle auf ein etwa 400 Millionen Jahre altes Gen zurückgehen, aus dem sie durch Genverdopplung entstanden sind [60]. PRL wird im menschlichen Genom von einem Gen auf Chromosom sechs kodiert, das aus 10 Kilobasen besteht und sich in fünf Exons und vier Introns gliedert [76]. Berwaer et al. konnten zeigen, dass die Transkription von PRL durch zwei verschiedene Promotoren unabhängig voneinander bewirkt werden kann [6], wobei der Ort des Ablesens den Promotor bestimmt. Das Ablesen der PRL-Desoxyribonukleinsäure (DNA) in der Hypophyse ist streng an die Anwesenheit des Pituitary-specific transcription factors 1 (Pit-1) gebunden. Pit-1 ist ein Transkriptionsfaktor, der spezifisch für die Hypophyse ist. Er reguliert hier die Expression von Wachstumshormonen, TSH und PRL. Die regulatorische DNA-Sequenz für die hypophysäre PRL-Synthese liegt auf dem Exon 1b des PRL-Gens. Die mRNA von PRL aus extrahypophysären Syntheseorten wie der Plazenta ist 150 Nukleotide länger als die in hypophysären Zellen entstehende. Dennoch ist das resultierende Peptid plazentaren Ursprungs nicht von dem hyophysären Ursprungs zu unterscheiden, was durch posttranslatorische Modifikation bedingt sein könnte [29]. Dem entspricht eine weitere nicht translatierte Region in 5’-Richtung. Di Matta et al. [24] fordern daher die Existenz eines weiteren Prolaktin-Promotors auf einem Exon in 5’-Richtung, das meist als Exon 1a bezeichnet wird. Berwaer et al. lokalisierten das Exon 1a bei 5840 bp in 5’-Richtung von dem Beginn des Exons 1b entfernt und fanden bei 375/-212 bp Bindungsstellen für lymphozytäre Proteine [6]. Ferner stellten sie fest, dass die Aktivität dieses Promotors unabhängig von Pit-1 8 1.2 Struktur und molekularbiologische Eigenschaften von Prolaktin 1 EINLEITUNG ist. Dieses steht im Einklang mit der Feststellung von Gellersen et al. [28], dass extrahypophysäre Freisetzung von PRL nicht von Stimulantien der Hypophyse wie TRH, epidermal growth factor oder Bromocritpin beeinflusst wird, sehr wohl aber von Glukokortikoiden und zyklischem Adenosinmonophosphat (cAMP). Berwaer et al. fanden in der Promotorregion für extrahypophysäres Prolaktin cAMP responsive elements (CREs) (TgACg) u.a. in der Nähe der TATAA-Box (-20 bis 25) [6] (siehe Abbildung 1). −100 A CAg CAA AAA CAg gAA gTT CAC TCT gTA AgC TTC TCC CAC CCT TCT CCC CTg AAg CAg gCC ATA AAA gAA TCC TC T gAC g TT TCT ATA AAg TAg −1 gTT CA T A Abbildung 1: Ausschnitt aus der Sequenz des Exon 1a Promotors; cAMP responsive element gelb unterlegt; Abbildung nach [6] Die hPRL-cDNA ist 914 Nukleotide lang, das Prohormon 227 Aminosäuren mit einem Signalpeptid aus 28 Aminosäuren, so dass das fertige hPRL eine Länge von 199 Aminosäuren erreicht [26]. Das Molekül ist aus einer Aminosäuren-Kette mit drei intramolekularen Disulfidbrücken zwischen sechs Cystein-Resten aufgebaut (Cys4 Cys11, Cys58 Cys174, Cys191 Cys199). Die Molekülmasse von hPRL beträgt etwa 23.000 Dalton. 50% der Aminosäuren bilden α-Helices, und der Rest bildet Schleifen. Man nimmt an, dass die Struktur von hPRL im Raum durch vier lange α-Helices in antiparalleler Anordnung gebildet wird [40] (siehe Abbildung 2). Neben der vorherrschenden 23.000 Dalton schweren Form des hRPL existieren verschiedene Varianten, die zum einen durch alternatives Spleißen, vielmehr jedoch durch proteolytisches Schneiden generiert werden, wobei eine 14-, 16- und 22 Kilodalton schwere Form entstehen kann. Die genaue Bedeutung dieser Proteolyseprodukte ist bisher wenig bekannt. Weiter können Makroprolaktine durch Dimerisation und Polymerisation oder durch Bindung an Proteine wie Immunglobuline (Igs) entstehen. Die hochmolekularen Varianten des hPRL sind im Allgemeinen biologisch weniger aktiv. Ebenfalls wird durch Phosphorylierung und Glykolysierung die biologische Aktivität des hPRL herabgesetzt. Eine mögliche Erklärung für die herabgesetzte biologische Aktivität hochmolekularer Prolaktine könnte eine Speicherfunktion sein. 9 1.3 Bereits bekannte Orte der Prolaktinsynthese beim Menschen 1 EINLEITUNG Abbildung 2: Molekulare Struktur von PRL; links die Vorderseite und rechts die Rückseite der Prolaktinstruktur niedrigster Energie; Abbildung entnommen aus [40] 1.3 Bereits bekannte Orte der Prolaktinsynthese beim Menschen Wie bereits erwähnt ist der Hypophysenvorderlappen bekanntester Ort der Prolaktinsynthese. Ontogenetisch stammen die laktotropen Zellen zusammen mit den somatotropen und thyrotropen von einer Pit-1-abhängigen Zelllinie hypophysärer Zellen ab. Im Hypothalamus von Ratten wurde Prolaktinsynthese-Aktivität in den dorsomedialen, ventromedialen, supraoptischen und paraventrikulären Kernen nachgewiesen [26]. Wahrscheinlich geht die Synthese im Hypothalamus unabhängig von der hypophysären Synthese vonstatten. Stimulierend wirken Steroidhormone aus dem Ovar, Angiotensin II und VIP. Die biologische Bedeutung dieses Prozesses ist weitgehend unbekannt. 1.3.1 Prolaktinsynthese in der menschlichen Plazenta, Dezidua und Endometrium Die Plazenta ist schon seit langem als ein Syntheseort prolaktinartiger Proteine (Prolactin like proteins, PLP) bekannt. Diese sind jedoch strukturell nicht identisch mit hPRL [26]. Endometriumzellen in der Proliferationsphase können durch Progesteron vermittelt hPRL synthetisieren. Ebenfalls sind hierzu Stromazellen von dezidualisiertem Endometrium während der frühen und späten Schwangerschaft befähigt. Diese Zellen reagieren dabei beson10 1.4 Prolaktinrezeptoren 1 EINLEITUNG ders auf eine intrazelluläre Erhöhung des Spiegels an cAMP mit einer vermehrten Prolaktinausschüttung, während eine alleinige Stimulation mit Progesteron keinen Effekt zeigt [74]. 1.3.2 Prolaktinsynthese in der menschlichen Glandula mammaria Zellen der weiblichen Brustdrüse in der Laktationsphase können zum einen hypophysäres Prolaktin aufnehmen zum anderen aber auch selbst synthetisieren. In der Milch findet sich zum Teil hypophysäres Prolaktin, aber auch hPRL, das in der Glandula mammaria selbst synthestisiert wurde. Der Darm des Neonaten kann während eines kurzen Zeitintervalls intakte Proteine absorbieren, sodass etwa 20% des Prolaktins in der Milch die Zirkulation des Neugeborenen erreichen. Dem hPRL in der Milch wird eine wichtige Funktion bei der Reifung des neuroendokrinen Systems und des Immunsystems zugeschrieben [26]. 1.3.3 Prolaktinsynthese im Immunsystem Schon 1989 berichtete Russell erstmalig von einem Prolaktin-ähnlichen Produkt aus Lymphozyten [67]. T-Zell Aktivatoren wie Phythämagglutinin (PHA) und Phorbol-12-myristyl13-acetat (PMA) sowie eine erhöhte Konzentration an intrazellulärem cAMP scheinen hierbei den Promotor für Prolaktin in Lymphozyten zu aktivieren [64]. Auch von Prostaglandin E2 als einem Stoff, der über eine Erhöhung des cAMP-Spiegels wirkt, ist berichtet, dass es in mononukleären Zellen des peripheren Blutes (PBMC) eine Synthese von PRL bewirken soll [30]. Neueste Ergebnisse zeigen, dass TNF-α den extrahypophysären PRL Promotor in leukämischen Zellen aktiviert, allerdings scheint der Mechanismus hierfür nicht eine Aktivierung von NF-κB sondern von Proteinkinase C zu sein [31]. Welche Bedeutung dem von Immunzellen synthetisierten PRL zukommen könnte, ist bisher nicht bekannt. Vor allem die Frage, ob PRL in biologisch relevantem Ausmaß vom Immunsystem selbst gebildet werden kann, bedarf weiterer Untersuchungen. 1.4 1.4.1 Prolaktinrezeptoren Struktur des Prolaktinrezeptors Der Prolaktinrezeptor ist ein einfaches membrangebundenes Protein, das zur Klasse I der Zytokinrezeptor-Superfamilie gehört. Diese Superfamilie umfasst Rezeptoren für verschiedene Interleukine, Granulocyte-Colony Stimulating Factor (G-CSF), Granulocyte Macrophage11 1.4 Prolaktinrezeptoren 1 EINLEITUNG Colony Stimulating Factor (GM-CSF), Erythropoietin (EPO) u.a. Zytokinrezeptoren vom Typ I - auch Hämopoietin-Rezeptoren - enthalten eine oder mehrere Domänen mit zwei konservierten Paaren von Cystein-Resten und einer proximal zur Membran gelegenen Sequenz von Tryptophan - Serin - X - Tryptophan - Serin (WSXWS), wobei X eine beliebige Aminosäure darstellt. Solche Rezeptoren binden klassischerweise Zytokine, die aus vier α-Helices aufgebaut sind (siehe Abbildung 3). Abbildung 3: Schema eines Zytokinrezeptors vom Typ I Die konservierten Eigenschaften des Rezeptors bilden Strukturen, die die α-Helices der Zytokine binden, während die spezifischen Eigenschaften der einzelnen Zytokine durch Aminosäurereste bestimmt werden, die von Rezeptor zu Rezeptor differieren. Der Prolaktinrezeptor besteht aus einer extrazellulären, einer 24 Aminosäuren langen transmembranären und einer intrazellulären Domäne. Die extrazelluläre Domäne kann entsprechend dem o.g. in eine Subdomäne D1 (NH2 -terminal) mit konservierten Disulfidbrücken zwischen Cysteinresten und eine membranproximale Subdomäne D2 mit WSXWS-Motiv unterteilt werden. Die Funktion der transmembranären Domäne bei der Aktivierung des Prolaktinrezeptors ist bisher unbekannt. So gut wie alle Isoformen des Prolaktinrezeptors besitzen in der intrazellulären Domäne eine Box 1, während die Box 2 bei der kurzen Isoform fehlt [26]. Box 1 ist eine membranproximale Region, die aus acht Aminosäuren besteht und reich an Prolin- und hydrophoben Resten ist. Die Box 2 ist weniger konserviert als Box 1 [7]. 1.4.2 Exprimierung des PRL-Rezeptors in unterschiedlichen Geweben Prolaktinrezeptoren (PRLRs) finden sich auf fast allen Zellen des menschlichen Körpers, so auch auf vielen peripheren Zellen des Immunsystems sowie in Thymus, Milz, Lymphknoten 12 1.4 Prolaktinrezeptoren 1 EINLEITUNG und Knochenmark [45]. Tabelle 1 bietet eine Zusammenstellung der Zellen und Gewebe, in denen PRLRs exprimiert werden. Zelle bzw. Gewebe Zelle bzw. Gewebe Zelle bzw. Gewebe Zentrales Nervensystem Gehirn Rinde Hippocampus Plexus choroideus Striatum Ductus cochlearis Corpus callosum Hypothalamus Astrozyten Gliazellen Retina Olfaktorisches System Ganglien Hypophyse Vorderlappen Intermediärlappen Nebennierenrinde Haut Epidermis Haarfollikel Schweißdrüsen Knorpel und Knochen Chondrozyten Knorpelsubstanz Osteoblasten Lunge Niere Rinde Lymphatisches Gewebe Milz Thymus Helferzellen Epitheliale Zellen Lymphozyten T B Makrophagen Ganglien Intestinale Zellen Sexualorgane Weiblich Ovar Ovum Granulosazellen Thekazellen Corpus luteum Tuba uterina Glandula mammaria Epitheliale Zellen Milch Tumoren Uterus Endometrium Plazenta Amnion Herz Herzmuskel Vorhof Skelettmuskel Braunes Fettgewebe Leber Hepatozyten Kupffer-Zellen Glandula submandibularis Glandula submaxillaris Pankreas Langerhans’sche Inseln Gastrointestinaler Trakt Ösophagus Magen Darm Duodenum Jejunum Ileum Colon Sexualorgane männlich Testis Germinalzellen Spermatozoen Leydig-Zellen Sertoli-Zellen Epididymis Vesicula seminalis Prostata Tabelle 1: Zusammenstellung der Organe und Gewebe, in denen PRLRs exprimiert werden; Tabelle entnommen aus [7] 13 1.4 Prolaktinrezeptoren 1 EINLEITUNG Unter den peripheren Zellen des Immunsystems exprimieren T- und B-Lymphozyten PRLRezeptoren. Durch PRL oder IL-2 induzierte Aktivierung dieser Zellen nimmt die Dichte an PRLR an der Oberfläche zu [18]. Sun et al. konnten 2004 zeigen, dass vermutlich auch NK-Zellen Rezeptoren für PRL an ihrer Membran besitzen. PRL kann NK-Zellen in Kostimulation mit IL-2 oder IL-15 aktivieren [72]. 1.4.3 PRLR-Isoformen Das Gen, das für den Prolaktinrezeptor kodiert, liegt auf Chromosom 5 und enthält 10 Exons. Die Synthese der Rezeptoren geschieht je nach Gewebe über drei verschiedene Promotoren. Die unterschiedlichen Isoformen der Prolaktinrezeptoren differieren in ihrer zytoplasmatischen Domäne, während die extrazelluläre Domäne identisch ist. Abhängig von der Anzahl der Aminosäuren, aus denen die zytoplasmatische Domäne besteht, unterscheidet man eine lange, eine intermediäre und eine kurze Isoform des PRLR. Zudem ist eine lösliche Isoform des PRLR bekannt (PRL binding protein, PRLbp), die nicht in der Zellmembran verankert ist [7] (siehe Abbildung 4). Abbildung 4: Isoformen des PRLR; alle Isoformen besitzen eine identische extrazelluläre Domäne, die den Liganden bindet. Subdomäne D1 enthält zwei Cystein-Paare (C-C), Subdomäne D2 das WSMotiv (als grüne Box dargestellt). Box 1 (organe dargestellt) findet sich in der zytoplasmatischen Domäne aller Isoformen. Abbildung entnommen aus [7] 14 1.4 1.4.4 Prolaktinrezeptoren 1 EINLEITUNG Aktivierung des Prolaktinrezeptors Jedes Prolaktin-Molekül besitzt zwei Bindungsstellen (Binding Sites). Zunächst bindet Bindungsstelle 1 (Helices 2 und 4) an das Rezeptor-Molekül. Diese Interaktion ist Bedingung für die Bindung von Bindungsstelle 2 (Helices 1 und 3) an ein weiteres Rezeptor-Molekül unter Homodimerisierung des Rezeptors. Es resultiert ein trimerer Komplex aus zwei RezeptorMolekülen und einem Prolaktin-Molekül [26], siehe Abbildung 5. Abbildung 5: Räumliche Struktur und Bindungsstellen von PRL und des PRLR; (A) Bindungsstellen des PRL-Moleküls für den PRL-Rezeptor (B) 3-D Struktur des Rezeptors; die Bindungsstelle für das PRL-Molekül ist mit Binding Site gekennzeichnet; Abbildung entnommen aus [7] Die intrazelluläre Domäne des Prolaktinrezeptors ist mit einer Tyrosinkinase namens Januskinase 2 (Jak2) assoziiert, die vermutlich über ein Adapterprotein mit der src kinase homology domain 3 (SH3) der Box 1 verbunden ist. Durch den engen räumlichen Kontakt bei der Dimerisierung kommt es zur Transphosphorylierung der Januskinasen. Diese phosphorylieren anschließend Tyrosinreste des Prolaktinrezeptors (siehe Abbildung 6). 1.4.5 Mit dem Prolaktinrezeptor assoziierte Signaltransduktion 1.4.5.1 Signaltransduktion über Signal transducers and activators of transcription (STATs): Zunächst interagieren die phosphorylierten Tyrosinreste des aktivierten Zytokinrezeptors mit der src homology 2 (SH2)-Domäne von zytoplasmatischen Proteinen namens Signal transducers and activators of transcription (STATs) wie in 1.4.4 beschrieben. 15 1.4 Prolaktinrezeptoren 1 EINLEITUNG Abbildung 6: Mechanismus der Prolaktinrezeptor-Aktivierung; Aktivierung des PRLR durch Homodimerisierung zweier Rezeptoren nach Bindung eines PRLMoleküls; Abbildung entnommen aus [26] Diese werden anschließend durch Rezeptor-assoziierte Januskinasen phosphoryliert, was zu Homo- und Heterodimerisierung der STATs unter Beteiligung der SH2-Domänen führt. Die Dimere können in den Kern translozieren, wo sie Transkriptionsfaktoren, wie Nucelar factor κB (NF-κB) und Interferon-Regulatory-Factor 1 (IRF-1) aktivieren. Bindungsstellen an Promotoren finden sich für IRF-1 u.a. an dem Interleukin (IL)-12 p40 Gen-Promotor und für NF-κB am Tumor-Nekrose-Faktor (TNF)-α-Promotor. Dieser oben beschriebene Reaktionsweg wird meist von Zytokinen eingeschlagen, die von T-Zellen nach Antigenkontakt freigesetzt werden und ist der wichtigste im Zusammenhang mit Signaltransduktion über den Prolaktinrezeptor (siehe Abbildung 8). Im Rahmen der Signaltransduktion über den Prolaktinrezeptor sind STAT1, STAT3 und v.a. STAT5 von Interesse [35]. Eine Übersicht über die STATs bietet Tabelle 22. Von STAT5 sind zwei Isoformen, STAT5a und STAT5b, bekannt. Die Sequenzen dieser beiden Moleküle zeigen eine Übereinstimmung von etwa 95%. Der entscheidende Unterschied liegt in den 8 C-terminalen Aminosäuren, zudem ist STAT5b am C-terminalen Ende 12 Aminosäuren kürzer als STAT5a [47]. Ein wichtiger negativer Regulator der Aktivierung von STAT5 ist die Src homology 2containing protein tyrosine phosphatase-1 (SHP-1). Dieses Protein wird v.a. in hämatopoetischen Zellen, aber auch PBMC exprimiert. SHP-1 bindet dabei an den PRL-Rezeptor / Jak2-Komplex, was zur Phosphorylierung C-terminaler Tyrosinreste führt. Dadurch werden 16 1.4 Prolaktinrezeptoren STAT STAT1 STAT2 STAT3 STAT4 beteiligte Zytokine Interferon α/β, Interferon γ Interferon α/β IL-6, IL-10 IL-12 STAT5a STAT5b STAT5a und 5b Prolaktin Wachstumshormon IL-2, IL-7, IL-9 zusätzlich o.g. STAT6 IL-4 1 EINLEITUNG Phänotyp der jeweiligen knockout-Maus Defekt der angeborenen Immunität; keine Antwort auf IFNs Defekt der Virusabwehr nicht lebensfähig Defekt in der TH1 -Entwicklung und der IFN-γProduktion Laktationsdefekt Zwergwuchs Laktationsdefekt, Zwergwuchs, Defekt der T-Zell-Proliferation unter Einfluss von IL-2 Defekt in TH2 -Entwicklung Defekt der IL-4 abhängigen Ig-Isotypen Tabelle 2: Übersicht über STATs, frei nach [1] growth factor receptor-bound protein 2 (Grb2) und das mit Grb2 assoziierte Protein Suppressor of cytokine signalling-1 (SOCS-1) aktiviert. SOCS-1 bindet sodann an Jak2, was zu deren Inaktivierung und damit zu einer verminderten Aktivierung von STAT5 führt (siehe Abbildung 36, Kapitel 4.1.3) [55]. Luo et al. konnten zeigen, dass STAT5b die Induktion des IRF-1 Promotors durch NF-κB hemmt [48]. NF-κB liegt in ruhenden Zellen inaktiv im Zytoplasma vor, wo es als Dimer an ein oder mehrere Proteine namens inhibitors of κB (IκBs) gebunden ist. Bei Aktivierung wird NF-κB aus diesem Komplex freigesetzt und kann durch die Kernporen in den Kern translozieren. Der nukleäre Export ist energieabhängig. NF-κB wurde ursprünglich als Transkriptionsfaktor verstanden, welcher an der Synthese der leichten Kette von Immunglobulinen beteiligt ist und B-Zell-spezifische Genexpression reguliert (daher der Name). Mittlerweile weiß man, dass diesem Transkriptionsfaktor als einem ubiquitär vorhandenen eine zentrale Bedeutung für das Immunsystem zukommt. Unter anderem kommt NF-κB eine entscheidende Rolle bei proinflammatorischen Prozessen wie Sepsis und SIRS zu. Hier spielt es bei der Antwort verschiedenster Zellen auf Zytokine wie TumorNekrose-Faktor und IL-1 eine Rolle. Des Weiteren ist NF-κB an der T-Zellaktivierung beteiligt, indem es die Transkription von IL-2 und IFN-γ initiiert. IRF-1 gehört zur neunköpfigen Familie der IRF-Proteine. Es ist an der Regulation der Synthese einer Vielzahl immunmodulatorischer Proteine beteiligt. Zudem spielt IRF-1 eine Rol- 17 1.4 Prolaktinrezeptoren 1 EINLEITUNG le im Zusammenhang mit der Progression des Zellzyklus, Apoptose und Tumorsuppression. IRF-1 knock out-Mäuse zeigen multiple Defekte der TH 1-Zellen, der CD8-T-Zellen und der NK-Zellen. Beim Menschen sind Defekte dieses Gens mit einer hohen Inzidenz an myelodysplastischen Syndromen und Leukämien korreliert [79]. Abbildung 7 stellt die vielzähligen Funktionen von IRF-1 zusammenfassend dar. Abbildung 7: IRF-1 ist ein multifunktionaler Transkriptionsfaktor Die dargestellte Zelle könnte einer T-, B- oder NK-Zelle, einem Makrophagen, einer Antigenpräsentierenden dendritischen Zelle oder einer γδT-Zelle entsprechen; nach [79] 1.4.5.2 Signaltransduktion über alternative Wege: Zusätzlich können Adapterproteine wie Shc / Grb2 / SOS über phosphorylierte Tyrosinreste an den PRL-Rezeptor binden, was zur Aktivierung der Ras/Raf/MAPK Kaskade führt. Weiter spielen Mitglieder der Src kinase-Familie eine Rolle, insbesondere eine Aktivierung der PI 39-Kinase über Fyn. Diesen beiden Signaltransduktionswegen über den PRLR wird eine Bedeutung im Rahmen der Proliferation und des Überlebens von Lymphozyten zugeschrieben, während der Weg 18 1.5 Immunmodulatorische Wirkung von Prolaktin 1 EINLEITUNG über Jak/Stat im Zusammenhang mit der Zelldifferenzierung der Lymphozyten und der Beeinflussung anderer Zellen durch Freisetzung von Zytokinen steht [19]. Box 1 der intrazellularen Rezeptordomäne ist des Weiteren an einer Aktivierung eines Tyrosinkinase-abhängigen Kalium-Ionen-Kanals durch Jak2 beteiligt, während das C-terminale Ende dieser Domäne an der Freisetzung von Inositol-1,3,4,5,-tetrakisphosphat (Ins(1,3,4,5)P4) und Inositolhexakisphosphat (InsP6) beteiligt ist, welche wiederum spannungsabhängige Kalzium-Kanäle öffnen (siehe Abbildung 8). 1.5 1.5.1 Immunmodulatorische Wirkung von Prolaktin Spezifische und unspezifische Immunantwort Die unspezifische - oder auch angeborene - Immunität stellt eine schnelle Antwort auf Mikroorganismen dar, die unabhängig von dem Erreger immer gleich abläuft. Die Mechanismen werden durch Strukturen in Gang gesetzt, die mehreren Gruppen von Mikroorganismen gemeinsam sind. Die Komponenten der unspezifischen Immunantwort sind • physikalische und chemische Barrieren wie Epithelien und antimikrobielle Substanzen, die von epithelialen Oberflächen gebildet werden, • Phagozyten wie Neutrophile und Makrophagen und Natürliche Killerzellen (NK-Zellen), • Bluteiweiße wie Komplement und andere Entzündungsmediatoren, • Zytokine, die die Aktivität der Zellen der unspezifischen Immunantwort beeinflussen und koordinieren. Die höher entwickelte erworbene und spezifische Immunität setzt vorherigen Kontakt zwischen Immunsystem und Antigen voraus und wird mit jeder weiteren Exposition effizienter und stärker. Die Komponenten sind Lymphozyten und ihre Produkte. Man unterscheidet innerhalb der erworbenen Immunität zwischen humoraler und zellulärer Immunantwort. Mediator der humoralen Antwort sind Antikörper, welche von B-Zellen produziert werden. Diese erkennen lösliche Antigene über Rezeptoren an ihrer Zelloberfläche und entwickeln sich zu Antikörper-sezernierenden Zellen. Zudem erkennen T-Helferzellen (CD4-Zellen) Antigene auf der Oberfläche Antigen-präsentierender Zellen des Wirtes und setzen Zytokine frei, welche die Aktivierung von B-, T-Lymphozyten und Makrophagen sowie Entzündungsmechanismen bewirken. Unter den T-Helfer-Zellen werden zwei Subpopulationen, die TH1und TH2-Zellen, unterschieden. Hauptsächliches Prinzip der TH1-Antwort ist die Freisetzung von IFN-γ und damit die Entwicklung einer proinflammatorischen Immunantwort. 19 1.5 Immunmodulatorische Wirkung von Prolaktin 1 EINLEITUNG Abbildung 8: Signaltransduktion über den PRL-Rezeptor;Abbildung entnommen aus [26] 20 1.5 Immunmodulatorische Wirkung von Prolaktin 1 EINLEITUNG TH2-Zellen sezernieren vorrangig IL-4 und IL-5 sowie IL-10, IL-13 und IL-18. Die zelluläre Immunität wird durch T-Killer-Zellen (CD8-Zellen) und NK-Zellen vermittelt. Sie ist primär gegen intrazelluläre Erreger gerichtet wie z.B. Viren, welche in Phagozyten und anderen Wirtszellen intrazellulär überleben, wo sie für Antikörper nicht erreichbar sind. 1.5.2 Immunologische Bedeutung der Zytokine TNF-α, IFN-γ, IL-12 und IL-10 Für die Untersuchungen der immunmodulatorischen Wirkungen von PRL wurden die Zytokine TNF-α, IFN-γ und IL-12 als Repräsentanten proinflammatorisch wirkender Stoffe und IL-10 als typisch antiinflammatorisches Zytokin bestimmt. Interleukin 12 (IL-12) ist immanent wichtig für die zelluläre Immunantwort. Es wird v.a. von B-Zellen und in geringerem Ausmaß von T-Zellen und Granulozyten gebildet. Ein wichtiger Induktor für die Bildung von IL-12 sind Bakterien bzw. LPS als Bestandteil der Zellwand gramnegativer Bakterien. IL-12 bewirkt eine Differenzierung naiver T-Zellen zu TH1-Zellen sowie die Synthese von IFN-γ und TNF-α durch diese. Es aktiviert NK-Zellen. Damit ist es ein klassisches proinflammatorisches Zytokin. Interferon-γ (IFN-γ) wird hauptsächlich von zytotoxischen T-Zellen, TH1-Zellen und NKZellen sezerniert. Es ist ein wichtiger Inhibitor viraler Replikation, induziert MHC-Expression und aktiviert Makrophagen. IFN-γ wirkt bezüglich seiner antiproliferativen Eigenschaften synergistisch mit TNF-α. Tumor-Nekrose-Faktor-α (TNF-α) ist benannt nach seiner zytolytischen und zytostatischen Wirkung auf Tumorzellen. Zudem steigert es die phagozytotische und zytotoxische Eigenschaften Neutrophiler Granulozyten und wirkt chemotaktisch auf diese Zellen. Es aktiviert das vaskuläre Endothel und erhöht dessen Permeabilität, was zu einem Übertritt von IgG, Komplement und Zellen in das umgebende Gewebe führt. Systemisch induziert es Fieber, mobilisiert Metabolite und führt zum Schock. Interleukin-10 (IL-10), welches von T-Zellen und Makrophagen freigesetzt wird, ist ein typisches Zytokin, dem eine entscheidende Bedeutung im Rahmen der TH2-Antwort zukommt. Es inhibiert die Sekretion von TNF-α und verhindert damit eine überschießende Antwort auf LPS. In Anwesenheit von IL-4 wirkt es zudem proliferatorisch auf Mastzellen. 1.5.3 Wirkung von Prolaktin Bereits 1972 vermuteten Chen et al. eine Funktion von Prolaktin auf das Immunsystem. Sie zeigten, dass exogen hinzugeführtes PRL die herabgesetzte Thymusfunktion bei PRLdefizienten Mäusen wieder verbessern kann [16]. Nagy et al. stellten fest, dass Hypophysektomie oder Supprimierung der PRL-Sekretion durch Bromocriptin zu einer Beeinträchtigung 21 1.5 Immunmodulatorische Wirkung von Prolaktin 1 EINLEITUNG der humoralen und zellulären Immunantwort führen. Diese kann durch Prolaktingabe aufgehoben werden [58, 57]. In Zellkulturen wirkt PRL immunmodulatorisch auf T- [5] und B- [3] Zellen, Makrophagen [5], NK-Zellen [51] und dendritische Zellen. Die immunmodulatorische Wirkung von PRL hängt hierbei vom Zelltyp, dem Differenzierungsstadium der Zellen und dem die Zelle umgebenden Milieu des Umfeldes ab. Vielfach wurde für PBMC [15] und NK-Zellen [52, 53] gezeigt, dass diese Zellen unter Stimulation mit PRL vermehrt Interferon γ (IFN-γ) sezernieren. Zudem zeigen PRL-stimulierte NK-Zellen eine erhöhte zytolytische Aktivität gegenüber der erythroleukämischen Zelllinie K562 [54]. Die Ausschüttung von PRL durch die Adenohypophyse wird zum einen durch den Hypothalamus gesteuert, zum anderen können IL-1, IL-2 und IL-6 die PRL-Produktion stimulieren, während IFN-γ und Endothelin-3 inhibitorisch wirken [17]. 1.5.4 PRL als Mitogen Die durch ein Antigen induzierte klonale Expansion von T-Lymphozyten ist eine wesentliche Komponente einer effektiven Immunantwort. Im Rahmen von in vitro-Experimenten konnte gezeigt werden, dass PRL wesentlich für das Ausmaß der Antwort von T- und B-Zellen auf Antigene oder Mitogene ist [20]. Ebenfalls agiert PRL als Komitogen für NK-Zellen und Makrophagen [19]. 1.5.5 PRL als antiapoptotischer Faktor Während Stressperioden wird von der Hypophyse sowohl vermehrt PRL als auch vermehrt Corticotropin Releasing Factor sezerniert. Ein wichtiger Mechanismus der immunsuppressiven Wirkung von Glukokortikoiden ist die Induktion von Apoptose in Populationen von T-Zell-Progenitorzellen. An murinen Thymozyten konnte gezeigt werden, dass die Dexamethason-induzierte Apoptose durch PRL antagonisiert werden kann. So scheinen PRL und Glukokortikoide ihre Wirkungen gegenseitig zu antagonisieren. Immunologische Homöostase kann somit als ein Gleichgewicht der gegenläufigen Effekte von PRL und Glukokortikoiden aufgefasst werden (siehe Abbildung 9). Diesem Antagonismus könnte eine entscheidende Bedeutung für das Immunsystem bei der Pathogenese verschiedener Autoimmunkrankheiten zukommen. 22 1.5 Immunmodulatorische Wirkung von Prolaktin 1 EINLEITUNG Abbildung 9: Antagonismus von PRL und Glukokortikoiden; Dex = Dexamethason Während Glukokortikoide die Apoptose von Lymphozyten induzieren, stimuliert PRL die Lymphozytenproliferation; Abbildung entnommen und verändert aus [19] 23 1.6 1.6 Zielsetzung der vorliegenden Arbeit 1 EINLEITUNG Zielsetzung der vorliegenden Arbeit Ziel dieser Arbeit ist die Untersuchung der möglicherweise zytokinartigen Eigenschaften des Hormons PRL. Das heißt, dass zum einen Aspekte einer potentiellen PRL-Synthese durch Immunzellen beleuchtet werden, zum anderen die Wirkung von PRL auf PBMC untersucht werden soll. Aspekte der Synthese von PRL durch PBMC sollen in Stimulationsversuchen mit Chlorophenylthio-(cpt)cAMP, PMA, PHA, PRL, LPS und entsprechenden Kostimulationen geprüft werden. Um eine umfassende Darstellung der Thematik liefern zu können, wird die Synthese auf möglichst vielen unterschiedlichen Ebenen der Proteinsynthese in menschlichen Zellen untersucht. Die Nachweismethoden erfassen dabei die Ebene der Aktivierung von Transkriptionsfaktoren (via EMSA), der Bildung der messenger Ribonukleinsäure (mRNA) (via Gene Expression (GE)Array) und der Synthese des fertigen Proteins (via enzyme-linked immuno sorbent assay (ELISA)). Die Wirkung von PRL auf PBMC soll einerseits bezüglich des Einflusses auf die Ausschüttung anderer Zytokine wie IFN-γ, IL-10, IL-12 und TNF-α via ELISA untersucht werden. Andererseits wird die Aktivierung von Transkriptionsfaktoren mittels EMSA geprüft und Fragen der Aktivierung von STATs via Dotblot beleuchtet. Ein weiterer zu untersuchender Aspekt ist das Expressionsverhalten von PRLR auf PBMC unter unterschiedlichen immunologischen Situationen. Ziel ist, eine Aussage darüber treffen zu können, ob PRL als Zytokin bezeichnet werden kann und - falls nicht - in welcher Hinsicht diesem Hormon zytokinartige Eigenschaften zukommen. 24 2 2 2.1 2.1.1 MATERIAL UND METHODEN MATERIAL UND METHODEN Material Geräte Analysenwaage 2002MPI (Sartorius, Göttingen) CO2 -Inkubator water-jacketed 3029 (Forma Scientific, Labotec, Göttingen) Dampf-Groß-Sterilisator Hydromat E 14 (Webeco, Bad Schwartau) Digitaler Auto-Radiograph (EG & G Berthold, Bad Wildbad) Dotblotter SRC 96 D Minifold I (Schleicher & Schuell, Dassel) Elecsys 2010 (Roche, Mannheim) ELISA-Reader (Anthos Labtec Instruments, Salzburg/Österreich) ELISA-Reader ThermoMax microplate reader (Molecular Devices, MWG Biotech, München) ELISA-Waschgerät (Biotest, Dreieich) Filmentwickler Crontex TSA (DuPont, Bad Homburg) Folieneinschweißgerät Polystar 100 GE (Ritsche + Herfurth, Hamburg) Gefriertruhe Bio-Freezer (Forma-Scientific, Labotec, Göttingen) Gelelektrophoresekammer Gelelectrophoresis Apparatus GNA-100 (Pharmacia LKB, Freiburg) Gelelektrophoresekammer (Micro Bio Tec Brand, Gießen) Gelelektrophoresekammer (Novex Novel Experimental Technology, San Diego, Californien, USA) Geltrockner Aldo-Xer (Schütt, Göttingen) Geltrockner SGD 2000 (Thermo Servant) 25 2.1 Material 2 MATERIAL UND METHODEN Heißluftsterilisator 2712 (Kottermann, Uetze-Hänigsen) Hybridisationsofen (Bachhofer Laboratoriumsgeräte, Reutlingen) Laborwaage 12116MP (Sartorius, Göttingen) Laborwaage LC 6200S (Sartorius, Göttingen) Netzgerät 3000/150 (Pharmacia LKB, Freiburg) pH-Meter pHM83 Autocal (Radiometer, Kopenhangen/DK) Pipette Combi-Pipette Typ 3180 (Eppendorf, Hamburg) Pipetten Multikanal, 5-50 µl / 50-300 µl (Titertek, ICN, Meckenheim) Pipetten Vario-Mikroliterpipetten 0,5-10 µl, 10-100 µl, 100-1000 µl (Eppendorf, Hamburg) Schüttler H S 250 basic (IKA Labortechnik, Staufen) Sterile Werkbank (Nuaire, Plymouth, MN/USA) Strahlendetektor LB 122 β,γ-Detector (Berthold, Wildbad) Strahlendetektor LB 1200 (Berthold, Wildbad) Thermocycler Thermomixer 5436 (Eppendorf, Hamburg) Vakuumkonzentrator Speed Vac Concentrator (Bachofer, Reutlingen) Vakuum-Pumpe Vacu Gene Pump (Pharmacia LKB, Freiburg) Vollblut-Zellzähler T-660 (Coulter Electronics, Krefeld) Zentrifuge Kühlzentrifuge: Centrikon T324 (Kontron, Hamburg) Zentrifuge Minifuge: Centrifuge 5415C (Eppendorf, Hamburg) Zentrifuge Omnifuge 2.0.RS (Heraeus Christ, Osterode) Zentrifuge Tischzentrifuge Sigma 2MK (Sigma Laborzentrifugen, Osterode) Zentrifuge Zytozentrifuge Cytospin-2 (Shandon, Frankfurt) 26 2.1 2.1.2 Material 2 MATERIAL UND METHODEN Laborbedarf Einmalspritzen, steril 2, 5 und 10 ml (Becton Dickinson, Heidelberg) Einschweißfolie Hybridization Bags (Life Technologies, Berlin) Filterpapier (Schleicher & Scuell, Dassel) Filmkassetten Hypercassettes Autoradiographie Cassettes (Amersham, Braunschweig) Frischhaltefolie (herkömmliche Haushaltsfrischhaltefolie) Gel-Bloting Papier GB 002 (Schleicher & Scuell, Dassel) Gewebekulturflaschen, beschichtet, 50, 260 und 800 ml (Nunc, Roskilde/DK) Gewebekulturplatten, 6- und 24-Loch-Platten (Falcon, Heidelberg) Gewebekulturschalen 100x20 mm (Falcon, Heidelberg) Hybridisationszylinder (Anfertigung der Wissenschaftlichen Werkstätten der Universität Lübeck) Kanülen 0,5x25 Nr. 18 (Becton Dickinson, Heidelberg) Kunststoffröhrchen, steril, mit Schraubverschluss, 15 und 50 ml (Sarstedt, Nürmbrecht) Kunststoffröhrchen, steril, mit Verschlusskappe, 5 ml (Greiner, Nürtingen) Latex-Einmalhandschuhe (Ansell, München) Mikroliterspritze (Hamilton, Bonaduz/Schweiz) Mikrotiterplatten, 96-Loch-Flach- und Rundboden (Falcon, Heidelberg) Mikrotiterplatten, 96-Loch-Flach- und Rundboden MAXISORP (Nunc, Roskilde/DK) Mikrotiterplatten 96-Loch, Flachboden, fluoreszenzarm (Millipore, Eschborn) Petrischalen für Gewebekulturen, 145 x 20 mm (Greiner, Nürtingen) Pipettenspitzen, 1-10 µl, kristall (Greiner, Nürtingen) 27 2.1 Material 2 MATERIAL UND METHODEN Pipettenspitzen, 10-100 µl, gelb (Greiner, Nürtingen) Pipettenspitzen, 100-1000 µl, blau (Greiner, Nürtingen) Pipettenspitzen, Combitips steril, 2,5 µl / 5 µl / 12,5 µl (Eppendorf, Hamburg) Pipettenspitzen, Comforttips, 10 µl / 200 µl (Eppendorf, Hamburg) Pipettenspitzen, PPN Precision Tip, 10-250 µl (Linbro, Amsterdam/Niederlande) Pipettenspitzen, Sterilfilter-Tips 0,5-10 µl (Biozym Diagnostik, Oldendorf) Pipettenspitzen, Sterilfilter-Tips 10-100 µl (Biozym Diagnostik, Oldendorf) ProbeQuantG-50 Micro Columns (Pharmacia LKB, Freiburg) Reaktionsgefäße 1,5 ml (Sarstedt, Nürmbrecht) Reaktionsgefäße 2 ml (Eppendorf, Hamburg) Reaktionsgefäße mit Schraubdeckel 1,5 ml (Sarstedt, Nürmbrecht) Röntgenfilm Biomax MX Scientific Imaging Film (Kodak, Rochester/USA) Röntgenfilm TMDG 1, P-MAT (Kodak, Rochester, USA) RNA-Konzentrierungs-Filter Microcon YM-30 (Millipore, Bedford/USA) Sarstedt Monovetten (Nürnbrecht) Stangenpipetten, steril 5, 10 und 25 ml (Greiner, Nürtingen) Zellschaber 18 cm (Falcon, Heidelberg) Zellschaber 25 cm (Costar, Cambridge, Massachussets, USA) 28 2.1 Material 2.1.3 2 MATERIAL UND METHODEN Zellkultur-Reagenzien, -Medien und -Zusätze Aqua ad injectabile, 10 ml-Ampullen (Braun, Heidelberg) Fetales Kälberserum, FCS, Myoklone plus-Qualität (Life Technologies, Berlin) Ficoll-Trennlösung 1,077 g/ml (Seromed Biochrom, Berlin) Bovines Insulin > 27 USP units/mg (Sigma, Deisenhofen) Kulturmedium RPMI 1640 (Seromed Biochrom, Berlin) L-Glutamin 200 mM (Seromed Biochrom, Berlin) Penicillin 10000 E/ml / Streptomycin 10000 µg/ml (Seromed Biochrom, Berlin) Phosphat Buffered Saline, PBS, ohne Ca2+ und Mg2+ (Life Technologies, Berlin) 2.1.4 2.1.4.1 Reagenzien für molekularbiologische Methoden Enzyme Oligonukleotidlabeling-Kit Ready To Go T4-Polynucleotid-Kinase für die Bandshift-Experimente (Pharmacia LKB, Freiburg) Multiples Myeloma Reverse Transkriptase 50 u/µl (Promega) ReactionReady Sweet PCR master mix (SuperArray) Ribonuklease Inhibitor Rnasin 40 u/µl (Promega) 2.1.4.2 Nukleotide und Nukleinsäuren α-32 P-dCTP, 3000 Ci/mmol (Amersham, Braunschweig) γ-32 P-ATP, 3000 Ci/mmol (Amersham, Braunschweig) dATP Li-Salz 100 mM (Roche, Mannheim) dGTP Li-Salz 100 mM (Roche, Mannheim) dTTP Li-Salz 100 mM (Roche, Mannheim) dCTP Li-Salz 10 mM (Perkin Elmer) DTT, 0,1 M (Promega, Heidelberg) Heringsperma-DNA, sonifiziert (Pharmacia LKB, Freiburg) diverse Oligonukleotide für die Bandshift-Versuche (siehe Tabelle 4; TIB Molbiol, Berlin; Santa Cruz Biotechnology, Heidelberg) 29 2.1 Material 2 MATERIAL UND METHODEN 2.1.4.3 Zelllinie, Prolaktin und Mitogene Re, Rc und Ra LPS aus E. coli, Stämme F515, J5 und K12 (extrahiert nach der Phenol-Chloroform-Ether-Methode in der Abt. f. Biophysik des Forschungszentrums Borstel, Referenz: [27]) Phythämagglutinin (PHA) (Sigma Chemie, Deisenhofen) Phorbol-12-myristyl-13-acetat (PMA) (Sigma Chemie, Deisenhofen) Humanes rekombinantes Prolaktin (R&D Systems GmbH, Wiesbaden-Nordenstadt) Duktale Mammakarzinomzellen (Zelllinie T-47D, ATCC American Type Culture Collection, Rockville, Maryland, USA) 2.1.5 2.1.5.1 Immunologische Reagenzien Antikörper für Dotblot anti-STAT5a-IgG, sc-1081 (monoklonales Kaninchen IgG, Santa Cruz Biotechnology, Heidelberg) anti-STAT5b-IgG, sc-1656 (monoklonales Maus IgG, Santa Cruz Biotechnology, Heidelberg) anti-STAT1-IgG, sc-346 (monoklonales Kaninchen IgG, Santa Cruz Biotechnology, Heidelberg) anti-Maus-IgG, sc-2008 (polyklonales Ziegen IgG, AP-konjugiert, Santa Cruz Biotechnology, Heidelberg) anti-Kaninchen-IgG, sc-2007 (polyklonales Ziegen IgG, AP-konjugiert, Santa Cruz Biotechnology, Heidelberg) 2.1.6 Testkits Die Testkitbestandteile sind nicht im Einzelnen aufgeführt. BandShift Kit (Pharmacia LKB, Freiburg) Elecsys humanes PRL (Roche, Mannheim) ELISA human TNF-α (R&D Systems GmbH) 30 2.1 Material 2 MATERIAL UND METHODEN ELISA human IFN-γ (Bender MedSystems, Wien/Österreich) ELISA human IL-10 (Bender MedSystems, Wien/Österreich) ELISA human IL-12 (Bender MedSystems, Wien/Österreich) First strand cDNA Synthesis Kit (SuperArray) Gene Expression Array Customer Array (Firma) RNA-Isolierungskit RNeasy Mini Kit (Quiagen, Hilden) RNA-Isolierungskit Purescript (Gentra, Minneapolis/USA) Protein-Bestimmungs-Kit Bio-Rad Protein Assay Dye Detergent Concentrate (Bio-Rad, München) RT PCR Primer Sets für PRLR, IFN-γ, PRL, IRF-1 (SuperArray) 2.1.7 Chemikalien und sonstige Reagenzien Alle außer den nachfolgend aufgeführten Chemikalien wurden in Analysenqualität von der Firma Merck (Darmstadt) bezogen: Aqua ad injectabilia, 10 ml-Ampullen (Braun, Heidelberg) Acrylamid-Bisacrylamid (30 %) (Sigma, Deisenhofen) Chlorophenylthio-cAMP (Biomol, Plymouth) Dodecylsulfat Na-Salz (Serva, Heidelberg) Dulbecco‘s Phosphate Buffered Saline DPBS (Bio Whittaker, Verviers/Belgien) Entwickler D 19 (Kodak, Rochester/USA) Färbelösung für Western blot Bio Rad Reagenz Ficoll 400 (Serva, Heidelberg) Fixierer Unifix (Kodak, Rochester/USA) Formaldehyddimethylacetal (Merck-Schuchardt, Hohenbrunn) 31 2.2 Methoden 2 MATERIAL UND METHODEN Gel-Ladepuffer 10x Loading Dye (250 mM Tris-HCl, pH 7,5, 0,2 % Bromphenol-blau, 0,2 % Xylenxylol, 40 % Glycerin; Pharmacia LKB, Freiburg) 2 β-Mercapto-Ethanol (Sigma, Deisenhofen) NP-40 (Nonided P40 Protein Grade Detergent; Calbiochem, San Diego, Kalifornien, USA) Pefablock SC Proteinaseinhibitor (Boehringer, Mannheim) Phenol, flüssig (Fluka, Neu-Ulm) Photoemulsion NTB-2 (Kodak, Rochester/USA) Poly dI-dC*Poly dI*dC (Pharmacia LKB, Freiburg) Rinderserumalbumin (BSA), Fraktion V (Boehringer, Mannheim) Substrat für Alkalische Phosphatase BCIP / NBT Alkaline Phosphatase Substrate Kit IV (Bio-Rad, München) Tetramethylethylendiamin (TEMED, Bio-Rad, München) 2.2 2.2.1 Methoden Zellpräparationen 2.2.1.1 Präparation von PBMC: Mononukleäre Zellen des peripheren Blutes (peripheral blood mononuclear cells, PBMC) wurden gewonnen aus Buffy Coats, einer angereicherten Leukozytenfraktion, die bei der Präparation von Erythrozytenkonzentraten und Blutplasma für Transfusionszwecke zurückbleibt. Die Isolierung erfolgte durch isopyknische Zentrifugation nach einer im Institut etablierten und in der Appendix (A.1) beschriebenen Methode. Dabei wurde eine Zellausbeute im Bereich von 1,0 - 1,5 x 109 Zellen/l erreicht. Die Zellkonzentration in dem Produkt wurde bestimmt mit Hilfe des Vollblut-Zellzählers T-660 (Coulter Electronics, Krefeld). 32 2.2 2.2.2 Methoden 2 MATERIAL UND METHODEN Zellkulturverfahren 2.2.2.1 Kulturen von PBMC: Das benötigte Endvolumen der Kultur von 4 ml wurde aus Zellsuspensionen mit einer Konzentration von 5 x 105 Zellen/ml auf sterile, pyrogenfreie 6Loch-Platten ausgesät. Als Kulturmedium verwendeten wir RPMI 1640 Medium der Firma Bio Whittaker mit Zusätzen (siehe Appendix, Kapitel A1.1). Es wurden jeweils möglichst geringe Volumina (insgesamt max. 10 %) der Stimulantien zugegeben. In Kontrollexperimenten wurde statt dessen das entsprechende Volumen des jeweiligen Mediums, das zur Verdünnung der Stimulantien bzw. Zusätze verwendet wurde, zugesetzt. Zum zeitlichen Ablauf der Kostimulation und den einzelnen Endkonzentrationen sei auf Kapitel 3 verwiesen. Die anschließende Inkubation erfolgte bei 37 ◦ C, 5 % CO2 und gesättigter Luftfeuchtigkeit ohne Medienwechsel bis zu 72 Stunden. 2.2.2.2 Vollblutansätze: Zur Erfassung der Effekte von PRL auf die Zytokinsynthese via ELISA nutzten wir Vollblutansätze nach einem im Institut etablierten und in [41] publizierten Verfahren. Blutproben wurden zehn gesunden Blutspendern in Sarstedt-Monovetten (15 IU Li-Heparin/ml Blut) abgenommen. 100 µl wurden zu 800 µl RPMI-Medium mit Zusätzen (siehe A1.1) pipettiert, das entweder 0 ng/ml, 100 ng/ml, 200 ng/ml oder 300 ng/ml rekombinantes PRL enthielt. Nach einer Stunde Vorinkubation wurden Aliquoten von 100 µl Kulturlösung oder LPS-Aliquoten (Endkonzentration: 100 ng/ml) hinzugegeben. Nach zwölf Stunden Inkubation wurden die Überstände für Messung von IL-10, IL-12 und TNF-α via ELISA abgenommen. 2.2.2.3 Präparation der Stimulantien: LPS und PMA wurden in RPMI-Medium unter sterilen Bedingungen ohne weitere Zusätze aliquotiert. PRL, cptcAMP, Noradrenalin und PHA wurden in sterilem Wasser aliquotiert. Dabei kamen die folgenden Endkonzentrationen zum Einsatz: 33 2.2 Methoden 2 MATERIAL UND METHODEN Stimulans Endkonzentration cptcAMP 50 µmol / 106 Zellen LPS 100 ng/ml Noradrenalin 100 ng/ml PHA 5 µg / 106 Zellen PMA 10 ng / 106 Zellen PRL 300 ng/ml Tabelle 3: Endkonzentration der Stimulantien in Zellkulturen von PBMC 2.2.2.4 Präparation von Mamma-Karzinom-Zellkulturen: Als Positivkontrolle einer PRLR-synthetisierenden Zelllinie dienten uns Zellen eines duktalen Mamma-Karzinoms (Zelllinie T-47D, ATCC). Die Stammkulturen wurden in Zellkulturflaschen in Konzentrationen von 2 x 105 Zellen/ml kultiviert. Die Zellen wurden zweimal wöchentlich nach mikroskopischer Kontrolle sedimentiert, gezählt, auf die entsprechenden Zellkonzentrationen eingestellt und in frisches Medium umgesetzt. Die Kulturmedien wurden mit folgenden Zusätzen versehen: Kulturmedium Zusatz RPMI 1640 10% 2 mM 1% 1,5 g/l 4,5 g/l 10 mM 1,0 mM 0,2 Units/ml FCS L-Glutamin Penicillin / Streptomycin Natriumbicarbonat Glucose HEPES Natriumpyruvat Rinderinsulin Tabelle 4: Zusätze des Kulturmediums für die Kultivierung der Mamma-Karzinom-Zelllinie 2.2.3 Präparation von Kernextrakten Prinzip der Herstellung von Kernextrakten ist die Lyse des Zellkerns mit Freisetzung des Kerninhalts inklusiv der Transkriptionsfaktoren und Trennung desselben vom übrigen Zellmaterial. 34 2.2 Methoden 2 MATERIAL UND METHODEN Zunächst werden die Zellen nach Stimulierung geerntet und gewaschen. Sodann werden im ersten Schritt die Zellmembranen lysiert und mit dem Zytosol entfernt. Die übrig bleibenden Zellkerne werden anschließend lysiert und das Karyoplasma freigesetzt. DNA sowie die Trümmer der Kernmembranen werden entfernt, wobei die Kernextrakte mit den darin enthaltenen Kernproteinen und Transkriptionsfaktoren im Überstand übrig bleiben. Aus etwa 4 x 106 Zellen wurden 50 µl Kernextrakt mit einer Protein-Konzentration von 4-6 µg/µl erreicht und in Aliquots von 10 µl bei -80 ◦ C tiefgefroren. Die Protein-Konzentration der Kernextrakte wurde mit Hilfe des Bio-Rad-Protein-Bestimmungs-Kits und des Hitachi-U3000-Photometers gemäß der Testanleitung des Herstellers durchgeführt. Für Einzelheiten zur Herstellung von Kernextrakten sei auf Kapitel A.2 der Appendix verwiesen. 2.2.4 Überstände Die Überstände der Stimulierungsexperimente enthielten die von den Immunzellen produzierten Zytokine und wurden zu deren Messung (via ELISA) steril abgenommen und bei -80◦ C ohne zwischenzeitliches Auftauen bis zur Messung tiefgefroren. 2.2.5 RNA-Extraktion Als Voraussetzung für das Arbeiten mit RNA müssen alle Materialien und Lösungen frei von RNAsen sein. Das heißt, dass die Materialien autoklaviert und acht Stunden bei 160 ◦ C sterilisiert sein müssen. Lösungen werden mit 0,02 % Diethylpyrocarbonat (DEPC) behandelt, was zu einer unspezifischen Alkylierung der RNAsen führt. Ferner sind sämtliche Arbeiten mit Einmal-Latex-Handschuhen durchzuführen, um eine nachträgliche Kontamination mit RNAsen zu vermeiden. Die RNA-Isolierung wurde entsprechend der Arbeitsvorschrift des Testherstellers mit Hilfe des RNA-Isolierungs-Kits von Invitrogen und des Kits von Quiagen durchgeführt. Durch spektralphotometrische Messung der optischen Dichte lässt sich sowohl die Menge als auch die Reinheit der zuvor isolierten RNA quantifizieren. Nukleinsäuren weisen bei einer Wellenlänge von 260 nm ein Maximum ihrer Lichtabsorption auf, während das von Proteinen bei 280 nm liegt. Die absolute Extinktion der Lösung in einer Quarzküvette gegen Wasser bei 260 nm ist dabei gemäß dem Lambert-Beerschen Gesetz linear vom RNA-Gehalt abhängig. Über den Quotienten beider Extinktionswerte (260 nm/280 nm) kann der Kontaminationsgrad der Probe mit Proteinen beurteilt werden. Der Quotient sollte zwischen 1,6 und 2,0 liegen. Die Proben wurden entweder sofort für die Gen-Expressions-Analyse eingesetzt oder bei 35 2.2 Methoden 2 MATERIAL UND METHODEN 80 ◦ C gelagert. Das Protokoll zur Bestimmung der Konzentration an RNA einer Probe ist in Kapitel A.3 der Appendix aufgeführt. 2.2.6 EMSA Ziel eines EMSA (electrophoretic mobility shift assays, im Folgenden auch als Bandshifts bezeichnet) ist der Nachweis von Wechselwirkungen von Nukleinsäuren mit Proteinen. Die in vivo vorliegenden Verhältnisse werden dadurch simuliert, dass Transkriptionsfaktoren aus dem Kern von stimulierten Zellen isoliert und diese dann mit in vitro synthetisierten, radioaktiv markierten Promotor-Abschnitten der Gene inkubiert werden, an die die Transkriptionsfaktoren in vivo binden (sogenannte Transkriptionsfaktor-Binderegionen oder - wie im Folgenden bezeichnet - Binding sites). Diese Bildung eines Komplexes aus Transkriptionsfaktor und Oligonukleotid kann aufgrund der dadurch entstehenden höhermolekularen Verbindungen mit nativer Acrylamid-Elektrophorese durch Trennung des Komplexes vom übrigen Reaktionsgemisch autoradiographisch sichtbar gemacht werden. Mit Hilfe der Bandshifts sollten Aussagen über die Aktivierung der Transkriptionsfaktoren NF-κB, IRF-1 und CRE binding protein (CREB) gewonnen werden. Eine normale autoradiographische Auswertung ermöglicht allenfalls semiquantitative Aussagen über das Ausmaß der Transkriptionsfaktor-Aktivierung, vergleichbar zur herkömmlichen RT-PCR. Wirkliche quantitative Aussagen sind nur mit Hilfe eines Instruments zur Bestimmung von Position, Intensität und Verteilung ionisierender Strahlung möglich. Dies ist mit einem Phosphoimager oder Digitalem Autoradiographen (DAR) möglich. 2.2.6.1 5´-32 P-Oligonukleotidlabeling: Die in Tabelle 5 aufgelisteten TranskriptionsfaktorPromotor-Binderegionen (Oligonukleotide) der verschiedenen Zytokin-Gene wurden für BandshiftExperimente verwendet. NF-κB ist ein ubiquitäres, innerhalb von Minuten aktivierbares Protein, das maßgeblich an der Aktivierung der verschiedenen Zytokin-Gene beteiligt ist und zur Entstehung einer proinflammatorischen Antwort entscheidend beiträgt (siehe auch Kapitel 2, Seite 17). IRF-1 ist ein Mitglied der Familie der IRF-Transkriptionsfaktoren, das am Entstehen einer antiviralen Immunantwort beteiligt ist. Zudem ist IRF-1 als Tumorsuppressor und Regulator von Zellzyklus und Apoptose bekannt [65]. Auch IRF-1 ist innerhalb von Minuten aktivierbar (siehe auch Kapitel 2, Seite 18). CREB (CRE binding protein) ist ein Transkriptionsfaktor, der über cAMP und Proteinkinase A aktiviert wird und die Transkription CRE-regulierter Gene bewirkt. Ein CRE-reguliertes Gen ist das PRL-Gen. 36 2.2 Methoden 2 MATERIAL UND METHODEN Oligonukle- Basensequenz (5’-XXX-3’) otid Zytokingen, auf dessen Promotor die Binderegion lokalisiert ist CREB S CTg AAg CAg gCC ATA AAA gAA TCC TCT gAC gTT TCT ATA AAg TAg CTA CTT TAT AgA AAC gTC AgA ggA TCC TTT TAT ggC CTg CTT CAg CRE in der ProlaktinPromotorregion IRF-1 S IRF-1 AS AgT TTC TAg TTT AAg TTT CCA Tgg AAA cTT AAA cTA gAA ACT IL-12 p40Gen-Promotor NF-κBC S gAT CCT CAg Agg ggA CTT TCC gAT G NF-κBC AS CAT Cgg AAA gTC CCC TCT gAg gAT C NF-κB-ConsensusSequenz/ TNF-α-Promotor Oct-1 S Oct-1 AS TCC ATG CAA ATG GAT ATC CAT TTG CAT GGA Oct-1-Kontrolle (unrelated DNA, Pharmacia) EBNA-1 S EBNA-1 AS TAG CAT ATG CTA TAG CAT ATG CTA EBNA-1-Kontrolle (Positivkontrolle, Pharmacia) CREB AS Tabelle 5: In den Bandshift-Experimenten verwendete Oligonukleotide. Erläuterungen: S: Sense; AS: Antisense; Referenzen: für NF-κB/TNF-α [75]; für IRF-1 [50]; für CREB [64].Hinweis: Die meisten o. g. Oligonukleotide wurden in Analysenqualität von TIB Molbiol (Berlin) bezogen. War der Lieferant Pharmacia (Freiburg), ist dies gesondert vermerkt. Von sämtlichen Oligonukleotiden wurde eine 50 nM-Stammlösung in aqua dest. hergestellt. EBNA = EpsteinBarr Virus Nuclear Antigen Das 5´-Endlabeling mit radioaktivem Phosphor (32 P, Amersham) dient der Detektierbarkeit der Oligonukleotide im Bandshift-Experiment und wurde mit Hilfe des Polynukleotidkinasekits der Firma Pharmacia nach deren Anleitung hergestellt (für detailliertere Angaben siehe A4.1). 2.2.6.2 Bindereaktion und elektrophoretische Trennung: Zum Bandshift werden etwa 10 µg Kernproteine zusammen mit 10 Mikrocurie radioaktiv markierter DNA eingesetzt. 37 2.2 Methoden 2 MATERIAL UND METHODEN Je nach Alter der radioaktiven Reagenzien entspricht dies etwa 2 µl. Während der Bindereaktion kommt es zur Bindung der Transkriptionsfaktoren aus dem Kernextrakt an den 32 P-markierten DNA-Abschnitt aus der Promotorregion des nachzuweisenden Gens. Dieses Reaktionsgemisch wird für ein bis zwei Stunden elektrophoretisch mit 200 V und 15 - 30 mA getrennt mit Hilfe eines Polyacrylamid-Gels, wobei die für das menschliche Auge unsichtbare Radioaktivität während des Laufvorganges zur Darstellung der Lauffront durch die Farbstoffe Bromphenol-blau und Xylenxylol ersetzt werden. Dabei laufen die ungebundenen Kernproteine und ungebundenen Oligonukleotide schneller im elektrischen Feld als die Komplexe aus Transkriptionsfaktor und gebundenem Oligonukleotid. Letztere lassen sich als radioaktiv detektierbare Bande sichtbar machen, während die ungebundenen Oligonukleotide als freie Radioaktivität am unteren Rand des Gels auftauchen (siehe Abbildung 10). Details zum Vorgehen sind im Anhang aufgeführt. 2.2.6.3 Trocknung, Detektion der Radioaktivität: Nach der elektrophoretischen Trennung wurden die Gele auf Filterpapier aufgebracht und im Geltrockner bei 95 ◦ C getrocknet. Die Auswertung erfolgte analog durch das Aufbringen eines Filmes in einer Filmkassette für 48-72 Stunden bei -80 ◦ C sowie autoradiographisch durch den Digitalen Autoradiographen. Zur Gewährleistung der Machbarkeit dieses Versuchsaufbaues wurde als Positiv-Kontrolle ein entsprechendes Experiment mit EBNA-1-DNA (EBNA-1 = Epstein-Barr Virus Nuclear Antigen-1) und EBNA-1-Extrakt (kloniert) durchgeführt. 38 2.2 Methoden 2 MATERIAL UND METHODEN Abbildung 10: Prinzip des EMSA; die freie DNA lagert sich unten ab, die Komplexe aus DNATranskriptionsfaktor-Antikörper als Supershift am weitesten oben; die Komplexe aus Transkriptionsfaktor und DNA befinden sich dazwischen; Abbildung aus dem Internet entnommen 2.2.7 Dotbloting Ziel des Dotblotings ist der Nachweis gesuchter Proteine mit Hilfe spezifischer Antikörper. Das Vorgehen entspricht im Allgemeinen dem beim Western Blot, doch werden beim Dotblot die zu untersuchenden Proteingemische nicht elektrophoretisch aufgetrennt, wodurch die zeitaufwendige Gelelektrophorese und der Umgang mit den giftigen Acrylamidgelen entfällt. Die zu untersuchenden Kernextrakte werden beim Dotbloting durch Vakuum direkt auf eine kleine runde Fläche (Dot) einer Membran aufgebracht. Anschließend wird mit einer Antikörperlösung gegen das gesuchte Protein, im zweiten Schritt mit einem gegen den ersten Antikörper gerichteten enzymverbundenen Antikörper (Alkalische Phosphatase-konjugiert) 39 2.2 Methoden 2 MATERIAL UND METHODEN inkubiert. Dabei findet eine Bindung des ersten Antikörpers (Primary) an das gesuchte Protein und des zweiten Antikörpers (Secondary) an den ersten statt. Dem zweiten Antikörper kann dann durch Zugabe einer Lösung Substrat für die Alkalische Phosphatase (BCIP / NBT Alkaline Phoshphatase Substrate Kit IV) angeboten werden, was zu einem farbigen Produkt verarbeitet wird. Die violetten Markierungen weisen somit die Anwesenheit des gesuchten Proteins im Kern nach. Wir verwendeten Gel-Bloting Papier der Firma Schleicher & Scuell, Dassel und kommerziell erhältliche Antikörper (Santa Cruz). Zur Feststellung der idealen Antikörperkonzentrationen stellten wir Verdünnungsreihen der ersten und zweiten Antikörper her und ermittelten so ein ideale Antikörper-Konzentrationen von 0,1 µg/ml in einer Lösung aus 10 % BSA für den ersten Antikörper und 1 µl/ml 10 % BSA-Lösung für den zweiten Antikörper. Die Inkubationen mit den Antikörpern fanden für den ersten Antikörper über Nacht und für den zweiten etwa 2 Stunden bei Raumtemperatur statt. Wir verwendeten erste Antikörper vom Kaninchen und von der Maus, die Zweitantikörper waren dementsprechend gegen Antikörper dieser Tiere gerichtet. Positive Kontrollen mit rekombinant hergestelltem, käuflichem Protein (STAT5b) führten zu dem erwarteten Nachweis durch den entsprechenden Antikörper. Zur negativen Kontrolle setzten wir proteinfreie Lösungen auf der Membran ein; weiterhin inkubierten wir versuchsweise ohne ersten oder ohne zweiten Antikörper und setzten rekombinante Proteine mit nicht darauf spezifische Antikörper ein (STAT5b mit Antikörper gegen STAT1). Alle dieser negativen Kontrollen führten zu keiner Markierung und bewiesen somit die Spezifität der Versuche. 2.2.8 ELISA (enzyme-linked immuno sorbent assay) Zur Messung der Zytokinkonzentration im Kulturüberstand diente die ELISA (enzymelinked immuno sorbent assay)-Technik. Das zu untersuchende Zytokin/Mediatorprotein (allgemein Antigen) bindet dabei im Rahmen einer Antigen-Antikörperreaktion an einen monoklonalen Primärantikörper (erster Antikörper), mit dem eine Mikrotiterplatte zuvor beschichtet wurde. Darauf erfolgt die Zugabe eines biotinilierten Sekundärantikörpers (zweiter Antikörper), der gegen ein weiteres Epitop des zu untersuchenden Zytokins gerichtet ist. Das Enzym (meistens eine avidingekoppelte Peroxidase) katalysiert eine Farbstoffreaktion mit einem Substrat (z. B. TMB). Die Geschwindigkeit der stattfindenden Reaktion ist innerhalb des Messbereichs proportional zur Enzymkonzentration und ermöglicht so durch photometrische Quantifizierung des Farbstoffumsatzes die Bestimmung der zuvor gebundenen Antigenmenge (siehe auch Abbil- 40 2.2 Methoden 2 MATERIAL UND METHODEN dung 11). Abbildung 11: Prinzip des Sandwich-ELISA; Abbildung entnommen aus http://www.biologie.de/Nuetzliches/FCI-CD/ausschnitte/KVs2.pdf Durch dieses sehr sensitive Verfahren können geringste Zytokinkonzentrationen gemessen werden (Nachweisgrenze je nach ELISA bei ca. 5 pg/ml), wobei in die Auswertung nur Werte einbezogen wurden, die eine mindestens ebenso große Absorption zeigten wie der kleinste Standard (beispielsweise zwischen 3 und 32 pg/ml bei Zytokinen). Die Durchführung erfolgte entsprechend der dem Testkit beigefügten Arbeitsvorschrift. 2.2.9 Elecsys Zur Messung der PRL-Konzentration in Kulturüberständen bedienten wir uns des PRLTestkits der Serie der Elecsys der Firma Roche. Auch bei dieser Methode bilden sich SandwichKomplexe. Der erste Antikörper ist biotinyliert und der zweite mit einem Ruthenium-Komplex markiert. Diese Mikropartikel werden in der Messzelle magnetisch auf der Oberfläche einer Elektrode fixiert. Das Anlegen elektrischer Spannung induziert sodann Chemolumineszenz, die mittels Photomultiplier gemessen wird. Die Ergebnisse werden anhand einer Kalibrationskurve ermittelt. Diese wird durch 2-Punkt-Kalibration und eine vom Hersteller mitgelieferte Masterkurve generiert. Der Nachweisbereich liegt zwischen 0,470-470 ng/ml, und ist damit sehr gut geeignet, endokrinologisch relevante PRL-Konzentrationen im Zellüberstand zu erfassen. Allerdings haben für die Messung von Zytokinen eingesetzte ELISAs untere Nachweisgrenzen im Pikogrammbereich und sind damit deutlich sensitiver. Ein ELISA mit solchen Nachweisgrenzen ist für PRL kommerziell nicht erhältlich. Die Durchführung erfolgte entsprechend der Arbeitsvorschrift des Testherstellers. 41 2.2 Methoden 2.2.10 2 MATERIAL UND METHODEN Nachweis der messenger RNA Eine weitverbreitete Möglichkeit, zu untersuchen, ob ein bestimmtes Gen in einer Zellpopulation aktuell transkribiert wird, ist der Nachweis der messenger RNA des gesuchten Genproduktes. Dazu wird die gesamte mRNA aus den zu untersuchenden Zellen isoliert (siehe Kapitel 2.2.5, Seite 35) und mit Hilfe des Enzyms Reverse Transkriptase in komplementäre DNA (cDNA) umgeschrieben. Die cDNA wurde in unseren Experimenten entweder direkt mittels Genexpressions-Analysen untersucht oder zunächst durch die Polymerase-KettenReaktion vervielfältigt und anschließend durch Gelelektrophorese identifiziert. 2.2.10.1 Nachweis mittels Polymerase-Ketten-Reaktion (PCR, polymerase chain reaction) Die Polymerase-Ketten-Reaktion (PCR) ist eine sehr gängige Methode, um kurze DNA-Abschnitte zu vervielfältigen. Mit jedem Zyklus der PCR erfolgt eine Verdopplung der entsprechenden DNA-Sequenz. Der zu amplifizierende Abschnitt der DNA wird durch die Auswahl der Primer festgelegt. Dies sind synthetisch hergestellte Oligonukleotide, deren Sequenz dem Anfang und dem Ende des zu vervielfältigenden DNA-Abschnittes entspricht. Ein Zyklus besteht aus drei Teilschritten. 1. Melting: Im ersten Schritt wird der DNA-Doppelstrang sowohl der Ausgangs-DNA als auch der Primer denaturiert und damit in seine Einzelstränge zerlegt. Dies geschieht durch Erwärmung des Reaktionsgemisches auf etwa 95◦ C, wobei es zur Lösung der Wasserstoffbrückenbindungen kommt, die für die Bildung des Doppelstranges verantwortlich sind. 2. Annealing: Die Temperatur des Reaktionsgemisches wird soweit gesenkt, dass sich die Primer an die Einzelstränge der DNA anlagern. Die hierfür erforderliche Temperatur hängt von den Primern ab und liegt meist 5◦ C unter ihrem Schmelzpunkt bei Werten um 50-60◦ C. 3. Elongation: Durch Erwärmung des Gemisches auf das Temperaturoptimum der DNAPolymerase (72◦ C) beginnt die Polymerase, komplementäre Nukleotide an die einzelsträngigen DNA-Stücke anzulagern. Den Anfang des entstehenden Stranges stellt der erste und den Endpunkt der zweite Primer dar. Die periodische Erwärmung und Abkühlung findet in einem Thermocycler statt. Nach meist 20-40 Zyklen werden die Reaktionsgemische dem Thermocycler entnommen und auf ein Agarosegel aufgetragen. Durch das Anlegen von Spannung kommt es zur Wanderung der Bestandteile ihrer Masse entsprechend im elektrischen Feld. Die vervielfältigten DNA-Stücke 42 2.2 Methoden 2 MATERIAL UND METHODEN bilden damit eine Bande, deren Lokalisation auf dem Gel von ihrer Masse und damit der Anzahl der Basenpaare abhängt. Parallel wird eine sog. Leiter aufgetragen, ein Gemisch aus DNA-Stücken definierter Größe, die ebenfalls durch das elekrische Feld ihrer Masse entsprechend aufgetrennt werden, und anhand derer die Anzahl von Basenpaaren einer Banden bildenden Struktur abgelesen werden kann. Wir synthetisierten cDNA aus der mRNA der Zellen unserer Versuchsreihen mit Hilfe des First Strand cDNA Synthesis Kit der Firma SuperArray (Versuchsanleitungen siehe Appendix). Für die PCR setzten wir Primer eines RT PCR Primer Sets der Firma SuperArray sowie mit dem Programm OligoExplorer kreierte Oligonukleotide ein (siehe Ergebnisteil). Als master mix für die PCR verwendeten wir ReactionReady Sweet PCR master mix der Firma SuperArray. 2.2.10.2 Nachweis mittels Genexpressions-Analysen (GEArray) Mit GenexpressionsAnalysen wird der Status der Genexpression einer Zellpopulation dargestellt. Dies geschieht unter Zuhilfenahme der mRNA der Zellen, die den aktuell in der Zelle transkribierten Genen entspricht. Diese mRNA wird aus den zu untersuchenden Zellen isoliert (siehe Kapitel 2.2.5) und mit Hilfe des Enzyms Reverse Transkriptase in DNA umgeschrieben und radioaktiv markiert. Man erhält somit komplementäre DNA (cDNA). Auf einer Nitrozellulosemembran sind vom Hersteller cDNA-Fragmente aufgetragen worden, die den einzelnen Genen entsprechen, die bei der Genexpressions-Analyse untersucht werden sollen. In einem Annäherungsschritt wird die cDNA den auf der Membranen aufgebrachten Genproben präsentiert. Tritt eine Hybridisierung zwischen der aus der mRNA synthetisierten cDNA und den auf der Membran aufgebrachten DNA-Genen ein, so wird dies sichtbar durch eine radioaktive Strahlung im definierten Bereich des aufgebrachten Genes auf der Membran. Autoradiographisch stellt sich dies als Punkt dar, wobei aus dem Hybridisierungsmuster auf der Membran sowie aus der Hybridisierungsintensität auf die Genexpression zurückgeschlossen werden kann. Durch dieses Verfahren werden eine quantitative sowie auch eine qualitative Aussage gemacht zu den Genen, die in der untersuchten Zellpopulation exprimiert werden. Die qualitative Aussage lässt sich durch das Vorhandensein eines radioaktiv strahlenden Bereiches auf der Membran tätigen. Dies zeigt eine Hybridisation der radioaktiv markierten cDNA an. Die quantitative Aussage entsteht durch den Vergleich der Expression verschiedener Gene der Zellen bei unterschiedlichen Situationen. So untersuchten wir die Genexpression bei 43 2.2 Methoden 2 MATERIAL UND METHODEN Abbildung 12: Prinzip des GEArrayAbbildung modifiziert nach der Anleitung des Herstellers PBMC im Ruhezustand und nach Zugabe verschiedener Stimulantien. Ein im Vergleich zu den Experimenten mit Zellen im Ruhezustand stärker radioaktiv strahlender Bereich auf der Membran deutet auf eine stärkere Expression des in diesem Bereich aufgebrachten Genes im stimulierten Zustand hin. Zum Versuchsaufbau sei auf die Versuchsanleitung der GEA-Kits der Firma Superarray verwiesen, deren Test-Kits wir zur Genexpressions-Analyse verwendeten (siehe Kapitel A.5 der Appendix). Eine kurze Zusammenfassung des Versuches wird nachfolgend wiedergegeben. Nach der RNA-Extraktion wurde die RNA mit Hilfe des aus Multiple-Myeloma-Zellen gewonnenen Enzyms Reverse Transkriptase in cDNA translatiert und mit 32 P radioaktiv markiert. Versuche mit 33 P, das ebenfalls für Genexpressions-Analysen empfohlen wird, führten zu wesentlich schwächeren Signalen und einer erschwerten Auswertung, so dass wir die Experimente mit einer Markierung mit 32 P durchführten. Gleichzeitig zur cDNA-Herstellung wird die Membran mit den darauf aufgebrachten Genen mit DNA aus Lachssperma bei 65 ◦ C in einem Waschpuffer inkubiert, um unspezifische Bindungen mit der DNA zu vermindern. Anschließend wird die cDNA mit dem gleichen Waschpuffer und unter Zusatz von Lachssperma-DNA für mehrere Stunden bei 65 ◦ C inkubiert. Nach mehreren Waschschritten mit Lösungen aus SDS und SSC (nähere Angaben in der Appendix) in verschiedenen 44 2.2 Methoden 2 MATERIAL UND METHODEN Konzentrationen ist die Membran fertig zur Auswertung. Dazu wird sie in feuchtem Zustand in Hybridisationsfolie eingeschweißt und entweder bei -20 ◦ C zur Belichtung einem Film ausgesetzt und mittels DAR autoradigraphisch ausgewertet. Die Membran kann durch Kochen mit einer konzentrierten SDS-Lösung zum erneuten Gebrauch wieder verwendet werden, indem die Hybridisierungen der auf der Membran aufgebrachten Gene mit der cDNA gelöst und die cDNA abgespült wird (Stripping). Ein Erfolg des Strippings wird durch die Exposition der gestrippten Membran mit einem Röntgenfilm dargestellt, der bei erfolgreichem Stripping keine Markierungen durch radioaktive Strahlen mehr aufweist. Nach der Entwicklung der GEArrays als Instrument zur Untersuchung der Genexpression im Jahre 1995 wurde deutlich, dass die Ergebnisse verschiedener Versuchsreihen mit unterschiedlichen Zellpopulationen nur bedingt miteinander vergleichbar sind und auch innerhalb einer Versuchsreihe die Ergebnisse deutlich divergieren können. Daher wurden Kriterien gesucht, deren Einhaltung die Ergebnisse verschiedener Versuchsreihen miteinander vergleichbar machen und die Genexpressions-Experimente als verlässliche Untersuchung in der Forschung etablieren. Dies führte zu den MIAME-Kriterien (Minimal Information about Microarray Experiments), die im Jahre 2001 in Miami formuliert und publiziert wurden [12]. Darin wird neben den technischen Anforderungen an die kommerziellen Anbieter von Tests für Genexpressions-Analysen auch eine fünfmalige Reproduktion der Ergebnisse von den Anwendern verlangt, bis deren Daten als verlässlich gelten und veröffentlicht werden dürfen. 2.2.11 Auswertung der Ergebnisse / Statistik Die dargestellten Ergebnisse basieren auf der Auswertung einer Reihe von Einzelexperimenten. Um den Ergebnisteil besser überschaubar und anschaulicher zu gestalten, sind die Ergebnisse als Mittelwerte ± Standardabweichung (standard deviation, SD) bzw. ± Standardfehler (standard error, SE) aus mehreren Einzelresultaten dargestellt. Mit Hilfe des “Kolmogorow-Smirnov Goodness of Fit” - Testes wurden die Stichproben hinsichtlich ihrer Normalverteilung untersucht. Die Unterschiede für den verteilungsunabhängigen Vergleich zweier verbundener Stichproben in der Mediatorproduktion bzw. in der mRNA- und Gen-Aktivierung wurden im Falle einer Normalverteilung mittels des “Student´s T”-Tests auf Signifikanz überprüft (** : p < 0,01; * : p < 0,05; Trend Tendenz: p < 0,10). Im Falle einer Nicht-Normalverteilung der Datensätze wurde der Vorzeichen-Rang-Test von Wilcoxon (Wilcoxon Matched-Pairs Signed-Ranks Test) verwendet. 45 2.2 Methoden 2 MATERIAL UND METHODEN Die Korrelationen zwischen biologischen und biophysikalischen Daten wurden unter Anwendung des Pearson-Tests (normalverteilte Daten) bzw. des Spearman-Tests (nicht normalverteilte Daten) analysiert. Sämtliche oben aufgeführten statistischen Testverfahren wurden mit dem Programm SPSS for windows durchgeführt. 46 3 3 ERGEBNISSE ERGEBNISSE Es wurde die Wirkung von PRL auf Immunzellen an Vollblutzellen und PBMC sowie eine mögliche Synthese von PRL durch Immunzellen an PBMC untersucht. 3.1 Wirkung von Prolaktin auf Immunzellen Im Rahmen der Untersuchung zur Wirkung von PRL auf Immunzellen wurden zunächst Experimente mit Vollblutzellen durchgeführt, um der Effekt auf die Gesamtheit der Blutzellen zu erfassen. Da in Vorversuchen Bandshift-Experimente an Vollblutkulturen methodische Schwierigkeiten aufwarfen, wurden die Untersuchungen an Transkriptionsfaktoren mit PBMC-Kulturen durchgeführt. Es wurde zunächst die Wirkung von PRL auf Vollblutzellen und anschließend den Effekt von PRL auf LPS-stimulierte Zellen untersucht. Im Vollblutansatz wurden Einflüsse von PRL auf die Synthese von IL-12, TNF-α, IL-10 und IFN-γ untersucht, Wirkungen auf eine mögliche Aktivierung der Transkriptionsfaktoren NF-κB und IRF-1 wurden an PBMC gemessen. 3.1.1 Einfluss von Prolaktin auf die Zytokinproduktion (IL-12, TNF-α, IL-10) im Vollblutansatz Zunächst wurde der Einfluss von Prolaktin auf die spontane Freisetzung der Zytokine IL-12, TNF-α und IL-10 im Vollblutansatz via ELISA untersucht. Es wurde mit Endkonzentrationen von 100 ng/ml, 200 ng/ml und 300 ng/ml an rekombinantem humanen PRL über einen Zeitraum von 12 Stunden inkubiert und die ZytokinKonzentrationen in den Überständen der einzelnen Ansätze mit der Konzentration im Überstand der Medium-Kontrolle verglichen. Bei Messungen an Vollblut zehn unterschiedlicher Blutspender ergaben sich keine statistisch signifikanten Unterschiede unter PRL-Inkubation im Vergleich zur Kontrolle, so dass sich folgern lässt, dass PRL allein nicht in der Lage ist, die Ausschüttung dieser Zytokine in merklichem Ausmaße zu beeinflussen. Desweiteren wurde ein etwaiger kostimulatorischer Effekt von PRL auf die LPS-induzierte Zytokinfreisetzung im Vollblutansatz betrachtet. Dieser Versuchsaufbau erfasst somit die Wirkung von PRL auf eine inflammatorische Immunantwort. Die Ansätze wurden eine Stunde mit 0 ng/ml, 100 ng/ml, 200 ng/ml und 300 ng/ml PRL vorinkubiert, anschließend wurde LPS (Endkonzentration: 100 ng/ml) hinzugegeben und für weitere 11 Stunden inkubiert. 47 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE Abbildung 13 zeigt die im ELISA gemessene Konzentration von IL-12 gegen die Endkonzentration von PRL im Ansatz aufgetragen, mit der vorinkubiert wurde. Abbildung 13: Einfluss von PRL auf die IL-12-Konzentration im Überstand von Vollblutkulturen nach LPS-Stimulation Dargestellt ist die IL-12-Sekretion LPS-stimulierter Vollblutzellen in Abhängigkeit von der PRLKonzentration, mit welcher vorinkubiert wurde. Es wurde mit PRL-Konzentrationen von 0, 100, 200 und 300 ng/ml für 1 h vorinkubiert und anschließend LPS (100 ng/ml) hinzugegeben. Die Überstände wurden nach 12 Stunden abgenommen. Abgebildet sind Box Plots mit Mittelwert (dicke Linie) und Median (dünne Linie). n = 10, statistische Signifikanzen gegen 0 ng/ml PRL sind mit (*) gekennzeichnet: p < 0,01. Die Konzentration von IL-12 wurde 11 Stunden nach Zugabe von LPS gemessen. Bei einer Fallzahl von n = 10 ergibt sich dabei ein signifikanter Anstieg der IL-12-Konzentration im Überstand bei Vorinkubation mit PRL von 2006 ± 247 pg/ml (2006 pg/ml ±12%) nach alleiniger Stimulation mit LPS auf 2617 ± 352 pg/ml (2617 pg/ml ±13%) bei Vorinkubation mit 300 ng/ml PRL. Es deutete sich eine Konzentrationsabhängigkeit des Effektes von der PRL-Konzentration, mit der vorinkubiert wurde, an, diese war jedoch nicht statistisch signi48 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE fikant. Abbildung 14: Einfluss von PRL auf die TNF-α-Konzentration im Überstand von Vollblutkulturen nach LPS-Stimulation Dargestellt ist die TNF-α-Sekretion LPS-stimulierter Vollblutzellen in Abhängigkeit von der PRLKonzentration, mit welcher vorinkubiert wurde. Es wurde mit PRL-Konzentrationen von 0, 100, 200 und 300 ng/ml für 1 h vorinkubiert und LPS (100 ng/ml) hinzugegeben. Die Überstände wurden nach 12 h abgenommen. Abgebildet sind Box Plots mit Mittelwert (dicke Linie) und Median (dünne Linie). n = 10, statistische Signifikanzen gegen 0 ng/ml PRL sind mit (*) gekennzeichnet: p < 0,01 Abbildung 14 zeigt die Konzentration von TNF-α im Überstand bei Vorinkubation mit PRL und LPS. Für eine Fallzahl von n = 10 ist die TNF-α-Konzentration im Überstand gegen die Endkonzentration an PRL, mit der vorinkubiert wurde, aufgetragen. Es zeigt sich ein signifikanter Anstieg der Konzentrationen von TNF-α im Überstand bei Vorinkubation mit PRL von 617 ± 72 pg/ml auf 746 ± 92 pg/ml. Eine Abhängigkeit dieses Effektes von der PRL-Konzentration im Ansatz besteht hier in Form eines Trends (p < 0,1). Zusamenfassend lässt sich feststellen, dass sowohl für TNF-α als auch für IL-12 ein kosti49 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE mulatorischer Effekt von PRL bei LPS-Stimulation vorliegt. Dabei konnten alle untersuchten PRL-Konzentrationen die TNF-α- und die IL-12-Sekretion signifikant erhöhen verglichen mit der Sekretion nach alleiniger Stimulation mit LPS (p < 0,01). Eine Zunahme dieses Effektes mit der PRL-Konzentration lässt sich für TNF-α in Form eines Trends feststellen, ist jedoch sowohl für TNF-α als auch für IL-12 statistisch nicht signifikant. Abbildung 15 zeigt die Konzentration von IL-10 im Überstand in Abhängigkeit von der PRL- Abbildung 15: Einfluss von PRL auf die IL-10-Konzentration im Überstand von Vollblutkulturen nach LPS-Stimulation Dargestellt ist die IL-10-Sekretion LPS-stimulierter Vollblutzellen in Abhängigkeit von der PRLKonzentration, mit welcher vorinkubiert wurde. Es wurde mit PRL-Konzentrationen von 0, 100, 200 und 300 ng/ml für 1 h vorinkubiert und LPS (100 ng/ml) hinzugegeben. Die Überstände wurden nach 12 h abgenommen. Abgebildet sind Box Plots mit Mittelwert (dicke Linie) und Median (dünne Linie). n = 10, statistische Signifikanzen gegen 0 ng/ml PRL sind mit (*) gekennzeichnet: p < 0,05 Endkonzentration im Ansatz. Die Konzentrationen von IL-10 ändern sich unter Vorinkubation mit PRL in Endkonzentrationen von 0 ng/ml, 100 ng/ml und 200 ng/ml nicht signifikant im Vergleich zu der nur mit LPS stimulierten Kontrolle. Bei einer PRL-Endkonzentration 50 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE von 100 ng/ml scheint ein Trend zur Minderung der IL-10-Synthese zu bestehen, dieser ist jedoch nicht statistisch signifikant. Bei Vorinkubation mit einer PRL-Konzentration von 300 ng/ml kommt es zu einer statistisch signifikanten Zunahme der IL-10-Konzentration (p < 0,05) von 15,79 ± 3,76 pg/ml (15,79 pg/ml ±24%) auf 20,87 ± 3,81 pg/ml (20,87 pg/ml ±18%). Dieser Effekt ist jedoch trotz seiner statistischen Signifikanz aufgrund der niedrigen Gesamtkonzentrationen an IL-10 nicht überzubewerten (siehe Abbildung 15). 3.1.2 Einfluss von PRL auf die IFN-γ-Freisetzung durch Vollblutzellen Sowohl unter Stimulation mit LPS als auch unter Stimulation mit PHA wirkt PRL auch auf die IFN-γ-Sekretion kostimulatorisch, während Stimulation mit PRL allein keine signifikante Erhöhung der IFN-γ-Konzentration bewirkt. Abbildung 16: Effekt von PRL auf die IFN-γ-Sekretion LPS-stimulierter Zellen im Vollblutansatz Dargestellt ist die IFN-γ-Sekretion LPS-stimulierter Vollblutzellen in Abhängigkeit von der PRLKonzentration, mit welcher vorinkubiert wurde. Es wurde mit PRL-Konzentrationen von 0, 5, 10 und 100 ng/ml für 1 h vorinkubiert und anschließend mit LPS (100 ng/ml) stimuliert. Die Überstände wurden nach 12 h abgenommen. Statistische Signifikanzen gegen 0 ng/ml PRL sind mit (*) markiert, Signifikanzen gegenüber der nächstniedrigeren PRL-Konzentration sind mit (#) markiert Abbildung 16 zeigt die Konzentration an IFN-γ im Überstand LPS-stimulierter Vollblutzellen aufgetragen gegen die Endkonzentration an PRL im Ansatz, mit welcher vorinkubiert wurde. Es konnten in Bezug auf die Sekretion von IFN-γ signifikante Effekte bereits bei 51 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE PRL-Endkonzentrationen von 5 ng/mlgemessen werden. Hier fand sich eine statistisch signifikante Abhängigkeit zwischen IFN-γ-Spiegeln und PRL-Endkonzentrationen (siehe Abbildung 16). 3.1.3 Einfluss von PRL auf die Kerntranslokation von (NF-κB und IRF-1) Die in Kapitel 3.1.2 dargestellten Ergebnisse zeigen, dass PRL allein keinen Einfluss auf die Zytokinsynthese in Vollblutzellen hat, wohl aber in Verbindung mit LPS. Daher wurde der Einfluss von PRL allein und mit Stimulation durch LPS auf eine Aktivierung von Transkriptionsfaktoren untersucht. Für diese Untersuchungen wurden drei unterschiedlich stimulierte Ansätze von PBMC verwendet und mit der als “K” deklarierten unstimulierten Kontrolle verglichen. Die im Folgenden als “PRL” und “PRL+LPS” bezeichneten Ansätze wurden zunächst mit PRL in einer Endkonzentration von 300 ng/ml stimuliert, während die Ansätze “K” und “LPS” ohne Stimulation mit PRL inkubierten. Nach einer Stunde wurde zu den Ansätzen “LPS” und “PRL+LPS” Lipopolysaccharid in einer Endkonzentration von 100 ng/ml zugegeben und alle Ansätze inkubierten für eine weitere halbe Stunde im Brutschrank. Es wurden demzufolge PBMC-Kulturen nach dem in Abbildung 17 dargestellten Schema angesetzt. Abbildung 17: Prinzip der Vorinkubation mit PRL und anschließenden Stimulation mit LPS Die Ansätze PRLünd PRL+LPS"werden mit einer PRL-Endkonzentration von 300 ng/ml für 1 h vorinkubiert. Anschließend werden die Ansätze LPSünd LPS+PRLmit LPS (100 ng/ml) für 30 min inkubiert. Nach 1,5 h Gesamtinkubationszeit werden die Kernextrakte extrahiert. Diese stimulierten Zellkulturen wurden auf Unterschiede bezüglich des Maßes an aktivierten Transkriptionsfaktoren untersucht. Weil aktivierte Transkriptionsfaktoren in den Kern trans52 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE lozieren, wurden mittels EMSA die Aktivität der Transkriptionsfaktoren NF-κB und IRF-1 in den Kernextrakten der einzelnen Ansätze bestimmt und verglichen sie mit der Kontrolle sowie untereinander. Für NF-κB bestimmten wir die Aktivität der Bindung des Transkriptionsfaktors an den TNF-α-Promotor, für IRF-1 an den IL-12-Promotor. Abbildung 18: Repräsentativer NF-κB-EMSA von n=3 Die Ansätze PRLünd PRL+LPS"wurden mit einer PRL-Endkonzentration von 300 ng/ml für 1 h vorinkubiert, anschließend wurden die Ansätze LPSünd LPS+PRLmit LPS (100 ng/ml) für 30 min stimuliert. Die Kernextrakte wurden nach 1,5 h Gesamtinkubationszeit extrahiert. Die Intensität der Banden ist ein Maß für die Bindungsaktivität von NF-κB an den TNF-α-Promotor K = unstimulierte Kontrolle, aqua = Wasserkontrolle (absolute Kontrolle) Abbildung 18 zeigt einen repräsentativen EMSA von drei durchgeführten Versuchen für NFκB. Die Intensität der spezifischen Banden ist dabei ein Maß für die Bindungsaktivität von NF-κB an den TNF-α-Promotor, die mit der Konzentration an NF-κB im Kernextrakt korreliert. Abbildung 19 zeigt einen repräsentativen EMSA für IRF-1 bei einer Fallzahl von n = 3. Die Intensität der spezifischen Banden entspricht hierbei der Bindungsaktivität von IRF-1 an den IL-12-Promotor und kann wiederum als Maß für die Konzentration von IRF-1 im Kern betrachtet werden. Den Abbildungen 18 und 19 ist zu entnehmen, dass mit PRL inkubierte Zellen (PRL) im Vergleich zur unstimulierten Kontrolle (K) eine erhöhte Bindungsaktivität für sowohl NF-κB als 53 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE auch IRF-1 aufweisen. Die Bindungsaktivität unter PRL-Inkubation entspricht in etwa der von mit LPS stimulierten PBMC. Abbildung 19: Repräsentativer IRF-1-EMSA von n=3 Die Ansätze PRLünd PRL+LPS"wurden mit einer PRL-Endkonzentration von 300 ng/ml für 1 h vorinkubiert, anschließend wurden die Ansätze LPSünd LPS+PRLmit LPS (100 ng/ml) für 30 min stimuliert. Die Kernextrakte wurden nach 1,5 h Gesamtinkubationszeit extrahiert. Die Intensität der Banden ist ein Maß für die Bindungsaktivität von IRF-1 an den IL-12-Promotor. K = unstimulierte Kontrolle Vorinkubation mit PRL und anschließende Stimulation mit LPS führt zu einer weiteren Zunahme der Bindungsaktivität von NF-κB und IRF-1. Die quantitative Erfassung der Signalintensitäten mittels DAR und anschließende statistische Auswertung liefert die in Abbildung 20 und Abbildung 21 dargestellten Ergebnisse. Abbildung 20 zeigt die Bindungsaktivität von NF-κB nach Stimulation im Vergleich zur unstimulierten Kontrolle als x-fache Verstärkung. Gezeigt sind Mittelwert und Standardfehler des Mittelwertes. Diese Darstellung erlaubt die Aussage, dass Stimulation mit LPS eine etwa 1,5fache Erhöhung der Bindungsaktivität von NF-κB - verglichen mit der unstimulierten Kontrolle - induziert, während Inkubation mit PRL ohne nachfolgende Stimulation eine etwa 2fache Erhöhung der Bindungsaktivität bewirkt (p < 0,05). Ein der Darstellung nach zu vermutende Trend, dass die Inkubation mit PRL höhere NF-κB-Spiegel induziert als Stimu54 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE lation mit dem starken Immunstimulans LPS, lässt sich statistisch signifikant jedoch nicht feststellen. In Verbindung mit LPS und Vorinkubation mit PRL werden mit etwa 3fach höheren Bindungsaktivitäten die höchsten Werte erreicht (p < 0,01). Der Effekt entspricht einem synergistischen (siehe Diskussion, Kapitel 4.1.3) Abbildung 20: Phosphoimager basierte quantitative Auswertung dreier EMSAs von NF-κB Dargestellt ist das Maß der Aktivierung von NF-κB in PBMC unter den beschriebenen Stimulationsbedingungen als x-fache Verstärkung der Signalintensität im Vergleich zur unstimulierten Kontrolle. Mittelwert und Standardfehler des Mittelwertes sind gezeigt * = signifikant versus unstimulierter Kontrolle oder entsprechender Probe: p < 0,05 K = unstimulierte Kontrolle Abbildung 21 zeigt die Phosphoimager-basierte quantitative Auswertung dreier EMSAs von IRF-1. Man sieht die Verstärkung der Signalintensität der spezifischen Bande für IRF-1 im Vergleich zur unstimulierten Kontrolle. Es ist eine etwa 1,5-fache Verstärkung der Signalintensität nach ausschließlicher Inkubation mit PRL als auch nach ausschließlicher Stimulation mit LPS (p < 0,05) zu erkennen. In Verbindung von Vorinkubation mit PRL und Stimulation mit LPS wird eine etwa 3,5-fache Verstärkung der Signalintensität erreicht (p < 0,01). Dies lässt einen Effekt vermuten, der den eines reinen additiven übersteigt und somit einem überadditiven-synergistischem entspricht (siehe Diskussion, Kapitel 4.1.3). 55 3.1 Wirkung von Prolaktin auf Immunzellen 3 ERGEBNISSE Abbildung 21: Phosphoimager basierte quantitative Auswertung dreier EMSAs von IRF-1 Dargestellt ist das Maß der Aktivierung von IRF-1 unter den dargestellten Stimulationsbedingungen als x-fache Verstärkung der Signalintensität im Vergleich zur unstimulierten Kontrolle. Mittelwert und Standardfehler des Mittelwertes sind gezeigt * = signifikant versus unstimulierter Kontrolle oder entsprechender Probe: p < 0,05 K = unstimulierte Kontrolle 3.1.4 Einfluss von PRL auf die Aktivierung von STAT5b in PBMC Aufgrund des offensichtlichen Klärungsbedarfs, der sich aus der Tatsache ergibt, dass es bei Inkubation mit PRL sowohl zur Aktivierung von NF-κB als auch von IRF-1 kommt, allerdings bei nachfolgender Stimulation mit LPS eine signifikante Änderungen der Zytokinsynthese bewirkt, wurde der Einfluss von PRL auf die Aktivierung von Signal transducers and activators of transcriptions (STATs) untersucht. Im Zusammenhang mit der Signaltransduktion über den PRL-Rezeptor ist STAT5b von enormer Bedeutung. Von STAT5b ist bekannt, dass es die NF-κB-vermittelte Signaltransduktion hemmt (siehe Einleitung). Es wurden daher Kernextrakte aus PBMC, welche nach dem in Abbildung 17 dargestellten Schema stimuliert worden waren, semiquantitativ mit einem Dotblot (n = 5) auf das Vorhandensein von STAT5b untersucht. STAT5b: Ein typischer Dotblot für STAT5b ist in Abbildung 22 dargestellt. Abbildung 22 ist zu entnehmen, dass sich die stärksten Indikatorreaktionen in den Kulturansätzen fanden, die allein mit PRL inkubiert wurden, was auf einen hohen Gehalt der Kernextrakte an STAT5b schließen lässt. In den Stimulationsansätzen mit LPS findet sich eine 56 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Abbildung 22: repräsentativer Dotblot mit Antikörpern gegen STAT5b (n = 5) Die Ansätze PRLünd PRL+LPS"wurden mit einer PRL-Endkonzentration von 300 ng/ml für 1 h vorinkubiert. Anschließend wurden die Ansätze LPSünd LPS+PRLmit LPS (100 ng/ml) für 30 min stimuliert. Die Kernextrakte wurden nach 1,5 h Gesamtinkubationszeit gewonnen. Die Farbintensität ist der Konzentration an STAT5b im Kernextrakt proportional. PBS = absolute Kontrolle deutlich geringere Farbintensität entsprechend einer geringeren Konzentration an STAT5b im Kernextrakt im Vergleich zur mit PRL inkubierten Probe kommt. Bei Vorinkubation mit PRL und nachfolgender Stimulation mit LPS kommt es zu einer stärkeren Farbreaktion als bei alleiniger LPS-Stimulation, aber zu einer geringeren Reaktion als bei alleiniger PRLInkubation. Durch das Zusammenwirken von PRL und LPS müssen also geringere Konzentrationen von STAT5b in den Kern transferiert werden als durch alleinige PRL-Inkubation. 3.2 Prolaktinsynthese durch PBMC Um einen möglichst sicheren und biologisch umfassenden Nachweis einer eventuellen Synthese von PRL durch PBMC zu führen, wurden drei unterschiedliche Ebenen der Generierung von Proteinen in eukaryontischen Zellen betrachtet. Im Einzelnen wurde gemessen: 1. die Aktivität von aus PBMC gewonnenen Kernextrakten bezüglich der Bindung an ein cAMP responsive element in der Promotorregion für extrahypophysäres PRL mittels EMSA, 2. die mRNA für extrahypophysäres PRL in PBMC mittels GEArray und RT-PCR, 3. die Konzentration an PRL in Kulturüberständen von PBMC. 57 3.2 Prolaktinsynthese durch PBMC 3.2.1 3 ERGEBNISSE Versuch des Nachweises einer möglichen PRL-Synthese auf Ebene der Transkriptionsfaktoren Wie bereits in der Einleitung dargestellt, gelang Berwaer et al. der Nachweis von CREs (TgACg) in der Promotorregion für extrahypophysäres Prolaktin u.a. in der Nähe der TATAABox (-20 bis 25) [6]. Für den Nachweis der Transkriptionsfaktoren mittels EMSA wurde als Oligonukleotid ein Abschnitt aus dem Exon 1a des Prolaktinspromotors, der den in Abbildung 1 gelb markierten Bereich enthält, verwendet. Inkubiert wurden PBMC mit PRL für 30 min, um eine eventuelle positive Rückkopplung einer etwaigen PRL-Synthese durch extrazelluläres PRL zu erfassen. Zudem wurden Experimente mit PHA-Stimulation durchgeführt. Stimuliert wurde entsprechend denen in Tabelle 6 aufgeführten Bedingungen. Ansatz Kontrolle (K) PHA PRL PHA 5 µg/ml - PRL 300 ng/ml Tabelle 6: Bedingungen für die Inkubation mit PRL und PHA; Inkubation über 30 min Abbildung 23 zeigt einen repräsentativen EMSAs (n = 5). Setzt man die Signalintensität der spezifischen Banden optisch in Beziehung zur Gesamtintensität der jeweiligen Spuren, so lässt sich feststellen, dass das Verhältnis dieser Größen nach Stimulation mit PHA und PRL in etwa dem der Kontrolle entspricht. Die quantitative Auswertung mittels Phosphoimager zeigte demzufolge keine statistischen Signifikanzen für die Beeinflussung der Signalintensität durch Stimulation mit PHA und PRL. Dementsprechend lässt sich die CRE-Bindungsaktivität des PRL-Promotors weder durch PHA noch durch PRL im Vergleich zur unstimulierten Kontrolle erhöhen. Weiter wurden PBMC mit cptcAMP, PMA, PHA und Noradrenalin (NA) mit den in Tabelle 3 aufgelisteten Endkonzentrationen über 20 min, 1 h, 3 h, 7 h, 10 h, 14 h, 17 h und 21 h stiumiert. Hier zeigte sich nach 20 min im Vergleich zur unstimulierten Kontrolle eine vermehrte Bindungsaktivität von CRE nach Stimulation mit cptcAMP allein oder cptcAMP + PHA und eine noch stärkere Bindungsaktivität bei Stimulation mit cptcAMP + PMA + PHA, jedoch nicht bei cptcAMP + PMA. Es wurde kein Anstieg der Bindungsaktivität für CRE nach Stimulation mit Noradrenalin gemessen. 58 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Abbildung 23: Repräsentativer EMSA nach Stimulation mit PHA und PRL von n = 5 Untersucht wurde die Aktivierung von Stimulation des Ansatzes PRL mit einer PRL-Konzentration von 300 ng/ml und des Ansatzes PHA mit einer PHA-Konzentration von 5 µg/ml über 30 min Die Intensität der spezifischen Banden bezogen auf die Intensität der gesamten Spur entsprechen für PHA und PRL etwa der der Kontrolle; K = unstimulierte Kontrolle aqua = Wasserkontrolle (absolute Kontrolle) 59 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Die Bindungsaktivität nahm mit der Stimulationszeit zu, erreichte bei 10 h ihr Maximum, um dann abzusinken und nach 21 h wieder das Niveau der unstimulierten Kontrolle zu erreichen. Ein repräsentativer EMSA von n = 5 für eine Stimulationszeit von 10 h ist in Abbildung 24 gezeigt. Abbildung 24: Repräsentativer EMSA nach Stimulation mit cptcAMP, PHA, PMA und NA aus n=5 Es wurde über 10 h mit den in Tabelle 3 aufgeführten Endkonzentrationen der einzelnen Stimulantien stimuliert. Die Intensität der spezifischen Banden bezogen auf die Intensität der gesamten Spur ist für cptcAMP+PHA+PMA deutlich höher als für die unstimulierte Kontrolle K = unstimulierte, aqua = Wasserkontrolle (absolute Kontrolle) 3.2.2 Versuch des Nachweises einer möglichen PRL-Synthese auf Ebene der mRNA Mittels GEArray sowie RT-PCR wurde die Fragestellung auf Ebene der mRNA untersucht. 3.2.2.1 Untersuchung mittels GEArray: Hierfür wurde ein kommerziell erhältlicher Customer Array, auf dem ein DNA-Stück aus dem plazentaren humanen PRL-Gen aufgetragen ist, verwendet. Zudem wurde mit dieser Methode die Expression von mRNA für IFN-γ, 60 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE IRF-1 und für den Prolaktinrezeptor untersucht. Als interne Kontrolle dafür, dass die untersuchte Probe auch tatsächlich RNA enthält, sind auf jeder Membran DNA-Abschnitte für Glyceraldehyd-3-phosphatdehydrogenase (GAPDH) und β-Aktin als typische HousekeepingGene, welche auch in anderen Methoden wie der PCR als solche verwendet werden, aufgetragen. GAPDH ist ein wichtiges Enzym der Glykolyse, das in fast allen Geweben in hohem Maße synthetisiert wird und damit als stimulationsunabhängige Positivkontrolle gilt. Auch β-Aktin wird von nahezu allen Geweben gebildet. In diesem Versuchsaufbau wurden ebenfalls zunächst mögliche rückkoppelnde Mechanismen von PRL auf seine Synthese durch Stimulation mit PHA, PRL und PHA + PRL untersucht. Die RNA-Extraktion wurde nach 30 min Stimulation gestartet. Abbildung 25 zeigt einen typischen GEArray aus n = 7 für mit PHA und PHA+PRL stimulierte PBMC. Es fanden sich in allen Versuchsreihen starke Signale für die Positivkontrollen und in mit PHA, PRL oder PHA + PRL inkubierten Ansätzen Signale für IFN-γ. In einer Versuchsreihe konnten wir im mit PHA+PRL inkubierten Ansatz RNA für IRF-1 nachweisen. In keinem der Versuche fanden sich Signale für PRL oder den PRL-Rezeptor. Abbildung 25: Repräsentativer Gearray nach Stimulation mit PHA und PRL aus n = 7 Stimulation über 30 min mit einer PHA-Konzentration von 5 µg/ml und einer PRL-Konzentration von 300 ng/ml. Es sind deutliche Signale für die beiden Positivkontrollen und bei PHA und PHA+PRL für IFN-γ zu sehen. K = unstimulierte Kontrolle 61 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Weiter wurden PBMC aus vier Buffycoats mit cptcAMP, PMA, PHA und Noradrenalin (NA) über 20 min, 1 h, 3 h, 7 h, 10 h, 14 h, 17 h, 21 h und 24 h mit den in Tabelle 3 aufgelisteten Endkonzentrationen stimuliert. Es zeigten sich in allen Ansätzen deutliche Signale für die beiden Positivkontrollen. In den mit cptcAMP + PMA, cptcAMP + PHA und cptcAMP + PMA + PHA stimulierten Ansätzen fanden sich zudem Signale für IFN-γ. Nach Stimulation mit cptcAMP + PHA + PMA trat weiter ein Signal für IRF-1 auf. Abbildung 26 zeigt einen typischen GEArray für Stimulation mit cptcAMP, cptcAMP+PHA, cptcAMP+PMA und cptcAMP+PHA+PMA. Unter allen Stimulationsbedingungen sind die beiden Positivkontrollen deutlich zu erkennen, weiter finden sich in den mit cptcAMP + PMA, cptcAMP + PHA und cptcAMP + PMA + PHA stimulierten Ansätzen Signale für IFN-γ. Ebenfalls zeigt sich das oben beschriebene Signal für IRF-1 nach Stimulation mit cptcAMP+PHA+PMA. Auch in diesem Versuchsaufbau wurde kein Signal für PRL oder seinen Rezeptor detektiert. Abbildung 26: Repräsentativer GEArray nach Stimulation mit cAMP, PHA und PMA Stimulation über 14 h; die Endkonzentrationen der Stimulantien sind in Tabelle 3 aufgelistet Es sind deutliche Signale für die beiden Positivkontrollen auf allen Membranen zu sehen; nach Stimulationen mit cptcAMP + PMA, cptcAMP + PHA und cptcAMP + PMA + PHA traten Signale für IFN-γ auf; in Kostimulation von cptcAMP mit PHA und PMA ist ein Signal für IRF-1 zu sehen. K = unstimulierte Kontrolle Da entsprechend einschlägiger Literatur [26] der PRLR von PBMC synthestisiert wird, sich aber in keinem unserer Experimente ein Signal für den PRLR fand, wurde als Positivkontrolle RNA aus duktalen Mamma-Karzinom-Zellen isoliert, um mögliche Fehler in dem Versuchssystem auszuschließen. Diese Zellen synthetisieren in hohem Maße den Prolaktinrezeptor. In dem entsprechenden GEArray zeigte sich ein deutliches Signal für den Prolaktin62 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE rezeptor neben den beiden Signalen für die Positivkontrollen (siehe Abbildung 27). Abbildung 27: Repräsentativer GEArray mit RNA aus Mamma-Karzinom-Zellen Es sind deutliche Signale für die beiden Positivkontrollen zu sehen, zudem ist ein vergleichbar starkes Signal für den PRLR zu erkennen 3.2.2.2 Untersuchungen mittels RT-PCR: Da die dargestellten Ergebnisse in Widerspruch zu den bisher in der Literatur publizierten Daten stehen, diese allerdings nicht auf Untersuchungen mittels GEArray, sondern auf RT-PCR-Daten beruhen (siehe Einleitung), sind Unterschiede in der Sensitivität der Methoden als Ursache für die Abweichung der dargestellten Ergebnisse von denen anderer Arbeitsgruppen anzunehmen. Es wurden daher dieselben Proben, die zuvor mittels GEArray untersucht worden waren, mittels RT-PCR untersucht. Als Primer wurden kommerziell erhältliche Testkits für PRL, IFN-γ, PRLR und IRF-1 der Firma Superarray eingesetzt. Diese entsprechen exakt den auf den GEArrays aufgetragenen Sequenzen. Als Kontrolle wurden für PRL und PRLR zudem Primer eingesetzt, die mit dem Programm Oligo Explorer (Version 1.1.0) erstellt worden waren, um ein Problem auf Ebene der verwendeten DNA-Sequenzen auszuschließen. Abbildungen 28 und 29 zeigen die Sequenzen und die Annealing-Temperaturen der eingesetzten Primer. 5’ - CTC TGG TGA AGT GTG TTT CC - 3’ upper primer 5’ - CTG AGG AGA GGT TAT GGA TG - 3’ lower primer Annealing-Temperatur: Tm (annealing) = 59,3◦ C Abbildung 28: Primer für PRL, upper primer blau unterlegt, lower primer gelb unterlegt Primer mit Programm Oligo Explorer 1.1.0 erstellt 63 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE 5’ - GCG ACC TTC ATT CAG ATA CC - 3’ upper primer 5’ - TCA GAA GTG GGA GGA AAG TC - 3’ lower primer Annealing-Temperatur: Tm (annealing) = 59,3◦ C Abbildung 29: Primer für PRL, upper primer blau unterlegt, lower primer gelb unterlegt Primer mit Oligo Explorer 1.1.0 erstellt Für die Synthese der cDNA wurden Proben derselben mRNA wie in den oben beschriebenen Versuchen mit dem Customer Array eingesetzt. Für die Stimulationsreihen mit PHA und PRL ergab sich nach elektrophoretischer Auftrennung des fluoreszierenden PCR-Produktes das in Abbildung 30 dargestellte Ergebnis. 64 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Abbildung 30: Ausschnitt aus der elektrophoretische Auftrennung des PCR-Gemisches nach Inkubation mit PHA und PRL Es wurde über 30 min mit einer PHA-Konzentration von 5 µg/ml und einer PRL-Konzentration von 300 ng/ml inkubiert. Bei gleicher Intensität der GAPDH-Banden ist ein deutliches Signal für IRF-1 nach PRL-Inkubation zu sehen und Signale für IFN-γ nach Stimulation mit PHA und PHA+PRL. Der PRLR zeigt das stärkste Signal in der unstimulierten Kontrolle gefolgt von der PRL-inkubierten Probe. Nach Stimulation mit PHA und PHA+PRL zeigen sich keine Signale. Bei alleiniger PRLInkubation ist in keinem der Ansätze ein Signal zu finden K = unstimulierte Kontrolle, OE = Der verwendete Primer wurde mit dem Oligo Explorer kreiert. Bei fehlender Angabe wurden Primer der Firma SuperArray benutzt. Es zeigt sich, dass wie in den GEArray-Experimenten auch hier weder in der unstimulierten Kontrolle noch nach Inkubation mit PHA und PRL oder PHA + PRL Signale für PRL zu sehen sind - weder bei Verwendung des Primers von SuperArray noch bei Benutzung des mit Oligo Explorer hergestellten. Die PCR des PRLR zeigt nach elektrophoretischer Auftrennung des Gemisches deutliche Signale für die unstimulierte Kontrolle und ein etwas schwächeres Signal bei der PRLInkubation bei etwa gleicher Intensität der GAPDH-Kontrolle in beiden Proben. Sowohl bei Stimulation mit PHA als auch bei Inkubation mit PHA und PRL lässt sich ein deutlich schwächeres Signal für PRLR erkennen als in der unstimulierten Kontrolle. Für den Transkriptionsfaktor IRF-1 ist ein deutliches Signal nach Inkubation mit PRL zu sehen, die übrigen Signale entsprechen denen der Kontrolle. Auch die zusätzliche Stimulation mit PHA wirkt keinesfalls additiv auf die IRF-1-Synthese, sondern vielmehr scheint die 65 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Wirkung von PRL hier durch zusätzliche Stimulation mit PHA vermindert zu werden. Die PCR für IFN-γ zeigt starke Signale für Stimulationen mit PHA und PHA+PRL, allerdings kein Signal bei alleiniger Inkubation mit PRL. Die Abbildungen 31 und 32 zeigen die Ergebnisse der PCR nach Stimulationsexperimenten mit cptcAMP, PMA und PHA. Die cDNA wurde aus derselben RNA synthetisiert, die auch mittels GEArrays untersucht wurde. Dementsprechend sind die Stimulationsbedingungen identisch mit den oben beschriebenen. Abbildung 31 zeigt das PCR-Ergebnis nach 7 h Stimulation der Zellkulturen mit cptcAMP, cptcAMP+PMA, cptcAMP+PHA und cptcAMP+PHA+PMA sowie die unstimulierte Kontrolle. Bei etwa gleicher Intensität der Banden der GAPDH-Kontrolle finden sich deutliche Signale für PRL nach Stimulation mit cAMP und cAMP+PHA, während die unstimulierte Kontrolle, cAMP+PHA und cAMP+PMA+PHA Signale von deutlicher geringerer Intensität zeigen. Ebenfalls sind Signale für den PRLR vorhanden, die etwa gleich intensiv sind mit leicht erhöhten Werten bei cAMP+PHA und cAMP. Abbildung 32 stellt das Ergebnis von Zellen desselben Buffycoats nach 14 h Stimulation dar. Für IFN-γ sieht man sehr starke Banden nach Stimulation mit cptcAMP+PHA und cptcAMP+PHA+PMA. Der PRLR liefert in der unstimulierten Kontrolle das stärkste Signal. Damit scheint die Neusynthese des PRLR im Vergleich zur unstimulierten Kontrolle nach 14 h Stimulation unter allen Stimulationsbedingungen abzunehmen. Dieses Ergebnis zeigte sich sowohl bei Verwendung des Primers der Firma SuperArray als auch bei Verwendung des mit dem Oligo Explorer entworfenen. Für PRL sind auch nach 14 h Stimulation Signale bei der Stimulation mit cptcAMP und cptcAMP+PHA zu sehen, allerdings tritt nach dieser Zeit auch ein Signal für die Stimulation mit cptcAMP+PMA auf, das in der Versuchsreihe das stärkste ist. Die Synthese von mRNA, die für PRL kodiert, scheint für letztere Bedingungen daher später ein Maximum zu erreichen als bei Stimulation mit cptcAMP+PHA oder cptcAMP. Dieses Ergebnis ließ sich mit beiden eingesetzten Primern für PRL reproduzieren. Für IRF-1 entsprechen die Signale in allen Versuchsreihen Signale denen der unstimulierten Kontrolle. 66 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Abbildung 31: Ausschnitt aus der elektrophoretischen Auftrennung des PCR-Gemisches nach Stimulation mit cptcAMP, PHA und PMA Stimulation über 7 h; die Endkonzentrationen der Stimulantien sind in Tabelle 2 aufgelistet Bei etwa gleicher Intensität der GAPDH-Banden sind die Banden für den PRLR in allen Stimulationen etwa entsprechend der Intensität der Bande der unstimulierten Kontrolle. Deutliche Banden für PRL sind bei Stimulation mit cptcAMP und cptcAMP+PHA zu sehen K = unstimulierte Kontrolle, cAMP = cptcAMP, CPP = cptcAMP+PHA+PMA OE = Der verwendete Primer wurde mit dem Oligo Explorer kreiert. Bei fehlender Angabe wurden Primer der Firma SuperArray benutzt. 67 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Abbildung 32: Ausschnitt aus der elektrophoretischen Auftrennung des PCR-Gemisches nach Stimulation mit cAMP, PHA und PMA Stimulation über 14 h; die Endkonzentrationen der Stimulantien sind in Tabelle 3 aufgelistet Bei etwa gleicher Intensität der GAPDH-Banden sind bei Stimulation mit cptcAMP+PHA und cptcAMP+PHA+PMA deutliche Banden für IFN-γ zu sehen. Die stärksten Signale für den PRLR treten bei der unstimulierten Kontrolle auf. Banden für PRL sind bei Stimulation mit cptcAMP, cptcAPMP+PMA und cptcAMP+PHA zu sehen, die stärkste Bande tritt hier bei Stimulation mit cptcAMP+PMA auf. Die IRF-1-Banden entsprechen in ihrer Intensität in allen Ansätzen der Intensität der Bande der unstimulierten Kontrolle. K = unstimulierte Kontrolle, cAMP = cptcAMP, CPP = cptcAMP+PHA+PMA OE = Der verwendete Primer wurde mit dem Oligo Explorer kreiert. Bei fehlender Angabe wurden Primer der Firma SuperArray benutzt. 68 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE Abbildung 33: Ausschnitt der elektrophoretischen Auftrennung des PCR-Gemisches aus cDNA von Mamma-Karzinom-Zellen Es ist ein deutliches Signal für den PRLR zu erkennen bei fehlendem Signal für PRL OE = Der verwendete Primer wurde mit dem Oligo Explorer kreiert. Bei fehlender Angabe wurden Primer der Firma SuperArray benutzt. Abbildung 33 zeigt das Ergebnis der PCR mit cDNA, die aus mRNA einer duktalen MammaKarzinom-Zelllinie gewonnen wurde. Hier findet sich ein deutliches Signal für den PRLR und kein Signal für PRL. Bei einer Zyklenzahl von 35 und Verwendung des Primers, der mit dem Oligo Explorer entworfen wurde, ist das Signal für den PRLR deutlich stärker als die entsprechenden Signale in den vorangegangenen Abbildungen. Da diese Probe auch zu einem Signal für PRLR im GEArray führte, lässt sich aufgrund der Signalunterschiede folgern, dass der Grund der Differenzen zu den Ergebnissen der GEArrays in der geringeren Sensitivität des GEArrays zu suchen ist (siehe Diskussion). 3.2.3 Versuch des Nachweises auf Ebene des Proteins Um zu klären, ob das fertige Protein PRL im Überstand der PBMC in endokrin bedeutsamen Konzentrationen nachzuweisen ist, benutzten wir einen ELISA von der Firma Roche (Elecsys-System). Wir stimulierten Zellen mit cAMP, cAMP + PHA, cAMP + PMA, cAMP + PHA + PMA und Noradrenalin unter denselben Bedingungen wie oben dargestellt. In den Überständen konnten wir - bei einer unteren Nachweisgrenze von 5,6 ng/ml - kein PRL detektieren. Geht man davon aus, dass das PRL von PBMC in denselben Konzentrationen wie 69 3.2 Prolaktinsynthese durch PBMC 3 ERGEBNISSE andere Zytokine, z.B. IL-12 und TNF-α, immunologisch akiv ist, so ist die Sensitivität des Elecsys zu gering, um Aussagen über die PRL-Synthese in PBMC treffen zu können. 70 4 4 DISKUSSION DISKUSSION In der vorliegenden Arbeit wurde das zytokinartige Verhalten des Hormons PRL untersucht. Zum einen wurde der Einfluss von PRL auf die Zellen des Immunsystems bzw. auf PBMC beleuchtet, zum anderen die Frage einer etwaigen Synthese von PRL durch Immunzellen. Wie in der Einleitung bereits dargestellt, ist die Zytokin-Sekretion durch Immunzellen nicht Folge einer Freisetzung aus intrazellulären Speichern, sondern Zytokine werden nach entsprechender Zellaktivierung neu synthetisiert und anschließend schlagartig freigesetzt. Da aufgrund der starken Bindung der Zytokine an ihren Rezeptor extrem geringe Mengen eines Zytokins genügen, um einen biologischen Effekt zu erzielen, können mit kommerziell erhältlichen Nachweissystemen für PRL, die endokrine und damit systemisch aktive Konzentrationen von PRL messen, keine definitive Aussage zu einer etwaigen Synthese immunologisch aktiver Konzentrationen von PRL durch PBMC getroffen werden. Da aber die Zytokin-Sekretion auf Neusynthese beruht, untersuchten wir die Synthese von PRL auf Ebene der Aktivierung von Transkriptionsfaktoren und der Bildung von mRNA für PRL. Eine weitere Eigenschaft von Zytokinen ist die Beeinflussung der Synthese anderer Zytokine. Diesbezüglich führten wir ausführliche Untersuchungen zum Einfluss von PRL auf die Synthese von TNF-α, IL-12, IFN-γ und IL-10 sowie auf die Aktivierung der Transkriptionsfaktoren IRF-1 und IFN-γ durch. Da Zytokine nicht antigenspezifisch sind, ist es von großer Wichtigkeit für die Spezifität des Immunsystems, dass die Empfindlichkeit der Zielzellen für ein Zytokin und damit die Expression von Zytokinrezeptoren durch äußere Signale reguliert werden kann. Wir untersuchten in diesem Zusammenhang die Neusynthese von PRLR unter unterschiedlichen Stimulationsbedingungen. Ein besonders interessanter Aspekt war hier die Frage des Verhaltens unter PRL-Inkubation. Zum einen könnte man eine verminderte Neusynthese im Sinne eines Schutzes vor Überaktivierung erwarten, wie es für Hormone typisch ist. Es wäre zum anderen auch plausibel, dass eine vermehrte Neusynthese von PRLR im Sinne einer gesteigerten Empfindlichkeit der Zielzelle für weitere PRL-Moleküle stattfindet, wie man es von Zytokinen kennt, die in deutlich geringeren Konzentrationen an einer immunologischen Synapse wirken. 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem Es sei deutlich darauf hingewiesen, dass bei unseren Untersuchungen zu der Wirkung von PRL auf Immunzellen hypophysäres humanes PRL verwendet wurde. Die Frage, ob sich etwaiges von Immunzellen synthetisiertes PRL genauso verhält, kann nach dem aktuellen 71 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION Stand der Literatur nicht beantwortet werden. In der Einleitung wurde in Kapitel 1.5 die in der Literatur vielfach vorbeschriebene immunmodulatorische Wirkung von hypophysärem PRL dargestellt. Da es jedoch zumindest scheinbar gegensätzliche Ergebnisse einzelner Arbeitsgruppen gibt, ist die in Kapitel 1.5 dargestellte Wirkung keinesfalls unumstritten. In Experimenten mit PRLR-defizienten Mäusen beispielsweise konnte die Arbeitsgruppe um Bouchard keine nennenswerten Immundefekte feststellen [9]. Im Folgenden sind zwei offensichtlich konträre Ergebnisse einander gegenübergestellt: Our results (...) directly show that the PRL/ In vivo scheint Prolaktin vielfältige Wir- PRLR ligand receptor system is not essen- kungen auf immunologische Funktionen tial for normal immune system develop- zu haben (...). ment. Bei Mäusen, denen einmal täglich Our data further demonstrate that PRLR mehrere Tage lang pharmakologische Do- signaling is not required for normal immu- sen von Prolaktin subkutan appliziert wur- nity and call into question the numerous re- de, zeigte sich bei den Lymphozyten in der ports attributing a major role for PRL in Milz ein dosisabhängiger Anstieg in B- und immunoregulation. T-Zellaktivität. Bouchard B, Ormandy CJ, Di Santo JP, Kelly PA., Immune system development and function in prolactin receptor-deficient mice, J Immunol. 1999, [9] Schedlowski, Tewes; Psychoneuroimmunologie, 1996, [69] Ob und wie diese Ergebnisse miteinander zu vereinbaren sind, ist Gegenstand aktueller Diskussionen. Ein möglicher Aspekt zur Erklärung ist, dass Zytokinfunktionen immer redundant sind und somit andere Zytokine die Funktion von PRL bei den knock out Mäusen übernehmen können [79]. Somit wäre der aufgezeigte Gegensatz nur ein scheinbarer. Ein Ziel unserer Untersuchungen war es, durch in vitro-Versuche die Wirkungen von PRL auf menschliche Immunzellen möglichst genau zu untersuchen, um Effekte für den Makroorganismus und Beeinflussungen im Mikromileu der Zelle fundiert diskutieren zu können. Dazu war es nötig, Signalverstärkungen und -auslöschungen auf Ebene der Transkriptionsfaktoren zu untersuchen. In der Literatur ist IRF-1 als ein von PRL aktivierter Transkriptionsfaktor weitaus am meisten und besten untersucht. Zu einer direkten Beeinflussung des nach dem aktuellen Wissensstand wichtigsten proinflammatorischen Transkriptionsfaktor NF-κB liegen bis auf die von uns erhobenen, in dieser Arbeit dargestellten und in [10] publizierten Daten bisher keine Ergebnisse vor. Es werden daher zunächst die bisher veröffentlichten Daten zu der Aktivierung von IRF-1 durch PRL dargestellt und im Hinblick auf unsere Ergebnisse zu IRF-1 und NF-κB diskutiert. 72 4.1 4.1.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION Aktivierung von IRF-1 durch PRL In der Einleitung wurde die Bedeutung von IRF-1 für das Immunsystem dargestellt. Das IRF-1-Gen wird in hohem Maße durch PRL aktiviert. Der Promotor enthält unter anderem Bindungsstellen für NF-κB, Specificity Protein-1 (Sp1) und eine Interferon γ activated site (GAS). Die meisten Zytokine beeinflussen das IRF-1 Gen durch Aktivierung von STATs, die wiederum entweder mit dem GAS Element interagieren oder durch Translokation von NFκB in den Zellkern das IRF-1 Gen aktivieren [79]. Wie in der Einleitung dargestellt, ist der Prototyp der Signaltransduktion nach Aktivierung des PRL-Rezeptors der Jak/STAT-Weg. Die funktionelle Aktivierung der STATs erfolgt dabei nicht nach dem “Alles-oder-NichtsPrinzip”, sondern wird vielmehr durch Interaktion mit Kernproteinen - den Koaktivatoren - moduliert. Am besten untersucht sind dabei die sogenannten Adapterproteine p300/CBP. Diese binden nicht direkt an DNA, sondern modulieren als größere Einheit viele Proteine in ihrer Aktivität, die wiederum an DNA binden [32]. Sie entscheiden dabei über die Reihenfolge, in der Gene einer Zelle transkribiert werden. Unterschiedliche Untereinheiten von p300/CBP scheinen mit STAT1 zu interagieren [32], eine dieser auch mit STAT5 [63]. Ob hierbei kompetetive Effekte auftreten, ist bislang nicht bekannt [79]. Die Beeinflussung des IRF-1 Gens durch PRL geschieht nun auf zwei unterschiedlichen Wegen [79]: 1. PRL aktiviert STAT1. Dieses transloziert in den Kern und bindet an das GAS-Element des IRF-1 Promotors, was dessen Aktivierung nach sich zieht. Zudem ist eine Sp1 Bindungsstelle entscheidend für die Aktivierung des IRF-1 Gens. Voraussetzung ist die Anwesenheit von p300/CBP. Wahrscheinlich wird das IRF-1 Gen durch die Bildung eines Komplexes aus Sp1, STAT1 und p300/CBP aktiviert. 2. PRL aktiviert STAT5b. Yu-Lee et al. diskutieren, dass Stat5b vermutlich das in begrenztem Ausmaß vorhandene p300/CBP verbraucht und damit die Bildung des Komplexes aus STAT1, Sp1 und p300/CBP verhindert [79]. Zudem kann durch eine TNF-α-vermittelte Aktivierung von NF-κB und anschließende Bindung von NF-κB an die entsprechende Bindungsstelle des IRF-1 Promotors das IRF-1 Gen aktiviert werden. Auch hierzu ist die Anwesenheit von p300/CBP Voraussetzung, so dass STAT5b die Wirkungen von NF-κB auf IRF-1 hemmen kann [79]. NF-κB wirkt synergistisch mit STAT1 auf die Aktivierung des IRF-1 Promotors, jedoch antagonisiert STAT5b die Wirkungen von NF-κB auf das IRF-1 Gen. Sowohl STAT1 als auch STAT5b werden durch PRL aktiviert [79]. Die beschriebenen Mechanismen sind in Abbildung 34 zusammengefasst. 73 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION Abbildung 34: Mechanismus der Regulation der Transkription des IRF-1 Gens durch PRL 1. Viele Mediatoren verstärken die IRF-1 Aktivierung durch PRL wie STAT1, Sp1 und p300/CBP 2. STAT5b inhibiert die Transkription von IRF-1 3. Der IRF-1 Promotor kann durch STAT1 und durch TNFα-aktiviertes NF-κB aktiviert werden. Somit kann PRL über STAT5b die TNF-α-induzierte IRF-1-Aktivierung inhibieren Abbildung modifiziert nach Yu-Lee L-Y. Stimulation of interferon regulatory factor-1 by prolactin. Lupus 10(2001), 591-599, [79] 74 4.1 4.1.2 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION Aktivierung von NF-κB durch PRL Die umfassende Bedeutung von NF-κB für das Immunsystem wurde bereits in der Einleitung dargestellt. Eine Aktivierung von NK-κB durch PRL ist bisher in der Literatur nicht beschrieben. Die Ergebnisse von Yu-Lee et al. legen ganz im Gegenteil eine indirekte negative Beeinflussung der NF-κB-vermittelten Wirkungen durch PRL nahe (siehe Kapitel 4.1.1) 4.1.3 Einordnung der eigenen Ergebnisse zur Beeinflussung von IRF-1 und NF-κB durch PRL Die in dieser Arbeit dargestellten Untersuchungen zeigen, dass PRL auch in PBMC den IRF-1 Promotor aktiviert. Dies entspricht den von Yu-Lee et al. publizierten Ergebnissen [79]. Dass die Aktivierung vom Ausmaß her dem außerordentlich starken Stimulus LPS entspricht, ist in der Tat sehr überraschend und wird im Hinblick auf die zu erwartende Aktivierung von Zytokinen diskutiert (siehe Kapitel 4.1.4). LPS bewirkt die Aktivierung und Translokation von NF-κB in den Kern. Da NF-κB den IRF-1-Promotor durch Bindung an seine NF-κB-Bindungsstelle aktiviert, liegt die Vermutung nahe, dass die Aktivierung des IRF-1 Gens unter LPS-Stimulation Folge der erhöhten intranukleären NF-κB-Spiegel ist. Unsere Ergebnisse zeigen, dass LPS sowohl NF-κB als auch IRF-1 um das etwa 1,5fache verglichen mit der unstimulierten Kontrolle aktiviert. Diese Daten stützen die Hypothese, dass eine durch LPS induzierte Aktivierung von IRF-1 Folge erhöhter intranukleärer Konzentrationen von aktiviertem NF-κB ist. Weiter konnten wir zeigen, dass eine PRL-Inkubation ebenfalls eine direkte Aktivierung und Translokation von NF-κB in den Zellkern zur Folge hat - und zwar in demselben bzw. eher noch höherem Ausmaß als LPS. Geht man unseren Ergebnissen zufolge davon aus, dass PRL NF-κB in vergleichbarem Ausmaß wie LPS aktiviert, und verknüpft man dies mit der Tatsache, dass PRL eine starke Aktivierung von STAT1 bewirkt [80], so sollte man sogar erwarten, dass PRL den IRF-1Promotor weitaus stärker aktiviert als LPS. Unseren Ergebnissen zufolge aktivieren PRL und LPS den IRF-1-Promotor jedoch in vergleichbarem Außmaß. Eine Erklärungsmöglichkeit für diesen scheinbar widersprüchlichen Sachverhalt ist die gleichzeitige Aktivierung von STAT1 und STAT5b durch PRL. Dabei wird die Wirkung von NF-κB auf den IRF-1-Promotor durch STAT5b stark limiert, gleichzeitig wird das IRF-1 Gen jedoch an anderer Stelle des Promotors unabhängig davon durch STAT1 aktiviert. In Kostimulation mit LPS und PRL wurde gezeigt, dass sowohl NF-κB als auch IRF-1 in ihrer Aktivität um das 3- bis 3,5-fache erhöht werden im Vergleich zur unstimulierten Kontrolle. Bezüglich der Aktivierung von NF-κB lässt sich dies als additive Wirkung der Ein75 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION zeleffekte von LPS und PRL auffassen. Für IRF-1 müsste man jedoch eigentlich - dem in Kapitel 4.1.1 dargestellten und in der Literatur beschriebenen Mechanismus [80, 79] folgend - eine Limitierung der Wirkung von NF-κB und STAT1 auf IRF-1 durch STAT5b erwarten. Die in unseren Ergebnissen nachgewiesene 3,5- bis 4-fache Erhöhung der intranukleären IRF-1 Spiegel bei Kostimulation mit PRL und LPS ist jedoch überadditiv synergistisch. In Kontradiktion zu dem von Yu-Lee et al. publizierten, in Kapitel 4.1.1 dargestellten Modell zeigen wir zumindest eine uneingeschränkte additive Wirkung, eher sogar einen superadditiven synergistischen Effekt, keinesfalls jedoch eine Effektlimitierung. In einer PCR im Rahmen ähnlicher Stimulationsexperimente mit PHA, führt Kostimulation von PHA+PRL zu einer Transkription von IRF-1 in ähnlichem Ausmaß wie alleinige Stimulation mit PHA. Die höchsten Konzentrationen an IRF-1 mRNA finden sich in diesem Versuchsaufbau bei alleiniger Inkubation mit PRL. Dementsprechend scheint die beschriebene superadditive synergistische Beeinflussung einer Reaktion auf inflammatorische Stimuli für eine Kostimulation mit LPS spezifisch zu sein. Diese Ergebnisse könnten durch ein Zusammenspiel folgender Mechanismen erklärt werden: 1. LPS und PRL induzieren durch additive Wirkung eine starke NF-κB-Aktivierung, sodass - relativ gesehen zum nur durch PRL aktivierten STAT5b - ein großer Überschuss an NF-κB vorliegt. Die Mengen an STAT5b im Kern reichen daher nicht aus, um die Wirkung von NF-κB, das zusätzlich durch LPS aktiviert wird, zu limitieren. 2. LPS und PRL bewirken eine geringere Aktivierung von STAT5b als PRL allein, so dass zusätzlich zum relativen Überschuss an NF-κB auch absolut gesehen weniger STAT5b im Kern vorhanden ist. Der erste Mechanismus würde einen additiven Effekt bei Kostimulation erklären, im Zusammenwirken mit dem zweiten auch eine gegenseitige Verstärkung der Einzelwirkungen. In Dotblot-Versuchen, die wir mit einer unstimulierten Kontrolle, PRL- und LPS-stimulierten PBMC sowie mit PRL und LPS- kostimulierten Zellen durchführten, fanden wir eine stark vermehrte Konzentration von STAT5b im Kern nach alleiniger Inkubation mit PRL verglichen mit der Kostimulation mit PRL und LPS. Dies stützt die Annahme einer verminderten Aktivierung von STAT5b bei Kostimulation. Dementsprechend lässt sich die Abbildung 34 wie in Abbildung 35 dargestellt erweitern. Ein möglicher Mechanismus zur LPS-spezifischen verminderten Aktivierung von Stat 5 bei Kostimulation ist die Inhibition über Src homology 2-containing protein tyrosine phosphatase1 (SHP-1) . Das Prinzip der Wirkung von SHP-1 auf Stat 5 wurde in der Einleitung dargestellt und ist in Abbildung 36 zusammengefasst. 76 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION Abbildung 35: Möglicher Mechanismus der Regulation der Transkription des IRF-1 Gens durch PRL und LPS in Kostimulation 1. PRL aktiviert das IRF-1 Gen durch Aktivierung von STAT1 und NF-κB. Die Akitivierung von STAT5b durch PRL limitiert diesen Effekt. 2. LPS wirkt kostimulatorisch zu PRL durch zusätzliche Aktivierung von NF-κB und verminderte Aktivierung von STAT5b Abbildung 36: Inhibitorische Rolle von SHP-1 in der Jak2/STAT5-Kaskade; 1. Bindung an Rezeptor / Jak2-Komplex 2. Phosphorylierung der SHP-1-Tyrosinreste und Aktivierung von Grb2 und SOCS-1 3. SOCS-1 bindet an Jak2 4. Hemmung der Aktivierung von STAT5 nach Minoo et al., A novel SHP-1/Grb2-dependent mechanism of negative regulation of cytokinereceptor signaling: contribution of SHP-1 C-terminal tyrosines in cytokine signalling; Blood, 103(4), 2004, [55] 77 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION SHP-1 ist ein Protein, von dem andererseits berichtet wurde, dass es in Zusammenhang mit Signaltransduktion von LPS über CD14/Toll-like-Rezeptor (TLR) 4 steht und hier eine Regulatorfunktion für das Ausmaß der inflammatorischen Reaktion einer Zelle auf LPS ausübt [61]. Es scheint damit wahrscheinlich, dass LPS über Aktivierung von SHP-1 auch die Inaktivierung von STAT5 bewirkt. Dieses entspräche einem LPS-spezifischen Effekt, der im Einklang mit unseren PCR-Ergebnissen bei Stimulationsreihen mit PHA und PRL steht. 4.1.4 Beeinflussung der Zytokinsynthese durch PRL Um die Bedeutung erhöhter Prolaktinspiegel im Blut für das Geschehen proinflammatorischer Prozesse zu erfassen, haben wir in Vollblutansätzen die Konzentrationen von TNF-α, IL-12, IFN-γ und IL-10 nach Stimulation mit PRL und LPS + PRL gemessen und mit der unstimulierten Kontrolle verglichen. Die Stimulation mit LPS + PRL stellt ein in vitro-Abbild des Einflusses von PRL auf einen bereits initiierten proinflammatorischen Prozess dar. Die alleinige Stimulation mit PRL zeigt den Effekt ohne weitere proinflammatorische Agentien. Unsere Versuchsreihen zeigten zunächst, dass die alleinige Inkubation mit PRL die Konzentration keines der gemessenen Zytokine in statistisch signifikantem Ausmaß beeinflusst. Dies scheint im Widerspruch zur vermehrten Aktivierung der Transkriptionsfaktoren NF-κB und IRF-1 zu stehen, denn schließlich sollten erhöhte IFN-γ-Spiegel als unmittelbare Folge vermehrter Aktivierung von IRF-1 entstehen. Es müssen hier noch weitere, bisher unbekannte Mechanismen eine Rolle spielen, durch die nach der Aktivierung der entsprechenden Transkriptionsfaktoren die Synthese des Zytokins inhibiert wird. Da wir auch in der PCR nach Stimulation mit PRL zwar starke Banden in der IRF-1-PCR, jedoch keine in der IFN-γ-PCR detektieren konnten, liegt die Vermutung nahe, dass diese Inhibierung durch Deaktivierung der Transkriptionsfaktoren stattfinden muss, da es nicht zur Synthese von mRNA zu kommen scheint, oder aber auch posttranskriptionelle Mechanismen wären denkbar. Für Zytokine wie IL-12, die als Folge der Aktivierung von NF-κB entstehen, könnte auch die Aktivierung von STAT5b durch PRL eine entscheidende Rolle spielen, da hier ein analoger Einfluss zur Limitierung der NF-κB-bedingten IRF-1-Aktivierung möglich wäre. In Kostimulation von PRL und LPS ergibt sich eine vermehrte Sekretion von TNF-α, IFN-γ und IL-12 verglichen zur alleinigen Stimulation mit LPS. Dabei besteht eine Abhängigkeit von der Konzentration an PRL. Diese Effekte entsprechen denen auf der Ebene der Transkriptionsfaktoren beschriebenen. Zusammenfassend sind die einzelnen Ergebnisse nochmals in Tabelle 7 dargstellt. 78 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem Stimulans PRL LPS PRL + LPS Wirkung auf die Freisetzung von TNF-α, IL-12, IFN-γ durch PBMC keine vermehrte Freisetzung im Vergelich zur unstimulierten Kontrolle = deutlich vermehrte Freisetzung im Vergleich zur unstimulierten Kontrolle ↑ vermehrte Freisetzung im Vergleich zur LPS-Stimulation ↑↑ 4 DISKUSSION Wirkung auf Transkriptionsfaktoren Aktivierung von NF-κB und IRF-1 ↑ Aktivierung von NF-κB und konsekutiv IRF-1 ↑ Addition der Einzeleffekte in Bezug auf die Aktivierung von NF-κB überadditiver synergistischer Effekt auf die Aktivierung von IRF-1 ↑↑ Tabelle 7: Wirkung von PRL auf proinflammatorische Prozesse 4.1.5 Zustände von Hyperprolaktinämie und mögliche klinische Bedeutungen Um den in vitro-Ergebnissen, nach denen PRL proinflammatorische Prozesse verstärkt, in vivo-Situationen zuzuordnen, werden im Folgenden markante Zustände erhöhter PRL-Spiegel diskutiert. 4.1.5.1 Tumoren der Adenohypophyse Einen weit bekannten Zustand mit erhöhten Prolaktinwerten im Serum stellt das Prolaktinom dar. Es handelt sich hierbei um den häufigsten endokrin aktiven Hypophysentumor. Man unterscheidet Mikroprolaktinome mit ProlaktinSerumkonzentrationen < 200 ng/ml und einer Tumorgröße von <1 cm Durchmesser von Makroprolaktinomen. Klinisch äußert sich dieser Tumor neben Zeichen der Raumforderung bei Frauen durch sekundäre Amenorrhö, Anovulation mit Sterilität, eventuell Osteoporose, Libidoverlust und in einigen Fällen Galaktorrhö, bei Männern durch Libido- und Potenzverlust mit eventueller Gynäkomastie. Inwieweit es hier zu einer relevanten Beeinträchtigung der Immunfunktion kommt, wird durchaus kontrovers beurteilt. Kardioglu et al. fanden bei diesen Patienten eine absolute und relative Verminderung der NK-Zellen und der CD3/CD25-Lymphozyten, wobei keine Korrelation zur Höhe der Prolaktinspiegel im Serum hergestellt werden konnte. Weiter kann die Zellzahl auch nicht durch 79 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION Behandlung mit dem Dopamin-Agonisten Bromocriptin angehoben werden, während es unter dieser Behandlung zu einem Anstieg der CD8-Zellen mit Erniedrigung des CD4/CD8Verhältnisses kommt. Diesen Ergebnissen entspricht eine Verschlechterung der zytotoxischen Funktion bei Hyperprolaktinämie [38]. In PBMC von Patienten mit Hypophysenadenomen findet man nur bei solchen mit Makroadenomen und hier nur auf CD8-Zellen eine erhöhte Dichte von Prolaktinrezeptoren, die wiederum nicht mit dem Prolaktinspiegeln im Serum korreliert. Auch die Reaktionsintensität von PBMC dieser Patienten auf T-Zell-Mitogene unterscheidet sich nicht von der mit Bromocriptin behandelter Patienten [44]. Allerdings scheint es durch die erhöhten Spiegel an hypophysärem PRL beim Adenom zu einer Beeinflussung der Chemotaxis Neutrophiler Granulozyten [25] und einer erhöhten Inzidenz von Schilddrüsen-Antikörpern zu kommen [34]. Zusammenfassend kann also gesagt werden, dass erhöhte PRL-Spiegel im Zusammenhang mit Hypophysenadenomen nicht mit wesentlichen Beeinträchtigungen der Immunität einhergehen. Ein möglicher Mechanismus ist eine erniedrigte Expression des Rezeptors auf Immunzellen. 4.1.5.2 Systemischer Lupus erythematodes (SLE) Der systemische Lupus erythematodes ist eine Systemerkrankung der Haut und des Gefäßbindegewebes zahlreicher Organe mit Vaskulitis / Perivaskulitis der kleinen Arterien und Arteriolen in Kombination mit Ablagerung von Immunkomplexen aus DNS, Anti-DNS, Komplement und Fibrin. Immer wieder wird von erhöhten PRL-Serumspiegeln im Zusammenhang mit dieser Erkrankung berichtet [56] [37]. Tatsächlich scheinen PBMC von Patienten mit SLE sehr empfindlich auf v.a. nur leicht erhöhte Konzentrationen von PRL zu reagieren. Bei Stimulation mit PRL kommt es hier zu einer starken IgG-Produktion, die mit der Krankheitsaktivität korreliert [36]. Es konnte beobachtet werden, dass T-Lymphozyten von Patienten mit SLE im Verhältnis zu solchen gesunder Spender zudem vermehrt PRL sezernieren. Dies wurde in Zusammenhang mit bestimmtem Genpolymorphismen im PRL-Gen bei SLE-Patienten gebracht [71]. 80 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION 4.1.5.3 Hyperprolaktinämie und Narkose Während der Vollnarkose kommt es - abhängig vom Narkotikum - zu einer unterschiedlich starken Ausschüttung von PRL [49][14, 2]. 20 Minuten nach Narkoseeinleitung konnten Brand et al. im peripheren Blut von Patienten eine Zunahme der Konzentration an proinflammatorischen Zytokinen messen sowie eine Veränderung der Zusammensetzung peripherer Blutzellen in Richtung der B- und T-Zellen feststellen [11]. V.a. Opioide bewirken kurzfristig eine extreme Zunahme der PRL-Plasmakonzentration, während gleichzeitig das Plasmakortisol abnimmt [14]. Bei opioidbasierter Narkoseführung konnten Crozier et al. sowohl unter balanzierter Anästhesie als auch unter totaler intravenöser Anästhesie (TIVA) eine bis zu 10-fache Zunahme der Prolaktinspiegel intraoperativ messen [21]. Unsere Versuche zeigen, dass verstärkte inflammatorische Reaktionen auf Stimuli wie LPS in vitro auch bei alleiniger Stimulation mit PRL auftreten und deuten damit daraufhin, dass die von Brand et al. gemessenen Effekte nicht nur Folge erniedrigter Kortisolspiegel, sondern ebenfalls Resultat erhöhter PRL-Spiegel sind. Während einer Operation am Darm kommt es regelmäßig zur Überwindung der Mukosabarriere durch LPS [8]. Hier könnte eine Narkose, die auf Opioiden basiert, über eine Erhöhung der Plasmakonzentration an PRL eine höhere intra- und postoperative Freisetzung von proinflammatorischen Zytokinen bewirken, was wiederum an der Pathophysiologie von septischem Schock und Multiorganversagen beteiligt sein kann. Gerade im Rahmen der Fast track surgery wird in den letzten Jahren bei Narkosen für abdominalchirurgische Eingriffe - bei Fehlen von Kontraindikationen - dazu übergegangen, Opioide durch Supplementierung der Analgesie durch regionale Verfahren einzusparen. Das bessere Outcome der entsprechenden Patienten auf alleinige Effekte von PRL zurückzuführen, wäre sicherlich vermessen und zudem falsch, doch lässt sich ein gewisser Miteffekt in Form einer geringeren “Anfälligkeit” solcher Patienten gegenüber proinflammatorischen Reizen durch verminderte PRL-Spiegel postulieren. Zudem bedeutet das Eintreffen im Operationstrakt für einen Patienten oft Stress, da nahezu jeder Mensch dies als eine extrem ungewohnte und damit auch Angst besetzte Situation empfindet. Wie bereits in der Einleitung erwähnt, stellt Stress einen Trigger für die hypophysäre Prolaktinfreisetzung dar. Dementsprechend bedeutet präoperativer Stress - zusätzlich zu 81 4.1 Einfluss von hypophysärem Prolaktin auf das Immunsystem 4 DISKUSSION negativen Auswirkungen auf das kardiovaskuläre System - eine vermehrte Disposition zu überschießenden Reaktionen auf inflammatorische Reize wie LPS. Hier helfen eine gute Prämedikation sowie eine ruhige Atmosphäre während der Narkoseeinleitung, den Patienten möglichst weit von Stressoren abzuschirmen. Auch sei hier wiederum das Konzept der Fast track surgery erwähnt, das ja letztendlich darauf basiert, den Stress des Patienten prä-, peri- und postoperativ zu minimieren. Auch hier geht weniger Stress mit niedrigeren PRLSpiegeln und einer verminderten Antwort auf proinflammatorische Reize einher. 4.1.5.4 Weitere Zustände von Hyperprolaktinämie Unter Behandlung mit Dopaminantagonisierenden Medikamenten wie Neuroleptika kommt es gehäuft zu Autoimmunphänomenen [73]. Ein möglicher Mechanismus wäre hier, dass Lymphozyten in dem durch PRL veränderten Zytokinmilieu vermehrt auf Signale reagieren, die normalerweise keine Immunreaktion bewirken. Zudem können Antigen-präsentierende-Zellen (APCs) unter Einfluss von PRL die Immunantwort abnormal stimulieren. Auch in Schwangerschaft und Postpartalperiode kommt es - neben vielen anderen hormonellen Veränderungen - zu einer ausgeprägten Hyperprolaktinämie. In dieser Zeit treten ebenfalls vermehrt Autoimmunkrankheiten auf bzw. der Verlauf vorbestehender Krankheiten wird negativ beeinflusst [62, 39]. Dieser Effekt ist vermutlich auch als eine Summation der Wirkung einzelner Hormone wie Cortisol und PRL auf Immunsystem zu begreifen. 4.1.6 Zusammenfassende Darstellung des Einflusses von PRL auf das Immunsystem Erhöhte PRL-Spiegel gehen in vivo häufig mit erniedrigten Kortisolspiegeln einher. Hier ist somit der Effekt von PRL isoliert schlecht messbar. Bezeichnend ist, dass bei Patienten mit Autoimmunerkrankungen wie Lupus erythematodes, Hashimoto-Thyreoiditis oder rheumatoider Arthritis signifikant erhöhte Prolaktinkonzentrationen im Serum gemessen werden können. Zudem ging aus Untersuchungen unserer Arbeitsgruppe hervor, dass nach Narkoseeinleitung sowohl erhöhte PRL- als auch erniedrigte Kortisolspiegel gemessen werden können und in Überständen entsprechender Vollblutproben nach in vivo Stimulation mit LPS erhöhte Konzentrationen an inflammatorischen Zytokinen auftreten. Unsere Experimente stellen diese Situation in vitro nach und lassen Aussagen über den isolierten Effekt von PRL zu. • Die Stimulation von Vollblutzellen mit bis zu 300 ng/ml PRL, was typischen PRL82 4.2 Prolaktinsynthese durch PBMC 4 DISKUSSION Plasmaspiegeln entspricht, die nach Narkoseeinleitung erreicht werden können, hat selbst keinen Effekt auf die Freisetzung von Zytokinen. Unter denselben Stimulationsbedingungen findet man in PBMC allerdings vermehrt NF-κB und IRF-1 im Zellkern. • Kostimulation mit LPS (100 ng/ml) bewirkt eine inflammatorische Reaktion, deren Ausmaß von der Konzentration an PRL abhängt. Die Verstärkung der Antwort auf LPS durch PRL findet sich auch auf Ebene der Transkriptionsfaktoren NF-κB und IRF-1. 4.2 Prolaktinsynthese durch PBMC Schon 1989 berichtete Russell erstmalig von einem Prolaktin-ähnlichem Produkt aus Lymphozyten [67]. Sabharwal et al. konnten 1992 zeigen, dass PBMC ein 60kD schweres PRL-ähnliches Protein synthetisieren und sezernieren. Diesem schreiben sie eine autokrine Funktion zu [68]. Reem et al. zeigten 10 Jahre nach Russell mittels EMSA, dass T-Zell Aktivatoren und eine erhöhte Konzentration an intrazellulärem cAMP den Promotor für Prolaktin in Lymphozyten aktivieren [64]. Die Arbeitsgruppe führte diesen Nachweis mit einer T-lymphozytären Jurkat-Zelllinie durch. Sie nutzten doppelsträngige Oligonukleotide aus dem Bereich des Exon 1a, der das in Abbildung 1 markierte CRE enthielt. Eine erhöhte intrazelluläre cAMPKonzentration zeigte dabei einen synergistischen Effekt mit PHA und PMA, während PHA und PMA allein kaum wirksam waren. Eine Transkriptionsinduktion konnte nach zwei Stunden festgestellt werden und erreichte nach ca. sechs Stunden ihr Maximum, um dann langsam abzunehmen. Gerlo et al. [30] wiesen 2003 an leukämischen Zelllinien und peripheren mononukleären Zellen (PBMC) eine Transkriptionsinduktion durch intrazellulär erhöhtes cAMP via RTPCR und Southernbloting als auch eine Translation über ELISA nach. Die Arbeitsgruppe fand erhöhte hPRL-mRNA-Konzentrationen in PBMC nach Stimulation mit dem zellmembrangängigen cAMP Chlorophenylthio-cAMP (cptcAMP). Stimulation mit PMA ergab eine Abnahme der basalen mRNA-Konzentration und in den meisten Fällen bewirkte PMA auch in Kostimulation eine Minderung des Effektes von cptcAMP. Zudem konnte die Arbeitsgruppe in einigen Fällen eine Zunahme der hPRL-mRNA-Konzentration nach Stimulation mit Prostaglandin E2, einem Stoff, der über eine Erhöhung des cAMP-Spiegels wirkt, feststellen. 2006 wiesen Gerlo et al. eine Aktivierung des extrahypophysären PRL-Promotors durch TNF-α über Proteinkinase C nach [31]. 83 4.2 Prolaktinsynthese durch PBMC 4 DISKUSSION Die Publikation von Gerlo et al. aus dem Jahr 2003 ist die einzige uns bekannte, in der ein positiver Nachweis des Proteins PRL via ELISA geführt wurde. Alle anderen Arbeitsgruppen führten den Nachweis ausschließlich über die Messung von Transkriptionsfaktoren (EMSA) oder RNA (RT-PCR). Für einen sicheren Nachweis der Synthese von PRL in PBMC untersuchten wir möglichst viele Schritte der Synthese von PRL. Die Synthese eines Proteins läuft in einer Zelle in der folgenden Reihenfolge ab (siehe Abbildung 37): 1. Aktivierung von Transkriptionsfaktoren 2. Transkription des gesamten Gens inklusive nicht-kodierender Abschnitte (Introns) 3. Capping, Polyadenylierung und Splicen des primären RNA-Transkripts 4. Die reife mRNA verlässt den Kern und wird an den Ribosomen translatiert Abbildung 37: Schematische Darstellung des Ablaufs der Proteinsynthese in einer Eukaryonten-Zelle Ein menschliches Gen besteht aus kodierenden (Exons) und nicht kodierenden Abschnitten (Introns). Nach Reifung der RNA verlässt diese den Kern und wird an den Ribosomen translatiert Abbildung aus dem Internet entnommen 84 4.2 Prolaktinsynthese durch PBMC 4 DISKUSSION Dementsprechend untersuchten wir PBMC auf die Aktivierung von Transkriptionsfaktoren, das Vorhandensein von mRNA und letztendlich bestimmten wir die PRL-Konzentration im Zellüberstand. 4.2.1 Aktivität der Transkriptionsfaktoren in Kernextrakten aus PBMC bezüglich der Bindung an den Prolaktin-Promotor Um den häufig geführten Nachweis einer Prolaktinsynthese über Transkriptionsfaktoren zu überprüfen, benutzten wir ebenfalls Oligonukleotide, die die in Abbildung 1 gelb unterlegte Sequenz des cAMP responsive element enthielten. In einer Versuchsreihe benutzten wir PMA, PHA und cptcAMP als Stimulantien, da von diesen berichtet ist, dass sie die Prolaktinsynthese in Immunzellen induzieren [30, 64]. Zusätzlich stimulierten wir mit Noradrenalin, weil von Immunzellen bekannt ist, dass sie β2 Rezeptoren besitzen [77], an welchen Katecholamine durch Erhöhung intrazellulärer cAMPSpiegel wirken. Dieser Sachverhalt ließ für uns eine Beeinflussung der PRL-Synthese in PBMC durch Katecholamine als wahrscheinlich erscheinen. In einer weiteren Versuchsreihe stimulierten wir mit humanem rekombinanten Prolaktin und mit PRL in Kostimulation mit PHA, um einen möglichen positiv rückkoppelnden Effekt von PRL auf seine eigene Synthese zu erfassen. In unseren Stimulationsexperimenten mit PHA, PMA und cptcAMP konnten wir die Ergebnisse von Reem et al. [64] für PBMC reproduzieren, nur maßen wir bereits nach 20 min eine vermehrte Bindungsaktivität von CRE im Vergleich zur unstimulierten Kontrolle nach Stimulation mit cptcAMP oder cptcAMP + PHA und eine noch stärkere Bindungsaktivität bei Stimulation mit cptcAMP + PMA + PHA, jedoch nicht bei Stimulation mit cptcAMP + PMA. Damit stehen unsere Untersuchungen bezüglich der Aktivität der Transkriptionsfaktoren ebenfalls im Einklang mit denen von Gerlo et al. [30] Nach Stimulation mit PRL, PHA und PRL+PHA konnten wir keine vermehrte Aktivität an Transkriptionsfaktoren im Vergleich zur unstimulierten Kontrolle messen. PRL scheint daher in Bezug auf die eigene Synthese nicht im Sinne eines Zytokins zu wirken, welche stimulierend auf die eigene Freisetzung im Sinne einer Effektpotenzierung agieren. Dies ist im Kontext der Wirkung von PRL als Hormon sinnvoll, um vor überschießenden Reaktionen zu schützen. Auch nach Stimulation mit Noradrenalin fand sich in den Kernextrakten keine erhöhte Bindungsaktivität der Transkriptionsfaktoren. Die Aktivierung von cAMP durch Noradrenalin scheint demnach nicht ausreichend für eine Aktivierung des PRL-Promotors zu sein. 85 4.2 4.2.2 Prolaktinsynthese durch PBMC 4 DISKUSSION Untersuchung von PBMC auf PRL-mRNA mittels GEArray und RT-PCR PBMC aus denselben Buffycoats, die wir auch für die Untersuchung der Transkriptionsfaktoren verwendet hatten, untersuchten wir auf das Vorhandensein von PRL-mRNA und PRLR-mRNA via GEArray. Wir konnten hier im Rahmen der Sensitivität des GEArrays keine PRL-mRNA detektieren, während wir für die Positivkontrollen GAPDH und β-Aktin deutliche mRNA-Signale sehen konnten. Als weitere Kontrolle für die Validität unserer Untersuchungen setzten wir eine Mamma-Karzinom-Zelllinie ein, die laut Angaben des Herstellers den Prolaktinrezeptor jedoch kein PRL exprimiert. Hier erhielten wir mittels GEArray wie erwartet ein deutliches Signal für PRLR-mRNA und keinen Nachweis von PRL-mRNA. Um den offensichtlichen Widerspruch zu klären, in dem unsere Ergebnisse zu denen von Reem et al. und Gerlo et al. [64, 30] stehen, untersuchten wir mRNA einiger der mittels GEArray getesteten Proben mittels RT-PCR für PRL. Wir setzten zum einen kommerzielle Primer der Firma SuperArray zum anderen den selbstkreierten Primer ein. Da wir mittels der PRL-PCR in einigen der Proben Signale erhielten, die mit der Literatur im Einklang stehen, lassen sich die negativen Ergebnisse mittels GEArray auf die unterschiedliche Sensitivität der einzelnen Methoden bei vermutlich sehr geringen PRL-Konzentrationen im Überstand zurückführen. Dabei traten auch in der unstimulierten Kontrolle nach PRL-PCR teilweise Signale auf, die allerdings recht schwach waren. Dementsprechend scheint auch im Ruhezustand von PBMC in geringen Mengen PRL mRNA transkribiert zu werden. Eine eventuelle Basalsynthese könnte einer Auffüllung intrazellulärer PRL-Speicher dienen. Dies entspräche der bereits von Sabharwal et al. für PRL-ähnliche Proteine postulierten Hypothese, dass PRL intrazellulär gespeichert wird und nach Stimulation im Sinne einer immunologischen Synapse an PRLRs bindet, um anschließend vermehrt synthetisiert zu werden [68]. Die Vorstellung einer Wirkung von PRL an einer immunologischen Synapse stünde auch im Einklang mit den im ELISA erhobenen Daten. Zudem konnten wir feststellen, dass intrazelluläre Erhöhung der Konzentration an cAMP tatsächlich im Vergleich zur unstimulierten Kontrolle zu vermehrter Transkription von PRLmRNA führt. Zustände erhöhter intrazellulärer cAMP-Konzentrationen stellten wir durch Stimulation mit zellmembrangängigem cptcAMP nach. Hier fanden wir die stärksten Signale nach Stimulation über 7h. Ein geringfügig stärkeres Signal trat bei Kostimulation mit cptcAMP und PHA auf. In Kostimulation mit PMA fanden wir ein Signal, das in seiner 86 4.2 Prolaktinsynthese durch PBMC 4 DISKUSSION Intensität etwa der Intensität des Signals der unstimulierten Kontrolle entspricht. Demzufolge scheint PMA im Gegensatz zu PHA die cAMP-induzierte Transkription des PRL-Gens inhibieren zu können, während PHA eine leichten kostimulatorischen Effekt ausübt. Diese Ergebnisse entsprechen unseren Resultaten der Untersuchungen an Transkriptionsfaktoren und gehen konform mit der Literatur [30]. Bei Stimulation über 14 h detektierten wir allerdings in cptcAMP + PMA kostimulierten Zellkulturen ein Signal in der PRL-PCR. Dieses war deutlich intensiver als die Signale für die alleinige Stimulation mit cptcAMP und cptcAMP+PHA. Es wäre also auch möglich, dass in Kostimulation mit PMA die cAMP-induzierte Transkription des PRL-Gens zu späteren Zeitpunkten verschoben ist. Erstaunlich ist, dass wir in keiner untersuchten Probe ein Signal für PRL bei Kostimulation von cAMP mit PHA und PMA fanden. Dies war unseren Untersuchungen an Transkriptionsfaktoren zufolge nicht zu erwarten und lässt eine noch unbekannte Beeinflussung der Transkription des PRL-Gens vermuten. Zusammenfassend lässt sich formulieren, dass es bei Erhöhung intrazellulärer cAMP-Spiegel zu einer verstärkten Synthese von PRL mRNA kommt. Dieser Effekt von cAMP wird in Kostimulation mit PHA leicht verstärkt. Allerdings sind die Mengen an mRNA, die entstehen, unterhalb der Nachweisgrenze eines GEArrays, was eine Synthese von PRL durch PBMC in für den Makroorganismen relevanten Konzentrationen unwahrscheinlich macht. 4.2.3 Untersuchung der Überstände von PBMC-Kulturen auf PRL mittels ELISA Aufgrund der schwierigen Interpretierbarkeit unserer Ergebnisse einerseits und der wenigen Publikationen, die von messbaren Konzentrationen an PRL im Überstand von stimulierten PBMC berichten, andererseits, bestimmten wir die PRL-Konzentration unserer Zellkulturüberstände via ELISA. Hier benutzten wir - wie Gerlo et al. [30] - den PRL Elecsys der Firma Roche. Wir fanden in keinem unserer Zellüberstände messbare PRL-Konzentrationen. Dies steht im Einklang mit der Tatsache, dass kaum Publikationen zu diesem Thema vorhanden sind, die von einem PRL-Nachweis in Überständen stimulierter Immunzellen gesunder Spender via ELISA berichten. Allerdings liegt die untere Nachweisgrenze dieses Testsystems bei PRL-Konzentrationen von 0,470 ng/ml. Zum Vergleich bewirkt eine Stimulation von Vollblutzellen mit LPS eine Endkonzentration an IFN-γ von etwa 0,4 ng/ml im Überstand. Dies zeigt eindrucksvoll, dass ein ELISA mit einer derart geringen Sensitivität für die 87 4.2 Prolaktinsynthese durch PBMC 4 DISKUSSION Messung an Zytokinen nur bedingt geeignet ist. Gerade wenn man davon ausgeht, dass in PBMC synthetisiertes PRL ausschließlich an einer immunologischen Synapse wirkt, ist das Ergebnis nicht verwunderlich. Allerdings zeigt es eindeutig, dass eine mögliche endokrine Wirkung von durch PBMC synthetisiertes PRL sehr unwahrscheinlich ist. 4.2.4 Zusammenfassende Darstellung Wir konnten in PBMC gesunder Blutspender nachweisen, dass Stimulation mit cptcAMP zu einer Aktivierung von Transkriptionsfaktoren führt, die an das CRE des PRL-Promotors binden. Dieser Effekt wird potenziert durch Kostimulation mit PHA und PHA+PMA, nicht jedoch durch Kostimulation nur mit PMA. Die in der Literatur beschriebenen Hinweise auf die Synthese von PRL-mRNA in PBMC gesunder Spender beruhen größtenteils auf Untersuchungen via PCR, die wir größtenteils bestätigen konnten. Unsere Untersuchungen mittels GEArray zeigten jedoch in keiner Stimulation Signale für PRL-mRNA. Wir erklären diesen Widerspruch dadurch, dass die Mengen an synthetisierten PRL-mRNA so gering sind, dass sie mit einer Nachweismethode einer Sensitivität der des GEArrays nicht erfasst werden. Unsere Untersuchungen via ELISA zeigen, dass PRL von PBMC nicht in einer für das endokrine System relevanten Konzentration sezerniert wird. Sie gehen konform mit den Ergebnissen des GEArray. Die mittels EMSA gewonnenen Ergebnisse zeigen jedoch sehr starke Banden. Wir müssen daher annehmen, dass die Mengen an mRNA, die synthetisiert werden, entweder nicht transkribiert werden oder in deutlich geringerer Menge als es dem Maß der Aktivierung der Transkriptionsfaktoren entspricht. Es scheint also Mechanismen zu geben, die die PRL-Synthese in PBMC gesunder Blutspender nach Aktivierung der Transkriptionsfaktoren inhibieren. Mögliche Mechanismen bedürfen der weiteren Untersuchung. Eine endokrine Wirkung von PRL, das in PBMC synthetisiert wird, schließen unsere Ergebnisse somit aus, eine autokrine Wirkung wäre jedoch möglich und auch wahrscheinlich. 4.2.5 Diskussion von autokrinen und parakrinen Wirkungen von möglicherweise in PBMC synthetisiertem PRL Geht man von der Hypothese aus, dass PRL zwar als Protein von PBMC gebildet wird, jedoch in sehr geringen Mengen, so sollte man an entsprechenden Zellkulturen einige Stunden nach Bildung der mRNA die Wirkungen des gebildeten PRL auf die eigene Zellkultur erwarten. Nimmt man unseren Ergebnissen zufolge an, dass cptcAMP+PHA und cptcAMP 88 4.3 Wirkung von hypophysärem PRL auf die Synthese von PRLR in PBMC 4 DISKUSSION stimulierte PBMC-Kulturen nach 7 h das Maximum der Synthese an PRL-mRNA erreichen, so sollte man nach 14 h Wirkungen des gebildeten PRL auf Ebene der mRNA detektieren können. Wie wir zeigen konnten, bewirkt die Zugabe von exogenem PRL eine Aktivierung des IRF-1-Promotors und eine Synthese von IRF-1-mRNA (siehe Kapitel 3.1). In unseren Versuchsreihen konnten wir weder nach Stimulation mit cptcAMP noch mit cptcAMP+PHA vermehrte Signale für IRF-1 in der IRF-1-PCR bis zu einer Stimulation von 14 h feststellen. Daher lässt sich annehmen, dass endogenes PRL, falls es von PBMC in geringsten Mengen synthetisiert wird, zumindest keine autokrinen Wirkungen entwickelt, die denen von exogenem hypophysärem PRL entsprechen. Auch eine mögliche autokrine Wirkung im Sinne einer vermehrten Synthese des PRLR scheint nicht zu bestehen, da die Signale für PRLR in der PRLR-PCR unabhängig von dem Auftreten von Banden für PRL in der PRL-PCR in den entsprechendne Versuchsreihen stets etwa denen der unstimulierten Kontrolle entsprachen. 4.3 Wirkung von hypophysärem PRL auf die Synthese von PRLR in PBMC Von einem Hormon erwartet man, dass bei erhöhten Plasmakonzentrationen die Rezeptordichte auf den Zielzellen abnimmt, von einem Zytokin könnte man erwarten, dass die Rezeptordichte zunimmt. Ersteres würde einen vermehrten Abbau von PRLR und eine verminderte Neusynthese, letzteres eine vermehrte Neusynthese bedeuten. Da das Maß an mRNA des PRLR in der Zelle dem Maß der Neusynthese entspricht, untersuchten wir unsere wie im Ergebnisteil beschrieben stimulierten Zellkulturen mit GEArray und RT-PCR auf PRL-mRNA. Die Schlussfolgerungen aus den Untersuchungen an Stimulationsreihen mit cptcAMP sind in Kapitel 4.2 dargestellt. Unsere Untersuchungen an PRL-, PHA- und PRL+PHA-stimulierten PBMCs zeigten das deutlichste Signal für die unstimulierte Kontrolle, ein abgeschwächtes Signal für PRL-Stimulation und die schwächsten Signale für PHA- und PHA+PRL-Stimulation. Eine diskrete Abschwächung der Signalintensität für PRL-Stimulation bedeutet letztendlich eine verminderte Synthese von PRLR-mRNA nach Stimulation mit exogenem PRL und entspräche dem Effekt eines klassischen Hormons. Eine extreme Abschwächung der Signalintensität und damit eine kaum vorhandene Neusynthese von PRLR finden wir nach Stimulation mit PHA. Dies könnte den Effekt erklären, dass unter Kostimulation mit PRL + PHA die PRL-induzierte IRF-1-Synthese gedrosselt wird, da durch eine stärkere Herunterregulation der Dichte an PRLRs auf der Zelloberfläche eine verminderte Aktivierung des IRF-1-Promotors über den PRLR bewirkt wird. Die PRL-induzierte Erniedrigung der Transkription des PRLR-Gens könnte den biologi89 4.4 Prolaktin - ein Zytokin ? 4 DISKUSSION schen Effekt haben, das Immunsystem vor zu hohen PRL-Konzentrationen aus der Hypophyse zu schützen - ein Effekt, der von klassischen Hormonen und ihren Zielzellen bekannt ist. Eine weitere Herunterregulation durch PHA könnte einen Schutz des Organismus vor überschießender Reaktion auf inflammatorische Reize durch PRL bedeuten. Eine interessante Frage, die in diesem Zusammenhang zu klären wäre, ist, inwiefern sich Stimulation mit LPS auf die PRLR-Dichte auf PBMC auswirkt. 4.4 Prolaktin - ein Zytokin ? Um diese Frage abschließend zu klären, sei nochmals auf die Definition von Abbas verwiesen [1]: • Die Zytokin-Sekretion ist ein kurzer, selbstlimitierender Prozess. Die Zytokine sind normalerweise nicht als präformierte Moleküle gespeichert und ihre Synthese ist gewöhnlich durch Transkription nach vorausgehender Zellaktivierung geregelt. Anschließend werden Zytokine schnell sezerniert und schlagartig freigesetzt. Ob Blutzellen in der Lage sind, PRL tatsächlich in sehr geringen Mengen zu synthetisieren, konnten wir nicht mit abschließender Sicherheit klären, da wir keinen Nachweis des Proteins führen konnten. • Die Wirkungen der Zytokine sind pleiotrop, d.h. ein Zytokin wirkt auf verschiedene Zelltypen, und redundant, d.h. einen bestimmten Effekt können mehrere Zytokine losgelöst voneinander bewirken. PRLRs sind - wie in der Einleitung dargestellt - auf den verschiedensten Zelltypen exprimiert und PRL entfalten diverse Wirkungen an diesen. Auch die Redundanz der Wirkung ist gegeben, da kein spezifischer PRL-Effekt bekannt ist. • Zytokine beeinflussen oft die Synthese und die Eigenschaften anderer Zytokine. PRL allein ist nicht in der Lage, die Synthese der von uns untersuchten Zytokine zu beeinflussen, doch verstärkt es eine proinflammatorische Antwort von Blutzellen auf LPS. • Die Wirkung der Zytokine kann lokal oder systemisch sein, v.a. autokrin und parakrin, aber bei massiver Freisetzung ist auch ein Übertritt in die Blutbahn mit Wirkung über längere Distanz möglich. 90 4.4 Prolaktin - ein Zytokin ? 4 DISKUSSION Eine autokrine oder parakrine Wirkung von eventuell in PBMC synthetisiertem PRL konnten wir nicht nachweisen. Die Frage nach der Bedeutung der PRL-Synthese durch Immunzellen bleibt ungeklärt. • Die Wirkung wird durch Bindung an spezifische Membranrezeptoren auf Zielzellen vermittelt, wobei die Dissoziationskonstanten zwischen 10−10 bis 10−12 M liegen. Durch diese starke Bindung reichen oft nur geringe Mengen eines Zytokins aus, um einen biologischen Effekt zu erzielen. Der PRLR als Zytokinrezeptor vom Typ I erfüllt diese Eigenschaften. • Äußere Signale regulieren die Expression von Zytokinrezeptoren und damit die Empfindlichkeit der Zielzelle für das Zytokin. Dies ist unabdingbar für die Spezifität des Immunsystems, da Zytokine nicht antigenspezifisch sind. Exogenes PRL und PHA sind in der Lage, die Transkription des PRLR-Gens zu beeinflussen. • Die zelluläre Antwort auf die meisten Zytokine beruht auf einer Änderung der Genexpression von Zielgenen. Die zelluläre Antwort auf PRL beruht v.a. auf vermehrter Expression con IRF-1. Es lässt sich also zusammenfassen, dass PRL zwar viele Eigenschaften eines Zytokins erfüllt, andere jedoch nicht. Derzeit PRL als Zytokin zu bezeichnen, wäre daher voreilig, aber von einem Hormon mit zytokinartigen Eigenschaften kann zu Recht gesprochen werden. 91 5 5 ZUSAMMENFASSUNG ZUSAMMENFASSUNG Prolaktin (PRL) ist ein Hormon, dem in vielerlei Hinsicht Eigenschaften eines proinflammatorischen Zytokins zukommen. Wie bei vielen Zytokinen wird die Wirkung von PRL über Zytokinrezeptoren vom Typ I vermittelt, die von Immunzellen exprimiert werden. Äußere Signale, die die Expression von dem PRL-Rezeptor (PRLR) und damit die Empfindlichkeit der Zielzelle für PRL beeinflussen, sind größtenteils noch nicht identifiziert. Wie Polymerase-Ketten-Reaktion (PCR)-Untersuchungen zeigten, scheint Stimulation mit PRL selbst die Neusynthese von PRLR negativ zu beeinflussen. Wie ein typisches Zytokin beeinflusst PRL die Synthese anderer proinflammatorischer Zytokine positiv, doch bedarf es hierzu der Kostimulation mit beispielsweise Lipopolysaccharid (LPS). Dies wurde an Hand der Zytokine Tumor-Nekrose-Faktor-α (TNF-α), Interleukin-12 (IL-12) und Interferon-γ (IFN-γ) mittels EnzymImmunoassay (ELISA) gezeigt. Allein hat PRL keine Wirkung auf die Sekretion von Zytokinen durch Zellen des peripheren Blutes, allerdings ist es Gel-Shift-Assays (electrophoretic mobility shift assays, EMSAs) zufolge in der Lage, Transkriptionsfaktoren wie Interferon-Regulatory-Factor-1 (IRF-1) und Nuclear Factor-κB (NF-κB) zu aktivieren. Dabei ist die Wirkung von NF-κB auf den IRF-1-Promotor und damit auf die Freisetzung von IFN-γ über die gleichzeitige Aktivierung von Signal transducer and activator of transcription 5b (STAT5b) durch PRL limitiert. Für die mangelnde Aktivierung der Synthese anderer Zytokine wie IL-12 und TNF-α trotz vermehrter Translokation von NF-κB in den Kern müssen weitere inhibitorische Prinzipien eine Rolle spielen, welche weiterer Untersuchungen bedürfen. Anders als klassische Zytokine scheint PRL jedoch nicht in relevantem Ausmaß von mononukleären Zellen des peripheren Blutes synthetisiert und sezerniert zu werden. EMSAs zufolge werden jedoch Transkriptionsfaktoren, die an den Prolaktin-Promotor binden, durch erhöhte intrazelluläre Spiegel an zyklischem Adenosinmonophpsphat (cAMP) in hohem Maße aktiviert. Zudem entstehen - wie PCR-Versuche zeigen - geringe Konzentrationen an PRL-mRNA. Die Synthese muss hier auf dem Weg von der Bindung des Transkriptionsfaktors an den Promotor zur Bildung der mRNA gedrosselt werden. Die Mechanismen hierfür bleiben unbekannt. Sollte PRL von PBMC sezerniert werden, dann müsste es an einer immunologischen Synapse und gänzlich anders als hypophysäres PRL auf PBMC wirken. Hypophysärem PRL kommt eine bedeutende Rolle für das Immunsystem zu. So lassen sich signifikant erhöhte PRL-Spiegel bei Patienten mit Autoimmunkrankheiten messen. Eine klinische Bedeutung der Beeinflussung proinflammatorischer Prozesse durch PRL wurde im Zusammenhang mit der Narkose untersucht. Hier erhöhen v.a. Opioide die hypophysäre PRL-Freisetzung, was eine vermehrte Disposition des Organismus für überschießende Reaktionen auf inflammatorische Stimuli wie LPS zur Folge hat. Es wäre somit denkbar, dass durch Einsparen von Opioiden über z.B. Supplementierung der Analgesie durch regionale Verfahren das Risiko einer systemischen inflammatorischen Reaktion im Rahmen einer verminderten Disposition für proinflammatorische Stimuli herabgesetzt wird. 92 APPENDIX A A PROTOKOLLE Protokolle Die Versuchsprotokolle basieren allesamt auf im Institut etablierten Methoden und wurden zum größten Teil in der Promotionsarbeit von Dr. Jürgen Luhm publiziert. A.1 Isolierung von PBMC Die mononukleären Zellen des peripheren Blutes (PBMC) lassen sich aus dem Buffycoat mittels Dichtegradientenzentrifugtion aus der Interphase - zwischen dem Serum und dem Trennmedium mit Erythrozyten und Granulozyten - gewinnen. Sie werden durch Absaugen isoliert, gewaschen und in gewünschter Konzentration im jeweiligen Kulturmedium suspendiert. A.1.1 Lösungen Wasch-Lösung (pH 7,4) Dichtegradienten-Lösung Kulturmedium A.1.2 PBS-Puffer Ficoll: 1,077 g/ml RPMI 1640 mit Zusatz - 10 % FCS (hitzeinaktiviertes fetales Kälberserum; 30min bei 56◦ C inaktiviert) - 1 % L-Glutamin - 1 % Penicillin/Streptomycin Protokoll • Buffycoat zu gleichen Teilen mit PBS verdünnen und das gleiche Volumen FicollTrennlösung damit überschichten • 20 min bei 600 g (20 ◦ C) zentrifugieren, dabei möglichst langsam beschleunigen und abbremsen • Zellfraktion aus der Interphase mittels Pipette absaugen • 2x in PBS waschen (300 g, 10 min, 20 ◦ C) • PBMC in Kulturmedium überführen und die Zellkonzentration am Vollblutzellzähler bestimmen • die benötigte Zellkonzentration (3 x 106 Zellen/ml für die Protein- und RNA-Ansätze bzw. 5 - 10 x 106 für die Bandshift-Ansätze) durch Verdünnung mit Medium herstellen 94 A.2 Protokoll zur Herstellung von Kernextrakten A.2 Protokoll zur Herstellung von Kernextrakten A.2.1 Erforderliche Reagenzien: A Extraktions-Puffer 1: 20 mM HEPES; pH=7,8 10 mM KCl 0,1 mM EDTA 0,5 mM Pefa Block (oder PMSF) 1 mM DTT Extraktionspuffer 2: 10 % (w/v) Nonidet NP40-Lösung PBS Extraktionspuffer 1 + 400 mM NaCl A.2.2 PROTOKOLLE Protokoll: • Stimulation von 4 x 106 Zellen und Inkubation für die jeweilige Zeit im Brutschrank • Kühlzentrifuge Centrikon T324 und für Eppendorf-Reagenzgefäße geeignete Zentrifuge Omnifuge 2.0RS vorkühlen • Zellen ernten: adhärente Monozyten mit dem Zellscraper abschaben, Zellen in ein steriles 15 ml Tube überführen, zweimal bei 1000 g und 4 ◦ C für 5 min waschen und dazu bei der zweiten Zentrifugation mit 15 ml PBS waschen • Überstand dekantieren, Röhrchen kopfüber auf Filterpapier stellen, Resuspension des Pellets in 1 ml eiskaltem PBS, Überführung der Suspension in ein 1,5 ml-EppendorfTube Die nachfolgend geschilderten Arbeitsvorgänge unbedingt auf Eis durchführen: • kurze (15 s, max. 30 s) Zentrifugation bei 13000 g, dabei extrem schnell arbeiten • Pellet vorsichtig in 500 ml eiskaltem Extraktionspuffer 1 resuspendieren (Vorsicht: Die Zellen dürfen kaum DNA verlieren) • 15 min auf Eis stellen (mind. 5 min, max. 2 h) • 25 - 30 ml 10 %ige NP-40-Lsg. dazugeben, das Eppendorf-Tube vorsichtig anschnippen, dabei wiederum extrem schnell arbeiten • kurze (15 - 30 s) Zentrifugation bei 13.000 g 95 A.3 Bestimmung des RNA-Gehaltes einer Probe A PROTOKOLLE • Überstand (enthält die RNA- sowie die Zytoplasmafraktion) schnell dekantieren (kann ggf. auch für RNA-Extraktion verwendet werden) • zum Pellet (= viskose, zumeist farblose Kernfraktion) 50 ml Extraktionspuffer 2 geben (nicht resuspendieren) • 15 min auf Eis stehen lassen (Kernlyse) • 5 min bei 4 ◦ C und 13.000 g zentrifugieren • ca. 70 ml Überstand (= Kernextrakt) vorsichtig abnehmen (Vorsicht: Pellet löst sich leicht ab!), in 3 Aliquots à 20 ml bei - 80 ◦ C einfrieren • außerdem ein 10 ml-Aliquot zur Proteinbestimmung abnehmen A.3 Bestimmung des RNA-Gehaltes einer Probe • in einer Quarzküvette 5 µl RNA-Lösung mit 95 ml ddH2 O-DEPC verdünnen (1:20Verdünnung) • bei 260 und 280 nm Extinktion gegen ddH2 O-DEPC photometrisch bestimmen • zum Errechnen des RNA-Gehaltes in µg/ml den Extinktionswert bei 260 nm mit 800 multiplizieren (dabei muß der Quotient E (260 nm)/E (280 nm) zwischen 1,6 und 2,0 liegen, optimal > 1,8) • Proben entweder sofort im GEArray einsetzen oder bei - 80 ◦ C lagern A.4 Bandshift-Experimente A.4.1 5´-32 P-Oligonukleotidlabeling Das 5´-Endlabeling erfolgte mit Hilfe eines wie folgt beschriebenen modifizierten ReadyTo-Go-T4-Polynukleotid-Kinase (PNK)-Kits der Firma Pharmacia. A.4.1.1 Protokoll: pro Ansatz: • 1 PNK-Kit-Eppendorf-Tube (umgerechnet auf 50 ml: 8 - 10 U PNK, 50 mM Tris-HCl (pH 7,6), 10 mM Magnesiumchlorid, 5 mM DTT, 0,1 mM Heringssperma-DNA, 0,1 mM EDTA (pH 8,0), 0,2 mM ATP, Stabilisatoren) verwenden • 25 µl aqua dest. hinzufügen 96 A.4 Bandshift-Experimente A PROTOKOLLE • 2 - 5 min bei Raumtemperatur inkubieren, anschließend mit der Pipette vorsichtig, aber sehr gründlich mischen • 10 pmol (mind. 5 pmol) jedes Oligonukleotids (= 5 µl bei einer Konz. von 2 pmol/ml; Vorverdünnung: 10 µl des 50 pmol/ml-Oligonukleotids + 240 µl aqua dest.) hinzufügen • 18 µl aqua bidest. ad Gesamtmenge 50 µl hinzufügen • 2 µl γ-ATP32 (3.000 Ci/mmol, 10 mCi/ml, 3,33 pmol/µl), hinzugeben (bis zum Dreifachen an Radioaktivität einsetzbar, um bessere Signale zu erhalten, dann entsprechend weniger Wasser nehmen; außerdem Halbwertzeit von 32 P (14,3 d) berücksichtigen!) • kurz anzentrifugieren • 30 min bei 37 ◦ C inkubieren • die beiden Oligonukleotide (Sense- und zugehöriges Antisense-Oligonukleotid) zusammengeben (Gesamtvolumen: 100 µl) • zum Stoppen des Labelings 5 min bei 95 ◦ C denaturieren, dann über Nacht oder einige Stunden bis auf Raumtemperatur abkühlen lassen, dabei lagern sich die beiden Oligonukleotide zusammen (Annealing) • Radioaktivität (cpm) im Szintillationszähler bestimmen • Reinigen von überschüssigem 32 P und der PNK, die bei der Bindereaktion stören würden, mit Hilfe der Probe Quant Micro Columns auf der Basis einer Gelfiltration gemäß der Anleitung des Herstellers A.4.2 Bindereaktion Während der Bindereaktion kommt es zur Bindung der Transkriptionsfaktoren aus dem Kernextrakt an den 32 P-markierten DNA-Abschnitt aus der Promotorregion des nachzuweisenden Gens. Nach Optimierung der verschiedenen nachfolgend skizzierten BindungsParameter in zahlreichen Vorversuchen wurden die Experimente unter folgenden Bedingungen durchgeführt: 97 A.4 Bandshift-Experimente A PROTOKOLLE Bindungsparameter Endkonzentration im 20 µl-Ansatz Ionenstärke der Ionen, die für die jeweiligen Protein-DNA-Wechselwirkungen erforderlich sind 100 mM Na, w/o Ca Stabilität 10 % Glycerin Reduktion 0,5 mM DTT Chelatoren, um Nukleasen zu inhibieren 1 mM EDTA pH-Wert 7,5-8,0 (10 mM Tris-HCl) Detergentien 0,05 % NP-40 Temperatur Raumtemperatur Ausschluss unspezifischer Interaktionen 2 µg Poly(dI-dC)*poly(dI-dC) Um zu gewährleisten, dass der Bandshift unter den gewählten Versuchsbedingungen prinzipiell funktioniert, wurde als Positiv-Kontrolle ein entsprechendes Experiment mit EBNA1-DNA (EBNA-1 = Epstein-Barr Virus Nuclear Antigen-1) und EBNA-1-Extrakt (kloniert) durchgeführt. A.4.2.1 Typische Konzentrationen der einzusetzenden Proteine bzw. DNAs: • Kernprotein-Extrakt: 5 mg/ml (allg. 0,1 - 10 mg/ml) • 32 P-Target-DNA: 1 ng/ml (allg. 0,1 - 2,5 ng/ml) mit 10000 - 20000 cpm • bei Vergleichsmessungen im Szintillationszähler die Einbaurate bestimmen (ca. 10 50 %) und die einzelnen Target-DNA so verdünnen, dass äquivalente Radioaktivitätsmengen eingesetzt werden A.4.2.2 Protokoll: Pipettierschema (Standard-Versuchsansatz): Reagenzien Volumen (µl) 10x Bindepuffer 2 50 % Glycerin 4 1 % NP-40 1 Poly dI-dC 1 98 A.4 Bandshift-Experimente A Reagenzien Volumen (µl) radioaktiv markierte DNA 2 aqua bidest. + Kern-Protein-Extrakt 10 PROTOKOLLE • Inkubation bei Raumtemperatur für 20 - 30 min, • dann 2 µl Gel-Ladepuffer hinzufügen und • Bandshift-Gel starten A.4.3 Bandshift-Gel und nachfolgende (digitale) Autoradiographie Im nativen Acrylamid-Gel werden die aufgetragenen Konstrukte elektrophoretisch aufgetrennt und anschließend einer Autoradiographie unterzogen. Dabei wandern die TranskriptionsfaktorDNA-Komplexe im elektrischen Feld entsprechend ihrem höheren Molekulargewicht langsamer als die im Überschuss vorhandene radioaktiv markierte DNA, wodurch es zum Shift der Bande letzterer kommt, dem sogenannten Bandshift. benötige Lösungen: 10x TBE-Puffer 10,8 g Tris 55 g Borsäure 40 ml 0,5 M EDTA pH 8,0 ad 1 l mit aqua dest. 0,25x TBE-Puffer 25 ml 10x TBE-Puffer ad 1 l mit aqua dest. Protokoll: Gel ein bis zwei Tage vor der Bindereaktion gießen • Glasplatten besonders intensiv putzen • großes Gel, ca. 25 cm lang, 1,5 mm dick, 4 % oder 8 %ig in 0,25 x TBE-Puffer wie folgt gießen: 99 A.4 Bandshift-Experimente A PROTOKOLLE – 51,25 ml (8 %iges Gel) bzw. 75,25 ml H2 O (4 %iges Gel) – + 12 bzw. 24 ml Acrylamid/Bisacrylamid 30 % (29+1) – + 2,25 ml 10 x TBE (10,8 g Tris, 55 g Borsäure, 40 ml 0,5 M EDTA, pH 8,0, ad 1 l mit aqua dest.) – + 90 µl TEMED – + 90 µl APS (40 %) zum radikalvermittelten Start des Polymerisierungsprozesses • Gel etwa 30 min polymerisieren lassen (bis zu einer Woche bei + 4 ◦ C haltbar) • Gel 30 min bei 200 - 220 V vorlaufen lassen, dann Stromstärke auf 15 mA regulieren • Proben aus der Bindereaktion mit 2 µl Ladefarbstoff versehen, gut mischen und auftragen • Elektrophorese bei 200 V für ca. 1 - 1,5 h, bis sich die vom Bromphenol-blau angezeigte Lauffront etwa 1 cm vom unteren Gelrand entfernt befindet (Vorsicht: keine Radioaktivität auslaufen lassen!) • 1 Glasplatte mit Gel-Spacer entfernen, Gel vorsichtig mit Bloting-Papier von der anderen Platte lösen und mit Frischhaltefolie abdecken • Gel auf dem Geltrockner für ca. 80 min trocknen (80 ◦ C); danach auf Hyperfilm-MP bei -80 ◦ C in einer Gefriertruhe für 5 - 72 h belichten; Film entwickeln und/oder digital mit dem DAR autoradiographieren 100 A.5 Genexpressions-Analyse A A.5 Genexpressions-Analyse A.5.1 Protokoll zur Herstellung der benötigten Lösungen 20x SSC 175,3 g NaCl 88,2 g Natriumcitrat-Dihydrat Wasser ad 900 ml pH auf 7,0 mit 1 M HCl einstellen Wasser ad 1000 ml 20x SDS 200 g SDS Wasser ad 1000 ml auf 65 ◦ C erhitzen um vollständige Lösung zu erreichen Wasch-Lösung 1 (pro Probe) 20 ml 20x SSC 10 ml 20x SDS Wasser ad 200 ml Wasch-Lösung 2 (pro Probe) 20x SSC 5 ml 20x SDS Wasser ad 200 ml dNTP-Gemisch 5 mM dATP 5 mM dGTP 5 mM dTTP 50 µM dCTP Buffer B 50 µl RT Buffer 1 µl 1 M DTT 50 µl dNTP-Gemisch A.5.2 PROTOKOLLE GEArray ASSAY Protokoll Bei allen Schritten sind unbedingt Handschuhe zu tragen!! Herstellung der RNA: Die gesamte RNA der zu untersuchenden Zellen wird mittels Qiagen RNeasy mini kit gemäß Anleitung des Herstellers aus den geernteten PBMC gewonnen. 101 A.5 Genexpressions-Analyse A Probensynthese: Synthese der cDNA - Probe mit α-32 P dCTP: RNA 5 - 10 µg GEAprimer Mix (buffer A) 2 µl RNase-freies Wasser ad 20 µl • In einem sterilen PCR-Gefäß mischen • Probe für 2 min in einen auf 70 ◦ C vorgewärmten Heizblock stellen • Probe auf 42 ◦ C kühlen und bei dieser Temperatur halten Herstellen des Labeling-Mix (20 µl pro RNA-Probe): Buffer B 8 µl α-32 P-dCTP (10 mCi/ml) 5 µl RNase inhibitor 1 µl MMLV Reverse Transkriptase (50 units/µl) 2 µl RNase-freies Wasser 4 µl Labeling-Reaktion: • Für 2 min den Labeling Mix bei 42 ◦ C erwärmen • je 20 µl Labeling Mix zu jeder vorbereiteten (s.o.) RNA-Probe geben • gut vermischen • Inkubation bei 42 ◦ C für 25 min Stoppen der Labeling-Reaktion durch Zugabe von 5 µl Buffer C. Denaturieren der somit hergestellten cDNA-Probe: • Buffer E auf 68 ◦ C erwärmen • Zugabe von 5 µl Buffer D • Inkubation für 20 min bei 68 ◦ C • Zugabe von 50 µl Buffer E • Inkubation für 10 min bei 68 ◦ C 102 PROTOKOLLE A.5 A.5.3 Genexpressions-Analyse A PROTOKOLLE Hybridisierung 1. Als ersten Arbeitsschritt des kompletten Versuchs 15 ml der GEAhyb Hybridisationslösung pro Probe auf 68 ◦ C erwärmen und zur vollständigen Lösung ein wenig Schwenken. 2. Lachs-Sperma-DNA 5 min bei 100 ◦ C denaturieren und dann sofort auf Eis stellen 3. Lachs-Sperma-DNA zur Hybridisationslösung geben (Endkonzentration: 100 µg DNA/ml); bis zum Benutzen bei 68 ◦ C halten 4. Anfeuchten der Membran mit deionisiertem Wasser und Platzieren der Membran in Hybridisationszylinder 5. 10 ml der mit Lachs-Sperma-DNA versehenen Hybridisationslösung dazugeben und 1-2 h bei 68 ◦ C und 5-10 rpm/min prähybridisieren lassen. 6. Anschließend die Prähybridisationslösung verwerfen 7. Die fertige denaturierte cDNA Probe wird zu den restlichen 5 ml Hybridisationslösung gegeben; Hybridisation im Hybridisationszylinder über Nacht bei 68 ◦ C und 5-10 rpm/min 8. Zweimaliges Waschen der Membran mit 100 ml vorgewärmter Waschlösung 1 für 30 min bei 68 ◦ C und 30-40 rpm/min 9. Zweimaliges Waschen der Membran mit 100 ml vorgewärmter Waschlösung 2 für 30 min bei 68 ◦ C und 30-40 rpm/min 10. Die Membran kann nun in Hybridisationsfolie eingeschweißt werden 103 B B EIGENE PUBLIKATIONEN UND KONGRESSBEITRÄGE Eigene Publikationen und Kongressbeiträge Brand JM, Frohn C, Cziupka K, Brockmann C, Kirchner H, Luhm J. Prolactin triggers proinfalmmatory immune responses in peripheral immune cells. Eur Cytokine Netw 2004 (15): 99-104 Luhm J, Cziupka K, Brockmann C, Kirchner H, Koritke P, Brand JM. Prolactin triggers pro-inflammatory immune responses in peripheral immune cells. Jahrestagung der DGFI 2004 Cziupka K, Luhm J, Brockmann C, Kirchner H, Brand JM. Beeinflussung der proinflammatorischen Immunantwort durch Prolaktin. Jahrestagung der NDGFI 2004 104 C C DANKSAGUNG Danksagung Herrn Prof. H OLGER K IRCHNER, Direktor des Institutes für Immunologie und Transfusionsmedizin, danke ich für die Bereitstellung des Arbeitsplatzes und die hervorragende Unterstützung bei der Anfertigung dieser Arbeit. Mein größter Dank gilt meinem Doktorvater Herrn PD Dr. rer. nat. J ÜRGEN L UHM für die Überlassung des Themas, meine labortechnische Ausbildung und die Einführung in die Welt der Forschung. Ohne seine aufmunternden Worte an den Tiefpunkten, die man während der Laborarbeit immer wieder erlebt, seine ständige Unterstützung und seine vielen Ratschläge hätte diese Arbeit nie entstehen können. Meinem Betreuer PD Dr. med. J ÖRG -M ATTHIAS B RAND danke ich für viele hilfreiche Diskussionen und Anregungen zum Thema dieser Arbeit aber auch zu anderen medizinischen Themen. Bei der Laborarbeit wurde ich von vielen MTAs unterstützt, wobei mein besonderer Dank P ETRA KORITKE und P ETRA G LESSING gilt. Mein besonderer Dank gilt meinen Eltern, die mich während der ganzen Zeit immer unterstützt und aufgemuntert haben und mir das Studium der Humanmedizin ermöglichten. 105 D D LEBENSLAUF Lebenslauf Name Geburtsdatum Geburtsort Eltern Geschwister Schulbildung 1987 - 1991 1991 - 1993 1993 - 1999 1996 1998 - 1999 1999 1999 Studium 1999-2005 2001 2002 2004 2005 12/05 - 10/06 Katharina Cziupka 20. Dezember 1980 Stade Günter Cziupka, Studienrat Angela Cziupka, Lehrerin Johannes Cziupka Besuch der Pestalozzi-Grundschule, Stade Besuch der Orientierungsstufe Camper Höhe, Stade Besuch des Gymnasiums Athenaeum, Stade Überspringen der 10. Klasse Teilnahme an allen Runden des deutschen Auswahlverfahrens zur 30. und 31. Internationalen Chemieolympiade Nominierung zur Teilnahme an der 31. Internationalen Chemieolympiade in Bangkok als Mitglied der deutschen Mannschaft; dort Gewinn einer Bronze-Medaille Erwerb der allgemeinen Hochschulreife (Notendurchschnitt 1,0) Studium der Medizin an der Universität zu Lübeck Ärztliche Vorprüfung Erster Teil der Ärztlichen Prüfung Zweiter Teil der Ärztlichen Prüfung Dritter Teil der Ärztlichen Prüfung Approbation als Ärztin Assistenzärztin an der Klinik für Allgemein-, Thorax-, Viszeral- und Kinderchirurgie des Universitätsklinikums Gießen-Marburg, Standort Gießen 106 D LEBENSLAUF seit 10/06 Assistenzärztin an der Klinik für Allgemeinchirurgie der Ernst-Moritz-Arndt-Universität, Greifswald seit 2002 Doktorandentätigkeit am Institut für Immunologie und Transfusionsmedizin der Universität zu Lübeck 2000 - 2005 Stipendiatin der Studienstiftung des deutschen Volkes 107 ABBILDUNGSVERZEICHNIS ABBILDUNGSVERZEICHNIS Abbildungsverzeichnis 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 Ausschnitt aus dem PRL-Promotor . . . . . . . . . . . . . . . . . . . . . . Molekulare Struktur von PRL . . . . . . . . . . . . . . . . . . . . . . . . Schema eines Zytokinrezeptors vom Typ I . . . . . . . . . . . . . . . . . . Isoformen des PRLR . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Räumliche Struktur und Bindungsstellen von PRL und des PRLR . . . . . Mechanismus der Aktivierung des PRLR . . . . . . . . . . . . . . . . . . IRF-1 ist ein multifunktionaler Transkriptionsfaktor . . . . . . . . . . . . . Signaltransduktion über den PRLR . . . . . . . . . . . . . . . . . . . . . . Antagonismus von PRL und Glukokortikoiden . . . . . . . . . . . . . . . . Prinzip des EMSA . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Prinzip des ELISA . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Prinzip des GEArrays . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Einfluss von PRL auf die IL-12-Konzentration im Überstand von Vollblutkulturen nach LPS-Stimulation . . . . . . . . . . . . . . . . . . . . . . . . Einfluss von PRL auf die TNF-α-Konzentration im Überstand von Vollblutkulturen nach LPS-Stimulation . . . . . . . . . . . . . . . . . . . . . . . . Einfluss von PRL auf die IL-10-Konzentration im Überstand von Vollblutkulturen nach LPS-Stimulation . . . . . . . . . . . . . . . . . . . . . . . . Effekt von PRL auf die IFN-γ-Sekretion LPS-stimulierter Zellen im Vollblutansatz . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Prinzip der Vorinkubation mit PRL und anschließenden Stimulation mit LPS Repräsentativer NF-κB-EMSA . . . . . . . . . . . . . . . . . . . . . . . . Repräsentativer IRF-1-EMSA . . . . . . . . . . . . . . . . . . . . . . . . Phosphoimager-basierte quantitative Auswertung dreier EMSAs von NF-κB Phosphoimager-basierte quantitative Auswertung dreier EMSAs von IRF-1 Dotblot mit Antikörpern gegen STAT5b . . . . . . . . . . . . . . . . . . . Repräsentativer EMSA nach Stimulation mit PHA und PRL . . . . . . . . Repräsentativer EMSA nach Stimulation mit cptcAMP, PHA, PMA und NA Repräsentativer GEArray nach Stimulation mit PHA und PRL . . . . . . . Repräsentativer GEArray nach Stimulation mit cAMP, PHA und PMA . . . Repräsentativer GEArray mit RNA aus Mamma-Karzinom-Zellen . . . . . Primer für PRL . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Primer für PRLR . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Ergebnis der PCR nach Inkubation mit PHA und PRL . . . . . . . . . . . . 108 9 10 12 14 15 16 18 20 23 39 41 44 48 49 50 51 52 53 54 55 56 57 59 60 61 62 63 63 64 65 ABBILDUNGSVERZEICHNIS 31 32 33 34 35 36 37 ABBILDUNGSVERZEICHNIS Ergebnis der PCR nach Stimulation mit cptcAMP, PHA und PMA über 7h . 67 Ergebnis der PCR nach Stimulation mit cAMP, PHA und PMA über 14h . . 68 Ergebnis der PCR mit cDNA aus Mamma-Karzinom-Zellen . . . . . . . . 69 Mechanismus der Regulation der Transkription des IRF-1 Gens durch PRL 74 Möglicher Mechanismus der Regulation der Transkription des IRF-1 Gens 77 durch PRL und LPS in Kostimulation . . . . . . . . . . . . . . . . . . . . Inhibitorische Rolle von SHP-1 in der Jak2/STAT5-Kaskade . . . . . . . . 77 Schematische Darstellung des Ablaufs der Proteinsynthese in einer EukaryontenZelle . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 84 109 LITERATUR LITERATUR Literatur [1] A BBAS , L ICHTMAN , AND P OBER. Cellular and Molecular Immunology. Saunders Company, Pennsylvania, 2004. [2] A RNETZ BB, L AHNBORG G, E NEROTH P, AND T HUNELL S. Age-related differences in the serum prolactin response during standardized surgery. Life Sci 35 (1984), 2675– 2680. [3] B ERCZI I, NAGY E, KOVACS K, AND H ORVATH E. Regulation of humoral immunity in rats by pituitary hormones. Acta Endocrinol 98 (1981), 506–513. [4] B ERN HA, AND N ICOLL CA. The comparative endocrinology of prolactin. Rec Prog Horm Res 24 (1968), 681–720. [5] B ERNTON EW, M ELTZER MS, AND H OLADAY JW. Suppression of macrophage activation and T-lymphocyte function in hypoprolactinemic mice. Science 239 (1989), 401–404. [6] B ERWAER M, M ARTIAL JA, AND DAVIS JR. Characterization of an up-stream promotor directing extrapituitary expression of the human prolactin gene. Mol Endocrinol 8 (1994), 635–642. [7] BOLE-FEYSOT C, GOFFIN V, EDERY M, BINART N, AND KELLY PA. Prolactin (PRL) and its receptor: Actions, signal transduction pathways and phenotypes observed in PRL receptor knockout mice. Endocrine Reviews 19, 3 (1998), 225–268. [8] B OLKE E, J EHLE PM, G RAF M, BAIER A, W IEDECK H, S TEINBACH G, S TORCK M, AND O RTH K. Inflammatory response during abdominal and thyroid surgery, a prospective clinical trial on mediator release. Shock 16 (2001), 334ff. [9] B OUCHARD B, O RMANDY CJ, D I S ANTO JP, AND K ELLY PA. Immune system development and function in prolactin receptor-deficient mice. J Immunol (1999). [10] B RAND JM, F ROHN C, C ZIUPKA K, B ROCKMANN C, K IRCHNER H, AND L UHM J. Prolactin triggers pro-infalmmatory immune responses in peripheral immune cells. Eur Cytokine Netw 15 (2004), 99–104. [11] B RAND JM, F ROHN C, L UHM J, K IRCHNER H, AND S CHMUCKER P. Early alterations in the number of circulating lymphocyte subpopulations and enhanced proinflammatory immune response during opipoid-based general anesthesia. Shock 20 (2003), 213–217. 110 LITERATUR LITERATUR [12] B RAZMA A, H INGAMP P, Q UACKENBUSH J, S HERLOCK G, S PELLMAN P, S TO ECKERT C, A ACH J, A NSORGE W, BALL CA, C AUSTON HC, G AASTERLAND T, G LENISSON P, H OLSTEGE FC, K IM IF, M ARKOWITZ V, M ATESE JC, PARKIN SON H, ROBINSON A, S ARKANS U, S CHULZE -K REMER S, S TEWART J, TAYLOR R, V ILO J, AND V INGRON M. Minimum information about a microarray experiment (MIAME)-toward standards for microarray data. Nat Genet 29 (2001), 365–371. [13] B RELJE TC, PARSONS JA, AND S ORENSON RL. Regulation of islet B-cell proliferation by prolactin rat islets. Diabetes 43 (1994), 263–273. [14] B ROCKMANN C, R AASCH W, AND BASTIAN C. Endocrine stress parameters during TIVA with remifentanil or sufentanil. Anaesthesiol Intensimed Notfallmed Schmerzther 35 (2000), 685–691. [15] C ESARIO TC, YOUSEFI S, C ARANDANG H, S ADATI H, L E J, AND VAZIRI N. Enhanced yields of gamma interferon in prolactin treated peripheral blood mononuclear cells. Proc Soc Exp Biol Med 205 (1994), 89–95. [16] C HEN HW, W EIER H, H EININGER HJ, AND H UEBNER RI. Tumorigenesis in strain DW/J mice and induction by prolactin of the groupspecific antigen of endogenous Ctype RNA tumor virus. J Natl Cancer Inst 49 (1972), 1145–1153. [17] C HIKANZA IC. Prolactin and neuroimmunomodulation: in vitro and in vivo observations. Ann NY Acad Sci 876 (1999), 119–130. [18] C LEVENGER CV, A LTMANN SW, AND P RYSTOWSKY MB. Requirement of nuclear prolactin for interleukin-2-stimulated proliferation of T lymphocytes. Science 5015 (1991), 77–79. [19] C LEVENGER CV, F REIER DO, AND K LINE JB. Prolactin receptor signal transduction in cells of the immune system. Journal of Endocrinology 157 (1998), 187–197. [20] C LEVENGER CV, RUSSEL DH, A PPASAMY PM, AND P RYSTOWSKY MD. Regulation of interleukin 2-driven T-lymphocyte proliferation by prolactin. Acad Sci USA 87 (1990), 6460–6464. [21] C ROZIER TA, M ÜLLER JE, Q UITTKAT D, S YDOW M, W UTTKE W, AND K ETTLER D. Effect of anaesthesia on the cytokine responses to abdominal surgery. Br J Anaesthiol 72 (1994), 280–285. 111 LITERATUR LITERATUR [22] D E V ITO WJ, O KULICZ WC, S TONE S, AND AVAKIAN C. Prolactin-stimulated mitogenesis of cultured astrocytes. Endocrinology 130 (1992), 2549–2556. [23] D IEDRICH , H OLZGREVE , J ONAT, S CHNEIDER , burtshilfe. Springer Verlag, Heidelberg, 2000. AND W EISS. Gynäkologie und Ge- [24] D I M ATTIA GE, G ELLERSEN B, D UCKWORTH ML, AND F RIESEN HG. Human prolactin gene expression: the use of an alternative noncoding exon in decidua and the IM-9-p3 lymphoblast cell line. J Biol Chem 251 (9999), 16412–16421. [25] F ORNARI MC, PALACIOS MF, D IEZ RA, AND I NTEBI AD. Decreased chemotaxis of neutrophils in acromegaly and hyperprolactinemia. Eur J Endocrinol 130 (1994), 463–468. [26] F REEMAN ME, K ANYICSKA B, L ERANT A, AND G YÖRGY N. Prolactin: Structure, function and regulation of secretion. Physiological Reviews 80 (2000), 1524–1631. [27] G ALANOS C, L UDERITZ O, AND W ESTPHAL O. A new method for the extraction of r lipopolysaccharides. Eur J Biochem 9 (1969), 245–249. [28] G ELLERSEN B, D I M ATTIA GE, F RIESEN HG, AND B OHNET HG. Regulation of prolactin secretion in the human B-lymphoblastoid cell line IM-9-p3 by dexamethasone but not other regulators of pituitary prolactin secretion. Endocrinology 125 (1989), 2853–2861. [29] G ELLERSEN B, D I M ATTIA GE, F RIESEN HG, AND B OHNET HG. Prolactin (PRL) mRNA from human decidua differs from pituitary PRL mRNA but resembles the IM9-p3 lymphoblast PRL transcript. Mol Cell Endocrinol 64 (9999), 127–130. [30] G ERLO S, VANDEN B ERGHE W, V ERDOOD P, H OOGHE -P ETERS EL, AND KOOIJ MAN R. Regulation of prolactin expression in leukemic cell lines and peripheral blood mononuclear cells. J Neuroimmunol 135 (2003), 107–116. [31] G ERLO S, V ERDOOD P, AND KOOIJMAN R. Tumor necrosis factor-α activates the extrapituitary PRL promotor in myeloid leukemic cells. Journal of Neuroimmunology 172 (2006), 206–210. [32] G LASS CK, ROSE DW, AND ROSENFELD MG. Nuclear receptor coactivators. Curr Opin Cell Biol 9 (1997), 222–232. 112 LITERATUR LITERATUR [33] H ORSEMAN ND, AND Y U -L EE L-Y. Transcriptional regulation by the helix bundle peptide hormones: Growth hormone, prolactin, and hematopoietic cytokines. Endocr Rev 15 (1994), 627–649. [34] I SHIBASHI M, K UZUYA N, S AWADA S, K ITAMURA K, K AMOI K, AND YAMAJI T. Anti-thyroid antibodies in patients with hyperprolactinemia. Endocrinol Jpn 38 (1991), 517–522. [35] JABBOUR HN, C RITCHLEY HO, AND B ODDY SC. Expression of functional prolactin receptors in nonpregnant human endometrium: janus kinase-2, signal transducer and activator of transcription-1 (STAT1) and STAT5 proteins are phosphorylated after stimulation with prolactin. J Clin Endocrinol Metab 83 (1998), 2545–2553. [36] JACOBI AM, ROHDE W, VOLK HD, D ORNER T, B URMESTER GR, AND H IEPE F. Prolactin enhances the in vitro production of IgG in peripheral blood mononuclear cells from patients with systemic lupus erythematosus but not from healthy controls. Ann Rheum Dis 60 (2001), 242–247. [37] JARA LJ, V ERA -L ASTRA O, M IRANDA JM, A LCALA M, AND A LVAREZ N EMEGYEI J. Prolactin in human systemic lupus erythematosus. Lupus 10 (2001), 748–56. [38] K ADIOGLU P, ACBAY O, D EMIR G, G AZIOGLU N, AND G UNDOGDU S. The effect of prolactin and bromocriptine on human peripheral immune status. J Endocrinol Invest 24 (2001), 147–151. [39] K ANIK WS, AND W ILDER RL. Hormonal alterations in rheumatoid arthritis including the effects of pregnancy. Rheum Dis Clin North Am 26 (2000), 805ff. [40] K EELER C, DANNIES PS, AND H ODSDON ME. The tertiary structure and backbone dynamics of human prolactin. J Mol Biol 328 (2003), 1105–1121. [41] K IRCHNER H, K LEINICKE C, AND D IGEL W. A whole-blood technique for testing production of human interferon by leukocytes. J Immunol Methods 28 (1982), 213ff. [42] K ROWN K, WANG Y, H O TWC, K ELLY PA, AND WALKER AM. Prolactin isoform 2 as an autocrine growth factor for GH3 cells. Endocrinology 131 (1992), 595–602. [43] L EHNINGER , N ELSON , scher Verlag, 1982. AND C OX. Prinzipien der Biochemie. Spektrum Akademi- 113 LITERATUR LITERATUR [44] L EITE - DE -M ORAES MC, T OURAINE P, K ELLY PA, K UTTENN F, AND DARDENNE M. Prolactin receptor expression in lymphocytes from patients with hyperprolactinemia or acromegaly. J Endocrinol 147 (1995), 353–359. [45] L EITE DMM, T OURAINE P, G AGNERAULT MC, S AVINO W, K ELLY PA, AND DAR DENNE M. Prolactin receptors and the immune system. Ann Endocrinol 56 (1995), 567–570. [46] L ÖFFLER , AND P ETRIDES. Biochemie und Pathobiochemie. Springer-Verlag, Heidelberg, 1998. [47] L IU X, ROBINSON GW, G OUILLEUX F, G RONER B, AND H ENNINGHAUSEN L. Cloning and expression of Stat5 and an additional homologue (Stat5b) involved in prolactin signal transduction in mouse mammary tissue. Proc Natl Acad Sci USA 92 (1995), 8831–8835. [48] L UO G, AND Y U -L EE L. Transcriptional inhibition by Stat5. Differential activities at growth-related versus differentiation-specific promoters. J Biol Chem 272 (1997), 26841–26849. [49] M ARANA E, A NNETTA MG, M EO F, PARPAGLIONI R, G ALEONE M, M AUSSIER ML, AND M ARANA R. Sevoflurane improves the neuroendocrine stress response during laparoscopic pelvic surgery. Can J Anesth 50 (2003), 348–354. [50] M ARUYAMA S, S UMITA K, S HEN H, K ANOH M, X U X, S ATO M, M ATSUMOTO M, S HINOMIYA H, AND A SANO Y. Identification of IFN regulatory factor-1 binding site in IL-12 p40 gene promoter. J Immunol 170 (2003), 997–1001. [51] M ATERA L. Endocrine, paracrin and autocrine actions of prolactin on immune cells. Life Sci 59 (1996), 599–614. [52] M ATERA L, C ONTARINI M, B ELLONE G, F ORNO B, AND B IGLINO A. Upmodulation of interferon-gamma mediates the enhancement of spontaneous cytotoxicity in prolactin-activated natural killer cells. Immunol 98 (1999), 386–392. [53] M ATERA L, AND M ORI M. Cooperation of pituitary hormone and prolactin with interleukin-2 and interleukin-12 on produktion of interferon-gamma by natural killer and t cells. Ann NY Acad Sci 917 (2000), 505–513. [54] M AVOUNGOU E, B OUYOU -A KOTET MK, AND K REMSNER PG. Effects of prolactin and cortisol on natural killer (NK) cell surface expression and function of human 114 LITERATUR LITERATUR natural cytotoxicity receptors (NKp46, NKp44, NKp30). Clinical and Experimental Immunology 139 (2004), 287–296. [55] M INOO P, Z ADEH MM, ROTTAPEL R, L EBRUN JJ, AND A LI S. A novel SHP1/Grb-2-dependent mechanism of negative regulation of cytokine-receptor signaling: contribution of SHP-1 C-terminal tyrosines in cytokine signalling. Blood 103 (2004), 1398ff. [56] M OSZKORZOVA L, L ACINOVA Z, M AREK J, M USILOVA L, AND D OHNALOVA A. Hyperprolactinaemia in patients with systemic lupus erythematodes. Clin Exp Rheumatol 20 (2002), 807–812. [57] NAGY E, AND B ERCZI I. Immunodeficiency in hypophysectomized rats. Acta Endocrinol 89 (1978), 530–537. [58] NAGY E, B ERCZI I, W REN E, A SA SL, AND KOVACS K. Immunomodulation by bromocryptine. Immunopharmacology 6 (1978), 231–243. [59] NANBU -WAKAO R, F UJITANI Y, M ASUHO Y, M URAMATSU M, AND WAKAO H. Prolactin enhances CCAAT enhancer-binding protein-beta (C/EBPbeta) and peroxisome proliferator-activated receptor gamma (PPAR gamma) messenger RNA expression and stimulates adipogenic conversion of NIH-3T3 cells. Mol Endocrinol 14 (2000), 307–316. [60] N IALL HD, H OGAN ML, S AUER R, ROSENBLUM IY, AND G REENWOOD FC. Sequences of pituitary and placental lactogenic and growth hormones: evolution from a primordial peptide by gene reduplication. Proc Natl Acad Sci USA 68 (1971), 866–870. [61] OKENWA C, REGO D, PANG NJ, AND KOZLOWSK M. Tyrosine phosphatase SHP-1 regulates LPS-induced cytokine networks involved in inflammation. Cell Mol Biol Lett 8 (2003), 590–591. [62] O LSEN NJ, AND KOVACS WJ. Hormones, pregnancy and rheumatoid arthritis. J Gend Specif Med 5 (2002), 28ff. [63] P FITZNER E, JAHNE R, W ISSLER M, S TOECKLIN E, AND G RONER B. p300/CREBbinding protein enhances the prolactin-mediated transcriptional induction through direct interaction with the transactivation domain of Stat5, but does not partcipate in the Stat5-mediated suppression of the glucocorticoid response. Mol Endocrinol 12 (1998), 1582–1593. 115 LITERATUR LITERATUR [64] R EEM GH, R AY DW, AND DAVIS JRE. The human prolactin gene upstream promotor is regulated in lymphoid cells by activators of T-cells and by cAMP. Journal of Molecular Endocrinology 22 (1999), 285–293. [65] ROMEO G, F IORUCCI G, C HIANTORE MV, P ERCARIO ZA, VANNUCCHI S, AND A FFABRIS E. IRF-1 as a negative regulator of cell proliferation. J Interferon Cytokine Res 22 (2002), 39–47. [66] ROSEN JM, W YSZOMIERSKI SL, AND H ADSELL D. Regulation of milk protein gene expression. A Rev Nutr 19 (1999), 407–436. [67] RUSSELL DH. New aspects of prolactin and immunity: a lymphocyte-derived prolactin like product and nuclear protein kinase-C activation. Trends Pharmocol Sci 10 (1989), 40–44. [68] S ABHARWAL P, G LASER R, L AFUSE W, VARMA S, L IU Q, A RKINS S, KOOIJMAN R, K UTZ L, K ELLEY KW, AND M ALARKEY WB. Prolactin synthesized and secreted by human peripheral blood mononuclear cells: An autocrine growth factor for lymphoproliferation. Proc Natl Acad Sci 89 (1992), 7713–7716. [69] S CHEDLOWSKI , AND [70] S CHMIDT-T HEWS , berg, 2000. T EWES. Psychoneuroimmunologie. 1996. AND L ANG. Physiologie des Menschen. Springer-Verlag, Heidel- [71] S TEVENS A, R AY DW, W ORTHINGTON J, AND DAVIS JR. Polymorphisms of the human prolactin gene - implications for production of lymphocyte prolactin and systemic lupus erythematosus. Lupus 10 (2001), 676–683. [72] S UN R, L I LA, W EI HM, AND TIAN ZG. Expression of prolactin receptor and response to prolactin stimulation of human NK cell lines. Cell Research 14, 1 (2004), 67–73. [73] TAKORABET L, ROPARS A, R ABY C, AND C HARREIRE J. Phenothiazine induces de novo MHC class II antigen expression on thyroid epithelial cell. a new mechanism for drug-induced autoimmunity. J Immunol 154 (1995), 3593ff. [74] TANG B, G ULLER S, AND G URPIDE E. Cyclic adenosine 3‘,5‘-monophosphat induces prolactin expression in stromal cells isolated from human proliferative endometrium. Endocrinology 133 (1993), 2197ff. 116 LITERATUR LITERATUR [75] T REDE NS, T SYTSYKOVA AV, C HATILA T, G OLDFELD AE, AND G EHA RS. Transcriptional activation of the human TNF-alpha promoter by superantigen in human monocytic cells: role of NF-kappa B. J Immunol 155 (1995), 902–928. [76] T RUONG AT, D UEZ C, B ELAYEW A, R ENARD A, P ICTET R, B ELL GI, AND M AR TIAL JA. Isolation and characterization of the human prolactin gene. EMBO J 3 (1984), 429–437. [77] X U B. The importance of beta-adrenergic receptors in immune regulation: a link between neuroendocrine and immune system. Med Hypotheses 56 (2001), 273–276. [78] Y U -L EE L- Y. Prolactin stimulates transcription of growth-related genes in Nb2 T lymphoma cells. Mol Cell Endocrinol 68 (1990), 21. [79] Y U -L EE L-Y. Stimulation of interferon regulatory factor-1 by prolactin. Lupus 10 (2001), 591–599. [80] Y U -L EE L-Y. Prolactin modulation of immune and inflammatory responses. Endocrinol Rev (2002), 435–455. 117