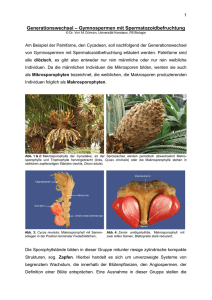
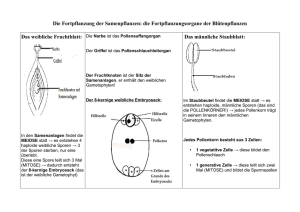
Vergleichende morphogenetische und entwicklungsgeschichtliche
Werbung

Vergleichende morphogenetische und entwicklungsgeschichtliche Untersuchungen an Samenanlagen Dissertation zur Erlangung des Grades eines Doktors der Naturwissenschaften der Fakultät für Biologie und Biotechnologie der Ruhr-Universität Bochum angefertigt im Lehrstuhl für Evolution und Biodiversität der Pflanzen vorgelegt von Julia Kunze, geb. Obermann aus Dortmund Bochum 2008 Referent Prof. Dr. Th. Stützel Koreferent Prof. Dr. R. Tollrian VERZEICHNISSE i INHALTSVERZEICHNIS 0. ABBILDUNGSVERZEICHNIS _________________________________________________V 1. EINLEITUNG ________________________________________________ 1 1.1. EVOLUTION DES INTEGUMENTS 1 1.1.1. THEORIE ZUR INTEGUMENTENTWICKLUNG 2 1.1.2. FOSSILE FUNDE: CUPULA, LAGENOSTOM UND CO. 3 1.1.3. DIE FUNKTION DES INTEGUMENTS 5 MORPHOLOGISCHE UNTERSUCHUNGEN 7 1.2.1. UNTERSUCHUNGEN AN URSPRÜNGLICHEN GYMNOSPERMEN 7 1.2.2. UNTERSUCHUNGEN AN URSPRÜNGLICHEN ANGIOSPERMEN 8 1.2. 1.3. ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 10 1.3.1. ENTWICKLUNGSGENE UND IHRE WIRKUNGSWEISE 10 1.3.2. MADS-BOX-GENE 11 1.3.3. DAS ABC-MODELL 13 1.3.4. DIE ÜBERTRAGUNG DES ABC-MODELLS AUF DIE GYMNOSPERMEN 14 1.4. ZIEL DER ARBEIT 16 2. MATERIAL UND METHODEN ________________________________ 18 2.1. HISTOLOGISCHE UNTERSUCHUNGEN 18 2.1.1. FIXIERUNG DES PFLANZENMATERIALS 18 2.1.2. LICHTMIKROSKOPISCHE UNTERSUCHUNGEN 18 2.1.3. RASTERELEKTRONENMIKROSKOPISCHE UNTERSUCHUNGEN 21 2.1.4. DIGITALE DOKUMENTATION 22 ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 23 MOLEKULARBIOLOGISCHE VORARBEITEN 23 2.2.1.1. In-silico-Analyse und Primerdesign 23 2.2.1.2. RNA-Isolierung und cDNA-Synthese 23 2.2. 2.2.1. VERZEICHNISSE ii 2.2.1.3. Nested PCR 24 2.2.1.4. Gelelektrophorese 24 2.2.1.5. Aufreinigung der DNA 25 2.2.1.6. Klonierung 25 2.2.1.7. M13-PCR und Plasmidcharakterisierung 25 2.2.1.8. Isolierung der Plasmid-DNA 26 2.2.1.9. DNA-Fällung und Sequenzierung 27 2.2.1.10. Synthetisierung der Sonde 27 2.2.1.11. Dot-Blot 28 2.2.2. IN-SITU-HYBRIDISIERUNG 29 2.2.2.1. RNasefreie Fixierung von Frischmaterial 29 2.2.2.2. Überführung in Wachs 30 2.2.2.3. Erstellung von Paraffinschnitten 31 2.2.2.4. Entparaffinierung und Prähybridisierung 31 2.2.2.5. Hybridisierung 32 2.2.2.6. Antikörper-Behandlung 33 2.2.2.7. Detektion 34 2.2.2.8. Stoppen der Reaktion 35 2.2.2.9. Lichtmikroskopie und Dokumentation 35 2.2.3. LÖSUNGSLISTE 35 2.2.4. HERSTELLERNACHWEISE 37 PFLANZENMATERIAL 38 ZAMIA AMBLYPHYLLIDIA (ZAMIACEAE) 38 2.3.1.1. Allgemeine Artbeschreibung 38 2.3.1.2. Beschreibung der untersuchten Individuen 39 GINKGO BILOBA (GINKGOACEAE) 41 2.3.2.1. Allgemeine Artbeschreibung 41 2.3.2.2. Beschreibung des untersuchten Individuums 42 MAGNOLIA STELLATA (MAGNOLIACEAE) 43 2.3.3.1. Allgemeine Artbeschreibung 43 2.3.3.2. Beschreibung des untersuchten Individuums 45 2.3. 2.3.1. 2.3.2. 2.3.3. VERZEICHNISSE iii 3. ERGEBNISSE ______________________________________________ 46 3.1. HISTOLOGISCHE UNTERSUCHUNGEN 46 3.1.1. INTEGUMENTENTWICKLUNG BEI ZAMIA AMBLYPHYLLIDIA 46 3.1.2. INTEGUMENTENTWICKLUNG BEI GINKGO BILOBA 53 3.1.3. INTEGUMENTENTWICKLUNG BEI MAGNOLIA STELLATA 59 ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 66 MOLEKULARBIOLOGISCHE VORARBEITEN 66 3.2.1.1. Primerdesign zur Gewinnung einer MADS-Box-C-Gen-Sequenz 66 3.2.1.2. RNA-Isolierung und cDNA-Synthese 70 3.2.1.3. Klonierung und Sequenzierung der gewonnen Genseqeuenzen 71 3.2.1.4. Synthetisierung der Sonde 76 3.2.1.5. Dot-Blot 77 IN-SITU-HYBRIDISIERUNG – METHODISCHE ERGEBNISSE 78 3.2.2.1. RNasefreie Fixierung von Frischmaterial 78 3.2.2.2. Überführung in Wachs 78 3.2.2.3. Erstellung von Paraffinschnitten 79 3.2.2.4. Entparaffinierung und Prähybridisierung 80 3.2.2.5. Hybridisierung 81 3.2.2.6. Antikörper-Behandlung 81 3.2.2.7. Detektion 82 IN-SITU-HYBRIDISIERUNG – INHALTLICHE ERGEBNISSE 84 3.2.3.1. Sporophylle von Zamia amblyphyllidia 84 3.2.3.2. Blätter von Zamia amblyphyllidia 89 3.2. 3.2.1. 3.2.2. 3.2.3. 4. DISKUSSION _______________________________________________ 91 4.1. EVOLUTION DES INTEGUMENTS 91 4.1.1. DIE TELOMTHEORIE UND DIE MORPHOLOGIE DES INTEGUMENTS 91 4.1.2. DIE FUNKTION DES INTEGUMENTS 94 4.1.3. ÄUßERES INTEGUMENT UND EPIMATIUM IM VERGLEICH 96 4.1.4. EIN NEUES KONZEPT ZUR EVOLUTION DES INTEGUMENTS 98 VERZEICHNISSE 4.2. iv ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 104 4.2.1. IN-SITU-HYBRIDISIERUNGEN AN SPOROPHYLLEN VON ZAMIA 104 4.2.2. IN-SITU-HYBRIDISIERUNGEN AN BLÄTTERN VON ZAMIA 106 5. ZUSAMMENFASSUNG _____________________________________ 107 6. LITERATURVERZEICHNIS __________________________________ 108 7. ANHANG__________________________________________________ 116 ERKLÄRUNG 116 LEBENSLAUF 117 DANKSAGUNG 118 VERZEICHNISSE v 0.1. Abbildungsverzeichnis Abb. 1: Schemazeichnung Samenanlage Abb. 2: Schematische Darstellung eines MADS-box-Gens des MIKC-Typs 12 Abb. 3: Knock-out-Mutanten (MADS-box-Gene) von Arabidopsis thaliana (L.) HEYNH. (aus: RIECHMANN & MEYEROWITZ, 1997, www.its.edu) 13 Abb. 4: Schema zur Funktion der MADS-box-Gene innerhalb der Angiospermen-Blüte 14 Abb. 5: Schematische Darstellung der Sporophylltypen der Cycadales 39 Abb. 6: Zapfen von Zamia amblyphyllidia D.W.STEV. 40 Abb. 7: Zamia amblyphyllidia D.W. STEV., BoGa Bochum 40 Abb. 8: Kurztriebe bei Ginkgo biloba L. 41 Abb. 9: Ginkgo biloba L., Ast mit „Tschitschi“, aufgenommen am MejiSchrein (Japan) 42 1 Abb. 10: Ginkgo biloba L. , Privatanwesen in Herdecke 43 Abb. 11: Blüten von Magnolia stellata (SIEBOLD & ZUCC.) MAXIM 44 Abb. 12: Karpell-/Samenentwicklung bei Magnolia stellata (SIEBOLD & ZUCC.) MAXIM 45 Abb. 13: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM in Blüte, BoGa Bochum 45 Abb. 14: Zamia amblyphyllidia D.W. STEV., REM- und LM-Aufnahmen 49 Abb. 15: Zamia amblyphyllidia D.W. STEV., LM- und REM-Aufnahmen 50 Abb. 16: Zamia amblyphyllidia D.W. STEV., LM-, REM und Binokular-Aufnahmen 51 Abb. 17: Zamia amblyphyllidia D.W. STEV., LM-Aufnahmen 52 Abb. 18: Ginkgo biloba L. , REM-Aufnahmen 55 Abb. 19: Ginkgo biloba L. , REM-Aufnahmen 56 Abb. 20: Ginkgo biloba L. , REM- und LM-Aufnahmen 57 Abb. 21: Ginkgo biloba L. , Fotografie und LM- und REM-Aufnahmen 58 VERZEICHNISSE vi Abb. 22: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM- und REM-Aufnahmen 62 Abb. 23: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM-Aufnahmen 63 Abb. 24: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM- und REM-Aufnahmen 64 Abb. 25: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM-, REM- und Binokular-Aufnahmen 65 Abb. 26: Aminosäuren-Alignment zu MADS-box-Genen der C-Funktionsklasse von Gymnospermen und ausgewählten Angiospermen 67 Abb. 27: Gelelektrophorese zum 1. Schritt der „nested-PCR“ mit den Primern „1-mads_for“ und „Poly-T“ 68 Abb. 28: Gelelektrophorese zum 2. Schritt der „nested-PCR“ mit den Primern „2-C_for“ und „Poly-T“ 68 Abb. 29: Schrittweise Ermittlung eines degenerierten Reverse-Primers „primer-mads_rev“ 69 Abb. 30: Gelelektrophorese zum 1. Schritt der „nested PCR“ mit den Primern „1-mads_for“ und „mads_rev“ 70 Abb. 31: Gelelektrophorese zum Folgeschritt der „nested PCR“ mit den Primern „2-C_for“ und „mads _rev“ 70 Abb. 32: Gelelektrophorese zur Plasmid-DNA nach M13-PCR der Klonierungsprodukte des 2. PCR-Schrittes einer „nested-PCR“ mit den Primern „2-C_ for“ und dem Poly-T-Reverse-Primer 71 Abb. 33: Sequenzierungsergebnis der M13-PCR von Klon 3 72 Abb. 34: Gelelektrophorese zur Plasmid-DNA nach M13-PCR der Klonierungsprodukte des 2. PCR-Schrittes einer „nested-PCR“ mit den Primern „2-C_for“ und „primer-mads_rev“ 73 Abb. 35: Nukleotidsequenz-Alignment der Klone 3, 7, 8 und 9 mit den MADSbox-C-Genen der nah verwandten Gymnospermen Cycas edentata L. und Ginkgo biloba L. 74 Abb. 36: In-silico-Analyse zu den Nukleotidsequenzen der Klone eines MADSbox-C-Gens von Zamia amblyphyllidia D.W. STEV. 75 Abb. 37: Nukleotidsequenz-Alignment der Klone 3, 7, 8, 9 und 13 76 Abb. 38: Schematische Darstellung des Plasmids im Bereich des Inserts 77 VERZEICHNISSE vii Abb. 39: „Dot-Blot“ nach erfolgter Färbereaktion 77 Abb. 40: Blattgewebe nach der Behandlung mit Proteinase K 80 Abb. 41: In der In-situ-Hybridisierung unterschiedlich intensiv gefärbte Gewebeschnitte 83 Abb. 42: Zamia amblyphyllidia D.W. STEV., In-situ-Hybridisierung an weiblichen reproduktiven Strukturen (LM-Aufnahmen) 87 Abb. 43: Zamia amblyphyllidia D.W. STEV., In-situ-Hybridisierung an weiblichen reproduktiven Strukturen (LM-Aufnahmen) 88 Abb. 44: Zamia amblyphyllidia D.W. STEV., In-situ-Hybridisierung an Blättern (LM-Aufnahmen) 90 Abb. 45: Samenschuppe von Saxegothaea conspicua LINDL., REM (Quelle: RESTEMEYER 2002) 97 Abb. 46: Morphologische Reihe zur Entwicklung der Samenanlagen bei Lagarostrobus franklinii (HOOK f.) QUINN, REM (Quelle: RESTEMEYER 2002, Beschriftung ergänzt) 97 Abb. 47: Schemazeichnung zur Entwicklung des Integuments 100 Abb. 48: Längshalbierte Samenanlage von Zamia amblyphyllidia D.W.STEV. zum Zeitpunkt der Bestäubungsreife 101 Abb. 49: Rekonstruktionen fossiler Samenanlagen 103 EINLEITUNG 1. EINLEITUNG 1.1. EVOLUTION DES INTEGUMENTS 1 Die Evolution der Spermatophyta als ein wichtiger Schritt der Pflanzen zum Leben an Land ist eines der in der Geschichte der Botanik am meisten diskutierten Rätsel, dem schon Generationen von Wissenschaftler(inne)n auf die Integument Spur zu kommen versuchen. Fest steht aber, dass die Mikropyle Entwicklung der Samenanlage und die damit von einer feuchten Umgebung unabhängige Befruchtung einen Nucellus entscheidenden Schritt zur Anpassung an ein trockenes MakroHabitat darstellt. Einhergehend mit der Evolution der prothallium Samenanlage verlief die Evolution des Integuments. Als Archegonium Integument wird eine Hüllstruktur um die Samenanlage Abb. 1: Schemazeichnung mit Öffnung, der so genannten Mikropyle, bezeichnet. Samenanlage Das Integument umgibt bei allen rezenten Spermatophyta den Nucellus, in dem sich das Makroprothallium mit den Eizellen enthaltenden Archegonien entwickelt (Abb. 1). Hat eine Befruchtung stattgefunden, spricht man nicht mehr von der Samenanlage, sondern vom Samen (WAGENITZ, 2003). Die ersten Landgänger in der Evolution waren vor ca. 450 Millionen Jahren einfache Gefäßpflanzen wie die fossile Gattung Cooksonia, die eine erste Differenzierung in Anhangsorgane mit unterschiedlichen Funktionen zeigt (BANKS 1971). So ist der für die im Wasser lebenden Vorfahren typische thallöse Aufbau einem Aufbau aus gabelig verzweigten Telomen gewichen, die von einer Protostele, einem ursprünglichen Leitbündeltyp, durchzogen sind. Die Funktion der Wurzel wurde durch Triebe, die meist parallel zum Boden und manchmal sogar abwärtsgerichtet verliefen, übernommen. Die daran inserierenden Rhizoide dienten der Aufnahme von Wasser und Mineralien (ZIMMERMANN 1969). Da auf dieser Entwicklungsstufe kaum Festigungsgewebe ausgebildet waren, zeichneten sich die ersten Landpflanzen durch eine Wuchshöhe von maximal 10 cm aus. Wie alle Vertreter der Rhyniales, einer fossilen Ordnung der Psilophytopsida (Urfarne), trugen sie Sporangien (EDWARDS 1970a). Ein grundlegender Schritt für die Evolution der Samenanlage ist die Heterosporie, bei der im Mikrosporangium viele kleine männliche und im Makrosporangium nur wenige große weibliche Sporen gebildet werden. Einhergehend mit der Differenzierung in die so genannten Mikro- und Makrosporen fand eine Reduktion der gametophytischen Generation statt, da diese aufgrund der in der Spore in großen Mengen enthaltenen, EINLEITUNG 2 gut verfügbaren Nährstoffe nun ohne photosynthetische Energiegewinnung Gameten ausbilden kann. Dabei sind die Makrosporen die wesentlich größeren Sporen, da sie zur Sicherstellung der anfänglichen Entwicklung von der Zygote zum Sporophyten über eine bessere Ausstattung mit Reservestoffen verfügen (ZIMMERMANN 1969). Die Heterosporie ist im Laufe der Evolution mehrfach unabhängig voneinander entwickelt worden und ist rezent innerhalb der Pteridophyta bei den Lycopodiopsida (z.B. bei Isoetes oder Selaginella) wie auch den Filicopsida (z.B. bei Salvinia oder Pillularia) vorzufinden. Ausgehend von der Heterosporie fand eine weitere Reduktion der Sporenmenge bis auf eine einzige Makrospore statt, die die gesamte Makroprothallienentwicklung in sehr stark reduzierter Form innerhalb des Sporangiums durchläuft. Mit diesem am Sporophyten bleibenden Prothallium, das zudem in das Sporangium eingeschlossen ist, ist die Vorstufe zur Samenanlage entwickelt. Ebenfalls stark reduziert findet die Entwicklung des Mirkoprothalliums im Sporangium statt. Es verbleibt jedoch nicht am Sporophyt, sondern wird in der Spore ausgebreitet. Rezent ist diese weit entwickelte, spezielle Form der Endosporie bei Selaginella rupestris (L.) SPRING und Selaginella apoda (L.) SPRING beobachtete worden, die im Normalfall allerdings wie alle anderen Selaginella-Arten auch beide Sporentypen aus den Sporangien entlassen. Mit dem „Sesshaftwerden“ der Makrospore im Sporangium (HOFMEISTER 1851) ist der erste Schritt zur Samenanlage vollzogen. Als Samenanlage bezeichnet man den Komplex aus Makroprothallium, umgebenden Nucellus (= Makrosporangium) und Integument, wobei die Zahl der Integumente innerhalb der Spermatophyten variiert: die Gymnospermen haben konstant ein Integument, während bei den Angiospermen ein bis drei Integumente anzutreffen sind. Über die Evolution des Integuments gibt es verschiedene Theorien, die allerdings alle aufgrund fehlender handfester vergleichender oder entwicklungsgeschichtlicher Grundlagen im Spekulativen geblieben sind. 1.1.1. THEORIE ZUR INTEGUMENTENTWICKLUNG Das bekannteste und derzeit allgemein anerkannte Modell zur Evolution der Spermatophyta liefert ZIMMERMANN (1930) im Rahmen seiner Telomtheorie. Er postuliert mit seiner Theorie, dass sämtliche pflanzlichen Organe aus gabelig verzweigten Trieben entstanden sind. Die Endglieder dieser Gabeläste werden als Telome, die Abschnitte zwischen zwei Gabelungen als Mesome bezeichnet. Durch eine Reihe so genannter Elementarprozesse leitet ZIMMERMANN alle bekannten morphologischen Strukturen aus diesem Verzweigungssystem ab. Einer der Elementarprozesse ist beispielsweise die Übergipfelung, bei der einige Telome stärker im Wachstum gefördert werden als andere, ein anderer ist die Planation, bei der alle Telome in eine Ebene rücken. Von EINLEITUNG 3 letzterem ausgehend kann eine Verwachsung stattfinden, zudem ist eine Reduktion aller Telom-Zustände möglich, so dass z.B. reduzierte Schuppen„blätter“ entstehen können. Der letzte der Elementarprozesse ist die so genannte Einkrümmung, durch die ZIMMERMANN die Entstehung von Sporophyllen mit randständigen Sporangien bei vielen fossilen Pteridophyten oder der rezenten Gattung Equisetum erklärt. ANDREWS (1961) überträgt die Telomtheorie von ZIMMERMANN derart auf die Samenanlagenentwicklung, dass das Integument seiner Interpretation nach im Verlauf der Evolution aus sterilen, an der Basis des Sporangiums stehenden Hülltelomen durch Verwachsungsprozesse entstanden ist. ANDREWS Erklärung des Integuments als ein Verwachsungsprodukt aus sterilen Hüllen dient schon seit vielen Jahrzehnten als ein grundlegendes Modell zur Evolution der höheren Pflanzen. Wahrscheinlich basiert ZIMMERMANNS 1930 im Rahmen der Telomtheorie veröffentlichte Modellvorstellung zur evolutiven Entwicklung der frühen Samenanlagen auf den Ergebnissen von wie auch die weiterführende Interpretation ANDREWS (1961) auf morphologischen Studien an Angiospermen, da die Entwicklung der Integumente bei vielen Angiospermen von der Basis der Samenanlage ausgeht und sich so die in der Telomtheorie formulierte Verschmelzung basal stehender Hülltelome problemlos auf die Samenanlagenentwicklung bei Angiospermen projizieren lässt. Zu dem Zeitpunkt, als die Telomtheorie verfasst wurde, fehlten vergleichende Untersuchungen an Gymnospermen fast völlig und sind auch heute eher spärlich. Das bezüglich des Wachstums der Integumente bei Angiospermen bekannte Schema wurde nach der Entdeckung des Integuments bei den Gymnospermen kurzer Hand auf diese übertragen. 1.1.2. FOSSILE FUNDE: CUPULA, LAGENOSTOM UND CO. Fossile Funde und ihre Rekonstruktionen bilden die Grundlage für Evolutionsstudien bezüglich der Integumententwicklung. Bereits weit vor der Evolution des Integuments sind biologische Entwicklungen wie das Verbleiben des Makroprothalliums im Makrosporangium oder die Evolution der Bestäubung, die beispielsweise die Bildung einer Bestäubungskammer mit einschließt, aufgetreten (ROTHWELL & SCHECKLER 1988). Daher fokussiert sich das Interesse auf die Funde fossiler primitiver Gymnospermen, den evolutiv ersten Pflanzen mit samenanlagenähnlichen Strukturen, d.h. mit einem von einer Hüllstruktur umgebenen Makrosporangium. Eben diese Strukturen dienen als eindeutiges Merkmal zur Abgrenzung von den fossilen Pteridophyten des oberen Devons. Eine hypothetische Entwicklungsreihe von einem von mehreren Lappen umgebenen Makrosporangium zu einem vollständig umhüllten lässt sich anhand der von LONG (1959, 1960a, 1960b) veröffentlichten fossilen Funde aus Berkwickshire, Schottland, erstellen. Die aus dem Unterkarbon stammenden Arten Genomosperma EINLEITUNG 4 kidstoni mit zahlreichen unverwachsenen, lang ausgezogenen Lappen an der Basis eines Makrosporangiums und Genomosperma latens mit mehreren basal stehenden Lappen, die sich über dem distalen Ende des Makrosporangiums zusammenneigen, können in einer evolutiven Entwicklungsreihe als Vorläufer für die von fast vollständig miteinander verwachsenen Lappen umgebenen Makrosporangien von Eurystoma angulare und die bis auf eine apikale Öffnung vollkommen umschlossenen Makrosporangien bei Stamnostoma huttonense interpretiert werden. Die bei St. huttonense anzutreffende Struktur ist funktionell als Samenanlage mit Integument und Nucellus anzusehen. Bei zahlreichen primitiven, fossilen Samenanlagen sind verschiedene Modifikationen am Integument oder Makrosporangium zu erkennen. Auswüchse am distalen Ende des Makrosporangiums scheinen der Aufnahme des windverbreiteten Pollens zu dienen (TAYLOR 1981, NIKLAS 1982). Ist dieser Bereich röhren-, becher- oder glockenförmig umgebildet, so spricht man von einer Salpinx wie sie z.B. fossil bei Eurystoma angulare anzutreffen ist. Eine andere vom Makrosporangium gebildete Struktur mit vermutlich gleicher Funktion ist das auch nur fossil bekannte Lagenostom, das sich aus zwei Komponenten zusammen setzt: zum einen aus einer zentralen Säule, die von der ausgezogenen Spitze des Makrosporangiums gebildet wird und somit der beschriebenen Salpinx entspricht, und zum anderen vom eigentlichen Lagenostom, einer Struktur, die die Mittelsäule becher- oder ringförmig umgibt. Da das nach oben weit geöffnete Lagenostom starr ist, drückt sich das Makrosporangium im weiteren Wachstum in den vom Lagenostom gebildeten Hohlraum hinein und nimmt so den dort angesammelten Pollen auf (STEWART & ROTHWELL 2001). Eine solche Struktur ist zum Beispiel bei Physostoma elegans (STEWART 1983) und zahlreichen weiteren fossilen Funden erkennbar (GIFFORD & FOSTER 1996). Die Abwandlungen in der Ausbildung des Integuments sind ebenfalls sehr vielfältig. So sind neben den abgeplatteten fossilen Samenanlagen mit geflügeltem Integument wie bei Spermolithus devonicus (CHALONER et al. 1977) auch rundliche Samenanlagen mit einer auffälligen äußeren Struktur zu finden. Bei Conostoma kestospermum ist beispielsweise eine bandförmig um die Samenanlage gewundene, unregelmäßig begrenzte Struktur ausgebildet (TAYLOR & LEISMAN 1963). Die bisher erwähnten fossilen primitiven Samenanlagen stehen wie die nahezu alle bekannten Samenanlagen des frühen Karbons auch in einem gabelig verzweigten Achsensystem, dessen Achsen eine becherförmige Struktur bilden. Diese wird als Cupula bezeichnet und darüber definiert, dass sie die Samenanlagen einzeln (z.B. bei Runcaria heinzelinii, GERIENNE et al. 2004) oder in Gruppen (z.B. bei Moresnetia zalesskyi, FAIRON-DEMARET & SCHECKLER 1987) vollständig oder nur teilweise einhüllt EINLEITUNG 5 (ROTHWELL & SCHECKLER 1988). Die Achsen der Cupula können dabei miteinander verschmolzen oder völlig frei sein. Darüber hinaus hüllt die Cupula nicht in jedem Fall den ganzen Samenanlagenkomplex ein, sondern umgibt oft auch, wie beispielsweise bei Archaeosperma arnoldii, zwei sich gegenüberstehende Komplexe jeweils nur zur Hälfte, so dass die Cupulae zusammen die Samenanlagen fast vollständig umgeben (PETTITT & BECK 1968). Oft ist der Cupula eine sekundäre Struktur aufgelagert. Bei Lagenostoma lomaxi handelt es sich dabei z.B. um über die ganze Außenseite der Cupula zerstreut verteilte Drüsen (GIFFORD & FOSTER 1996 nach EMBERGER 1944). Aus evolutionstheoretischer Sicht ist lange Zeit spekuliert worden, ob es sich bei der Cupula um einen Vorläufer des Karpells handelt (STEWART & ROTHWELL 2001). Diese Interpretation basiert auf der Annahme WETTSTEINS (1935), dass die ursprünglichsten Karpelle einsamige Gynoceen mit basaler Plazentation und orthotropen Samenanlagen gewesen seien. Dies hat WETTSTEIN veranlasst, die Polysonaceae oder auch die Casuarinaceae als basal zu betrachten. Molekulare Daten stellen jedoch durchweg Arten mit mehreren Karpellen und marginaler Plazentation an die Basis des Systems der Angiospermen, so dass WETTSTEINS Annahme längst widerlegt ist. Unabhängig davon ist die Cupula aber als eine wichtige Struktur zur Klärung der Evolution der Angiospermen zu betrachten. 1.1.3. DIE FUNKTION DES INTEGUMENTS Die Verlagerung der ursprünglich freien gametophytischen Generation in das Makrosporangium stellt einen deutlichen Vorteil für den Gametophyten dar. Im Sporangium kann sich das Makroprothallium vor äußeren Umwelteinflüssen geschützt entwickeln und ist dadurch sowohl länger lebensfähig als auch über einen längeren Zeitraum befruchtungsfähig (TAYLOR 1982). Im Makrosporangium findet die Befruchtung in völliger Unabhängigkeit von atmosphärischem Wasser statt (STEWARD 1983). Da das Prothallium im Sporangium keine eigene Photosynthese betreibt, sondern allein über den Sporophyten ernährt wird, ist es bis auf die generativen Strukturen vollständig reduziert. Das Makrosporangium mit dem Makroprothallium ist von wenigstens einem Integument umgeben, dem die Schutzfunktion für den Komplex zugesprochen wird (TAYLOR 1982). Ein anderer gedanklicher Ansatz, der die Schutzfunktion des zu Beginn der Makroprothallienentwicklung sehr dünnen Integuments vernachlässigt, befasst sich mit der Öffnungsweise des Sporangiums bei den Spermatophyta. Innerhalb der Pteridophyta sind verschiedene Öffnungsmechanismen des Sporangiums zur Sporenentlassung zu finden. Den Anulus, der aus dickwandigen Zellen besteht, die von dünnwandigen EINLEITUNG 6 Zellen unterbrochen werden, findet man bei den Filicatae, den Echten Farnen. Bei der Austrocknung des Sporangiums verhindern die verdickten Zellwände, dass die Anulus-Zellen einschrumpfen, wodurch die zwischen diesen starren Zellen liegenden dünnwandigen Zellen, die so genannten Stomium-Zellen, beim trocknungsbedingten Zusammenziehen des gesamten Sporangiums einer Sollbruchstelle gleich einreißen. Der Anulus kann das Sporangium sowohl ringförmig umgeben als auch nur auf eine bestimmte Region konzentriert sein. Die zu den Filicatae gehörenden Salviniales, die Wasserfarne, verfügen über ein Sporangium, das mit drei Klappen aufplatzt und eine sich ebenfalls 3-klappig öffnende Spore mit bereits keimendem Prothallium enthält (BRESINSKY 2002). Das Sporangium der Spermatophyta, das einen Funktionswandel zum Nährgewebe für das sich entwickelnde Prothallium vollzogen hat, verfügt über keinen der genannten Öffnungsmechanismen. Der einzige Kontakt zur Umgebung ist über die Bestäubungskammer gegeben, die direkt unter der Öffnung im Integument, der Mikropyle, liegt. Bei fossilen Samenanlagen ist es vielfach eine Aufgabe des Integuments gewesen, Pollen und Archegonien zusammenzubringen. Wie bereits beschrieben ist zu diesem Zweck das Integument eine starre Struktur, die nach oben trichterförmig ausgebildet ist, was die Sammlung vieler Pollenkörner erleichtert, um die Chance auf arteigenen Pollen zu erhöhen (STEWART & ROTHWELL 2001). Nach der Befruchtung ist ein an das Wachstum des Nucellus bzw. Embryos angepasstes Wachstum des Integuments erforderlich (ROTHWELL & SCHECKLER 1988). Die meisten rezenten Gymnospermen benötigen für die Bestäubung einen Bestäubungstropfen zur Aufnahme des Pollens (STÜTZEL & RÖWEKAMP 1999b), denn sie verfügen im Gegensatz zu den Angiospermen nicht über eine Narbe als rezeptives Gewebe, weil die Narbe eine Ausbildung der die Samenanlagen einhüllenden Fruchtblätter ist. Da es vorteilhaft ist, den Bestäubungstropfen möglichst groß zu gestalten und möglichst lange zu exponieren, um die Trefferquote für ein einziges notwendiges arteigenes Pollenkorn zu erhöhen, muss der Tropfen zum einen sehr viskos sein und durch irgendeine Struktur gehalten werden. Die Viskosität wird durch einen extrem hohen Zuckergehalt im Bestäubungstropfen erreicht, wohingegen das Integument als verantwortlich für die Haltestruktur gesehen wird. Insbesondere die evolutiv ersten Samenanlagen tragenden Pflanzen wurden durch Pollenkörner bestäubt, die bewegliche Spermatozoide entließen, die sich aktiv durch den Bestäubungstropfen zu den Archegonien bewegen konnten (THOMAS & SPICER, 1987). EINLEITUNG 1.2. 7 MORPHOLOGISCHE UNTERSUCHUNGEN Die oben vorgestellte Telomtheorie von ZIMMERMANN (1930) (siehe 1.1.1.) dient seit vielen Jahrzehnten als ein grundlegendes Modell zur Evolution der höheren Pflanzen und ist sowohl in Schulbüchern als auch in der Wissenschaftsliteratur abgedruckt, ohne dass aber bislang versucht worden wäre, dieses Modell durch morphologische Studien zu überprüfen. Für die Rekonstruktion der Evolution der Spermatophyta wie auch für die Verifizierung der dazu vorhandenen Modelle ist es jedoch unabdinglich, die Strukturen der Samenanlagen eingehender zu untersuchen. In der Literatur sind zahlreiche Studien zur Entwicklung der reproduktiven Strukturen von verschiedenen Arten sowohl der Gymnospermen als auch der Agiospermen zu finden. Die meisten dieser Studien sind älteren Datums und beinhalten ausführliche Beschreibungen und sehr detaillierte Zeichnungen. Die Informationen beziehen sich jedoch häufig nur auf einzelne Organe oder bestimmte Abschnitte aus der gesamten Organentwicklung, so dass es oftmals sehr problematisch ist, in der Literatur zu der jeweiligen aktuellen Fragestellung passende Daten zu finden. Erschwerend kommt hinzu, dass die meist kunstvoll angefertigten Zeichnungen sowohl vom allgemeinen Wissensstand zur Zeit der Entstehung als auch von der Interpretation des Verfassers beeinflusst sein können. Die nachstehenden beiden Punkte geben einen knappen Überblick über die vorhandenen Daten bezüglich der Entwicklung der Integumente bei den Angio- und Gymnospermen aus der Literatur wie auch aus unveröffentlichten Arbeiten der Arbeitsgruppe am Lehrstuhl. 1.2.1. UNTERSUCHUNGEN AN URSPRÜNGLICHEN GYMNOSPERMEN Morphologische Untersuchungen zur Entwicklung der Samenanlagen von den rezent vorkommenden stammesgeschichtlich ältesten Vertretern der Spermatophyten, den Gymnospermen, wurden bereits zu Beginn des letzten Jahrhunderts angefertigt und anhand von Schemazeichnungen oder sehr detaillierten Zeichnungen dokumentiert. Hervorzuheben sind hierbei die Arbeiten von VELENOVSKÝ (1910), GOEBEL (1923) und CHAMBERLAIN (1935), die an verschiedenen Gymnospermen geforscht haben. Um jedoch die Telomtheorie von ZIMMERMANN (1930) zur Evolution des Integuments aus miteinander verschmolzenen Telomlappen zu überprüfen, liegt es auf der Hand, die Untersuchungen an den Arten zu beginnen, bei denen eine zwei- oder mehrlappige Mikropyle anzutreffen ist. Hier wäre es am ehesten zu erwarten, dass eine Bildung des Integuments durch die kongenitale Verwachsung ursprünglich noch getrennter Telomrudimente nachweisbar ist. Die verfügbaren morphogenetischen Arbeiten von MUNDRY (2000) sowohl zur Entwicklung der Samenanlagen bei Gnetum gnemon L. EINLEITUNG 8 mit einer mehrlappigen Mikropyle als auch zur Samenanlagenentwicklung von Pinus mugo TURRA mit einer zweilappigen Mikropyle ließen aber keine derartigen Verwachsungsprozesse erkennen. Innerhalb der rezenten Gymnospermen werden die Ginkgoaceae und Cycadaceae als die ursprünglichsten Familien angesehen. Ein Indiz für die Urtümlichkeit dieser Art ist die Spermatozoidbefruchtung, wie sie bei Landpflanzen außerhalb der Spermatophyten nur noch bei den Bryophyten und Pteridophyten anzutreffen ist. Für die Betrachtung der Evolution des Integuments sind besonders Vertreter dieser extrem ursprünglichen Familien von Interesse. Sehr umfangreiche Arbeiten zu Ginkgo biloba L. und der Cycadacee Zamia floridana A. DC sind von CAROTHERS (1907) und SMITH (1910) durchgeführt und in detaillierten Zeichnungen festgehalten worden. Allerdings liegen bei diesen oftmals gerade die entscheidenden Strukturen im Grenzbereich des Auflösungsvermögens der zur Präparation bzw. Untersuchung eingesetzten Stereomikroskope, so dass es im Laufe der Wissenschaftsgeschichte zahlreiche Beispiele von fehlerhaften Darstellungen gegeben hat. In neueren Arbeiten sind die aktuellen Untersuchungsergebnisse zum größten Teil fotografisch dokumentiert und liegen so als Rohdaten vor (GIFFORD & FOSTER 1996). Unabhängig davon, auf welche Weise die Daten dokumentiert worden sind, sind sehr selten lückenlose Entwicklungsgänge aufgezeigt. Demzufolge sollen im Rahmen der vorliegenden Arbeit Untersuchungen mit modernster morphologischer Methodik zu den beiden Spermatozoid befruchteten Arten Ginkgo biloba (Ginkogaceae) und Zamia amblyphyllidia (Cycadaceae) anhand von vollständigen Entwicklungsreihen vorgestellt werden. 1.2.2. UNTERSUCHUNGEN AN URSPRÜNGLICHEN ANGIOSPERMEN Die Angiospermen stellen ein bei Weitem intensiver bearbeitete Gruppe dar als die Gymnospermen. Es gibt unzählige Veröffentlichungen zu anatomischen und morphogenetischen Untersuchungen. Morphologische Arbeiten zu Angiospermen sind dabei aber oft sehr alt (z.B. GOEBEL 1923). Die aktuelleren Arbeiten auf morphologischer Ebene befassen sich vornehmlich nicht mehr mit der Histologie oder Morphogenese verschiedener Organe sondern mit der Expression der für die Organbildung nötigen Entwicklungsgene und oftmals auch mit den entsprechenden Knock-Out-Mutanten (ROBINSON-BEERS et al. 1992, KIM et al. 2005, usw.). Ebenso viele Untersuchungen befassen sich allein mit Gensequenzen, um phylogenetisch verwertbare Aussagen für Familien (z.B. Azuma et al. 2001) oder sogar für das gesamte Florenreich (z.B. DOYLE & DONOGHUE 1986) treffen zu können. Dabei ist das Augenmerk entweder auf Nutzpflanzen oder auf sich schnell reproduzierende Pflanzen gerichtet. Erstere zu untersuchen kann Perspektiven für eine effektivere wirtschaftliche Nutzung bieten, EINLEITUNG 9 letztere erfreuen sich zur Untersuchung der reproduktiven Strukturen oder der genetischer Variabilitäten großer Beliebtheit, da ihr Entwicklungszyklus von nur wenigen Wochen schnelle Ergebnisse fördert. Da die an der Basis des Stammbaums der Angiospermen stehende Unterklasse der Magnoliidae weder über einen kurzen Entwicklungszyklus noch über viele nennenswerte Nutzpflanzen verfügt, wurden in den letzten Jahrzehnten keine weitgefächerten Studien an diesen durchgeführt. Lediglich zur Blütenentwicklung liegen umfangreiche Werke von ERBAR & LEINS (u.a. 1981, 1983, 1994, 2000) vor. In diesen Arbeiten wird aber nicht näher auf die Entwicklung der Samenanlage eingegangen, die gerade für die im Rahmen dieser Arbeit untersuchte Fragestellung von Interesse ist. Innerhalb der Unterklasse der Magnoliidae sind es insbesondere die Magnoliaceen, die viele ursprüngliche Merkmale aufweisen. So verfügen sie z.B. über einfache Blätter ohne Stipeln und einen urtümlichen Aufbau der Blüte mit schraubig angeordneten Tepalen, Stamen und Karpellen, wobei die beiden letztgenannten in einer nicht festgelegten, aber immer großen Anzahl vorhanden sind. Die Karpelle entwickeln meist mehrere Samenanlagen, von denen aber häufig nur eine heranreift. In neueren molekularen Stammbäumen treten die Amborellales, Nympheales und Illiciales als die basalsten Ordnungen der Magnoliidae auf (KADEREIT 2002). Aufgrund der Materialverfügbarkeit im Botanischen Garten Bochum wird in der vorliegenden Arbeit das Augenmerk auf die Entwicklungsreihe der Samenanlagen von Magnolia stellata (SIEBOLD & ZUCC.) MAXIM gerichtet. Die Paleoherb-Hypothese von STEBBINS (1974), die besagt, dass an der Basis der Angiospermen vermutlich krautige Pflanzen gestanden haben, spielt in diesem Falle keine Rolle, obwohl sie durch die ersten molekulare Stammbäume von DONOGHUE & DOYLE (1989) untermauert worden sind. 1928 diskutierte ARBER bereits inhaltlich diese Thematik, die Jahrzehnte später als Paleoherb-Hypothese formuliert wurde, unter Verweis auf LOTSY (1906) und stellte dabei fest, dass es aufgrund des langsamen Entwicklungszyklus aber insbesondere die baumförmigen Arten sind, die dazu tendieren, ursprüngliche Blütenmerkmale zu konservieren. In Einklang damit sind bei Magnolia stellata zahlreiche als ursprünglich angesehene Merkmale zu finden und möglicherweise noch weitere zu erwarten. Die meisten der wissenschaftlichen Arbeiten zu den Magnoliaceen, auf die wir heute zurückgreifen können, sind älteren Datums und verfügen oft über sehr detailreiche Zeichnungen, die sich zumeist jedoch auf die Embryologie, die im botanischen Sinne als Entwicklung des Gametophyten und des Embryos aufgefasst wird, beziehen. Die für die in dieser Arbeit untersuchte Fragestellung wichtigen Entwicklungsstadien von Nucellus und Integument sind in diesen Arbeiten nur marginal erwähnt oder gänzlich unbeachtet geblieben (EARLE 1938, LEINFELLNER 1967, DE BOER & BOUMAN 1972). Der derzeit aktuellste Artikel stammt von DE-XING XIAO & FENG-XIA XU aus 2006 und EINLEITUNG 10 dokumentiert die Entwicklung des Makroprothalliums fotografisch. Weitere zahlreiche Fotografien zur Erforschung der Samenanlagenentwicklung und der Morphologie des äußeren Integuments sind in einer 1994 veröffentlichten Arbeit von UMEDA, IMAICHI & KATO enthalten und dienen als Vergleichsbasis für die eigenen Untersuchungen. Ein zusätzlicher Punkt, der die Familie der Magnoliaceae in Blickfeld des evolutiven Interesses rücken lässt, ist die Tatsache, dass ihr reifer Same mit seiner fleischigen, oft intensiv gefärbte Hülle den Samen vieler Cycadeen sehr ähnlich ist, obwohl sich diese Hüllstruktur nicht aus homologen Strukturen innerhalb der Samenanlage bildet. Ein ähnliches Endprodukt liefert auch das Epimatium der Podocarpaceae, dessen Entwicklung bereits mit neusten Methoden studiert wurde (RESTEMEYER 2002, RIEGER 2002). Durch einen Vergleich der Entwicklung der genannten Strukturen, soll geklärt werden, ob das äußere Integument der Angiospermen möglicherweise einer Struktur entspricht, die bereits von der Schwestergruppe, den Gymnospermen, bekannt ist. 1.3. ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN Manchmal entstehen morphologische Fragestellungen, bei denen man mit herkömmlichen histologischen und morphologischen Untersuchungsmethoden an die Grenzen stößt. Hierbei ist es äußerst reizvoll, zu versuchen, ob man mit neuen Methoden, bei denen die verschiedenen Organstrukturen anhand der für ihre Entwicklung aktiven Gene verglichen werden, weiter kommt. Auf entwicklungsgenetischer Basis lassen sich in manchen Fällen Unterscheidungen zwischen Geweben treffen, die hinsichtlich ihres mikroskopisch erkennbaren Differenzierungsgrades nicht trennbar sind. Diese Untersuchungsmethode, die auch im Rahmen der vorliegenden Arbeit zum Einsatz kommt, wird In-situ-Hybridisierung genannt. Mittels der In-situ-Hybridisierung kann die zeitlich und räumlich begrenzte Expression von Entwicklungsgenen in Gewebeschnitten nachgewiesen werden. Die Methode ist risikobehaftet, da sie nicht ganz einfach ist. Außerdem verlangt sie, dass bei den Untersuchungen Material verwendet wird, dass zum richtigen Entwicklungszeitpunkt gesammelt wurde. Allerdings ist es vorab unmöglich, zu sagen, wann welche Gene exprimiert werden. 1.3.1. ENTWICKLUNGSGENE UND IHRE WIRKUNGSWEISE Als Entwicklungsgene werden Gene bezeichnet, die steuernd in einen Entwicklungsprozess eingreifen. Vieler dieser Gene gehören den so genannten Homöobox-Genen EINLEITUNG 11 an, die schon längere Zeit als verantwortlich für ontogenetische Vorgänge bei Tieren bekannt sind. Allen Homöobox-Genen ist ein sehr stark konservierter Bereich - die so genannte Homöobox - gemeinsam, die für die Homöo-Domäne der Homöo-DomänProteine kodiert, die wiederum der DNA-Bindung dient (GEHRING et al. 1994). Hierbei ist es selten ein einzelnes Gen, das eine spezielle Entwicklung hervorruft, sondern viel mehr eine Reaktionskaskade, bei der speziell aufeinander abgestimmt Gene gemeinsam agieren. Sie sind dafür verantwortlich, dass die Zellen eines Individuums, die alle über das gleiche Erbgut verfügen, dennoch andersartig ausgebildet sind. In Abhängigkeit von einem Entwicklungsprozess oder vom physiologischen Zustand einer Pflanze werden die jeweils charakteristischen Gene aktiviert, während andere Gene reprimiert werden. Eine räumlich und zeitlich differenzierte Genaktivität führt zu verschiedenartigen Zelltypen, die ihrerseits für die Bildung unterschiedlicher Gewebe stehen (WEILER 2002). Ein typisches Beispiel für eine Reaktionskaskade liefern THEISSEN et al. (2000) für die Blütenbildung beim pflanzlichen Modellorganismus Arabidopsis thaliana (L.) HEYNH. basierend auf zahlreichen genetische Untersuchungen. Hier sind es fünf nacheinander in Abfolge aktivierte Gene, die benötigt werden, um aus einem undifferenzierten Meristem Blütenorgane zu bilden. Auf verschiedene Umweltreize hin werden zuerst Gene, z.B. das Gen LD (LUMINIDEPENDENS), exprimiert, deren Genprodukte die Expression verschiedener Meristemidentitätsgene zur Bildung des Blütenmeristems induzieren. Die Transkription der Organidentitätsgene wird erst zwei Schritte später durch die Produkte der zwischengeschalteten intermediären Gene aktiviert. Organidentitätsgene wirken bei der genetischen Determinierung des Blütenmeristems zu Petalen und Sepalen (bzw. Tepalen), Stamina und Karpellen oft in Kombination mit anderen Organidentitätsgenen (siehe auch 1.3.3). Genauer spezifiziert werden diese Blütenorgane durch die nachgeschalteten so genannten „Downstream genes“, bei denen es sich wie bei den Organidentitätsgenen um MADS-box-Gene handelt. 1.3.2. MADS-BOX-GENE Eine Gruppe der Entwicklungsgene stellen die MADS-box-Gene dar. Sie kodieren für Transkriptionsfaktoren, die DNA in Form von Proteindimeren binden. Ihre Bezeichnung leitet sich aus dem Akronym der ersten vier Gene ab, die aus dieser großen Genklasse beschrieben wurden. Es handelt sich dabei um das Gen „MCM1“ aus der Bäckerhefe Saccharomyces cerevisiae, um „AGAMOUS“ aus Arabidopsis thaliana, um „DEFICIENS“ aus Antirrhinum majus L., einer Art, die neben A. thaliana häufig für genetische Untersuchungen an Pflanzen genutzt wird, und um das menschliche Gen „SRF“, den so genannten Serum Response Factor. EINLEITUNG 12 MADS-box-Gene treten wie zuvor aufgelistet nicht nur im Pflanzen- sondern auch im Tierreich und bei den Pilzen auf, so dass davon ausgegangen werden kann, dass sie sich im Laufe der Evolution vor der Aufspaltung in die einzelnen Stämme entwickelt haben (THEISSEN et al. 2000). Ein weiteres Indiz für das enorme Alter der MADS-boxGene ist die hoch konservierte ca. 60 Aminosäuren umfassende MADS-Domäne, die bei allen Vertretern nahe zu identisch ist. Der genaue Ursprung ist jedoch ungewiss. Sicher ist nur, dass es vor der evolutionären Trennung von Pflanzen und Tieren zu einer Genverdopplung gekommen sein muss, da es zwei unterschiedliche Typen von MADS-box-Genen gibt: den „ARG80 – bzw. MEF2-Typ“, der bei Tieren und Pilzen anzutreffen ist, und den „MIKC-Typ“, der in Pflanzen vorkommt (ALVAREZ-BUYLLA et al. 2000a). Aufgrund des Vorkommens im gesamten Pflanzenreich sind MADS-boxGene für systematische Analysen gut verwertbar. Der Name “MIKC-Typ” leitet sich von den aufeinander folgenden Genomabschnitten, den MADS-, Intervening-, Keratinlike- und C-terminale-Domänen, ab, die über mehr oder weniger konstante Aminosäurenzahlen verfügen. Die hoch konservierte MADSDomäne ist 57 Aminosäuren lang und bindet DNA sequenzspezifisch. Der MADSDomäne folgt ein kurzer eingeschobener DNA-Abschnitt, die so genannte I-Domäne (= intervening-domaine), von variabler Länge. Die K-Domäne wiederum, deren Name sich von der sehr großen Ähnlichkeit mit dem Keratin-Molekül ableitet, umfasst eine konservierte Region von 70 Aminosäuren. Sie bildet eine amphipatische DNA-Helix, eine Struktur, die sehr oft in den Aktivierungsdomänen von Transkriptionsfaktoren enthalten ist. Somit ist anzunehmen, dass die K-Domäne der verbesserten ProteinProtein-Interaktion dient. Beim letzten Abschnitt, der C-terminalen-Domäne handelt es sich um eine Aminosäurenfolge, die sowohl in der Länge als auch in der Sequenz sehr stark zwischen verschiedenen MADS-box-Genen variiert (PURRUGGANAN et al. 1995). In Abbildung 2 ist ein MADS-box-Gen des in Pflanzen vorkommenden MIKCTyps schematisch dargestellt. MADS -Domäne “Intervening” -Region Keratinähnlicher Abschnitt C-terminales Ende 20 As Abb. 2: Schematische Darstellung eines MADS-box-Gens des MIKC-Typs EINLEITUNG 1.3.3. 13 DAS ABC-MODELL MADS-box-Gene sind nahezu überall in Pflanzen vorzufinden (ALVAREZ-BUYLLA et al. 2000b), nehmen aber eine besondere Rolle bei der Blütenbildung ein. MEYEROWITZ et al. erforschten dies zu Anfang der 90er Jahre anhand von Knock-out-Mutanten an Arabidopsis thaliana, der Modellpflanze der Pflanzengenetik. Sie stellten fest, dass sich die MADS-box-Gene in unterschiedliche Funktionsklassen (A-, B- und C-MADSbox-Gene) untergliedern lassen, die für die unterschiedlichen Organidentitäten der einzelnen Wirtel innerhalb einer Angiospermen-Blüte stehen (COEN & MEYEROWITZ 1991). Bei einer Ausschaltung der A-Funktion entstehen Pflanzen mit deformierten Petalen und Sepalen. Wird die B-Funktion unterdrückt, sind die mittleren Blütenwirtel betroffen, d.h. Petalen werden zu Sepalen und Stamina zu Karpellen umgewandelt. Der Verlust der C-Funktion betrifft die generativen Strukturen, so dass im Zentrum der Blüte anstelle von Stamina und Karpellen Petalen und Sepalen stehen. Aufgrund der allgemein bekannten so genannten „gefüllten Blüten“ ist dieser Funktionsverlust der prominenteste. Die in Abbildung 3 gezeigten Knock-out-Mutanten zu A. thaliana sind einer Veröffentlichung von RIECHMANN & MEYEROWITZ (1997) entnommen. Abb. 3: Knock-out-Mutanten (MADS-box-Gene) von Arabidopsis thaliana (L.) HEYNH. (aus: RIECHMANN & MEYEROWITZ, 1997, www.its.edu) ap1-1: Knock-out-Mutante der A-Funktion; pi-1: Knock-out-Mutante der B-Funktion; ag-1: Knockout-Mutante der C-Funktion Anhand von Doppelmutanten lassen sich eindeutige Rückschlüsse ziehen, welche der A-, B- und C-Funktionen in welcher Kombination die Entwicklung der einzelnen Blütenorgane steuern. Die alleinige Expression von MADS-box-A-Genen ist für die Bildung von Sepalen zuständig. MADS-box-A-Gene in Kombination mit MADS-boxB-Genen führen zur Bildung von Tepalen. Werden in einem Blütenwirtel neben den B-Genen auch die MADS-box-C-Gene eingeschaltet, so entstehen Stamina. Für die EINLEITUNG 14 Bildung von Karpellen ist lediglich die C-Funktion zuständig (WEIGEL & MEYEROWITZ 1994). Über die MADS-box-C-Gene hinaus sind weitere Gene, die D-Funktionsgene, unabdingbar, um die Entwicklung der Samenanlagen zu steuern (MODRUSAN et al. 1994, COLOMBO et al. 1995), so dass das von COEN & MEYEROWITZ (1991) erstellte Schema zu den Wirkorten der verschiedenen MADS-box-Gen-Funktionen ergänzt werden kann (Abb. 4). THEISSEN (2000) fügt diesem Modell eine weitere Funktion hinzu, die E-Funktion, die an der Entwicklung der Organe der inneren Blütenwirtel mitwirkt, d.h. die B-, C- und D-Funktion unterstützt. Sepalen A Tepalen Stamina Karpelle Samenanlagen A B B C C C D Abb. 4: Schema zur Funktion der MADS-box-Gene innerhalb der Angiospermen-Blüte 1.3.4. DIE ÜBERTRAGUNG DES ABC-MODELLS AUF DIE GYMNOSPERMEN Das oben dargestellte ABC-Modell zur Genregulierung der Blütenentwicklung durch MADS-box-Gene basiert auf zahlreichen Untersuchungen vor allem an den Modellpflanzen Arabidopsis thaliana und Antirrhinum majus und bezieht sich somit auf eine klassische Angiospermen-Blüte. Da molekulargenetische Untersuchungen an MADSbox-Genen auf eine evolutive Trennung von Angiospermen und Gymnospermen vor bereits rund 300 Millionen Jahren (BECKER et al. 2000, ZHANG 2004) hinweisen, ist anzunehmen, dass die genannten Gene in ihrer getrennten Weiterentwicklung einen Funktionswandel vollzogenen haben, der die stark abweichende Morphologie von Angiospermen- und Gymnospermen-Blüte bedingt. Aufgrund der langen getrennten Entwicklung und der abweichenden Morphologie ist nicht zwangsläufig zu erwarten, dass die bei Angiospermen gefundenen Gene in gleicher Form bei Gymnospermen anzutreffen sind. Aus diesen Gründen ist eine direkte Übertragung des ABC-Modells auf die Gymnospermen unmöglich. Erschwerend kommt hinzu, dass sich die größte Zahl der molekulargenetischen Arbeiten zu Gymnospermen mit der Phylogenie und Systematik dieser Gruppe beschäftigt und sich daher mit den dafür typischerweise EINLEITUNG 15 genutzten Genen befasst. GADEK et al. (2000) z.B. verwenden die Chloroplastengene matK und rbcL für die Klärung der verwandtschaftlichen Beziehungen innerhalb der Cupressaceae s.l., WANG et al. (2000) bedienen sich u. a. des mitochondrialen nad5Gens zur systematischen Analyse der Pinaceen. Die ersten Untersuchungen zu den bei Angiospermen entdeckten Blütenidentitätsgenen an Gymnospermen zeigen, dass auch Gymnospermen über MADS-box-Gene verfügen, die sich anhand von Sequenzvergleichen den bekannten Gen-FunktionsKlassen der Angiospermen zuordnen lassen. Da es von der Samenkeimung bis zur ersten Zapfenbildung bei Gymnospermen fünf bis 30 Jahre dauern kann, können die gefundenen MADS-box-Gene anders als bei Arabidopsis nicht innerhalb von wenige Jahre andauernden Forschungsprojekten über Knock-out-Mutanten auf ihre Funktion hin untersucht werden. Daher wird hier oftmals auf die Methode der Real-time-PCR zurückgegriffen, mittels der man eine quantitative Aussage über die in den einzelnen, präparierten Pflanzenteilen exprimierten Gene treffen kann (FUKUI et al. 2001, JAGER et al. 2003). Eine andere Möglichkeit bietet die experimentelle Herstellung von transgenen Arabidopsis-Pflanzen mit den Genen, die bei Gymnospermen isoliert worden sind. Die Mutationen an den transgenen Pflanzen lassen Rückschlüsse auf die GenFunktionen zu (SUNDSTRÖM & ENGSTRÖM 2002). Die präzise Lokalisierung des Wirkortes der isolierten Gene erfolgt mittels der in der Angiospermenforschung weit verbreiteten In-situ-Hybridisierung. Diese Methode ist für Gymnospermen wegen des Vorhandenseins von sekundären Pflanzenstoffen in der Handhabung wesentlich problematischer. Trotz allem wurden derartige Versuche an mehreren Taxa der Pinaceen, u.a. Pinus radiata D.DON. (MOURADOV et al. 1998 & 1999), Picea mariana (MILL.) BR., ST. & PB. (RUTLEDGE et al. 1998) und Picea abies (L.) KARST (TANDRE et al 1998, SUNDSTRÖM et al. 1999, CARLSBECKER 2004), sowohl an vegetativen als auch an generativen Knospen durchgeführt. Außer den Pinaceen sind beispielsweise die Gnetaceae Gnetum gnemon L. (WINTER et al. 1999, BECKER 2000) und die Cycadaceae Cycas edentata DE LAUB. (ZHANG et al. 2004) mittels der In-situ-Hybridisierung bearbeitet worden. Bei der letztgenannten Art, der ursprünglichen Gymnospermen Cycas edentata, sind bisher nur die männlichen reproduktiven Strukturen und die reifen Samen untersucht worden. In der Entwicklung befindliche weibliche Zapfen sind insbesondere in den jungen Stadien sehr schwer zugänglich und werden pro Individuum nur in sehr geringer Zahl oder sogar in Einzahl gebildet. Dies erklärt, dass eine Untersuchung der weiblichen reproduktiven Strukturen in ihrer Entwicklung noch aussteht. EINLEITUNG 1.4. 16 ZIEL DER ARBEIT Das Ziel der Arbeit ist es, ein schlüssiges Modell zur Evolution der Samenanlage und insbesondere des Integuments zu entwickeln. Ein solches Modell darf sich nicht wie die klassischen Modelle auf eine formale Ableitung von verschiedenen Phänotypen beschränken, sondern muss im Sinne eines gradualistischen Darwinismus die kontinuierliche Funktionsveränderung einer Struktur mit einbeziehen. Zu diesem Zweck werden morphologische Studien an Gymnospermen durchgeführt und mit den bereits vorliegenden Arbeiten zu Gymnospermen wie auch Untersuchungen an Angiospermen verglichen, um anhand der Unterschiede und Gemeinsamkeiten Rückschlüsse auf die Entstehung und Funktion des Integuments ziehen zu können. Um mehr Licht ins Dunkel der Evolution zu bringen, werden darüber hinaus fossile Funde eingehend studiert und von den herkömmlichen Modellvorstellungen losgelöst interpretiert. Ein besonderer Schwerpunkt wird hierbei auf die Funktion der jeweiligen Strukturen und ihren Funktionswandel gelegt. Anhand von entwicklungsgenetischen Studien an ausgewählten Gymnospermen soll überprüft werden, welche Teile des Modells zur Blütenentwicklung der Angiospermen auf ihre Schwester- und Ausgangsgruppe, die Gymnospermen, übertragbar sind. Die Untersuchungen sollen zudem Klarheit verschaffen, ob es sich um Gene gleichen Ursprungs handelt, die bei Gymnospermen und Angiospermen die Entwicklung der generativen Strukturen steuern, oder ob unabhängig voneinander Gene mit ähnlicher Funktion und Struktur im Laufe der Evolution entstanden sind. Dies kann zur Klärung der Homologie der Samenanlagen von Gymnospermen und Angiospermen beitragen, die bislang unkritisch als gegeben angenommen wird und den Morphologen als Basis zur systematischen Einordnung beider Gruppen zu den Spermatophyten dient. Auch der genetisch arbeitende Taxonom fasst sie aufgrund von Chlorplasten-Genen als Monophylum zusammen. Das macht die Homologie plausibel, ohne sie jedoch zu beweisen. Ein umfassenderes Wissen über die Wirkungsweise von MADS-box-Genen bei Gymnospermen wird dabei helfen zu erkennen, ob die Gennetzwerke ebenso komplex sind wie bei Angiospermen. Darüber hinaus soll durch den zeitlichen und räumlichen Aktivitätsnachweis von einem speziellen Blütenentwicklungsgen, dem MADS-box-CGen, ein Nachweis über die Gewebespezifität des Integuments der Samenanlage der Gymnospermen gewonnen werden, um über die entwicklungsgenetische Klärung der Integumentbildung Rückschlüsse auf die Evolution dieser bedeutenden Struktur ziehen zu können. Die hierfür notwendige Methode der In-situ-Hybridisierung ist für den Lehrstuhl Evolution und Biodiversität der Pflanzen der Ruhr-Universität Bochum EINLEITUNG 17 gänzlich neu. Aufgrund dessen, dass die bei Gymnospermen meist in recht großer Menge vorhandenen sekundären Pflanzenstoffe - vielfach phenolische Verbindungen - schon bei einfacheren histochemischen Verfahren äußerst störend sein können, ist bereits schon vor der Entscheidung für die Anwendung der In-situ-Hybridisierung klar gewesen, dass die etablierten Protokolle für Arabidopsis thaliana und andere Modellorganismen der Angiospermen nicht direkt auf die Gymnospermen übertragbar sein würden. Daher ist nicht allein die Etablierung der Methode der In-situ-Hybridisierung am Lehrstuhl ein Arbeitsziel, sondern vor allem die Entwicklung eines Protokolls, das zu reproduzierbaren Ergebnissen führt. Größe und Generationsdauer bringen dabei Schwierigkeiten mit sich, die analog zu denen zu sehen sind, die entstünden, wenn das zoologische Forschungsobjekt für Entwicklungsgenetik nicht Drosophila sondern ein Elefanten wäre. MATERIAL UND METHODEN 18 2. MATERIAL UND METHODEN 2.1. HISTOLOGISCHE UNTERSUCHUNGEN 2.1.1. FIXIERUNG DES PFLANZENMATERIALS Das Pflanzenmaterial wurde nach der Probenentnahme zum besseren Eindringen der Fixierlösung und der Reduktion des Probenvolumens soweit wie möglich bis zur zu untersuchenden Struktur frei präpariert und unverzüglich in FAA, einem FormalinEthanol-Eisessig-Gemisch, für drei Tage fixiert. Größere Objekte mit einem Volumen von mehr als einem halben Kubikzentimeter wurden für wenigstens fünf Tage im Fixativ belassen. 100 ml dieses Fixatives setzen sich wie folgt zusammen: Ethanol 70 % 90 ml Eisessig (99-100%ige Essigsäure) 5 ml Formalin 40 % 5 ml Zur Lagerung der Proben nach der Fixierung erfolgte die Überführung des Materials in 70%iges Ethanol. Die gegebenenfalls weiteren Präparationen der Proben für die folgenden Untersuchungen wurden in 70%igem Ethanol unter Zuhilfenahme eines Stereomikroskops der Firma ZEISS (Stemi SV11) bei bis zu 40facher Vergrößerung durchgeführt. 2.1.2. LICHTMIKROSKOPISCHE UNTERSUCHUNGEN Für die histologischen Untersuchungen sind Serienschnitte in Paraffintechnik erstellt worden. Die dazu notwendige Entwässerung der zu untersuchenden Objekte erfolgte in zwei Schritten, sofern die Proben nicht bereits in 70%igem Ethanol lagerten: Ethanol 70 % kurz spülen Ethanol 70 % mindestens 24 Stunden Bei der schrittweisen Überführung der Proben in HISTOWAX (HISTOLAB PRODUCTS AB) nach GERLACH (1984) dient Tertiäres Butanol als Intermedium. Da der Schmelzpunkt von Tertiärem Butanol bei 25°C liegt, wurden die Proben in einem auf 37°C temperierten Wärmeschrank für je mindestens 24 Stunden in den unten aufgeführten Lösungen gelagert. Dabei diente der in Lösung IV vorhandene Farbstoff Eosin der MATERIAL UND METHODEN 19 Anfärbung der Objekte, damit diese während der folgenden Schritte im Wachs besser sichtbar waren und sich somit im weiteren Arbeitsverlauf besser handhaben ließen. Im Verlauf des späteren Färbevorgangs ist das Eosin durch Alkohol wieder vollständig herausgelöst worden. 100 ml der zur Überführung benötigten Lösungen setzen sich wie in untenstehender Tabelle aufgelistet zusammen: Lösung I Lösung II Lösung III Lösung IV Lösung V Lösung VI Tertiäres Butanol abs. (in ml) Ethanol (96 %) vergällt (in ml) Aqua dest. (in ml) Eosin 20 35 55 100 100 100 50 50 45 - 30 15 - ja - Die in reines Tertiäres Butanol überführten Proben wurden zusammen mit diesem Intermedium in Glasbehälter mit Deckeln übertragen und die Glasbehälter dann mit HISTOWAX-Kügelchen (HISTOLAB PRODUCTS AB) aufgefüllt. Damit das HISTOWAX (Schmelzpunkt bei ca. 58°C) in die Proben eindringen konnte, wurden sie zugedeckt zwei bis drei Tage in einem an das Abluftsystem angeschlossenem Wärmeschrank bei 63°C gelagert. Anschließend sind die Proben mit abgenommenem Deckel für weitere zwei bis drei Tage im Wärmeschrank belassen worden, um das gesundheitsschädliche Tertiäre Butanol vollständig verdampfen zu lassen. Anschließend erfolgte das Einbetten der Objekte in Wachs (GERLACH 1984). Das Wachsblöckchen wurde nach dem Erkalten um das einzelne Objekt herum zu gleichmäßig-parallelkantigen Quadern geschnitten, die auf kleine Holzblöcke als Halterung aufgeschmolzen worden sind. Die Holzblöcke wurden anschließend in ein Rotationsmikrotom MOD 1130/Biocut (REICHERT-JUNG) mit automatischem Objektrückzug eingespannt. Mittels des Mikrotoms wurden von den Objekten 7-10 µm dicke Serienschnitte angefertigt. Das Aufkleben der erhaltenen Schnittbänder auf Objektträger mit Eiweißglycerin erfolgte nach MAYR (aus GERLACH 1984). Bevor die Schnitte gefärbt werden konnten, mussten sie für einige Stunden in einem Wärmeschrank bei 40°C auf den Objektträgern antrocknen. Das Färben der Serienschnitte erfolgte mittels der Safranin-Astrablau-Färbung nach GERLACH (1984). Zum Entparaffinieren ist allerdings anstelle von Histol das weniger MATERIAL UND METHODEN 20 intensiv nach Orange riechende ROTICLEAR (ROTH) verwendet worden. Als erstes wurden die Schnitte in einer absteigenden Alkoholreihe auf die Färbung in wässriger Lösung vorbereitet: Roticlear I 10 Minuten Roticlear II 10 Minuten Roticlear/Isopropanol abs. (1:1) 5 Minuten Isopropanol abs. 5 Minuten Ethanol 96 % 5 Minuten Ethanol 70 % 2 Minuten Ethanol 50 % 2 Minuten Aqua dest. 2 Minuten Die Zusammensetzung der Safranin- und Astrablau-Lösungen in der nun folgenden Färbung entspricht mit einer Ausnahme den Lösungen A und B bei GERLACH (1984): abweichend von der Rezeptur bei GERLACH ist der Lösung B, d.h. der Färbelösung Astrablau, zur Vermeidung von Pilzinfektionen eine Messerspitze Phenol zugesetzt worden. Die verwendete Safranin-Lösung färbt die DNA und verholzte Zellwände, die viel Lignin eingelagert haben, rot an. Der Farbstoff Astrablau hingegen bewirkt eine Blaufärbung reiner Cellulosewände. Astrablau-Lösung 5 Minuten Aqua dest. kurz spülen Aqua dest. kurz spülen Aqua dest. kurz spülen Safranin-Lösung 5 Minuten Aqua dest. kurz spülen Aqua dest. kurz spülen Aqua dest. kurz spülen Die zunächst überfärbten Serienschnitte wurden anschließend nach Augenmaß mit stark verdünnter Salzsäure differenziert: Aqua dest. + 20 Tropfen HCl (5 %) kurz spülen Um die differenzierten Schnitte auf das Eindeckeln vorzubereiten mussten sie wieder entwässert und in ROTICLEAR überführt werden: MATERIAL UND METHODEN 21 Ethanol 70 % kurz spülen Ethanol 96 % kurz spülen Isopropanol abs. 2 Minuten Roticlear IV 5 Minuten Roticlear V 5 Minuten Roticlear VI 5 Minuten Für die Erstellung von Dauerpräparaten wurden die Objektträger mit den safraninastrablau-gefärbten Serienschnitten mit ENTELLAN NEU (MERCK) bedeckt und unter Vermeidung von Luftblässchen mit Deckgläsern eingedeckt. Die Untersuchung der erstellten Dauerpräparate erfolgte mit einem Lichtmikroskop Axioplan der Firma ZEISS in Hellfeld-Durchlicht-Mikroskopie. Die Ergebnisse wurden über eine daran angeschlossene Digitalkamera ColorView (SOFT IMAGING SYTEMS) fotografisch dokumentiert. Da jedoch viele Objekte die Größe des Kameraausschnitts überschritten, war eine Montage aus bis zu 36 Einzelbildern mittels der Anwendung MIA (Multiple Image Alignment) des speziell für Bildbearbeitung und Bildverwaltung ausgelegten Computerprogramms analySIS der Firma SOFT IMAGING SYSTEM DIGITAL erforderlich. 2.1.3. RASTERELEKTRONENMIKROSKOPISCHE UNTERSUCHUNGEN Zur Vorbereitung auf die Rasterelektronenmikroskopie wurden die Objekte mittels der Critical-Point-Trocknung nach GERSTERBERGER & LEINS (1978) getrocknet. Dazu wurde das präparierte Material zunächst für mindestens 24 h in FDA (FormaldehydDimethylacetat) chemisch entwässert. FDA dient bei der Critical-Point-Trocknung als ein Intermedium und wird im Verlauf der Trocknung gegen das bei der Trocknung eingesetzte CO2 ausgetauscht, das im Critical-Point-Dryer CPD 030 (BALZERS) bei 50 bar und 10°C im flüssigen Aggregatzustand vorliegt. Im weiteren Verlauf der CriticalPoint-Trocknung wird bei 73.8 bar und 31°C der kritische Punkt für das Medium CO2 erreicht und ein direkter Übergang vom flüssigen zum gasförmigen Aggregatzustand findet statt. Der direkte Phasenübergang verhindert Schrumpfungsartefakte wie sie bei einer Trocknung z.B. in einem Wärmeofen entstehen würden. Um ein Zerbersten der Proben bei plötzlicher Abnahme des extremen Innendrucks in der Trocknungskammer zu verhindern, wird das das CO2 langsam kontinuierlich abgelassen. Die getrockneten Präparate wurden abhängig von ihrer Größe entweder mit Leit-C (NEUBAUER) oder Leit-Tabs (PLANO) auf massive Aluminiumzylinder aufgeklebt. Um MATERIAL UND METHODEN 22 für die Rasterelektronenmikroskopie ein Abfließen der Elektronen zu gewährleisten, wurden die Objekte mittels eines Sputtergerätes SCD (BALZERS) hauchdünn mit Gold besputtert. In der Regel beträgt die Besputterungsdauer zwischen drei und fünfzehn Minuten. Eine dreiminütige Besputterung ist beispielsweise bei kleinen und wenig strukturierten Objekten wie den jungen Stadien von Zamia amblyphyllidia D.W. STEV. ausreichend gewesen, wohingegen ältere Entwicklungsstadien derselben Art bis zu fünfmal länger besputtert werden mussten. Die Durchführung der rasterelektronenmikroskopischen Untersuchung der wie oben beschrieben vorbereiteten Objekte erfolgte mittels des über die Sekundärelektronendetektor-Technologie verfügenden Rasterelektronenmikroskops DSM 950 der Firma ZEISS. Die Ergebnisse wurden digital mit Hilfe des Programms DIPS (DIGITAL IMAGE PROCESSING SYSTEM 2.2) erfasst. Die Aufnahmen erfolgten bei einer Auflösung von 2000x2000 Pixel. 2.1.4. DIGITALE DOKUMENTATION Die für diese Arbeit erstellten digitalen Fotos sowohl vom Habitus der untersuchten Pflanzen als auch von Pflanzenteilen im Detail sind entweder mit einer Digitalkamera COOLPIX 990 der Firma NIKON oder mit einer Digitalkamera PowerShot S2 IS der Firma CANON und der dazugehörenden Software aufgenommen und aufgearbeitet worden. Für einige Detailaufnahmen wurden die Objekte unter der Stereolupe Stemi SV 11 (ZEISS) mit angeschlossener Digitalkamera Colorview (SOFT IMAGING SYSTEMS) abfotografiert. Diese Aufnahmen wie auch die Aufnahmen der lichtmikroskopischen und rasterelektronenmikroskopischen Untersuchungen sind mit Hilfe des Computerprogramms Adobe Photoshop 7.0 zu Bildtafeln zusammengestellt und beschriftet worden. MATERIAL UND METHODEN 2.2. ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 2.2.1. MOLEKULARBIOLOGISCHE VORARBEITEN 23 Das Ziel der entwicklungsgenetischen Untersuchungen ist die Durchführung einer Insitu-Hybridisierung. Hierzu ist vorab die Synthetisierung einer Hybridisierungs-Sonde erforderlich. Die hierfür nötigen grundlegenden Arbeitsschritte sind mit der Methode dargestellt, die sich im Laufe der Versuche als die erfolgreichste heraus gestellt hat. 2.2.1.1. IN-SILICO-ANALYSE UND PRIMERDESIGN Der Begriff „In-silico-Analyse“ beschreibt als Überbegriff sowohl die Sequenzsuchen im Internet als auch die Computerprogramm gestützten Verarbeitungen dieser, die letztendlich als Hilfsmittel zum Design von Oligo-Nukleotid-Primern genutzt werden. Die für diese Arbeit verwendeten Sequenzen zu MADS-box-C-Genen von wenigen Gymnospermen und zahlreichen Angiospermen stammen aus der Datenbank des „National Center for Biotechnology Information“ (NCBI). Unter der Web-Adresse http: //www.ncbi.nlm.nih.gov/ ist ein freier Zugriff auf alle Sequenzen möglich. Die Sequenzen sind mit dem Programm BIOEDIT Version 7.0.1. aligniert worden. Aus den Bereichen mit den am deutlichsten übereinstimmenden Gensequenzen sind die Oligo-Nukleotid-Sense- und Anti-Sense-Primer gewählt worden. Ebenfalls in dieses Alignment eingebaut wurden die mir von Dr. Francisco Vergara-Silva zur Verfügung gestellten Primer. Die Primer wurde von der MWG BIOTECH AG (Web-Adresse: http:// www.mwg-biotech.com) synthetisiert. 2.2.1.2. RNA-ISOLIERUNG UND CDNA-SYNTHESE Für die Isolierung der RNA aus pflanzlichem Gewebe wird extrem frisches Material benötigt, das möglichst frei von RNasen ist. Hierzu erfolgte die Materialentnahme und die erforderliche Präparation mit sterilem Werkzeug, gefolgt vom sofortigen Schockgefrieren in flüssigem Stickstoff um die RNase-Aktivität einzudämmen. Das Pflanzenmaterial wurde in einem Mörser unter Zugabe von flüssigem Stickstoff gemörsert. Anschließend wurde aus den nun mechanisch aufgeschlossenen Zellen unter Zuhilfenahme des „RNeasy Plant Mini Kit“ der Firma QUIAGEN die Gesamt-RNA isoliert. Um in den folgenden Schritten mit stabilerer DNA arbeiten zu können wurde die RNA unter Zuhilfenahme des „3´RACE System for Rapid Amplification of cDNA Ends“Kits von INVITROGEN in cDNA umgeschrieben. MATERIAL UND METHODEN 24 2.2.1.3. NESTED PCR Bei der so genannten „nested PCR“, der verschachtelten Polymerase Kettenreaktion, werden zwei verschiedene Primer-Paarungen in zwei aufeinander folgenden PCRSchritten genutzt, um ein spezifischeres Ergebnis zu erzielen. Im Rahmen dieser Arbeit wurden im ersten PCR-Schritt unspezifische, degenerierte Primer für MADSbox-Gene im Allgemeinen eingesetzt. Das Ergebnis dieser PCR mit Temperaturgradienten im Thermocycler PCRExpress (HYBAID) diente als Grundlage für die darauf folgende PCR, bei der nun spezifischere, degenerierte Primer für MADS-box-C-Gene zum Einsatz gekommen sind. Diese Verfahrensweise wurde mehrfach abgewandelt, um das Ergebnis immer wieder zu optimieren. Das Basisprotokoll für einen PCRAnsatz sieht folgendermaßen aus: Puffer: 5,0 µl MgCl2: 3,0 µl H20: 36,5 µl Forward Primer: 1,0 µl Reverse Primer: 1,0 µl dNTPs: 1,0 µl cDNA: 2,0 µl taq: 0,5 µl Die für alle im Rahmen dieser Arbeit durchgeführten PCRs eingesetzten Substanzen MgCl2, dNTP-Mixe, Puffer und Taq-Polymerasen stammen sowohl von der Firma INVITROGEN als auch von PROMEGA. Von letztgenanntem Anbieter wurden ebenfalls die aufeinander abgestimmten „5X Green GoTaq Reaction Buffer“ bzw. „5X Green GoTaq Flexi Reaction Buffer“ in Kombination mit der „GoTaq DNA Polymerase“ verwendet. 2.2.1.4. GELELEKTROPHORESE Die Gelelektrophorese dient der Auftrennung der in einer PCR amplifizierten DNAFragmente die durch die Einlagerung von Ethidiumbromid unter UV-Licht im Gel sichtbar werden. Hierzu wurde aus TBE-Puffer und Agarose ein 1%iges Agarosegel hergestellt, auf das die PCR-Proben vermischt mit einem Ladepuffer (Bromophenol Blue Loading Solution, PROMEGA) sowie einem Marker (Gene RulerTM 100bp DNALadder Plus, FERMENTAS) für die Fragmentlängenbestimmung aufgetragen worden sind. Die Laufzeit der Gelelektrophorese betrug bei 100 V und 175 mA ungefähr 20 bis 30 Minuten. MATERIAL UND METHODEN 25 2.2.1.5. AUFREINIGUNG DER DNA Bevor die aus einer PCR gewonnene DNA für weitere Versuche eingesetzt werden kann, muss sie aus dem Gel herausgelöst und aufgereinigt werden. Die mit einem sterilen Skalpell aus dem Gel ausgeschnittenen Banden gewünschter Fragmentlänge wurden in mehreren Zentrifugationsschritten aufgereinigt und behandelt, so dass die PCR-Fragmente anschließend in H2O gelöst vorlagen. Die Aufreinigungen wurden unter Verwendung der beiden Kits „QIAquick Gel Extraction Kit (50)“ von QUIAGEN und „Wizard SV Gel and PCR Clean-Up System“ von PROMEGA durchgeführt. 2.2.1.6. KLONIERUNG Die Klonierung dient der exponentiellen Vermehrung der aufgereinigten DNA-Fragmente. Dazu wurden die gewünschten DNA-Fragmente in ein Plasmid, einen so genannten Vektor (pGEM-T Easy, PROMEGA), ligiert. Die ligierten Plasmide wiederum wurden in kompetente Escherichia coli-Zellen (Stamm JM 109) transformiert. Die Transformierungen wie auch die vorangegangene Ligationen sind entsprechend der Anleitung des Kits „pGEM-T und pGEM-T Easy Vector Systems“ der Firma PROMEGA vorgenommen worden. Bevor die E. coli-Zellen ausplattiert worden waren, sind sie für 1,5 Stunden bei 37°C in 950 µl LB-Medium angewachsen. Von dieser Lösung wurden 100 ml und 200 ml abgenommen und auf zwei LB+X-Gal-Platten verteilt, auf denen die Bakterien für einige Tage bei 37°C weiter gewachsen sind. Da die Insertionsstelle im lacZ-Gen des aufgenommen Plasmids liegt, ist bei den ein Insert enthaltenden Bakterienkolonien der X-Gal-Abbau gestört und sie verfärben sich anders als die Bakterienkolonien mit intakter ∃-Galactosidase nicht blau, sondern bleiben weiß. Die weißen, Insert enthaltenden Kolonien wurden von den Platten gepickt, um zum einen Mittels der M13-PCR (siehe 2.2.1.7.) die Länge der aufgenommen Inserts zu bestimmen und zum anderen die für die PCR gepickten Kolonien für die Plasmidisolierung zu kultivieren. Die Kultivierung erfolgte durch Inkubation der Kolonien in 300 µl - 600 µl LB+AMP-Nährmedium unter Schütteln im Wärmeschrank bei 37°C. 2.2.1.7. M13-PCR UND PLASMIDCHARAKTERISIERUNG Bei der M13-PCR setzt man ein spezielles Primer-Paar ein, das vor und hinter der Insertionsstelle im Plasmid ansetzt und somit während der PCR nur das Insert und einen kurzen, definierten Bereich des Plasmid-Genoms vervielfältigt. Die Sequenzen der im Rahmen der vorliegenden Arbeit genutzten Primer lauten zum einen für den MATERIAL UND METHODEN 26 M13-forward-Primer 5`-GTTTTCCCAGTCACGAC-3´ und zum anderen für den M13reverse-Primer 5`-CAGGAAACAGCTATGAC-3`. Pro gepickter Kolonie ist folgender Ansatz pipettiert worden: Puffer: 3 µl MgCl2: 3 µl H20: 13 µl dNTPs: 2 µl M13_for: 3 µl M13_rev: 3 µl taq-Polymerase: 1 µl gelöste Bakterien: 2 µl (bei direktem Lösen der gepickten Kolonien im PCR-Ansatz Zugabe von 2 µl H2O) Als Plasmidcharakterisierung bezeichnet man eine PCR mit einem Mix kurzer Primer, die bei unterschiedlichen Klonen an unterschiedlichen Stellen im Genom ansetzten, so dass sich unterschiedliche Bandenmuster im Gel ergeben. Läuft parallel ein bereits sequenziertes Insert im Gel mit, ist durch einen Abgleich der Bandenmuster eine Schlussfolgerung auf die Gensequenz der einzelnen Klone insofern möglich, als das sich sagen lässt, ob einer der unbekannten Klone dasselbe Insert wie der schon bekannte Klon enthält. Pro Klon wurde folgender PCR-Ansatz zusammengestellt: Puffer: 3 µl MgCl2: 3 µl H20: 15 µl dNTPs: 2 µl Primer-Mix: 5 µl Plasmid-DNA: 1 µl taq: 1 µl 2.2.1.8. ISOLIERUNG DER PLASMID-DNA Die Isolierung der Plasmid-DNA aus den Klonen mit gewünschter Fragmentlänge bzw. Bandenmuster erfolgte mit Hilfe des „NucleoSpin 96 Plasmid Kits“ (MACHERREYNAGEL) und des Kits „Wizard Plus SV Minipreps DNA Purification System“ der Firma PROMEGA unter geringfügigen Abwandlung wie z.B. einer verlängerten Inkubationszeit zur Bindung an die Membranen. MATERIAL UND METHODEN 27 2.2.1.9. DNA-FÄLLUNG UND SEQUENZIERUNG In Anschluss an die so genannte Miniprep wurde die erhaltene DNA-Menge über die Extinktionsmessung bei den Wellenlängen 260 nm für die DNA-Konzentration und 280 nm für den Reinheitsgrad der Probe mit einem Extinktionsmessgerät der Firma EPPENDORF (Biophotometer) ermittelt. Für die DNA-Fällung werden 2 µg mit wie folgt zusammen pipettiert und bei -20°C für 2 Stunden gelagert: Natriumacetat 1/10-tel Volumen der DNA EtOH p.a., -20°C 2,5-fache Volumen der DNA Anschließend ist für 30 Minuten bei 4°C zentrifugiert und der Überstand verworfen worden. Nach der Zugabe von 600 µl bis 1000 µl 70%igen Ethanol auf Eis wurde für weitere 5 Minuten zentrifugiert, der Überstand verworfen und die Probe im Wärmeschrank für etwa 20 Minuten zum Trocknen belassen. Zur Sequenzierung wurde die gefällte DNA an die MWG BIOTECH AG (Web-Adresse: http://www.mwg-biotech.com) geschickt, von wo die Ergebnisse der Sequenzierung per E-Mail übermittelt worden sind. Die erhaltenen Sequenzen werden mit bekannten Sequenzen aus der Datenbank des „National Center for Biotechnology Information“ (Web-Adresse: http://www.ncbi.nlm.nih.gov/) abgeglichen. Die vom „NCBI“ erstellten Stammbäume ermöglichen zudem eine Einordnung zu übergeordneten Gengruppen. 2.2.1.10. SYNTHETISIERUNG DER SONDE Basierend auf den Sequenzierungsergebnissen wurde das PCR-Produkt der M13PCR eines Klons als Template für die Synthetisierung der Sonde eingesetzt. Vorab wurde anhand einer Extinktionsmessung im Extinktionsmessgerät Biophotometer der Firma EPPENDORF die einzusetzende Menge an DNA ermittelt. Es wurden zwei Ansätze pipettiert: zum einen der Sonden-Mix mit der T7-Polymerase, zum anderen der Kontrollen-Mix mit der SP6-Polymerase. Die Kontrolle hat in der In-situHybridisierung als negativ Kontrolle Verwendung gefunden. Im Rahmen dieser Arbeit ist mit Digoxigenin-markierten Sonden gearbeitet worden, die mit Hilfe des „DIG labeling Kit“ von ROCHE synthetisiert worden sind. Gefällt wurden Sonde und Kontrolle durch Zugabe der untenstehenden Komponenten und anschließendem Schockgefrieren in flüssigem Stickstoff oder der Lagerung für 2 Stunden bei -20°C. MATERIAL UND METHODEN 28 Der Ansatz für die Fällung setzte sich folgendermaßen zusammen: Ammoniumacetat 1/10tel Volumen der RNA Glykogen 1 µl EtOH p.a., -20°C 100 µl Nach dem Gefrieren sind die Proben für 30 Minuten bei 4°C zentrifugiert worden, der Überstand wurde abgenommen, 100 µl 70%iger Ethanol zugegeben und nochmals 15 Minuten bei 4°C zentrifugiert. Nach der Abnahme des Überstandes und einer mindestens 5-minütigen Trocknung im Wärmeschrank bei 37°C wurden die Pellets von Sonde und Kontrolle in je 50 µl DEPC-Wasser resuspendiert und zur weiteren Lagerung bei -80°C weggefroren. 2.2.1.11. DOT-BLOT Der Dot-Blot dient der Bestimmung der für die In-situ-Hybridisierung einzusetzenden Menge an Sonde. Dafür wurden in Anlehnung an ZACHGO (2002) je 1 µl Sonde in unterschiedlichen Konzentrationen (1:10, 1:100, 1:1000, 1:10.000) und 1 µl KontrollRNA (aus: „RevertAidTM First Strand cDNA Synthesis Kit“ von FERMENTAS) auf eine Filtermembran „Schleicher & Schuell Protran Nitrocellulose Membranes“ der Firma WHATMAN gegeben und auf untenstehende Weise unter Zuhilfenahme einiger Komponenten des „DIG Nucleic Acid Detection Kit“ (ROCHE) behandelt. 0.2M NaOH 5 Minuten 2x SSPE 5 Minuten Puffer 1 + 0,1% Tween20 5 Minuten Puffer 1 + 0,1% Tween20 + 0,5 % Blockingreagenz 20 Minuten Puffer 1 + anti-DIG-Lösung (1:3000) 50 Minuten Puffer 1 + 0,1% Tween20 5 Minuten Puffer 1 + 0,1% Tween20 10 Minuten Puffer 1 + 0,1% Tween20 15 Minuten Puffer 2 5 Minuten Der letzte Inkubationsschritt erfolgte in 10 ml Puffer 2, in denen eine Färbetablette „SIGMA FASTTM BCIP/NBT“ der Firma SIGMA-ALDRICH gelöst worden war. Nach etwa 10 Minuten war eine Färbung erkennbar. Die Farbintensität der unterschiedlichen Sonden-Konzentrationen wurde mit der Färbung der Kontroll-RNA abgeglichen, um die für die In-situ-Hybridisierung einzusetzende Menge an Sonde zu ermitteln. MATERIAL UND METHODEN 2.2.2. 29 IN-SITU-HYBRIDISIERUNG Die im Rahmen dieser Arbeit durchgeführten In-situ-Hybridisierungen basieren auf dem Laborprotokoll „In Situ Hybridization Protocol using Digoxigenin-labeled probes on tissue sections (Stand April 2003)“ der Arbeitsgruppe von Dr. Stefan Gleissberg am Institut für Spezielle Botanik der Universität Mainz. Es wurde von GLEISSBERG und Dr. Erwin Groot in Anlehnung an das Protokoll von ZACHGO (2002) entwickelt. Auf die in den einzelnen In-situ-Hybridisierungen vorgenommenen Modifikationen wird im Ergebnisteil eingegangen. 2.2.2.1. RNASEFREIE FIXIERUNG VON FRISCHMATERIAL Die Materialsammlung für die In-situ-Hybridisierung erfolgte zur Vermeidung einer Kontamination der Proben mit RNasen unter möglichst sterilen Bedingungen. Um die Aktivität der immer in lebendem Gewebe vorkommenden RNasen zu hemmen, ist das Pflanzenmaterial gekühlt zur unmittelbaren Präparation unter einer Stereolupe transportiert worden, sofern die Präparation der Proben nicht am Ort der Probenentnahme mittels sterilem Werkzeug und einer sterilen Glasplatte als Arbeitsfläche erfolgten konnte. Direkt in Anschluss an die Präparation wurden die Objekte in das vorab angesetzte, gekühlte Fixativ überführt, so dass zwischen Probenentnahme und Fixierbeginn nicht mehr als 30 Minuten vergangen sind. Für die ausstehenden In-situ-Hybridisierungen wurde auf zweierlei Weise fixiert: zum einen in FAA, einem Formalin-Ethanol-Eisessig-Gemisch (siehe 2.1.1., jedoch mit Fixierung bei 4°C), und zum anderen entsprechend dem unten stehendem Rezept mit Paraformaldehyd nach ZACHGO (2002). Beide Fixative wurden vor der Fixierung aus RNasefreien Chemikalien frisch angesetzt. 16 % Paraformaldehyd 10x PBS DEPC-Wasser 10 ml 4 ml 26 ml Um das Eindringen des Paraformaldehyd-Fixatives zu beschleunigen, wurde in den Fällen ein Vakuum angelegt, in denen die zu fixierenden Proben nicht von alleine im Fixativ abgesunken sind oder Proben mit einem Volumen von mehr als einem halben Kubikzentimeter fixiert wurden. Unabhängig vom Anlegen eines Vakuums betrug die Fixierdauer mindestens 12 Stunden bei 4°C. Deutlich länger wurde aber nicht fixiert, da spätestens nach dieser Zeit von einer vollständigen Durchfixierung des Materials auszugehen ist und eine weitere Verlängerung der Fixierdauer keinen Einfluss auf das Ergebnis hat. MATERIAL UND METHODEN 30 2.2.2.2. ÜBERFÜHRUNG IN WACHS Für die schrittweise Überführung der mit Paraformaldehyd fixierten Proben in HISTOWAX (HISTOLAB PRODUCTS AB) bzw. Paraplast Plus (TYCO HEALTHCARE/ KENDALL) diente Roti-Histol (ROTH) als Intermedium. Als erstes wurden die Proben direkt in Anschluss an die Fixierung mit dem im Fixativ enthaltenen 10x PBS bei weiterhin 4°C gewaschen. Danach durchliefen sie eine aufsteigende Alkoholreihe zur Entwässerung bei Raumtemperatur. Zur besseren Durchmischung der Lösungen wurden die Proben während untenstehender Reihe gewippt. Der dem 95%igen Ethanol zugesetzte Farbstoff Eosin (WALDECK DIVISION CHROMA) diente der Anfärbung der Objekte, damit diese im Wachs besser sichtbar wurden und sich somit besser im Wachsblock für das Schneiden am Mikrotom ausrichten ließen. Im Verlauf der In-situ-Hybridisierung ist das Eosin durch Alkohol wieder vollständig herausgelöst worden. 10x PBS 30 Minuten bei 4°C 10x PBS 30 Minuten bei 4°C Ethanol 30 % mind. 30 - 60 Minuten Ethanol 50 % mind. 30 - 60 Minuten Ethanol 70 % mind. 30 - 60 Minuten Ethanol 85 % mind. 30 - 60 Minuten Ethanol 95 % mind. 30 - 60 Minuten Ethanol 100 % mind. 30 - 60 Minuten Ethanol 100 % mind. 30 - 60 Minuten Ethanol 100 % : Roti-Histol (1:1) mind. 60 Minuten Roti-Histol mind. 60 Minuten Roti-Histol mind. 60 Minuten Die im Formalin-Ethanol-Eisessig-Gemisch (FAA) fixierten Proben wurden auf Eis mit 70%igem Ethanol gewaschen und anschließend für mindestens 24 Stunden in 70%igem Ethanol gelagert, so dass obige Entwässerungsreihe erst vom Schritt im 85%igem Ethanol durchlaufen worden ist. Die in Roti-Histol überführten Proben wurden in Glasbehälter übertragen, mit RotiHistol bedeckt und mit mindestens der doppelten Menge an geschmolzenem HISTOWAX bzw. Paraplast Plus aufgefüllt. Damit das HISTOWAX (Schmelzpunkt bei ca. 58°C) bzw. Paraplast Plus (Schmelzpunkt bei ca. 56°C) in die Proben eindringen kann, wurden die Proben bei knapp 60°C gelagert. Eine Woche lang erfolgte durch MATERIAL UND METHODEN 31 das tägliche Abschütten und Wiederauffüllen von Wachs unter Vermeidung eines Trockenfallens der Proben und durch einen einmaligen Austausch des Glasbehälters die Beseitigung des Intermediums Roti-Histol. 2.2.2.3. ERSTELLUNG VON PARAFFINSCHNITTEN Zuerst wurden die in Wachs überführten Objekte mittels steriler Keramikschälchen eingebettet. Nach dem Erkalten wurde das Wachs um das einzelne Objekt herum zu Quadern geschnitten und die Objekte auf Holzblöcke als Halterung aufgeschmolzen. Die Holzblöcke sind anschließend in ein Rotationsmikrotom MOD 1130/Biocut der Firma REICHERT-JUNG eingespannt worden. Es wurden Schnitte von 10 µm Dicke angefertigt, die einzeln abgenommen und mit Hilfe steriler Pinzetten auf spezielle, Polylysin-beschichtete Objektträger POLYSINETM der Firma ERIE SCIENTIFIC COMPANY übertragen wurden. Zum Strecken der Schnitte wurden die Objektträger auf eine vor Staub geschützte Wärmeplatte (40 – 50°C) gelegt und einige Tropfen DEPC-Wasser darauf gegeben. Vor der weiteren Behandlung mussten die Schnitte über Nacht antrocknen; eine längere Lagerung bei 4°C ist möglich. 2.2.2.4. ENTPARAFFINIERUNG UND PRÄHYBRIDISIERUNG Als Vorbereitung auf die eigentliche In-situ-Hybridisierung wurden die Schnitte als erstes mit Roti-Histol entparaffiniert und in einer absteigenden Alkoholreihe für die folgenden Schritte in wässrigen Lösungen präpariert, die wiederum dem Andauen der Gewebeschnitte (Proteinase K-Behandlung) und der Erhöhung der Permeabilität der Zellmembranen (Triethanolaminen mit Essigsäure-Anhydriden) dienen, damit die Sonde während der Hybridisierungsreaktion besser ins Gewebe Eindringen kann. Roti-Histol I (2x genutzt) 5 Minuten Roti-Histol II (1x genutzt) 5 Minuten Roti-Histol III (neu) 5 Minuten Roti-Histol : Ethanol (1:1) 5 Minuten Ethanol 100 % 5 Minuten Ethanol 100 % 5 Minuten Ethanol 95 % in NaCl 0,85 % 2 Minuten Ethanol 85 % in NaCl 0,85 % 2 Minuten Ethanol 70 % in NaCl 0,85 % 2 Minuten Ethanol 50 % in NaCl 0,85 % 2 Minuten Ethanol 30 % in NaCl 0,85 % 2 Minuten MATERIAL UND METHODEN 32 0.2 M HCl 20 Minuten DEPC-H20 10 Sekunden DEPC-H20 10 Sekunden 2x SSPE 20 Minuten bei 70°C 5 µg/ml Proteinase K 20 Minuten bei 37°C 2X SSPE 5 Minuten Für den nächsten Inkubationsschritt wurden die Proben in 100 mM Triethanolamine (TEA) gesetzt und tropfenweise mit Acetanhydriden (Endkonzentration 0,5 %) unter Rühren angereichert. 100 mM TEA + 0,5 % Acetanhydride 15 Minuten unter Rühren 2X SSPE kurz Spülen 2X SSPE kurz Spülen NaCl 0,85 % kurz Spülen Ethanol 30 % kurz Spülen Ethanol 50 % kurz Spülen Ethanol 70 % kurz Spülen Ethanol 85 % kurz Spülen Ethanol 95 % kurz Spülen Ethanol 100 % kurz Spülen Ethanol 100 % kurz Spülen 2.2.2.5. HYBRIDISIERUNG Die eigentliche Hybridisierungsreaktion findet im feucht-warmen Klima statt. Für die dafür benötigte Feuchtekammer wurde mit 4x SSPE-Puffer getränktes Filterpapier in eine dicht verschließbare, sterile Box gelegt. Zur Vermeidung des direkten Kontakts der Objektträger mit dem Filterpapier wurden Plastikpipetten als Unterlagen für die Objektträger genutzt. Bevor die Sonde auf die Schnitte gegeben worden ist, wurden die Objektträger in der „Humid chamber“ für 30 Minuten bei 55°C inkubiert. Für die Hybridisierung wurde die Sonde verschieden stark mit der Hybridisierungslösung verdünnt (1:100, 1:500, 1:1000). Dazu pipettierte man einen kleinen Ansatz von 50 µl, wobei die Sondenmenge jedoch vorab auf das Endvolumen von 200 µl bis 400 µl Hybridisierungslösung pro Objektträger berechnet worden ist. Der Ansatz wurde 2 Minuten bei 80°C inkubiert, anschließend für 5 Minuten auf Eis gelagert und MATERIAL UND METHODEN 33 kurz abzentrifugiert. Danach wurde der Ansatz mit der entsprechenden Menge an Hybridisierungslösung aufgefüllt und bei 55°C gelagert. Die Hybridisierungslösung ist jedes Mal möglichst frisch zusammen pipettiert worden. 2 ml Hybridisierungslösung setzen sich wie folgt zusammen: Formamide 1000 µl 50 % Dextran Sulfat 500 µl 10x Salze 250 µl Hefe-tRNA (gelöst in TE-Puffer) 25 µl 50x Dehnhardtsreagenz 50 µl DEPC-H2O 175 µl Bevor die Hybridisierungslösung blasenfrei auf die Objektträger gegeben wurde, sind kleine Deckglassplitter als Abstandhalter zwischen Objektträger und Deckglas gelegt worden, damit die Hybridisierungslösung einen gleichmäßigen Film auf den Schnitten bilden konnte. Die Gewebeschnitte inkubierten über Nacht bei 55°C in der luftdicht verschlossenen Probenkammer. Auf die gleiche Weise ist mit den Negativ-Kontrollen in einer separaten Kammer verfahren worden. 2.2.2.6. ANTIKÖRPER-BEHANDLUNG Um die Gewebeschnitte mit den an die Sonde bindenden Antikörpern behandeln zu können, mussten als erstes mit vorgewärmten 3x SSPE die Deckgläser von den Objektträger gespült werden wie auch die Hybridisierungslösung in untenstehender Reihe von den Schnitten gewaschen werden musste. Darüber hinaus wurden die frei in der Lösung vorhandenen Sonden-Fragmente durch die Behandlung mit RNase A verdaut. 3x SSPE 30 Minuten bei 45°C 3x SSPE 30 Minuten bei 45°C 3x SSPE 30 Minuten bei 45°C NTE 20 Minuten bei 37°C NTE mit 20 µg/ml RNase A 30 Minuten bei 37°C NTE 5 Minuten bei 37°C NTE 5 Minuten bei 37°C 1,5x SSPE 30 Minuten bei 52°C MATERIAL UND METHODEN 34 1x SSPE 30 Minuten bei52°C 0,4x SSPE 30 Minuten bei 52°C In den nächsten Inkubationsschritten wurden unspezifische Bindungen der restlichen vorhandenen Sonde blockiert und die Schnitte auf die eigentliche Behandlung mit den Antikörpern vorbereitet. Puffer 1 pur 5 Minuten Puffer 1 mit 0,5 % Blocking Reagenz 30 Minuten Puffer 1 mit 1% BSA und 0,1% Tween 20 30 Minuten Für die Inkubation mit den Anti-Digoxigenin-AP konjugierten Antikörpern aus dem „DIG Nucleic Acid Detection Kit“ der Firma ROCHE wurden diese 1:3000 in Puffer 1 mit 1%igem BSA verdünnt. Bei der Präparation der Objektträger und der Probenkammern wurde wie unter 2.2.2.5. verfahren, jedoch ist nun das Filterpapier für die Antikörperbehandlung mit DEPC-H2O anstelle von SSPE getränkt worden. Nach der Zugabe von je 300 µl Antikörperlösung inkubierten die Gewebeschnitte für 1 Stunde bei Raumtemperatur in den feuchten Klimakammern. 2.2.2.7. DETEKTION Bei der Dektektionsreaktion werden die Antikörper über die spezifische Bindung des Farbstoffes „SIGMA FASTTM BCIP/NBT“ (SIGMA-ALDRICH) sichtbar. Da die Antikörper wiederum an die Sonde gebunden sind, ist eine räumliche Zuordnung der Expression des mittels der Sonde gesuchten Gens möglich. Bevor jedoch der Farbstoff auf die Gewebeschnitte gegeben werden kann, müssen die unspezifisch gebundenen bzw. ungebundenen Antikörper von den Objektträgern gewaschen werden. Dazu wurden die Objektträger 4-mal hintereinander für je 20 Minuten in Puffer 1 mit 0,1% Tween 20 und anschließend einmal für 5 Minuten in Puffer 2 mit 0,1% Tween 20 bei Raumtemperatur inkubiert. Die Objektträger wurden nach Probe und Kontrolle sortiert in zwei Probenkammern (vergleiche 2.2.2.6) gegeben, mit Deckglassplittern präpariert und mit mindestens 200 µl Detektionslösung bedeckt. Die Detektionslösung besteht aus einer Tablette „SIGMA FASTTM BCIP/NBT“ (SIGMA-ALDRICH), die in 10 ml DEPC-Wasser unter ca. 2,5-stündiger Rotation bei 37°C vor Licht geschützt gelöst wird. Nach Zugabe der Detektionslösung mussten die Gewebeschnitte für 1-4 Tage vollständig abgedunkelt bei Raumtemperatur inkubieren, bis eine deutliche Farbreaktion zu erkennen war. Gegebenenfalls musste etwas Färbelösung nachgegeben werden. MATERIAL UND METHODEN 35 2.2.2.8. STOPPEN DER REAKTION Sobald eine deutliche Färbung in den Proben erkennbar war, wurde die Reaktion mit Aqua dest. abgestoppt, indem man die Deckgläschen vorsichtig mit viel Aqua dest. von den Objektträgern spülte. In Anschluss daran durchliefen die Objektträger eine aufsteigende Alkoholreihe, um anschließend mit Deckgläschen und ENTELLAN NEU (MERCK) eingedeckt zu werden. Aqua dest. 5 Minuten Ethanol 30 % 30 Sekunden Ethanol 50 % 30 Sekunden Ethanol 70 % 30 Sekunden Ethanol 85 % 30 Sekunden Ethanol 95 % 30 Sekunden Ethanol 100 % 30 Sekunden Ethanol 100 % 30 Sekunden 2.2.2.9. LICHTMIKROSKOPIE UND DOKUMENTATION Lichtmikroskopisch untersucht wurden die Dauerpräparate mit dem Lichtmikroskop Axioplan der Firma ZEISS mittels Hellfeld-Durchlicht-Mikroskopie. Die fotografische Dokumentation der Ergebnisse der In-situ-Hybridisierungen erfolgte über die daran angeschlossene Digitalkamera ColorView (SOFT IMAGING SYTEMS). Bei Objekten, die die Größe des Kameraausschnitts überschritten, wurde mit Hilfe der Programms MIA (Multiple Image Alignment) des Computerprogramms analySIS der Firma SOFT IMAGING SYSTEM DIGITAL ein Bild aus mehreren Einzelbildern zusammengesetzt. 2.2.3. LÖSUNGSLISTE Im Folgenden werden nur die Lösungen aufgelistet, auf die in den vorangegangenen Abschnitten noch nicht ausführlicher eingegangen wurde. Zusätzliche Lösungen, die in Reinform verwendet wurden, sind den Herstellernachweisen im folgenden Kapitel 2.2.4. zu entnehmen. 0.5 M EDTA 4,653g EDTA.2H2O (Titriplex) auf 25 ml DEPC-Wasser auffüllen und bei pH 12.0 lösen, pH 8.0 mit Essigsäure und NaOH einstellen MATERIAL UND METHODEN 36 LB-Amp-X-Gal-IPTG-Platten Ampicillin (20 g), X-Gal (8 g), IPTG (10 g), Agarose (28 g) mit LB-Medium, pH 7.5 auf 200 ml auffüllen LB-Medium Trypton (10 g), Hefeextrakt (5 g), NaCl (5 g) auf 1000 ml mit H2O auffüllen, pH 7.5 NTE 0.5 M NaCl, 10 mM Tris-HCl, 1mM EDTA, pH 8.0 10x PBS-Stocklösung 1.3 M NaCl, 0.07 M Na2HPO4, 0.03 M NaH2PO4 in 1 l DEPC-Wasser lösen Proteinase K Puffer 20 mM Tris-HCl, 2 mM CaCl2, pH 7.0 Puffer 1 100 mM Tris-HCl, 150 mM NaCl, pH 7.5 Puffer 2 100 mM TRIS base, 100 mM NaCl, 50 mM MgCl2, pH 9.5 (Frisch ansetzten!) 10x Salze 5 M NaCl (60 ml), 1 M Tris-HCl, pH 6.5 (10 ml), 0,5 M EDTA (10 ml), NaH2PO4.2H2O (7,8 g), Na2HPO4 (7,1 g) auf 100 ml mit DEPC-Wasser auffüllen 20x SSPE 3 M NaCl, 20 mM EDTA, 200 mM NaH2PO4.2H2O, pH 7.4 10x TBE-Puffer 890 mM Tris, 890 mM Borsäure, 20 mM EDTA TE-Puffer 10 mM Tris-HCl pH 7.5, 1 mM EDTA, pH8.0 1 M Tris-HCl pH 7.5 MATERIAL UND METHODEN 2.2.4. 37 HERSTELLERNACHWEISE Im Folgenden werden die im Rahmen des molekulargenetischen Teils dieser Arbeit verwendeten Substanzen mit den dazugehörigen Herstellern aufgelistet, sofern diese nicht bereits im Text in den Kapiteln 2.2.1. und 2.2.2. genannt wurden. Substanz Hersteller Acetanhydrid, 1 l BSA Fraktion V, 25 g CaCl2, 1kg Chloroform, 1 l Denhardt´s Lösung, 5 ml DEPC, 20 ml Dextransulfat (Natriumsalz), 25 g Eosin, 10 g Ethanol absolut, 1 l Ethanol p.a., 1 l Formaldehyd (37%), 1 l Formamid, 1 l HCl (36-38%), 1 l HCl 2M, 1 l MgCl2 . 6 H2O (MolBiol), 250 g Na2HPO4 . 2 H2O, 1kg NaCl, 1kg NaH2PO4 . H2O, 1 kg NaOH 4Mol, 250 ml NaOH-Pellets, 1 kg Paraformaldehyd 16%, 10 x 10 ml-Amp. Paraplast, 1 kg Proteinase K, 5 ml RNase A, 100 mg Titriplex (EDTA), 250 g Triethanolamine, 100 ml Tris (MolBiol), 500 g TrisHCl (MolBiol), 250 g Tween 20 (MolBiol), 250 ml Yeast tRNA, 25g Sigma-Aldrich Applichem J.T.Baker, Baker analyzed J.T.Baker, Baker analyzed Applichem Applichem Applichem Waldeck Division Chroma VWR - Normapur Merck J.T.Baker, Baker analyzed VWR - Normapur J.T.Baker, Baker analyzed Applichem Applichem J.T.Baker, Baker analyzed J.T.Baker, Baker analyzed J.T.Baker, Baker analyzed Riedel-de Haen J.T.Baker, Baker analyzed Electron Microscopy Sciences Kendall Applichem Applichem Merck Fluka/Biochemika Applichem Applichem Applichem Invitrogen MATERIAL UND METHODEN 2.3. PFLANZENMATERIAL 2.3.1. ZAMIA AMBLYPHYLLIDIA (ZAMIACEAE) 38 2.3.1.1. ALLGEMEINE ARTBESCHREIBUNG Bei der Art Zamia amblyphyllidia D.W.STEV. handelt es sich um einen Vertreter der Cycadales, eine evolutiv sehr alte Gymnospermen-Ordnung, die allgemein hin auch als Palmfarne bekannt sind. Diese Art weist wie alle anderen Vertreter der Ordnung viele Merkmale der ursprünglichen, fossilen Gymnospermen auf. Zum einen ist das die Ausbildung beweglicher Spermatozoide, die rezent neben den Cycadales nur noch bei Ginkgo biloba L. anzutreffen ist, zum anderen ist es die einfache, bei vielen Cycadales den Baumfarnen stark ähnelnde Erscheinung aus einem mit Blattbasen besetzten Stamm mit einem Blattschopf aus großen Wedeln. Dieser Habitus spiegelt sich in der deutschen Bezeichnung Palmfarne wider. Z. amblyphyllidia sieht aufgrund des kurzen, unmittelbar oberhalb der Erdoberfläche verzweigten Stamms allerdings nur begrenzt palmenartig aus. Distal an jedem Stammende steht ein Blattschopf aus maximal 15 Wedeln, die eine Länge von 150 cm erreichen können und aus bis zu 40 gleichmäßig verteilten Fiederpaaren bestehen. Die einzelnen Fiedern sind schmallanzettlichen und im distalen Viertel leicht gezähnt. Die Cycadales hatten ihren weltweiten Verbreitungshöhepunkt vor rund 150 Millionen Jahren im Mesozoikum. Rezent sind die 185 Arten der Cycadales in ihrem Vorkommen eng auf die tropischen Breiten aller Kontinente beschränkt. Die Gattung Zamia umfasst insgesamt 54 Arten, die alle in Mittelamerika zwischen Florida und Bolivien verbreitet sind. Innerhalb dieses Bereichs ist das natürliche Verbreitungsgebiet der einzelnen Art stark eingeschränkt. Z. amblyphyllidia kommt auf Jamaika, Kuba und im Norden Puerto Ricos vor. Das Verbreitungsgebiet ist mit dem von Z. angustifolia JACQ., Z. pygmea SIMS, Z. pumila L. und Z. portoricensis URBAN vergleichbar (WHITELOCK 2002). Da eine starke Ähnlichkeit dieser Arten untereinander besteht wurde Z. amblyphyllidia erstmals 1987 von STEVENSON als eigene Art beschrieben. Im rein vegetativen Zustand sind die vom Habitus her sehr ähnlichen Arten Z. pumila und Z. amblyphyllidia lediglich anhand der stärkeren Zähnung der Fiedern bei Z. pumila auseinander zu halten. Weibliche, generative Exemplare lassen sich zudem anhand der Färbung der Samen unterscheiden, die bei Z. amblyphyllidia dunkelrot und bei Z. pumila orange ist. Neben der Familie der Zamiaceae gehören auch die Cycadaceae und Stangeriaceae zu den Cycadales, die nach JONES (1993) aus der Radiation aus wenigen Reliktarten hervorgegangen sind. Die drei Familien lassen sich am einfachsten in Hinblick auf die MATERIAL UND METHODEN 39 Ausbildung ihrer Sporophylle wie folgt klassifizieren. Die Cycadaceae bilden ein Sporophyll mit einem sterilen, phylloiden Endabschnitt und mehreren seitlich ansetzenden Samenanlagen aus (Abb. 5A). Bei den Stangeriaceen ist der sterile Endabschnitt stark reduziert und die Zahl der Samenanlagen ist auf zwei festgelegt (Abb. 5B). Die Zamiaceen, deren phylloider Sporophyllabschnitt zu einem schmalen, kompakten Schild reduziert ist, haben zwei oberhalb des Sporophyllstiels inserierende Samenanlagen mit - abweichend von den anderen beiden Familien der Cycadales - zur Zapfenachse weisende Mikropylen (Abb. 5C). A Abb. 5: B C Schematische Darstellung der Sporophylltypen der Cycadales A: Cycas revoluta L., Cycadaceae - Sporophyll mit phylloidem Endabschnitt; B: Stangeria eriopus (KUNZE) BAILL., Stangeriaceae – Sporophyll mit sehr stark reduziertem phylloiden Abschnitt, der die Samenanlagen bedeckt; C: Zamia amblyphyllidia D.W.STEV., Zamiaceaea – Sporophyll mit schildartig komprimiertem Endabschnitt 2.3.1.2. BESCHREIBUNG DER UNTERSUCHTEN INDIVIDUEN Sämtliches entnommenes Pflanzenmaterial von Zamia amblyphyllidia stammt aus dem Botanischen Garten der Ruhr-Universität Bochum. Dort werden die Pflanzen in Kübelkulturen in Gewächshäusern gehalten. Im Bestand des Botanischen Gartens Bochum befinden sich sowohl mehrere weibliche als auch männliche Exemplare von Z. amblyphyllidia, einer Art, die wie alle Cycadales diözisch ist. Eine Differenzierung der Pflanzen in männliche und weibliche Individuen ist allein vom Habitus unmöglich, so dass das die Zapfen als generative Merkmale herangezogen werden müssen. Die schlanken Zapfen der männlichen Pflanzen werden bis zu 8 cm lang und haben eine zylindrische, zur Spitze hin verschmälerte, abgerundete Form. Zum Stäuben weichen die rot-braunen Sporophylle auseinander (Abb. 6A). Die viel kompakter wirkenden, zylindrischen weiblichen Zapfen werden etwa 15 cm lang und erreichen dabei einen Durchmesser von 4-6 cm. Der Apex der zur Bestäubungsreife dunkelbraunen und später eher gräulichen Zapfen ist zu einer langen sterilen Spitze ausgezogen (Abb. 6B,C). Die einzelnen Sporophylle, die nur zum Zweck der Bestäubung kurz unterhalb MATERIAL UND METHODEN 40 des Apex auseinander weichen (Abb. 6B), stehen während der gesamten Samenentwicklung extrem kompakt. Sie werden erst durch die Größenzunahme der Samen auseinander gedrängt und geben den Blick auf die rot gefärbte Sarkotesta der Samen frei (Abb. 6C). A B C Abb. 6: Zapfen von Zamia amblyphyllidia D.W.STEV. A: männlicher Zapfen zum Zeitpunkt des Stäubens; B: Bestäubungsreifer weiblicher Zapfen mit ringförmig auseinander gewichenen Sporophyllen; C: Weiblicher Zapfen mit reifen Samen. Da im botanischen Garten Bochum mehrere weibliche Exemplare vorhanden sind, wurde abhängig von der gewünschten Zapfengröße von unterschiedlichen Pflanzen gesammelt, die in Größe und Alter dem in Abbildung 7 gezeigten Exemplar entsprechen. Für die Erstellung einer lückenlosen Entwicklungsreihe wurde auf fixiertes Material zurückgegriffen, das schon in den Jahren 1998, 2000, 2002 und 2003 gesammelt worden war. Die Materialsammlung für die In-situ-Hybridisierung erfolgte anders als zur Erstellung einer Entwicklungsreihe punktuell. So wurde im Juli 2005 ein kaum differenzierter Zapfen aus einem Vegetationskegel herauspräpariert. Im Mai und November 2006 wurden je zwei Zapfen unterschiedlichen Alters mit deutlich entwickelten Sporophyllen entnommen. In 2007 erfolgte im Februar neben der Sammlung eines Abb. 7: Zamia amblyphyllidia D.W. ganz jungen, gerade ausdifferenzierten Zapfens STEV., BoGa Bochum auch die Entnahme junger Blätter und Fiedern für die In-situ-Hybridisierung. Für die RNA-Isolierungen wurden aus den Zapfen frei präparierte Samenanlagen verwendet, die direkt nach den jeweiligen Entnahmen im Februar, Juli und November 2005 verarbeitet wurden. MATERIAL UND METHODEN 2.3.2. 41 GINKGO BILOBA (GINKGOACEAE) 2.3.2.1. ALLGEMEINE ARTBESCHREIBUNG Ginkgo biloba L. ist die einzige rezent vorkommende Art der monotypischen Familie der Ginkgoaceae. Es handelt sich um einen sommergrünen Baum mit monopodialem Wuchs, der eine Höhe von 40 m erreicht (SCHÜTT et al. 2002). Im späten Frühjahr treiben kräftig grün gefärbte Blätter aus, die sich im Verlauf des Sommers grüngräulich verfärben und im Herbst leuchtend gold-gelb erscheinen. Das Artepitheton „biloba“ leitet sich von der Form der Blätter ab, da die Blätter oftmals durch eine tiefe Einbuchtung in der Mitte des oberen Spreitenrandes „zweilappig“ sind (Abb. 8A). Neben diesen „biloben“ Blättern sind an jedem Baum gleichzeitig immer zahlreiche fächerförmige Blätter ohne eine derartige Zweiteilung vorhanden (Abb. 8A). Sie alle verfügen über eine Gabelnervatur. Die Gabelnervatur, bei der sich alle Blattgefäße dichotom verzweigen, ist unter den rezenten Pflanzen nicht weit verbreitet. An den Langtrieben stehen die Blätter spiralig angeordnet, während sie an den Kurztrieben aufgrund der stark gestauchten Internodien gebüschelt wirken (Abb. 8A). An den Kurztrieben der diözischen Ginkgo-Bäume sind entweder die männlichen oder die weiblichen reproduktiven Strukturen zu finden. Die männlichen Individuen bilden lockere Kätzchen aus, die den Kätzchen von Weiden ähnlich sind (Abb. 8B). Bei den Weibchen stehen an den Kurztrieben bis zu einem Dutzend Samenanlagen tragende Strukturen, die jeweils aus einem Träger und meist zwei Samenanlagen bestehen. In den meisten Fällen gelangt nur eine der beiden Samenanlagen zur Reife (Abb. 8C). Das Integument reifer Samen entwickelt sich zu einer inneren stark verholzten Schicht und einer äußeren Schicht, die eine gelbliche, fleischige Hülle mit großen Mengen an Buttersäure bildet. Dieser Buttersäureanteil führt beim Faulen der Samen zu einem extrem unangenehmen Geruch, der an ranzige Butter erinnert. A B C Abb. 8: Kurztriebe bei Ginkgo biloba L. A: Vegetativer Kurztrieb mit gabelnervigen Blättern; B: Männlicher Kurztrieb mit sog. „Kätzchen“; C: Weiblicher Kurztrieb mit Sporangiophoren. MATERIAL UND METHODEN 42 Aufgrund seiner in Form und Farbe besonderen Blätter und des sehr hohen Alters, das einzelne Bäume erreichen können, gelten Ginkgo-Bäume in ganz Asien als heilig. In China soll es Bäume geben, die fast 4000 Jahre alt sind. Sie sind mit einem Stammumfang von annähernd 17 m zu imposanten Bäumen gewachsen. Eine weitere interessante Erscheinung, die an den sehr alten Ginkgo-Exemplaren zu sehen ist, sind die so genannten „Tschitschi“ (Abb. 9). Als „Tschitschi“ werden die verholzten, zylindrischen Auswüchse an den Astunterseiten bezeichnet. Diese AusAbb. 9: Ginkgo biloba L., Ast mit wüchse wachsen positiv geotrop Richtung Erde, „Tschitschi“, abgelichtet am Meji-Schrein (Japan) wo sie wurzeln und neue Sprosse bilden können (FUJI 1998). Nicht weniger interessant als das Alter einzelner Individuen ist das erdgeschichtliche Alter der Gattung Ginkgo. Ginkgo biloba ist der einzige bis heute überdauernde Vertreter der Ginkgoales, die im Mesozoikum mit rund 20 Gattungen eine große Formenvielfalt ausgebildet hatten. Funde aus dem Jura belegen, dass der rezente Ginkgo seit ca. 150 Millionen Jahren nahezu unverändert ist. Daher hat DARWIN den für die Wissenschaft so interessanten Baum als „lebendes Fossil“ bezeichnet. Das weltweite Verbreitungsgebiet der Ginkgoales während ihrer Blütezeit im Mesozoikum, ist auf ein natürliches Vorkommen im östlichen China reduziert worden. Von dort aus wurde der Ginkgo-Baum im 11. Jahrhundert n. Chr., der Blütezeit des Buddhismus, in Japan, Korea und Nordchina eingeführt und kultiviert. Die erste Artbeschreibung erstellte KAEMPFER 1712. 1730 wurde dann das erste Exemplar von Ginkgo biloba auf den europäischen Kontinent nach Utrecht gebracht. Von dort aus erfolgte, insbesondere durch die Kultivierung in Botanischen Gärten, eine rasche Ausbreitung innerhalb ganz Europas. Heute ist der Ginkgo ein häufig angepflanzter Straßenbaum, da er nicht nur gegen Insekten- und Pilzbefall sondern auch gegen die starke Luftverschmutzung resistent ist (SCHULTZEMOTEL 1992). 2.3.2.2. BESCHREIBUNG DES UNTERSUCHTEN INDIVIDUUMS Die im Botanischen Garten der Ruhr-Universität Bochum angepflanzten Exemplare von Ginkgo biloba sind erst rund 25 Jahren alt und haben die Geschlechtsreife noch nicht erreicht, so dass diese Bäume bislang nicht als Weibchen oder Männchen identifiziert werden konnten. Aus diesem Grunde bilden sie bislang auch noch keine MATERIAL UND METHODEN 43 generativen Strukturen aus, die jedoch insbesondere für die im Rahmen dieser Arbeit untersuchte Fragestellung von Interesse sind. Daher stammt sämtliches verwendetes Material von einem ca. 80 Jahre alten weiblichen Exemplar von einem Privatanwesen in Herdecke (Abb. 10). Das Material von dort wurde sowohl für die licht- als auch für die rasterelektronenmikroskopischen Untersuchungen von Dezember 2002 bis Juni 2003, im Abstand von 14 Tagen gesammelt und fixiert, um die Anlegung und die Entwicklung der Integumente vollständig dokumentieren zu können. Während der Winterruhe erfolgte die reduzierte Materialsammlung in einer vierwöchigen Sammelfrequenz. Im Frühling 2007 erfolgten weitere Materialentnahmen mit unmittelbar anschließender RNasefreier Fixierung für noch ausstehende In-situ-Hybridisierungen. Für die dafür vorab notwendigen Vorarbeiten wurde gleichzeitig Material zur RNA-Isolierung schockAbb. 10: Ginkgo biloba L., Privatanwesen in Herdecke gefroren und im Labor aufgearbeitet. 2.3.3. MAGNOLIA STELLATA (MAGNOLIACEAE) 2.3.3.1. ALLGEMEINE ARTBESCHREIBUNG Magnolia stellata (SIEBOLD & ZUCC.) MAXIM gehört zur Familie der Magnoliaceae, die innerhalb der Magnoliopsida der Unterklasse der Magnoliidea angehört, die ausschließlich größere Holzpflanzen umfasst. Die Magnoliopsida stellen zusammen mit den Liliopsida und Asteriopsida die drei Klassen der Angiospermen dar. Letztere Klasse grenzt sich durch das Vorhandensein von tricolpaten Pollen von den beiden erstgenannten Klassen mit monosulcaten Pollen ab. Die laut kladistischen Analysen den Magnoliopsida nah stehenden Liliopsida lassen sich aufgrund ihrer Einkeimblättrigkeit aus den dikotylen Magnoliopsida ausgliedern. Zu der M. stellata beinhaltenden Ordnung Magnoliales zählen die in den Tropen verbreiteten Familien der Degeneriacaeae, Himantandraceae, Annonaceae und Myristicaeae. Das natürliche Verbreitungsgebiet der Magnoliaceae erstreckt sich reliktär über die Nordhemisphäre von den Tropen bis in die warm-temperierten Zonen (BRESINSKY 2002). So gibt es rezent ca. 80 Arten, die in Ost-Asien, Nord- und Mittel-Amerika und im Himalaja beheimatet sind (KRÜSSMANN 1976). Bei den bei uns in den gemäßigten Breiten in großer Vielfalt MATERIAL UND METHODEN 44 anzutreffenden Magnolien handelt es sich um Anpflanzungen. Den Verbreitungshöhepunkt hatten die Magnoliaceen im Tertiär mit einem Vorkommen auf der gesamten Nordhalbkugel. Dabei zeigten sie ein typisch arkto-tertiäres Verbreitungsmuster, das durch die Wanderung der Gletscher und die Kontinetaldrift hervorgerufen worden ist (WEN 2001). Allerdings stammen zahlreiche fossile Belege bereits aus der Kreide, was ein Indiz für das hohe erdgeschichtliche Alter dieser Familie ist (KRUSE 1992). M. stellata ist ein häufig angepflanztes, langsam wachsendes Ziergehölz mit strauchförmigem Wuchs, das in unseren Breiten meist die maximale Höhe von 8 m erreicht. Aufgrund seiner kräftigen, weißen Blüten, die sich vor dem Austrieb der Laubblätter öffnen, erfreut sich M. stellata bei uns immer größerer Beliebtheit. Die im Durchmesser ca. 7-8 cm großen, aus 12-15 schmalen Petalen zusammengesetzten Blüten werden durch dicht behaarte Knospenschuppen vor der Winterkälte geschützt bevor sie sich im zeitigen Frühjahr öffnen und den Blick auf die zahlreichen Staubgefäße und Fruchtblätter freigeben (Abb. 11A-C). Die 4-10 cm langen und 3-5 cm breiten Laubblätter von M. stellata sind verkehrt-eiförmig mit keilförmiger Basis und einer abgerundeten bzw. stumpfen Spitze (KRÜSSMANN 1976). A B C Abb. 11: Blüten von Magnolia stellata (SIEBOLD & ZUCC.) MAXIM A: Blüte kurz vorm Öffnen, umhüllt von den stark behaarten Knospenschuppen, die bereits geöffnet sind; B: geöffnete ungegliederte Blütenhülle in lateraler Ansicht; C: Blick in die Blütenmitte mit Staubgefäßen und den zentralen Fruchtblättern, von denen nur die Narben zu erkennen sind. Die Fruchtblätter sind spiralig um die säulenförmig ausgezogene Blütenachse, die so genannte Columella, angeordnet und bilden pro Karpell je zwei Samenanlagen aus, von denen oftmals eine oder auch beide Samenanlage unbefruchtet bleiben und sich somit auch nicht weiter entwickeln (Abb. 12A). Während des weiteren Wachstums platten sich die Samenanlagen gegeneinander ab, so dass sie im reifen Zustand halbkugelförmig erscheinen (Abb. 12B). Die reife Frucht ist eine verholzte Sammelfrucht, deren einzelne Karpelle sich dorsizid öffnen. Aus den geöffneten Karpellen hängen die reifen Samen zur besseren Präsentation an den Wandverstärkungen der MATERIAL UND METHODEN 45 Schraubentracheiden des vertrockneten Funiculs heraus, die sich unter dem Gewicht der Samen entspiralisieren (Abb. 12C, Pfeil). A B C Abb.12: Karpell-/Samenentwicklung bei Magnolia stellata (SIEBOLD & ZUCC.) MAXIM A: Gynoceum mit unbefruchteten und stark angeschwollenen befruchteten Karpellen, Tepalen und Stamina bereits abgefallen; B: zwei gegeneinander abgeplattete Samen im am Rücken aufgeplatzten Karpell; C: Fruchtstand mit reifen Samen, die teilweise schon an einem dünnen Faden heraushängen (Pfeil). 2.3.3.2. BESCHREIBUNG DES UNTERSUCHTEN INDIVIDUUMS Sämtliches Pflanzenmaterial von Magnolia stellata stammt von einem einzigen etwa 3-4 m hohen Exemplar aus dem Botanischen Garten der Ruhr-Universität Bochum, das in Abbildung 13 gezeigt ist. Bei den gesammelten Pflanzenteilen handelt es sich um Blütenknospen, die sowohl für die licht- als auch rasterelektronenmikroskopische Untersuchung entnommen wurden. Die Materialentnahme erfolgte kontinuierlich von August 2005 bis Mai 2006 mit einer von der Witterung und Vegetationsphase abhängigen Sammelfrequenz von zwei bis vier Wochen. In unregelmäßigen Abständen ist zudem auch an einigen Sammeldaten Material Abb. 13: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM in Blüte, BoGa Bochum für noch ausstehende In-situ-Hybridisierungen RNasefrei gesammelt und fixiert worden. Die Sammlung der reifen Samen für die morphologischen Untersuchungen erfolgte gegen Ende September 2007. Ergänzt wurde die Sammlung durch Material von einem 8 m hohen, ungewöhnlich gut tragenden Exemplar von M. stellata, das im Stadtpark Bochum angepflanzt ist. ERGEBNISSE 3. ERGEBNISSE 3.1. HISTOLOGISCHE UNTERSUCHUNGEN 3.1.1. INTEGUMENTENTWICKLUNG BEI ZAMIA AMBLYPHYLLIDIA 46 Die im Folgenden aufgezeigte Entwicklungsreihe zur Integumentbildung beginnt mit sehr jungen Sporophyllen von gerade einmal 1,2 mm Länge und 0,8 mm Breite, die an einem 12 mm langen und 6 mm breiten Zapfen stehen. Da die sehr jungen Zapfen noch von den Blattbasen der Wedel verborgen sind, mussten sowohl die alten als auch die sehr jungen, noch eingerollten Wedel vor der Materialentnahme entfernt werden. Oftmals stirbt nach dieser Prozedur der ganze betroffene Seitenast ab. Bei diesen jungen Sporophyllen ist bereits beidseits des Stiels je ein SamenanlagenPrimordium zu sehen (Abb. 14A). Die undifferenzierten Samenanlagen zeigen in der distalen Hälfte, die der Zapfenachse zugewandt ist, ringsum die Makrosporenmutterzelle einen zentral gelegenen Bereich von erhöhter Zellteilungsaktivität. Dies ist an der hohen Dichte des Cytoplasmas und des Fehlens einer Vakuole in diesen Zellen erkennbar (Abb. 14B). Bei nur geringfügig weiter entwickelten Sporophyllen, die auf dieselbe aufwändige Weise präpariert worden sind, ist in den histologischen Untersuchungen im Längsschnitt zu erkennen, dass sich das Primordium in dieser Zone in Integument und Nucellus differenziert, wobei der Nucellus reicher an Plasma erscheint als das umgebende Integument (Abb. 14D). Die Entwicklung des Integuments beginnt damit, dass auf der distalen Fläche der Samenanlage eine ringförmige Zone im Wachstum zurückbleibt. In der Aufsicht auf das Primordium ist diese Zone als ringförmige Einbuchtung zu erkennen (Abb. 14C). Auch im Längsschnitt ist diese Einbuchtung zu erahnen (Abb. 14D, Pfeile). Bei den in etwa dem gleichen Zeitraum desselben Jahres gesammelten Sporophyllen lässt sich bereits deutlich zwischen Integument und Nucellus differenzieren. In diesem Stadium ist bereits eine Mikropyle in Form einer kreisrunden Öffnung auf der der Zapfenachse zugewandten Seite der Samenanlage erkennbar. Sie misst annähernd 100 µm, was etwa ein Sechstel der Breite der Samenanlage ausmacht, und lässt den Blick auf den Nucellus frei (Abb. 14E). Integument und Nucellus zeigen ein Längenwachstum oberhalb der Zone, in der beide Strukturen kongenital verwachsen sind. Der Bereich, in dem das Integument nun frei ist, macht etwa ein Fünftel der Länge der gesamten Samenanlage aus (Abb. 14F). Die Differenzierung des Primordiums in Integument und Nucellus setzt sich ebenso in basaler Richtung als histologische Differenzierung fort, die der im freien Abschnitt des Integuments gleicht. Durch die beschriebenen ERGEBNISSE 47 Differenzierungsvorgänge ist nun über die ganze Samenanlage von der Mikropyle bis zum Funiculus eine Unterscheidung zwischen Nucellus und Integument problemlos möglich. Zunächst entwickeln sich Nucellus und Integument in gleichem Maße, so dass bei einer weiteren Größenzunahme keine wesentlichen Verschiebungen der Größenverhältnisse innerhalb der Samenanlage zu verzeichnen sind (Abb. 15A, B). Erst später findet das Längenwachstum der Samenanlage überwiegend im distalen Bereich der Samenanlage statt, dort wo das Integument frei ist. Aus dieser Wachstumsverlagerung resultiert, dass der Nucellus nur noch bis etwa zur Hälfte mit dem Integument verwachsen ist, bevor die Entwicklung des Makroprothalliums beginnt. Durch ein vorangegangenes verstärktes Streckungswachstum des Integuments im Vergleich zum Nucellus umschließt das Integument den Nucellus nun vollständig (Abb. 15C). Die Mikropyle, die ihre Größe nicht wesentlich verändert hat, macht aufgrund der starken Größenzunahme der gesamten Samenanlage auf 12 mm in der Breite nun weniger als ein Zehntel der distalen Fläche der gesamten Samenanlage aus (Abb. 15D). In diesem Stadium bildet das Integument am Mikropylenrand unregelmäßige Zipfel aus, die aufeinander zu wachsen, bevor sie - wie später noch beschrieben - zu einer röhrenförmigen Struktur auswachsen, die den Bestäubungstropfen hält. Durch diese Zipfel wird die Mikropyle zwischenzeitlich verengt (Abb. 15D,F). Zeitgleich mit den bisher beschriebenen Entwicklungsvorgängen nimmt das Integument an Mächtigkeit zu. Über der Nucellusspitze bleibt durch die 200µm dicke Zellschicht des Integuments ein Kanal als Mikropyle frei (Abb. 15E, F). Dieses Stadium ist Ende Oktober erreicht. Zu diesem Zeitpunkt setzt die weitere Differenzierung innerhalb des Nucellus mit einer Größenzunahme der Makrospore ein (Abb. 15E). Bis Anfang Dezember zieht sich das Integument rund um die Mikropyle zu einer kleinen Spitze aus (Abb. 16A, B), die sich in der letzten Januarhälfte zu einer Röhre ausformt (Abb. 16C). Parallel dazu wächst der Nucellus mit der Spitze in die Mikropyle ein (Abb. 16A, C). Zu diesem Zeitpunkt beginnt die Alveolenbildung, d.h. durch die Bildung von Zellwänden entwickelt sich das vielkernige Makroprothallium zu einem vielzelligen Gewebe (Abb. 16E). Vor der Alveolenbildung liegen die Zellkerne von zwölf simultanen Kernteilungen frei innerhalb der Wand der ursprünglichen Makrospore vor (Makrosporenwand ohne Zellkerne, Abb. 16F). Ab Mitte Februar ist im Nucellus im Bereich zwischen Makroprothallium und Mikropyle unmittelbar unterhalb letzterer die Bestäubungskammer ausgebildet (Abb. 17A). Nach der Bestäubung wachsen einige Zellen des inneren Mikropylenrandes in die Mikropyle ein und verschließen diese dadurch (Abb. 17B, Pfeil). In diesem Stadium sind bereits die im Längsschnitt deutlich erkennbaren Archegonien im Makroprothallium entwickelt (Abb. 17C). ERGEBNISSE 48 Zeitgleich mit der Ausbildung der ausgezogenen Mikropyle Ende Oktober lässt sich eine Differenzierung des Integuments in zwei Schichten erkennen (Abb. 17D). Die Zellen der äußeren Schicht sind sehr cytoplasmareich, wohingegen die Zellen der inneren Schicht stark vakuolisiert sind. Der äußerste Zipfel der Mikropyle wird fast vollständig von der äußeren Schicht des Integuments gebildet, während die innere Integumentschicht im basalen Bereich der Samenanlage die deutlich mächtigere ist (Abb. 17E). In der Übergangszone zwischen den Schichten verläuft bei den älteren Stadien eine dünne Schicht stark cytoplasmahaltiger Zellen (Abb. 17D, Pfeil). Diese Schicht wird sich zum Sklerenchym entwickeln. Die innerste Integumentschicht, die sich nicht bis in den Mikropylenbreich erstreckt (siehe Abb. 17A), besteht aus stark vakuolisierte Zellen. Sie ist histologisch mit dem Nucellus vergleichbar und wird wie dieser ebenfalls als Nährschicht für das wachsende Makroprothallium aufgebraucht. Die äußerste Schicht des Integuments entwickelt sich bei Reifung der Samenanlage zu einer fleischigen Sarkotesta. Rund um die Samenanlage ziehen sich Leitbündel vom Funiculus bis zur Mikropyle durch die außen liegende Schicht des Integuments. Die Leitbündel verzweigen sich aus einem Strang, der aus dem Stiel des Sporophylls im Bogen zur Samenanlage zieht (Abb. 17E). Im Bereich des Funiculus sind die Leitbündel noch sehr kräftig und werden in Richtung Mikropyle immer schwächer. Bei den Leitbündeln handelt es sich um den so genannten kollateral geschlossenen Leitbündel-Typ, bei dem das Xylem und das Phloem direkt aneinandergrenzen. Wie üblich ist das Xylem nach innen und das Phloem nach außen orientiert. An letzteres schließt zum Rand der Samenanlage hin eine Schicht von parenchymatischen Zellen an (Abb. 17F). ERGEBNISSE Abb. 14: Zamia amblyphyllidia D.W.STEV., REM- und LM-Aufnahmen A: Primordium einer Samenanlage Mitte Juni; B: Quergeschnittenes Primordium Anfang Juli; C: Samenanlagenprimordium Ende Juli; D: Längsgeschnittenes Primordium Ende Juli; E: Samenanlage Anfang August; F: Längsschnitt durch Samenanlage Anfang August (I = Integument; Mi = Mikropyle; Mz = Makrosporenmutterzelle; Nu = Nucellus; PrSa = Primordium Samenanlage; St = Stiel) 49 ERGEBNISSE Abb. 15: Zamia amblyphyllidia D.W.STEV., LM- und REM-Aufnahmen A: Schwach entwickelte Samenanlage Ende Oktober im Längsschnitt; B: Samenanlage Ende Oktober (schwach entwickelt) in der Aufsicht; C: Längsgeschnittene Samenanlage Ende Oktober; D: Aufsicht auf Samenanlage Ende Oktober; E: Längsschnitt durch weit entwickelte Samenanlage Ende Oktober; F: Detail Mikropyle einer weit entwickelten Samenanlage Ende Oktober (I = Integument; Mi = Mikropyle; Ms = Makrospore; Nu = Nucellus; Sa = Samenanlage; St = Stiel) 50 ERGEBNISSE Abb. 16: Zamia amblyphyllidia D.W.STEV., LM-, REM- und Binokular-Aufnahmen A: Längsgeschnittene Samenanlage Anfang Dezember; B: Mikropyle im Detail Anfang Dezember; C: Längsschnitt durch Samenanlage Mitte Januar; D: Detail Mikropyle Anfang Januar; E: Detail Alveolenbildung im Makroprothallium Ende Januar, Pfeil auf Mikrotubuli zwischen den Zellkernen; F: frei präparierte, mit Safranin angefärbte Makrosporenwand ohne Zellkerne Mitte Januar (I = Integument; Mi = Mikropyle; Mp = Makroprothallium; Ms = Makrospore; Nu = Nucellus; Zk = Zellkern) 51 ERGEBNISSE Abb. 17: Zamia amblyphyllidia D.W.STEV., LM-Aufnahmen A: Längsschnitt Samenanlage Mitte Februar, Pfeil auf Bestäubungskammer; B: Mikropyle Mitte März im Längsschnitt; C: Längsgeschnittene Samenanlage Mitte März; D: Detail Integument Mitte März, Pfeil auf dünne Sklerenchymschicht; E: jüngere Sporophyllhälfte im Längsschnitt; F: Leitbündel im Detail (A = Archegonium, Bk = Bestäubungskammer; Hk = Harzkanal; I = Integument; Lb = Leitbündel; Mp = Makroprothallium; Ms = Makrospore; Nu = Nucellus; Ph = Phloem; pZ = parenchymatische Zellen; St = Stiel; Xy = Xylem) 52 ERGEBNISSE 3.1.2. 53 INTEGUMENTENTWICKLUNG BEI GINKGO BILOBA Anfang Dezember, bevor die kältebedingte Ruhephase die Entwicklung der Samenanlagen unterbricht, hat die Differenzierung des Integuments noch nicht begonnen. Die Primordien der Samenanlagen am distalen Ende der Sporangiophoren sind aber bereits in der aufpräparierten Knospe zu erkennen (Abb. 18A). Die noch vollkommen undifferenzierten Primordien erscheinen in der Frontalansicht annähernd rund (Abb. 18B). In der Aufsicht auf den Vegetationskegel erkennt man jedoch, dass sie bedingt durch die Platzverhältnisse innerhalb der Knospe seitlich abgeplattet sind (Abb. 18A). Im Zuge der fortschreitenden Entwicklung wächst verstärkt die vom Vegetationskegel abgewandte Fläche, so dass sich in der Aufsicht eine mehr oder weniger dreieckige Form des Primordiums ergibt. Im Februar beginnt an der zum Knospenrand weisenden Fläche des Primordiums die Differenzierung in Integument und Nucellus (Abb. 18C, Pfeil) mit einer kreisförmigen Zone, die im Wachstum zurückbleibt (Abb. 18D, E). Dadurch entwickelt sich bis Ende Februar eine tiefe Kerbe, die den Nucellus vom Integument trennt (Abb. 18F). In der weiteren Entwicklung strecken sich Integument und Nucellus und die Samenanlage nimmt zeitgleich dazu an Umfang zu, so dass die Mikropyle in Relation zur gesamten Samenanlage kleiner wird. Ende Februar nimmt die Mikropyle in etwa die Hälfte der distalen Fläche der Samenanlage ein (Abb. 19A). Durch das weitere Wachstum der Samenanlage erscheint die Mikropyle bei den am weitesten entwickelten Sporangiophoren innerhalb der Knospe von Ende März nur noch wie eine winzige Öffnung, obwohl die Breite der Mikropyle seit der Ausdifferenzierung im Februar unverändert etwas mehr als 100 µm beträgt (Abb. 19B). Im eben beschriebenen Stadium Ende März bildet sich von der Zone ausgehend, in der die Samenanlagen am distalen Ende des Sporangiophors aneinandergrenzen, ein ringförmiger Wulst (Abb. 19C). Dieser Wulst verdickt sich während der folgenden Wochen und umwächst die Basis der Samenanlage vollständig (Abb. 20A). Er ähnelt rein histologisch eher dem Stiel des Sporangiophors als dem Integument (Abb. 20B). Im weiteren Wachstum der Samenanlage verändert die Mikropyle ihre anfänglich kreisrunde Form, da das Wachstum des Integuments unregelmäßig verläuft. Durch ein verstärktes Wachstum an den seitlichen Rändern der Mikropyle ergibt sich oft eine zweilippige Form (Abb. 19D). Wachsen die Ränder noch weiter aufeinander zu, entsteht eine schlitzförmige Mikropyle (Abb. 19E). Die Ausformung der Mikropyle als ein zweilippiges Integument ist nur eine Entwicklungsmöglichkeit. Häufig entstehen durch das Wachstum entlang des gesamten Integumentrandes oder das Aufeinanderzuwachsen mehrerer Lappen unregelmäßig geformte Mikropylen (Abb. 19F). ERGEBNISSE 54 Ende April, wenn sich die Knospe öffnet, ist das Integument um die Mikropyle ein kleines Stück weit ausgezogen (Abb. 20A). Dadurch entsteht ein Freiraum zwischen dem Integument und dem distalen Ende des Nucellus (Abb. 20B). Im Zentrum des Nucellus ist bereits die Makrosporenmutterzelle zu sehen. Der Bereich des Nucellus, der sich distal der Makrosporenmutterzelle erstreckt, ist nicht mit dem Integument verwachsen. Die Verwachsungszone beider Strukturen macht in diesem Stadium ca. zwei Drittel der Länge der gesamten Samenanlage aus. Anfang Mai ist das Integument zu einem langen Zipfel ausgezogen (Abb. 20C). Der Nucellus hat distal eine Spitze ausgebildet, die in den entstandenen Zwischenraum eingewachsen ist. Im Bereich der ausgezogenen Nucellusspitze ist die Bestäubungskammer zu finden, die in die Mikropyle hineinragt (Abb. 20D). Im basalen Abschnitt des Nucellus entwickelt sich das Makroprothallium. Durch eine deutliche Streckung der Samenanlage distal des Makroprothalliums sind Integument und Nucellus nur noch in der basalen Hälfte der Samenanlage miteinander verwachsen. Zeitgleich zur Ausbildung der Makrospore ist auch die gesamte basale Region gewachsen. In den folgenden Wochen wächst die Spitze des Integuments bei einer Größenzunahme der gesamten Samenanlage nicht weiter aus, jedoch verdickt sich der Rand des Integuments rund um die Mikropyle (Abb. 20E). Darüber hinaus ist der Integumentrand leicht nach außen gebogen, wie auch im histologischen Längsschnitt zu sehen ist (Abb. 20F). Durch diese trichterförmige Ausgestaltung kann die Mikropyle Bestäubungstropfen tragen, die viel größer sind als der Durchmesser der Mikropyle (Abb. 21A). Im Laufe des Reifungsprozesses verschließt sich die Mikropyle und die ausgezogene Spitze des Integuments ist durch die auf die anderen Bereiche beschränkte Dickenzunahme am reifen Samen nicht mehr deutlich auszumachen und nur aufgrund einer Einbuchtung im fleischigen Integument erkennbar (Abb. 21B). In etwa zeitgleich mit der Ausbildung der Bestäubungskammer Anfang Mai zeigt sich eine Differenzierung des Integuments in zwei Schichten (Abb. 20D, 21C). Die innere Schicht besteht aus stärker vakuolisierten Zellen, die Zellen der äußeren Schicht sind cytoplasmareicher. Die äußere Schicht, die allein die Mikropyle formt, ist die mächtigere Schicht. Durch diese ziehen sich zahlreiche Harzkanäle rings um den Nucellus. Einen Monat später ist von der inneren Schicht nichts mehr erkennbar, wohingegen die äußere Schicht mächtiger geworden ist. Ebenso sind die Harzkanäle deutlich großlumiger als noch im Mai (Abb. 21D). Leitbündel sind im Integument weder in den Längs- noch in den Querschnitten zu erkennen. Die Versorgung der Samenanlagen erfolgt durch eines der zwei kräftigen Leitbündel, die sich durch den Stiel des Sporangiophors zur Basis je einer Samenanlage ziehen (Abb. 21E). Von außen sind dem Integument kristalline Feinstrukturen aufgelagert (Abb. 21F). ERGEBNISSE Abb. 18: Ginkgo biloba L., REM-Aufnahmen A: Spiralige Anlegung der Blätter und Sporangiophore am frei präparierten Vegetationskegel Anfang Dezember; B: Sporangiophor Anfang Dezember, in der Achsel eines abpräparierten Blattes; C: Sporangiophor mit beginnender Differenzierung des Samenanlagenprimordiums (Pfeil) Mitte Februar; D: Detailausschnitt der Differenzierungszone eines Samenanlagenprimordiums Mitte Februar; E: Weiter entwickeltes Samenanlagenprimordium Mitte Februar; F: Mikropyle Ende Februar im Detail (B = Blatt; I = Integument; Nu = Nucellus; PrSa = Primordium Samenanlage; Sph = Sporangiophor; St = Stiel) 55 ERGEBNISSE Abb. 19: Ginkgo biloba L., REM-Aufnahmen A: Aufsicht auf Samenanlagen Anfang März; B: Aufsicht auf Samenanlagen Ende März; C: Samenanlagen Ende März, Pfeil auf ringförmige Wulst an der Basis der Samenanlage; D: Samenanlage mit zweilippigem Integument Ende März; E: Schlitzförmige Mikropyle einer Samenanlage Anfang April im Detail; F: Detailbild einer unregelmäßig geformten Mikropyle Anfang April (I = Integument; Mi = Mirkopyle; Nu = Nucellus) 56 ERGEBNISSE Abb. 20: Ginkgo biloba L., REM-und LM-Aufnahmen A: Samenanlage Ende April; B: Längsschnitt durch Samenanlage Ende April; C: Samenanlage Anfang Mai; D: Samenanlage von Anfang Mai im Längsschnitt, Pfeil weist auf (artfremden?) Pollen in der Bestäubungskammer; E: Trichterförmige Mikropyle Ende Mai im Detail; F: Längsschnitt durch Samenanlage Ende Mai (Bk = Bestäubungskammer; I = Integument; Mi = Mikropyle; Ms = Makrospore; Nu = Nucellus; Sa = Samenanlage; W = Wulst) 57 ERGEBNISSE Abb. 21: Ginkgo biloba L., Fotografien, LM- und REM-Aufnahmen A: Seitenansicht einer Samenanlage mit Bestäubungstropfen; B: Detailaufnahme einer zugewachsenen Mikropyle bei einer reifenden Samenanlage; C: Detail des Integuments einer längsgeschnittenen Samenanlage Anfang Mai; zwischen den Pfeilen jeweils eine Integumentschicht; D: Längsschnitt durch eine Samenanlage Anfang Juni; E: Längsgeschnittenes Sporangiophor Ende April, Pfeile auf Leitbündel; F: Kristalline Strukturen auf der Epidermis des Integuments (Hk = Harzkanal; Mi = Mikropyle; Nu = Nucellus; Sa = Samenanlage; St = Stiel; W = Wulst) 58 ERGEBNISSE 3.1.3. 59 INTEGUMENTENTWICKLUNG BEI MAGNOLIA STELLATA Die Entwicklung der Samenanlage beginnt gegen Ende des Sommers. Die gesamte Blüte ist zu diesem Zeitpunkt in einer etwa 5 mm langen endständigen Blütenknospe verborgen. Sie öffnet sich erst im Frühling des Folgejahres. Die Abbildung 22A zeigt die generativen Strukturen einer Blüte von Ende August im Längsschnitt. Es wurden alle Knospenschuppen und Blütenblätter wie auch die basal stehenden Staubblätter abpräpariert. Anhand der restlichen Stamen ist zu erkennen, dass diese aus sehr kurzen Filamenten und extrem lang gestreckten Antheren von ca. 2,5 mm Länge in diesem jungen Stadium aufgebaut sind. Der Querschnitt durch eine Anthere macht sichtbar, dass die Antheren aus zwei Theken bestehen, die durch ein Konnektiv verbunden werden. Jede Theke setzt sich aus zwei Pollensäcken zusammen (Abb. 22B). Im abgebildeten Stadium sind die Pollensäcke erst 100 µm breit und enthalten noch keinen Pollen, sondern die Mirkosporenmutterzellen, aus denen sich je vier Pollenkörner entwickeln werden. In Abbildung 22B ist ein längsgeschnittenes Karpell von Ende August mit einem noch völlig undifferenzierten Samenanlagenprimordium gezeigt (Abb. 22C). Auch anhand der rasterelektronischen Bilder zu freipräparierten geringfügig älteren Primordien der Samenanlagen von Anfang September ist sowohl in der Aufsicht wie auch anhand der Detailaufnahme keinerlei Differenzierung zu erkennen (Abb. 22D, E). Die Primordien der Samenanlagen sind im undifferenzierten Zustand etwa 50 µm breit und erstrecken sich über 100 µm entlang der Karpellwand. Die Karpelle haben auf Höhe der Samenanlagenprimordien eine Breite von 400 µm. Bis Ende September nehmen die Karpelle und Samenanlagenprimordien weder an Größe zu noch verändert sich der Differenzierungsgrad der Primordien deutlich (Abb. 23A). Dennoch enthalten einige der nur geringfügig weiter entwickelten Karpelle im basalen Abschnitt der Blütenachse Samenanlagenprimordien mit einer beginnenden Differenzierung. Der in Abbildung 23B dargestellte Längsschnitt durch ein Karpell Ende September zeigt ein solches Samenanlagenprimordium, bei dem sich das innere Integument als eine erste kleine Wölbung vom restlichen Nucellus absetzt. Bereits zwei Wochen später, Anfang Oktober, ist diese Differenzierung sogar schon deutlich an weniger weit entwickelten Primordien zu erkennen. Die Anfang Oktober weniger weit entwickelten Karpelle sind im Bereich der Samenanlagen Primordien bis zu 500 µm Breite gewachsen; die Primordien messen dabei knapp 75 µm in der Breite und rund 85 µm in der Höhe (Abb. 23C). Im Detailbild in Abbildung 23D ist zu erkennen, dass sich zentral im oberen Bereich des Nucellus die Makrosporenmutterzelle entwickelt hat. Sie hebt sich hell von den übrigen Nucelluszellen ab, da sie weniger Cytoplasma enthält. Auf Höhe der Makrosporenmutterzelle ist nun auch das innere Integument erkennbar. Die weit entwickelten Samenanlagen- ERGEBNISSE 60 primordien von Anfang Oktober sind 80 µm breit und ohne den nun abzugrenzenden Funiculus ca. 100 µm hoch. Sie sitzen in Karpellen von 600 µm Breite. Beim in Abbildung 23E gezeigten Karpellquerschnitt ist eine Samenanlage im Karpell erkennbar. Allerdings wird anhand der angefertigten Serienschnitte deutlich, dass ein Karpell meistens zwei Samenanlagen enthält, von denen eine jedoch verkümmern kann. Bei den weiter entwickelten Samenanlagen von Anfang Oktober ist ein einseitiger basaler Auswuchs der Samenanlage an der dem Funiculus gegenüber liegenden Seite sichtbar (Abb. 23E). Dieser basale Auswuchs, der wie eine Verlängerung des Funiculus wirkt, stellt den Entwicklungsbeginn des äußeren Integuments dar. Die Makrosporenmutterzelle hat sich im Vergleich zu den erst weniger weit entwickelten Samenanlagen desselben Sammeldatums nicht weiter entwickelt. Einen Monat später, Anfang November, ist die Differenzierung der Samenanlage in Nucellus, inneres Integument und äußeres Integument problemlos zu erkennen (Abb. 24A). Das äußere Integument setzt sich als Wulst nun auch bis zum Funiculus weiter fort. In Kombination mit der rasterelektronenmikroskopischen Aufnahme einer zwei Wochen älteren Samenanlage ist zu erkennen, dass sich das innere und äußere Integument mehr oder weniger ringförmig um den Nucellus ziehen. Dabei ragt der Nucellus zwischen den beiden Integumenten hervor (Abb. 24B). Aufgrund der ringförmigen Entwicklung des äußeren Integuments haben die Samenanlagen ihre Breite im Laufe von vier bis sechs Wochen mit nun 160 µm mehr als verdoppelt. Auch an Höhe haben sie in demselben Zeitraum mit einem Wachstum um etwa 50% auf ca. 150 µm deutlich zugenommen. Die beschriebene ringförmige Struktur ist beim äußeren Integument anfangs noch im Stielbereich unterbrochen und nur als ein erheblich niedrigerer Wall fortgesetzt, wie anhand der Abbildung 23D zu erkennen ist. Die dort gezeigte Samenanlage stammt von Anfang Februar und ist problemlos mit den weit entwickelten Samenanlagen von Mitte Dezember vergleichbar, da die Art Magnolia stellata eine Winterruhe einlegt. In Abbildung 24C ist ein Längsschnitt durch eine weit entwickelte Samenanlage von Mitte Dezember dargestellt, bei dem erkennbar ist, dass die Makrosporenmutterzelle an Größe zugenommen hat und nicht mehr wie anfänglich knapp 6 µm sondern gut 20 µm breit ist. Die Makrosporenmutterzelle nimmt jedoch nicht nur an Breite zu, sondern verändert auch ihre Form. Sie entwickelt sich von einer kugeligen zu einer lang gestreckten Zelle (Abb. 24E). Ende Februar misst sie gut 20 µm mal 30 µm. Der Nucellus, das innere und das äußere Integument haben sich im apikalen Bereich gestreckt, so dass die Makrosporenmutterzelle als Folge davon tiefer im Nucellus gelegen scheint. Parallel dazu ist das äußere Integument stärker als die anderen Strukturen in entgegengesetzter Richtung gewachsen und stellt nun die mächtigste ERGEBNISSE 61 Struktur der Samenanlage dar. In der seitlichen Ansicht macht das äußere Integument etwa 60% der Gesamthöhe der Samenanlage aus (Abb. 24F). Die bisher dargestellten Entwicklungsstadien beziehen sich jeweils, sofern nicht anders erwähnt, auf das am weitesten entwickelte Stadium zu diesem bestimmten Sammelzeitpunkt. Anhand der in Abbildung 25A gezeigten Samenanlagen von Mitte März wird aber deutlich, dass zwei Samenanlagen einer Blüte sogar innerhalb eines Karpells stark voneinander abweichend entwickelt sein können. Dass der gezeigte Größenunterschied kein durch die Schnittebene hervorgerufenes Artefakt ist, wird durch die zwei Samenanlagen desselben Sammeldatums in Abbildung 25B deutlich. Sie sitzen gemeinsam in einem Karpell, das für die rasterelektronenmikroskopischen Untersuchungen aufpräpariert wurde. Die rechte Samenanlage ist nur annähernd 200 µm hoch, die linke Samenanlage hat eine Höhe von mindestens 250 µm. Eine derart weit entwickelte Samenanlage wie letztere ist auch in Abbildung 25C gezeigt. Im Vergleich zu einer Samenanlage von Ende Februar (Abb. 24E) haben sich der Nucellus und die beiden Integumente weiter gestreckt, so dass der unverwachsene Bereich von Nucellus und den Integumenten nicht mehr knapp ein Drittel sondern nun gut die Hälfte der Gesamthöhe der Samenanlage ausmacht (Abb. 25C). In der seitlichen Ansicht wird deutlich, dass das Wachstum der Samenanlage auf der vom Funiculus abgewandten Seite stärker gefördert wird (Abb. 25D). Am reifen Samen Ende September sind die beschriebenen Entwicklungsvorgänge nicht mehr nachzuvollziehen. Man erkennt lediglich eine zweigeteilte Strukturierung in eine harte, schwarz gefärbte Samenschale, die als Sklerotesta bezeichnet wird, und eine fleischige, den Samen umgebenden Sarkotesta, die rot gefärbt ist (Abb. 25E). An einem Samen, der in der Mitte längs durchgetrennt wurde, sieht man nach einer kurzen Antrocknungszeit, dass der gesamte Aufbau jedoch vielschichtiger ist (Abb. 25F). Das äußere Integument, das den größten Teil der Sarkotesta bildet, lässt eine Trennschicht erkennen, die sich ringsum den Embryo von der Spitze des Nucellus bis zum Funiculus zieht. Von dieser Trennschicht aus nach innen gelegen grenzt das innere Integument an, das die Samenschale bildet. Von der Samenschale umhüllt ist der Nucellus, der als Nährgewebe für den Embryo dient und von diesem bereits fast vollständig aufgezerrt ist. Der Funiculus ist vollständig ausgetrocknet. Lediglich die Leitbündel sind erhalten und als fädige Struktur an der Basis des reifen Samens zu erkennen (Abb. 25F). An diesen Leitbündeln, die unter dem Gewicht des Samens entspiralisiert sind, hängt der reife Samen zur besseren Präsentation für endozoochore Verbreiter aus dem Karpell heraus. ERGEBNISSE Abb. 22: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM- und REM-Aufnahmen A: Längsschnitt durch das Gynoeceum und Androceum einer Blütenknospe Ende August; B: Anthere im Querschnitt; C: Längsschnitt durch ein junges Karpell Ende August; D: Aufsicht auf die beiden Samenanlagenprimordien in einem aufpräparierten Karpell Anfang September; E: Detailansicht der apikalen Zone eines Primordiums Anfang September (An = Anthere; Ba = Blütenstandsachse; Fi = Filament; K = Karpell; Kon = Konnektiv; PrSa = Primordium Samenanlage; Ps = Pollensack) 62 ERGEBNISSE Abb. 23: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM-Aufnahmen A: Quergeschnittenes Karpell Ende September; B: Längsschnitt durch ein weit entwickeltes Karpell Ende September – Pfeil weist beim Samenanlagenprimordium auf beginnende Bildung des inneren Integuments; C: Querschnitt durch ein Karpell Anfang Oktober; D: Detailabbildung eines schwach entwickelten Samenanlagenprimordiums Anfang Oktober; E: Quergeschnittenes Karpell Anfang Oktober; F: Samenanlage Anfang Oktober im Detail (aI = äußeres Integument; iI = inneres Integument; K = Karpell; Mz = Makrosporenmutterzelle; Nu = Nucellus; PrSa = Primordium Samenanlage) 63 ERGEBNISSE Abb. 24: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM- und Rem-Aufnahmen A: Längsgeschnittene Samenanlage von Anfang November im Detail; B: seitliche Ansicht einer Samenanlage Mitte Dezember; C: Samenanlage im Längsschnitt, Mitte Dezember; D: Aufsicht auf eine Samenanlage Anfang Februar; E: Längsschnitt durch Samenanlage Anfang Februar; F: Samenanlage von Ende Februar in seitlicher Ansicht (aI = äußeres Integument; iI = inneres Integument; Mz = Makrosporenmutterzelle; Nu = Nucellus) 64 ERGEBNISSE Abb. 25: Magnolia stellata (SIEBOLD & ZUCC.) MAXIM, LM-, REM- und BinokularAufnahmen A: Querschnitt durch Karpell Mitte März; B: Aufsicht auf die Samenanlagen eines aufpräparierten Karpells Ende März; C: Längsgeschnittene Samenanlage Mitte März; D: Samenanlage in seitlicher Ansicht Anfang April; E: reifer Samen von Anfang November mit aufpräparierter Sarkotesta; F: Längsgeschnittener reifer Samen Ende September (aI = äußeres Integument; E = Endosperm; iI = inneres Integument; K = Karpell; Mp = Makroprothallium; Mt = Makrosporentetrade; Nu = Nucellus) 65 ERGEBNISSE 3.2. ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 3.2.1. MOLEKULARBIOLOGISCHE VORARBEITEN 66 Eine wesentliche Aufgabe der vorliegenden Arbeit bestand darin, ein Protokoll für die In-situ-Hybridisierung an Gymnospermen zu erstellen. Aus diesem Grund wird auch sehr ausführlich auf die einzelnen dafür notwendigen Arbeitsschritte eingegangen, ungeachtet dessen, dass diese Vorgehensweise äußerst unüblich für molekularbiologische Arbeiten ist. In Hinblick auf die detaillierte Ergebnisdarstellung zu den In-situHybridisierungen (3.2.2.) sind die Ergebnisse der molekularbiologischen Vorarbeiten in diesem Kapitel ebenso ausführlich aufgezeichnet. Sämtliche Arbeiten, die ein Genbzw. S1-Labor bedingen, wurden in den Laboren der „AG Parasitologie“ von Prof. Dr. Schaub am Lehrstuhl für Evolutionsökologie und Biodiversität der Tiere der RuhrUniversität Bochum durchgeführt. 3.2.1.1. PRIMERDESIGN ZUR GEWINNUNG EINER MADS-BOX-C-GEN-SEQUENZ Das in Abbildung 26 gezeigte Aminosäuren-Alignment basiert auf Ergebnissen der In-silico-Suche in der Datenbank des „National Center for Biotechnology Information“ (NCBI) zu allen von Gymnospermen verfügbaren MADS-box-C-Gen-Sequenzen ausgehend vom 2003 von JAGER et al. veröffentlichten AGAMOUS-like MADS-box-Transkriptionsfaktor von Ginkgo biloba (Ginkgoaceae). Neben diesem sind im Alignment drei weitere AGAMOUS-like MADS-box-Transkriptionsfaktoren von Picea mariana (Pinaceae), das dal2-Gen von Picea abies (Pinaceae), das GGM3-Gen von Gnetum gnemon (Gnetaceae) und die MADS-box-Transkriptionsfaktoren von Cycas edentata (Cycadaceae) und den zwei Pinaceen Pinus resinosa und Pinus radiata enthalten. Ergänzt werden die aus Gymnospermen isolierten Gensequenzen durch MADS-boxC-Funktions-Gene von Oryza sativa (Poaceae), Malus x domestica (Rosaceae) und Zea mays (Poaceae). Das Alignment (Abb. 26) umfasst sowohl die MADS-, K– und IDomäne als auch knapp 100 Aminosäuren der in der Länge variablen C-terminalenRegion, die durch die rot und schwarz geschriebenen Aminosäurekürzel farblich voneinander abgegrenzt sind. Es bildet die Grundlage für das Primerdesign von MADSbox-C-Gen-Primern für Zamia amblyphyllidia (Cycadaceae). Ebenfalls farbig markiert sind in diesem Alignment die mir von Dr. F. Vergara-Silva zur Verfügung gestellten Primer „1-mads_for“ (blau), ein MADS-box-Gen spezifischer PCR-forward-Primer, und „2-C_for“ (violett), ein C-Gen spezifischer Forward-Primer für die so genannte „nested PCR“. Um die Oligonukleotid-Primer im AminosäurenAlignment einzuordnen, ist vorab ein Alignment auf Nukleotidsequenzbasis der oben ERGEBNISSE 67 genannten Gene erstellt und mittels des Programms BIOEDIT in die in Abbildung 26 vorliegende Form umgeschrieben worden. Es wird deutlich, dass der MADS-box-Gen spezifische Primer „1-mads_for“ in der für alle MADS-box-Gene hoch konservierten MADS-Domäne gelegen ist, wohingegen der C-Gen spezifische Primer „2-C_for“ im Übergang von der variablen I-Domäne zur konservierten K-Domäne ansetzt. ________________________MADS-Domäne_______________________________ I-Domäne_ C.edentata --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANNSVKRTIERYKKTCAD-NT C.edentate --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANNSVKRTIERYKKTCAD-NT G.biloba --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANNSVKRTIDRYKKTCAD-NS P.mariana ------KIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANHSVKRTIERYKKTCVD-NN P.mariana --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANHSVKRTIERYKKTCVD-NN P.abies --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANHSVKRTIERYKKTCVD-NN P.mariana --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANHSVKRTIERYKKTCVD-NN P.resinosa --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANHSVKRTIERYKKTCVD-NN P.radiata --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANHSVKRTIERYKKTCVD-NN G.gnemon --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEFANNSVKRTIERYRKTCAD-NN M.domestica RKLGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSNRGRLYEYANNSVKGTIERYKKASAD-SS Z.mays ---------------TTSRQVTFCKRRNGLLKKAYELSVLCDAEVALVVFSSRGRLYEYANNSVKSTIERYKKANSD-SS O.sativa --MGRGKIEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEYANNSVKSTVERYKKANSD-TS Z.mays GGRGKGKTEIKRIENTTNRQVTFCKRRNGLLKKAYELSVLCDAEVALIVFSSRGRLYEYANNSVKGTIERYKKATSDNSS primer1-mads_for _______ __ K-Domäne_______________________ C.edentata QGGAISESNSQ-YWQQEAGK LRQQIDILQNAN-RHLMGDALTSLSVKELKQLEIRLERGLSRVRSKKNEMLLEEIEIMQ C.edentata QGGAISESNSQ-YWQQEAGK LRQQIDILQNAN-RHLMGDALTSLSVKELKQLEIRLERGLSRVRSKKNEMLLEEIEIMQ G.biloba QGGAISECNSQ-YWQQEAGK LRQQIDILQNAN-RHLMGDALTSLSVKELKQLEIRLERGISRVRSKKNEMLLEEIEIMQ P.mariana HGGAISESNSQ-YWQQEAGK LRQQIEILQNAN-RHLMGDGLTALNIKELKQLEVRLEKGIGRVRSKKNEMLLEEIDIMQ P.mariana HGGAISESNSQ-YWQQEAGK LRQQIEILQNAN-RHLMGDGLTALNIKELKQLEVRLEKGIGRVRSKKNEMLLEEIDIMQ P.abies HGGVISESNSQ-YWQQEAGK LRQQIEILQNAN-RHLMGDGLTALNIKELKQLEVRLEKGIGRVRSKKNEMLLEEIDIMQ P.mariana HGGAISESNSQ-YWQQEAGK LRQQIEILQNAN-RHLMGDGLTALNIKELKQLEVRLEKGIGRVRSKKNEMLLEEIDIMQ P.resinosa HGGAISESNSQ-YWQQEAGK LRQQIEILQNAN-RHLMGDGLTALNIKELKQLEVRLEKGISRVRSKKNEMLLEEIDIMQ P.radiata HGGAISESNSQ-YWQQEAGK LRQQIDILQNAN-RHLMGDGLTALNIKELKQLEVRLEKGISRVRSKKNEMLLEEIDIMQ G.gnemon QGGAIAESNAQ-YWQQEAVK LKQQIDVLNNQI-RHYMGECLQSMTIKELKQLEGKLEKGLGRVRSKRNEKLLEDIDTLQ M.domestica NTGSVSEASTQ-YYQQEAAK LRARIVKLQNDN-RNMMGDALNSMSVKDLKSLENKLEKAISRIRSKKNELLFAEIEYMQ Z.mays NSGTVAEVNAQ-YYQQESSK LRQMIHSLQNANTRNIVGDSIHTMGLRDLKQMEGKLEKAIIKIRARKNELLYAEVDYMQ O.sativa NSGTVAEVNAQ-HYQQESSK LRQQISSLQNANSRTIVGDSINTMSLRDLKQVENRLEKGIAKIRARKNELLYAEVEYMQ Z.mays AAGTIAEVTIQ-HYKQESARLRQQIVNLQNSN—-RALIGDSITTMSHKELKHLETRLDKALGKIRAKKNDVLCSEVEYMQ primer2-C_for primer-mads _____________________ ___C-terminale Domäne______ __________________________ C.edentata RREHILLAENQFLRTKIAE-YE-SNQNTNV-LIPGP--EFDALP---AFDSR-NFLHANLIEAA--AHHYTQQDQAALQL C.edentata RREHILLAENQFLRTKIAE-YE-SNQNTNV-LIPGP--EFD ALP--AFDSR-NFLHANLIEAA--AHHYTQQDQAALQL G.biloba RREHILLAENQFLRTKIAE-CE-SSQNAN--MLPGP--EFD ALP--GFDSR-HFLHASIMD----AHHYAQQDQTALQL P mariana RREHILIQENEILRSKIAE-CQ-NSHNTN--MLSAP--EYD ALP--AFDSR-NFLHANLIDAA---HHYAHQEQTTLQL P.mariana RREHILIQENEILRSKIAE-CQ-NSHNTN--MLSAP--EYD ALP--AFDSR-NFLHANLIDAA---HHYAHQEQTTLQL P.abies RREHILIQENEILRSKIAE-CQ-NSHNTN--MLSAP--EYD ALP--AFDSR-NFLHANLIDAA---HHYAHQEQTTLQL P.mariana RREHILIQENEILRSKIAE-CQ-NSHNTS--MLSAP--EYD ALP--AFDSR-NFLHANLIDAA---HHYAHQEQTTLQL P.resinosa RREHILIQENEILRSKIAE-CQ-NSHNTN--MLSAP--EYD ALP--AFDSR-NFLHANLIDAA---HHYAHQEQTTLQL P.radiata RREHILIQENEILRSKIAE-CQ-NSHNTN--MLSAP--EYD ALP--AFDSR-NFLHANLIDAA---HHYAHQEQTTLQL G.gnemon RREDNLIRENEYIRNKIAE-CQ-SHQHAN--MLTAAAVEYD AIPA-AYDSR-NFMHANLIEAAAAHHHYAQQEQTALHL M.domestica KRELDLHNNNQLLRAKIAE-NERASRTLNV-MAGGGTSSYD ILQSQPYDSR-NYFQVNALQPN--HQYNPRHDQISLQL Z.mays KREMDLQTDNMYLRSKIAESNETGQPAMHMTMGAPPTSEYD HMA--PFDSR-NFLQV---SMPQ--HYSHQLQPTTLQL O. sativa KREVELQNDNMYLRSKVVE-NERGQQPLNM-MGAASTSEYD HMVNNPYDSR-NFLQVNIMQQPQ--HYAHQLQPTTLQL Z. mays RREMELQNDNLYLRSRVDE-NERAQQTANM-MGAPSTSEYQ QHGFTPYDPIRSFLQFNIVQQPQ--FYSQQEDRKDFND _rev Abb. 26: Aminosäuren-Alignment zu MADS-box-Genen der C-Funktionsklasse von Gymnospermen und ausgewählten Angiospermen Aminosäureabkürzungen in rot und schwarz zur optischen Abgrenzung der Domänen (I = Intervening, K = Keratin-like); blau: Forward-Primer für MADS-box-Gene allgemein; violett: Forward-Primer speziell für C-Gene; grau: für Gymnospermen übereinstimmende Sequenzbereiche; grün: Reverse-Primer ERGEBNISSE 68 Die ersten „nested PCRs“ mit den aufeinander abgestimmten Primern „1-mads_for“ und „2-C_for“ in Kombination mit Poly-T-Oligonukleotiden als Reverse-Primer führten zu keinem Ergebnis. Nach dem ersten PCR-Schritt waren in der Gelelektrophorese keine deutlichen Banden von definierter Fragmentlänge auszumachen, sondern ein Gemisch aus DNA-Fragmenten von einer Länge zwischen 100 und 300 bp (Abb. 27). M 47,8 50,2 51,5 52,9 54,4 55,8 [°C] primer1-mads_for Abb. 27: Gelelektrophorese zum 1. Schritt der „nested-PCR“ mit den Primern „1-mads_for“ und „Poly-T“ (M = Marker) M 50,2 51,5 52,9 54,4 55,3 55,8 [°C] primer2C_for Abb. 28: Gelelektrophorese zum 2. Schritt der „nested-PCR“ mit den Primern „2C_for“ und „Poly-T“ (M = Marker) Bei Einsatz dieser PCR-Produkte in der Folge-PCR waren zwar deutliche Banden auszumachen, jedoch entsprachen die Fragmentlängen nicht denen der gesuchten Sequenzlänge (Abb. 28). Die Klonierung und Sequenzierung der DNA-Fragmente, die der gewünschten Sequenzlänge mit einer Differenz von ca. 100 bp am nahesten kamen, führte zu einem Ergebnis, das leider in keinem Zusammenhang mit MADSbox-Genen stand (siehe 3.2.1.3.). Dieser Umstand machte die Synthetisierung eines spezifischeren Reverse-Primers erforderlich. Dazu wurde im Alignment eine Region gewählt, in der zwischen allen Sequenzen der Gymnospermen eine 100%-ige Übereinstimmung der Aminosäurenabfolge existiert und die zudem noch viele Aminosäuren enthält, die nur über wenige Basen-Tripletts kodiert werden. Die gefundene Aminosäurensequenz wurde in die dazugehörenden Nukleotidsequenzen umgeschrieben, zu denen dann wiederum der Komplementärstrang in 3`-5`Richtung gebildet wurde, um auf diesem basierend entsprechend dem internationalen Code für degenerierte Primer die Sequenz für den Reverse-Primer „primer-mads_rev“ ermitteln zu können (Abb. 29). Dieser Primer ist in den folgenden nested PCRs eingesetzt worden. ERGEBNISSE 69 A) Umschreiben der Sequenz(EEIEIMQRREH)in die jeweiligen Triplett-Codes E G A E A G G A I A G A T E T C A G A I A G A T M T C A A T Q G C A R A G C A G R T C A G A G C G A T C A G A G B) Umschreiben der Tripletts in den „Reverse-Strang“ A A C G T A G T T A C A A G G T T A C A A A G G A A G G A C) Umschreiben des „Reverse-Strang“ in den „Komplementärstrang“ und degenerierten Prime-Code T T G C A T C A A T G T T C C T G C A T D A T G T Y A T T C C T T C T C C T A T Y T C D A T Y T C Y degen. Primer-Code Abb. 29: Schrittweise Ermittlung eines degenerierten Reverse-Primers „primer-mads_rev“ A) Aminosäuresequenz der Primer-Region mit darunter stehenden möglichen Triplett-Codons (grau unterlegt); B) Umschreibung des grau unterlegten Bereichs aus A) von der 5´-3´-Richtung in die entgegengesetzte 3´-5´-Richtung; C) Bildung des Komplementärstranges zu B) unter Berücksichtigung aller möglichen Nukleotide und Übertragung in den degenerierten Primercode für die nicht eindeutigen Nukleotidpositionen (gelb unterlegt in der grün markierten Primersequenz) Die nun folgende „nested PCRs“ unter Einsatz des in Abbildung 29 beschriebenen Reverse-Primers „primer-mads_rev“ führte mit den beiden wie zuvor nacheinander eingesetzten Primern „1-mads_for“ und „2-C_for“ zum erwarteten Ergebnis von einer isolierten Gensequenz mit ca. 200 bp Länge. Im ersten Schritt der „nested PCR“ wurden zwei auf unterschiedliche Weise gewonnene cDNAs (siehe 3.2.1.2.) als Templates in der Temperaturgradienten-PCR eingesetzt. In der Gelelektrophorese (Abb. 30) zeigte sich, dass die meisten PCR-Produkte im gleichen Längenbereich liegen. Da die genaue Länge der MADS-box-Gene von Zamia amblyphyllidia jedoch unbekannt ist, wurden für den darauf folgenden PCR-Schritt die PCR-Produkte mit der variabelsten Fragmentlänge eingesetzt (Ansatz 2 bei 48.9°C). Diese ebenfalls mit einem Temperaturgradienten durchgeführte PCR ergab in der Gelelektrophorese in allen Ansätzen eine gut sichtbare, klar definierte Bande bei einer Basenlänge von etwas mehr als 200 bp (Abb. 31). ERGEBNISSE 48,9 50,2 51,5 70 52,9 54,4 M 48,9 50,2 51,5 52,9 54,4 M 49,8 50,2 [°C] primer1-mads_for primer-mads_rev Abb. 30: Gelelektrophorese zum 1. Schritt der „nested PCR“ mit den Primern „1-mads_for“ und „mads_rev“ (M = Marker; links und rechts je zwei unterschiedliche cDNAs als Templates für PCR) 51,5 52,9 54,4 [°C] primer2C_for primer-mads_rev Abb. 31: Gelelektrophorese zum Folgeschritt der „nested PCR“ mit den Primern „2-C_for“ und „mads _rev“ (M = Marker) 3.2.1.2. RNA-ISOLIERUNG UND CDNA-SYNTHESE Die Isolierung der RNA wie auch die anschließende cDNA-Synthese sind jeweils auf zweierlei Weisen vorgenommen worden. Zum einen wurde das Material für die RNAIsolierung entsprechend der Anleitung des „RNeasy Plant Mini Kits“ von QUIAGEN in Stickstoff gemörsert, in ein 2 ml-Eppendorfgefäß überführt und unter Verwendung des RLC-Puffers aufgeschlossen. Zum anderen wurde eine Methode getestet, bei der das Pflanzenmaterial direkt im Eppendorfgefäß unter Zugabe vom RLC-Puffer mit einem Mini-Pistill zerkleinert wurde. Mit einer RNA-Ausbeute von ca. 50 µg/ml im Vergleich zu 154,7 µg/ml RNA (bzw. in späteren Versuchen bis zu 224,5 µg/ml RNA) bei der Durchführung entsprechend des „RNeasy Plant Mini Kit“-Protokoll erwies sich die alternative Methode als weniger praktikabel, so dass sie in Kapitel 2 „Material und Methoden“ unerwähnt bleibt. Im Folgenden ist mit der RNA weitergearbeitet worden, die mit Hilfe des Kits isoliert wurde. Bei der cDNA-Synthese wurde ebenfalls eine Alternative zur cDNA-Synthese mittels eines Kits („3´RACE System for Rapid Amplification of cDNA Ends“, INVITROGEN) getestet, indem 1 µl RNA mit 1 µl dT18VN-Oligonukleotiden und 11 µl Aqua dest. für 10 Minuten bei 70°C inkubiert wurde, bevor dem Ansatz 2 µl dNTPs, 4 µl Puffer (5x) und 1 µl Reverse Transkriptase zugegeben wurden, um 1 Stunde lang bei 42°C zu inkubieren. Nach den anschließenden 10 Minuten Inkubationszeit bei 70°C ist die ERGEBNISSE 71 Reaktion abgeschlossen. Wie der Abbildung 30 zu entnehmen ist, wurde in der PCR bei dieser Methode (linke Seite) ein weniger breites Spektrum an DNA als bei der Isolierung mittels des oben erwähnten Kits (rechte Seite) amplifiziert, so dass auch diese Methode keine Erwähnung im Kapitel 2 der vorliegenden Arbeit findet. 3.2.1.3. KLONIERUNG UND SEQUENZIERUNG DER GEWONNENEN GENSEQUENZEN Die im Rahmen der unter 3.2.1.1. beschriebenen „nested PCR“ gewonnenen DNAFragmente wurden nach der Gelelektrophorese aus dem Gel ausgeschnitten und eluiert, um sie in einen pGEM-T-Easy-Vektor zu ligieren und dieses Produkt dann in kompetente Zellen zu transformieren, die auf Nährplatten gegeben wurden, um sich – und somit auch das Insert – exponentiell zu vermehren. Das „Picken“ der Insert enthaltenden Kolonien ist auf zwei Weisen vorgenommen worden. Entweder nimmt man die Kolonien ab und löst sie in 40 µl H2O, von denen 2 µl für einen PCR-Ansatz verwendet werden und der Rest zu 600 µl LB+AMP-Nährmedium gegeben wird, oder man löst die mittels eines abgeflämmten Zahnstochers abgenommenen Kolonien direkt in 30 µl eines PCR-Ansatzes und gibt die restlichen am Zahnstocher haftenden Kolonien mitsamt des Zahnstochers in 300 µl LB+AMP-Nährmedium. Bei beiden Methoden folgte eine Inkubation der Kolonien unter Schütteln im Wärmeschrank bei 37°C. Anhand der Trübung der Nährlösung lässt sich auf die Stärke des Bakterienwachstums schließen. Beide Methoden zeigten vergleichbare positive Ergebnisse. Der oben erwähnte PCR-Ansatz dient der Fragmentbestimmung des Inserts mittels der M13-PCR, bei der spezielle Primer eingesetzt werden, durch die das Insert und ein definierter, kurzer Bereich ringsum das Insert vervielfältigt werden, so dass eine anschließende Gelelektrophorese Aufschluss über die Insertlänge gibt. Eine „nested PCR“ ohne den speziellen Reverse-Primer „primer-mads_rev“ führte zu zahlreichen Klonen mit einer Insertlänge von ca. 900 bp (Abb. 32). Abb. 32: Gelelektrophorese zur Plasmid-DNA nach M13-PCR der Klonierungsprodukte des 2. PCR-Schrittes einer „nestedPCR“ mit den Primern „2-C_ for“ und dem Poly-T-ReversePrimer M 1 2 3 4 5 M13-PCR, Inserts Klone 1-9 6 7 8 9 Klon 1, 3, 4, 5, 6, 7 und 9 zeigen eine Insertlänge von ca. 900 bp, Klon 2 ist ca. 400 bp lang, Klon 8 ist ca. 250 bp lang. (M = Marker, 1-9 = Klon-Nr.) ERGEBNISSE 72 Die Länge der variablen C-terminalen Sequenz des gesuchten MADS-box-C-Gens von Z. amblyphyllidia ist unbekannt. Der Abgleich mit bekannten Sequenzen anderer Gymnospermen lässt jedoch eine Länge von mehr als 500 bp vermuten, so dass die die Klone mit kürzerem Insert (= Klon 2 und 8) verworfen wurden. Von den übrigen Klonen wurde Klon 3 exemplarisch für eine Sequenzierung in einer Übernacht-Kultur weitervermehrt, „geprept“ (siehe 2.2.1.8.), gefällt und zur Sequenzierung versandt. Das Sequenzierungsergebnis eines exemplarischen Klons sah so aus (Abb. 33): madsC_Klon3 Vektor TATGCATCCAACGCGTTGGGAGCTCTCCCATATGGTCGACCTGCAGGCGGCCGCGAATTCACTAGTGATT Insert GGCCACGCGTCGACTAGTACCGGCTGGTCCGCGTTGTTCGAACCCACGACGGGGCCGACGACCGCGCCTA CCGCTGCGTGTCGCAGGAGGAGGACCCCGAGGGGATTGTGGGGATTTCGCTGTCCAAGGATCTCATGGTC ATCGCCGGGCGGGCGCTGAGATCCAACATTACCTCGCTGGGGCCCCTCGTGCTCCCCCTTTCCGAGCAGA TTCTGTACGCCCTGTCGGCGCTGAGGAGGAAATGGGCGGATCCGAAGGCGAGGCTTTATGTGCCGGACTT CAAGCGCGCGTTCGAGCATTTCTGCATCCACGCGGGGGGGAGGGCCGTCATCGACGAGCTGGAGAAGAAT TTGTCGCTGACGGAGGAGCAGGTGGAGGCCTCGCGGATGACGCTCTACCGCTTCGGGAACACCTCATCGT CCTCGCTGTGGTACGAATTGGCTTATATTGAAGCCAAGGGGAGGATGCGGGGGGGGGATCGGGTGTGGCA GATCGCGTTCGGGAGCGGATTCAAGTGCAATAGCGCCGTTTGGCAGGCGCTTCGCACGGTGAGGA Vektor ??? Abb. 33: Sequenzierungsergebnis der M13-PCR von Klon 3 schwarz: Insert = Produkt des zweiten PCR-Schritts einer „nested-PCR“ mit den Primern „2-C_for“ und einem Poly-T-Reverse-Primer; rot: Vektor In der ermittelten Sequenz (Abb. 33) war nicht der gesamte Vektor zu finden, was auf ein zu langes Insert für die Sequenzierung schließen lässt. Der Sequenzabgleich über „NCBI“ (siehe 2.2.1.9.) ergab eine relativ hohe Sequenzübereinstimmung von 76% mit zahlreichen Nukleotidsequenzen von am Aufbau von Fettsäuren beteiligten Enzymen aus unterschiedlichen Gruppen des Pflanzenreiches. Beispiele daraus sind das der Sequenz ähnlichste „putative very long chain fatty acid condensing enzyme CUT1“ aus der Monokotylen Hordeum vulgare L. (Poaceae), die „3-ketoacyl-CoASynthase“ aus der Dikotylen Eranthis hyemalis (L.) SALISB. (Ranunculaceae) oder die „beta-ketoacyl-CoA-Synthase“ aus dem Lebermoos Marchantia polymorpha L. (Marchantiaceae). Da diese Enzyme in keinem Zusammenhang mit MADS-box-Genen stehen, wurde die „nested PCR“ wie unter 3.2.1.1. beschrieben modifiziert. ERGEBNISSE 73 Die PCR-Produkte der „nested PCR“ mit den Primern „1-mads_for“ (MADS-box-Gen spezifischer PCR-forward-Primer) im ersten Schritt und „2-C_for“ (Forward-Primer für MADS-box-C-Gene) im Folgeschritt sowie dem Reverse-Primer „primer-mads_rev“ führten zu DNA-Fragmenten gewünschter Länge, die wie bereits oben beschrieben aufgearbeitet wurden. Abbildung 34 zeigt die Gelelektrophorese mit den Produkten der M13-PCR von jeweils fünf gepickten Klonen pro Nährplatte, die einmal mit 100 µl Bakterienlösung und einmal mit 200 µl Bakterienlösung versetzt wurde. Abb. 34: Gelelektrophorese zur Plasmid-DNA nach M13-PCR der Klonierungsprodukte des 2. PCR-Schrittes einer „nestedPCR“ mit den Primern „2C_for“ und „primer-mads_rev“ Links vom Marker (M) Klone von einer Nährplatte mit 100 µl Auftrag, rechts davon Klone von einer Platte mit 200 µl Auftrag an Bakterienlösung 1 2 3 4 5 M 6 M13-PCR, Inserts Klone 1-5 & 6-10 7 8 9 10 Klone 3, 5, 6-9 sind ca. 250 bp lang, Klon 1 ist etwas kürzer, Klon 2 etwas länger; Klon 4 enthält wenig DNA; Klon 10 zeigt eine Doppelbande. Die Klone 1, 3 und 6 bis 9 wurden in einer so genannten „Miniprep“ aufgearbeitet, die isolierte DNA gefällt und zur Sequenzierung versandt. Die erhaltenen Ergebnisse der Sequenzierung ergaben für die Klone 1 und 6 keine verwertbaren Sequenzen. Klon 6 enthielt eine Nonsens-Sequenz, die in der Blast-Search im NCBI zu gar keinem Ergebnis führte, wohingegen es sich bei Klon 1 um eine verunreinigte Probe handelt, die ein Insert enthielt, das mit dem Gen „Def1“ bzw. „defensin“ aus der Raubwanze Triatoma brasiliensis übereinstimmt. T. brasiliensis ist ein Überträger der ChagasKrankheit und stellt ein Forschungsobjekt der AG Parasitologie von Prof. Dr. Schaub am Lehrstuhl für Evolutionsökologie und Biodiversität der Tiere der Ruhr-Universität Bochum dar. Die übrigen Klone 3, 7, 8 und 9 zeigen untereinander eine hohe Übereinstimmung in ihren Sequenzen (Abb. 35) und lassen beim Abgleich mit den im „NCBI“ verfügbaren Nukleotidsequenzen mittels der so genannten „megablast“, eine Suchform nach hoch ähnlichen Sequenzen, eine eindeutige Zuordnung zu den MADS-box-C-Genen der Spermatozoid befruchteten Gymnospermen Cycas edentata L. (Cycadaceae) und Ginkgo biloba L. (Ginkgoaceae) zu. In der Abbildung 35 sind eine Sequenzen von C. edentata und beide Sequenzen von G. biloba mit den Klonen 3, 7, 8 und 9 aligniert. ERGEBNISSE 74 Dabei sind die übereinstimmenden Nukleotide aller Sequenzen rot unterlegt. Die Bereiche, die nur für die Klone und die aus der gleichen Ordnung wie Z. amblyphyllidia stammende Art C. edentata übereinstimmen, sind in pink unterlegt. An fünf Nukleotidpositionen passt die bei Z. amblyphyllidia gefundene Sequenz besser zu den Sequenzen von G. biloba als zu C. edentata. Diese Positionen sind durch ein darunter stehendes Ausrufungszeichen markiert. Die Sequenzabschnitte, die nur für die Klone identisch sind, sind in hellblau unterlegt. Basen innerhalb der Klonsequenzen, die von den übrigen abweichen, sind in grün farblich hervorgehoben. Klon 3 Klon 7 Klon 8 Klon 9 C. edentata G. biloba G. biloba *589 -------AGTACTGGCAACAGGAGGCAGGAAAACTCAGGCAACAAATTGACATTGTACAAAA -------AGTACTGGCAGCAGGAGGCAGGAAAACTCAGGCAACAAATTGACATTGTACAAAA -------AGTATTGGCAACAGGAGGCAGGAAAACTCAGGCAACAAATTGACATTGTACAAAA -------AGTACTGGCAGCAGGAGGCAGGAAAACTCAAGCAACAAATTGACATTGTACAAAA AATTCTCAGTACTGGCAACAGGAGGCAGGAAAACTCAGGCAGCAGATTGACATTCTACAAAA AATTCTCAGTACTGGCAACAGGAGGCAGGAAAACTGAGACAGCAAATTGATATTCTGCAAAA AATTCTCAGTACTGGCAACAGGAGGCAGGAAAACTGAGACAGCAAATTGATATTCTGCAAAA ! Klon 3 Klon 7 Klon 8 Klon 9 C. edentata G. biloba G. biloba *651 *700 TGCTAACAGACACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGC TGCTAACAGACACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGC TGCTAACAGACACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGC TGCTAACAGACACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGC TGCTAACAGACACCTAATGGGAGATGCACTCACATCTTTAAGCGTAAAGGAACTTAAGCAGC TGCAAATAGACACTTGATGGGGGACGCGCTTACATCTTTAAGTGTAAAGGAGCTTAAGCAGC TGCAAATAGACACTTGATGGGGGACGCGCTTACATCTTTAAGTGTAAAGGAGCTTAAGCAGC ! Klon 3 Klon 7 Klon 8 Klon 9 C. edentata G. biloba G. biloba *713 *750 TTGAAATTCGACTTGAAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTC TTGAAATTCAACTTGAAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTC TTGAAATTCGACTTGAAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTC TTGAAATTCGACTTGAAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTC TTGAAATTCGACTAGAAAGAGGCCTCAGCCGAGTACGATCAAAGAAGAATGAAATGCTGCTC TAGAAATTCGACTTGAGAGGGGCATTAGCAGGGTTCGATCAAAGAAGAATGAAATGTTGCTT TAGAAATTCGACTTGAGAGGGGCATTAGCAGGGTTCGATCAAAGAAGAATGAAATGTTGCTT ! ! Klon 3 Klon 7 Klon 8 Klon 9 C. edentata G. biloba G. biloba *775 *800 GAAGAGATAGAGATTATGC------------------------------------------GAAGAAATAGAAATAATGC------------------------------------------GAGGAAATAGAAATAATGC------------------------------------------GAAGAAATAGAAATAATGC------------------------------------------GAAGAGATCGAGATTATGCAAAGAAGGGAACACATATTACTAGCCGAGAATCAGTTTCTTCG GAGGAGATAGAGATTATGCAAAGAAGGGAACACATATTACTGGCGGAGAACCAGTTTCTTCG GAGGAGATAGAGATTATGCAAAGAAGGGAACACATATTACTGGCGGAGAACCAGTTTCTTCG ! Abb. 35: Nukleotidsequenz-Alignment der Klone 3, 7, 8 und 9 mit den MADS-box-C-Genen der nah verwandten Gymnospermen Cycas edentata L. und Ginkgo biloba L. Das Alignment zeigt einen Ausschnitt aus einem ca. 1200 bp (= GBM2-Gen von G. biloba) langen Alignment. Die übertragenen Nukleotidpositionen sind „*“ markiert; rot = für alle Sequenzen identische Nukleotide; pink = identische Sequenz der Klone mit C. edentata; hellblau = identische Klonsequenzen; grün = abweichende Nukleotidposition der Klone; „!“ = Übereinstimmung der Klone mit G. biloba ERGEBNISSE 75 Die In-silico-Analyse in den Datenbanken des „NCBI“ mit der so genannten „blastn“Suche, die nicht nur hoch ähnliche Sequenzen erfasst, führte darüber hinaus zu zahlreichen Übereinstimmungen der im Zuge dieser Arbeit gewonnenen Klonsequenzen mit MADS-box-C-Genen anderer Pflanzen (Abb. 36). Die größten Ähnlichkeiten sind nach den beiden oben erwähnten Gymnospermen G. biloba und C. edentata zu den Pinaceen zu finden, die ebenfalls Vertreter der Gymnospermen sind. Abb. 36: In-silico-Analyse zu den Nukleotidsequenzen der Klone eines MADS-box-C-Gens von Zamia amblyphyllidia D.W.STEV. Die Abbildung stellt einen Screenshot zur In-silico-Analyse im „NCBI“ dar. In rot umrandet sind alle Sequenzen, die von Gymnospermen stammen. Bei Betrachtung der Ergebnisse der In-silico-Analyse in Abbildung 36 fällt auf, dass eine MADS-box-C-Gen-Sequenz von C. edentata als höchste Übereinstimmung mit der Klonsequenz an erster Stelle angezeigt wird, die anderer aber erst gegen Ende der übrigen Gymnospermensequenzen aufgelistet ist. Das basiert auf der Ergebnisdarstellung. Es besteht zwar eine sehr hohe Übereinstimmung der Gensequenz im isolierten Bereich des MADS-box-C-Gens von Z. amblyphyllidia mit der AG-Sequenz von C. edentata („Max ident“ = 94%), jedoch liegt aufgrund der vielfachen Länge der C. edentata-Sequenz die Suchüberlappung („Query coverage“) bei nur 82%. ERGEBNISSE 76 3.2.1.4. SYNTHETISIERUNG DER SONDE Als Template für die Sondensynthese wurde das PCR-Produkt der M13-PCR von einem frisch abgenommenen Klon verwendet, der auf denselben Platten wie Klon 3, 7, 8 und 9 gewachsen ist. Von diesen Platten wurden mehrere Klone abgenommen und mittels einer Plasmidcharakterisierung, einer PCR mit einem Mix aus zahlreichen kurzen Primern, auf ihre Inserts hin überprüft, da man anhand des Bandenmusters in der Gelelektrophorese durch den Abgleich mit den charakteristischen Bandenmuster der bereits sequenzierten Klone auf das Insert der noch unbekannten Klone schliessen kann. Von den Klonen, die ein identisches Bandenmuster zeigten, wurde ein Klon ausgewählt und das Insert in einer M13-PCR vervielfältigt und zur Kontrolle zum Sequenzieren versandt. Dieser als Template für die Sondensynthese ausgewählte Klon 13 lässt sich problemlos mit den bekannten Klonen alignieren und zeigt keine vermehrten Abweichungen von diesen (Abb. 37). Klon Klon Klon Klon Klon 3 7 8 9 13 AGTACTGGCAACAGGAGGCAGGAAAACTCAGGCAACAAATTGACATTGTACAAAATGCTAACAGAC AGTACTGGCAGCAGGAGGCAGGAAAACTCAGGCAACAAATTGACATTGTACAAAATGCTAACAGAC AGTATTGGCAACAGGAGGCAGGAAAACTCAGGCAACAAATTGACATTGTACAAAATGCTAACAGAC AGTACTGGCAGCAGGAGGCAGGAAAACTCAAGCAACAAATTGACATTGTACAAAATGCTAACAGAC AGTACTGGCAGCAGGAGGCAGGAAAACTCAGGCAGCAAATTGACATTGTACAAAATGCTAACAGAC Klon Klon Klon Klon Klon 3 7 8 9 13 ACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGCTTGAAATTCGACTTG ACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGCTTGAAATTCAACTTG ACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGCTTGAAATTCGACTTG ACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGCTTGAAATTCGACTTG ACCTAATGGGAGATGCACTTACATCTTTAAGCGTAAAAGAACTTAAGCAGCTTGAAATTCGACTTG Klon Klon Klon Klon Klon 3 7 8 9 13 AAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTCGAAGAGATAGAGATTATGC AAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTCGAAGAAATAGAAATAATGC AAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTCGAGGAAATAGAAATAATGC AAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTCGAAGAAATAGAAATAATGC AAAGAGGCCTTAGCCGAGTAAGATCGAAGAAGAACGAAATGCTTCTCGAAGAAATAGAAATCATGC Abb. 37: Nukleotidsequenz-Alignment der Klone 3, 7, 8, 9 und 13 Das Alignment zeigt die gesamte Insert-Sequenz zu einem MADS-box-Gen der C-Funktionsklasse von Zamia amblyphyllidia; rot = für alle Sequenzen identische Nukleotide; grün = abweichende Nukleotidpositionen Die Synthetisierung der Sonde wurde wie unter 2.2.1.11. beschrieben vorgenommen, da es sich bei dem für die Klonierung verwendeten Plasmid um den pGEM-T-EasyVektor handelt. Bei diesem ist zwischen dem Insert und der Ansatzstelle für den Forward-M13-Primer die Promotorregion für die T7-Polymerase zur Synthetisierung der Sonde gelegen. Die Promotorregion für die SP6-Polymerase zur Synthese der Negativkontrolle, der so genannten Sense-Sonde, liegt im Plasmid zwischen dem Insert und der Ansatzstelle für den Reverse-Primer der M13-PCR (Abb. 38). ERGEBNISSE Vektor 77 M13_for T7 Vektor Insert Vektor Sp6 M13_rev Vektor Abb. 38: Schematische Darstellung des Plasmids im Bereich des Inserts Das Plasmid (= pGEM-T-Easy-Vector) ist in gelb dargstellt; rot = Ansatzstellen der M13-PCRPrimer; grün = Promotorregionen für die T7- bzw. SP6-Polymerasen; blau = Insert Die RNA-Gehaltsmessung in Anschluss an die Fällung und Lösung der Sonde ergab eine Sondenkonzentration von 230 µg/ml. Die Konzentration der Kontrolle lag sogar bei 350 µg/ml. Bei den späteren Sondensynthesen auf Basis desselben Templates wurden keine RNA-Gehaltsmessungen zur Konzentrationsbestimmung durchgeführt, sondern die für die In-situ-Hybridisierung einzusetzende Menge mittels des Dot-Blots (siehe 3.2.1.5.) ermittelt. 3.2.1.5. DOT-BLOT Beim Dot-Blot ermittelt man die für die In-situ-Hybridisierung einzusetzende Menge an Sonde, indem die Reaktion zwischen Sonde, Antikörpern und Färbelösung auf einem speziellen Filterpapier wie unter 2.2.1.12. beschrieben in vitro getestet wird. In Abbildung 39 ist eine solche Filtermembran nach erfolgter Reaktion dargestellt. Dieser Dot-Blot zur zweiten synthetisierten Sonde zeigte, dass die beiden Verdünnungen von 1:10 und 1:100 die besten Ergebnisse in 1: 10 1:1000 x der In-situ-Hybridisierung erwarten lassen, 1:100 1:10.000 wie anhand von den zwei dunklen Punkten zu erkennen ist. Die Detektionsreaktion mit der 1:1000 verdünnten Sonde fiel deutlich abgeschwächter aus. Bei der Verdünnung von 1:10.000 ist keine Farbreaktion mehr erkennbar. Um sparsam mit der erstellten Sonde bzw. Kontrolle zu arbeiten, ist in den Abb. 39: „Dot-Blot“ nach erfolgter Färbereaktion nachfolgenden In-situ-Hybridisierungen die Verdünnung von 1:100 oder maximal 1:50 eingesetzt worden. Nachdem auch diese Sonde nicht mehr zur Verfügung stand, ist eine neue Sonde synthetisiert worden, die ebenfalls in einer Konzentration von 1:100 in der In-situ-Hybridisierung zum Einsatz kam. ERGEBNISSE 3.2.2. 78 IN-SITU-HYBRIDISIERUNG – METHODISCHE ERGEBNISSE Die Übertragung des unveröffentlichten Protokolls „In Situ Hybridization Protocol using Digoxigenin-labeled probes on tissue sections (Stand April 2003)“ des Labors von DR. STEFAN GLEISSBERG am Institut für Spezielle Botanik der Universität Mainz von krautigen Angiospermen auf die stark mit sekundären Pflanzenstoffen belastete Gruppe der Gymnospermen machte einige Modifikationen in der Versuchsdurchführung erforderlich, die nun im Folgenden aufgezeigt und gegebenenfalls unmittelbar diskutiert werden, so dass der Ergebnisteil 3.2.2. im Kapitel 4 („Diskussion“) keine weitere Erwähnung findet. Die untenstehende Gliederung richtet sich der Einfachheit halber streng nach den Gliederungspunkten unter 2.2.2. („In-situ-Hybridisierung“) im Kapitel 2 („Material und Methoden“). 3.2.2.1. RNASEFREIE FIXIERUNG VON FRISCHMATERIAL Nach der Probenentnahme unter Kühlung und möglichst sterilen Bedingungen wurde für die ausstehenden In-situ-Hybridisierungen auf zwei verschiedene Weisen fixiert. Eine Methode ist die Paraformaldehyd-Fixierung nach ZACHGO (2002), die auch bei GLEISSBERG (2003) zum Einsatz kommt, die andere Methode stellt die seit Jahren bei morphologischen Untersuchungen bewährte Fixierung mit einem ebenfalls frisch und RNase frei angesetztem Formalin-Ethanol-Eisessig-Gemisch (FAA) dar. Für alle im Rahmen der vorliegenden Arbeit untersuchten Objekte hat sich die herkömmliche FAA-Fixierung bewährt, die jedoch durch die Kühlung der Proben durch Eis während des Anlegens eines Vakuums, das für ein schnelleres Eindringen des Fixatives sorgt, verfeinert wurde. Diese Fixierungsart ist mit am Lehrstuhl für Evolution und Biodiversität der Pflanzen grundsätzlich verfügbaren Substanzen durchführbar und zudem noch kostengünstiger als die Fixierung mit Paraformaldehyd, so dass sie bei gleichen Ergebnissen bei den In-situ-Hybridisierungen der Fixierung nach ZACHGO (2002) vorzuziehen ist. 3.2.2.2. ÜBERFÜHRUNG IN WACHS Das Hochführen ins Intermedium Roti-Histol zur Überführung in Wachs entsprechend des Protokolls (siehe 2.2.2.2.) verursachte trotz der zum Teil verdreifachten Inkubationszeiten während der Entwässerungsreihe keinerlei Probleme. Als problematisch stellte sich erst die Überführung in Wachs dar. Unabhängig von der angewandten Fixierung zeigten die Objekte im Paraplast Plus (TYCO HEALTHCARE/ KENDALL), dem bei GLEISSBERG (2003) angegebenen Histologiewachs, extreme Schrumpfungsartefakte und waren nicht mehr für die ausstehenden In-situ-Hybridisierungen zu nutzen. ERGEBNISSE 79 Daher wurde auf das herkömmliche Produkt zur Überführung von morphologischen Untersuchungsobjekten in Wachs, HISTOWAX der Firma HISTOLAB PRODUCTS AB, zurückgegriffen. Möglicherweise liegt der Grund für die Schrumpfungsartefakte nicht am Paraplast Plus an sich sondern am Wärmeschrank, in dem die Proben lagern. Der Wärmeschrank hat eine starke Schwankungsbreite von mehreren Grad Celcius um die eingestellte Temperatur von 58°C, bei der ein Erstarren des Paraplast Plus (Schmelzpunkt bei ca. 56°C) vermieden werden sollte. Ungünstigerweise darf laut Datenblatt des Herstellers Paraplast Plus im Gegensatz zu HISTOWAX (Schmelzpunkt bei ca. 58°C) keinen Temperaturen über 62°C ausgesetzt sein, da dann mit Veränderungen der Wachsstruktur gerechnet werden muss. Bezüglich der Übertragung der Proben in Glasbehälter und die damit einhergehende Überführung in Wachs wurden mehrere Methoden getestet. Beim Übertragen in die Glasbehälter wurden die Proben zuerst mit Roti-Histol bedeckt und anschließend ist entweder mit flüssigem Wachs oder mit Wachsflocken (Paraplast Plus) bzw. Wachskügelchen (HISTOWAX) aufgefüllt worden. Ebenso wurde beim täglichen Wechsel entweder mit geschmolzenem oder festem Wachs das abgeschüttete Wachs wieder aufgefüllt. Als die praktikabelste Methode erwies sich die im Protokoll nach GLEISSBERG (2003) aufgeführte Methode der Zugabe von flüssigem Wachs, da hier ein sehr schnelles Eindringen des Wachses gewährleistet ist. 3.2.2.3. ERSTELLUNG VON PARAFFINSCHNITTEN Die Paraffinschnitte für die In-situ-Hybridisierungen wurden am Rotationsmikrotom MOD 1130/ Biocut (REICHERT-JUNG) erstellt, an dem ebenfalls die Serienschnitte für die morphologischen Untersuchungen angefertigt werden. Abweichend wurde jedoch das Transportband abmontiert, um einer Verunreinigung der Proben mit RNasen vorzubeugen. Alle anderen Mikrotomteile wurden mit Chloroform gereinigt. Angefertigt wurden Einzelschnitte bzw. kurze Schnittbänder, die auf zweierlei Weise gestreckt wurden. Die erste Methode, die im Labor von Dr. Stefan Gleissberg zum Einsatz kommt, sieht folgendermaßen aus. Die Einzelschnitte werden in eine mit DEPCWasser gefüllte, sterile Wanne gegeben, die auf einer Wärmeplatte steht, so dass das Wasser angewärmt ist. Haben sich die Schnitte im Wasser gestreckt, werden sie nacheinander mit einem Objektträger aus dem Wasser gefischt und mitsamt des Objektträgers zum Trocknen auf die Wärmeplatte gelegt. Diese Methode bietet bei sehr kleinen Objekten den Vorteil, dass nur gezielte Schnitte aufgenommen werden. Bei größeren Objekten, wie sie im Rahmen dieser Arbeit genutzt wurden, ist man aber nicht auf Einzelschnitte angewiesen, da die wichtigen Strukturen oft in vielen aufeinander folgenden Serienschnitten zu erkennen sind. Aus diesem Grund kam bei ERGEBNISSE 80 der vorliegenden Arbeit nach anfänglichen Tests mit der eben geschilderten Methode die durch die morphologischen Untersuchungen vertraute Methode zum Tragen. Bei dieser werden die Schnittbänder auf Objektträgerlänge gekürzt oder mehrere kurze Stücke hintereinander auf die Objektträger gelegt, anschließend mit einigen Tropfen DEPC-Wasser versehen und zum Strecken auf einer Wärmeplatte (50°C) platziert. Zwischen Wärmeplatte und Objektträger wurde vorab eine sterile Glasplatte gelegt. Der Erfolg beider Methoden hängt entscheidend von der Wahl des verwendeten Objektträgers ab. Die Objektträger müssen mit Polylysin beschichtet sein, damit die Schnitte auf ihnen haften bleiben. Daher wurden spezielle, Polylysin-beschichtete Objektträger bestellt. Mit dem zuerst ausgewählten Produkt gab es insbesondere bei der zweiten Streckungsmethode Probleme, da die Objektträger derart beschichtet waren, dass das Wasser darauf keinen gleichmäßigen Film gebildet hat, sondern sich bei Kontakt mit dem Objektträger unmittelbar wieder tropfenförmig zusammengezogen hat. Aus diesem Grund war nicht nur die Streckung der Schnitte unmöglich, sondern stellte sich zudem das Problem, dass das Wasser beim Zusammenziehen die Schnitte mitzog, so dass diese umklappten, miteinander verschmolzen oder zum Teil zerrissen. Erst durch den Einsatz der Objektträger POLYSINETM der Firma ERIE SCIENTIFIC COMPANY, die die Adhäsionskräfte zwischen dem Objektträger und dem Wasser verstärken, waren diese Probleme behoben. 3.2.2.4. ENTPARAFFINIERUNG UND PRÄHYBRIDISIERUNG Die Entparaffinierung wie auch die absteigende Alkoholreihe zur Vorbereitung der Schnitte auf die eigentliche In-situ-Hybridisierung wurden nicht variiert, da diese Schritte vergleichbar mit der Lösungsreihe zur Entparaffinierung und Vorbereitung auf wässrige Lösungen von den morphologischen Untersuchungsmethode (siehe 2.1.2.) sind, die für die im Rahmen der In-situ-Hybridisierungen untersuchten Objekte erprobt ist. Abwandlungen waren allerdings bei der Proteinase K-Behandlung nötig. Diese Behandlung dient dem Andauen der Schnitte und soll wie auch der Einsatz von Triethanolaminen mit Acetanhydriden zur Erhöhung der Permeabilität der Zellmembranen ein problemloseres Eindringen der Sonde während der Hybridisierungsreaktion gewährleisten. Allerdings sind unter Verwendung Abb. 40: Blattgewebe nach der der Konzentrationsangaben von GLEISSBERG (2003) Behandlung mit Proteidie Schnitte zu stark angedaut worden, so dass das nase K ERGEBNISSE 81 Gewebe zum Teil völlig zerstört war (Abb. 40). Nach verschiedenen von der anfangs entsprechend des Protokolls verwendeten Konzentration von 10 µg/ml Proteinase K abweichenden Ansätzen erwies sich die Konzentration von 5 µg/ml Proteinase K als die geeigneteste Konzentration. Hierbei war ein einfaches Eindringen der Sonde in den Schnitt gegeben, ohne dass das Gewebe zu stark zerstört wurde. An der Triethanolamin-Lösung (100 mM) mit 0,5%-igen Essigsäure-Anhydriden ist wie auch an den darauf folgenden Lösungen einschließlich der aufsteigenden Alkoholreihe nichts weiter abgewandelt worden. 3.2.2.5. HYBRIDISIERUNG Die eigentliche Hybridisierung wurde strikt entsprechend des In-situ-HybridisierungsProtokolls von GLEISSBERG (2003) durchgeführt, da dies ein sehr sensibler Schritt im gesamten Versuch ist. Variiert wurden allerdings die Konzentrationen der Sonde von Verdünnungen zwischen 1:50 und 1:500 und das Volumen der auf die Objektträger gegebenen Sonden-Hybridisierungslösung-Mixe auf bis zu 400 µl, um das Versuchsergebnis unablässig zu optimieren. Darüber hinaus fand die Hybridisierungsreaktion anders als im Protokoll vorgegeben nicht in einem mit Parafilm abgedichtetem Gefäß aus Glas statt, sondern in einer dicht verschließbaren Box aus Kunststoff. Diese Box hat zwar den Nachteil, dass eine Sterilisation durch Heißluft nicht möglich ist und sie daher durch Chloroform sterilisiert werden musste, dafür ließ sich in dieser dicht verschließbaren Kunststoffbox hervorragend ein feucht-warmes Klima schaffen, wie es für die Hybridisierung nötig ist. Letztendlich ist noch anzumerken, dass die halbstündige Inkubation der Schnitte in den „Humid chamber“ vor Zugabe der Sonde nach GLEISSBERG (2003) eine Mindestangabe zu sein scheint, damit die Objektträger im feucht-warmen Klima in der Box mit 4x SSPE benetzt werden. Während einer Insitu-Hybridisierung wurde aufgrund äußerer Umstände diese Inkubationszeit von 30 Minuten um ein Vielfaches auf mehrere Tage gestreckt, während denen regelmäßig kontrolliert wurde, ob die Kammer noch feucht genug ist. Der Versuch wurde trotz dieser Unterbrechung gemäß dem Protokoll fortgesetzt und führte letztendlich zu einem positiven Ergebnis mit einem allerdings nur sehr schwachen Färbesignal. 3.2.2.6. ANTIKÖRPER-BEHANDLUNG Nach einigen In-situ-Hybridisierungen, die von der zeitlichen Abfolge der AntikörperBehandlung und Detektion den Angaben von GLEISSBERG (2003) entsprachen, wurde die Versuchsdauer derart gekürzt, dass in Anschluss an die 60-minütige Inkubation der Gewebeschnitte in einer feuchten Klimakammer bei Raumtemperatur zur Antikörper-Reaktion direkt mit der Detektion fortgefahren wurde. So wird die Lagerung ERGEBNISSE 82 der Proben über Nacht bei 4°C nach den vier 20-minütigen Waschschritten in Puffer 1 mit 0,1% Tween 20 umgangen. Da nach der Inkubation und den Waschschritten, die zur Vorbereitung auf den Folgeschritt in Puffer 2 mit 0,1% Tween20 dienen, direkt die Detektion folgt, sind die genannten vier Waschschritte im Abschnitt „Material und Methoden“ (Kapitel 2) dem Abschnitt „Detektion“ (siehe 2.2.2.7.) zugeordnet. Durch die Unterbindung der langen Lagerung der Gewebeschnitte über Nacht wurde nicht nur die Versuchsdauer um einen Tag gekürzt, sondern auch die Gefahr vermindert, dass die Objektträger und die darauf befindlichen Gewebeschnitte austrocknen oder auf andere Weise Schaden nehmen könnten. 3.2.2.7. DETEKTION Für die Detektionsreaktion, bei der ein Farbstoff spezifisch an die Antikörper bindet, wurden verschiedene Färbemöglichkeiten getestet. So wurde wie im In-situ-Hybridisierungs-Protokoll nach GLEISSBERG (2003) angegeben die Färbetablette „SIGMA FASTTM BCIP/NBT“ der Firma SIGMA-ALDRICH verwendet. Diese Färbetabletten sind auf unterschiedliche Weisen gelöst worden. So wurde zum einen je eine Tablette in 10 ml Polyvinylalkohol gegeben, da dieser Stoff als Reaktionsbeschleuniger für die Detektion gilt (ZACHGO 2002). Eine weitere Variante stellt das Lösen in 10 ml Aqua dest. dar, das die Handhabung der gelösten Tablette erleichtert, da es im Gegensatz zum Polyvinylalkohol nicht viskos ist. Zu guter Letzt wurde auch die vom „Dot Blot“ (siehe 2.2.1.12.) übernommene Lösungsweise in 10 ml Puffer 2 ausprobiert, deren Vorteil darin liegen kann, dass das Färbeprodukt in der Lösung ist, in der auch die zu detektierenden Gewebeschnitte kommen. Neben der „SIGMA FASTTM BCIP/NBT“Färbetablette ist auch die in dem „DIG Nucleic Acid Detection Kit“ der Firma ROCHE beinhaltete NBT/BCIP-Stocklösung entsprechend der dem Kit beiliegenden Anleitung verwendet worden. Von allen im Rahmen dieser Arbeit getesteten Detektionslösungen wurden mit der letztgenannten, d.h. der im Detektionskit enthaltenen Färbelösung, die schlechtesten Ergebnisse erzielt, da bei allen Proben keine Färbung erkennbar war. Am zweitschlechtesten schnitt die Methode der in Polyvinylalkohol gelösten Färbetablette ab, obwohl hier deutliche Färbereaktionen zu erkennen waren. Das Problem bei dieser Methode lag an anderer Stelle. Durch die starke Viskosität des Polyvinylalkohols hat sich die Färbetablette nicht vollständig gelöst, so dass kleine Klumpen in der Lösung verblieben, die auf die Objektträger gegeben wurde. Diese Klumpen führten zu teilweise sehr fleckigen Färbeergebnissen. Die besten Resultate wurden mit Aqua dest. und Puffer 2 als Lösemedium für die BCIP/NBT-Färbetablette erzielt. Der Einfachheit halber hat sich im Laufe der In-situ-Hybridisierungen das Lösen in Aqua dest. durch- ERGEBNISSE 83 gesetzt. Die Abbildung 41 zeigt die unterschiedlichen Farbergebnisse zweier In-situHybridisierungen anhand von Digitalaufnahmen jeweils eines Kontroll-Objektträgers und eines Objektträgers, der mit Sonde behandelt wurde. Dabei handelt es sich bei beiden Abbildungen (41A, B) um ein Versuchsergebnis nach Einsatz einer in Aqua dest. gelösten Färbetablette, wie auch anhand der gleichmäßigen Färberesultate zu erkennen ist. In diesem Falle beruht die extrem unterschiedliche Färbung also nicht auf dem zur Färbereaktion eingesetzten Medium, sondern auf einer Kombination aus der Menge der bei der Hybridisierung eingesetzten Sonde und der Einwirkzeit, die von Versuch zu Versuch variiert worden ist. A B Abb. 41: In der In-situ-Hybridisierung unterschiedlich intensiv gefärbte Gewebeschnitte Die Abbildung zeigt die Ergebnisse zweier In-situ-Hybridisierungen an Sporophyllen von Zamia amblyphyllidia - der Kontroll-Objektträger ist jeweils oben, der mit der Sonde behandelte Objektträger darunter; A) intensiv gefärbtes Hybridisierungsergebnis; B) schwach gefärbtes Ergebnis einer In-situ-Hybridisierung. Das Stoppen der Reaktion (siehe 2.2.2.8.) war ein unproblematischer Vorgang am Ende des mehrtägigen In-situ-Hybridisierungsversuchs, so dass nicht weiter darauf eingegangen wird, wie auch die Lichtmikroskopie und Dokumentation (siehe 2.2.2.9.) als eine Standardmethode der etablierten morphologischen Untersuchungen keiner weiteren Beachtung bedarf. ERGEBNISSE 3.2.3. 84 IN-SITU-HYBRIDISIERUNG – INHALTLICHE ERGEBNISSE Unter diesem Gliederungspunkt werden die Ergebnisse der In-situ-Hybridisierungen an verschiedenen Strukturen von Zamia amblyphyllidia anhand ausgewählter lichtmikroskopisch dokumentierter Gewebeschnitte dargestellt. 3.2.3.1. SPOROPHYLLE VON ZAMIA AMBLYPHYLLIDIA Die Abbildung 42 zeigt die Ergebnisse einer In-situ-Hybridisierung zu MADS-box-CGenen an Sporophyllen von Zamia amblyphyllidia. Aus diesem Versuch resultierte eine intensive Färbung der mit Sonde behandelten Gewebeschnitte. Die in Abbildung 43 dargestellten Hybridisierungsergebnisse zu Sporophyllen bei Z. amblyphyllidia zeigen eine weniger intensive Färbung. In Anbetracht des nur geringfügig weniger fortgeschrittenen Entwicklungsstandes der Sporophylle ist dennoch mit einer vergleichbaren Expressionsstärke zu rechnen. Die in Abbildung 42 gezeigten Sporophylle wurden Anfang Mai gesammelt, in FAA fixiert und in HISTOWAX überführt. Der Stand der Entwicklung ist mit den von Ende Dezember bis Mitte Januar für die morphologischen Untersuchungen gesammelten Sporophyllen vergleichbar (Abb. 16). Integument und Nucellus sind deutlich voneinander zu differenzieren, da sie nur in den basalen zwei Dritteln der Samenanlage miteinander verwachsen sind. Zudem ist die Makrospore bereits in ein vielkerniges Stadium übergegangen, ohne dass bislang eine Zellwandbildung stattgefunden hat. Eine Bestäubungskammer ist noch nicht ausgebildet. Dies ist das Stadium, in dem zahlreiche Entwicklungen sowohl in der Makrospore als auch im Nucellus initiiert werden. In der Makrospore wird in Kürze die Alveolenbildung einsetzen, nach der die Archegonienentwicklung beginnen kann. Vorher wird der Nucellus in die Mikropyle einwachsen und unterhalb dieser Region eine Bestäubungskammer ausbilden (Abb. 11). Die Abbildungen 42A und B zeigen jeweils einen Längsschnitt durch ein Sporophyll von Z. amblyphyllidia des oben beschriebenen Entwicklungsstandes. Das Sporophyll hat bereits eine Breite von gut 7 mm und eine Höhe von knapp 8 mm erreicht. Von der Höhe entfallen 60% auf die Mächtigkeit des Schildes des Sporophylls während die beiden Samenanlagen lediglich eine Länge von gut 3 mm bei einer Breite von maximal 2,5 mm erreicht haben. Die mit der MADS-box-C-Gen-Sonde behandelte Probe (Abb. 42A) zeigt eine deutliche Violettfärbung, die sich über den gesamten Längsschnitt erstreckt. Die Bereiche von besonders intensiver Färbung sind die Bereiche besonders hoher Zellteilungsaktivität (z.B. in der Makrospore), besonders großer Zelldichte (z.B. äußere Integumentschicht) und die Bereiche mit Zellen, die ERGEBNISSE 85 viele sekundäre Inhaltsstoffe enthalten (z.B. die bräunlich erscheinenden Haare am Sporophyll). Dieses Färbungsmuster ist auch in der Detailaufnahme der mit Sonde behandelten Samenanlage erkennbar (Abb. 42 C). Die Kontroll-Längsschnitte, die abgesehen von der Zugabe einer MADS-box-C-GenSonde wie die Proben (Abb. 42 A, C) behandelte wurden, zeigen keinerlei künstliche Färbung, so dass sie blass-bräunlich erscheinen (Abb. 42 B, D). Auch hier sind im Längsschnitt durch das gesamte Sporophyll die Bereiche größerer Zelldicht wie auch die Zellen mit sekundären Inhaltsstoffen deutlich dunkler, so dass das Schild des Sporophylls auch ohne eine histologische Färbung gut erkennbar ist (Abb. 42 B). In der sehr blassen Detailaufnahme der Samenanlage (Abb. 42 D) sind sowohl die äußere Integumentschicht als auch die Zellkerne der Makrospore erheblich schwerer auszumachen als in der Probe (Abb. 42 C). Die Detailbilder zu Ausschnitten aus den Integumentwänden zeigen die Unterschiede im Gewebe von Probe (Abb. 42 E) und Kontrolle (Abb. 42 F) auf zellulärer Ebene. Bei der Kontrolle sehen die Zellkerne blass-braun gefärbt aus, wohingegen sie bei der Probe eine intensiv violette Färbung angenommen haben. Auch das Cytoplasma, das bei der Kontrolle auch in der Detailaufnahme kaum erkennbar ist, erscheint in der Probe deutlich violett. Das in Abbildung 43 dargestellte Sporophyll von Z. amblyphyllidia stammt nicht vom selben Objektträger bzw. aus demselben In-situ-Hybridisierungsversuch wie das in Abbildung 42 gezeigte Sporophyll. Es wurde in einem anderen Versuchsdurchlauf hybridisiert, bei dem die Inkubationszeit der Färbereaktion abgekürzt worden war. Die Sammlung der Probe sowie ihre unmittelbare Fixierung mit Paraformaldehyd fanden Anfang November statt. Wie beim zuvor beschriebenen Sporophyll (Abb. 42) wurde als histologisches Paraffin HISTOWAX verwendet. Das Ende November gesammelte Sporophyll (Abb. 43) ist vom Entwicklungsstand her gut mit den Anfang Dezember für morphologische Untersuchungen gesammelten Sporophyllen gleichzusetzen (Abb. 15) und daher nur geringfügig jünger als das in Abbildung 42 gezeigte Sporophyll. Dieses erkennt man zum einen an der geringeren Größe des Sporophylls, das knapp 6 mm breit und nur rund 6 mm hoch ist, und den kleineren Samenanlagen, die erst 2 mm breit und gerade 2,7 mm hoch sind. Zum anderen ist bei diesen Samenanlagen auch die Makrospore in Relation zur gesamten Samenanlage kleiner. Mit einem Durchmesser von 600 µm macht sie gerade mal ein Drittel der Gesamtbreite der Samenanlage auf Höhe der Makrospore aus, wohingegen die Makrospore beim geringfügig weiter entwickelten Sporophyll schon einen Anteil von 45% an der Breite der Samenanlage erreicht. ERGEBNISSE 86 Unabhängig von den entwicklungsbedingten Unterschieden zwischen den beiden Sporophyllen aus Abbildung 42 und 43 und der stark abweichenden Farbintensität, sind die Hybridisierungsergebnisse bezüglich der Unterschiede zwischen der Probe und der Kontrolle sehr gut vergleichbar. Im Längsschnitt der mit der MADS-box-CGen-Sonde behandelten Probe ist eine nur schwache Violettfärbung des gesamten Sporophylls zu erkennen (Abb. 43A). Dennoch ergibt sich auch hier eine HellDunkel-Verteilung innerhalb des Sporophylls. Wie beim zuvor beschriebenen Sporophyll erscheinen die Bereiche mit besonders großer Zelldichte, die Bereiche mit hoher Zellteilungsaktivität und die eingelagerten sekundären Pflanzenstoffen deutlich dunkler im Vergleich zu den sehr hell erscheinenden großlumigen Zellen. Auch die Samenanlage, die mit der Sonde behandelt worden ist, zeigt diese Farbverteilung deutlich. Das Integument setzt sich aus einer dunkleren äußeren und einer helleren inneren Schicht zusammen und innerhalb der Makrospore sind die dunkel gefärbten Zellkerne erkennbar (Abb. 43 C). Die beschriebene Farbverteilung ist allerdings ebenfalls bei den Längsschnitten, die in der In-situ-Hybridisierung als Negativ-Kontrolle verwendet wurden, vorhanden (Abb. 43 B, D). Jedoch variiert die Färbung hier nicht von einem dunklen zu einem hellen Violett, sondern beschränkt sich auf die natürlicher Weise vorhandene Braunfärbung des gesamten Sporophylls. Kleine unregelmäßig geformte violette Flecken auf den Kontroll-Schnitten sind deutlich als aufgelagerte Strukturen erkennbar, die keine Farbreaktion innerhalb des Gewebes darstellen. Eine derartige Auflagerung ist auch in Abbildung 43 A an der rechten Samenanlage zu sehen. Die Farbunterschiede zwischen Probe und Kontrolle, die in den Längsschnitten durch das Sporophyll bzw. durch jeweils eine Samenanlage nur schwer zu erkennen sind, sind in den Detailaufnahmen (Abb. 43 E, F) gut ersichtlich. Die Detailaufnahmen des Integumentgewebes, aufgenommen von je einem als Probe (Abb. 43 E) und einem als Kontrolle (Abb. 43 F) behandelten Objektträger, zeigen die Färbungsunterschiede auf zellulärer Ebene deutlich. Wie bei der Kontrolle der oben beschriebenen In-situHybridisierung mit dem intensiven Färbeergebnis (Abb. 42) sind auch hier in dieser Kontrolle lediglich die Zellkerne als dunkle, braune Strukturen auszumachen (Abb. 43 F). Die Zellwände erscheinen sehr blass, das Cytoplasma ist fast nicht zu erkennen. Bei der Probe ist insbesondere das Cytoplasma deutlich violett angefärbt, so dass auch hier unabhängig von der Intensität der Färbung die Hybridisierung erfolgreich verlaufen ist (Abb. 43 E). ERGEBNISSE Abb. 42: Zamia amblyphyllidia D.W.STEV., In-situ-Hybridisierung (LM-Aufnahmen) A: Längsschnitt durch schwach entwickeltes Sporophyll Anfang Mai (Probe); B: Schwach entwickeltes Sporophyll Anfang Mai im Längsschnitt (Kontrolle); C: Längsschnitt durch die Samenanlage eines schwach entwickelten Sporophylls Anfang Mai (Probe); D: Längsgeschnittene, schwach entwickelte Samenanlage Anfang Mai (Kontrolle); E: Detail des Integumentgewebes der Probe; F: Detail des Integumentgewebes der Kontrolle (I = Integument; Ms = Makrospore; Nu = Nucellus; Sa = Samenanlage) 87 ERGEBNISSE Abb. 43: Zamia amblyphyllidia D.W.STEV., In-situ-Hybridisierung (LM-Aufnahmen) A: Längsgeschnittenes Sporophyll Anfang November (Probe); B: Längsschnitt durch ein Sporophyll Anfang November (Kontrolle); C: Samenanlage Anfang November im Längsschnitt (Probe); D: Längsgeschnittene Samenanlage Anfang November (Kontrolle); E: Detail des Integumentgewebes der Probe; F: Detailansicht des Integumentgewebes der Kontrolle (I = Integument; Ms = Makrospore; Nu = Nucellus; Sa = Samenanlage) 88 ERGEBNISSE 89 3.2.3.2. BLÄTTER VON ZAMIA AMBLYPHYLLIDIA In Abbildung 44 sind die Ergebnisse einer In-situ-Hybridisierung dargestellt, bei der längsgeschnittene Blattfiedern eines jungen Fiederblatts von Zamia amblyphyllidia untersucht wurden. Neben diesen Blattlängsschnitten wurden in der für die Abbildung 44 ausgewählten In-situ-Hybridisierung auch Längsschnitte von Sporophyllen von Z. amblyphyllidia hybridisiert. Da diese zu einem Hybridisierungsergebnis führten, das dem in Abbildung 43 gleicht, werden diese im Folgenden nicht dargestellt. Abbildung 44 A zeigt die Hälfte eines Längsschnitts durch ein junges Fiederblatt, das in der In-situ-Hybridisierung mit der MADS-box-C-Gen-Sonde behandelt wurde. Die Kontrolle, die abgesehen von der Zugabe der Sonde auf die gleiche Weise wie die Probe behandelt wurde, wird in Abbildung 44 B gezeigt. Bei beiden Schnitten ist das dünne Gewebe teilweise stark zerstört worden. Darüber hinaus ist sowohl bei der Probe als auch bei der Kontrolle keine Farbreaktion erkennbar. Beide Schnitte wirken zwar auf den ersten Blick leicht violett gefärbt, doch erweist sich dieser Eindruck bei genauerer Untersuchung als falsch (Abb. 44 A, B). Bei der Betrachtung der Detailabbildungen auf zellulärer Ebene, die jeweils von der Probe (Abb. 44 C) und der Kontrolle (Abb. 44D) einen Gewebeausschnitt im Bereich eines Leitbündels sowie ein Stück Epidermis zeigen, ist deutlich zu erkennen, dass keine dieser Zellen violett angefärbt ist. Weder die Zellkerne noch das Cytoplasma haben die violette Detektionslösung angelagert. Kräftig gefärbt erscheinen lediglich einige Zellen des Leitbündels, da sie sekundäre Pflanzenstoffe beinhalten. Jedoch handelt es sich in diesem Fall um eine Braun- und nicht um eine Violettfärbung. Die verdickten Zellwände der Epidermis wirken grau. ERGEBNISSE Abb. 44: Zamia amblyphyllidia D.W.STEV., In-situ-Hybridisierung (LM-Aufnahmen) A: Quergeschnittene Fieder eines jungen Blattes (Probe); B: Querschnitt durch eine junge Blattfieder (Kontrolle); C: Detailaufnahme eines Leitbündels und des umliegendes Blattgewebes einer Blattfieder im Querschnitt (Probe); D: Leitbündel mit umliegendem Gewebe einer quergeschnittenen Blattfieder im Detail (Kontrolle) (E = Epidermis; Lb = Leitbündel) 90 DISKUSSION 4. DISKUSSION 4.1. EVOLUTION DES INTEGUMENTS 4.1.1. DIE TELOMTHEORIE UND DIE MORPHOLOGIE DES INTEGUMENTS 91 Die Integumententwicklung ist im Rahmen der vorliegenden Arbeit am Beispiel der beiden Gymnospermen Zamia amblyphyllidia D.W.STEV. und Ginkgo biloba L. wie auch an der Angiospermen Magnolia stellata (SIEBOLD & ZUCC.) MAXIM in Hinblick auf die 1930 von ZIMMERMANN aufgestellte Telomtheorie untersucht worden. Wie bereits in der Einleitung (1.1.1.) beschrieben, besagt die Telomtheorie in Bezug auf die Samenanlagenentwicklung, dass das Integument im Laufe der Evolution aus sterilen Hülltelomen an der Basis von Sporangien durch Verwachsungsprozesse entstanden ist. Zahlreiche Rekonstruktionen fossiler Funde scheinen diese These zu stärken. Es ist allerdings schwierig dabei heraus zu finden, ob unbewusste und unbeabsichtigte Interpretationen in den sowohl handwerklich als auch gedanklich anspruchsvollen Rekonstruktionen stecken, da die bekanntesten Werke zu den frühesten fossilen Samenanlagen erst Jahrzehnte nach der Formulierung der Telomtheorie verfasst worden sind (ANDREWS 1963; GILLESPIE et al. 1981; VEGA & ARCHANGELSKY 1997 und andere). Jedoch stammt die Vorstellung, dass die Bildung des Integuments von der Basis der Samenanlage ausgeht, aller Wahrscheinlichkeit nach nicht allein von der Betrachtung fossiler Funde. Es scheint eher nahe liegend, dass die Darstellung der Samenanlagenentwicklung im Rahmen des Telom-Modells auf den damals bereits in großer Zahl vorliegenden Beobachtungen der Samenanlagenentwicklung bei Angiospermen basiert, da bei diesen zumindest die Entwicklung des äußeren Integuments deutlich erkennbar von der Basis der Samenanlage ausgeht. Ein bekanntes Werk, das gewiss auch schon ZIMMERMANN bei der Formulierung der Telomtheorie vorlag, stammt von COULTER & CHAMBERLAIN aus dem Jahr 1912 und befasst sich mit der Samenanlagenentwicklung bei Angiospermen u. a. am Beispiel von Capsella bursapastoris (L.) MED.. Die Arbeit von COULTER & CHAMBERLAIN dient auch heute noch als Vorlage für die Darstellung der Samenanlagenentwicklung bei Angiospermen in den meisten Lehrbüchern, da sich seit der Veröffentlichung vor knapp einem Jahrhundert Generationen von Standardlehrwerken auf diese beziehen. Die Entwicklung des Integuments bei den zwei untersuchten Gymnospermen Ginkgo biloba und Zamia amblyphyllidia erfolgt nicht durch eine Verwachsung von Integumentlappen, die an der Basis der Samenanlage gebildet werden. Die Beobachtung eines solchen Verwachsungsprozesses wäre jedoch innerhalb der Gymnospermen DISKUSSION 92 am ehesten bei diesen als ursprünglich angesehenen Gymnospermen und ihren eng verwandten Arten zu erwarten gewesen. Die monotypische Familie der Ginkgoaceae und die Ordnung der Cycadales, zu denen Zamia amblyphyllidia gehört, sind nämlich die einzigen rezenten Vertreter der Spermatophyta bei denen die Befruchtung über Spermatozoide erfolgt. Bei den untersuchten Arten handelt es sich beim Integument um eine ringförmige Struktur, die als ganzes angelegt wird und sich gleichmäßig weiterentwickelt, so dass die in Hinblick auf die Telomtheorie geforderten späteren Verwachsungsprozesse nicht auch nur im Ansatz erkennbar sind. Auch bei Arten, wo die Mikropyle in zwei Arme oder Lappen ausgezogen ist (z.B. Pinus oder Picea, MUNDRY 2000) oder sogar weiter aufgeteilt sein kann, wie beispielsweise bei Gnetum (MERSMANN 1998), handelt es sich bei der Ausformung der Mikropyle immer um eine ontogenetisch späte Aufteilung einer ursprünglich glatten, ringförmigen Integumentanlage. Darüber hinaus erfolgt die Differenzierung des Integuments bei beiden im Rahmen der vorliegenden Arbeit untersuchten Gymnospermen von der Spitze der Samenanlagenprimordien und nicht von ihrer Basis aus, wie das für ein Integument zu erwarten wäre, das entsprechend ZIMMERMANNs Telomtheorie aus an der Basis der Samenanlage stehenden Hülltelomen im Verlauf der Evolution entstanden sein soll. Bei Zamia amblyphyllidia ist zu diesem Zeitpunkt die Makrosporenmutterzelle bereits im Nucellus zu erkennen. Laut Untersuchungen von QUISUMBING (1925; zitiert nach SCHNARF 1937) setzt die Entwicklung der Makrosporenmutterzelle jedoch erst nach der Differenzierung des Samenanlagenprimordiums in Nucellus und Integument ein. Dieser Fall trifft auf Ginkgo biloba zu. Hier ist die Makrosporenmutterzelle im Nucellus erst nach der Initiierung des Integuments erkennbar. Für beide Arten trifft aber zu, dass die Makrosporenmutterzelle und das sich daraus entwickelnde Makroprothallium, von dem sich letztendlich der wachsende Embryo ernährt, innerhalb des Nucellus proximal vom Ansatzpunkt des Integuments gelegen sind. Die reifen Samen von Ginkgo und Zamia amblyphyllidia zeigen eine Dreischichtigkeit innerhalb des Integuments. Die innerste Schicht gleicht dem Nucellusgewebe und wird von Embryo als Nährgewebe aufgezehrt. Daran anschließend ist eine dünne, aber sehr harte Schicht, die so genannte Sklerotesta, zu finden. Die äußere Schicht entwickelt sich bei beiden Gymnospermen zu einer fleischigen Hülle, die als Sarkotesta bezeichnet wird. Eine ähnlich aufgebaute Hülle um den Embryo findet man auch bei den Taxaceae. Jedoch wird bei diesen die fleischige Schicht nicht aus einer Schicht des Integuments sondern aus einer separaten Struktur gebildet, die sich an der Basis der Samenanlage entwickelt und als Arillus bezeichnet wird. Bei der Gattung Taxus ist diese Struktur nicht mit dem Samen verwachsen, wohingegen bei der Gattung Cephalotaxus eine vollständige Verwachsung beider Strukturen zu beobachten ist (MUNDRY 2000). DISKUSSION 93 Ähnlich stellt sich die Integumententwicklung bei der Angiospermen Magnolia stellata dar. Anders als gemeinhin angenommen, konnte im Rahmen dieser Arbeit nicht gezeigt werden, dass beide Integumente an der Basis der Samenanlage entstehen. Ganz im Gegenteil dazu lassen die im Kapitel 3.1.3. gezeigten Ergebnisse für das innere Integument auf eine Entwicklung schließen, die sehr stark an die Entwicklung des Integuments der beiden untersuchten Gymnospermen-Arten erinnert. Auch die wenigen in der Literatur verfügbaren Arbeiten zur Samenanlagenentwicklung bei Magnolia widersprechen diesem Ergebnis nicht. Jedoch hat keiner der Autoren einen Vergleich zur Samenanlagenentwicklung bei Gymnospermen gezogen. DE BOER und BOWMAN (1972) beispielsweise geben nicht an, auf welcher Höhe des Samenanlagenprimordiums die Entwicklung des inneren Integuments initiiert wird, da sie vor allem daran interessiert sind, dass dessen Entwicklung im subdermalem Gewebe mit periklinen Zellteilungen beginnt und somit ein deutlicher Unterschied zur Entwicklung des inneren Integuments mit Ursprung in der dermalen Zellschicht bei Capsella bursa-pastoris vorliegt (ROTH 1957). Den Zeichnungen von DE BOER und BOWMAN ist jedoch zu entnehmen, dass das innere Integument in der Mitte des Primordiums der Samenanlage und nicht an der Basis entsteht. Deutlicher erkennbar ist dies anhand der lichtmikroskopischen Aufnahmen zur Samenanlagenentwicklung bei Magnolia grandiflora L. von UMEDA et al. (1994), die zudem die einzige Vergleichsbasis für die eigenen Ergebnisse bieten. Aus den Aufnahmen ist ersichtlich, dass die Entwicklung des inneren Integuments wie bei Magnolia stellata auf Höhe der Makrosporenmutterzelle beginnt. UMEDA et al. beschreiben diesen Entwicklungsursprung des inneren Integuments als das Erscheinen des inneren Integuments rings um den Apex des Samenanlagenprimordiums. Ein basaler Ursprung des inneren Integuments ist somit auch für Magnolia grandiflora auszuschließen. Lichtmikroskopische Aufnahmen zur Samenentwicklung bei der nicht zur Gattung Magnolia gehörenden Art Manglietia decidua Q.Y.ZHENG (Magnoliaceae) bestätigen die im Zuge dieser Arbeit gefundenen Ergebnisse bei Magnolia stellata ebenfalls (XIAO & XU 2006). Bei Magnolia stellata ist zum Zeitpunkt der Initiierung des inneren Integuments im Nucellus bereits die Makrospore zu erkennen. Sie liegt wie bei den beiden untersuchten Gymnospermen in etwa auf Höhe der Ansatzstelle des inneren Integuments. Bis zum Einsetzten der Bildung des äußeren Integuments hat sich die Makrosporenmutterzelle vergrößert, jedoch noch nicht geteilt. Soweit die oben genannten Arbeiten auch Bezug auf die Entwicklung des äußeren Integuments nehmen, belegen sie dessen basalen Ursprung. In Ergänzung dazu zeigen die eigenen Untersuchungen, dass das äußere Integument anders als das innere Integument nicht ringförmig sondern als ein am Funiculus unterbrochener Ring angelegt wird. Dies kann als Folge der zu diesem Zeitpunkt bereits weitgehend DISKUSSION 94 anatropen Lage der Samenanlage gesehen werden. Der Ursprungsort des äußeren Integuments entspricht der in Hinblick auf die Telomtheorie geforderten basal an der Samenanlage beginnenden Entwicklung. Jedoch fehlt wie beim inneren Integument eine Bildung aus „Lappen“ oder Telomresten. In ebenso deutlichem Widerspruch zur Telomtheorie steht aber der Anlegungszeitpunkt des äußeren Integuments, da auch die Telomtheorie verlangt, dass alle Strukturen an einer Achse in der Abfolge von unten nach oben angelegt werden. Auf die Samenanlagenentwicklung übertragen bedeutet dies, dass zuerst das äußere Integument, daraufhin das innere Integument und zum Schluss der Nucellus gebildet werden müssten. Allerdings wird das äußere Integument erst angelegt, nachdem die Entwicklung des inneren Integuments bereits eingesetzt hat. Diese offensichtliche Unstimmigkeit bleibt schon bei ANDREWS (1961), der die Telomtheorie ZIMMERMANNs auf die Evolution der Samenanlagen projizierte, unbeachtet. Insgesamt sind bei der Untersuchung der Entwicklung der Integumente von Magnolia stellata - abgesehen vom Entwicklungsursprung des äußeren Integuments an der Basis der Samenanlage - keinerlei Hinweise auf eine Verwachsung aus mehren Integumentlappen zu finden, so dass auch diese morphologische Entwicklungsreihe ebenfalls keine Indizien zur Stärkung eines Telomkonzeptes für die Entstehung des Integuments liefert. 4.1.2. DIE FUNKTION DES INTEGUMENTS Eine in der Wissenschaft unkritisch reflektierte These zur Funktion des Integuments besagt, dass das Integument die Samenanlage schützen soll. In den frühen Stadien, in denen die Integumentbildung einsetzt, scheint ein Schutz der Samenanlage durch das Integument jedoch aus zweierlei Gründen eher unwahrscheinlich. Zum einen ist die als Integument bezeichnete Struktur noch parenchymatisch weich, zum anderen werden die Nährstoffe in das Endosperm erst nach der Bestäubung oder sogar erst nach der Befruchtung eingelagert. Da also die Samenanlagen erst mit Nährstoffen ausgestattet werden, wenn die Entwicklung eines Embryos zu erwarten ist, macht dieser reduzierte Nährstoffaufwand einen besonderen Schutz der Samenanlage zum Zeitpunkt der Bestäubung überflüssig, da sie zu diesem Zeitpunkt nicht attraktiver für Herbivore als die übrigen Blütenbestandteile ist. Hingegen ist es biologisch sinnvoll, den reifen Samen zu schützen. Dies kann am einfachsten durch die zu diesem Zeitpunkt äußerste Schicht übernommen werden. Erst der reifende Samen wird durch das Integument geschützt, wenn er in etwa seine endgültige Größe erreicht hat. Die schützende Sklerenchymschicht umschließt dabei immer den Embryo und dessen Nährgewebe. Diese harte Samenschale kann die äußerste Schicht sein, sie kann aber auch - wie bei den Ginkgoales, den Cycadales und den Magnoliales – von einer fleischigen, der zoochoren Ausbreitung dienlichen Schicht, der Sarkotesta, umgeben DISKUSSION 95 sein. Es ist daher zu fragen, ob nicht andere Ursachen und funktionelle Zwänge zur Entwicklung des Integuments geführt haben. Sicherlich besteht eine erste und entscheidende Funktion des Integuments bei den rezenten Gymnospermen in der Exponierung des Bestäubungstropfens, der bei den meisten Gymnospermen zur Pollenaufnahme notwendig ist. Anders als Angiospermen bilden Gymnospermen keine Narbe als rezeptives Gewebe aus, da die Narbe eine Bildung der Fruchtblätter ist, die wiederum nur bei den Angiospermen zu finden sind. Bei den Gymnospermen wird der Pollen daher in der Regel durch dem Bestäubungstropfen aufgenommen. Zieht sich der Bestäubungstropfen durch Resorption oder Austrocknung in die Mikropyle zurück, nimmt er den aufgenommenen Pollen mit. Dabei wird nicht nach der Art des Pollens selektiert. Daher ist es vorteilhaft, den Bestäubungstropfen so groß wie möglich zu gestalten und so lange wie möglich zu exponieren, um die Wahrscheinlichkeit zu erhöhen, arteigenen Pollen aufzunehmen. Zum einen besteht der Bestäubungstropfen aus einer durch einen geringen Gehalt an Zuckern leicht viskosen, wässrigen Lösung, die das Zerfließen des Tropfens bis zu einer gewissen Tropfengröße verhindert. Zum anderen ist mit der trichterförmigen Ausformung der Mikropyle durch das Integument eine Struktur geschaffen worden, die dem Bestäubungstropfen einen zusätzlichen Halt verleiht. Würde der Tropfen, der zum größten Teil aus der Zellflüssigkeit der zur Bildung der Bestäubungskammer aufgelösten Zellen besteht, direkt aus dem Nucellus austreten, wäre es der Pflanze nur möglich, einen sehr kleinen und vor allem sehr flachen Tropfen zu halten. Hinzu kommt, dass die Nucellusränder der lysogenen Öffnungszone äußert unregelmäßig geformt sind und daher auch keine ausgeprägte Adhäsionszone zwischen dem Bestäubungstropfen und der Samenanlage bieten wie es eine glatte Struktur tun würde. Daher ist es nicht unwahrscheinlich, dass im Laufe der Evolution eine glatte, starre Konstruktion rings um den sich lysogen öffnenden Bereich des Nucellus entwickelt wurde, die schon vor dem Austreten des Bestäubungstropfens gebildet wird, damit sie diesen sofort stützen kann. Mit Hilfe einer solchen Konstruktion kann der Bestäubungstropfen deutlich größer und runder gestaltet und auch länger und somit erfolgreicher exponiert werden. Nicht nur durch die trichterförmige, starre Ausbildung der Mikropyle sondern auch durch die Wendung der gesamten Samenanlage wurden verschiedene Wege zur Exponierung von besonders großen Bestäubungstropfen geschaffen. Durch die Umwendung der Samenanlage wird der Bestäubungstropfen zusätzlich in einen vor Wind und Verdunstung geschützten Raum verlagert. Im Zuge der Evolution sind verschiedenartige Ausbildungen der Mikropyle geschaffen worden wie beispielsweise die papillöse Mikropyle der Lacrioideae oder Mikropyle von Pinus, die zwei Ärmchen ausbildet. Die große Radiation dieses Organs ist ein starkes Indiz für die enorme Bedeutung, die das Integument für den Bestäubungsvorgang bei den DISKUSSION 96 Gymnospermen einnimmt. Daraus darf allerdings nicht zwangsläufig geschlossen werden, dass das Integument zur Bildung einer Haltestruktur für den Bestäubungstropfen zielgerichtet evoluiert wurde. Ein weiterer Denkansatz zur funktionellen Evolution des Integuments befasst sich mit dem Öffnungsmechanismus des Nucellus, der mit dem Makrosporangium der Farne zu homologisieren ist. Die Sporangien der Farne öffnen beispielsweise über AnulusZellen. Bei einigen rezenten Pteridophyten wie z.B. Trichomanes sind diese Zellen ringförmig um das distale Ende des Sporangiums angeordnet. Da solche Strukturen in den Samenanlagen der rezenten Gymnospermen aber nicht einmal rudimentär zu erkennen sind, ist zu schlussfolgern, dass eine andere Struktur die Öffnungsfunktion übernommen haben muss, damit dem Pollen bzw. den aus dem Pollen entlassenen Spermatozoiden ein Weg zu den sich im Sporangium entwickelnden Archegonien frei gehalten wird. Die Funktion des Integuments könnte also bei den ursprünglichen Samenanlagen darin gelegen haben, als Ergänzung zum extrem weichen Gewebe des Nucellus eine starrere Struktur zu bilden, die mittels einer speziellen Öffnung (= Mikropyle) den Kontakt zur Außenwelt sichert. Dieser Kontakt ist dadurch notwendig geworden, dass anders als bei den Farnen keine Sporen aus den Sporangien mehr entlassen wurden und außerhalb keimten, sondern die ganze Sporenentwicklung mit Makroprothalliumsentwicklung und Bildung der Archegonien in stark reduzierter Form in den Nucellus verlagert worden ist. Durch diese Verlagerung ins Innere der Pflanze musste gewährleistet sein, dass die Mikrosporen ebenfalls in den Nucellus gelangen können. 4.1.3. ÄUßERES INTEGUMENT UND EPIMATIUM IM VERGLEICH Bei der Betrachtung der Entwicklung des äußeren Integuments von Magnolia stellata fällt auf, dass diese sehr stark an die Entwicklung vom Epimatium (Podocarpaceae) erinnert. Das Epimatium ist wie auch der Arillus der Taxaceae bzw. Phyllocladaceae eine oftmals fleischige und intensiv gefärbte Hüllstruktur, die die Samenanlage zum Teil oder auch vollständig umgibt. Die Ontogenese der weiblichen Zapfen einiger Podocarpaceen wurde bereits eingehend im Rahmen einer Diplomarbeit und einer Dissertation am Lehrstuhl für Evolution und Biodiversität der Pflanzen untersucht (RIEGER 2002, RESTEMEYER 2002). In Bezug auf die Ähnlichkeit der Entwicklung des Epimatiums zum Wachstum des äußeren Integuments von Magnolia stellata sind zwei Arten der Podocarpaceae hervorzuheben: Lagarostrobus franklinii (HOOK f.) QUINN und Saxegothaea conspicua LINDL. Diese beiden Arten stehen jeweils in einer monotypischen Gattungen. Bei Lagarostrobus franklinii handelt es sich um einen langsam wachsenden mittelgroßen Baum, der in Tasmanien beheimatet ist. Die Art DISKUSSION 97 Saxegothaea conspicua kommt im Süden Chiles sowie im angrenzenden Gebiet zu Argentinien vor und wächst zu kleinen bis mittelgroßen Bäumen aus (FARJON 1998). Wie auch bei der Initiierung des äußeren Integuments von Magnolia stellata beginnt die Entwicklung des Epimatiums bei den beiden oben genannten Podocarpaceen mit einer mehr oder weniger ringförmigen Struktur an der Basis der Samenanlage, die zu diesem Zeitpunkt bei den drei verglichenen Arten eine beginnende Differenzierung in Nucellus und Integument zeigt. Dieser Auswuchs an der Basis der Samenanlage ist zu Beginn noch nicht ringförmig. Bei Magnolia stellata ist dieser Ring im Bereich des Funiculus unterbrochen (vgl. Abb. 24D). Bei beiden Podocarpaceen ist dieser Ring in der Zone abgeplattet, in der die Samenanlage der Samenschuppe anliegt. Abbildung 45 zeigt eine sehr junge, kaum differenzierte Samenanlage von Saxegothaea conspicua auf der aus dem Zapfen freipräparierten Samenschuppe für ein besseres räumliches Verständnis in der Übersicht (RESTEMEYER 2002). In Abbildung 46 ist ein Ausschnitt aus der vollständigen morphologischen Reihe zur Anlegung der Samenanlagen von Lagarostrobus Abb. 45: Samenschuppe von franklinii aus der Dissertation von RESTEMEYER Saxegothaea conspidargestellt. Vergleicht man diese rasterelektronencua LINDL., REM (Quelle: RESTEMEYER 2002) mikroskopischen Aufnahmen mit den Aufnahmen zur Entwicklung der Samenanlagen bei Magnolia stellata (Abb. 24, 25), ist die verblüffende Ähnlichkeit dieser Entwicklungen nicht zu übersehen. E E E I I Nu Nu I Abb. 46: Morphologische Reihe zur Entwicklung der Samenanlagen bei Lagarostrobus franklinii (HOOK f.) QUINN, REM Die in der Aufsicht erkennbaren Strukturen sind in den übernommen Abbildungen C-E jeweils von außen nach innen: Epimatium (E) Integument (I) Nucellus (Nu) (Quelle: RESTEMEYER 2002, Beschriftung ergänzt) DISKUSSION 98 Zur Klärung der nun zwangsläufig aufkommenden Frage, inwieweit das Epimatium von Lagarostrobus franklinii bzw. auch von Saxegothaea conspicua mit dem äußeren Integument von Magnolia stellata vergleichbar ist, müssen die Funktionen der beiden Strukturen geklärt sein. Hierbei wird es schwierig, da zur Funktion beider Strukturen nur sehr unvollständige Daten in der Literatur vorliegen. Im Allgemeinen wird aber angenommen, dass das äußere Integument bei Magnolia stellata als Attraktion für samenausbreitende Tiere dient. Dafür spricht neben der auffällige Färbung und der fleischigen Beschaffenheit auch die Präsentation der reifen Samen außerhalb des Karpells (vgl. Abb. 12C). Darüber hinaus unterstützt die im Rahmen dieser Arbeit untersuchte Samenanlagenentwicklung bei Magnolia stellata die Annahme, dass das äußere Integument maßgeblich an der Umwendung der Samenanlage beteiligt ist. Besonders die letzte Funktion lässt sich nach Auswertung der Entwicklungsreihen von Lagarostrobus franklinii und Saxegothaea conspicua ebenso auf das Epimatium projizieren. Betrachtet man das äußere Integument der Angiospermen als eine dem Epimatium der Podocarpaceen homologe Struktur, würde das bedingen, dass beide Strukturen auf eine gemeinsame Vorläuferstruktur zurückzuführen sind, die vor der evolutiven Trennung von Gymnospermen und Angiospermen existiert haben muss. Ebenso ist denkbar, dass die „Hülle“ um die Samenanlage aus einer konvergenten Entwicklung resultiert. Allein bei den Gymnospermen muss eine Hüllstruktur in Form vom Arillus der Taxaceae, dem Arillus der Phyllocladaceae und dem Epimatium der Podocarpaceae mehrfach im Evolutionsverlauf entwickelt worden sein, da diese drei Familien innerhalb der Systematik der Gymnospermen deutlich getrennt voneinander stehen. So erscheint es mehr als wahrscheinlich, dass auch das äußere Integument der Angiospermen keine abgeleitete Struktur darstellt, sondern als ein Neuerwerb dieser Gruppe zu betrachten ist. So ist innerhalb der Angiospermen eine derartige Struktur ein zweites Mal quasi als drittes Integument, wie z.B. der fleischige Arillus der Passiofloraceen, entstanden. Trotz der verblüffenden, aber wahrscheinlich funktionsbedingten Ähnlichkeit, also nicht zwangsläufig von einer Homologie auszugehen. 4.1.4. EIN NEUES KONZEPT ZUR EVOLUTION DES INTEGUMENTS Die Untersuchungen zur Integumententwicklung an den zwei Gymnospermen-Arten Ginkgo biloba und Zamia amblyphyllidia, die in unterschiedlichen je ursprünglichen Ordnungen der Gymnospermen stehen, sowie an der über zahlreiche ursprüngliche Merkmale verfügenden Angiospermen Magnolia stellata, ergeben keine Bestätigung für die Telomtheorie nach ZIMMERMANN bezüglich der Entwicklung der Integumente als ein Verwachsungsprodukt an der Basis der Samenanlage stehender Hülltelome. Aus diesem Grund soll im Folgenden ein anderer denkbarer Evolutionsweg für die Struktur „Integument“ aufgezeigt werden. Bei diesem Denkansatz wird davon ausge- DISKUSSION 99 gangen, dass die im Laufe der Ontogenese nachvollziehbaren Entwicklungsschritte auf vergleichbare Weise ebenfalls im Laufe der Evolution durchlaufen worden sind. Dem entsprechend ist der evolutive Ursprung des Integuments nicht in einer basalen Hüllstruktur der Samenanlage zu suchen, sondern handelt es sich beim Integument vielmehr um eine Bildung des Nucellus. Ausgangsform war vermutlich eine anulusartige Struktur wie sie rezent in hoch spezialisierter Form bei einigen Pteridophyten, z.B. innerhalb der Ordnung Schizaeales, zu finden ist. Eine vergleichbare ringförmige Struktur kann verhindern, dass die lysogen entstandene Sporangienöffnung durch das Einsinken der weichen, parenchymatischen Ränder wieder verschlossen wird. Durch eine entsprechende Ausformung des Randes erlaubt dieser die Bildung sehr großer Bestäubungstropfen. So gesehen ist die Mikropyle ein Teil der Öffnungsstruktur des Makrosporangiums, das bei den Spermatophyten als Nucellus bezeichnet wird. Zum Zeitpunkt, zu dem die Mikropyle gebildet wird, ist bereits eine Mehrschichtigkeit des Integuments zu erkennen. Interessanter Weise wird die Mikropyle nur von der Hüllstruktur, nicht aber von der späteren Nährschicht gebildet (vgl. Abb. 16A,C oder 20F). Dabei bildet der innen liegende Bereich der Mikropyle, der zu der Struktur umgeformt wird, die den Bestäubungstropfen hält, eine extrem glatte Oberfläche und somit eine gute Adhäsionszone für den Bestäubungstropfen aus. Zudem ist dieser Bereich die einzige hydrophile Zone der Epidermis der Samenanlage. Ansonsten ist die Samenanlage wie auch alle übrigen Teile des Zapfeninneren hydrophob, so dass der Bestäubungstropfen die angrenzenden Oberflächen nicht benetzt, sondern als ein Tropfen in der Mikropyle akkumuliert. Die oben beschriebenen, für die Evolution der Samenanlage postulierten Vorgänge lassen sich anhand der Ontogenese der untersuchten Arten Zamia amblyphyllidia, Ginkgo biloba und Magnolia stellata nachvollziehen. So bildet der Nucellus bei allen Arten am distalen Ende eine ringförmige Struktur aus, die im weiteren ontogenetischen Entwicklungsverlauf die ursprüngliche Nucellusspitze übergipfelt und auch die Mikropyle bildet. Die beschriebenen Vorgänge, die sich auf den distalen Bereich der Samenanlage beschränken, lassen sich in der Aufsicht nachvollziehen. Weitere Differenzierungsvorgänge innerhalb der Samenanlage, die sich in basaler Richtung erstrecken, können nur anhand lichtmikroskopischer Untersuchungen nachvollzogen werden. Durch diese wird deutlich, dass auch in dieser Richtung eine Umbildung des äußeren Nucellusgewebes derart satt findet, dass dieses Gewebe histologisch dem Gewebe im Mikropylenbereich gleicht. Dementsprechend sind in der Ontogenese die Integumente von Zamia amblyphyllidia und Ginkgo biloba wie auch das innere Integument von Magnolia stellata durch eine Differenzierung des Nucellus entstanden. Aus dieser Tatsache lässt sich schließen, dass der evolutive Weg zum Integument ebenfalls über eine Umbildung des Nucellus bzw. des Makrosporangiums verlaufen DISKUSSION 100 ist. Die Schemazeichnung in Abbildung 47 verdeutlicht die Beteiligung des Nucellus an der Entwicklung des Integuments. A B C Abb. 47: Schemazeichnung zur Entwicklung des Integuments A: Die Initialisierung des Integuments (grün) erfolgt am distalen Ende des Nucellus (orange); B: Integumentbildung durch Ausdifferenzierung des äußeren Nucellusgewebes in basaler Richtung; C: Relationen von Nucellus (orange), Sklerotesta (schwarz) und Sarkotesta (grün) an einer reifen Samenanlage Ein weiteres Indiz für die oben aufgestellte These bietet der reifende Samen. Anhand von längs halbierten Samen lassen sich – insbesondere nach kurzer Antrocknung – die einzelnen Integumentschichten deutlich von einander unterscheiden. Eine solche Samenanlage zum Zeitpunkt der Bestäubungsreife von Zamia amblyphyllidia ist in der Abbildung 48 gezeigt. Die Abbildung 48A dient der Übersicht. In Abbildung 48B hingegen sind die entscheidenden Strukturen, auf die im Folgenden eingegangen wird, farbig markiert. Wie in der Literatur z.B. bei SMITH 1910 beschrieben ist das Integument in eine fleischige Sarkotesta (in grün markiert) und eine harte Sklerotesta (Markierung in rot) differenziert. Diese beiden Schichten bilden das Integument einer reifen Samenanlage. Bei der in Abbildung 48 gezeigten Samenanlage zum Zeitpunkt der Bestäubung ist überall im Bereich, in dem das Integument nicht mit dem Nucellus verwachsen ist, eine dritte Integumentschicht (in gelb markiert) zu erkennen. Diese dritte Schicht ist nach innen gelegen und ragt nicht bis in die Mikropyle hinein. Rein histologisch ist sie nicht vom Nucellusgewebe zu unterscheiden. Da sie während der weiteren Entwicklung der Samenanlage vom Embryo als Nährgewebe aufgebraucht wird, unterscheidet sie sich auch in ihrer Funktion nicht vom restlichen Gewebe des Nucellus. Aufgrund der funktionellen und histologischen Übereinstimmung von der in Abbildung 48B gelb markierten dritten Integumentschicht und dem Nucellus ist davon auszugehen, dass es sich bei dieser Schicht um eine Bildung des Makrosporangiums und nicht um eine separate Struktur handelt. Betrachtet man die Herkunft dieser einen Integumentschicht, die sich zunächst nicht vom übrigen Integument unterscheidet, ist davon auszugehen, dass es sich auch bei der Sarkotesta und der Sklerotesta um keine separate Struktur sondern ebenfalls um eine Bildung des Nucellus handelt. DISKUSSION A 101 B Abb. 48: Längshalbierte Samenanlage von Zamia amblyphyllidia D.W.STEV. zum Zeitpunkt der Bestäubungsreife A) Bestäubungsreife Samenanlage differenziert in Integument (I) und Nucellus (N) mit Makroprothallium (Mp) sowie Bestäubungskammer (Pfeil) B) Samenanlage wie bei A) mit farbig hervorgehoben Integumentschichten: grün = Sarkotesta; rot = Sklerotesta; gelb = dem Nucellus histologisch gleichende Nährschicht für den Embryo Basierend auf der Ontogenese, Histogenese und Morphologie des Integuments ist unerlässlich ein neues Konzept zur Evolution der Samenanlage aufzustellen. Anders als bisher angenommen handelt es sich beim Integument nicht um eine separate Struktur, die an der Basis der Samenanlage gebildet wird und diese umwächst, sondern um eine Struktur, die ihren Ursprung an der Spitze des Nucellus hat und sich gänzlich aus diesem entwickelt. Darüber, weshalb die Evolution überhaupt ein Integument hervor gebracht hat, lässt sich nur spekulieren. Als sehr wahrscheinlich erscheint es aber, dass nach dem Sesshaft werden des Makrosporangiums an der Mutterpflanze ein System hervorgebracht werden musste, mit dessen Hilfe die Pollenkörner auch unabhängig vom atmosphärischen Wasser am Sporangium haften blieben und die zur Keimung notwendigen feuchten Bedingungen vorfanden. Diese wäre durch eine lysogene Öffnungsweise des Sporangiums, das bei den Spermatophyta als Nucellus bezeichnet wird, umsetzbar. Die Bildung einer lysogenen Öffnung des Sporangiums alleine dürfte aber unzweckmäßig gewesen sein, da das dünne Gewebe wieder in sich zusammenfällt und die gebildete Öffnung somit selbst wieder verschließen würde. Aufgrund dessen ist anzunehmen, dass - möglicherweise aus einer gemeinsamen Vorläuferstruktur des Anulus der Pteridophyten abgeleitet - eine Struktur entstanden ist, mit der das dünne Gewebe im Öffnungsbereich stabilisiert werden konnte. Aus dieser stabilisierenden Struktur hat sich im Laufe der Evolution das rezente Integument durch Differenzierungsvorgänge in den äußeren Schichten des Nucellusgewebes entwickelt. DISKUSSION 102 Einhergehend mit dem oben beschriebenen Konzept zur Evolution der Samenanlage müssen zahlreiche fossile Funde nochmals neu interpretiert und teilweise auch neu untersucht werden. Die eingangs im Kapitel 1.1.2. beschriebenen fossilen Strukturen „Cupula“ und „Lagenostom“ bzw. „Salpinx“ stehen aber nicht im Widerspruch zum im Rahmen dieser Arbeit neu entwickelten Evolutionskonzept. Vielmehr können diese fossilen Rekonstruktionen zur Untermauerung des Konzepts herangezogen werden. Wie schon durch TAYLOR (1981) und NIKLAS (1982) beschrieben, sind bei zahlreichen primitiven, fossilen Samenanlagen Modifikationen am Makrosporangium zu finden, die der Aufnahme des windverbreiteten Pollens dienlich zu sein scheinen. Sind diese Auswüchse becher-, röhren- oder glockenförmig ausgebildet, werden sie als Salpinx bezeichnet. Besteht die Struktur zur Aufnahme des Pollens aus der so genannten Mittelsäule und einem umgebenden Becher bzw. Ring, spricht der Paläobotaniker von einem Lagenostom (z.B. STEWART 1983). Beide Strukturen, Lagenostom und Salpinx, lassen sich als unterschiedliche Entwicklungsstufen der Integumentevolution in das neue Konzept integrieren. Die Salpinx stellt dabei eine sehr ursprüngliche Öffnungsstruktur dar, die eher als eine primitive Struktur zur Stabilisierung der lysogen entstandenen Makrosporangienöffnung anzusehen ist. Eine etwas weiter entwickelte Ausformung der Öffnungsregion stellt das Lagenostom dar, wenn die „Mittesäule“ als Nucellusspitze interpretiert wird, die von einer stabilisierenden Struktur umgeben wird. Die „becher- oder ringförmige“ Bildung des fossilen Lagenostoms, die an der Spitze des Makrosporangiums steht und die Mittelsäule umgibt (siehe auch STEWART & ROTHWELL 2001 oder GIFFORD & FOSTER 1996), entspricht im Rahmen des neuen Evolutionskonzepts der Mikropyle der rezenten Spermatophyta (Abb. 49). In der obigen Ausführung zur Interpretation fossiler Samenanlagen werden bereits die fossilen Makrosporangien mit einer Salpinx oder einem Lagenostom am distalen Ende als Samenanlagen interpretiert. Diese Interpretation steht im Widerspruch zu der in der Paläobotanik anerkannten Definition für die primitiven, fossilen Samenanlagen ähnlichen Strukturen, die sich auf die Hüllstruktur um ein Makrosporangium stützt (ROTHWELL & SCHECKLER 1988). Löst man sich von dem Gedanken, dass das Integument aus einer mehr oder weniger verwachsenen Hüllstruktur an der Basis eines Makrosporangiums entstanden ist, lassen sich diese Strukturen ebenfalls neuartig interpretieren. Dabei verliert beispielsweise die von LONG (1959, 1960a, 1960b) aufgestellte hypothetische Entwicklungsreihe zur Bildung des Integuments nicht an Bedeutung. Es muss lediglich davon ausgegangen werden, dass es sich bei der aus mehreren Lappen verwachsenen Struktur an der Basis des Makrosporangiums nicht um ein Integument handelt, sondern vielmehr um eine Struktur, die die aus Makrosporangium und Salpinx bzw. Lagenostom bestehende Samenanlage umgibt. Diese Struktur könnte ein sehr primitiver Vorläufer der Blütenhülle der Angiospermen sein. DISKUSSION 103 Betrachtet man den u.a. von LONG beschrieben Komplex aus Samenanlage und Hüllstruktur als Samenanlage mit einer primitiven Blütenhülle, d.h. als eine primitive eingeschlechtige Blüte, muss in dem Zug auch die Cupula weitergehend interpretiert werden. ROTHWELL & SCHECKLER (1988) definieren die Cupula als eine Struktur, die aus einem verzweigten Achsensystem hervorgeht und die Samenanlagen becherförmig einzeln oder in Gruppen vollständig oder nur zum Teil einhüllt. Diese Struktur kann dabei sehr vielfältig gestaltet sein. Bei fossilen Arten, bei denen von der Cupula nur eine einzelne „primitive Blüte“ umhüllt wird, lässt sich unter Betrachtung der rezenten Blüten die Cupula problemlos als der erste Schritt zu einer zweiten Blütenhülle interpretieren, um beispielsweise die Attraktivität der „Blüte“ zu erhöhen. Ein funktioneller Zusammenhang ist z.B. bei der 1944 von EMBERGER beschriebenen Cupula von Lagenostoma lomaxi herzustellen, denn diese Cupula ist möglicherweise als Anlockung für potentielle Bestäuber auf der Außenseite mit zerstreut verteilten Drüsen besetzt. Die Interpretation der Cupula als zweite Blütenhülle lässt sich auch auf die Fossilien übertragen, bei denen die Cupula nicht nur eine sondern mehrere Samenanlagen mit primitiver Blütenhülle umschließt. Die beschriebene Erscheinung könnte als erster Schritt zu mehrkarpelligen Blüten interpretiert werden. Genauso ist es aber auch denkbar, dass eine Cupula aus ökonomischen Gründen mehr als eine Samenanlage umhüllt. Die Abbildung 49 zeigt anhand von Rekonstruktionen fossiler Funde die bislang als gültig angesehenen Interpretationen der einzelnen Strukturen und farblich (blau) hervorgehoben die Neuinterpretationen, wie sie im Rahmen dieser Arbeit entwickelt worden sind. Lagenostom Integument Mittelsäule Nucellusspitze Integument Hüllstruktur (Blütenhülle?) Cupula Cupula Integument Hüllstruktur Makroprothallium Makroprothallium A B Abb. 49: Rekonstruktionen fossiler Samenanlagen A) Schemazeichung einer fossilen Samenanlage mit Lagenostom (Quelle: ROTHWELL & SCHECKLER, in BECK 1988, Beschriftung ergänzt) B) Rekonstruktion des Cupula-Komplexes von Archaeosperma arnoldii nach Pettit & Beck 1968 (Quelle: STEWARD & ROTHWELL 1993, Beschriftung ergänzt) DISKUSSION 4.2. ENTWICKLUNGSGENETISCHE UNTERSUCHUNGEN 4.2.1. IN-SITU-HYBRIDISIERUNGEN AN SPOROPHYLLEN VON ZAMIA 104 Die Ergebnisse der In-situ-Hybridisierung an Sporophyllen von Zamia amblyphyllidia zum MADS-box-C-Gen zeigen, dass dieses Gen in der gesamten Samenanlage wie auch im ganzen Sporophyll exprimiert wird (Abb. 42,43). Diese Versuchsergebnisse stehen in keinem Widerspruch zu den Ergebnissen von In-situ-Hybridisierungen mit verschiedenen MADS-box-C-Genen an Angiospermen. Sie lassen sich problemlos in das ABC-Modell von COEN & MEYEROWITZ (1991) integrieren, das auf der Basis von Knockout-Mutanten zu MADS-box-Genen der Klassen A, B und C erstellt worden ist. In diesem Modell wird den MADS-box-C-Genen eine Beteiligung an der Steuerung der Entwicklungsprozesse aller generativen Strukturen der Blüte zugesprochen. Die alleinige Expression von C-Genen führt zur Bildung von weiblichen Strukturen, d.h. von Karpellen. Findet die Expression eines C-Gens in Kombination mit einem MADSbox-B-Funktionsgen statt, so entwickeln sich die männlichen Strukturen der Blüte, d.h. Staubgefäße. Daher überrascht es nicht, dass ein Gen aus der MADS-box-CFunktionsklasse auch in der gesamten reproduktiven Struktur einer Gymnosperme zu finden ist. Studien über das Vorhandensein von MADS-box-Genen aus den drei verschiedenen Funktionsklassen bei Gymnospermen lagen schon zu Beginn der vorliegenden Arbeit vor. Allerdings ist dabei nur vereinzelt auf die räumliche und zeitliche Verteilung der Genexpression innerhalb der Samenanlage bzw. ihrer Trägerstruktur eingegangen worden, so dass diese mittels der Methode der In-situ-Hybridisierung geklärt werden sollte. Aus den Ergebnissen der Untersuchungen an Zamia amblyphyllidia lässt sich ableiten, dass das im Rahmen dieser Arbeit isolierte MADS-box-C-Gen gleichmäßig im gesamten Sporophyll einschließlich der Samenanlage exprimiert wird. Es konnte auch keine gesteigerte Expression in den Bereichen festgestellt werden, in denen zum jeweiligen Sammelzeitpunkt entscheidende Entwicklungsschritte statt gefunden haben. Im in der Abbildung 43A gezeigten Sporophyll ist beispielsweise zu erwarten gewesen, dass die Samenanlage eine stärkere Expression des MADS-box-C-Gens aufweist als das Schild und der Stiel des Sporophylls, da sich zum Sammelzeitpunkt innerhalb der Samenanlage eine Vielzahl von verschiedenen Entwicklungsschritten vollziehen wohingegen im Schild und Stiel lediglich eine Größenzunahme statt findet. Eine ebenso gleichmäßige Expression eines MADS-box-C-Gens (CyAG) ist auch am reifen Samen einer nahverwandten Art, der Cycadaceae Cycas edentata L., deutlich zu erkennen (ZHANG et al. 2004). Wie schon von den Angiospermen bekannt werden bei den Gymnospermen ebenfalls MADS-box-Gene der C-Funktionsklasse in allen DISKUSSION 105 generativen Strukturen exprimiert. BECKER et al. (2003) belegen dies anhand von Insitu-Hybridisierungen an männlichen Zapfen der Gymnospermen Gnetum gnemon L.. CARLSBECKER et al. (2004) sprechen dem Gen DAL1 aus der Gymnospermen Picea abies (L.) H. KARST., das den MADS-box-C-Genen sehr ähnlichen ist, eine wichtige Rolle beim Übergang vom juvenilen zum adulten Stadium der generativen Strukturen zu. Damit könnte die Expression von MADS-box-C-Genen sowohl in weiblichen als auch in männlichen Strukturen erklärt werden. Die im Rahmen dieser Arbeit gewonnen Ergebnisse von In-situ-Hybridisierungen mit einem MADS-box-C-Gen aus Zamia amblyphyllidia an Sporophyllen bzw. Samenanlagen dieser Art, lassen sich problemlos in die bisherigen Forschungsergebnisse anderer Arbeitsgruppen eingliedern. Sie lassen sich ebenfalls bestens in das bereits im Kapitel 4.1.4. dargelegte neue Konzept zur Evolution der Samenanlage einfügen, da Nucellus und Integument auch bezüglich der Expression des MADS-box-C-Gens keinerlei Unterschiede aufweisen, so dass die Ergebnisse den Ansatz untermauern, dass das Integument eine Bildung des Nucellus und keine separate Struktur darstellt. Weitaus differenzierter stellen sich die Ergebnisse von In-situ-Hybridisierungen mit MADS-box-C-Genen an jungen weiblichen Zapfen verschiedener Pinaceen dar. Bei Picea abies (TANDRE et al. 1998), Picea mariana (MILL.) BSP. (RUTLEDGE et al. 1998) und Pinus radiata DON. (MOURADOV et al. 1999) ist jeweils deutlich zu erkennen, dass die Expression der MADS-box-C-Gene nur auf die Samenschuppe begrenzt ist. Die In-situ-Hybridisierungen bei Picea mariana wurden zudem an Stadien durchgeführt, bei denen auf der Samenschuppe bereits eine Samenanlage entwickelt ist. Hier ist eine verstärkte Genexpression innerhalb der Samenanlage besonders im Nucellus verglichen mit der gesamten Samenschuppe zu verzeichnen. Wie bereits erwähnt, konnten derartige Expressionsmuster anhand von In-situ-Hybridisierungen an Zamia amblyphyllidia nicht ermittelt werden. Aufgrund dieser erhaltenen relativ undifferenzierten Expressionsmuster lassen sich keine Aussagen über die genregulatorischen Vorgänge bei der Entwicklung der einzelnen Strukturen innerhalb der Samenanlage treffen, so dass hier eine Genregulation der Entwicklungsprozesse der Samenanlage mittels anderer, untergeordneter Gene zu vermuten ist. Bei Angiospermen werden z.B. die so genannten MADS-box-D-Gene als verantwortlich für die Entwicklung der Samenanlagen (MODRUSAN ET AL. 1994, COLOMBO ET AL. 1995) diskutiert. Für eine weitere Klärung der Wirkungsweise der MADS-box-Gene bei Zamia amblyphyllidia oder auch anderen Cycadeen müssen in Zukunft vielfältige In-situ-Hybridisierungen mit MADS-box-Genen aus den verschiedenen Funktionsklassen B, B-sister (BECKER 2000), C und D durchgeführt und ausgewertet werden. Des Weiteren ist interessant, innerhalb der Gymnospermen nach MADS-box-Genen der A-Funktion zu suchen, da diese MADS-box-Gene bei den Angiospermen bislang nur in Zusammenhang mit der DISKUSSION 106 Entwicklung der Blütenhülle, die eine Bildung der Angiospermen ist, nachgewiesen wurden. Darüber hinaus besteht in Hinblick auf die oben dargestellten In-situ-Hybridisierungsergebnisse zu den Pinaceen Picea abies, Picea mariana und Pinus radiata in Bezug auf zwei Fragen Klärungsbedarf. Zum einen ist nicht einsichtig, weshalb bei den genannten Pinaceen eine differenzierte Genexpression der MADS-box-C-Gene innerhalb der Samenanlage zu erkennen ist und im Gegensatz dazu die Expression bei Zamia amblyphyllidia innerhalb der ganzen Samenanlage konstant wirkt. Zum anderen sollte auch anhand von In-situ-Hybridisierungen mit MADS-box-C-Genen an jungen weiblichen Zapfen von Zamia amblyphyllidia überprüft werden, ob diese Gene im ganzen Zapfen exprimiert werden oder ob in der Sprossachse wie bei den zum Vergleich zur Verfügung stehenden Pinaceen keine MADS-box-C-Genexpression nachweisbar ist. Anhand von derartigen vergleichenden Untersuchungen können gegebenenfalls Aussagen über eine differenzierte Funktion der MADS-box-C-Gene innerhalb verschiedener Gruppen der Gymnospermen getroffen werden bzw. eine Einteilung in Unterklassen der MADS-box-C-Gen- vorgenommen werden. 4.2.2. IN-SITU-HYBRIDISIERUNGEN AN BLÄTTERN VON ZAMIA Die In-situ-Hybridisierungsversuche an sehr jungen, kaum ausdifferenzierten Blättern sowie an jungen Fiedern bereits ausdifferenzierter Wedel, dienten zur Kontrolle des im Rahmen dieser Arbeit isolierten MADS-box-C-Gens. Den Erwartungen nach sollte dieses Gen nur in generativen Strukturen exprimiert werden, nicht aber im Spross, in den Blättern oder in den Wurzeln. Um auszuschließen, dass es sich beim überall im Sporophyllgewebe nachzuweisenden MADS-box-Gen nicht um ein funktionsloses, ubiquitär exprimiertes Genduplikat handelt, sind in mehreren In-situ-Hybridisierungen parallel zu den histologischen Schnitten von Samenanlagen bzw. Sporophyllen auch Schnitte von RNase frei fixiertem Blattgewebe mitgeführt und auf dieselbe Weise wie das zu untersuchenden Material behandelt worden. Diese Vorgehensweise diente der Kontrolle für den Fall, dass das Blattgewebe zwar ein negatives Hybridisierungsergebnis zeigt, aber der Grund z.B. auch in einer fehlerhaften Versuchsdurchführung zu suchen sein könnte. Zeigen die parallel mitgeführten Samenanlagen bzw. Sporophylle eine positive Hybridisierungsreaktion an, kann man sich sicher sein, dass ein negatives Ergebnis bezüglich des Blattgewebes nicht auf Mängel bei der Versuchsdurchführung zurück zu führen ist. Aus dem Grund können die in der Abbildung 44 exemplarisch gezeigten In-situ-Hybridisierungsergebnisse zu Blattgewebe von Zamia amblyphyllidia als Beweis dafür gewertet werden, dass im Rahmen der vorliegenden Arbeit tatsächlich ein MADS-box-C-Gen aus der als sehr ursprünglich angesehenen Gymnospermen Zamia amblyphyllidia isoliert worden ist. ZUSAMMENFASSUNG 5. 107 ZUSAMMENFASSUNG Das im Rahmen dieser Arbeit erstellte Konzept zur Evolution der Samenanlage steht vollkommen losgelöst von der Telomtheorie ZIMMERMANNS, die eine Bildung des Integuments aus sterilen Telomlappen an der Basis der Samenanlage postuliert. Ganz im Gegensatz dazu konnte anhand von morphologischen Studien an als ursprünglich angesehenen Gymno- bzw. Angiospermen gezeigt werden, dass das Integument an der Spitze des Nucellus initiiert wird und vollständig aus umgewandelten Nucellusgewebe aufgebaut ist. Somit ist das Integument keine separate Struktur. Es ist vielmehr eine Struktur, die allein vom Nucellus gebildet wurde, um der lysogen entstandenen Öffnung an dessen apikalen Ende Festigkeit zu verleihen. Dieses neu entwickelte Konzept steht nicht im Widerspruch zu den fossilen Funden, die sich basierend auf dieser These neu und zum Teil sogar sinnvoller interpretieren lassen. So stellen die fossilen Strukturen Salpinx und Lagenostom keine Randerscheinungen der Evolution dar, sondern sind Indizien dafür, dass die Evolution des Integuments ebenso abgelaufen ist, wie die Ontogenese dieser Struktur, die sich anhand von morphologischen Studien bei rezenten Spermatophyta nachvollziehen lässt. Die Cupula, eine andere fossile Struktur, die bei vielen primitiven, fossilen Samenanlagen beschrieben ist, kann demnach als eine primitive Blütenhülle betrachtet werden. Ein anderer Aspekt der vorliegenden Arbeit hat sich damit befasst, den Ursprung dessen, was im Allgemeinen als Integument bezeichnet wird, über die histologischen Methoden hinaus mit Mitteln der Entwicklungsgenetik als eine Bildung des Nucellus zu beweisen. Zu diesem Zweck ist die Methode der In-situ-Hybridisierung am Lehrstuhl für Evolution und Biodiversität der Pflanzen der Ruhr-Universität Bochum im Rahmen dieser Arbeit etabliert worden. Dazu wurde nicht nur ein vollständiges Labor eingerichtet, sondern auch ein Laborprotokoll für In-situ-Hybridisierungen an Gymnospermen entwickelt. Dieses Protokoll ist permanent optimiert worden, so dass es in Zukunft als Vorlage für In-situ-Hybridisierungen an Gymnospermen genutzt werden kann. Die Entwicklung eines speziell auf Gymnospermen abgestimmten Protokolls war notwendig, da sich die Standardprotokolle von In-situ-Hybridisierungen an Angiospermen aufgrund der vielen sekundären Pflanzenstoffe bei Gymnospermen nicht auf diese übertragen ließen. Die erhaltenen Ergebnisse der In-situ-Hybridisierungen an Sporophyllen und Samenanlagen von Zamia amblyphyllidia lassen sich nahtlos in die aktuellen Forschungsergebnisse anderer Arbeitsgruppen zu MADS-box-Genen einfügen. Zudem untermauern die gewonnen In-situ-Hybridisierungsergebnisse das bereits oben dargestellte Konzept zur Evolution der Samenanlage, das im Rahmen der vorliegenden Arbeit entwickelt worden ist. LITERATURVERZEICHNIS 6. 108 Literaturverzeichnis ALVAREZ-BUYLLA, E. R. , LILJEGREN, S. J. , PELAZ, S. , GOLD, S. J. , BURGEFF, C. , DITTA, G. S. , VERGARA-SILVA, F. & YANOFSKY, M. F. (2000b): MADS-box gene evolution beyond flowers: expression in pollen, endosperm, guard cells roots and trichomes. Plant J. 24(4): 1-11. ALVAREZ-BUYLLA, E. R. , PELAZ, S. , LILJEGREN, S. J. , GOLD, S. J. , BURGEFF, C. , DITTA, G. S. , DE POUPLANA, L. R. , MARTINEZ-CASTILLA, L. & YANOFSKY, M. F. (2000a): An ancestral MADS-box gene duplication occurred before the divergence of plants and animals. PNAS. Vol. 97. No. 10: 5328-5333. ANDREWS, H. N. (1961): Studies in Paleobotany. John Wiley, New York. ANDREWS, H. N. (1963): Early seed plants. Science 142 (3594): 925-931. AZUMA, H. , GARCIA-FRANCO, J. G. , RICO-GRAY, V. & THOEN, L. B. (2001): Molecular Phylogeny of the Magnoliaceae: the Biogeography of tropical and temporate disjunctions. Am. J. Bot. 88(12): 2275-2285. BANKS, H. P. (1971): Occurrence of Cooksonia, the oldest vascular land plant macrofossil, in the Upper Silurian of New York state. J. Indian Bot. Soc. 50A: 227-235. BECKER, A. (2000): Charakterisierung phylogenetisch informativer MADS-Box-Gene aus der Gymnosperme Gnetum gnemon L.. Diss., Köln. BECKER, A. , SAEDLER, H. & THEISSEN, G. (2003): Distinct MADS-box gene expression patterns in the reproductive cones of the gymnosperm Gnetum gnemon. Dev. Genes. Evol. 213: 567-572. BECKER, A. , WINTER, K.-U. , MEYER, B. , SAEDLER, H. & THEISSEN, G. (2000): MADSBox Gene Diversity in Seed Plants 300 Million Years Ago. Mol. Biol. Evol. 17(10): 1425-1434. BRESINSKY, A. (2002): Übersicht des Pflanzenreiches - Embryophyten. In: SITTE, P. , WEILER, E. W. , KADEREIT, J. W. , BRESINSKY, A. & KÖRNER, C.: Straßburger – Lehrbuch der Botanik, 35. Aufl. Spektrum Akademischer Verlag Heidelberg, Berlin LITERATURVERZEICHNIS 109 CARLSBECKER, A. , TANDRE, K. , JOHANSON, U. , ENGLUND, M. & ENGSTRÖM, P. (2004): The MADS-Box gene DAL1 is a potential mediator of the juvenile-to-adulttransition in Norway spruce (Picea abies). Plant J. 40: 546-557. CAROTHERS, I. E. (1907): Development of ovule and female gametophyt in Ginkgo biloba. Botanical Gazette. 43:116. CHALONER, W. G. , HILL, A. J. & LACEY, W. S. (1977): First Devonian platyspermic seed and its implication in gymnosperm evolution. Nature 265: 233-235. CHAMBERLAIN, C. J. (1935) : Gymnosperms : Structure and Evolution. University of Chicago Press, Chicago. COEN, E. S. & MEYEROWITZ, E. M. (1991): The war of the whorls: genetic interactions controlling flower development. Nature. Vol. 353: 31-37. COLOMBO, L. , FRANKEN, J. , KOETJE, E. , VAN WENT, J. , DONS, H. J. M. , ANGENENT, G. C. & VAN TUNEN, A. (1995): The Petunia MADS Box Gene FBP11 Determines Ovule Identity. Plant Cell. Vol. 7: 1859-1868. COULTER, J. M. & CHAMBERLAIN, C.J. (1912): Morphology of angiosperms. D. Appleton, New York. DE BOER, R. & BOUMAN, F. (1972): Integumentary studies in the Polycarpicae. II. Magnolia stellata and Magnolia virginiana. Acta Bot. Neerl. 21: 617-629. DE-XING XIAO & FENG-XIA XU (2006): Megasporogenesis and development of female gametophyt in Manglietia decidua (Magnoliaceae). Ann. Bot. Fennici 43: 437444. DONOGHUE, M.J. & J.A. DOYLE (1989): Phylogenetic studies of seed plants and angiosperms based on morphological characters. In: B. FERNHOLM, K. BREMER, & H. JORNVALL (eds.): The Hierarchy of Life. Nobel Symposium 70. Elsevier Publ., Amsterdam EARLE, T. T. (1938): Origin of seed coats in Magnolia. Am. J. Bot. 25: 221-223. EDWARDS, D. (1970a): Fertile Rhyniophytina from the Lower Devonian of Britain. Paleontol. 13: 451-461. ERBAR, C. & LEINS, P. (1981): Zur Spirale in Magnolien-Blüten. Beitr. Biol. Pflanzen 56: 225-241. LITERATURVERZEICHNIS 110 ERBAR, C. & LEINS, P. (1983): Zur Sequenz von Blütenorganen bei einigen Magnoliiden. Bot. Jahrb. Syst. 103: 433-449. ERBAR, C. & LEINS, P. (1994): Flowers in Magnoliidae and the origin of flowers in other subclasses of the angiosperms. I. The relationships between flowers of Magnoliidae and Alismatidae. Plant Syst. Evol. 8: 193-208. FAIRON-DEMARET, M. & SCHECKLER, S. E. (1987): Typification and redescription of Moresnetia zalesskyi STOCKMANS, 1948, an early seed plant from the Upper Famennian of Belgium. Science de la Terre. 57: 183-199. FARJON, A. (1998): World checklist and bibliography of conifers. Royal Botanical Gardens Kew, London. FUJI, K. (1895): On the nature and origin of the socalled „chichi“ (Nipple) of Ginkgo biloba. Bot. Mag. (Tokyo) 9: 444-450 FUKUI, M. , FUTAMURA, N. , MUKAI, Y. , WANG, Y , NAGAO, A. & SHINOHARA, K. (2001): Ancestral MADS Box Genes in Sugi, Cryptomeria japonica D.Don (Taxodiacae), Homologous to the B Function Genes in Angiosperms. Plant Cell Physiol. 42(6): 566-575. GADEK, P. A. , ALPERS, D. L. , HESLEWOOD, M. M. & QUINN, C. J. (2000): Relationships within Cupressaceae sensu lato: a combined morphological and molecular approach. Am. J. Bot. 87(7): 1044-1057. GEHRING, W. J. , AFFOLTER, M. & BÜRGLIN, T. (1994): Homeodomaine proteins. Annu. Rev. Biochem. 63:487-526. GERLACH, D. (1984): Botanische Mikrotechnik. 3. Aufl. Thieme, Stuttgart GERRIENNE, P. , MEYER-BERTHAUD, B. , FAIRON-DERMAT, M. , STREEL, M. & STEEMANS, P. (2004): Runcaria, a Middle Devonian Seed Plant Precursor. Science 306: 856858. GESTERBERGER, P. & LEINS, P. (1978): Rasterelektronenmikroskopische Untersuchungen an Blütenknospen von Physalis Philadelphia (Solanaceae). Ber. Dt. Bot. Ges. 91: 381-387. GIFFORD, E. M. & FOSTER, A. S. (1996): Morphology and Evolution of Vascular Plants. 3. Aufl. W. H. Freeman and Company, New York. LITERATURVERZEICHNIS 111 GILLESPIE, W. H. , ROTHWELL, G. W. & SCHECKLER, S. E. (1981): The earliest seeds. Nature. Vol. 293. No. 5832: 462-464. GOEBEL, K. (1923): Organographie der Pflanzen. Teil 3. Gustav Fischer, Jena. HOFMEISTER, W. (1851): Vergleichende Untersuchungen der Keimung, Entfaltung und Fruchtbildung höherer Kryptogamen. Leipzig. JAGER, M. , HASSANIN, A. , MANUEL, M. , LE GUYADER, H. & DEUTSCH, J. (2003): MADSBox Genes in Ginkgo biloba and the Evolution of the AGAMOUS family. Mol. Biol. Evol. 20(5): 842-854. JONES, D. L. 1993. Cycads of the world. Reed Books, Chatswood KADEREIT, J. W. (2002): Evolution und Stammesgeschichte der Angiospermen – ein Überblick. In: SITTE, P. , WEILER, E. W. , KADEREIT, J. W. , BRESINSKY, A. & KÖRNER, C.: Straßburger – Lehrbuch der Botanik, 35. Aufl. Spektrum Akademischer Verlag Heidelberg, Berlin KIM, S. , KOH, J. , YOO, M.-J. , KONG, H. , HU, Y. , MA, H. , SOLTIS, P. S. & SOLTIS, D. E. (2005): Expression of floral MADS-box genes in basal angiosperms: implications for the evolution o floral regulators. Plant J. 43: 724-744. KRUSE, J. (1992): Magnoliales. In: FUKAREK, F. , HAMMER, K. , HANELT, P. & SCHULTZEMOTEL, J.: Urania Pflanzenreich – Blütenpflanzen 1, 1. Aufl. Urania Verlag Leipzig, Jena, Berlin. KRÜSSMANN, G. (1976): Handbuch der Laubgehölze, Band II, E-Pro. Verlag Paul Parey, Berlin und Hamburg. LEINFELLNER, W. (1967): Über die Karpellen verschiedener Magnoliales. IV. Magnolia und Michelia (Magnoliaceae). Österr. Bot. Z. 114: 73-83 LEINS, P. (2000): Blüte und Frucht - Aspekte der Morphologie, Entwicklungsgeschichte, Phylogenie, Funktion und Ökologie. Schweizerbart'sche Verlagsbuchhandlung, Stuttgart. LONG, A. G. (1959): On the Structure of Calymmatotheca kidstoni Calder (emended) and Genomosperma latens gen. et sp. nov. from the Calciferous Sandstone Series of Berwickshire. Trans. Roy. Soc. Edin. 64(2): 29-44. LONG, A. G. (1960a): Stamnostoma huttonense gen. et sp. nov. – a Pteridosperm seed and cupule from the Calciferous Sandstone Series of Berwickshire. Trans. LITERATURVERZEICHNIS 112 Roy. Soc. Edin. 64(9): 201-215. LONG, A. G. (1960b): On the Structure of Samaropsis scotica Calder (emended) and Eurystoma angulare gen. et sp. nov. Petrified Seeds from the Calciferous Sandstone Series of Berwickshire. Trans. Roy. Soc. Edin. 64(12): 261-281. MERSMANN, H. (1998): Blütenentwicklung bei Gnetidae. Dipl.-Arb., Bochum MODRUSAN, Z. , REISER, L. , FELDMANN, K. A. , FISCHER, R. L. & HAUGHN, G. W. (1994): Homeotic Transformation of Ovules into Carpel-like Structures in Arabidopsis. Plant Cell. Vol.: 333-349. MOURADOV, A. , GLASSICK, T. V. , HAMDORF, B. , MURPHY, L. C. , MARLA, S. S. , YANG, Y. & TEASDALE, R. D. (1999): Family of MADS-Box-Gene Expressed Early in Male and Female Reproductive Structures of Monterey Pine. Plant Physiol. 117: 5561. MOURADOV, A. , HAMDORF, B. , PELAZ, S. , TEASDALE, R. D. , KIM, J. T. , WINTER, K.-U. & THEISSEN, G. (1999): A DEF/GLO-like MADS-Box-Gene From a Gymnosperm: Pinus radiata Contains an Ortholog of Angiosperm B Class Floral Hometic Genes. Developmental Genetics 25: 245-252. MUNDRY, I. (2000): Morphologische und morphogenetische Untersuchungen zur Evolution der Gymnospermen. Bibl. Bot. 152: 1-90. NIKLAS, K. J.: The Influence of Paleozoic Ovule and Cupule Morphologies on Wind Pollination. Evolution 37(5): 968-986 PETTITT, J. M. & BECK, C.B. (1968): Archaeosperma arnoldii – a Cupulate Seed from the Upper Devonian of North America. Cont. Mus. Paleontol. Univ. Mich. 22: 139154. PURRUGGNAN, M. D. , ROUNSLEY, S. D. , SCHMIDT, R. J. & YANOFSKY, M. F. (1995): Molecular Evolution of Flower Development: Diversification of the Plant MADSBox Regulatory Gene Family. Genetics. 140: 345-356. RESTEMEYER, J. (2002): Morphologische und morphogenetische Untersuchungen zur Phylogenie und Evolution der Podocarpaceae und Phyllocladaceae. Diss., Bochum. RIECHMANN, J. L., MEYEROWITZ, E. M. (1997): MADS domain proteins in plant development. Biol. Chem. 378: 1079–1101. LITERATURVERZEICHNIS 113 RIEGER, M. (2002): Morphologischer und histologischer Vergleich der Zapfenentwicklung bei Podocarpaceae und Phyllocladaceae. Dipl.-Arb., Bochum ROBINSON-BEERS, K. , PRUITT, R. E. & GASSER, C. S. (1992): Ovule Development in Wild-Type Arabidopsis and Two Female-Sterile Mutants. Plant Cell 4: 12371249. ROTH, I. (1957): Die Histogenese der Integumente von Capsella bursa-pastoris und ihre morphologische Bedeutung. Flora 145: 212-235. ROTHWELL, G. W. & SCHECKLER, S. E. (1988): Biology of Ancestral Gymnosperms. In: BECK, C. E.: Origin and evolution of gymnosperms, Columbia University Press, New York RUTLEDGE, R. , REGAN, S. , NICOLAS, O. , FOBERT, P. , CÔTÉ, C. , BOSNICH, W. , KAUFFELDT, C. , SUNOHARA, G. , SEGUIN, A. & STEWART, D. (1998): Characterization of an AGAMOUS homologue from the conifer black spruce (Picea mariana) that produces floral homeotic conversions when expressed in Arabidopsis. Plant J. 15(5): 625-634. SCHNARF, K. (1937): Anatomie der Gymnospermen-Samen. In K. LINSBAUER: Handbuch der Pflanzenanatomie, Band X/1. Gebrüder Borntraeger, Berlin. SCHULTZE-MOTEL, J. (1992): Gymnospermae. In: FUKAREK, F. , SCHULTZE-MOTEL, J. & SIEGEL, M.: Urania Pflanzenreich – Moose, Farne, Nacktsamer, 1. Aufl. Urania Verlag Leipzig, Jena, Berlin. SCHÜTT, P. , SCHMUCK, H. J. & STIMM, B. (2002): Lexikon der Baum- und Straucharten – Das Standardwerk der Forstbotanik. Nikol Verlag, Hamburg. SMITH, F. G. (1910): Development of the ovulate strobilus and young ovule of Zamia floridana. Botanical Gazette. 50:128-141. STEBBINS, G. L. (1974): Flowering Plants: Evolution Above the Species Level. Harvard University Press, Cambridge, Massachusetts. STEWART, W. N. & ROTHWELL (2001): Paleobotany and the Evolution of Plants. 2. Ed., Cambridge University Press, Cambridge STEWART, W. N. (1983): Paleobotany and the Evolution of Plants. Cambridge University Press, Cambridge LITERATURVERZEICHNIS 114 STÜTZEL, TH. & RÖWEKAMP, I. (1999b): Bestäubungsbiologie bei Nacktsamern (Gymnospermen). In: ZIZKA, G. & SCHNECKENBURGER, S.: Blütenbiologie – faszinierendes Miteinander von Pflanzen und Tieren. – Kleine SenckenbergReihe 33: 107-117. Frankfurt. SUNDSTRÖM, J. , CARLSBECKER, A. , SVENSSON, M. E. , SVENSON, M. , THEISSEN, G. & ENGSTRÖM, P. (2004): MADS-Box Genes Active in Developing Pollen Cones of Norway Spruce (Picea abies) are Homologous to the B-Class Floral Homeotic Genes in Angiosperms. Developmental Genetics 25: 253-266. TANDRE, K. , SVENSON, M. , SVENSSON, M. E. & ENGSTRÖM, P. (1998): Conservation of gene structure and activity in the regulation of reproductive organ development of conifers and angiosperms. Plant. J. 15(5): 615-623. TAYLOR, T. N. & LEISMAN, G. A. (1963): Conostoma kestospermum, a new species of Paleozoic seed from the Middle Pennsylvanian. Am. J. Bot. 50(6): 574-580. TAYLOR, T. N. (1982): Reproductive Biology in Early Seed Plants. BioScience 32: 2328 THEISSEN, G. (2000): Development of floral organ identity: stories from the MADS house. Curr. Opin. Plant Biol. 4: 75-85. THEISSEN, G. , BECKER, A. , DI ROSA, A. , KANNO, A. , KIM, J. T. , MÜNSTER, T. , WINTER, K.-U. & SAEDLER, H. (2000): A short history of MADS-box genes in plants. Plant Mol. Biol. 42: 115-149. THOMAS, B. A. & SPICER, R. A. (1987): The evolution and paleobiology of land plants, Croom Helm, London. UMEDA, A. , IMAICHI, R. & KATO, M. (1994): Ovular development and morphology of the outer integument of Magnolia grandiflora (Magnoliaceae). Am. J. Bot. 81(3): 361-367. VEGA, J. C. & ARCHANGELSKY, S. (1997): The first Gondwana Carboniferous compound cupule and associated seeds. A preliminary note. Review of Palaeobotany and Palynology. 99: 55-59. VELENOVSKÝ, J. (1910): Vergleichend Morphologie der Pflanzen. III. Teil. Fr. Rivnác. Prag. LITERATURVERZEICHNIS 115 WAGENITZ, G. (2003): Wörterbuch der Botanik, 2 Aufl. Spektrum Akademischer Verlag Heiderlberg, Berlin. WANG, X.-Q. , TANK, D. C. & SANG, T. (2000): Phylogeny and Divergence Time in Pinaceae: Evidence from Three Genomes. Mol. Biol. Evol. 17(5): 773-781. WEIGEL, D. & MEYEROWITZ, E. M. (1994): The ABC`s of floral homeotic genes. Cell 78: 203-209. WEILER, E. (2002): Physiologie. In: SITTE, P. , WEILER, E. W. , KADEREIT, J. W. , BRESINSKY, A. & KÖRNER, C.: Straßburger – Lehrbuch der Botanik, 35. Aufl. Spektrum Akademischer Verlag Heidelberg, Berlin WEN, J. (2001): Evolution of Eastern Asian-Eastern North American Biogeographic Disjunctions: A Few Additional Issues. Int. J. P. Sci. 162 (6):117-122 WETTSTEIN, R. 1935. Handbuch der Systematischen Botanik. Franz Deuticke, Leipzig, Wien. WHITELOCK, L. M. (2002): The Cycads. Timber Press, Portland. WINTER, K.-U. , BECKER, A. , MÜNSTER, T. , KIM, J. T. , SAEDLER, H. & THEISSEN, G. (1999): MADS-box genes reveal taht gnetophyts are more closely related to conifers than to flowering plants, PNAS 96: 7342-7347. XIAO, D.-X. & XU, F.-X. (2006): Megasporogenesis and development of female gametophyte in Manglietia decidua (Magnoliaceae). Ann. Bot. Fennici 43: 437444. ZACHGO, S. (2002): In situ hybridization. In: GILMARTIN, P. M. & BOWLER, C.: Molecular Plant Biology, Vol. 2. Oxford University Press, Oxford. ZHANG, P. , TAN, H. T. W. , PWEE, K.-H. & KUMAR, P.P. (2004): Conservatin of class C function during 300 million years of evolution from gymnosperms to angiosperms. Plant J. 37: 566-577. ZIMMERMANN, W. (1959): Die Phylogenie der Pflanzen. 2. Aufl. Gustav Fischer, Stuttgart. ZIMMERMANN, W. (1969): Die Geschichte der Pflanzen, 2. Aufl. Georg Thieme Verlag, Stuttgart. ANHANG 7. 116 ANHANG ERKLÄRUNG Hiermit erkläre ich, dass ich die Arbeit selbstständig verfasst und bei keiner anderen Fakultät eingereicht und dass ich keine anderen als die angegebenen Hilfsmittel verwendet habe. Es handelt sich bei der heute von mir eingereichten Dissertation um fünf in Wort und Bild völlig übereinstimmende Exemplare. Weiterhin erkläre ich, dass digitale Abbildungen nur die originalen Daten enthalten und in keinem Fall inhaltverändernde Bildbearbeitung vorgenommen wurde. Bochum, den 01.04.2008 ______________________________ (Unterschrift) ANHANG 117 LEBENSLAUF Name: Julia Gabriela Kunze, geb. Obermann Anschrift: Fahrendelle 23e, 58455 Witten Tel.: 02303/423888 Mail: [email protected] Geburtsdatum: Geburtsort: Staatsangehörigkeit: Konfession: Familienstand: 29.09.1978 Dortmund-Hörde deutsch evangelisch verheiratet Ehepartner: Eltern: Christian Kunze - Studienrat Doris Obermann, geb. Göbel - Lehrerin i. R. Frank Obermann - Schulleiter i. R. Eva Christine Weberink - Juristin Geschwister: Schulbesuch: Abschluss: Studium: Abschluss: 1985 – 1989: Jahnschule (Grundschule), Kamen-Methler 1989 – 1998: Städtisches Gymnasium, Kamen 08.06.1998: Abitur (Note 1,9) WS 1998/99 – SS 2003: Studiengang Biologie an der Ruhr-Universität Bochum 07.08.2003: Diplom (Note 1,2) Bisherige Tätigkeiten: 09/2003 – 05/2004: WHK am LS für Spezielle Botanik, Ruhr-Universität Bochum 06/2004 – 11/2006: Promotionsstipendiatin des „Allgemeinen Promotionskollegs der RUB“ am LS für Spezielle Botanik 12/2006 – 06/2007: WHK am LS für Spezielle Botanik, Ruhr-Universität Bochum 07/2007 – 03/2008: Fertigstellung der Promotionsarbeit Aktuelle Tätigkeit: Seit 01/2008: Lehramtsanwärterin für GHR am Studienseminar Dortmund ANHANG 118 DANKSAGUNG Mein Dank gilt ... ... selbstverständlich an erster Stelle meinem Betreuer Prof. Dr. Thomas Stützel für die Überlassung des spannenden und facettenreichen Themas. ... ebenfalls meinem Korreferenten Prof. Dr. Ralf Tollrian, der so spontan bereit war, das Korreferat zu übernehmen. Danke! ... Prof. Dr. Gerd Schaub und seiner gesamten Arbeitsgruppe (AG Parasitologie am Lehrstuhl für Evolutionsökologie und Biodiversität der Tiere), da ich das dortige S1-Labor mitsamt des gesamten Equipments für meine molekulargenetischen Vorarbeiten nutzen durfte. ... in Zusammenhang mit letzterem Punkt ganz besonders Dr. Peter Waniek, der mir die molekularbiologische Arbeitsweise nahe gebracht und geduldig meine ersten Schritte auf diesem weiten Feld begleitet hat. Nach Peters Aufbruch ins ferne Brasilien wurde Dr. Markus Deckers ein immer wichtigerer Ansprechpartner für kurze, aber dringende labortechnische Anfragen. Danke dafür! ... allen Mitgliedern des Lehrstuhls für Evolution und Biodiversität der Pflanzen. Sie alle haben meinen Uni-Alltag in den letzten vier Jahren entscheidend beeinflusst und zum häufig sehr fröhlichen Arbeitsklima am Lehrstuhl sowie auf Exkursionen beigetragen. ... aber ganz besonders einer hier an dieser Stelle hervorzuhebenden Mitarbeiterin des Lehrstuhls: Dipl.-Biol. Nicole Hille. Da Nicole ein methodisch gleiches Thema wie ich in ihrer Promotion bearbeitet hat, konnten wir gemeinsam den neuen und oft auch steinigen Weg zur In-situ-Hybridisierung betreten und uns so gegenseitig durch Höhen und Tiefen ziehen. Was hätte ich alleine nur gemacht ... ... zu guter Letzt natürlich auch meinem ganzen Freundeskreis, meinem Rausche und dem Rest meiner Familie, die irgendwann aufgegeben haben, mich nach dem Fortgang meiner Arbeit zu fragen und mich so in Ruhe und mit vollstem Verständnis für meinen chronischen Zeitmangel promovieren ließen. ... vorsichtshalber auch noch all denen, die ich eventuell an dieser Stelle vergessen habe, denen ich aber dennoch sehr dankbar bin wie z.B. den lieben CafêtenFrauen oder den geduldigen Herren im Chemikalienlager. Vielen Dank!