No Slide Title
Werbung
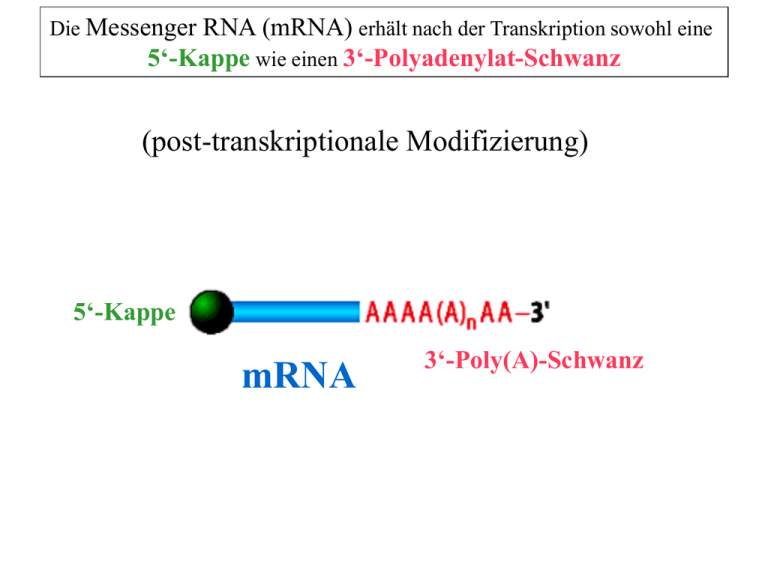
Die Messenger RNA (mRNA) erhält nach der Transkription sowohl eine 5‘-Kappe wie einen 3‘-Polyadenylat-Schwanz (post-transkriptionale Modifizierung) 5‘-Kappe mRNA 3‘-Poly(A)-Schwanz 3‘ Polyadenylat-Schwanz der Polyadenylatschwanz wird erst zum Ende der Transkription von einem Enzym (PolyA-Polymerase) an das gespaltende 3‘-Ende angehängt (Transkriptions-Termination) 3‘ n = 200 - 300 Nukleotide Spaltung durch Endonuclease Der polyA-Schwanz 1. erhöht Stabilität der mRNA 2. wichtig beim Export der mRNA ins Zytoplasma Spaltsignal 3‘ UTR ORF mRNA mRNA 5‘ 5‘ 5‘ 5‘ Die Polyadenylierung der mRNA DNA mRNA RNA-Polymerase QuickTime™ and a Cinepak decompressor are needed to see this picture. polyA-Polymerase Der Poly(A)-Schwanz erlaubt die einfache und schnelle biochemische Reinigung von poly(A)+ RNA durch Affinitätschromatographie Säule mit poly(U) gebunden an kleine Sepharose-Kügelchen reine poly(A)+ RNA >Northern, cDNA-Synthese, RT-PCR Die Bildung der 5‘-Kappe an der mRNA erfolgt nur bei Eukaryonten vom Gen kodiert später angehängt 7 5‘ ungewöhnliches 5‘-5‘Triphosphat der 5‘-Kappe wird noch während der Transkription von den entsprechenden Enzymen (“capping enzymes“) an das 5‘-Ende angehängt die 5‘-Kappe erhöht die Stabilität der mRNA - Kontrolle der GenExpression 5‘ 2‘ 3‘ die 5‘-Kappe ist wichtig beim Export der mRNA in das Zytoplasma und bei der anschließenden Translation (Anlocken von Initiationsfaktoren der Translation) RNA-Turnover De-Adenylierung und Entfernung der 5‘-Kappe sind Signale zum Abbau der mRNA 5‘-Kappe “decapping enzyme“ Fragen aus der schriftlichen Physikumsprüfung Die Cap-Struk tur („Kop fgruppe“ ) euka ryontischer RNA -Moleküle (A) wir d bei der posttranslation alen Proze ssierung abg espalt en (B) befinde t sich am 5 ’-Ende d er mRNA und en thält einen methyl ie rten Guany lr est (C) wir d durch d ie RNA-Polymerase ll bei de r Initi ation de r Transk ription syn thetisi ert (D) blockiert bei der Translation d ie Peptidyltransferase-Reaktion ist ein Produkt der Trans esterifizierung beim Spleißvorgang ……eine weitere post-transkriptionale Veränderung das Spleißen der Prä-mRNA Die Entdeckung des Spleiß-Phänomens bis 1977 hatte man die Vorstellung, daß die DNA-Sequenz eines Gens co-linear mit der Aminosäure-Sequenz ist als man aber die reife mRNA von bestimmten Genen isolierte (polyU-Affinitätschromatographie) und mit der entsprechenden DNA-Matritze hybridisierte und die DNA::RNAHybride im Elektronenmikroskop sichtbar machte, gab es eine unerwartete Entdeckung >>>> die gereinigte mRNA war viel kürzer als die DNA-Matritze NNN N Intron Exon N N N N C T A TACGGATCGTA N N N N C T A N ACAGTAGGCTATAAC AUGCCUAGCAU-UGUCAUCCGAUAUUG Die Struktur des Hühner-Ovalbumin-Gens Elektronenmikroskopie Zeichnung E mRNA DNA-Matritze die Schleifen A-G sind die 7 Introne, die im primären Transkript noch vorhanden sind, später aber bei der Reifung der mRNA herausgeschnitten werden. das Entfernen der Introne wird als Spleißen (“splicing“) bezeichnet AUG Intron Exon Stop Prä-mRNA 8000 Nts Intronsequenzen werden durch Spleißen entfernt reife mRNA Translation Protein (Ovalbumin) Chromosom mit 1.5 x 108 Nucleotid-Basenpaaren - ca. 3000 Gene 0.5% des Chromosoms enthält Gene >>> 15 Gene ein Gen mit 105 Nucleotid-Basenpaaren Promoter Intron Exon Transkription Prä-mRNA Spleißen mRNA Transkription Bildung der 5‘-Kappe Exon Intron Spleißosome prä-mRNA Spleißen Polyadenylierung mRNA Intron > das Spleißen ist ein äußerst komplizierter Vorgang - ca. 300 Komponenten beteiligt > der Spleiß-Enyzm-Komplex wird Spliceosome genannt > die meisten Gene höherer Eukaryonten haben Introne (bis zu 40 pro Gen) > die Exone sind weniger als 1000 BP lang, in der Regel 100-200 Nukleotide (entspricht 30-50 Aminosäuren) > die Größe der Introne schwankt von 50 - 20 000 Nukleotide > die DNA, die den Intronen entspricht, ist daher viel mehr als die DNA, die für die Exone kodiert (Gen > 50.000 BP; mRNA > 5.000 BP) die Entfernung des Introns aus der Prä-mRNA muß äußerst präzise ablaufen, damit die reife mRNA korrekt für das entsprechende Protein kodiert die Präzision muß auf ein Nukleotid genau sein (molekulares Skalpell) mRNA Korrekt gespleißt korrektes Spleißen prä-mRNA fehlerhaftes Spleißen > frameshift Mutation >andere Aminosäuresequenz mRNA falsch gespleißt Wie schneidet das Spliceosom derart korrekt die Introne aus der Prä-mRNA? man findet beim Vergleich der Exon/Intron-Übergänge bzw. Intron/Exon-Übergänge Consensus-Sequenzen Consensus-Sequenz A G G U A A G U…………………..C A G G 5‘-Spleißstelle 3‘-Spleißstelle Allgemeine Regeln bei den Spleiß-Consensus-Bereichen • die Basensequenz eines Introns beginnt • Introne haben noch eine wichtige interne Stelle, die stets ca. 20 - 50 Nukleotide stromaufwärts von der 3‘ AG-Spleißstelle liegt und Verzweigungsstelle (“branch site“) genannt wird. In Hefe lautet die Verzweigungsstelle nahezu immer UACUAAC (TACTAAC-Box) AG GUAAG 5‘-Spleißstelle GU und endet mit AG UACUAAC “branch site“ .................CAG G 3‘-Spleißstelle • Bereiche zwischen der 5‘ und 3‘ Spleißstelle und der Verzweigungsstelle sind relativ unwichtig für das Spleißen des Introns • durch Mutationen in jeder dieser drei wichtigen Regionen kann es zur Hemmung des Spleißens bzw. zu einem veränderten Spleißen kommen • falsches Spleißen verursacht auch Krankheiten, z. B. einige Formen von Thalassämien = erbliche Blutkrankheiten (Anämien) >>> defekte Hämoglobinsynthese (1) Normale Globin prä-mRNA Exon 1 Intron 1 mRNA Exon 2 Exon 1 Exon 2 Intron 2 Exon 3 Exon 3 Protein verlängertes Exon (2) Mutierte Globin prä-mRNA Exon 1 Intron 1 mRNA Exon 2 Exon 1 Exon 2 Intron 2 Exon 3 Punktmutation Stop Exon 3 kein Häm 128 Protein b-Thalassämien (1) Exon 2 -----CCTATTGGTCTATTTTCCACCCTTAG GCTG------- Exon 3 Mutation (2) Exon 2 normale Spleißstelle -----CCTATAG GTCTATTTTCCACCCTTAG GCTG--neue Spleißstelle Stop Codon Exon 3 am Spleißvorgang sind viele Komponenten beteiligt Spleiß-Enzyme U-snRNA (Uridin-rich, small nuclear RNA) U1 U-snRNAs + Proteine U2 5‘-Kappe 3‘ U-snRNPs (Snurps) U4 U5 U6 biochemisch handelt es sich beim Spleißen um die Spaltung und Neubildung von Phosphodiester-Bindungen = Transesterifizierungs-Reaktionen (ohne Energie) das Spleißen beginnt mit einem nukleophilen Angriff der 2‘-OH-Gruppe des konservierten Adenins innerhalb des Introns auf die 3‘-5‘ Phosphodiester-Bindung am 5‘ Exon/Intron-Übergang Intron Exon22 Exon konserviertes A in TACTAAC-Sequenz es entsteht das Lasso-Intermediat (“lariat“) 5‘ Verzweigung 3‘ U-snRNPs (Snurps) U1 und U2 die 5‘-Spleißstelle und die Verzweigungsstelle U1 verläßt das Spliceosom U4, U5 und U6 lagern sich zusammen die verschiedenen U-snRNPs reagieren nacheinander mit den verschiedenen Spleißstellen, bis sich ein funktionsfähiges Spliceosom ausgebildet hat bei der Bindung von U4, U5 und U6 kommt es zum 5‘-Spleißen U6 hält Exon 1 im Spliceosom (Lasso-Bildung “lariat“) U2 und U4 verlassen das Spliceosom U5 und U6 verlassen das Spliceosom Ex1 Ex2 Wie können die Snurps am Spleißvorgang teilnehmen? U1 die snRNA-Moleküle haben an den Stellen, an denen keine Proteine gebunden sind, einzelsträngige RNA-Bereiche, die komplimentär zu den Consensus-Bereichen im Intron sind U1 bindet an die 5‘-Spleißstelle 5‘ 3‘ U2 bindet an die Verzweigungsstelle U5 bindet an die 3‘-Spleißstelle U1 U2 U1 hemmendes Oligonukleotid GUCCAUUCA Exon 1 Exon 2 Intron 5‘-Spleißstelle Verzweigungsstelle 3‘-Spleißstelle Hemmung des Spleißvorgangs durch Auto-Immunantikörper Hemmung des in vitro Spleißens durch Auto-Immunantikörper (Patienten mit systemischem Lupus erythematodes) Autoimmun-Krankheit: Antikörper gegen zelleigene Proteine; z. B. gegen die Sm-Proteine der U-snRNPs Gelenke und die meisten Organsysteme stark beeinträchtigt >>>> Schlüsselrolle der U-snRNPs beim Spleißen entdeckt mit Hilfe dieser AK Der Mechanismus des Prä-mRNA-Spleißens QuickTime™ and a Animation decompressor are needed to see this picture. Fragen aus der schriftlichen Physikumsprüfung Fragen aus der schriftlichen Physikumsprüfung Restriktionsendonuk leasen (A) sind am Spleißen von mR NA-Vorläufermolekülen b eteiligt (B) integrieren reve rse Transkripte der Retrovir us-RNA ins Geno m der Wirt sze ll e (C) spalten die 5’-Cap-Struktu r vom Ende de r mRNA (D) entfernen d ie Poly(A )-Sequenz vom 3’-Ende d er mRNA (E) sind Enzyme, die in Bak terien Phagen-D NA spalt en Fragen aus der schriftlichen Physikumsprüfung Fragen aus der schriftlichen Physikumsprüfung 9 kb = 9 000 BP/3 = 3 000 AS x 100 Da = 300 000 Da Fragen aus der schriftlichen Physikumsprüfung 52 Ein Gen enthält zwei Exons: Die Anzahl der Basen in der codierenden Sequenz beträgt 300 im Exon 1 und 600 im Exon 2. Die mittlere relative Molekülmasse der von diesen Exons codierten Aminosäuren ist 110/Aminosäure. Welche relative Molekülmasse wird für das codierte Protein mit einem Fehler von plus/minus 1000 bestimmt? (A) (B) (C) (D) (E) 3 000 10 000 33 000 100 000 330 000 Warum gibt es Introne in der prä-mRNA, die beim Reifung zur mRNA entfernt und abgebaut werden? Welche physiologische Bedeutung hat das Spleißen? Welche physiologische Bedeutung hat das Spleißen? warum Gene Introne haben ist in vielen Fällen noch unklar (Introne sind letztendlich Abfall!) Man kennt physiologisch-relevante Spleißvarianten: (1) alternatives Spleißen: erlaubt, aus einem einzigen Gen mehrere Genprodukte zu erhalten (Dogma 1 Gen > 1 Protein) (2) Trans-Spleißen: 5‘-Exon eines Gens wird an das 3‘-Exon eines anderen Gens gehängt >> dies ermöglicht der Zelle neue Genprodukte (Proteine) mit neuer Funktion zu schaffen Alternatives Spleißen (z. B. 3‘-alternativ) Exon 1 Exon 2 Alt.Exon Alt.Exon Intron Intron Alt.Exon Intron 3‘ Exon 1 Intron 5‘ Exon 1 Exon 2 Alt.Exon 3‘ Exon 2 Beispiele für alternatives Spleißen findet man bei den Immunglobulinen, Myosine, Hormonen etc. Der Lebenszyklus einer mRNA QuickTime™ and a Animation decompressor are needed to see this picture.