Fachinformation 42 (10/11) | Molekulare Onkologie
Werbung
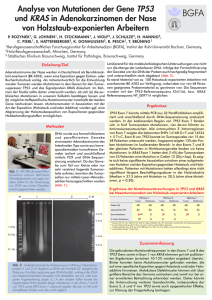
fachinfo42_20111011_LV2006-Master 12.10.11 14:57 Seite 1 Fachinformation 42 (10/11) | Molekulare Onkologie Next Generation Sequencing (NGS) in der Molekularen Pathologie Dipl.-Ing. (FH) Tanja Hinrichsen, Prof. Dr. med. Barbara Dockhorn-Dworniczak Wissenschaftlicher Hintergrund Bei der Behandlung von soliden Tumoren nimmt der Einsatz von molekularen Antikörpern (mAB) oder „Small Molecules“ (z.B. Tyrosinkinase-Inhibitoren, TKI) immer breiteren Raum ein. Der Behandlungserfolg ist allerdings abhängig vom Mutationsstatus bestimmter Gene. Während beim kolorektalen Karzinom (CRC) nur Patienten mit Wildtypsequenz im KRAS(Exon 2 und 3) bzw. BRAF-Gen (Exon 15) auf eine Therapie mit den mABs Cetuximab (Erbitux) oder Panitumumab (Vectibix) ansprechen, profitieren beim Nicht-kleinzelligen Lungenkarzinom (NSCLC) vor allem Patienten mit Mutationen im EGFR-Gen (Exon 18, 19, 20 und 21) von einer Therapie mit den TKI Erlotinib (Tarceva) und Gefitinib (Iressa). Allerdings können bestimmte Mutationen (sog. Escape-Mutationen) auch zur TherapieResistenz führen. Auch Patienten mit NSCLC, bei denen eine KRAS-Mutation vorliegt, zeigen ein Nichtansprechen gegenüber TKI. Patienten mit Gastrointestinalen Stromatumoren (GIST) weisen häufig Mutationen im c-KIT- (Exon 9, 11, 13 und 17) und im PDGFRa-Gen (Exon 12, 14 und 18) auf. Diese Mutationen können Einfluß auf die Sensitivität einer Behandlung mit Imatinib (Gleevec) nehmen bzw. Resistenzen gegenüber TKI verursachen. Tumorentität Gen KRAS Exon Mutationsstatus Therapiemöglichkeiten 2, 3 Wildtyp Cetuximab, Panitumumab BRAF 15 Wildtyp Cetuximab, Panitumumab Nicht-kleinzelliges Lungenkarzinom (NSCLC) EGFR 18, 19, 20, 21 mutiert* Erlotinib, Gefitinib KRAS 2, 3 Wildtyp Erlotinib, Gefitinib Gastrointestinale Stromatumoren (GIST) C-KIT 9, 11, 13, 17 mutiert* Imatinib PDGFRa 12, 14, 18 mutiert* Imatinib malignes Melanom BRAF 15 mutiert Vemurafenib kolorektales Karzinom (CRC) * auch Mutationen beschrieben, die zu einer Therapieresistenz führen Derzeit wird in der Routinediagnostik nicht der gesamte codierende Bereich der genannten Gene untersucht, da die Mutationsrate niedrig, die Mutationssuche aufwändig ist und das Untersuchungsmaterial meist eine Mischung zwischen Tumor- und Normalgewebe darstellt. Mit der herkömmlichen Sanger-Sequenzierung können Mutationen übersehen werden, da für eine sichere Detektion mindestens 20% der untersuchten DNA verändert sein muß. Durch NGS ist es jetzt möglich, mit hohem Durchsatz einzelne Templates (d.h. Einzelmoleküle) der zu untersuchenden DNA klonal zu amplifizieren und zu sequenzieren. Mit dieser Methode werden in einem parallelen Ansatz mehrere Gene bzw. Zielsequenzen in einem Tumor gleichzeitig mit hoher Abdeckung (Coverage) analysiert, wodurch Datenqualität und Informationsgehalt erheblich verbessert werden. Weiterhin können mit dieser Technologie Mutationen mit einer hohen Sensitivität vor einem Wildtyp-Hintergrund detektiert werden. Bei einer 1.000-fachen Abdeckung beispielsweise wird jedes Nukleotid der Zielsequenz 1.000-fach sequenziert und einzeln ausgelesen. Dies entspricht einer Nachweisgrenze von ca. 5% an veränderten DNA-Molekülen. Durch die klonale Amplifikation von Einzelmolekülen aus dem DNA-Gemisch werden sog. Allelic Dropouts vermieden. Auch dies trägt zu einer Erhöhung der Sensitivität bei. Z ENTRUM FÜR H UMANGENETIK UND L ABORATORIUMSMEDIZIN Dr. Klein und Dr. Rost n LOCHHAMER STR . 29 82152 M ARTINSRIED Tel. +49.89.895578-0 n fachinfo42_20111011_LV2006-Master 12.10.11 14:57 Seite 2 Fachinformation 42 (10/11) | Molekulare Onkologie Abb.: Deletionsnachweis (c.1652_1666del; p.Pro551_Val555del) in Exon 11 des C-KIT-Gens mit Sanger-Sequenzierung (links) und NGS (rechts). Die Datenqualität ist bei NGS besonders in Mischgewebe wie z.B. Tumoren der konventionellen Sanger-Sequenzierung überlegen. Indikation Therapieentscheidung bei: Diagnose CRC NSCLC GIST malignes Melanom Gen KRAS EGFR C-KIT BRAF Mutationsnachweis Exon 2, 3 Exon 18, 19, 20, 21 Exon 9, 11, 13, 17 Exon 15 Gen BRAF KRAS PDGFRa Mutationsnachweis Exon 15 Exon 2, 3 Exon12, 14, 18 Anforderung/Versand nach Rücksprache Material Je nach Tumorgröße 3-5 je 10 µm-Schnitte aus FFPE-Gewebe fixiert auf Objektträgern, sowie ein gesonderter Objektträger mit Tumor für die histopathologische Begutachtung oder Paraffinblöcke vom Tumor Methode Das eingesandte Material wird histopathologisch untersucht, entsprechende Tumorareale identifiziert und markiert, sowie für die DNA-Isolierung mikrodisseziiert. Nach PCRAmplifikation der Zielregionen wird mittels Emulsions-PCR weiter angereichert und klonal auf dem GS Junior (454 Life Sciences, Branford, CT, USA) sequenziert. Dauer der Untersuchung 7 Tage Literatur Heinemann et al, Cancer Treatment Reviews, 35:262 (2009) / Jönsson et al, BMC Clinical Pathology, 9:8 (2009) / Neumann et al, Pathol Res Pract, 205:858 (2009) / Richman et al, J Clin Oncol 27:5931 (2009) / Kalikaki et al, Lung Cancer 69:110 (2009) / Lasota et al, Histopathology, 53:245 (2008) In Zusammenarbeit mit dem ZENTRUM FüR PATHOLOGIE KEMPTEN-ALLGäU Prof. Dr. med. Barbara Dockhorn-Dworniczak, Dr. med. Claus Hirte, Prof. Dr. med. Laszlo Füzesi Robert-Weixler Str.48, 87439 Kempten, phone: +49-831-51299-0, fax: +49-831-51299-40 Zentrum für Humangenetik dr. klein und dr. rost und LaboratoriumsmediZin n LocHHamer str. 29 n 82152 martinsried