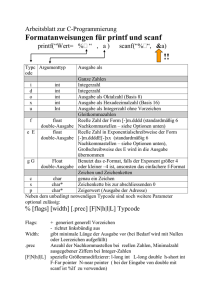
Graphalgorithmen
Werbung

ACM ICPC
Praktikum
Kapitel 10: Graphalgorithmen
Übersicht
•
•
•
•
•
•
•
Bäume
Minimale Spannbäume
Zusammenhang
Kürzeste Wege
Kreise
Planare Graphen
Netzwerkfluss und bipartites Matching
Bäume
• Ein Baum ist ein zusammenhängender Graph, der keine
Kreise enthält.
• Jeder Baum mit n Knoten hat
also genau n-1 Kanten.
• Ein Baum hat oft einen ausgezeichneten Knoten, der die
Wurzel repräsentiert.
• Jeder Knoten mit Grad 1 im
Baum heißt Blatt.
Bäume
• Gewurzelter Baum:
gerichteter Baum, in dem
jeder Knoten bis auf Wurzel
Eingrad 1 hat.
• Binärer Baum:
Jeder Knoten hat Ausgrad
2 oder 0.
• Spannbaum eines Graphen
G=(V,E): Teilgraph von G,
der ein Baum ist und alle
Knoten von G enthält.
Minimaler Spannbaum
• Gegeben: Graph G=(V,E) mit Kantenkosten
c:E ! IR
• Gesucht: Spannbaum T in G mit minimaler
Summe der Kantenkosten
• Algorithmen:
Starte mit leerer Kantenmenge. Wiederhole, bis
Spannbaum erreicht:
– Kruskal: füge Kante mit minimalem Gewicht unter
Wahrung der Kreisfreiheit hinzu
– Prim: erweitere Baum um minimale Kante
Prims Algorithmus
1. #define
MAXV 100
2. #define MAXDEGREE 50
3. typedef struct {
4.
int v;
5.
int weight;
6. } edge;
/* maximum number of vertices */
/* maximum outdegree of a vertex */
/* neighboring vertex */
/* edge weight */
7. typedef struct {
8.
edge edges[MAXV+1][MAXDEGREE];
/* adjacency info */
9.
int degree[MAXV+1];
/* outdegree of each vertex */
10.
int nvertices;
/* number of vertices in the graph */
11.
int nedges;
/* number of edges in the graph */
12. } graph;
Prims Algorithmus
1.
int parent[MAXV];
/* discovery relation */
2. prim(graph *g, int start)
3. {
4.
int i,j;
5.
bool intree[MAXV];
6.
int distance[MAXV];
7.
int v;
8.
int w;
9.
int weight;
10.
int dist;
11.
12.
13.
14.
15.
for (i=1; i<=g->nvertices; i++) {
intree[i] = FALSE;
distance[i] = MAXINT;
parent[i] = -1;
}
16.
17.
distance[start] = 0;
v = start;
/* counters */
/* is the vertex in the tree yet? */
/* distance vertex is from start */
/* current vertex to process */
/* candidate next vertex */
/* edge weight */
/* best current distance from start */
Prims Algorithmus
1.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19. }
while (intree[v] == FALSE) {
intree[v] = TRUE;
for (i=0; i<g->degree[v]; i++) {
w = g->edges[v][i].v;
weight = g->edges[v][i].weight;
if ((distance[w] > weight) && (intree[w] == FALSE)) {
distance[w] = weight;
parent[w] = v;
}
}
v = 1;
dist = MAXINT;
for (i=1; i<=g->nvertices; i++)
if ((intree[i] == FALSE) && (dist > distance[i])) {
dist = distance[i];
v = i;
}
}
Prims Algorithmus
1. main()
2. {
3.
graph g;
4.
int i;
5.
read_graph(&g,FALSE);
6.
prim(&g,1);
7.
printf("Out of Prim\n");
8.
9.
10.
11.
12.
13. }
for (i=1; i<=g.nvertices; i++) {
/*printf(" %d parent=%d\n",i,parent[i]);*/
find_path(1,i,parent);
}
printf("\n");
MST Probleme
• Maximum Spanning Tree: Maximiere
Kosten eines Spannbaums.
• Minimum Product Spanning Tree:
Minimiere Produkt der Kantenkosten
(´ minimiere Summe der Logarithmen)
• Minimum Bottleneck Spanning Tree:
Minimiere maximale Kantenkosten
(MST minimiert auch max. Kantenkosten)
Zusammenhang
• Ein ungerichteter (gerichteter) Graph ist (stark)
zusammenhängend, wenn es einen
ungerichteten (gerichteten) Weg zwischen zwei
beliebigen Knotenpaaren gibt.
• Jeder Graph, der auch nach Löschung eines
beliebigen Knotens noch zusammenhängend ist,
heißt zweifach zusammenhängend.
• Eine Kante, dessen Löschung den Graphen in
zwei Teilgraphen zerteilt, heißt Brücke.
Test auf Zusammenhang
• Einfacher Zusammenhang: DFS, BFS
• Starker Zusammenhang: Finde gerichteten Kreis
mittels DFS. Schrumpfe solch einen Kreis zu
einem einzelnen Knoten und wiederhole, bis
kein gerichteter Kreis mehr gefunden. Ergibt
einzelnen Knoten:
Graph start zusammenhängend.
• k-facher Zusammenhang: Teste, ob jedes
Knotenpaar Fluss der Größe >=k hat.
Kürzeste Wege
• Gegeben: Graph G=(V,E) mit
Kantenkosten c:E ! IR
• Weg von v nach w ist kürzester Weg,
wenn Summe der Kantenkosten minimal.
• Single-source-shortest-path: Dijkstra
Idee: arbeite ähnlich zu Prim, um einen
kürzeste-Wege-Baum aufzubauen
• All-pairs-shortest-path: Floyd-Warshall
Idee: verwende Matrixmultiplikation
Dijkstras Algorithmus
1.
int parent[MAXV];
2. dijkstra(graph *g, int start)
3. {
4.
int i,j;
5.
bool intree[MAXV];
6.
int distance[MAXV];
7.
int v;
8.
int w;
9.
int weight;
10.
int dist;
/* discovery relation */
/* was prim(g,start) */
/* counters */
/* is the vertex in the tree yet? */
/* distance vertex is from start */
/* current vertex to process */
/* candidate next vertex */
/* edge weight */
/* best current distance from start */
11.
12.
13.
14.
15.
for (i=1; i<=g->nvertices; i++) {
intree[i] = FALSE;
distance[i] = MAXINT;
parent[i] = -1;
}
16.
17.
distance[start] = 0;
v = start;
Dijkstras Algorithmus
1.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20. }
while (intree[v] == FALSE) {
intree[v] = TRUE;
for (i=0; i<g->degree[v]; i++) {
w = g->edges[v][i].v;
weight = g->edges[v][i].weight;
if (distance[w] > (distance[v]+weight)) {
distance[w] = distance[v]+weight;
parent[w] = v;
}
}
v = 1;
dist = MAXINT;
for (i=1; i<=g->nvertices; i++)
if ((intree[i] == FALSE) && (dist > distance[i])) {
dist = distance[i];
v = i;
}
}
/*for (i=1; i<=g->nvertices; i++) printf("%d %d\n",i,distance[i]);*/
Dijkstras Algorithmus
1. main()
2. {
3.
graph g;
4.
int i;
5.
6.
read_graph(&g,FALSE);
dijkstra(&g,1);
7.
8.
9.
for (i=1; i<=g.nvertices; i++)
find_path(1,i,parent);
printf("\n");
10. }
Floyd-Warshall Algorithmus
•
•
#define MAXV
#define MAXDEGREE
•
#define MAXINT
•
•
•
•
•
typedef struct {
int v;
int weight;
bool in;
} edge;
/* neighboring vertex */
/* edge weight */
/* is the edge "in" the solution? */
•
•
•
•
•
•
typedef struct {
edge edges[MAXV][MAXDEGREE];
int degree[MAXV];
int nvertices;
int nedges;
} graph;
/* adjacency info */
/* outdegree of each vertex */
/* number of vertices in the graph */
/* number of edges in the graph */
•
•
•
•
typedef struct {
int weight[MAXV+1][MAXV+1];
int nvertices;
} adjacency_matrix;
/* adjacency/weight info */
/* number of vertices in the graph */
100
50
/* maximum number of vertices */
/* maximum outdegree of a vertex */
100007
Floyd-Warshall Algorithmus
1.
2.
3.
4.
5.
6.
7.
8.
initialize_adjacency_matrix(adjacency_matrix *g)
{
int i,j;
/* counters */
g -> nvertices = 0;
for (i=1; i<=MAXV; i++)
for (j=1; j<=MAXV; j++)
g->weight[i][j] = MAXINT;
}
9. read_adjacency_matrix(adjacency_matrix *g, bool directed)
10. {
11.
int i;
/* counter */
12.
int m;
/* number of edges */
13.
int x,y,w;
/* placeholder for edge and weight */
14.
15.
initialize_adjacency_matrix(g);
scanf("%d %d\n",&(g->nvertices),&m);
16.
17.
18.
19.
20.
21. }
for (i=1; i<=m; i++) {
scanf("%d %d %d\n",&x,&y,&w);
g->weight[x][y] = w;
if (directed==FALSE) g->weight[y][x] = w;
}
Floyd-Warshall Algorithmus
1.
2.
3.
print_graph(adjacency_matrix *g)
{
int i,j;
4.
5.
6.
7.
8.
9.
10.
11. }
for (i=1; i<=g->nvertices; i++) {
printf("%d: ",i);
for (j=1; j<=g->nvertices; j++)
if (g->weight[i][j] < MAXINT)
printf(" %d",j);
printf("\n");
}
12. print_adjacency_matrix(adjacency_matrix *g)
13. {
14.
int i,j;
/* counters */
15.
16.
17.
18.
19.
20.
•
}
/* counters */
for (i=1; i<=g->nvertices; i++) {
printf("%3d: ",i);
for (j=1; j<=g->nvertices; j++)
printf(" %3d",g->weight[i][j]);
printf("\n");
}
Floyd-Warshall Algorithmus
1. floyd(adjacency_matrix *g)
2. {
3.
int i,j;
4.
int k;
5.
int through_k;
6.
7.
8.
9.
10.
11.
12.
13. }
/* dimension counters */
/* intermediate vertex counter */
/* distance through vertex k */
for (k=1; k<=g->nvertices; k++)
for (i=1; i<=g->nvertices; i++)
for (j=1; j<=g->nvertices; j++) {
through_k = g->weight[i][k]+g->weight[k][j];
if (through_k < g->weight[i][j])
g->weight[i][j] = through_k;
}
Floyd-Warshall Algorithmus
1. main()
2. {
3.
adjacency_matrix g;
4.
5.
read_adjacency_matrix(&g,FALSE);
print_graph(&g);
6.
floyd(&g);
7.
print_adjacency_matrix(&g);
8. }
Kreise
• Alle Graphen, die keine Bäume sind,
enthalten Kreise.
• Eulerkreis: Kreis, der jede Kante genau
einmal durchläuft.
• Hamiltonscher Kreis: Kreis, der jeden
Knoten genau einmal durchläuft.
Eulerkreis
• Eulerkreis im ungerichteten Graphen:
Existiert, wenn alle Knoten geraden Grad haben.
Algorithmus: Beginne bei beliebigem Knoten,
erweitere Kreis um beliebige Kante, bis wieder
am Ausgangspunkt.
Wiederholung ergibt kantendisjunkte Kreise, die
beliebige verschmolzen werden können.
• Eulerkreis im gerichteten Graphen:
Existiert, falls für jeden Knoten der Eingrad
gleich dem Ausgrad ist. Dann wie oben.
Hamiltonscher Kreis
• NP-vollständiges Problem, d.h. es gibt
aller Voraussicht nach keinen
Polynomialzeitalgorithmus dafür.
• Backtracking (mittels Durchlauf aller
möglichen Knotenpermutationen) kann
verwendet werden, falls Graph genügend
klein ist.
Planare Graphen
• Ein Graph ist planar, falls die Knoten und Kanten
so in 2D-Raum eingebettet werden können,
dass es keine Kantenüberschneidungen gibt.
• Ein Baum ist ein planarer Graph.
• Sei n Anzahl Knoten, m Anzahl Kanten und f
Anzahl Facetten, dann gilt n-m+f=2.
• Jeder planare Graph erfüllt m <= 3n-6.
• Ein Graph ist planar genau dann, wenn er
keinen K3,3 oder K5 als Graphminor enthält.
Netzwerkfluss
• Gegeben: gerichteter Graph G=(V,E) mit
Kantenkapazitäten c:E ! IR+ und Quell-Ziel-Paar
(s,t)
• Gesucht: Fluss f:E ! IR+ mit maximalem
Flusswert von s nach t.
• Fluss f ist legal, falls
– (u,v) f(u,v) = (v,w) f(v,w) für alle v 2 V n {s,t}
– f(e) <= c(e) für alle e 2 E
• Flusswert von f ist (s,w) f(s,w) - (u,s) f(u,s)
Netzwerkfluss
• O.B.d.A. sei G ein einfacher gerichteter
Graph, ansonsten Transformation:
neu
• Residuales Netzwerk von f: G’=(V,E’) mit
c’:E ! IR+, wobei c’(u,v)=c(u,v)-f(u,v) falls
(u,v) 2 E, c’(u,v) = f(v,u) falls (v,u) 2 E, und
sonst c’(u,v) = 0.
Netzwerkfluss
• Augmentierender Pfad p=(v1,…,vk) in G’:
Pfad mit positiven Kantenkosten
• Residuale Kapazität von p:
cp = mini c’(vi,vi+1)
• Neuer Fluss f’ =f+p:
f’(u,v) = f(u,v)+cp falls (u,v) 2 p und
f’(u,v) = f(u,v)-cp falls (v,u) 2 p
• Es gilt: Flusswert von f’ > Flusswert von f
und f maximal , kein augm. Pfad in G’
Ford-Fulkerson Algorithmus
1. #define
MAXV
2. #define MAXDEGREE
100
50
/* maximum number of vertices */
/* maximum outdegree of a vertex */
3. typedef struct {
4.
int v;
5.
int capacity;
6.
int flow;
7.
int residual;
8. } edge;
/* neighboring vertex */
/* capacity of edge */
/* flow through edge */
/* residual capacity of edge */
9. typedef struct {
10.
edge edges[MAXV][MAXDEGREE];
11.
int degree[MAXV];
12.
int nvertices;
13.
int nedges;
14. } flow_graph;
/* adjacency info */
/* outdegree of each vertex */
/* number of vertices in the graph */
/* number of edges in the graph */
Ford-Fulkerson Algorithmus
1.
2.
3.
4.
5.
6.
main()
{
flow_graph g;
int source, sink;
int flow;
int i;
/* graph to analyze */
/* source and sink vertices */
/* total flow */
/* counter */
7.
8.
scanf("%d %d",&source,&sink);
read_flow_graph(&g,TRUE);
9.
netflow(&g,source,sink);
10.
print_flow_graph(&g);
11.
12.
13.
flow = 0;
for (i=0; i<g.nvertices; i++)
flow += g.edges[source][i].flow;
14.
15. }
printf("total flow = %d\n",flow);
Ford-Fulkerson Algorithmus
1.
2.
3.
4.
initialize_graph(g)
flow_graph *g;
{
int i;
5.
6.
7.
8.
}
9.
10.
11.
12.
13.
14.
15.
read_flow_graph(g,directed)
flow_graph *g;
bool directed;
{
int i;
int m;
int x,y,w;
/* graph to initialize */
/* counter */
g -> nvertices = 0;
g -> nedges = 0;
for (i=0; i<MAXV; i++) g->degree[i] = 0;
16.
17.
18.
19.
20.
21.
22. }
initialize_graph(g);
scanf("%d %d\n",&(g->nvertices),&m);
for (i=1; i<=m; i++) {
scanf("%d %d %d\n",&x,&y,&w);
insert_flow_edge(g,x,y,directed,w);
}
/* graph to initialize */
/* is this graph directed? */
/* counter */
/* number of edges */
/* placeholder for edge and weight */
Ford-Fulkerson Algorithmus
1. insert_flow_edge(flow_graph *g, int x, int y, bool directed, int w)
2. {
3.
if (g->degree[x] > MAXDEGREE)
4.
printf("Warning: insertion(%d,%d) exceeds degree bound\n",x,y);
5.
6.
7.
8.
9.
g->edges[x][g->degree[x]].v = y;
g->edges[x][g->degree[x]].capacity = w;
g->edges[x][g->degree[x]].flow = 0;
g->edges[x][g->degree[x]].residual = w;
g->degree[x] ++;
10.
11.
12.
13.
14. }
if (directed == FALSE)
insert_flow_edge(g,y,x,TRUE,w);
else
g->nedges ++;
Ford-Fulkerson Algorithmus
1.
2.
3.
edge *find_edge(flow_graph *g, int x, int y)
{
int i;
/* counter */
4.
5.
6.
for (i=0; i<g->degree[x]; i++)
if (g->edges[x][i].v == y)
return( &g->edges[x][i] );
7.
8.
return(NULL);
}
9. add_residual_edges(flow_graph *g)
10. {
11.
int i,j;
12.
13.
14.
15.
16. }
/* counters */
for (i=1; i<=g->nvertices; i++)
for (j=0; j<g->degree[i]; j++)
if (find_edge(g,g->edges[i][j].v,i) == NULL)
insert_flow_edge(g,g->edges[i][j].v,i,TRUE,0);
Ford-Fulkerson Algorithmus
1.
2.
3.
print_flow_graph(flow_graph *g)
{
int i,j;
4.
5.
6.
7.
8.
9.
10.
11.
12.
13. }
/* counters */
for (i=1; i<=g->nvertices; i++) {
printf("%d: ",i);
for (j=0; j<g->degree[i]; j++)
printf(" %d(%d,%d,%d)",g->edges[i][j].v,
g->edges[i][j].capacity,
g->edges[i][j].flow,
g->edges[i][j].residual);
printf("\n");
}
14. bool processed[MAXV];
15. bool discovered[MAXV];
16. int parent[MAXV];
17. bool finished = FALSE;
/* which vertices have been processed */
/* which vertices have been found */
/* discovery relation */
/* if true, cut off search immediately */
Ford-Fulkerson Algorithmus
1. initialize_search(g)
2. flow_graph *g;
traverse */
3. {
4.
int i;
5.
6.
7.
8.
9.
10. }
/* graph to
/* counter */
for (i=1; i<=g->nvertices; i++) {
processed[i] = FALSE;
discovered[i] = FALSE;
parent[i] = -1;
}
Ford-Fulkerson Algorithmus
1.
2.
3.
4.
5.
bfs(flow_graph *g, int start)
{
queue q;
int v;
int i;
/* queue of vertices to visit */
/* current vertex */
/* counter */
6.
7.
8.
init_queue(&q);
enqueue(&q,start);
discovered[start] = TRUE;
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24. }
while (empty(&q) == FALSE) {
v = dequeue(&q);
process_vertex(v);
processed[v] = TRUE;
for (i=0; i<g->degree[v]; i++)
if (valid_edge(g->edges[v][i]) == TRUE) {
if (discovered[g->edges[v][i].v] == FALSE) {
enqueue(&q,g->edges[v][i].v);
discovered[g->edges[v][i].v] = TRUE;
parent[g->edges[v][i].v] = v;
}
if (processed[g->edges[v][i].v] == FALSE)
process_edge(v,g->edges[v][i].v);
}
}
Ford-Fulkerson Algorithmus
1. bool valid_edge(edge e)
2. {
3.
if (e.residual > 0) return (TRUE);
4.
else return(FALSE);
5. }
6.
7.
8.
9.
process_vertex(v)
int v;
{
}
10. process_edge(x,y)
11. int x,y;
12. {
13. }
/* vertex to process */
/* edge to process */
Ford-Fulkerson Algorithmus
1.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
find_path(start,end,parents)
int start;
/* first vertex on path */
int end;
/* last vertex on path */
int parents[];
/* array of parent pointers */
{
if ((start == end) || (end == -1))
printf("\n%d",start);
else {
find_path(start,parents[end],parents);
printf(" %d",end);
}
}
13. int path_volume(flow_graph *g, int start, int end, int parents[])
14. {
15.
edge *e;
/* edge in question */
16.
edge *find_edge();
17.
18.
19.
20.
21.
22.
23. }
if (parents[end] == -1) return(0)
e = find_edge(g,parents[end],end);
if (start == parents[end])
return(e->residual);
else
return( min(path_volume(g,start,parents[end],parents), e->residual) );
Ford-Fulkerson Algorithmus
1. augment_path(flow_graph *g, int start, int end, int parents[], int volume)
2. {
3.
edge *e;
/* edge in question */
4.
edge *find_edge();
5.
if (start == end) return;
6.
7.
8.
9.
10.
11.
e = find_edge(g,parents[end],end);
e->flow += volume;
e->residual -= volume;
12.
13. }
augment_path(g,start,parents[end],parents,volume);
e = find_edge(g,end,parents[end]);
e->residual += volume;
Ford-Fulkerson Algorithmus
1. netflow(flow_graph *g, int source, int sink)
2. {
3.
int volume;
/* weight of the augmenting path */
4.
add_residual_edges(g);
5.
6.
initialize_search(g);
bfs(g,source);
7.
volume = path_volume(g, source, sink, parent);
8.
9.
10.
11.
12.
13.
14. }
while (volume > 0) {
augment_path(g,source,sink,parent,volume);
initialize_search(g);
bfs(g,source);
volume = path_volume(g, source, sink, parent);
}
Anwendungen
• Maximum Matching in bipartiten Graphen.
• Gegeben: Graph G=(V,E)
• Matching: Teilmenge E’ ½ E, so dass jeder
Knoten max. Grad 1 hat.
• G bipartit genau dann, wenn zweifärbbar
(d.h. zwei Farben reichen, Knoten so zu
färben, dass keine zwei adjazenten
Knoten dieselbe Farbe haben)