4 Chiroptische Methoden
Werbung
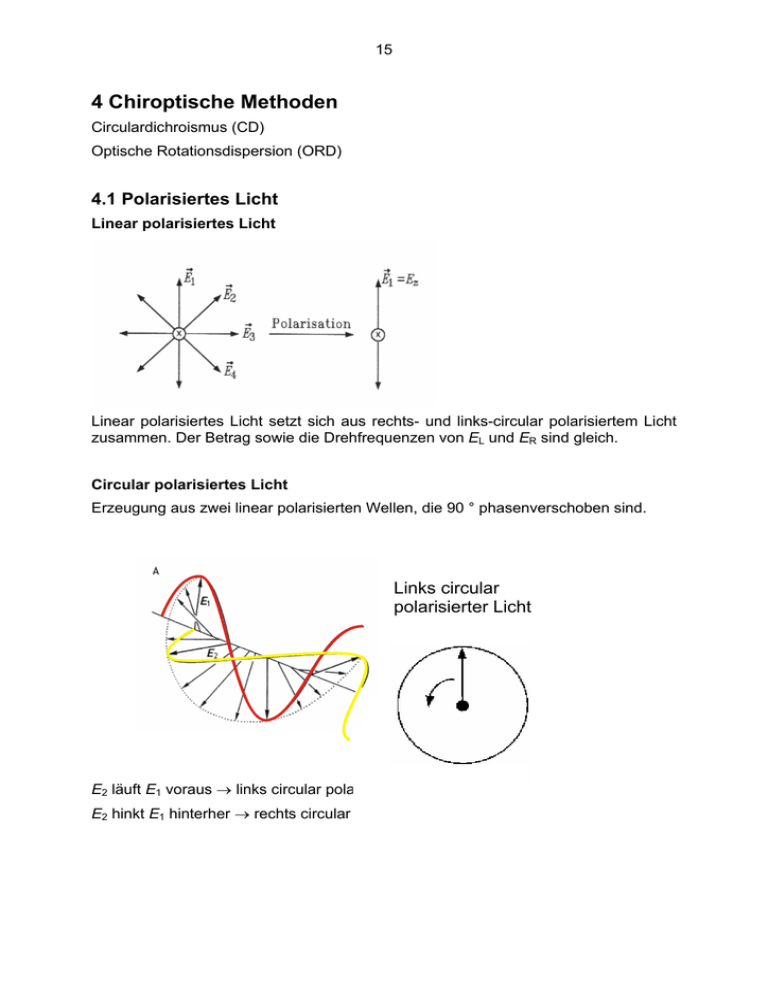
15 4 Chiroptische Methoden Circulardichroismus (CD) Optische Rotationsdispersion (ORD) 4.1 Polarisiertes Licht Linear polarisiertes Licht Linear polarisiertes Licht setzt sich aus rechts- und links-circular polarisiertem Licht zusammen. Der Betrag sowie die Drehfrequenzen von EL und ER sind gleich. Circular polarisiertes Licht Erzeugung aus zwei linear polarisierten Wellen, die 90 ° phasenverschoben sind. Links circular polarisierter Licht E E2 läuft E1 voraus → links circular polarisiertes Licht E2 hinkt E1 hinterher → rechts circular polarisiertes Licht 16 4.2 Circulardichroismus Die Extinktionskoeffizienten für rechts- und links circular polarisiertes Licht sind für eine optisch aktive Substanz unterschiedlich. Messaufbau Monochromator Eλ E λ,45° Billings- ER,L Sample cell Detector θ Modulator λ Double refracting material Computer Optical axes Optical axes Extraordinary beam Ordinary beam ⇒ different velocities Erzeugung von circular polarisiertem Licht mit dem λ/4-Plättchen bestehend aus einem doppelbrechenden Material definierter Dicke. Doppelbrechende Materialien sind aufgrund ihrer Gitterstruktur optisch anisotrop. In ihnen breitet sich das Licht inverschiedenen Richtungen mit unterschiedlichen Geschwindigkeiten aus. Bei Eintritt eines Lichtstrahls wir dieser in zwei Teilstrahlen aufgespalten, einen ordentlichen und einen außerordentlichen Strahl., die in aufeinander senkrecht stehenden Ebenen polarisiert sind. Wenn Licht senkrecht zur optischen Achse auftrifft, dann breiten sich die beiden Strahlen in der gleichen Richtung aus, aber ihre Geschwindigkeiten sind unterschiedlich. Sie verlassen den 17 Kristall mit erin Phasendifferenz, die von dessen Plattendicke und von der Wellenlänge des einfallenden Lichts abhängt. Benutzt man ein solches Plättchen mit einer Dicke von λ/4, so treten zwei senkrecht aufeinander stehende Strahlen, die um 90 ° phasenverschoben sind, aus dem Plättchen aus. Optisch aktive Moleküle Ein elektronischer Übergang ist erlaubt, wenn ein von Null verschiedenes Übergangsdipolmoment existiert. el µ21 = Ψ 2 | µ el | Ψ 1 Die Oszillatorstärke D21 = µ12el ⋅ µel21 ∼ ε (ν ) dν ∫ ν band Einige Übergänge sind elektronisch verboten, aber magnetisch erlaubt. Diese haben ein nicht verschwindendes magnetisches Übergangsmoment: m µ21 = Ψ 2 | µm | Ψ 1 Die Rotationsstärke ( Θ ν ) ∫ ν( )dν m R21 = Im µ 21el ⋅ µ21 ∼ band In symmetrischen Molekülen ist das magnetische Übergangsmoment immer senkrecht zum elektrischen Übergangsmoment, deshalb ist R21 = 0. Es muss eine Parallelkomponente von µel zu µm existieren. Jedes Molekül, welches kein Symmetriezentrum besitzt zeigt optische Aktivität (Quantentheorie der optischen Aktivität von Rosenfeld, 1929) Coronen Hexahelicen (optisch inaktiv) (optisch aktiv) 18 Was wird gemessen? Die Messgröße ist die Differenz der Extinktionskoeffizienten des rechts und links polarisierten Lichts. Also gilt nach Lambert-Beer: ∆E = EL − ER = ( ε L − ε R ) c d Angegeben wird allerdings die Elliptizität Θ: Θ= 1 180° ln10 (ε L − ε R ) c d π 4 b E ER Θ EL | EL |≈ TL b = ER + ΕL a a = ER − EL | ER |≈ TR | E |: Länge des E-Feld-Vektors des circular polarisierten Lichts (R: rechts, L: links) T : Transmission tan Θ = ER − EL ER + ΕL erweitern mit tanΘ = ( = TR − TL TR + TL TR + TL = a b ) liefert TR − TL TR + 2 TR TL + TL Der Unterschied zwischen TR und TL ist klein, daraus folgt: tan Θ ≅ Θ und TR + 2 TR TL + TL ≅ 4T 19 Θ= ∆T 4T mit ∆T = TR − TL Nach Lambert gilt: logT = −EL ( ) log T + ∆T = −ER und Reihenentwicklung um ∆T = 0: ( ) ln T + ∆T = lnT + ( ∆T T ) log T + ∆T = logT + ∆T 1 ⋅ T ln10 EL − ER = −(ER − EL ) = logT + ∆E = EL − ER = Θ= ∆T 4T = ∆T T ⋅ ∆T 1 ∆T 1 ⋅ − logT = ⋅ T ln10 T ln10 1 ln10 1 ⋅ ln10 ⋅ ∆E 4 ∆E = ε c d = ( ε L − ε R ) c d Θ= 1 180° ln10 (ε L − ε R ) c d π 4 Spezifische Elliptizität [ Θ] = Θ in cd grad cm2 g-1 zur Berechnung benötigt man die genaue Konzentration bei Proteinen: ε190 = 8.500 -11.400 M-1 cm-1 pro Aminosäurerest (nicht genau genug) ε280 (in 6 M GuHCl) = x Trp ⋅ 5.690 M-1 + y Tyr ⋅ 1.280 M-1 Molare Elliptizität [Θ]M = [Θ] M 100 in grad cm2 dmol-1 (100 hat historische Gründe) M = molare Masse des Moleküls 20 Mean residue weight ellipticity [Θ]MRW = [Θ] MRW 100 in grad cm2 dmol-1 MRW = mittlere molare Masse des Monomers des Polymers 4.2.1 Elektronische CD von Proteinen Circulardichroismus wird in dem Bereich der elektronischen Übergange der Moleküle gemessen. UV-Spektrum CD-Spektrum Poly-L-Alanin (α-Helix) Poly-L-Alanin (α-Helix) Bestimmung von Sekundärstrukturen in Proteinen Sind über ein Protein keine kristallographischen Daten verfügbar, so hilft die CDSpektroskopie, die Sekundärstrukturanteile in einem Protein zu bestimmen. 21 Poly-L-Lysin Annahme: Ein CD-Spektrum eines Proteins ist die Summe der CD-Spektren der einzelnen Sekundärstrukturelemente A) Bestimmung von Sekundärstrukturen mittels Referenzspektren von Poly-L-Lysin ΘT = χhelix Θhelix ( λ ) + χ sheet Θsheet ( λ ) + χrandom Θrandom ( λ ) B) Bestimmung von Sekundärstrukturen mittels Referenzspektren eines ProteinDatensatzes (Röntgenkristallstrukturdaten) CONTIN (S. Provencher, Comput. Phys. Commun. 1982, 27, 213-227, 229-242) SELCON3 (N. Sreerama & R.W. Woody, J.Mol.Biol. 1994, 242, 497-507) Varselec (L.A. Compton, L. A. & W. C. Johnson, Anal. Biochem. 1986, 155, 155167. P. Manavalan & W. C. Johnson, Anal. Biochem. 1987, 167, 76-85.) K2D - neural network method (Andrade et al. Prot Engineering 1993, 6, 383-390) 22 Hämoglobin Hemoglobin Tumor necrosis factor Eco RI α-Helix: β-Sheet: RC: α-Helix: β-Sheet: RC: 68 % 5% 27 % 68 % 0% 32 % Tumor Nekrose-Faktor Hemoglobin Tumor necrosis factor α Eco RI Endonuclease α-Helix: β-Sheet (antiparallel): β-Sheet (parallel): β-Turns: RC: 33 % 20 % 5% 17 % 27 % α-Helix: β-Sheet (antiparallel): β-Sheet (parallel): β-Turns: RC: 26 % 20 % 8% 25 % 21 % 23 Kinetische Betrachtung der Proteinfaltung und -entfaltung Rückfaltung von Lysozym nach Guanidiumhydrochlorid (6 M) Verdünnung. Insertion von Peptiden in Lipidmembranen offene Kreise: POPC geschlossene Kreise POPG cB: gebundenes Peptid cf: freies Peptid cL: Gesamt-Lipidkonzentration 24 4.2.2 Elektronische CD von Nukleinsäuren AMP: Purine besitzen keine Asymmetrie und demnach ist das Molekül optisch inaktiv. Eine leichte Asymmetrie wird in das Nukleotid durch die N-glykosidische Bindung zur Desoxyribose eingeführt, so dass optische Aktivität im CD-Spektrum beobachtet werden kann. dApA: Das Dinukleotid zeigt durch die Kopplung der beiden Basen bereits deutlich optische Aktivität. Poly-A: In einem Polymer liegt eine starke Kopplung vor, so dass die Elliptizität stark zunimmt. Zudem tragen die Anordnung der Basen im Raum zusätzlich zur optischen Aktivität bei. Sekundärstruktur von DNA CD-Spektren von DNA geben Aufschluss über die Konformation der DNA in Lösung. Pohl und Jovin (1972) entdeckten mittels CD-Spektroskopie die Z-Form der DNA.