Vorlesung Biophysik I - Molekulare Biophysik W. Kremer
Werbung

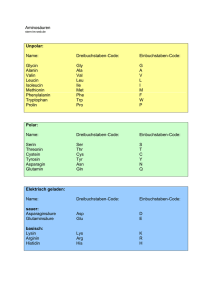
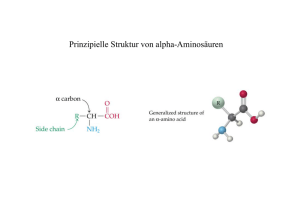
Vorlesung Biophysik I - Molekulare Biophysik W. Kremer • • • • • • • 24.10. 31.10. 07.11. 14.11. 21.11. 28.11. 05.12. • • 12.12. 19.12. • 09.01. • 16.01. • • • 23.01. 30.01. 06.02. Zelle Biologische Makromoleküle I Biologische Makromoleküle II Nukleinsäuren-Origami (DNA, RNA) Aminosäuren-Origami (Protein-Nanotechnologie) Molekulare Motoren Methoden zur Strukturbestimmung: Magnetische Resonanzspektroskopie I Grundlagen Magnetische Resonanzspektroskopie II - Mehrdimensionale NMR- Spektroskopie Magnetische Resonanzspektroskopie III – Proteinstrukturbestimmung, Dynamik und Bewegung, ESR-Spektroskopie Röntgenstrukturanalyse I – Streuung von Wellen, Faltungstheorem, Pattersonfunktion, Phasenproblem Röntgenstrukturanalyse II - Synchrotonstrahlung, zeitaufgelöste Kristallographie Röntgenkleinwinkelstreuung Elektronenmikroskopie I – Elektronenoptik, Kontrastentstehung und Bildinformation Elektronenmikroskopie II – Kristalline Objekte, Tomographie Rastertunnel- und Rasterkraftmikroskopie Quintessenz: Die Zelle ist aus einer Vielzahl von biologischen Makromolekülen aufgebaut und deren Interaktion und Organisation bestimmt die jeweilige physiologische und biologische Funktion. Ziel: Die Identifizierung dieser biologischen Makromoleküle und die Bestimmung der Struktur und der Interaktionspartner. Physikalische Untersuchungsmethoden für die Visualisierung und Bestimmung der Zellkompartimente und dann der einzelnen Makromoleküle auf atomarer Ebene. www.pdb.org Warum sind 3D-Strukturen wichtig?? Lehrsatz: Die räumliche Struktur von biologischen Makromolekülen bestimmt deren biologische Funktion. Beweis: Denaturierung ( = Zerstörung der geordneten 3D-Struktur) von Proteinen führt zum Funktionsverlust. Aktuelle Fragen: Wie kann man aus der räumliche Struktur von biologischen Makromolekülen deren biologische Funktion ableiten? Inwieweit bestimmt die biologische Funktion die räumliche Struktur von biologischen Makromolekülen? ff f f f Strukturelle Biologie Wie kann man die 3D-Struktur aus der Sequenz berechnen (vorhersagen)? f f f f f f Bioinformatik Bestimmung der räumlichen Struktur von Objekten Beugungsmethoden: - optische Mikroskopie - Elektronenmikroskopie - Röntgenkristallographie - Neutronenkristallographie → Wellenlänge λ < d NMR-Methoden: - MR-Tomographie - NMR-Strukturbestimmung → Wellenlänge λ >> d Modularer Aufbau biologischer Makromoleküle ist wichtiges Organisationsprinzip: Mit wenigen einfachen Elementen lassen sich komplexe Strukturen aufbauen. senkt Informationsbedarf senkt Syntheseaufwand Notwendig: standardisierte Verknüpfungen Modularität kann auf verschiedenen Ebenen angewandt werden (Elementarbausteine, Domänen) Hauptgruppen biologischer Makromoleküle Proteine Bausteine: Aminosäuren Nucleinsäuren Bausteine: Nucleotide Polysaccharide (Kohlenhydrate) Bausteine: Monosaccharide Lipide Bausteine: u.a. Fettsäuren Proteine (Polypeptide) Aminosäure Aminosäure Peptidbindung Aminosäure Peptidbindung Nucleinsäuren (Polynucleotide) Nucleotid Nucleotid Phosphodiesterbindung Nucleotid Phosphodiesterbindung Kovalente Verbindungen Kovalente Verbindungen (2) Kovalente Verbindungen (3) Chemische Verbindungen Chemische Verbindungen (2) Aminosäuren (Bausteine der Proteine/Peptide) Peptidbindung Aminosäuren (1) Aminosäuren (2) Die proteinogenen Aminosäuren Aminosäure EinbuchDreibuch- Polarität der Relative stabencode stabencode Seitenkette Häufigkeit [%] 1 Alanin A Ala unpolar 9.0 Arginin R Arg positiv 4.7 geladen Asparagin N Asn polar 4.4 Asparaginsäure D Asp negativ 5.5 geladen Cystein C Cys polar 2.8 Glutamin Q Gln polar 3.9 Glutaminsäure E Glu negativ 6.2 geladen Glycin G Gly unpolar 7.5 Histidin H His positiv 2.1 geladen2 Isoleucin I Ile unpolar 4.6 Leucin L Leu unpolar 7.5 Lysin K Lys positiv 7.0 geladen Methionin M Met unpolar 1.7 Phenylalanin F Phe unpolar 3.5 Prolin P Pro unpolar 4.6 Serin S Ser polar 7.1 Threonin T Thr polar 6.0 Tryptophan W Trp unpolar 1.1 Tyrosin Y Tyr polar 3.5 Valin V Val unpolar 6.9 1 2 Relative Häufigkeit der Aminosäuren in nicht-verwandten Proteinen. Bei neutralem pH liegt gewöhnlich ein Gleichgewicht zwischen positiv geladener und polarer Form der Seitenkette vor. Nukleotide (Bausteine der Nukleinsäuren) Basen (Komponenten der Nukleotide) Zucker (Komponenten der Nukleotide) Nomenklatur der Nukleotide Phosphodiesterbindung => Nukleinsäuren Weitere Funktionen von Nukleotiden Polysaccharide Glykosidische Bindung Monosaccharid Glykosidische Bindung Monosaccharid Monosaccharid Glykosidische Bindung Glykosidische Bindung Monosaccharid Monosaccharide Ringformation Ringformation Disaccharide Komplexe Oligosaccharide und Polysaccharide Lipide Lipid Lipid Lipid Lipid Lipid Lipid Fettsäuren Carboxylgruppen und Phospholipide Selbstorganisation von Lipidaggregaten Glykosilierte Lipide Weitere Lipide/Steroide Hauptgruppen biologischer Makromoleküle Proteine Bausteine: Aminosäuren Nucleinsäuren Bausteine: Nucleotide Polysaccharide (Kohlenhydrate) Bausteine: Monosaccharide Lipide Bausteine: u.a. Fettsäuren Protein structures are determined through weak non-covalent interactions Protein structures are determined through weak non-covalent interactions Schwache nicht-kovalente Wechselwirkungen Proteine Nukleinsäuren Polysaccharide (Kohlenhydrate) Lipide Vorlesung Biophysik I - Molekulare Biophysik W. Kremer • • • • • • • 24.10. 31.10. 07.11. 14.11. 21.11. 28.11. 05.12. • • 12.12. 19.12. • 09.01. • 16.01. • • • 23.01. 30.01. 06.02. Zelle Biologische Makromoleküle I Biologische Makromoleküle II (anderer Termin !!!!!) 21.11 Nukleinsäuren-Origami (DNA, RNA) hier auch anderer Termin !!!!! 21.11 Aminosäuren-Origami (Protein-Nanotechnologie) Molekulare Motoren Methoden zur Strukturbestimmung: Magnetische Resonanzspektroskopie I Grundlagen Magnetische Resonanzspektroskopie II - Mehrdimensionale NMR- Spektroskopie Magnetische Resonanzspektroskopie III – Proteinstrukturbestimmung, Dynamik und Bewegung, ESR-Spektroskopie Röntgenstrukturanalyse I – Streuung von Wellen, Faltungstheorem, Pattersonfunktion, Phasenproblem Röntgenstrukturanalyse II - Synchrotonstrahlung, zeitaufgelöste Kristallographie Röntgenkleinwinkelstreuung Elektronenmikroskopie I – Elektronenoptik, Kontrastentstehung und Bildinformation Elektronenmikroskopie II – Kristalline Objekte, Tomographie Rastertunnel- und Rasterkraftmikroskopie