Graphen - Fakultät Statistik
Werbung

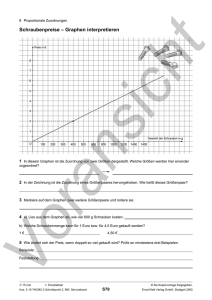
Graphen Graphen Seminar Bioinformatik Franziska Schwabe Fakultät Statistik TU Dortmund 3. Juli 2009 1 / 45 Graphen Gliederung 1 Graphentheorie Denitionen Spezielle Graphen Algorithmen 2 Bioconductor Software für Graphen Das Paket Das Paket Das Paket graph RBGL Rgraphviz 2 / 45 Graphen Was sind Graphen? Denition [wikipedia.de] Ein Graph besteht in der Graphentheorie anschaulich aus einer Menge von Punkten, zwischen denen Linien verlaufen. Die Punkte nennt man Knoten oder Ecken, die Linien nennt man meist Kanten, manchmal auch Bögen. Auf die Form der Knoten und Kanten kommt es im allgemeinen dabei nicht an. Knoten und Kanten können auch mit Namen versehen sein, dann spricht man von einem benannten Graphen. Wo werden Graphen verwendet? Bioinformatik Operations Research 3 / 45 Graphen Wie sieht ein Graph aus? s p q r 4 / 45 Graphen 5 / 45 Graphen 6 / 45 Graphen Beispiel GO-Graph Kategorisierung von Gen-Produkten Grundlage: gerichteter azyklischer Graph Kind- und Elter-Objekte in R: GO.db, GOstats 7 / 45 Graphen Graphentheorie Denitionen Notation Graph G = (V , E ) V: nicht-leere Menge von Knoten (engl. vertex) E: nicht-leere Menge von Kanten (engl. edges) |V |: Anzahl der Knoten ( ): V G Knotenset von G oder Menge der Knoten von G /U beschreibt Graph H mit V (H ) = V (G )/V (U ) und ( ) = E (G )/F (G , U ) mit F (G , U ) das Kantenset von der Kanten aus G , die Endpunkte in V (U ) haben. Graph H heiÿt G E H Untergraph von G Graph J heiÿt Teilgraph von G , falls V (J ) = V (G ) und ( ) ⊂ E (G ) E J 8 / 45 Graphen Graphentheorie Denitionen Kanten Kanten stehen für binäre Verbindungen zwischen zwei Knoten. Spezielle Kanten proper edge self-loop: verbindet Knoten mit sich selbst multi edge : mehrere Kanten haben dieselben Endpunkte directed edge : Kante mit zugehöriger Richtung Benennung von Knoten und Kanten Standardknotenbenennung: Eins-zu-eins Abbildung zwischen den ganzen Zahlen von 1 bis |V | und den Knoten von G Standardkantenbenennung: Benenne die Kante zwischen Knoten 1 und 2 mit 1 und so fortlaufend. Standardbenennung berücksichtigt keine multi edges und keine self-loops. sinnvoll für die Anwendung von Algorithmen auf Graphen 9 / 45 Graphen Graphentheorie Denitionen Kantenfolge Eine Kantenfolge W = hv0 , e1 , v1 , ..., v n −1 , en , vn i ist eine Abfolge von Knoten und Kanten, sodass die Endpunkte von Kante ei vi −1 und vi für i = 1, ..., n sind. Länge der Kantenfolge = ( Anzahl der abgelaufenen Kanten ungewichtete Kanten Summe der Kantengewichte gewichtete Kanten geschlossene Kantenfolge ⇔ v0 =v n Knoten v heiÿt erreichbar von Knoten u ⇔∃ Kantenfolge W von u zu v → Ein Graph G heiÿt verbunden ⇔ Zwischen je zwei Knoten besteht eine Kantenfolge. 10 / 45 Graphen Graphentheorie Denitionen Kantenfolge Sind alle Kanten paarweise verschieden, so heiÿt die Kantenfolge Pfad. Sind alle Knoten paarweise verschieden, so heiÿt die Kantenfolge Weg. Ein Weg mit v0 =v n heiÿt geschlossener Weg oder Kreis. 11 / 45 Graphen Graphentheorie Spezielle Graphen Spezielle Graphen Sind in einem Graphen G = (V , E ) die Kanten gerichtete → Kanten ( directed edges ), so heiÿt der Graph G gerichteter Graph oder Digraph. → Ein Graph G (bzw. Digraph G ) zusammen mit einer Abbildung c :E →R heiÿt ein (kanten-) gewichteter Graph (bzw. (kanten-) gewichteter Digraph). Ein Graph G heiÿt zusammenhängend, wenn es zwischen je zwei Knoten von G eine Kantenfolge gibt. Maÿ: Konnektivität k (G ) = minimale Anzahl der Kanten, deren Beseitigung einen unverbundenen Graphen verursacht ( )=l k G Graph G heiÿt l -verbunden oder l -zusammenhängend. 12 / 45 Graphen Graphentheorie Spezielle Graphen Bipartite Graphen Die Knoten des bipartiten Graphen G = (V , E ) können in zwei Sätze U und W geteilt werden, so dass jede Kante in E eine ungerichtete Beziehung zwischen einem Knoten in U und einem Knoten in W darstellt. Es existieren keine Kanten zwischen den Elementen innerhalb von U oder W allgemein gebräuchliche Darstellung: Die n m = |W | = |U | kreuz Adjazenzmatrix A mit ( Ai ,j = 1 es gibt Kante e von ui nach wj 0 sonst i = 1, ..., n; j = 1, ..., m. Gewichtungen oder Richtungen der Kanten können einfach eingepasst werden. 13 / 45 Graphen Graphentheorie Spezielle Graphen Beispiel für einen bipartiten Graphen PubMed und LocusLink PubMed: Datenbank für medizinische und biologische Paper LocusLink: Gen-Datenbank PM = Knotenmenge LL = Knotenmenge Verbindung durch Kante besteht: Gen kommt in Paper vor Adjazenzmatrix A mit Spalte = Paper und Zeile = Gen 14 / 45 Graphen Graphentheorie Spezielle Graphen Hypergraphen eng verbunden mit bipartiten Graphen generalisieren das Graphkonzept für die Spezialisierung von Beziehungen der Form eine zu vielen und viele zu vielen Ein Hypergrah ist deniert durch das Paar (V , E ), wobei V ein Knotenmenge und E eine Menge von Hyperkanten zwischen den Knoten. Jede Hyperkanten verbindet gleichzeitig mehrere Knoten. Gerichtete Azyklische Graphen Gerichtete Graphen ohne Kreise wichtige Klasse der gerichteten Graphen sind die Basis graphischer Modelle Beispiel in strukturierenden Konzepten: GO 15 / 45 Graphen Graphentheorie Spezielle Graphen Zufallsgraphen wichtig für statistisches Testen Generierung von Zufallsgraphen in Bioconductor: festgesetzte Menge an Knoten zufällig generierte Kanten Allgemein gebräuchliches Vorgehen: 1 Festsetzen der Anzahl an Konten 2 Festsetzen der erforderlichen Anzahl von Kanten 3 Graph wird durch Ziehen von Kanten ohne Zurücklegen aus einer Menge von n 2 = n(n − 1)/2 möglichen Kanten generiert. Zufallsgraphen können auch in Form von der Ausgangs- und Eingangsgrad Verteilungen der Knoten modelliert werden. Ein dritter Typ von Zufallsgraphen wird generiert durch die Annahme der Existenz einiger Knoteneigenschaften, die die Anwesenheit und möglicherweise das Gewicht der Kanten zwischen ihnen bestimmen. 16 / 45 Graphen Graphentheorie Algorithmen Erforschung und zugehörige Algorithmen Strategien, um Eigenschaften der Knoten oder Wege von speziellem Interesse zu entdecken Vorgehen: Start bei einem bestimmten Knoten und passiere alle Kanten und die übrigen Knoten auf spezielle Art und Weise 1 Breadth First Search (BSF): Besuch aller angrenzenden Knoten, bevor deren Nachfolger besucht werden. 2 Depth First Search (DFS): Ausgehend von einem Knoten wird der Knoten besucht, der tiefer im Graphen liegt. 17 / 45 Graphen Graphentheorie Algorithmen Zusammenhängende Untergruppen Bedürfnis nach einem Begri für zusammenhängende Untergruppen 1 n-Cliquen 2 k -Plex 3 k -Kern 4 λ-Menge Gs G bezeichnet im folgenden einen Untergraphen von = (V , E ) mit Knoten Vs und Kanten Es , deniert als Elemente aus E mit beiden Endknoten in Vs . 18 / 45 Graphen Graphentheorie Algorithmen n -Cliquen Subgraph mit Knoten Vs so, dass der Abstand d (v , u ) zwischen Knoten u und v die Forderung d (v , u ) Knoten v , u ∈V s ≤n für alle erfüllt k -Plex Maximaler Untergraph, in dem jeder Knoten nicht weniger als vs −k benachbarte Knoten hat ( ) degs u bezeichne den Grad des Knoten u innerhalb des Untergraphs Vs ⇒ deg (u ) ≥ v − k ∀ u ∈ V s s s 19 / 45 Graphen Graphentheorie Algorithmen k -Kern ähnlich wie ein k -Plex deniert Hauptunterschied: Minimale Anzahl an Kanten, die im Graphen existieren müssen, ist speziziert stattdessen: Maximalanzahl nicht notwendig λ-Menge Suche nach Regionen, in denen die Konzentration von Kanten zwischen den Knoten höher ist als die Konzentration von Kanten von dieser Region aus zum Rest des Graphen. λ(w , u ) bezeichne die Minimalanzahl zu entfernender Kanten, so dass kein Weg zwischen den Knoten u und v besteht. Für Graph G , ,w ∈ W u v = (V , E ) ist W /V eine λ-Menge, ∈ V /W : λ(u , v ) ≥ λ(w , l ). falls für alle und l Die Elemente einer λ-Menge müssen nicht benachbart sein. 20 / 45 Graphen Bioconductor Software für Graphen Wünsche 1 Erzeuge Datenstrukturen, die Graphen repräsentieren und leicht im bioinformatischen Arbeiten und in statistischer Software verwendet werden können. 2 Finde Algorithmen, für die Analyse von Graphen Beispiele: Kürzester-Weg, Schnittmengen Bestimmung, Zusammenhangsmaÿe, Spaltung. 3 Gebraucht werden Methoden für Layout, Benennung und Visualisierung von Graphenstrukturen. Biconductor Pakete 1 2 3 graph RBGL Rgraphviz 21 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Was kann graph? Inhalt: fundamentalen Klassen und Methoden zur Handhabung von Graphen RBGL und Rgraphviz beruhen auf den von graph erzeugten Datenstrukturen Graphen haben verschiedene aber äquivalente Darstellungen Eignung einer Darstellungsform ist abhängig von Faktoren wie: Gröÿe des Graphen, erwünschte Anwendung ⇒ von Interesse: Abdeckung extremer Bereiche der Graphengröÿe Graphendarstellung in R: Auistung seiner Knoten und Kanten Alternative Darstellung: Adjazenzmatrix (besonders geeignet für Digraphen) graph beinhaltet auch Möglichkeiten, die Darstellung ineinander umzurechnen 22 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Arbeiten mit graph R-Code library("graph") e <- c("s", "p", "q", "r") k <- list(s=list(edges=c("p","q")), p=list(edges=c("p","q")), q=list(edges=c("p","r")), r=list(edges=c(ÿ"))) g <- new("graphNEL", nodes = e, edgeL = k, edgemode= "directed") 23 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Wie sieht ein Graph aus? s p q r 24 / 45 Graphen Bioconductor Software für Graphen Das Paket graph g A graphNEL graph with directed edges Number of Nodes = 4 Number of Edges = 7 nodes(g) "s" "p" "q" "r" edges(g) $s "p" "q" $p "p" "q" $q "p" "r" $r "s" 25 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Graph a b e f g c d 26 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Nachbarschaft und Erreichbarkeit adjacent = angrenzend adj(g2, "c") $c "a" "b" "d" accessible = erreichbar acc(g2, c("b", "f")) $b a c b 3 1 2 $f g 1 27 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Abbildung: KEGG 28 / 45 Graphen Bioconductor Software für Graphen Das Paket graph R-Code data("integrinMediatedCellAdhesion") class(IMCAGraph) s <- acc(IMCAGraph, "SOS") Ergebnisse Protein Anzahl überwundener Kanten Ha-Ras 1 Raf 2 MEKI 3 ... 29 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Ausgänge R-Code deg <- degree(IMCAGraph)$outDegree deg[which.max(deg)] Ergebnis ITGB hat 6 ausgehende Kanten 30 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Sicherung Funktionen die Manipulationen durchführen, die den Graphen nachhaltig verändern, werden an einer Kopie durchgeführt. clearNode combineNodes 31 / 45 Graphen Bioconductor Software für Graphen Das Paket graph Zufallsgraphen Generierung in R randomEGraph randomNodeGraph randomGraph Beispiel nodeNames <- sapply(0:99, function(i) sprintf("N%02d",i)) rg <- randomEGraph(nodeNames, edges = 50) 32 / 45 Graphen Bioconductor Software für Graphen Das Paket graph N66 N58 N00 N34 N89 N65 N55 N04 N59 N54 N72 N86 N46 N06 N71 N29 N11 N22 N61 N95 N07 N28 N01 N39 N25 N19 N60 N45 N73 N32 N13 N94 N53 N50 N93 N36 N41 N99 N18 N74 N76 N05 N42 N49 N03 N75 N24 N92 N09 N84 N82 N47 N37 N48 N02 N10 N23 N90 N69 N40 N70 N98 N97 N77 N14 N35 N80 N31 N12 N64 N27 N67 N88 N26 N16 N51 N83 N44 N08 N33 N56 N68 N38 N52 N20 N96 N17 N79 N30 N91 N15 N21 N57 N43 N78 N81 N85 N62 N63 N87 33 / 45 Graphen Bioconductor Software für Graphen Das Paket RBGL Was kann RBGL? BFS und DFS Kürzeste Wege Minimale Bäume Aussagen zur Konnektivität Maximaluss Algorithmen viele weitere Funktionen 34 / 45 Graphen Bioconductor Software für Graphen Das Paket RBGL Kürzester Weg set.seed(123) rg2 <- randomEGraph(nodeNames, edges = 100) fromNode <- "N43" toNode <- "N81" sp <- sp.between(rg2, fromNode, toNode) sp[[1]]$path [1] "N43" "N08" "N88" "N73" "N50" "N89" [7] "N64" "N93" "N32" "N12" "N81" sp[[1]]$length [1] 10 35 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz Was kann Rgraphviz? verschiedene Layouts Allgemeine Eigenschaften von Graphen, wie Gröÿe und Hintergrundfarbe Eigenschaften der Knoten, wie Beschriftungen, Füll- und Rahmenfarbe Eigenschaften der Kanten, wie Beschriftungen, Linienstil und Farben Eigenschaften können für einzelne Knoten und Kanten oder für alle gewählt werden 36 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz 3 Layout Typen 1 dot layout hierarchisches Layout für gerichtete Graphen 2 neato layout möglichst keine Überlappung und dieselbe Länge der Kanten 3 twopi layout zyklische Anordnung um ein Zentrum 37 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz dot layout N00 N01 N03 N05 N06 N10 N04 N08 N02 N09 N07 38 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz neato layout N09 N10 N03 N06 N04 N02 N01 N07 N00 N08 N05 39 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz twopi layout N04 N07 N00 N05 N09 N06 N02 N08 N10 N03 N01 40 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz Farbspiel nodeA <- list(fillcolor = "lightblue") edgeA <- list(color = "goldenrod") attrs <- getDefaultAttrs(list(node=nodeA, edge =edgeA)) plot(g, attrs = attrs) 41 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz Graph mit Farben s p q r 42 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz Veränderung der Knoten- und Kanteneigenschaften globA <- list(node = list(width = "3", height = "1", shape = "box")) nodeA <- list(label = c(p = "YNL201C", q = "PpH3", r = "YBL046W", s = ßpt5"), shape = c(p = " ellipse", q = "circle")) edgeA <- list(label = c("p~q" = ÿtrong", "r~s" = "weak"), color = c("p~q" = "red", "r~s" = "blue")) plot(g, attrs = globA, edgeAttrs = edgeA, nodeAttrs = nodeA) 43 / 45 Graphen Bioconductor Software für Graphen Das Paket Rgraphviz Graph mit Knoten- und Kanteneigenschaften Spt5 YNL201C strong weak Pph3 YBL046W 44 / 45 Graphen Literatur Literatur Battista,G. D., Eads, R., Tamassia, R., et al.(1999): Graph Drawing: Algorithms for the visualisation of graphs., Prentice Hall, Upper Saddle River Genry, J.Carey, V.Ganser, E.(2004): Laying out pathways with Rgraphvi.z, R News, 4(2): 14-18, URL http://CRAN.R-project.org/doc/Rnews Gentlemann, R., Carey, V., et al.(2005): Bioinformatics and Computational Biology Solutions Using R and Bioconductor, Springer- Verlag, Berlin. 45 / 45