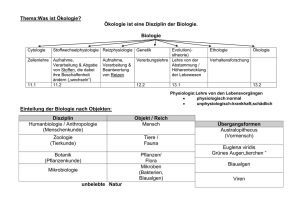
Endbericht: Synthetische Biologie
Werbung

Synthetische Biologie am Prüfstand Schule HLFS Ursprung Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 ZUSAMMENFASSUNG INHALTSVERZEICHNIS Zusammenfassung ........................................................................................... 3 Motivation ....................................................................................................... 5 Was ist Synthetische Biologie? ......................................................................... 6 Enzyme ............................................................................................................ 8 Synthetische Enzyme ........................................................................................... 10 Die Katalase ......................................................................................................... 11 Bacillus subtilis .............................................................................................. 12 Die Arbeit im Labor - Katalase aus Bacillus subtilis .................................... 14 Ergebnisse ...................................................................................................... 18 Auswertung mittels Proteingelen (SDS) ............................................................... 19 Messungen am Massenspektrometer ................................................................... 21 Diskussion zur Biosicherheit mit Experten .................................................. 22 Schuljahre 09/10 10/11 Impressum: Höhere land- und forstwirtschaftliche Schule HLFS Ursprung Ursprungstr. 4 5161 Elixhausen 0043 662 480301 http://www.ursprung.at http://synbio.ursprung.at Kontakt: [email protected] 2 Max-Planck-Institut Blog .............................................................................. 24 Erlernte Methoden ................................................................................................ 27 Eindrücke / Statements ................................................................................. 30 Sponsoren / Partner ....................................................................................... 34 Team................................................................................................................ 35 Synthetische Biologie (kurz: SynBio) ist ein neues, aufstrebendes Forschungsgebiet, dem sich weltweit immer mehr WissenschaftlerInnen widmen. Es beschäftigt sich mit der Herstellung von Genen und Proteinen, die als synthetische Produkte maßgeschneiderte Eigenschaften haben können. Die Synthetische Biologie verspricht, unsere Alltagswelt auf ähnliche Weise zu verändern wie es die Synthetische Chemie ein Jahrhundert zuvor getan hat. So selbstverständlich heute künstliche Produkte der chemischen Industrie, wie z.B. Kunststoffe, unser Leben in allen Bereichen (oft unbewusst) prägen, so selbstverständlich könnten in wenigen Jahrzehnten Produkte der Synthethischen Biologie Bestandteil unseres Alltags sein. Fest steht, dass früher oder später die Synthetische Biologie die klassische Gentechnik ablösen wird. Man erhofft sich vieles von der SynBio - welche der in sie gesetzten Erwartungen sie erfüllen wird, ist allerdings ungewiss. Ein Teilgebiet der SynBio befasst sich mit dem Einbau von nicht-kanonischen (= synthetischen) Aminosäuren in Proteine. Man versucht hier gezielt, die Bedeutung bestimmter Codewörter auf der DNA (die bei allen Lebewesen gleich sind) so zu verändern, dass Anstelle von kanonischen Aminosäuren synthetische eingebaut werden. Da ein Organismus Aminosäuren dieser Art nicht selbst herstellen kann, muss man ihm diese über das Nährmedium im Fermenter anbieten. Werden die nicht-kanonischen Aminosäuren tatsächlich vom Organismus aufgenommen und zum Bau von Proteinen verwendet, entstehen sogenannte ‚synthetische Eiweißmoleküle‘, die neuartige - mehr oder weniger nützliche - Eigenschaften haben können. Interessant ist, dass die Natur die immer- gleichen 20 kanonischen Aminosäuren selbstverständlich in unterschiedlicher Reihenfolge und Anzahl - ‚verbaut‘, der Wissenschaft jedoch über 700 verschiedene bekannt sind. In diesem Vergleich wird deutlich, wie viele Möglichkeiten es für ForscherInnen gibt, neue Proteine und Enzyme zu gestalten bzw. welches Potential in der Synthetischen Biologie steckt. 17 SchülerInnen der HLFS Ursprung setzten es sich zum Ziel, eine synthetische Katalase zu produzieren. Die Katalase ist ein Enzym, also ein Biokatalysator, der für Zellen schädliches - Wasserstoffperoxid neutralisiert. Auf Anfrage der SchülerInnen bot das Max-Planck-Institut für Biochemie in München (MPI) seine Hilfe und sein Know-How an. Und tatsächlich: Zur Überraschung des Projektteams und vor allem der WissenschaftlerInnen vom MPI gelang es, im Schullabor mittels eines auxotrophen Bacillus subtilis-Stammes 3 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 MOTIVATION Durch zahlreiche Artikel in verschiedenen Zeitungen (z.B. „Die Zeit“, „Profil“ , „Der Spiegel“ , „Spektrum der Wissenschaft“, ...) wurde uns klar: Synthetische Biologie ist ein brandheißes Thema! Schnell war unser Interesse geweckt und wir beschlossen, unser heuriges Projekt diesem neu aufkommenden Forschungszweig zu widmen. eine solche Katalase zu erzeugen. (Auxotroph bedeutet, der verwendete Bakterienstamm kann mehrere lebensnotwendige Aminosäuren nicht selbst herstellen und muss diese über die Nährlösung erhalten - d.h. unser Bakterienstamm würde ohne die künstliche Nährlösung nicht überleben.) Die Massenspektrometrie am MaxPlanck-Institut zeigte den Einbau der fremden Aminosäure Ethionin an mindestens sieben Stellen in der Aminosäurekette des Proteins. Es handelt sich damit um die erste synthetische Katalase weltweit! Welche neuen Eigenschaften dieses Enzym aufweist muss nun erforscht werden: Wie aktiv ist diese synthetische Katalase? Unter welchen Umgebungsbedingungen arbeitet sie? Wie gut ist sie industriell einsetzbar? Wurde die Anti-Aging-Wirkung verstärkt? Weiters haben unsere Forschungen gezeigt, dass Bacillus subtilis sehr einfach für die Methoden der SynBio verwendet werden kann. Dies war bisher „unerforschtes Land“. Schließlich interessierte uns auch der ethische Aspekt am Thema SynBio. Wir erfuhren, dass es in Österreich in diesem Bereich noch kaum gesetzliche Regelungen gibt. Angesichts der zahlreichen Risiken, die die Synthetische Biologie neben ihren Chancen gewiss mit sich bringt, sahen wir hier dringenden Forschungs- und Aufklärungsbedarf! Wir setzten uns deshalb zum Ziel, mit unserem Projekt auch eine öffentliche Diskussion zum Themenkomplex Synthetische Biologie auszulösen. Die brei- 4 WissenschaftlerInnen aus aller Welt sehen ein unglaubliches Zukunftspotential in dieser Technologie, die unser Leben in einigen Jahren ähnlich verändern könnte wie es die chemische Industrie vor einem Jahrhundert mit neuen, künstlichen Werkstoffen getan hat. Und man denke nur einmal darüber nach, wie sehr unser Leben durch die Einführung von künstlichen Werkstoffen in Form von Verpackungen, Baustoffen, Klebstoffen, Isolierungen, etc. umgekrempelt wurde. Ohne diese Materialien wäre der heutige Alltag nicht vorstellbar. te Bevölkerung sollte angemessen über die neue Technologie informiert werden und sich eine Meinung bilden können. Zum Auftakt dieses Vorhabens luden wir am 14. Dezember 2009 Dr. Markus Schmidt von der Organisation für Internationalen Dialog und Konfliktmanagement (IDC) in Wien und Dr. Nediljko Budisa vom Max-Planck-Institut - den „Shootingstar“ der Synthetischen Biologie in Deutschland - an die HLFS Ursprung ein. Keine kritische Frage sollte unbeantwortet bleiben. Dr. Markus Schmidt leitet das EU-Projekt „Synbiosafe“, welches eine Diskussion um Ethik und Sicherheit der Synthetischen Biologie europaweit anstoßen soll. Er war von unserem Schulprojekt so begeistert, dass er uns seine Hilfe anbot beim Vorhaben, auf die nicht ausreichend gefestigte Gesetzeslage und die Schwierigkeiten der Risikoabschätzung im Zusammenhang mit der Synthetischen Biologie aufmerksam zu machen. Alle aeroben Lebewesen brauchen Katalasen, um schädliches Wasserstoffperoxid abzubauen. Dieses entsteht beispielsweise als unerwünschtes Nebenprodukt bei der Oxidation von Fettsäuren und zerfällt in freie Radikale, die das Genom und Zellproteine schädigen. Außerdem trägt Wasserstoffperoxid zur Alterung der Zellen bei. Katalasen spalten Wasserstoffperoxid in harmlosen Luftsauerstoff (O2 ) und Wasser und machen es dadurch ungefährlich. Somit ist die Katalase gewissermaßen ein AntiAging-Enzym. in Proteinen zu verändern oder sie komplett neu aufzubauen. Die Eigenschaften des daraus entstehenden Proteins sind nicht unbedingt vorauszusehen. Von sehr nützlichen bis hin zu sehr gefährlichen Produkten ist alles möglich. Aufgrund dieser Tatsache kamen wir auf die Idee, auf fehlende Richtlinien für die Risikoabschätzung und daraus folgende mögliche Gefahren hinzuweisen. Sollte sich das vermutete Potential bestätigen und der damit verbundene prognostizierte Boom der Synthetischen Biologie eintreten, wäre bald jeder/jede KonsumentIn mit diesem Thema konfrontiert. Spätestens dann wäre es unumgänglich, sich mit der Problematik der SynBio lich auch, Anreize zu einer dringend notwendigen, fundierten Sicherheits- und Ethikdiskussion zu schaffen. auseinanderzusetzen. Wir aber denken: je früher man sich darüber Gedanken macht, desto besser! Während andere Nationen schon eifrig an dieser Technik forschen - die USA geben das Tempo vor - sieht Österreich nur zu. Grund dafür dürfte nicht zuletzt die negative Grundstimmung zur Gentechnik im Lande sein, die auch auf die Synthetische Biologie ‚übertragen‘ wird. Ziel unseres Projektes ist es also schließ- nicht natürlichen Aminosäuren ein golden leuchtendes, synthetisches Protein geschaffen hat.) Und was wäre ein Projekt, wenn man nicht selbst im Labor die diskutierten Techniken erforschen und hautnah ausprobieren könnte! Bei unseren Recherchen stolperten wir immer wieder über das Max-Planck-Institut (MPI) für Biochemie in Martinsried, das sich bereits intensiv mit SynBio befasst hat. Mit der Idee, mit Forschern vom MPI gemeinsam Versuche im Schullabor durchzuführen, wendeten wir uns an Dr. Nediljko Budisa. (Er ist es, der aus dem Green-Fluorescent-Protein durch den Austausch von Als wir beim österreichischen Gesundheitsministerium um Bewilligung für unser Projekt ansuchten, wurde uns mitgeteilt, dass es keinerlei Gesetze in Bezug auf die Verwendung von synthetischen Aminosäuren für neue Proteine gibt und dass (wie in unserem Fall bei der Herstellung synthetischer Enzyme) jeder mehr oder weniger nach Gutdünken handeln kann. Bisher ist die Risikoabschätzung bei gentechnisch veränderten Organismen ungefähr so definiert: Man nimmt die ungefährliche Qualle Aequorea victoria und gewinnt aus dieser das bekannte GreenFluorescent-Protein. Die zugehörige Erbanlage für dieses Protein schleust man in ein ebenso ungefährliches Bakterium ein. Das Ergebnis ist ein völlig harmloser und grün leuchtender Organismus, der nun keinen größeren Überlebensvorteil gegenüber vorher hat. Eine solche genetische Veränderung gilt als unbedenklich. Mit Hilfe der Synthetischen Biologie wird es nun möglich, die Aminosäurenabfolge Er war von unserem Tatendrang und unserem Interesse begeistert und willigte sofort ein, mit uns synthetische Enzyme zu kreieren. Umso motivierter stürzten wir uns in die Arbeit - wir würden mit Profis arbeiten und von ihnen viel Neues über diesen Forschungszweig erfahren. 5 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 WAS IST SYNTHETISCHE BIOLOGIE? Die Synthetische Biologie stellt einen sehr jungen Forschungszweig dar, dessen Möglichkeiten sehr vielfältig sind. Eine mögliche Definition für den Aufgabenbereich der Synthetischen Biologie lautet: Die Entwicklung biologischer Systeme und Organismen mit Hilfe standardisierter Bausteine und ingenieurswissenschaftlicher Prinzipien. Obwohl sich die Synthetische Biologie grundsätzlich mit ähnlichen Materialien und Methoden wie die Gentechnik befasst, bestehen doch große Unterschiede zwischen diesen Gebieten. Die konventionelle Gentechnik orientiert sich an bereits bestehende Formen des Lebens - sie tauscht beispielsweise einzelne Gensequenzen zwischen unterschiedlichen Organismen aus, transferiert sie usw.. Die Synthetische Biologie hingegen nimmt es in Angriff, Lebensformen, biologische Baustoffe, Biomoleküle, Enzyme (Katalysatoren) zu entwerfen und herzustellen, wie sie in der Natur nicht vorkommen. SynBio kann ein hilfreiches Werkzeug sein bei der Diagnose oder Behandlung von schwerwiegenden Erkrankungen, wie zum Beispiel Krebs. der Herstellung von neuen, maßgeschneiderten Biomaterialien mit Eigenschaften, die anders nicht zu erreichen wären. der Erzeugung von neuen Enzymen, die z.B. sogar in der Lage sein könnten, aus Stroh oder anderen agrarischen Abfallstoffen wertvollen Treibstoff zu erzeugen. der kostengünstigen Produktion von Medikamenten, wie sie vor allem für Entwicklungsländer von Bedeutung ist - ein synthetisch erzeugtes Medikament gegen Malaria kommt demnächst auf den Markt! der Herstellung von Medikamenten, die auf den individuellen Patienten zugeschnitten sind. 6 Ziele der Synthetischen Biologie Herstellung von „minimalen Organismen“: Noch tummeln sich keine von Grund auf künstlich geschaffenen Organismen in den Petrischalen. Ein Weg dahin ist aber das sogenannte „Minimal Genom“. Dieses stellt man aus Bakterien her, die bereits ein sehr kleines Genom aufweisen. Im Zuge langer Versuchsreihen schaltet man diesen Lebewesen nun nach und nach „unnötige“ (d.h. nicht lebensnotwendige) Gene ab. Außerdem wird DNA entfernt, die keine für das Leben notwendige Information enthält. Das Endergebnis ist ein Organismus, der nur mehr jene Erbinformation enthält, die ihm ein Überleben in einer definierten Nährlösung unter Laborbedingungen ermöglicht. Was also übrig bleibt, kann als Grundgerüst oder Chassis wie bei einem Auto für beliebige Aufbauten dienen, so der Grundgedanke. Diese Methode nennt sich „Top-Down“-Ansatz. Damit kommt man dem Verständnis dessen, was Leben eigentlich ausmacht, näher. Andererseits gewinnt man eine Plattform, in die man biologische Schaltkreise einsetzen kann. Fest steht, dass früher oder später die Synthetische Biologie die klassische Gentechnik wenn nicht ablösen, so doch zumindest ergänzen wird. Und man erhofft sich viel von der SynBio, auch wenn noch ungewiss ist, welche der in sie gesetzten Erwartungen sie tatsächlich erfüllen können wird. Auf jeden Fall ist es unseres Erachtens höchste Zeit, dass sich auch Politik und Gesellschaft mit jenen Fragen befassen, die die Synthetische Biologie aufwirft. Z.B.: Dürfen wir mit ingenieurwissenschaftlichen Methoden in das Leben eingreifen? Wie weit betrachten wir artifizielle (künstliche) biologische Systeme noch als‚ lebende Materie‘? Darf man Leben ‚synthetisieren‘? Wie kann man die Vermeidung von Unfällen, z.B. das versehentliche Freisetzen der SynBio-Organismen garantieren? Wie kann man einem Bioterrorismus entgegenwirken? Wie kann man das Risiko einschätzen? Wer soll das Risiko einschätzen? die Kontrollelemente zu charakterisieren, die für das Engineering von Mikroben entscheidend sind, damit schließlich Forscher diese DNA-Teile in synthetischen Organismen mischen und abgleichen können, um neue Wirkstoffe, Treibstoffe oder Chemikalien zu produzieren. Weltweit gibt es bereits mehrere Unternehmen, die DNA in größerem Maßstab vollautomatisch synthetisieren und Forschern zur Verfügung stellen - beispielsweise die in Regensburg (Deutschland) beheimatete Geneart AG. Herstellung neuer Eiweiße: Ein anderer Zweig der SynBio verfolgt die Herstellung noch nie dagewesener Eiweiße als Rohstoff und Bio-Katalysatoren (Enzyme) durch am Computer erstellte Proteinmodelle oder den Einbau von nicht-proteinogenen, also vom Leben nicht verwendeten Aminosäuren (Das war unser Ansatz bei der Arbeit im Labor). Neue Speichermoleküle Schließlich gibt es auch den Ansatz, eine künstliche DNA herzustellen, die anstatt der Desoxyribose ein anderes Zuckermolekül verwendet, z.B. eine Hexose. Das Ergebnis wäre eine HNA. Auf diese Weise könnte man Lebewesen erschaffen, die nur im Labor - gewissermaßen einer Parallelwelt - existieren können. Da keine DNA mehr als Erbgutspeicher verwendet würde, könnten sich diese Organismen nicht mehr in die freie Natur ‚einmischen‘. So könnte man wahrscheinlich die Gefahr der versehentlichen Freisetzung von gefährlichen Laborbakterien in den Griff bekommen. Vor allem dieser Aspekt zeigt, wie groß das Möglichkeitsspektrum der Synthetischen Biologie ist. Kreation künstlicher Zellen Im Unterschied zur schrittweisen Reduktion von Genen wird beim „BottomUp“-Ansatz versucht, eine von Grund auf künstliche Zelle („Protozelle“) mit den wichtigsten Merkmalen des Lebens wie der Interaktion mit der Umwelt und einem funktionierenden Stoffwechsel zu schaffen. Eine Zellmembran ist relativ leicht mit Fettbläschen nachzubauen. Es ist bereits gelungen, in solche zellartigen Bläschen Biomoleküle einzuschleusen, um eine Interaktion zu erwirken. In einem anderen Fall konnte man einen Stoffwechsel in Gang bringen. Design standardisierter biologischer Bausteine: Ein weiterer wichtiger Ansatzpunkt der Synthetischen Biologie besteht darin, Datenbanken aufzubauen, in der sämtliche Gen-Bausteine exakt beschrieben werden, um sie irgendwann einfacher und schneller miteinander kombinieren zu können. Es wird hier der Versuch unternommen, Quellen: „Leben 2.0, Biologie aus dem Baukasten“, www.zukunftswissen.apa.at, Mario Wasserfaller, 12.02.2010 „Schöpfung im Labor“, Der Spiegel 4.1.2010 „Leben vom Reißbrett“, Spektrum der Wissenschaft, Nov.2008 „Den Kode des Lebens erweitern“, Spektrum der Wissenschaft, Jän.2009 7 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 ENZYME Enzyme sind eine Untergruppe der Proteine (= Eiweiße). Als biologische Katalysatoren (d.h. als Stoffe, die eine Reaktion auslösen oder beschleunigen ohne selbst dabei verbraucht zu werden) sind sie bei nahezu jedem Stoffwechselprozess in einer lebenden Zelle beteiligt. So sind sie zum Beispiel verantwortlich für die Replikation von DNA. In Bakterien, Pflanzen, Tieren oder beim Menschen verdauen Enzyme beispielsweise Zucker, Fette und Eiweiße. Sie helfen uns auch dabei, Giftstoffen und Krankheiten zu trotzen oder unsere Zellen mit Sauerstoff zu versorgen. Kurzum sind Enzyme essenzielle Bestandteile allen Lebens. Doch auch in der Industrie finden Enzyme längst vielfältig Verwendung. Erzeugt von Bakterien spalten sie bspw. in der Bioethanolerzeugung Maisstärke zu Glukose, machen Obst für die Saftgewinnung weicher oder verringern die Temperaturentwicklung chemischer Reaktionen, wodurch unglaubliche Mengen an Energie eingespart werden können. Doch was sind Enzyme gentechnisch betrachtet? Ein Enzym besteht aus Aminosäuren, die in langen Ketten angeordnet sind. Es gibt 20 kanonische Aminosäuren. Die Größe von Enzymen variiert zwischen 100 und 30.000 Aminosäuren. Welche Funktion ein Enzym hat, ist von der Anordung der Aminosäuren abhängig. Diese Information über ein Eiweiß ist auf der DNA in Form eines Gens gespeichert und wird bei Bedarf auf ein mRNA-Molekül (d.i. eine transportierbare Kopie der DNA) umgeschrieben und als Matrix für die Synthese neuer Enzyme verwendet. Diese Synthese vollzieht sich in den Ribosomen, welche als Eiweißfabriken der Zelle anzusehen sind. Durch unterschiedliche Triplets (d.s. die 20 kanonischen Aminosäuren 8 drei aufeinander folgende Basen = Codon) auf dem mRNA-Strang ‚weiß‘ das Ribosom, welche Aminosäuren verknüpft werden müssen. Das mRNA-Molekül wird zwischen den zwei Untereinheiten des Ribosoms eingeklemmt, ein Triplet nach dem anderen abgelesen und die Aminosäurenkette Stück für Stück verlängert, bis das Protein fertig strukturiert und gefaltet seine Aufgabe aufnehmen kann. Ein Beispiel Wie bereits erwähnt schützen uns Enzyme auch vor dem Einfluss von Giftstoffen. Einer dieser Giftstoffe, der eine sehr große Wirkung auf den Menschen hat und trotzdem immer wieder in den Körper gelangt ist Ethanol - besser bekannt als Alkohol. Ist der Alkohol ins Blut und in die Leber übergegangen, reagiert der Körper sofort mit der Produktion eines Enzyms (Alkoholdehydrogenase), das Ethanol zu ungefährlichen Stoffen abbaut. Der genaue Ablauf der Umwandlung des Ethanols sieht folgendermaßen aus: Es werden zwei Wasserstoffatome vom Ethanolmolekül abgetrennt und daraus ein weit weniger toxisches Acetaldehyd hergestellt. Der Körper kann dieses nun weiter verstoffwechseln bzw. ausscheiden. Jedes Enyzm hat dafür ein sogenanntes ‚aktives Zentrum‘, eine kleine Einbuchtung an der Proteinoberfläche, wo die katalytische Reaktion durch die Herabsetzung der Aktivierungsenergie abläuft. 20 Aminosäuren BasenTriplett /Codon auf mRNA Alanin Ala GCU GCC GCA GCG Arginin Arg CGU CGC CGA CGG AGA AGG Asparagin Asn AAU AAC Asparaginsäure Asp GAU GAC Cystein Cys UGU UGC Glutamin Gln CAA CAG Glutaminsäure Glu GAA GAG Glycin Gly GGU GGC GGA GGG Histidin His CAU CAC Isoleucin Ile AUU AUC AUA Leucin Leu CUU CUC CUA CUG UUA UUG Lysin Lys AAA AAG Methionin Met AUG Phenylalanin Phe UUU UUC Prolin Pro CCU CCC CCA CCG Serin Ser UCU UCC UCA UCG AGU AGC Threonin Thr ACU ACC ACA ACG Tryptophan Trp UGG Tyrosin Tyr UAU UAC Valin Val GUU GUC GUA GUG Stop UAA UAG UGA Tabelle: Aminosäuren mit Abkürzung und dazugehörigen Codons 9 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 Die Katalase Synthetische Enzyme Ein Teilgebiet der Synthetischen Biologie befasst sich mit der Herstellung von Enzymen, die die Evolution als solche nie hervorgebracht hätte. Dies ist ‚notwendig‘, weil das Leben zwar perfekt an die Bedingungen in der Natur angepasst ist, nicht jedoch an industrielle Verfahrenstechniken. Man bedient sich dabei der Technik des Enzym-Engineerings, bei dem ein Enzym am Computer als Modell erstellt und theoretisch verbessert wird. Anschließend verändert man das Erbgut eines Bakteriums nach der Aminosäurenabfolge des Computermodells. Dieses Bakterium erzeugt nun das Enzym, das die Ingenieure/ IngenieurInnen zu ihrem Nutzen ‚kreiert‘ haben. Weil uns diese moderne Technologie nicht zur Verfügung stand, griffen wir für die Herstellung ‚unseres‘ Enzyms auf eine andere Methode zurück. Grundlage dieser Methode ist, dass es möglich ist, gewissermaßen das Repertoire der kanonischen Aminosäuren zu erweitern: Entzieht man dem Nährmedium spezieller, eigens ausgesuchter Bakterien eine Standardaminosäure und fügt anstelle dessen eine nicht-kanonische, jedoch strukturell sehr ähnliche Aminosäure hinzu, so besteht eine gewisse Wahrscheinlichkeit, dass die ‚eingeschmuggelte‘ Aminosäure in die Proteinsynthese einbezogen wird. Das Ergebnis kann in diesem Fall ein Enzym sein, das eine abgewandelte Struktur besitzt und deshalb neue Eigenschaften hat. Man muss erwähnen, dass diese Methode nur mit sehr wenigen Bakterienstämmen funktioniert. Ein Beispiel dafür ist das GFP, das grün fluoreszierende Protein, das erstmals 1961 von dem Japaner Osamu Shimomura aus der Qualle Aequorea victoria isoliert wurde. Dieses Protein lässt den Organismus, der es in sich trägt, unter UV-Licht-Einfluss grün leuchten. Wenn dieses Eiweiß an andere Enzyme geheftet wird kann man intrazelluläre Vorgänge - wie zum Beispiel die Teilung des Zellkernes und die damit verbundene Verdopplung des Chromosomensatzes - unter dem Mikroskop betrachten. Das GFP kann durch Umprogrammierung auch gelb oder cyan leuchten. Durch den Einbau von nichtkanonischen Aminosäuren können auch noch andere Farben (wie z.B. Gold) erreicht werden. Dies gelang erstmals dem berühmten Forscher Dr. Nedilko Budisa vom MPI, unserem Projektpartner und Mentor. Ein weiteres Beispiel aus der Arbeitsgruppe von Dr. Budisa stellt die synthetische Lipase dar. Stück für Stück wurde eine Amino- Ethionin säure nach der anderen ausgetauscht bis 14 nicht-natürliche Bausteine in das Enzym eingeflochten waren, wodurch seine Aktivität selbst bei niedrigeren Temperaturen unglaublich gesteigert werden konnte. Wenn man bedenkt, welche Vielfalt die Natur mit den 20 ihr zur Verfügung stehenden Bausteinen hervorgebracht hat, und welche Möglichkeiten in den ca. 680 nicht verwendeten Aminosäuren stecken, so kann man sich leicht vorstellen, welche Rolle Protein-Engineering bzw. die Synthetische Biologie in der Zukunft spielen könnten. Die nicht-kanonische Aminosäuren Norleucin und Ethionin werden zur experimentellen Untersuchung von Proteinstrukturen und -funktionen verwendet. Aminoacyl-tRNA-Synthetasen, wichtige Enzyme in der Proteinbiosynthese, können getäuscht werden, indem man ihnen anstelle ihrer normalen Substrate bestimmte unbiologische Aminosäuren anbietet. So werden Ethionin und Norleucin an solchen Positionen in Proteine eingebaut, die normalerweise Methionin einnehmen würde. Wir versuchten mit diesen beiden Aminosäuren unsere Katalase zu verändern. Norleucin Wasserstoffperoxid ist ein sehr starkes Oxidationsmittel, das als Desinfektionsund Bleichmittel verwendet wird. Es neigt dazu, in Wasser und Sauerstoff zu zerfallen. Wenn das geschieht, wird ein einzelnes Sauerstoffatom (O) abgespalten, das damit zum freien Radikal wird. Ein freies Radikal ist hochreaktiv und damit zytotoxisch gegenüber vielen Zellen. Wasserstoffperoxid kann deshalb zur Abtötung von Bakterien und anderen Mikroorganismen verwendet werden. Auch im menschlichen Körper kommt Wasserstoffperoxid in geringen Mengen vor, z.B. in Peroxisomen (= Zellorganellen, die als Entgiftungsapparat von in sauerstoffhaltiger Umgebung lebenden Organismen dienen - sie sind in der Leber, den Nieren und den roten Blutkörperchen vorhanden). Weiters entsteht Wasserstoffperoxid beim Abbau von Purinen (Bestandteile des Erbgutes, der DNA) sowie bei der Oxidation von Fettsäuren und Kohlenhydraten. Im zweiten Schritt werden sowohl Wasserstoffperoxid als auch das Enzym reduziert, Sauerstoff wird oxidiert und damit als Produkt neben einem weiteren Wassermolekül freigesetzt. Bei einer zu hohen Konzentration von Wasserstoffperoxid kommt es zur - meist unerwünschten - Graufärbung von Haaren. Dies aufgrund eines Effekts, den man sich gleichzeitig bei der Blondierung bzw. beim Färben der Haare zu Nutze macht. Eine zu hohe Wasserstoffperoxid-Konzentration könnte aber auch für neurologische Erkrankungen verantwortlich und manchmal sogar die Ursache für Diabetes sein. Allgemein gesagt führt Wasserstoffperoxid bei höheren Lebewesen zu Zell-Alterungsprozessen, bei Einzellern zum Zelltod. Die meisten Organismen schützen sich gegen diesen Stoff durch Enzyme, die Wasserstoffperoxid in die ungefährlichen Stoffe Luftsauerstoff (O2) und Wasser (H2O) spalten. Katalase ist eines dieser Enzyme, mit dem viele Lebewesen Wasserstoffperoxid abbauen können. Die Katalase schützt vor den oben genannten Erkrankungen und ist somit das Anti-Aging-Enzym schlechthin. So zeigten WissenschaftlerInnen, dass Mäuseherzzellen um bis zu 20% länger leben, wenn sie eine erhöhte Katalase-Produktion aufweisen. Katalase wird - in Kombination mit dem Enzym Glucoseoxidase - sehr häufig in der Lebensmittelindustrie als konservierendes ‚System‘ eingesetzt, zum Beispiel zur Konservierung von Backwaren, Getränken, Mayonnaise, Eiklar und Milchprodukten. In der Kosmetikindustrie wird Katalase als Anti-Aging-Enzym verwendet. Das Enzym wandelt in der Haut mehrere Peroxide und freie Radikale in Sauerstoff und Wasser um. Durch die Reduzierung der Anzahl freier Radikale verlangsamt sich der Alterungsprozess der Hautzellen, wodurch die Haut länger glatt und jünger aussieht. Die Reaktion, in der Wasserstoffperoxid zu Sauerstoff und Wasser umgewandelt wird, erfolgt in zwei Schritten. Im ersten Schritt wird H2O2 reduziert und das Enzym oxidiert, Wasser entsteht. H2O2 + Katalase (ox.,+O) = H2O + O2 + Katalase (red.) Die Summengleichung lautet: 2H2O2 = 2H2O + O2 Katalase wird durch Fermentation mit Hilfe von verschiedenen Pilz- und Bakterienarten gewonnen. Mindestens zwei Katalase-Präparate werden in Europa mit gentechnisch veränderten Pilzkulturen (Aspergillen) hergestellt, von denen eines auch in der Lebensmittelindustrie verwendet wird. frei nach: http://www.transgen.de/datenbank/enzyme/ 89.katalase.html http://www.agera-freising.de/pceg.htm http://de.wikipedia.org/wiki/Katalase H2O2 + Katalase (red.) = H2O + Katalase (ox.,+O) frei nach http://de.wikipedia.org/wiki/Norleucin 10 11 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 BACILLUS SUBTILIS Allgemeines Das Bacillus subtilis, auch Heubazillus genannt, ist ein aerobes Bodenbakterium. Insofern sein Vorkommen ubiquitär ist kann es aber durchaus auch aus Wasser und Luft isoliert werden. Besonders wichtig für uns ist, dass es nicht pathogen ist und somit keine Gefährdung für Menschen und andere Lebewesen darstellt. Das Bacillus subtilis ist stäbchenförmig und begeißelt. Durch die Begeißelung ist eine schnellere Fortbewegung gewährleistet, die besonders bei der Flucht vor gefährlichen Umweltbedingungen, sogenannten „Stresssituationen“ (pH-Wert Änderungen, Nährstoffmangel, Hitze, ...), von großer Bedeutung ist. Zum Überleben des Bacillus subtilis tragen aber auch Sporen im Zellinneren bei, sogenannte Endosporen. Sie sind ein besonderes Merkmal und dienen nicht der Fortpflanzung, sondern alleine dem Überdauern. Mit Hilfe der Endosporen kann das Bacillus subtilis Extremsituationen wie z.B. Hitze oder Trockenheit standhalten. Wie auch alle anderen Bakterien ist das Heubazillus ein ‚Weltmeister der Vermehrung‘. Bei optimalen Bedingungen kann es eine Verdopplungszeit der Zellen (= Generationszeit) von nur 26 Minuten erreichen, wobei 45 Minuten als normal zu betrachten sind. Das bedeutet, nach 10 Stunden hätten sich aus einer einzelnen Bakterie nzelle bei einer Generationszeit von 26 Minuten bereits 8 Millionen entwickelt, bei einer Generationszeit von 45 Minuten 8000. Anwendung von Bacillus subtilis In der Landwirtschaft wird das Bacillus subtilis als Fungizid (FZB24®) für Samen von Baumwolle, Gemüse, Erdnüssen und Sojabohnen eingesetzt. Es besiedelt die Rhizosphäre der Pflanzen und schützt diese damit vor Verpilzung - es konkurriert mit dem Pilz. Weiters produziert es flüchtige organische Verbindungen, die dem Pilzbefall entgegenwirken. Auch in der Medizin ist das Heubazillus nicht ohne Bedeutung. Es wird in Medikamenten gegen chronische Hautkrankheiten sowie gegen Verdauungsstörungen wie Durchfall und Darmentzündungen eingesetzt (z.B. in Utilin®, Utilin N® und Bactisubtil®). schen aus. Bacillus subtilis wird der 1. Risikogruppe zugeordnet. Außerdem hat Bacillus subtilis den von der Food and Drug Administration (FDA) vergebenen GRAS-Status (= Generally Recognized As Safe). Das bedeutet, dass das Bakterium gesundheitlich unbedenklich, hygienisch einwandfrei und technologisch wirksam ist. ~ 87% der Nukleotide haben eine codierende Funktion. Die Datenbank zeigt das gesamte Genom von Bacillus subtilis. Man kann vom Beginn bis zum Ende, gereiht nach Basenpaarnummern, durchblättern. Das Genom von Bacillus subtilis besteht aus 4214810 Basenpaaren. Ein Gen ist durchschnittlich 893 Nukleotide lang. Das längste Gen hat 14793 Nukleotide, das kürzeste hingegen nur 63. View chromosomal locations Ebenfalls eine Rolle spielt Bacillus subtilis in der Herstellung von WaschmittelEnzymen und der Synthese des Vitamins Riboflavin (Vitamin B2) sowie des Antibiotikums Bacitractin. Das Heubazillus produziert von Natur aus Riboflavin. Durch gentechnische Veränderungen wurde hier die Produktion auf die 300.000fache Menge gesteigert. Neben Escherichia coli wird Bacillus subtilis auch zur Produktion von Amylase eingesetzt, die zum Stärkeabbau dient. Hinsichtlich ihrer Sicherheit werden Mikroorganismen in insgesamt vier Gruppen eingeteilt. Während Gruppe 1 kein Risiko für den Menschen darstellt, geht von der Gruppe 4 ein hohes Risiko für den Men- Quellenangabe: http://de.wikipedia.org/wiki/Bacillus_subtilis http://microbio1.biologie.uni-greifswald.de:8080/institute/85 http://mikrobiologie.uni-graz.at/lehre_server/MOL102/Lab_Praxis_02_handout.pdf http://www.transgen.de/lebensmittel/mikroorganismen/595.doku.html Fotocredits: http://de.academic.ru/pictures/dewiki/98/bacillus_subtilis.jpg http://de.wikipedia.org/w/index.php?title=Datei:Bacillus_subtilis_Gram.jpg&filetimestamp=20050127095612 12 13 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 ARBEIT IM LABOR KATALASE AUS BACILLUS SUBTILIS Das Ziel unserer Laborarbeit war die Expression von Katalase in Gegenwart der nicht-natürlichen Aminosäuren Ethion und Norleucin. Das verwendete Laborbakterium (Bacillus subtilis Stamm 1A55, bezogen vom Bacillus Genetic Stock Center, http://www.bgsc.org/) sollte die nichtkanonischen Aminosäuren in seinen Stoffwechsel miteinbeziehen und schließ- lich ein synthetisches Enzym ins Nährmedium abgeben. Der von uns verwendete Bacillus subtilis-Stamm war Methionin-, Tryptophan-, Tyrosin- und Phenylalaninauxotroph und konnte somit außerhalb des künstlich hergestellten Nährmediums nicht überleben. Die Methionauxotrophie des ausgewählten Bakterienstammes wollten wir nutzen. Wir mischten sechs verschiedene Nährmedien. Drei auf Basis des BakteriumStandardmedium, genannt LB, und drei auf ein anderes Standardmedium, genannt M9. Bei LBM mischten wir Methion als natürliche Aminosäure bei, bei LBN ersetzen wir Methionin mit Norleucin, bei LBE mit Ethionin. Nach dem gleichem Schema stellten wir M9M, M9N, M9E her.Jeweils LBM und M9M mit dem der natürlichen Aminosäurenzusammensetzung sollten unsere Referenzproben sein, ob unsere Bacillusstamm überhaupt Amylasen erzeugt, also die sogenannte Nullprobe. Die Mischrezepte finden Sie in Tabelle 1. Obwohl unser Bacillus Stamm sicher war, wurden alle Maßnahmen zur Vermeidung einer Kontamination der Außenwelt sehr gewissenhaft und genau ausgeführt: Alle bei den Arbeiten verwendeten Einmalgerätschaften wurden im Autoklaven oder Certoklaven inaktiviert und anschließend weggeworfen. Lösungen mit speziellen Färbemittel wurden einer Sondermüllentsorgung zugeführt. Ebenfalls wurden alle Organismen durch Hitzesterilisation inaktiviert. Die Laborgeräte, Arbeitsbereiche, Labortische etc. wurden mit Desinfektionslösung gereinigt bzw. die Reinraumbank zusätzlich nach der Reinigung und Desinfektion mit UV-Licht behandelt. Anlegen der Vorkultur 20 μl des auf Trockeneis gelagerten Glycerin-Stocks des Bacillus subtilisStammes 1A55 werden zum Anlegen einer Vorkultur verwendet. Dazu werden 10ml des LB Mediums (fertig gemischt kommerziell erhältlich) unter der Reinraumbank in ein steriles 50ml Falconröhrchen gefüllt. Danach wird das Medium mit dem Glycerin-Stock mittels einer Pipette beimpft. 14 Es ist auf absolute Sterilität zu achten. Jedes verwendete Gerät ist entweder abzuflammen, mit UV-Licht zu behandeln oder mit 70%-Ethanol abzuwischen. Auch sind Handschuhe zu tragen, die ebenfalls desinfiziert werden sollten. Die beimpften Medien werden über Nacht bei 37°C in den Schüttelinkubator gestellt. Die Röhrchen sind leicht schräg zu platzieren, damit eine gute Durchmischung mit Luft gewährleistet ist. Der Deckel wird nur soweit festgeschraubt, dass er gerade noch sicher hält. Auf diese Weise kann die Luft außerhalb und innerhalb es Röhrchens zirkulieren. Bestimmen der optischen Dichte Um die optische Dichte der Probe - und damit die Menge der darin enthaltenen Keime - feststellen zu können, werden unter sterilen Bedingungen 0,9ml keimfreien Nährmediums in eine Küvette überführt. Dazu kommen 0,1ml des beimpften und bebrüteten Nährmediums, das zuvor gut gevortext/durchmischt werden muss. Mit Hilfe eines Pho- tometers wird bei 550nm Absorption die optische Dichte bestimmt. Als Blindprobe dient 1ml LB-Medium. Auch hier sollte auf steriles Arbeiten geachtet werden, damit keine Fremdkeime in die Vorkultur gelangen. Bei der Küvette muss man nicht mehr auf eine sterile Arbeitsweise achten - sie wird ohnehin entsorgt. Ansetzen der Hauptkultur Zunächst muss das Volumen für das Inokulum für die Hauptkultur berechnet werden. Dabei sollte die optische Dichte der Hauptkultur 0,1 betragen. VInokulum = (VHK*Start OD550) / OD550VK Unter sterilen Bedingungen wird das Nährmedium (100ml LB, 200ml M9) in die autoklavierten Schikanenkolben gefüllt. Danach wird das berechnete Inokolum aus der Vorkultur in den Schikanenkolben pipettiert. Dabei ist wieder auf absolut sauberes Arbeiten zu achten und der Schikanenkolben außerhalb der Reinraumbank immer mit Alufolie abzudecken. Die Ränder und Deckel sämtlicher Gefäße müssen nach jedem Öffnen und vor jedem Schließen abgeflammt werden. Der Schikanenkolben mit dem nun beimpften Nährmedium wird bei 37°C in den Schüttelinkubator gestellt. Dabei ist die Drehzahl so zu wählen, dass das Medium gut schäumt und somit viel Sauerstoff zu der Kultur gelangt. Nach 1h, 3h, 5h, 7h, 20h, 22h, und 24h wird jeweils 1ml der Zellsuspension unter sterilen Arbeitsbedingungen in eine Küvette überführt und die optische Dichte (und damit das Zellwachstum) bestimmt. Optische Dichte und Zeit sind wichtige Faktoren für die Erstellung einer Wachstumskurve. Die Proben werden anschließend in der Epifuge bei maximaler Geschwindigkeit 2 Minuten abzentrifugiert und die Zellüberstände mit einer Pipette in ein Eppendorfergefäß überführt, worin sie gekühlt gelagert werden. 15 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 Zellernte und Gewinnung der Katalase Sobald die optische Dichte abzufallen beginnt hat das Sterben der Zellen begonnen. Es ist rasch zu handeln, weil nun Proteasen (= Enzyme, die fähig sind, Enzyme abzubauen) in das Medium gelangen, die das gesuchte Enzym Katalase zerstören können. Die Zellsuspension wird, nun nicht mehr steril, in 50ml Falconröhrchen aufgeteilt und in der Zentrifuge bei 3500rpm 10 Minuten abzentrifugiert. Der Zellüberstand wird nun - ohne das Zellpellet aufzuwirbeln - in einen Faltenfilter abdekandiert. Der auf diese Weise gewonnene Zellüberstand enthält sämtliche in den Bakterien expressionierten Proteine. Das Filtrat versetzten wir mit Ammoniumsulfat, um das Ausfallen der Katalase herbeizuführen. Diese Flüssigkeit wurde noch insgesamt zweimal gefiltert. Mithilfe eines Dialyseschlauches trennten wir das zuvor zugegebene Ammoniumsulfat von der Katalase. Mit einem SDSGel trennten wir alle Proteine in unserem Substrat auf und kühlten es für die spätere Analyse in der Massenspektrometrie am Max-Planck-Institut ein. Tabelle 1: Medien LB Medium 0.5% (w/v) yeast extract 1% (w/v) Bacto tryptone 1% (w/v) NaCl 5 g/l 10 g/l 10 g/l LB+ Medium 0.1% (w/v) glucose L-Phenylalanin L-Tyrosin L-Tryptophan 0.5% (w/v) yeast extract 1% (w/v) Bacto tryptone 1% (w/v) NaCl 1 g/l 50 mg/l 50 mg/l 50 mg/l 5 g/l 10 g/l 10 g/l LBM Medium LB+ Medium 100 ml L-Methionin 50 mg/l Methionin in LB+ Medium auflösen und sterilfiltrieren; 2 ml in ein steriles 2 ml Eppi aliquotieren („Blank“) & bei 4 °C lagern; bald verbrauchen; kann über Nacht bei 4 °C gelagert werden. LBE Medium LB+ Medium 100 ml L-Ethionin 10 g/l Ethionin in LB+ Medium auflösen und sterilfiltrieren; 2 ml in ein steriles 2 ml Eppi aliquotieren („Blank“) & bei 4 °C lagern; bald verbrauchen;kann über Nacht bei 4 °C gelagert werden. LBN Medium LB+ Medium 100 ml L-Norleucin 10 g/l Norleucin in LB+ Medium auflösen und sterilfiltrieren; 2 ml in ein steriles 2 ml Eppi aliquotieren („Blank“) & bei 4 °C lagern; bald verbrauchen; kann über Nacht bei 4 °C gelagert werden. 16 5x M9 Konzentrat Na2HPO4·2 H2O 42.5 g/l KH2PO4 15 g/l 5 g/l NH4Cl NaCl 2.5 g/l pH Wert mit NaOH auf 7.0 einstellen; autoklavieren und bei Raumtemperatur lagern. 100x Spurenelementelösung MnCl2·4 H2O 100 mg/l ZnCl2 170 mg/l 43 mg/l CuCl2·2 H2O CoCl2·6 H2O 60 mg/l Na2MoO4·2 H2O 60 mg/l Sterilfiltrieren und lichtgeschützt bei Raumtemperatur lagern. 100 mM CaCl2 CaCl2·2 H2O 1.47 g / 100 ml Autoklavieren und bei Raumtemperatur lagern. 1M MgSO4 MgSO4·7H2O 24.6 g / 100 ml Autoklavieren und bei Raumtemperatur lagern. 50 mM FeCl3 FeCl3·6H2O 1.35 g / 100 ml Autoklavieren und bei Raumtemperatur lagern. M9 Medium M9M Medium 5x M9 Konzentrat 200 ml/l 100x Spurenelementelösung 10 ml/l 100 mM CaCl2 1 ml/l 1 M MgSO4 1 ml/l 1 ml/l 50 mM FeCl3·6H2O L-Phenylalanin 50 mg/l L-Tyrosin 50 mg/l L-Tryptophan 50 mg/l Glucose 5 g/l Mit destilliertem H2O auf 1 l auffüllen, Glucose und Aminosäuren auflösen, sterilfiltrieren und bald verbrauchen; kann über Nacht bei 4 °C gelagert werden. M9 Medium 200 ml L-Methionin 50 mg/l Methionin in M9 Medium auflösen und sterilfiltrieren; 2 ml in ein steriles 2 ml Eppi aliquotieren („Blank“) & bei 4 °C lagern; bald verbrauchen; kann über Nacht bei 4 °C gelagert werden. M9E Medium M9 Medium 200 ml L-Ethionin 50 mg/l Ethionin in M9 Medium auflösen und sterilfiltrieren; 2 ml in ein steriles 2 ml Eppi aliquotieren („Blank“) & bei 4 °C lagern; bald verbrauchen; kann über Nacht bei 4°C gelagert werden. M9N Medium M9 Medium 200 ml L-Norleucin 50 mg/l Norleucin in M9 Medium auflösen und sterilfiltrieren; 2 ml in ein steriles 2 ml Eppi aliquotieren („Blank“) & bei 4 °C lagern; bald verbrauchen; kann über Nacht bei 4 °C gelagert werden. 17 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 ERGEBNISSE Um zu bestimmen, ob es sich bei dem von unserer Bacillus subtilis-Kultur erzeugten Enzym auch tatsächlich um eine echte Katalase handelte (bzw. ob diese wirklich synthetische Aminosäuren in ihre Struktur integriert hatte) waren weitere Untersuchungsschritte notwendig. Die erste, noch relativ einfache Analyse der Zellüberstände und der darin gelösten Proteine, die das Bakterium während seiner Wachstumszeit erzeugt hatte, wurde mittels eines SDS-Proteingels durchgeführt. Durch den nicht all zu großen Materialaufwand konnten wir diesen Versuch selbst, in unserem Schullabor, durchführen. Die dafür benötigten Acrylamid-Gelplatten stellte uns dennoch das Max-Planck-Institut zur Verfügung, da die Herstellung in einer Schülergruppe aufgrund der stark toxischen Wirkung des Acrylamids nicht ganz ungefährlich gewesen wäre. 20 μl der zu untersuchenden Probe (gesammelte Zellüberstände, Präzipitate und Dialysate) wurden mit 5 μl des SDS-Probenpuffers versetzt, die Flüssigkeit durchmischt und mittels kurzen Abzentrifugierens in der Spitze des Eppendorfergefäßes gesammelt. Anschließend erfolgte eine fünfminütige Inkubation bei 95°C. Dabei lagerte sich der stark lipophile (= fettliebende = wasserhassende) Teil des Sodiumdodezylsulfates (SDS) an die Oberfläche eines jeden Eiweißes an. Da sich in der wässrigen Lösung die Ionenbindung löste erhielt das gesamte Dodezylsulfat eine negative Ladung und damit auch das daran haftende Eiweiß. Die Lösung (mit den nun negativ geladenen Proteinen) wurde nochmals durchmischt, abzentrifugiert und auf das Gel aufgetragen. Jetzt wurde für zwei Stunden eine 130 V Spannung angelegt. Die dabei unten entstehende positive Ladung übte eine Anziehungskraft auf die negativ geladenen Eiweiße aus. Diese wurden nun - abhängig von ihrer Größe - mehr oder weniger stark auf dem Weg durch das Gel gebremst und dementsprechend aufgetrennt. Sobald die Lauffront den unteren Bereich des Gels erreicht hatte wurde die Spannung abgeschaltet, das Gel von den Trägerplatten entfernt, eingefärbt und wieder entfärbt. Auf dem Leuchttisch konnte 18 man nun gut die Banden der nach ihrer Größe aufgespaltenen Proteine sehen. Mit Hilfe des an beiden Seiten aufgetragenen Markers, dessen Banden standardisiert durch das Gel mitwanderten, konnte man die Masse in kDa (Kilodalton) gut abschätzen. Erfreulicherweise stellte sich heraus, dass bei allen untersuchten Proben eine deutliche Bande auf der Höhe der 55 KDa Marke zu erkennen war - was der Größe der von uns gesuchten Katalase entsprach. Jedoch war dieses Ergebnis noch kein Beweis dafür, dass es sich bei dem gefundenen Protein tatsächlich um Katalase handelte und nicht um ein Eiweiß gleicher Größe. Dies war zwar unwahrscheinlich, wäre aber durchaus möglich gewesen. Schon gar nicht hatte das SDS-Gel Aufschluss gegeben, ob in die Katalase die nicht-kanonischen Aminosäuren Ethionin bzw. Norleucin eingebaut worden waren. Um dies herauszufinden, bedurfte es einer massenspektrometrischen Untersuchung. Da die dafür benötigten Gerätschaften jedoch deutlich über unserem Budget lagen (und für die Untersuchung außerdem eine ausgebildete Fachperson unerlässlich war), übernahm das Max-Planck-Institut diese Analyse für uns. Um einen Stoff massenspektrometrisch zu untersuchen, durchläuft seine Probe mehrere komplizierte Schritte: Zunächst Auswertung mittels Proteingelen (SDS-Gele) wird das Analyt durch verschiedenste Methoden ionisiert. Das heißt, es wird teilweise in seine Bestandteile zerlegt, denen Elektronen aus der Hülle entfernt werden, worauf sie eine positive Ladung annehmen. Wie stark diese Ladung ist hängt von mehreren Faktoren ab und dient zur Bestimmung des Stoffes. Anschließend werden die Ionen ähnlich wie beim Verfahren der Chromatographie, jedoch nach ihrem Masse-Ladung-Verhältnis aufgetrennt. Daraufhin erfolgt die Messung der aufgespaltenen Ionenpakete. Diese bewegen sich nacheinander über eine Detektorenplatte, die ihre Ladung feststellt und als sogenannte Peaks festhält. Um das massenspektrographische Ergeb- 1. Auftritt von Katalase im Kulturmedium Abbildung 1. Katalase Expression während des Bacillus subtilis-Wachstums. Es wurden Kulturüberstände von Bacillus subtilis zu unterschiedlichen Zeitpunkten der Wachstumsphase entnommen. Die Nummern der einzelnen Spuren geben den Zeitpunkt der Probennahme (in Stunden) nach der Kulturinokulation an. Beachten Sie das Auftreten der Katalase mit zunehmender Kulturzeit (rot gestrichelter Kasten). S (Standard): Fermentas Prestained protein marker (#Sm0671), Auftragsmenge: 5μl; M9M: Bacillus subtilis Kultur, Medium: Minimalmedium M9, Zugegebene Aminosäure: Methionin; LBM: Bacillus subtilis Kultur, Medium: Vollmedium LB, Zugegebene Aminosäure: Methionin; LBN: Bacillus subtilis Kultur, Medium: Vollmedium LB, Zugegebene Aminosäure: Norleucin. Es wurden jeweils 32 μl Kulturüberstand auf das Gel aufgetragen (+ 8 μl 5 x SDS Auftragspuffer). In den drei beispielhaft angegebenen Kulturen M9 mit Methionin, LB mit Methionin und LB mit Norleucin ist ein Auftreten der Katalase zwischen 7 und 20 Stunden nach der Kulturinokulation deutlich zu beobachten (siehe Abbildung 1, rotgestrichelter Kasten). Abb. 1 Abbildung 2. Katalaseexpression nach 21 Stunden. Es wurden die Bacillus-subtilisKulturüberstände nach Ende der Expressionszeit aufgetragen. S (Standard): Fermentas Prestained protein marker (#Sm0671), Auftragsmenge: 5μl; LB: Vollmedium LB; M9: Minimalmedium M9; M: Methionin; E: Ethionin; N: Norleucin. Es wurden jeweils 20 μl Kulturüberstand auf das Gel aufgetragen (+ 5 μl 5 x SDS Auftragspuffer). Nach Abschluss der Katalaseexpression wurden zum Vergleich alle Kulturüberstände auf ein Gel aufgetragen. Katalase war in allen Proben vorhanden (siehe Abbildung 2, rotgestrichelter Kasten). Vergleicht man den Gesamtproteingehalt der sechs Proben so zeigt sich, dass alle Kulturüberstände in etwa gleich viel Protein enthielten. Daraus kann gefolgert werden, dass die Bandenintensitäten der Katalase in den einzelnen Proben direkt miteinander verglichen werden können. Gleiche Mengen an Katalase sind einerseits in den Überständen LBN, M9M, M9E und M9N und andererseits in den Überständen LBM und LBE enthalten. Vergleicht man die zwei Gruppen an Überständen untereinander, enthalten LBM und LBE ca. dreimal soviel Katalase wie LBN, M9M, M9E und M9N. Zur näheren Untersuchung wurde die Katalase aus den Kulturüberständen weiter aufgereinigt. nis auswerten zu können ist es nötig, die Ladungen der einzelnen Ionen zu kennen. So kann die Anzahl der Ionen einer Größe anhand der gemessenen Ladung ermittelt werden. Das heißt: Wenn Größe und Ladung der von uns gesuchten synthetischen Aminosäure bekannt sind, so kann mithilfe der massenspektrometrischen Analyse bestimmt werden, ob und wie oft die gewünschte Manipulation des Enzyms vorliegt. Man erhält die genaue Aminosäurenkette und sieht, an welcher Stelle ein Austausch mit nicht-kanonischen Aminosäuren geklappt hat. Abb. 2 19 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 Messungen am Massenspektrometer Abb. 3 2. Aufreinigung der Katalase Abbildung 3. Zweistufige Ammoniumsulfatfällung der Katalase. S (Standard): Fermentas Prestained protein marker (#Sm0671), Auftragsmenge: 5μl; M9E: Bacillus subtilis Kultur, Medium: Minimalmedium M9, Zugegebene Aminosäure: Ethionin; LBE: Bacillus subtilis Kultur, Medium: Vollmedium LB, Zugegebene Aminosäure: Ethionin; Ü1: Überstand der Kultur; P1: Präzipitat bei Ammoniumsulfatzugabe zu 50 %; P2: Präzipitat bei Ammoniumsulfatzugabe zu 80 %; D1: Dialysat von Präzipität P1; D2: Dialysat von Präzipität P2. Es wurden jeweils 20 μl Kulturüberstand auf das Gel aufgetragen (+ 5μl 5 x SDS Auftragspuffer). In einer ersten Stufe wurden die Überstände (siehe Abbildung 3, Ü1) zu einer Endkonzentration von 50% mit Ammoniumsulfat versetzt. Das Präzipitat (P1, siehe Abbildung 3) wurde durch Filtration abgetrennt. Zum erhaltenen zwei- 20 ten Überstand wurde weiteres Ammoniumsulfat zu einer Endkonzentration von 80% zugegeben. Das Präzipitat (P2, siehe Abbildung 3) wurde durch Filtration abgetrennt. P1 und P2 wurden anschließend in 10 ml Trispuffer resuspendiert und über Nacht bei 4°C gegen Trispuffer dialysiert (Dialysate D1 und D2, siehe Abbildung 3). Abbildung 3 zeigt die Aufreinigung exemplarisch für die Kulturüberstände M9E und LBE. Durch den ersten Schritt der Ammoni- umsulfatfällung (Präzipitate P1) konnten kontaminierende Proteine aus der Probe entfernt werden. Vor allem das im Überstand (Ü1) am stärksten auftretende Protein bei ca. 37 kDa konnte fast gänzlich entfernt werden (siehe schwarze Pfeile in Abbildung 3). Die Katalase konnte durch die weitere Zugabe von Ammoniumsulfat aus dem Kulturüberstand ausgefällt werden (siehe rote Pfeile in Abbildung 3). Durch die anschließende Dialyse wurde die Katalase wieder in Lösung gebracht (siehe Dialysate D2 in Abbildung 3). Vergleicht man das Verhältnis von Katalase zu Gesamtprotein in Ü1 und D2 lässt sich feststellen, dass die Katalase durch die zweistufige Ammoniumsulfatfällung gegenüber den anderen Proteinen im Kulturüberstand stark aufkonzentriert wurde. Die Dialysate D2 wurden in der Folge für weitere Analysen herangezogen. Aus Zeitgründen konnte bis zur Fertigstellung dieses Berichts nur die massenspektrometrische Analyse der Proben LBE durchgeführt werden. Massenspektrometrische Analyse der Probe LBE. Die Proben wurden mit der sogenannten nano-LC/MS/MS Methode analysiert. Hierbei werden die zu analysierenden Proteinbanden aus dem SDS-PAGE Gel ausgeschnitten (siehe Abbildung 3 schwarzer, gestrichelter Kasten) und anschließend mit einem Proteine zerschneidenden Enzym, der Protease Trypsin, verdaut. Diese erzeugt kurzkettige Peptide aus dem langkettigen Protein. Die kurzkettigen Peptide werden anschließend chromatographisch aufgetrennt (nano-LC) und ihre Masse in einem ersten massenspektrometrischen Schritt (/MS/) bestimmt. Danach folgt ein Schritt, bei dem die einzelnen Peptide weiter in noch kleinere Teile fragmentiert werden. Die Masse dieser noch kleineren Teile der Peptide wird daraufhin durch einen zweiten massenspektrometrischen Schritt (/MS) aufgezeichnet. Aus den Massen der kleinen Peptidfragmente kann nun die Aminosäuresequenz der Pep- tide bestimmt werden. Daraus kann wiederum die Sequenz des Ausgangsproteins zusammengesetzt werden. Bei der Analyse des Einbaus von nicht-natürlichen Aminosäuren macht man es sich zu Nutze, dass beim Einbau die Masse der einzelnen Peptide des Proteins verändert wird. Dies kommt daher, dass die nicht-natürlichen Aminosäuren andere Massen besitzen als die natürlichen Aminosäuren, z.B. Methionin: 149,21 Da und Ethionin: 163,24 Da. Abbildung 4. Aminosäurepositionen, an denen ein Einbau der nicht-natürlichen Aminosäure Ethionin detektiert werden konnte. Die in der massenspektrometrischen Analyse gefundenen Peptide sind blau unterlegt. Schnittstellen der Protease Trypsin sind schwarz unterstrichen. Methionine sind in der Aminosäuresequenz rot markiert. Methioninpositionen, an deAbb. 4 nen ein Einbau von Ethionin nachgewiesen werden konnte, sind mit einem roten Stern gekennzeichnet. Abbildung 4 zeigt die Zusammenfassung der massenspektrometrischen Analyse der Katalase aus der LBE Kultur. Die roten Sterne kennzeichnen die Positionen im Protein, an denen Ethionineinbau nachgewiesen werden konnte. Allerdings ist der Ethioninanteil im Vergleich zum Methioninanteil noch gering (siehe Analysedetails im Anhang). Der Einbaugrad soll nun im weiteren Verlauf des Projekts am MaxPlanck-Institut für Biochemie verbessert werden. Hierfür soll als erster Schritt die Katalase in ein plasmidbasiertes Expressionssystem überführt werden, was für Bacillus subtilis im Zusammenhang mit nicht-natürlichen Aminosäuren bisher noch nicht untersucht wurde. 21 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 DISKUSSION ZUR BIOSICHERHEIT MIT EXPERTEN „What I cannot create, I do not understand, but: Do I understand, what I can create?“ Richard Feynman Die Synthetische Biologie bietet ungeahnte Möglichkeiten, ähnlich der Computerrevolution oder der synthetischen Chemie zuvor. So stellt zum Beispiel die Regensburger Firma Geneart DNABausteine her, die in nicht allzuferner Zukunft zur Synthetisierung künstlichen Lebens beitragen könnten. Organismen mit speziellen Fähigkeiten könnten von Menschenhand designed werden. Natürlich ist das nach dem heutigen Stand der Technik noch nicht möglich, laut Experten jedoch eine denkbare Entwicklung in naher Zukunft. Aber ist dies überhaupt aus ethischer Sicht vertretbar? Dürfen wir ins Handwerk des Schöpfers eingreifen? Maßt die Menschheit sich an, Gott zu spielen? Die Synthetische Biologie wirft ein weiteres Mal Fragen wissenschaftlicher Verantwortung auf. Besonders überrascht waren wir, dass es in Österreich derzeit keine klare Gesetzes- grundlage gibt bezüglich der Sicherheit im Zusammenhang mit Synthetischer Biologie bzw. in unserem Fall mit synthetischen Proteinen. Wir konnten unser Projekt ohne Probleme oder gesetzliche Einschränkungen durchführen. Besonders wichtig war es uns in unserem Projekt deshalb auch, auf die Sicherheitsaspekte der SynBio näher einzugehen. Am 14.12.2009 organisierten wir eine Abendveranstaltung, in der Fragen zur SynBio allgemein, aber auch zur Sicherheit und zur ethischen Vertretbarkeit erläutert werden sollten. Wir wollten die Menschen dazu anregen, sich eine eigene Meinung zu bilden - denn mit dem Strom schwimmen kann jeder. Selbst ein Statement zu formulieren und dieses auch begründen zu können ist weitaus schwieriger! Um die Diskussion auf einem fachlich hohen Niveau führen zu können, luden wir zwei Experten aus dem Gebiet der SynBio ein: Dr. Nediljko Budisa vom Max-Planck- Institut in München, den „Shootingstar“ der Synthetischen Biologie in Deutschland, und Dr. Markus Schmidt von der Organisation für Internationalen Dialog und Konfliktmanagement (IDC) in Wien. Dr. Schmidt leitet das EU-Projekt „Synbiosafe“, welches eine Diskussion über die Sicherheit der Synthetischen Biologie europaweit anstoßen soll. Die beiden gaben uns mit ihren Präsentationen einen Einblick in die Lehre des „Lebens vom Reißbrett“. Im Anschluss stellten sie sich unseren kritischen Fragen zur Thematik: • Welche Möglichkeiten bietet die Synthetische Biologie? • Wie kann man Risikofaktoren wie zum Beispiel die unbeabsichtigte Freisetzung von synthetischen Lebewesen - in den Griff bekommen? • Wie kann man die Öffentlichkeit in die Diskussion einbinden und dazu anregen, sich eine eigene Meinung darüber zu bilden? Es entbrannte eine nicht enden wollende Fragerunde, die viele zusätzliche Informationen lieferte und viele Unklarkeiten beseitigte. Wir hörten z.B., dass in den USA das MIT-Boston mit einem Wettbewerb namens iGEM - für „Undergraduates“ - die Synthetische Biologie an junge StudentInnen und SchülerInnen heranbringt. So entstanden zum Beispiel ein Escherichia coli-Bakterium mit Bananengeruch, eine synthetische Botox-Hautcreme oder ein Bakterium, welches die menschlichen Ausscheidungen je nach Ernährung und Krankheit unterschiedlich einfärbt. Das beweist, dass die Synthetische Biologie bereits in den „Undergraduate“-Stufen verstanden und angewendet werden kann, die Lehre also gewissermaßen vom Forschungslabor ins „Laienkochstudio“ übertragbar ist. Schon SchülerInnen können sich intensiv mit der Wissenschaft befassen und Nützliches damit bewirken! 22 Und tatsächlich schicken sich auch Privatpersonen an, bei der Konstruktion von Leben „mitzuspielen“. In Garagen und Wohnzimmern werken bereits „Biohacker“ mit Zellkulturen und Petrischalen nach dem „Do-it-Yourself “-Prinzip und bauen im Schnellverfahren biologische Systeme. Ziel der HobbyforscherInnen ist die Schaffung eines „biologischen Werkzeugkastens für die Garage“. Offensichtliches Gefahrenpotenzial besteht natürlich darin, dass die neuen Organismen durchaus Reißaus in die freie Natur nehmen könnten. Die Konsequenzen für die endemische Flora und Fauna kann noch niemand in vollem Umfang abschätzen, dazu ist das Forschungsgebiet zu komplex und neu. Der Sicherheitsforscher Schmidt sieht aber in dieser enthusiasmierten Szene jedenfalls eine „Demokratisierung der Biotechnologie“. Es sind nicht mehr allein die großen Konzerne, die patent- und profitgetrieben die Forschungsthemen bestimmen, sondern pragmatische Ideen vor dem Hintergrund des Open-Source-Gedankens. Wie könnten jedoch die Auswirkungen der SynBio aussehen wenn die Mittel dafür in falsche Hände geraten? Wie sieht es mit „Bioterror“ u.ä. aus? Eine Ahnung von den Gefahren gibt zum Beispiel folgender Fall: Einem Journalisten des „Guardian“ ist es in einem Versuch gelungen, sich einen Abschnitt des Pocken-Erbguts zu bestellen. Er wurde anstandslos beliefert. Außerdem ist es beispielsweise längst gelungen, das Genom von Polio- oder Grippeviren zu synthetisieren. Firmen, die Bio-Bricks kommerziell anbieten, müssen genaue Aufzeichnungen führen und ihre Datenbanken untereinander abgleichen. Sonst könnte es etwa geschehen, dass sich gewiefte Verbrecher bei unterschiedlichen Anbietern die Bausteine eines bestimmten Krankheitserregers besorgen und diese dann nur noch „zusammensetzen“ müssen ... Natürlich gibt es auch vielversprechende Möglichkeiten, die Sicherheit im Umgang mit synthetisch verändertem Material zu gewährleisten. Um die versehentliche Frei- setzung von synthetischen Organismen zu verhindern, könnte man gezielt Schutzmechanismen einbauen, die ein Leben außerhalb des Labors für die Bakterien unmöglich machen. Sogenannte Minimalorganismen etwa, die selber kaum mehr Stoffwechsel betreiben können, sondern alle Nährstoffe im Substrat benötigen, würden in freier Natur nicht überleben können. Deren Umwelt wäre alleinig das Labor oder der Fermenter. Biologische Firewalls: Die Erbsubstanz natürlichen Lebens, die in der Doppelhelix der DNA festgeschrieben ist, besteht aus einer sich ständig wiederholenden Sequenz aus Desoxyribose, einer Base (A für Adenin, G für Guanin, C für Cytosin und T für Thymin) und Phosphor. „Tauscht man nun die Desoxyribose der DNA aus und ersetzt sie mit beispielsweise einer Hexose, hat man ein neues genetisches System geschaffen, in diesem Fall eine ‚HNA‘. Kein natürlicher Organismus könnte diesen HNA lesen, wäre für diese also unsichtbar - eine biologische Firewall. Ein solcher Organismus läuft mit einem neuen, parallelen Betriebssystem“, so der Experte. sollen PR-Desaster vermieden werden, wie sie etwa bei genveränderten Pflanzen und zum Teil bei der Nanotechnologie geschehen sind. „Früher hat es da phasenweise eine große Arroganz der WissenschafterInnen gegeben. Heute sind Forschung und Industrie vorsichtiger, schließlich steht die Zukunft der Bio-Ökonomie auf dem Spiel“, meint Schmidt. Textzitate auch aus „Leben 2.0, Biologie aus dem Baukasten“, www.zukunftswissen.apa.at, Mario Wasserfaller, 12.02.2010 Den Hoffnungen, die in die Synthetische Biologie gesetzt werden, stehen nicht unbedeutende ethische und die Sicherheit betreffende Bedenken gegenüber. Was in der öffentlichen Darstellung rund um die Synthetische Biologie jedoch besonders auffällt, ist die von Beginn an relativ offene Diskussionskultur. Fast in vorauseilendem Gehorsam wird auch seitens der Industrie und Wissenschaft die Technikfolgenabschätzung und Diskussion forciert. Damit 23 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 MAX-PLANCK-INSTITUT BLOG Im Rahmen der Arbeiten mit den Experten vom Max-Planck-Insitut an der HLFS Ursprung bekamen zwei SchülerInnen das Angebot, in den Semesterferien 2010 nach München zu fahren und Seite an Seite mit hochrangigen WissenschaftlerInnen zu forschen. Wir waren ganz aufgeregt und gespannt, wer denn da fahren dürfe. Immerhin bestand das Team ja aus 17 Mitgliedern und viele wären gerne dabei gewesen. Wir beschlossen, das Problem mit einer demokratischen Wahl zu lösen. Ein anonymer Poll im Wiki leistete da hervorragende Dienste. Nach der ersten Abstimmungsrunde lagen drei von uns ex aequo und eine Stichwahl war nötig. Schließlich durften Simone Reiter und Michael Grömer die Reise antreten. Sonntag, 7. Februar 2010: Wir sind gut in München angekommen! Miche Hösl, Doktorand am Max-Planck-Institut (MPI) für Biochemie, hat uns tollerweise direkt am Bahnhof abgeholt und uns auch gleich den Weg zu unserer Pension in Planegg gezeigt. Das hätte sonst womöglich etwas länger gedauert. Anschließend sind wir noch gemütlich auf eine Pizza gegangen, während uns Miche eine Menge an Fragen bezüglich Uni, Doktorarbeit und der Wissenschaft allgemein beantwortete. Wirklich interessant, wie das alles so funktioniert - und gar nicht so einfach zu durchblicken. Man merkte total, welch begeisterter Wissenschaftler er ist. 24 Montag, 8. Februar 2010: Gespannt starteten wir in den Tag! Ob wir das MPI wohl wirklich finden würden? Hatte uns Miche am Vortag alles gut genug erklärt? Vollkommen ausgeschlafen (und mit einem super Frühstück im Bauch) machten wir uns auf den Weg. Tatsächlich lief alles nach Plan … Beim MPI angekommen meldeten wir uns bei der Rezeption an und bekamen sogleich ohne Probleme Ausweise, die uns den Aufenthalt für die gesamte Woche genehmigten. Ein Durchruf der Rezeptionistin zu Dr. Nediljko Budisas Abteilung genügte, und schon kam Miche, um uns den Weg zu zeigen. Er führte uns durch wirklich verwirrende Gänge hin zum Labor- und Bürotrakt, wo er uns gleich Nediljko, Lena und anderen Arbeitsgruppenmitgliedern vorstellte. Schon läutete die Glocke: Wir sind doch hier nicht in der Schule, oder? Verwunderung … So ist das also: Jeden Montag um 10 Uhr gibt es eine „Lagebesprechung“ der gesamten Abteilung. Nachdem das Rätsel um den verschwindenden Kaffee besprochen war, bot sie optimale Gelegenheit, uns - die Jungen hier - vorzustellen … Dann hieß es „Auf geht’s!“ Schließlich waren wir hier, um etwas zu lernen. Wir machten es uns also mit Miche in der Teeküche gemütlich und bekamen eine ausführliche Einführung über unsere bevorstehenden Arbeiten. Natürlich nützten wir diese Gelegenheit auch gleich aus, um viele Fragen zum Thema loszuwerden. Im Lauf des Vormittages stellten wir ein Nährmedium her, gossen Agarplatten und SDS-Gele zum Untersuchen verschiedenster Proteine. Solche Gele kannten wir bereits von den Labortagen an unserer Schule, damals hatten wir diese jedoch nicht selbst hergestellt, weil dafür giftiges Acrylamid benötigt wird. Wir hatten bis zu diesem Tag nicht gewusst, dass so ein SDS-Gel immer aus zwei Teilen besteht, nämlich dem Trenn- und dem Sammelgel. Einen Teil unserer Aufmerksamkeit gewann gleich einmal das Photometer ... ein wahres Designerstück der Laborwelt! Die Mittagspause wurde gemeinsam mit ein paar anderen ForscherInnen in der Teeküche verbracht. Dabei wurde uns bewusst, wie wichtig so eine Mittagspause ist, wenn sie - wie hier - dem Austausch und der Beratung unter den WissenschaftlerInnen dient. Danach konnten wir unsere bereits fest gewordenen SDS-Gele und Agarplatten verräumen bzw. lagerfähig machen. Da es für heute keine passenden Aufgaben mehr gab, wurden wir bereits gegen 15 Uhr entlassen und konnten so noch eine Tour durch München starten. Und schon wieder waren wir gespannt - was wohl der nächste Tag so bringen würde? Wenn alles nach Plan liefe, würde uns morgen Lena Strube, eine Bachelor-Studentin, einiges zeigen. Naja, erstmal hieß es jetzt schlafen und uns erholen, sodass wir morgen voller Kräfte und viel Aufnahmevermögen erneut in das Abenteuer starten können würden. Dienstag, 9. Februar 2010: Wieder einmal ausschlafen … Arbeitsbeginn um 10 Uhr - einfach Spitze, so ein später Start in den Tag! Heute hatten wir volles Programm: Mit Lena führten wir eine Transformation von Bacillus subtilis mit einem Plasmidvektor durch und stellten einige DNA-Gele her. Mit Miche befüllten wir die am Vortag gegossenen Proteingele, färbten und entfärbten diese um sie anschließend auswerten zu können. Außerdem halfen wir Miche noch bei der Durch- führung von Restriktionsverdau-Reaktionen, wobei unsere Pippetierfähigkeit auf die Probe gestellt wurde. Und das Photometer faszinierte uns wieder! Auf Knopfdruck spuckt es dir deine Messwerte aus und macht somit das ständige Herumtragen eines Blockes überflüssig. Zum Neidisch-werden sind aber auch die Bunsenbrenner, die sich von alleine, ganz ohne Feuerzeug, ein- und auch wieder ausschalten. Das „Tratschen“ in der Teeküche schätzten wir jetzt schon sehr: Es entstanden immer äußerst spannende Mittwoch, 10. Februar 2010: Ein echt spannender Tag! Wir durften mit Miche mittels His-Tag das GFP (green fluorescent protein) aufreinigen. Dieser Vorgang wird auch mit unseren im Schullabor erzeugten Enzymen stattfinden. Das GFP befand sich im Zellinneren. Um daran zu kommen, wurden die Zellen durch Ultraschallwellen zerstört und anschließend zentrifugiert, sodass Zellwandreste etc. abgeschieden werden konnten. Im nächsten Schritt bereiteten wir eine Nickelsäule vor. Als wir dann die Proteinlösung überführten, konnte man eine deutliche Grünfärbung der Säule erkennen. Dieses Protein mit seiner kräftigen giftgrünen Farbe ist wirklich faszinierend! Die Säule bindet durch die enthaltenen Nickelionen die mit His-Tag versehenen Proteine. Da auch andere, nicht gewünschte Proteine an der Säule schwach binden können, werden diese nur leicht haftenden Proteine vor dem Äquilibrieren noch durch mehrere Waschgänge gelöst. Dann wurde eluiert, was bedeutet, dass man die an der Säule mit dem His-Tag spezifisch gebundenen Proteine „erntete“, also von der Säule löste. Diese sollten jetzt rein sein. Per Photometermessung bestimmten wir auch gleich die Proteinkonzentration, die uns gute Arbeit bestätigte. Das aufgereinigte Protein sowie auch die einzelnen Waschlösungen würden wir dann morgen auf ein Gel auftragen. Miche nahm uns heute außerdem noch auf eine „Wanderung“ durch das echt riesengroße MPI mit: zu Lissy, der Herrscherin über das Massenspektrometer. Gespräche und Diskussionen. Diesmal bekamen wir einen Einblick, wie es in der Wissenschaft wirklich so läuft und was man als Wissenschaftler auf keinen Fall machen sollte: Anscheinend kommt es immer wieder mal vor, dass ForscherInnen etwas veröffentlichen, was sie nur ahnen, jedoch noch nicht fertig bewiesen haben - allein des Ruhmes wegen! Eigentlich unglaublich, oder? Da so etwas in den meisten Fällen früher oder später auffliegt und innerhalb von Tagen die gesamte Wissenschaftsszene Bescheid weiß, sind solche „inkorrekten Behauptungen“ aber ziemlich riskant und haben schon viele Jobs gekostet. Nach diesem Tag war auch endlich das Rätsel über die Funktion des Massenspektrometers, zumindest im Groben, geklärt. Miche hat sich wirklich bemüht, uns die äußerst komplexe Funktionsweise etwas näher zu bringen, aber ganz werden wir es wohl nie verstehen. Jedenfalls ist es einfach unvorstellbar, was heutzutage schon alles möglich ist! Es ist wirklich beeindruckend, was dieses nicht gerade kleine Gerät leisten kann! Aus nur wenigen μl einer Probe ermittelt es die Masse eine Moleküls fast auf den kDa (KiloDalton) genau. 25 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 Erlernte Methoden Donnerstag, 11. Februar 2010: Schön langsam bekamen wir einen Überblick über die am häufigsten gebrauchten Labortechniken. Nachdem wir tags zuvor Proteine aufgereinigt hatten, war heute DNA an der Reihe. Diesmal wurden die Zellen chemisch (durch NaOH) aufgeschlossen. Durch einen Puffer fielen störende Proteine und Kohlenhydrate aus, welche wir abzentrifugierten. Die DNA blieb im Puffer gelöst und konnte direkt in die vorbereiteten Säulen überführt werden. Nach dem Äquilibrieren wurde die DNA zuerst mit Isopropanol und anschließend mit Ethanol gewaschen. Zuletzt haben wir die DNA getrocknet und in Wasser gelöst. Mit dem Nanotrop konnte die genaue Konzentration bestimmt werden. Zum Abschluss dieses Tages ließen wir dann ein Gel mit den gestrigen Proben von der GFP-Aufreinigung laufen und brachten das aufgereinigte GFP zur Massenspektrometer-Untersuchung. Mal schauen: Mit ein bisschen Glück war Lissy nicht zu beschäftigt und wir würden morgen schon die Messwerte bekommen ... 26 Freitag, 12. Februar 2010: Und schon ist eine Woche um! Wir waren beide sehr überrascht, wie schnell diese Semesterferien vergangen waren. Den Freitag nützten wir, um alle Ergebnisse dieser Woche vollständig auszuwerten und zu dokumentieren. Außerdem hatten wir noch das Glück, den echten wissenschaftlichen Vortrag eines Professors namens Dr. Mohamed A. Marahiel von der Universität in Marburg besuchen zu können. Er fasste in einer Stunde die Ergebnisse zusammen, die er mit seiner Arbeitsgruppe in 20 Jahren erarbeitet hatte. Echt spannend! Er hatte erforscht, dass es neben den Ribosomen noch einen anderen Weg gibt, wie kleine Proteine (Peptide) entstehen können. Auf diese Art und Weise werden zum Beispiel Antibiotika in Organismen produziert. Es war wirklich toll, einmal zu sehen, wie so etwas abläuft. Dass wir kaum etwas verstanden haben, lag wohl nicht nur an der englischen Sprache, sondern auch an fehlendem Fachwissen. Aber wir nahmen es nicht so tragisch - schließlich haben wir ja auch noch nicht studiert! Zum Abschluss gingen wir alle gemeinsam in die Mensa mittagessen. Bis wir unsere sieben Sachen beisammen hatten und mit der Verabschiedung von allen unseren netten „ArbeitskollegInnen“ fertig waren, war es 15 Uhr. Puh, geschafft! Gerade noch rechtzeitig, dass Miche noch zum Fußballspielen kam und wir unseren Zug erwischten. Etwas erschöpft waren wir jetzt schon, nach dieser Woche voll neuer Eindrücke und Erfahrungen. Zweifel bestand jedoch keiner: Die „geopferte“ Ferienwoche am MPI war ein voller Erfolg und hat sich auf jeden Fall ausgezahlt! Hätten wir diese Gelegenheit nicht genützt, wir hätten es sicherlich bereut. Es war eine einmalige Möglichkeit, Einblick in den Alltag des Forscherlebens zu gewinnen. Durch diese „Schnupperwoche“ bauten wir unser Fachwissen und unsere soziale Kompetenz gleichzeitig aus. Eine größere Hilfe zur Studienwahl nach der Matura kann es kaum geben! Expressionstest: Wir führten mit einem Barstar-Modellprotein einen Expressionstest durch. Barstar ist ein kleines Protein, synthetisiert von Bacillus amyloliquefaciens. Dieses Protein wurde zuvor von Miche schon vorbereitet, indem er ein Stoppcodon in seine Sequenz eingebaut hat. Durch den Einbau der synthetischen Aminosäure P-Benzoyl-L-Phenylalanin (Bpa) könnte dieses Stoppcodon überlesen werden. Erwartung: Wenn die Expression richtig funktioniert - das würde bedeuten dass das Stoppcodon überlesen und an seiner Stelle Bpa eingebaut wird - muss eine Reinigung von GFPuv: Bei der His-Tag-Methode wird die DNA-Sequenz des zu isolierenden Proteins um die Codons eines HisTags (His-Linker, bestehend aus sechs Histidinresten) ergänzt. Dieser HisTag bindet spezifisch an ein Säulenmaterial mit zweiwertigen Nickel-Ionen. Diese Spezifität gewährleistet, dass nur das Fusionsprotein an das Säulenmaterial bindet. Erwartung: Mittels der His-Tag-Methode soll es gelingen, das GFPuv effektiv aufzureinigen. Durchführung: • Aufschließen der Zellen mittels Ultraschall • Abzentrifugieren der Zellbestandteile • Vorbereiten der Nickel-Säule • Aufreinigung des Proteins aus dem Lysat (Binden des Proteins, Elution des Proteins) Bande bei ca. 10 kDa erscheinen. Durchführung: • Picken von Einzelkolonien und Ansetzen einer Flüssigkultur • Kontrolle des Wachstums durch Messen von OD bei 600 nm • wenn OD-Wert bei ca. 0.6, Induktion der Proteinexpression mit IPTG • Ernte der Zellen nach 4.5 h Expression bei 30 °C Ergebnis: Auf dem Gel ist keine Bande bei 10 kDa sichtbar. Folglich wurde das Stoppcodon nicht überbrückt und auch die synthetische Aminosäure Bpa nicht eingebaut. • Messung der Proteinkonzentration • Vorbereitung der Probe für die Massenspektrometrie • Fluoreszenz und UV Spektrum messen Ergebnis: Das 1. Foto des SDS-Gels wurde unter UV-Licht gemacht, hier sind die Banden kaum zu erkennen. Für das 2. Foto wurden die Proteine eingefärbt. S: Standard Proteinmarker (5μl), P: Zellpellet; D: Durchfluss; W 1-3: Waschgang 1-3; GFP: aufgereinigtes Protein/GFP. Auf dem 2. Bild kann man deutlich sehen, wie viele Proteine verschiedener Länge bereits bei dem Durchfluss und 1. Waschgang abgeschieden wurden. Beim 2. und 3. Waschgang war die Säule schon gesättigt, weshalb sich in den Waschlösungen auch geringe Mengen des GFPs befinden. Dies ist an den zwei Banden mit 25 kDa zu erkennen, welche auf gleicher Höhe mit der des aufgereinigten GFPs sind. Die dicke Bande in der Spalte GFP zeigt, dass die Aufreinigung gut funktioniert hat. 27 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 Im Massenspektrometer wurde die genaue Masse des aufgereinigten Proteins bestimmt. Anhand des folgenden Graphen ist ersichtlich, dass das GFP eine Masse von 28014 kDa hat. Transformation eines Shuttlevektors in Bacillus subtilis: Erwartungen: Der Bacillus-subtilis–Stamm, bei welchem das Gen zur Amylaseproduktion „ausgeschaltet“ wurde, sollte einen Shuttlevektor mit Amylasegen aufnehmen und so wieder zur Amylaseproduktion angeregt werden. Schüttler sollten die Zellen die Plasmide aufgenommen haben und somit auch antibiotikaresistent sein) • die Zellen werden auf eine Platte aufgestrichen, über Nacht müssen sich Kolonien bilden Durchführung: • die Bacillus subtilis-Kultur wir angesetzt • bevor die Zellen die stationäre Phase erreichen, werden sie geerntet (nach 5 h) • einem Teil der Zellen wird EDTA beigemengt (fördert eine spätere Aufnahme der Vektoren) • Plasmide werden dazugegeben (nach zwei Stunden bei 37 °C im Auf den Platten bildeten sich keine Kolonien, folglich wurde der Vektor nicht aufgenommen und die Zellen waren somit nicht antibiotikaressistent. Ein möglicher Grund könnte sein, dass wir EDTA verwendet haben und nicht EGTA, wie es im Protokoll steht. Ergebnis: In folgendem Diagramm ist das Wachstum der Zellen dargestellt: Aufreinigung von Plasmid- DNA/ Restriktionsverdau: Erwartung: Sauber aufgereinigte Plasmid- DNA und Banden mit der richtigen Größe am DNA-Gel. Durchführung: • Zellpellet lösen • Zellen werden chemisch aufgeschlossen, durch Zugabe eines weiteren Puffers fallen Kohlenhydrate, Proteine, etc. aus • durch Zentrifugieren kann die gelöste DNA ohne ausgefallenen Stoffen direkt in eine vorbereitete Säule überführt werden • nach dem Equilibrieren wird die DNA mit Isopropanol und Ethanol gewaschen • die getrocknete DNA wird in Wasser gelöst • mittels Nanotrop kann die Konzentration ermittelt werden • Restriktionsverdau • auf DNA-Gel auftragen 28 Ergebnisse: Es ist bei jeder Probe eine deutliche Bande zu erkennen. Bei den Proben 3, 4 und 5 hat das Restriktionsenzym zweimal geschnitten, was man an den zwei Banden je Reihe ablesen kann(bei 4500 und 800 bp). 29 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 EINDRÜCKE / STATEMENTS Toni: Gen- und Biotechnologie haben mich schon immer fasziniert. Sie bieten eine Menge von Vorteilen für die Landwirtschaft, die Lebensmittelindustrie und die Medizin, die man sich zu Nutze machen kann. Nun folgt die Synthetische Biologie, eine Wissenschaft, die noch viel mehr Potential aufweist! Einfach unglaublich! Man muss es selbst miterlebt haben, um glauben zu können, was der Austausch einer einzigen Aminosäure bei einem Enzym bewirken kann. Am spannendsten finde ich die Versuche mit dem neuem Enzym. Dafür wird eine Menge an Kreativität gefordert. Faktoren wie Temperatur, pH-Wert und viele weitere Bedingungen schaffen ein breites Spektrum für Tests, doch die Ideen gehen uns nicht aus. Die Spannung bleibt bis zum Endresultat aufrecht. Ob das „verbesserte“ Enzym später Verwendung finden wird, wird sich erst herausstellen. Ich bin mir sicher, dass die Synthetische Biologie in der Zukunft eine wichtige Rolle spielen wird. Die Wissenschaft schläft ja nicht! Simone: Ich bin das erste Mal bei einem GBT Projekt dabei und absolut begeistert! Zuvor hätte ich nie gedacht, dass so viel Arbeit einen so riesigen Spaß machen kann. Besonders toll fand ich die Zusammenarbeit mit den ForscherInnen vom MPI - Birgit und Miche. Sie lieferten uns auf jede unserer Fragen ausführliche Antworten, die auch für uns SchülerInnen verständlich waren. Ein weiterer toller Punkt: Unsere Ideen, egal wo und wann, waren immer erwünscht und wurden vor allem auch ernstgenommen. Wir durften von uns ausgedachte Versuche selber planen und dann auch durchführen, was die ganze Arbeit noch viel spannender machte. Auch die kritischen Sichtweisen wurden nie außer Acht gelassen und über mögliche Risiken wurde diskutiert - meiner Meinung nach ist das besonders wichtig, da man alles von zwei Seiten betrachten muss. Alles in allem ein echt super-interessantes, gut organisiertes Projekt! 30 Pilot: „Synthetische Biologie“, da fasziniert mich schon der Name, weil das ist ja quasi ein Widerspruch in sich. Als sich das bunt zusammengewürfelte Team aus 4 Klassen mit einem derartigen Engagement auf die Arbeit stürzte, sodass unsere Kooperationspartner vom Max-Planck-Institut Martinsried ins Schwitzen kamen, da wollten wir immer mehr: Eine synthetische Katalase produzieren, und dann noch Amylase, was für den Klimaschutz tun, neue Gesetze anregen, Sicherheitsfragen diskutieren u.v.m. Ein unglaubliche Dynamik entstand. Jeder brachte seine Stärken ein, jeder kämpfte, wenn es zeitlich eng wurde, in den Ferien, an Sonntagen in den Nächten wurde gearbeitet. Ein verschworenes Team, das auf keinen Fall mit Frankenstein in Verbindung gebracht werden möchte, weil: wer sowas sagt, hat nicht verstanden, was Synthetische Biologie ist und den Wert unserer Arbeit nicht erkannt. Julian: Über SynBio mach ich mir eigentlich erst Gedanken, seit ich erfahren habe, dass wir sie als Thema im heurigen Projekt bearbeiten werden. Ich habe mich ein bisschen im Internet erkundigt und war fasziniert, was nicht alles möglich ist. Ich dachte schon, das Bild, dass ich von der SynBio hatte, könnte nicht besser werden, doch umso mehr ich mit dem Projekt vertraut wurde, desto überzeugter von der SynBio war bzw. bin ich. Stellen Sie sich vor, man könnte die Außenanforderungen und die Umsatzkraft von Industrieenzymen nach Belieben verändern! Wissen Sie, wieviel Geld man damit sparen könnte? Ganze Arbeitsschritte in der Industrie könnte man auslassen und somit auch weniger Energie verbrauchen, was wiederum den Klimaschutz fördern würde. Man ist noch nicht soweit, dass man alles gezielt und schnell verändern kann, aber man sollte diese Art der Forschung auch nicht verbieten, nur weil einige Unwissende Angst haben und glauben die SynBio wäre eine Gefahr für die Umwelt. Wir zum Beispiel haben in unserem Projekt mit auxotrophen Organismen gearbeitet, die außerhalb ihres Nährbodens sofort sterben würden. Wie sollte sich diese neue Art der Forschung sonst entwickeln, es braucht einfach Zeit. Vielleicht bekommt die SynBio auch irgendwann einmal die Zustimmung der Menschheit und wird nicht mehr von Unwissenden als Frankenstein-Wissenschaft verdammt. Mich hat die SynBio gefesselt, das muss ich ehrlich zugeben. Ich bin zwar noch Schüler und kein Experte auf diesem Gebiet, doch anhand dessen, was ich im Internet und Zeitschriften gelesen habe, oder auch im Laufe des Projekts erfahren habe, finde ich, dass diese Wissenschaft unbedingt weitergeführt, verfeinert und genutzt werden muss. Doch dies wird nur funktionieren, wenn man den Menschen ihre Angst nimmt und Ihnen gleichzeitig nahelegt welch endloses Potenzial in der SynBio steckt. Unser Bericht über dieses Projekt ist zum Beispiel ein kleiner Schritt in diese Richtung. Da ich zum ersten Mal bei so einem Projekt mitarbeite, war ich von Anfang an begeistert wie gut unser Team zusammenarbeitet. Dankbar bin ich auch, dass Birgit und Michael vom Max-PlanckInstitut gekommen sind und uns in die gefinkelte Laborarbeit eingeführt, uns alles erklärten haben und auch auf unsere spezifischen Fragen stets eine Antwort wussten. Einen Dank spreche ich auch an Konrad Steiner aus, durch den dieses Projekt erst möglich wurde, das Spezialisten und SchülerInnen verband. Rupi: Ich habe mich schon immer gerne mit Naturwissenschaften beschäftigt und mich ein ums andere Mal von ihnen beindrucken lassen. Das waren Wissenschaften wie Physik, Chemie, Biologie oder zuletzt Gentechnik. Jetzt aber bin ich mitten in einem Projekt, das quasi dabei ist, einer neuen Naturwissenschaft, der Synthetische Biologie bzw. SynBio, Gestalt zu verleihen und das alles in Zusammenarbeit mit dem Max-Planck-Institut. Was für ein Gedanke! Seit neuestem faszinieren mich auch die Rechtsfragen, auch weil mein Bruder in diese Richtung studiert. Ab und zu bekomm‘ ich von ihm einen kleinen Vortrag bezüglich Recht und daraus konnte ich immer nur eins entnehmen: Es ist nahezu alles rechtlich geregelt. Im Fachjargon nennt man das „Gesetzesflut“ bzw. „Regelungswut“. Umso mehr war ich verwun- dert über die Tatsache, dass es zu SynBio noch keinerlei Gesetze/Regelungen gibt. Man darf also in diesem Bereich derzeit alles machen, solange es nicht gegen ein allgemein gültiges Gesetz verstößt. Das ist, was ich an unserem Projekt fast noch cooler finde: Dass wir vielleicht eine Botschaft aussenden können, um den Gesetzgeber auf einen aufstrebenden Zweig der Wissenschaft aufmerksam zu machen, damit Gesetze entstehen können, solange noch Zeit ist und nicht erst dann, wenn die Zeit drängt und unüberdachte Entscheidungen getroffen werden. Zum Schluss möchte ich noch Herrn Konrad Steiner hervorheben, der bei allen Projekten enorm viel Arbeit, Motivation, Verständnis und Kompetenz einbringt. Er ist derjenige, der aus den SchülerInnen das herauslockt, was sie am besten können. Norbert: Ich komme aus einer Landwirtschaft, daher lagen meine Interessen immer schon wo anders als in einem Labor. Bei diesem Projekt wirkte ich anfangs nur mit, weil ich einfach etwas Neues sehen wollte. Doch dann dauerte es nicht lange und ich fand wirklich gefallen am Projekt. Zum einen war es spannend zu sehen, mit welchen Möglichkeiten die Forscher in den Laboren arbeiten können, zum anderen war es für mich - und ich denke auch für alle anderen - eine große Freude, gemeinsam an einem so tollen Pro- jekt zu arbeiten. Bei der Vorbereitung erfuhren wir von unserem „Piloten“, Herrn Konrad Steiner, welchen Stellenwert dieser Themenbereich bereits auf der ganzen Welt hat. Leider wird für die Forschung im Bereich der Synthetischen Biologie in Österreich nicht viel getan. Daher finde ich es umso wichtiger, dass wir SchülerInnen jetzt schon viel Arbeit geleistet und wichtige Erkenntnise für die Forschung gewonnen haben. 31 Synthetische Biologie am Prüfstand Schule Michael: „Synthetische Biologie“: Als ich erfuhr, das würde unser Projektthema sein, war ich zuerst planlos. Mit dem Begriff Biologie konnte ich ja etwas anfangen. Aber was synthetisch in dem Zusammenhang bedeutete, war mir unklar. Auch deshalb war ich sehr gespannt auf das Projekt, auf die Ergebnisse mindestens genauso auf die Durchführung. Und meine Erwartungen sind nicht enttäuscht worden! Das Projekt war wissenschaftlich ein glatter Erfolg, was allerdings nicht heißen soll, dass wir immer nur streng wissenschaflich waren. Besonders die Gespräche mit unseren Kollegen vom MPI waren oft sehr interessant. Ich kann zurückblickend sagen, dass meine Unterhaltungen mit Miche von Polymerasen bis zu Scrubs reichten! Das Projekt war für mich in vielen Hinsichten lehrreich. Ich habe nicht nur labortechnische Kenntnisse gewonnen, sondern habe auch von einem Profi Filmen und Filme schneiden gelernt und bin mit vielen Programmen (Photoshop, Magix) vertraut geworden. Was kann man sich von einem Projekt mehr erwarten? HLFS Ursprung 2009 -2011 Max: Etwas völlig neues erschaffen? Ich habe mich immer schon für das Erfinden von Mechanismen interessiert. Bis ich mich von der Biologie faszinieren lies. Und nun: Beides kombiniert erfüllt den perfekten Lebenstraum. Synthetische Biologie. Unumstritten ein Forschungszweig der Zukunft. Und die Möglichkeit, die ersten Schritte an unserer Schule durchführen zu dürfen ... ein einziger Traum. Markus: Ich war am Beginn des Schuljahres sofort vom Thema SynBio begeistert, obwohl ich zum Start des Projektes noch nicht genau wusste, wo die wirklichen Unterschiede zwischen der herkömmlichen Gentechnik und der Synthetischen Biologie lagen. Doch als umso spannender erwies es sich für mich, diese doch so gravierenden Differenzen durch die Auseinandersetzung mit diesem hoch interessanten und zukunftsreichen Thema zu erkennen. Und was gibt es besseres, als mit dieser Technik gleich selbst im Labor zu arbeiten? Katharina: „SynBio? - Was ist das?“ Diese Frage stellte ich mir noch am Anfang des Schuljahres und ich wusste: Ich will darüber mehr wissen! Ich bin jetzt schon das zweite Mal bei einem GBT Projekt dabei und kann wirklich sagen, dass der Wissendurst danach einfach nicht mehr aufhört. Wie jedes Jahr gehört auch wieder ein motiviertes Team dazu, was auch heuer wieder der Fall war. Man kann wirklich sagen: Wenn das Team nicht so überzeugt und motiviert gewesen wäre, würden wir das heute nicht schreiben. Auch Hut ab vor Herrn Steiner, der uns immer unterstüzt hat und dieses Projekt überhaupt ermöglichte. Hanna: Auch heuer wurde im Freifach Gen- und Biotechnologie wieder ein tolles Projekt gestartet. Gen- und Biotechnologie klingt jetzt vielleicht für viele NEGATIV, jedoch steckt hinter diesen Wörtern noch viel, viel, viel mehr als man glauben könnte bzw. so 32 hört. Nach unserem Riesen-Erfolg mit dem letztjährigen Projekt „Wenn Lifestyle krank macht“ habe ich gleich gewusst: Beim neuen Projekt muss ich wieder dabei sein. Projekte sind jedes mal viel Arbeit, aber sie haben mir gezeigt, dass man mit einem gutem Team und Projektleiter und viel Motivation einiges auf die Beine stellen kann. Durch die Super-Zusammenarbeit innerhalb des Teams und mit dem Max-PlanckInstitut wurde auch dieses Projekt zum Erfolg und ein Riesen-Erlebnis für uns alle. Ein Einblick in eine neue Materie: Synthetische Biologie. Klingt vielleicht unheimlich, ist jedoch richtig interessant und wir waren erstaunt, was alles in unserem Schullabor möglich ist. Alle Ergebnisse wurden immer mit Spannung erwartet und natürlich wurde mit Fotos und Laborberichten alles genauestens dokumentiert. ;-) diesem Bereich sogar eine kleine Vorreiterrolle in Österreich einnehmen konnten. Auch im Labor konnten wir durch das Arbeiten mit echten Profis sehr viel lernen. Andi: In unserem heurigen Projekt ging es um „synthetische Biologie“. Da niemand von uns wirklich etwas mit diesem Begriff anfangen konnte, mussten wir uns erstmal in die Materie vertiefen. Wir hatten viele Ideen, viele Vorschläge, und nicht alles konnten wir unterbringen. Doch letztendlich war es ein sehr lehrreiches und spannendes Projekt. Besonders die Laborarbeit lag sehr in meinem Interesse. Lorenz: Während meiner ganzen Schulzeit haben mich naturwissenschaftliche Fächer wie Physik, Chemie, Biologie, Gen- und Biotechnologie brennend interessiert. Und auch am Beginn dieses Schuljahres machten wir uns wieder einmal auf die Suche nach einem ganz neuen, super-interessanten Thema. Schlussendlich entschieden wir, uns mit Synthetischer Biologie zu beschäftigen. Nach dem ersten Studieren von einigen Texten zu diesem Thema waren wir von den unbeschreiblichen Möglichkeiten fasziniert, die diese Technologie bietet. Natürlich machten wir uns sofort über die möglichen Risiken Gedanken, die sich bei einem unkontrollierten Gebrauch ergeben könnten. Ich finde es besonders wichtig, dass man sich mit neuen Technologien beschäftigt und diese versteht, lernt, einen kritischen Standpunkt einzunehmen, die Vorteile zu verstehen und die möglichen Risiken abzuschätzen. Ich hoffe, dass es, sobald das Thema „Synthetische Biologie“ öffentlich diskutiert wird, nicht wieder zu einer „Panikmache“ kommt, wie das bei der Gentechnik der Fall war. Eine vernünftige, kritische Diskussion über das Thema Synthetische Biologie wäre eines der wichtigsten Ziele in diesem Schulprojekt. An dieser Stelle möchte ich mich auch bei unserem Projektleiter Professor Dr. Konrad Steiner, dem Motor dieses Projekts, bedanken. Er stellte die nötigen Kontakte mit top WissenschaftlerInnen her, ohne deren Hilfe wir niemals so weit gekommen wären. Er trieb uns an und mit seiner Fähigkeit, mitreißende Motivationsreden zu halten, hat er das Gelingen unseres Experiments erst möglich gemacht. Hemets Flo: Wir wussten wenig über SynBio - dafür war die Arbeit mit dieser Technik umso spannender. Natürlich ist es sehr interessant, in einem komplett neuen Bereich zu arbeiten. Es freut mich sehr, dass wir in Klinga: Ich darf nun schon zum zweiten Mal bei einem Projekt des Gen- und Biotechnologie Freifaches an unserer Schule teilnehmen und bin abermals begeistert, was eine Truppe von forschungsmotivierten Schü- Sepp: Die Freude, dass ich heuer beim Großprojekt „SynBio“ mitarbeiten durfte, war besonders groß, da ich am letztjährigen Projekt aufgrund schulischer Komplikationen „nur“ in dem ebenso interessanten Kleinthemenpark meine Interesse an neuartigen Technologien nähren konnte. Aufgrund des hohen Andranges im Labor habe ich mich dafür entschieden, meine Kenntnisse im ethischen Bereich zu erweitern. lerInnen bewerkstelligen kann! Dieses Jahr widmeten wir uns einem neuen Wissenschaftszweig, der Synthetischen Biologie. Von Anfang an war ich überwältigt, welches Potential in der Lehre vom ‚Reißbrettleben‘ stecken könnte. Man bedenke nur die Möglichkeit, ein völlig neuartiges Lebewesen im Labor von Grund auf zu gestalten und zu erzeugen. Bakterien könnten Treibhausgase in Baustoffe verwandeln, Medikamente könnten so billig erzeugt werden wie noch nie - das Anwendungsspektrum der SynBio ist theoretisch unbegrenzt. Ein langjähriger Traum vieler WissenschaftlerInnen würde damit erfüllt. Natürlich ist die Technik dazu noch nicht ausgereift, jedoch nähern wir uns in riesigen Schritten vielversprechenden Ergebnissen. Ich hoffe, wir konnten mit unserer Arbeit das Verständnis für eine aufstrebende Technologie ein wenig erweitern, wobei wir speziell auch auf den Sicherheits- und Ethikaspekt aufmerksam machen wollten. Besonders viel Spaß hat mir auch wieder die Laborarbeit gemacht, wobei ein großer Dank den ForscherInnen vom Max-Planck-Institut gebührt. Danke für ein tolles Projekt! Birgit: Ich war sehr begeistert vom Enthusiasmus aller Teilnehmer. Eure Motivation war tatsächlich grenzenlos und Euer Forscherdrang überwältigend. Ich würde mir eine ähnliche Begeisterung auch bei meinen Studenten wünschen. Es hat mir sehr großen Spaß gemacht, mit Euch zu arbeiten, ich glaub wir waren ein echt gutes Team. Das schöne Resultat unserer Be- mühungen spricht ja auch für sich. Ich bin auch sehr beeindruckt, dass SchülerInnen sich mit einer ganz neuen Technologie so eingehend auseinandersetzen wollen. Einerseits fachlich durch das Erlernen neuer Methoden (von denen andere SchülerInnen meist nicht mal was im regulären Unterricht hören), andererseits aber auch in Bezug auf die ethischen Fragen, die neue Technologien immer aufwerfen. Die Diskussion mit Markus Schmidt hat diese Aspekte sehr gut beleuchtet. Mein Fazit: Das war ein rundum gelungenes Projekt! Cool, dass ich mitmachen durfte! Miche: Ich konnte während meiner Doktorarbeit bisher einige Erfahrungen mit der Betreuung von Studenten - sei es nun bei Praktika an der Uni oder bei Bachelor/ Masterarbeiten direkt bei uns im Labor - sammeln. Als ich mich zur Betreuung dieses Schülerprojekts bereit erklärte, wusste ich jedoch nicht genau, was auf mich zukommen würde. Dementsprechend überrascht war ich auch von der Professionalität, der Auffassungsgabe, dem Fachwissen und der Einsatzbereitschaft der ganzen Gruppe. Es hat mich wirklich sehr beeindruckt, was sich im Rahmen eines solchen Schulprojekts alles realisieren lässt, wenn die beteiligten Personen wirklich dahinterstehen und sich engagieren. So ein Projekt hätte ich mir zu Schulzeiten auch gewünscht! Mir hat es wirklich sehr großen Spaß gemacht, mit Euch zu arbeiten! Außerdem hab auch ich dabei eine Menge dazugelernt! 33 Synthetische Biologie am Prüfstand Schule HLFS Ursprung 2009 -2011 SPONSOREN / PARTNER TEAM HLFS URSPRUNG Max-Planck-Institut für Biochemie, Martinsried BRD Altendorfer Rupert Brunschmid Martin Gimpl Anton Gratzer Julian PD Dr. Nediljko Budisa Grömer Michael Habl Maximilian Harz Andreas Hemetsberger Florian Hemetsberger Norbert Klinger Florian Lichtmannsperger Katharina Pendl Markus Pfleger Johanna Prof. Dr. Konrad Steiner, Projektleitung Reiter Simone Schwaiger Lorenz Strobl Josef Lena Strube Dr. Birgit Wiltschi Mag. Michael Hösl Graphik, Film, Web Mag. (FH) Sonnleitner Daniela Stockinger Markus Mag. Russegger Harald Senior „Projektler“ Kreuzeder Andreas Strieder Emanuel Kontakt: [email protected] Anna und Univ.Prof. Dr. Hans Adam Gemeinde Elixhausen Bildungsförderungsfonds für Gesundheit und Nachhaltige Entwicklung 34 35