Evolutionsökologie
Werbung

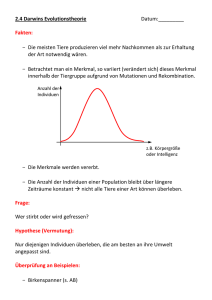
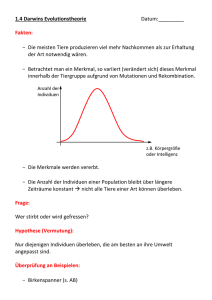
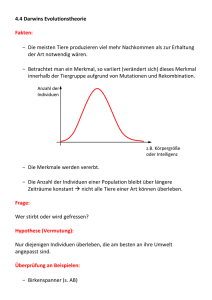
Evolutionsökologie Inhalt 8.1 Einleitung 8.2 Molekulare Ökologie: Unterschiede zwischen Artengenossen und Arten 8.3 Koevolutionäres Wettrüsten 8.4 Mutualistische Interaktionen Schlüsselkonzepte Dieses Kapitel soll › › die molekularen Marker vorstellen, die in der Ökologie verwendet werden; zeigen, wie mit diesen Markern die genetischen Unterschiede innerhalb von Arten und zwischen Arten untersucht werden können; › die Bedeutung des koevolutionären Wettrüstens zwischen Pflanzen und pflanzenfressenden Insekten und zwischen Parasiten und ihren Wirten für die Dynamik von Populationen aufzeigen; › auf die Rolle von mutualistischen Interaktionen und auf ihre entscheidende Bedeutung für die betreffenden Arten und nahezu alle Lebensgemeinschaften der Erde aufmerksam machen; › den Beitrag mutualistischer Beziehungen für eine Vielzahl von Bereichen vorstellen, von der Landwirtschaft über Verdauung und Wurzelsysteme bis zur Stickstofffixierung für Pflanzen. 8 298 Teil 3 Individuen, Populationen, Gemeinschaften und Ökosysteme Wir haben bereits festgestellt, dass in der gesamten Ökologie nichts einen Sinn ergibt, außer wenn man es im Lichte der Evolution betrachtet. Aber es gibt Bereiche in der Ökologie, wo Evolution wichtiger ist als in anderen. So kann es nötig sein, die Gene von Individuen im Detail zu analysieren oder zu unter­ suchen, welche Bedeutung Arten gegenseitig für ihre Evolution haben. 8.1 Einleitung In Kap. 2 haben wir klar gemacht, warum für dieses gesamte Buch der leicht veränderte, berühmte Spruch von Dobzhansky gilt, dass „nichts in der Ökologie einen Sinn ergibt, außer wenn man es im Lichte der Evolution betrachtet“. Aber Evolution ist mehr als die Grundlage der Ökologie (und des ganzen Rests der Biologie). In vielen Bereichen der Ökologie stehen evolutionäre Anpassungen durch natürliche Selektion tatsächlich im Mittelpunkt. Zur Beschreibung dieser Bereiche wird der Begriff „Evolutionsökologie“ verwendet. In einigen vorangegangenen Kapiteln wurden bereits Themen aus dem Bereich der Evolutionsökologie als wichtige Bestandteile von breiteren ökologischen Fragen behandelt. In Kap. 3 haben wir die Eigenschaften und die Bedeutung von Abwehrstrategien betrachtet, mit denen sich Pflanzen und Beutetiere gegen ihre Feinde verteidigen. In Kap. 5 haben wir gesehen, wie Life-history Strategien (d. h. der Ablauf von Wachstum und Reproduktion, etc.) nur im Zusammenhang mit den Lebensräumen verstanden werden können, in denen sie sich evolviert haben. In Kap. 6 haben wir interspezifische Konkurrenz als treibende evolutionäre Kraft für die Koexistenz oder die Verdrängung von Arten kennen gelernt. Und in Kap. 7 haben wir die Theorie des optimalen Nahrungserwerbs diskutiert: Die Evolution von Verhaltensstrategien, mit denen Räuber ihren Fortpflanzungserfolg maximieren und die gleichzeitig die dynamische Beziehung zur Beute formen. Die Aufzählung ist allerdings beileibe keine vollständige Übersicht über die Themen der Evolutionsökologie. Im vorliegenden Kapitel behandeln wir deshalb noch eine Reihe weiterer Aspekte (wobei die Gesamtliste dann immer noch nicht vollständig sein wird). Wir werden uns dabei besonders auf Koevolution konzentrieren: Artenpaare, die gegenseitig als treibende Selektionskraft auf ihre Evolution wirken. Der Aspekt des koevolutionären Wettrüstens zwischen Räuber und Beute wird in Abschn. 8.3 aufgegriffen. Dabei wird ein besonderer Schwerpunkt auf die Beziehungen zwischen Wirten und ihren Pathogenen gelegt werden: Jede Anpassung der Beute, welche den Angriff eines Räubers abwehrt oder vermeidet, bewirkt eine entsprechende Anpassung auf der Seite des Räubers, diese Abwehrmaßnahmen zu überwinden. Aber nicht alle koevolutionären Beziehungen sind feindlicher Natur. Viele Artenpaare leben mutualistisch und beide Partner profitieren, zumindest insgesamt betrachtet, von der Beziehung. Einige der wichtigsten dieser Mutualismen, z. B. Bestäubung, Korallen und Stickstofffixierung, werden in Abschn. 8.4 diskutiert. Wir beginnen allerdings nicht mit der Interaktion von Arten, sondern mit der evolutionären Differenzierung innerhalb von Arten und zwischen Arten. Dabei konzentrieren wir uns auf solche Differenzierungen, welche durch die neu entwickelten Methoden der Molekulargenetik untersucht und daher oft in den Bereich der „Molekularen Ökologie“ gerechnet werden. Kapitel 8 Evolutionsökologie 8.2 299 Molekulare Ökologie: Differenzierung innerhalb von Arten und zwischen Arten Meistens ist es für Ökologen völlig ausreichend, „Populationen“ oder „Arten“ so zu behandeln, als ob sie homogene, gegeneinander abgrenzbare Einheiten wären. So reden wir beispielsweise über die „Verbreitung des Indischen Elefanten“, wobei wir außer Acht lassen, dass die Art tatsächlich in getrennte Rassen oder Untergruppen zerfällt (Abb. 8.1). In manchen Fällen ist es aber notwendig zu wissen, wie stark sich die Individuen innerhalb einer Art oder die Individuen zweier Arten voneinander unterscheiden, um ihre Populationsdynamik zu verstehen und letztlich zu lenken. Besteht die Population eines Areals weitgehend aus Individuen, die hier geboren wurden oder aus Immigranten von anderen, unterscheidbaren Populationen? Wo genau endet die Verbreitung einer bestimmten Art und wo beginnt die Verbreitung einer ähnlichen, nah verwandten Art? Fälle wie diese lassen sich nur untersuchen, wenn man in der Lage ist festzustellen, wer mit wem enger verwandt ist (und wer nicht). Unsere Fähigkeit dazu ist abhängig davon, wie groß die Unterschiede zwischen Individuen sein müssen, so dass wir sie noch voneinander unterscheiden können, um festzustellen, woher sie gekommen sind oder wer ihre Eltern waren. In der Vergangenheit war das häufig schwierig, oft auch unmöglich. Nach den damals verwendeten, einfachen optischen Merkmalen sahen alle Individuen einer Art gleich aus. Sogar Individuen verschiedener Arten konnten oft Abb. 8.1 Verbreitung von zwei Verwandtschaftsgruppen des Indischen Elefanten Ele­ phas maximus, die sich nur mit Hilfe molekularer Marker voneinander unterscheiden lassen. Die Gruppen kommen in vielen Gegenden zusammen vor, sie sind aber so verschieden, dass ihre Populationsdynamik selbst dort, wo sie koexistieren, vermutlich unabhängig voneinander ist (nach Fleischer et al. 2001) Man muss wissen, wer mit wem enger verwandt ist 300 Teil 3 Individuen, Populationen, Gemeinschaften und Ökosysteme nur von erfahrenen Taxonomen unter dem Mikroskop unterschieden werden, z. B. anhand der männlichen Genitalien. Heutzutage können wir mit genetischen, molekularen Markern zwischen Populationen und sogar zwischen Individuen unterscheiden und sind daher viel besser in der Lage, solche Fragen zu untersuchen. Allerdings benötigen wir auch dazu Experten und eine aufwendige Ausrüstung. In Fenster 8.1 stellen wir einige der wichtigsten dieser molekularen Marker vor und zeigen, wofür sie verwendet werden. 8.1 Quantitative Aspekte M o l ek u l a r e M a r ke r Dies ist zwar nicht der geeignete Ort für einen CrashKurs in Molekularer Biologie oder den Methoden, mit denen im Labor molekulare Marker extrahiert, amplifiziert, aufgetrennt und analysiert werden. Trotzdem ist es nicht schlecht, zumindest eine gewisse Ahnung von den Eigenschaften dieser Marker zu haben und einige der technischen Begriffe und Abkürzungen zu kennen. Die meisten ökologischen Studien der letzten Zeit haben auf die eine oder andere Art DNA für molekulare Identifizierungen genutzt. Daher sollten wir zumindest wissen, dass ein DNA-Strang durch die Reihenfolge der Basen Adenin (A), Cytosin (C), Guanin (G) und Thymin (T) charakterisiert ist, aus denen er zusammengesetzt ist. In einem DNA-Doppelstrang bilden diese Basen immer die gleichen, einander gegenüberliegenden Paare aus A-T oder G-C. D ie A u s w a h l de s g eei g n e t e n M a r ke r s Die Verwendung von molekularen Markern in der Ökologie basiert auf der Tatsache, dass sich Individuen mehr oder weniger gut anhand von molekularen Variationen zwischen ihnen trennen lassen. Der Grund für diese Unterschiede sind letztlich Mutationen in der Reihenfolge der Basenpaare, die unabhängig davon auftreten, welche Folgen sie für den betreffenden Organismus haben. Was mit der Mutation passiert und dem Organismus, welcher sie trägt, hängt von dem Gleichgewicht zwischen Selektion und genetischer Drift ab, worunter man zufällige, ungerichtete Veränderungen der Genfrequenz von einer Generation zur nächsten versteht. Liegt die Mutation in einem wichtigen Bereich der DNA, welcher beispielsweise den entscheidenden Teil eines Enzyms kodiert, dann wird das Ergebnis wahrscheinlich durch die Selektion bestimmt. Eine ungünstige Mutation, und die meisten Mutationen in wichtigen DNA-Regionen sind ungünstig, wird sehr schnell verschwinden, weil der mutierte Organismus weniger fit ist als seine Artgenossen. Individuen einer Population unterscheiden sich in diesen Regionen daher nur wenig, und wenn doch, dann stellt der Unterschied wahrscheinlich eine adaptive Variation dar. Aber es gibt auch DNA-Regionen, die offenbar keine wichtigen Stellen eines Enzyms kodieren oder irgendeine andere Funktion haben, bei der die exakte Sequenz von Bedeutung ist. Variationen, welche in diesen Bereichen auftreten, werden daher als „neutral“ bezeichnet und im Laufe der Zeit können sich Mutationen in diesen Regionen anhäufen. Stellen wir uns zwei Nachkommen aus einer einzigen Paarung vor. Beide werden genetisch sehr ähnlich sein. Aber stellen wir uns weiter vor, dass jeder der Beiden sich weiter fortpflanzt und viele Generationen von Nachkommen hat. Mit jeder Generation häufen sich Mutationen an und die Nachkommen der Beiden werden sich immer stärker in solchen Regionen des Genoms unterscheiden, in denen Variationen neutral sind. Bei einer Momentaufnahme in der Zukunft sollten wir bestimmen können, welche Gruppen sich schon länger aufgetrennt haben und welche Gruppen fast noch nicht. Ob wir das können, hängt allerdings davon ab, wie schnell die DNA in der betreffenden Region mutiert: Geschieht es zu langsam, werden sich die Individuen kaum unterscheiden. Geschieht es zu Kapitel 8 Evolutionsökologie schnell, wird jedes Individuum einzigartig sein und es wird schwer, die Verwandtschaftsbeziehungen zu anderen zu bestimmen. Molekulare Marker sollten deswegen idealerweise so gewählt werden, dass die Mutationsrate zur Fragestellung passt. Um Wüstenrennmäuse zu differenzieren, die in verschiedenen Bauten leben, aber zur selben, lokalen Population gehören, sollte eine DNA-Region mit hoher Mutationsrate gewählt werden, mit großen Unterschieden zwischen aufeinanderfolgenden Generationen. Für eine Studie zu den Routen der Wiederbesiedelung von Europa durch den Braunbären in den letzten 10 000-12 000 Jahren seit dem Ende der letzten Eiszeit, welche zu den verschiedenen Populationen geführt haben, sollte eine Region mit relativ geringer Mutationsrate gewählt werden. P o ly me r a s e - K e t t e n r eak t i o n ( P o ly me r a s e c h ai n r ea c t i o n , P CR ) Die meisten molekular-ökologischen Arbeiten, in denen DNA aus Organismen extrahiert wird, nutzen die Polymerase-Kettenreaktion (PCR) um die Menge des untersuchten genetischen Materials zu vermehren und genügend Material für Analysen zur Verfügung zu haben. Da auf diese Weise sehr kleine Proben ausreichen, können Individuen auch nicht-invasiv untersucht werden, durch Proben aus Blut, Haaren, Kot oder einer Feder. Einfach dargestellt benötigt man für eine PCR sogenannte „Primer“, die an die DNA-Sequenz passen, welche man amplifizieren möchte. Bei der PCR-Reaktion, welche heutzutage komplett automatisiert abläuft, wird die ursprünglich doppelsträngige DNA zu Einzelsträngen denaturiert. Die Primer binden sich an diese einzelnen Stränge (man spricht von annealing) und ein Enzym, die DNA-Polymerase, kopiert die Sequenz zwischen den Primern. Dieser Ablauf wird 30- bis 40-mal wiederholt. Diese wiederholte Amplifizierung hat einen exponentiellen Verlauf. Daher entsteht aus einer ursprünglich nur sehr kleinen Menge an Ziel-DNA, welche sich inmitten von anderen, unerwünschten Sequenzen befindet, eine so große Menge, dass sie analysiert werden kann. Vorraussetzung ist es allerdings, dass nicht nur eine informative Zielregion der betreffenden DNA identifiziert wurde, sondern dass auch Primer vorliegen, die dazu passen. 301 K e r n - D N A u n d mi t o c h o n d r ia l e D N A Besonders in der Vergangenheit verwendeten viele Studien keine DNA aus dem Zellkern (welche in der Regel zu gleichen Teilen von den Eltern stammt und die Informationen für die überwiegende Mehrheit der Körperfunktionen eines Organismus enthält), sondern die relativ kurzen Stücke mitochondrialer DNA (mtDNA), die sich in den Mitochondrien im Zytoplasma jeder Zelle befinden. Der Hauptvorteil der mtDNA besteht darin, dass sie fast immer von der Mutter vererbt wird, von der ja das Zytoplasma des Eies stammt, und dass sie daher nicht der Rekombination unterworfen ist. Verwandtschaftslinien können deshalb leichter von Generation zu Generation verfolgt werden und außerdem ist die Mutationsrate höher als bei wichtigen, kodierenden Regionen der Kern-DNA, so dass eine feinere Differenzierung möglich ist. Auf der anderen Seite bietet mtDNA nur eine geringe Zahl an Zielregionen (Targets) für Primer, und da mtRNA nur von der Mutter vererbt wird, ist es unmöglich, die beiden Eltern eines Individuums in einer Population zu bestimmen. Daher werden heutzutage zusammen mit der mtDNA zunehmend auch Regionen der Kern-DNA verwendet und somit die Vorteile von beiden kombiniert. M ik r o s at e l l i t e n Innerhalb der Kern-DNA verwenden Molekularbiologen aber nicht nur Sequenzen (bzw. Gene), die für Proteine kodieren. Mikrosatelliten sind DNARegionen, in denen sich die gleichen drei oder vier Basen viele Male wiederholen. Vor und nach diesen Regionen befinden sich flankierende Bereiche, die für jeden Mikrosatelliten charakteristisch sind (Abb. 8.2a). Allele unterscheiden sich nun in der Anzahl der Wiederholungen und daher in der Länge der Mikrosatelliten-DNA. Mikrosatelliten von verschiedener Länge bewegen sich unterschiedlich schnell durch ein Gel (eine gallertartige Matrix), an dem Spannung angelegt wird, und können daher aufgetrennt werden (Elektrophorese). Innerhalb einer Population können Mikrosatelliten sehr verschieden sein. Durch Auswahl einer Reihe geeigneter Mikrosatelliten kann daher jedes Individuum einer Population eindeutig identifiziert werden (genetischer Fingerabdruck). Mikrosatelliten sind also besonders für eine Unterscheidung auf sehr feinem Niveau geeignet. 302 Teil 3 Individuen, Populationen, Gemeinschaften und Ökosysteme Se q u e n z ie r u n g Wurde die Zielregion der Kern- oder mitochondrialen DNA von vielen Individuen ausgewählt, extrahiert und amplifiziert, so benötigt man nun etwas, mit dem man die einzelnen Individuen voneinander unterschieden kann um beispielsweise festzustellen, wer mit wem enger verwandt ist. Angesichts der fortschreitenden Automatisierung der Analysen und den sinkenden Kosten wird dafür zunehmend die gesamte Gensequenz bestimmt. Wie oben erwähnt, sind verschiedene Regionen im selben Gen für die Funktion des Gens unterschiedlich wichtig (Abb. 8.2b). Manche Regionen sind bei allen Individuen, in allen Populationen oder oft sogar innerhalb derselben Art immer gleich. Diese Regionen werden als „konserviert“ (conserved) bezeichnet. Diese Regionen sind (vermutlich) für die Funktion des Gens am wichtigsten und spielen für eine Unterscheidung keine Rolle. Aber es gibt andere Regionen, in denen viel mehr Variation zu beobachten ist. Bei diesen Regionen ist anzunehmen, dass sie für die Funktion keine Rolle spielen (sie sind neutral) oder jedenfalls nur einer sehr schwachen Selektion ausgesetzt sind. Diese Regionen können verwendet werden, um Individuen und Populationen zu unterscheiden. a Re s t r ik t i o n s - F r a g me n t - L ä n g e n P o ly m o r p h i s m u s ( R F L P ) In der Vergangenheit wurden häufig bestimmte Enzyme, sogenannte Restriktionsendonukleasen verwendet, die bestimmte DNA erkennen und dort zerschneiden. Ein DNA-Strang wird auf diese Weise in Fragmente zerlegt. Individuen unterscheiden sich aufgrund von weitgehend neutralen Mutationen in der Lage dieser Stellen und daher auch in der Länge der DNA-Fragmente, welche durch den Einsatz der Restriktionsendonukleasen entstehen. Die DNAFragmente bei verschiedenen Individuen lassen sich nun wieder mit Hilfe der Elektrophorese vergleichen. Diese Variation ist als Restriktions-Fragment-LängenPolymorphismus (RFLP) bekannt. Da verschiedene Restriktionsendonukleasen die DNA an unterschiedlichen Erkennungssequenzen zerschneiden, hat jede Restriktionsendonuklease ihre eigenen Polymorphismen. Proben können nun mit einer ganzen Reihe dieser Enzyme behandelt werden, und die Individuen, welche am unterschiedlichsten sind, werden sich in der größten Anzahl an RFLPs unterscheiden. Der Nachteil dieser Methode besteht natürlich darin, dass sie nur einen sehr geringen Teil der vorhandenen Variation einer Sequenz verwendet. Abb. 8.2 a Der Begriff „Locus“ bezeichnet hier die Lage einer Region auf der gesamten DNASequenz. Ein „Allel“ ist die spezifische Variante der Sequenz, welche in einem bestimmten Flankierende Region Mikrosatellit Flankierende Region Fall an diesem Locus vorliegt. Erinnern wir uns, dass eine Sequenz aus zwei DNA-StränAllel mit acht Wiederholungen gen besteht, die durch Basenpaare verbunden sind: G ist immer mit C, und A ist immer mit T b gepaart. Die Abbildung zeigt zwei verschiedene Individuum 1 Allele an einem Locus eines Mikrosatelliten. Individuum 2 Individuum 3 Die Sequenz der sich wiederholenden Basen Individuum 4 ist unterschiedlich lang (rot), die flankierenden Individuum 5 Regionen sind dagegen identisch (schwarz). b Im Gegensatz dazu zeigt diese Abbildung die Basensequenz nur eines der beiden DNA-Stränge eines hypothetischen Gens (z. B. eines Gens, welches für ein Protein kodiert) bei fünf verschiedenen Individuen. Beachten Sie den Unterschied zwischen den identischen, konservierten Regionen an den beiden Enden (schwarz) und den variablen Regionen in der Mitte (rot). Die Unterscheidung zwischen Individuen muss anhand dieser variablen Region erfolgen Allel mit zehn Wiederholungen Kapitel 8 Evolutionsökologie 8.2.1 303 Unterschiede innerhalb einer Art Albatrosse, diese Weltenwanderer mit der größten Flügelspannweite aller heute lebenden Vogelarten, sind legendär und kommen in vielen Gedichten und Geschichten vor. Trotzdem wurden 2008 in der Roten Liste der IUCN (International Union for Conservation of Nature) 4 der bekannten 22 Albatrosarten als vom Aussterben bedroht eingestuft, 6 Arten sind stark gefährdet, 8 Arten sind gefährdet und nur 4 Arten sind lediglich auf der Vorwarnliste und gelten als gering gefährdet. Der Schwarzbrauenalbatros wurde kürzlich von Taxonomen in zwei Arten aufgetrennt: Den Campbell-Albatros Thalassarche impavida, der nur auf der Campbell-Insel zwischen Neuseeland und der Antarktis vorkommt, und Thalassarche melanophris, der in anderen Gegenden der Antarktis brütet, unter anderem auf den Falklandinseln, auf Südgeorgien und in Chile (Abb. 8.3a). Der Graukopfalbatros Thalassarche chrysostoma ist ungefähr genauso groß und brütet ebenfalls auf einer Reihe subantarktischer Inseln, einschließlich Südgeorgien. Die beiden Schwarzbrauenalbatrosarten sind meist im Bereich des Festlandsockels zu finden, während die Nahrungsgebiete der Graukopfalbatrosse sehr viel mehr „ozeanisch“ sind. Von beiden Arten wird angenommen, dass sie brutortstreu sind, und, wie die anderen Albatrosarten auch, zum Brüten in die Nähe ihres eigenen Geburtsortes zurückkehren. Angesichts der an allen Brutstätten Jahr für Jahr abnehmenden Individuenzahlen stellen sich nun die Fragen, ob diese einzelnen Populationen miteinander in Verbindung stehen oder getrennt sind und ob Schutzbemühungen die ganze Art umfassen sollten oder sich eher auf bestimmte Brutpopulationen beschränken sollten. Diese Fragen wurden für beide Arten anhand von mtDNA-Sequenzen und einer Gruppe von 7 Mikrosatelliten untersucht (Burg und Croxhall 2001). Die Ergebnisse der mtDNA-Analysen waren besonders eindeutig (Abb. 8.3b,c) aber auch die Daten der Mikrosatelliten ergaben das gleiche Bild. Für die Schwarzbrauenalbatrosse bestätigen die Daten die jüngste Abtrennung von T. impavida als eigene Art. Es zeigte sich aber auch, dass sich die beiden Arten auf den Campbell-Inseln miteinander paaren und Hybride produzieren. Überraschenderweise zeigten die Daten aber auch, dass T. melanophris auf den Falklandinseln eine Population bildet, die deutlich getrennt ist von einer Population auf Diego Ramirez (Chile), Südgeorgien und den Kerguelen, die trotz der Brutortstreue zu allen drei Standorten praktisch einheitlich ist. Im Unterschied dazu scheinen die Graukopfalbatrosse aller fünf Standorte mit ihrer größeren Flugreichweite eine einzige Brutpopulation zu bilden (Abb. 8.3c), auch hier obwohl sie brutortstreu sind. Aus der Sicht des Artenschutzes allerdings betrifft die wichtigste Schlussfolgerung der Studie die Art T. melanophris. Bislang wurde die Population auf den Falklandinseln aufgrund ihrer Größe und Stabilität als Versicherung gegen das Aussterben der Art betrachtet. Nun zeigen die molekularen Daten, dass die Falklandpopulation und die restlichen Populationen der Art nicht zusammen gehören und die Letzteren dadurch offenbar viel stärker vom Aussterben bedroht sind als vorher angenommen. (In Fenster 8.2 sehen wir, dass molekulare Marker in praktischen Fragen des Naturschutzes eine noch direktere Rolle spielen können.) Albatrosse Molekulare Marker im Artenschutz 304 Teil 3 Individuen, Populationen, Gemeinschaften und Ökosysteme a Campbell Inseln Antarktis Diego Ramirez Kerguelen Falklandinseln Südgeorgien Marion Inseln b T. melanophris (Falkland) DR E FI FI FI FI FI FI FI T. impavida FI F G mC BI BI mC BI BI A mC K BI iC D mC K BI C DR T. melanophris (Diego/Südgeorgien/Kerguelen) DR iC C iC mC DR M C DR K K M DR BI K C DR C BI iC BI M M K B M iC DR C C K M iC BI B mC iC iC BI K BI iC BI H M iC iC iC mC iC BI BI K DR A DR BI K DR DR FI FI M DR BI K FI FI mC c FI M M M BI Kapitel 8 Evolutionsökologie 305 Abb. 8.3a–c Differenzierung der Populationen der Schwarzbrauenalbatrosse Thalassarche impavida und T. melano­ phris sowie des Graukopfalbatros T. chrysostoma. a Standorte in der Sub-Antarktis, an denen Proben genommen wurden. b Die Verwandtschaftsbeziehungen von 73 Schwarzbrauenalbatrossen, basierend auf einer variablen Region der mtDNASequenz. Individuen des gleichen Standortes mit der exakt gleichen Sequenz sind mit einem Buchstaben bezeichnet (A, B, usw.) und in einem Oval dargestellt, dessen Größe der Anzahl der Individuen entspricht. Individuen mit einzigartiger Sequenz, die in keine dieser Gruppen fallen, sind folgendermaßen bezeichnet: BI (Südgeorgien), DR (Diego Ramirez, Chile), FL (Falklandinseln), K (Kerguelen), alle T. melanophris. mC (T. melanophris von den Campbell-Inseln), iC (T. impavida von den Campbell-Inseln). Die Kreuzschraffur auf den Verbindungslinien bezeichnet die Anzahl der Unterschiede zwischen den Individuen oder Gruppen. Die Proben fallen in drei Gruppen: T. impavida und T. melano­ phris von den Falklandinseln und T. chrysostoma von allen anderen Standorten. Beachten Sie jedoch, dass die Zuordnung zu den Gruppen, ebenso wie zu den Populationen, nicht perfekt ist. Darüber hinaus wurden einige Individuen von T. melanophris als Hybride von T. melanophris und T. impavida identifiziert. c Die Verwandtschaftsbeziehungen von 50 Graukopfalbatrossen, basierend auf einer variablen Region der mtDNA-Sequenz. Die Kodierung ist dieselbe wie in b mit zwei Ausnahmen: M steht für Marion-Inseln und C für Campbell-Inseln. In diesem Fall sind keine separierbaren Gruppen erkennbar (nach Burg und Croxall 2001) 8.2 Aktueller ÖKOnflikt F o r e n s i s c h e A n a ly s e de r He r k u n f t u n s e r e r Na h r u n g Wie wir in Kap. 12 noch ausführlicher diskutieren werden, gibt es immer häufiger Konflikte darüber, ob natürliche Populationen mancher Arten als Nahrungsquelle ausgebeutet oder eher geschützt werden sollen, entweder aus ethischen Gründen oder damit auch zukünftige Generationen noch etwas zu essen haben. In Kanada werden pazifische Lachsarten sowohl kommerziell (von der Industrie) als auch von Sportfischern intensiv ausgebeutet. Jeder der Nutzer hat dabei seine eigene Strategie, um sicherzustellen, dass auch in Zukunft noch Fische gefangen werden können. Wenn bestimmte Arten gut verfügbar sind, kann beispielsweise der Fang auf andere Fischarten vollständig eingestellt werden, so dass sie sich wieder fortpflanzen und ihre Bestände sich erholen können. Trotzdem ist die Nachhaltigkeit dieser Aktivitäten stark bedroht: Im Jahre 2002 wurde mit dem CohoLachs (Silberlachs, Oncorhynchus kisutch) aus dem Oberlauf des Fraser Rivers das erste Mal die Population einer Kanadischen Lachsart als „stark gefährdet“ eingestuft. Und viele andere Populationen benötigen ebenfalls Schutz. In einer idealen Welt wären politische Maßnahmen und damit das Management verschiedener Fischpopulation vollkommen effektiv. In der Realität aber kommt es zu illegaler Fischerei und es ist schwer, die Übeltäter auf frischer Tat zu ertap- pen. Eine alternative Methode oder zumindest eine weitere Strategie im Arsenal der Schutzbehörden besteht darin, illegal gefangene Fische an einer anderen Stelle in der Nahrungsmittelkette vom Fang zum Verbraucher zu identifizieren. Diese Strategie beruht auf molekularen Markern. Die zehn pazifischen Lachsarten der Gattung Oncorhynchus können durch eine RFLP-Analyse der Kern-DNA voneinander unterschieden werden (Withler et al. 2004). Die Ergebnisse einiger Untersuchungen im Fall des illegalen Besitzes von Lachsen sind in Tabelle 8.1 dargestellt. Im zweiten dargestellten Fall zeigte ein unzufriedener Küchenchef den Besitzer eines Restaurants bei den Behörden an. Einer der Fische wurde als Silberlachs identifiziert. Da das Tier offenbar nicht tiefgefroren war, konnte es nicht aus dem Vorjahr stammen, in dem der Fang noch legal war. Der Restaurantbesitzer erhielt seine gerechte Strafe. Mit Hilfe von Mikrosatelliten ist es möglich, sehr geringe Unterschiede zwischen Proben festzustellen, so dass man nicht nur zwischen Fischarten, sondern innerhalb einer Fischart mit einer gewissen Wahrscheinlichkeit sogar zwischen bestimmten Flüssen unterscheiden kann. Die Ergebnisse einiger solcher Untersuchungen sind in Tabelle 8.2 dargestellt. Im zweiten Fall wurde in 50 Fischdosen Rotlachs 306 Teil 3 Individuen, Populationen, Gemeinschaften und Ökosysteme gefunden, der illegal im Fraser River gefangen worden war. Der Angeklagte wurde zu 15 000 US-Dollar Strafe verurteilt. Bei ihm wurden 100 000 Fischdosen mit Lachs gefunden, im Verkaufswert von 300 000 bis 400 000 US-Dollar. Was halten Sie von der Höhe der verhängten Strafen? Wie schwerwiegend ist eine solche Straftat im Vergleich zu anderen Straftaten, wie Straßenraub oder dem Besitz von Drogen zum persönlichen Gebrauch? Sollte die Höhe der Strafe im Verhältnis zum ökonomischen Schaden stehen, den die betreffenden Fischereibetriebe erleiden oder sollte die Strafe eher ein Signal an all diejenigen darstellen, die sich weigern anzuerkennen, dass gefährdete Populationen geschont werden müssen, damit auch zukünftige Generationen noch etwas von ihnen haben? Tabelle 8.1 Artbestimmung von Lachsen anhand von biologischen Proben. Die Proben wurden von kanadischen Fischerei­ behörden aufgrund des Verdachtes genommen, dass die Fische aus illegalen Quellen stammen könnten Fall (Jahr) Untersuchte Gewebeart Identifizierte Art Ergebnis der Strafverfolgung Strafe ($) 1 (1995) Blut, Schuppen, Schleim aus dem Behälter Coho-Lachs Verurteilung 1 500 2 (1996) Muskulatur Keta-Lachs Verurteilung 1 800 Königslachs Coho-Lachs 3 (1998) Muskulatur Coho-Lachs Verurteilung ? 4 (1999) Muskulatur Atlantischer Lachs Keine Anklage – Königslachs Coho-Lachs 5 (2000) Muskulatur Coho-Lachs Schuldgeständnis 7 500 6 (2000) Muskulatur Rotlachs Verurteilung 1 000 (nach Wither et al. 2004) Tabelle 8.2 Bestimmung von Lachsen anhand von biologischen Proben. Die Proben wurden von Fischereibehörden aus den gleichen Gründen genommen wie in Tabelle 8.1. IF&T bezieht sich auf die Zuflüsse zum Oberlauf des Fraser (Interior Fraser) und Thompson Rivers Fall (Jahr) Identifizierte Lachsart Herkunft der Fische Ergebnis der Strafverfolgung Strafe ($) 1 (1998) Rotlachs 96,5% Fraser, 96,5% IF & T Schuldgeständnis 2 000 2 (1999) Rotlachs 100% Fraser, 100% IF & T Verurteilung 15 000 3 (1999) Königslachs 91,4% Fraser Keine Verurteilung, in Berufung 4 (2000) Rotlachs 100% Fraser, 100% IF & T Verurteilung 8 000 5 (2001) Rotlachs 97,8% Fraser, 97,8% IF & T Verurteilung 3 000 (nach Wither et al. 2004) http://www.springer.com/978-3-540-95896-3