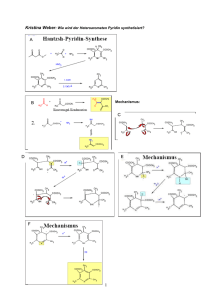
Kohlenhydrate
Werbung

Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Einleitung Kohlenhydrate Kohlenhydrate ist die Sammelbezeichnung für die als Naturstoffe sehr verbreiteten Polyhydroxyaldehyde (Aldosen) und Polyhydroxyketone (Ketosen) sowie höhermolekulare Verbindungen, die sich durch Hydrolyse in solche Verbindungen überführen lassen. Die Kohlenhydrate haben meist die Bruttoformel C n(H2O)n und lassen sich somit formal als „Hydrate des Kohlenstoffs“ auffassen. Diese Bezeichnung wurde beibehalten, obwohl sie bei einigen Kohlenhydraten nicht mehr berechtigt ist und man Kohlenhydrate kennt, die außer Kohlenstoff, Wasserstoff und Sauerstoff noch Stickstoff (z.B. Aminozucker) oder Schwefel enthalten. Die monomeren Aldosen oder Ketosen nennt man Monosaccharide, ihre Dimeren bis Decameren Oligosaccharide (Disaccharide, Trisaccharide usw.) und die makromolekularen Polysaccharide. Häufig werden die Mono- u. Oligosaccharide als „Zucker“ zusammengefaßt u. den Polysacchariden gegenübergestellt. Dies hat eine gewisse Berechtigung, denn die niedermolekularen Kohlenhydrate sind süßschmeckende, wasserlösliche, meist kristalline Verbindungen. Bei den Monosacchariden leitet sich die Benennung als Triosen, Tetraosen, Pentosen, Hexosen von der Anzahl der im Molekül vorhandenen Kohlenstoff-Atome ab. In den Oligosacchariden sind 2–10 Monosaccharid-Moleküle durch glykosidische Bindungen zu größeren Molekülen vereinigt. Die einfachsten Oligosaccharide sind die Disaccharide, von denen drei frei vorkommen, die von großer Bedeutung sind, nämlich Saccharose (Rohrzucker, Rübenzucker), Lactose (Milchzucker) und Maltose (Malzzucker). Von besonderer Bedeutung sind Maltose und Cellobiose, die bei der Hydrolyse von Stärke bzw. Cellulose entstehen. Die Bindung zwischen den Monosacchariden ist O-glykosidisch, doch kann daran nur eine der beiden Halbacetal-HydroxyGruppen beteiligt sein. Tatsächlich besitzen viele Disaccharide (z.B. die Maltose) reduzierende Eigenschaften wie die Monosaccharide, was bedeutet, daß einer der Zuckerreste Halbacetal-Form besitzt. Sind die beiden Monosaccharide jedoch über ihre anomeren C-Atome gekoppelt (z.B. im Rohrzucker), so ähnelt das Disaccharid einem Acetal, wirkt deshalb nicht reduzierend. Analog den Disacchariden sind auch die übrigen Oligosaccharide aufgebaut. Die Polysaccharide lassen sich in zwei Gruppen gliedern, nämlich in die als Gerüststoffe für Pflanzen und einige Tiere dienenden Polysaccharide und in die Reservestoffe, die bei Bedarf im Organismus durch bestimmte Enzyme in Freiheit gesetzt werden. Beide Gruppen sind hochmolekulare Polymere, die oft nur aus einer einzigen Pentose oder Hexose aufgebaut sind. Im Gegensatz zu solchen Homoglykanen (Beisp.: Cellulose, Stärke, Glykogen) sind die Heteroglykane aus verschiedenartigen Monosacchariden aufgebaut. Die Kohlenhydrate spielen in der Natur eine vielfältige Rolle. Sie werden von den Pflanzen in ungeheuren Mengen durch Photosynthese produziert. Daneben werden sie auch im tierischen Organismus nicht nur als Reservestoff (Glykogen) benötigt, sondern sind unentbehrlich als Bestandteile der Nucleinsäuren, der Glykolipide, Glykoproteine, Glykosphingoside u. dgl. – in der Immunologie schreibt man den Kohlenhydrat-Anteilen wichtige Erkennungs-Funktionen bei AntigenAntikörper-Reaktionen zu. Glykolyse Hierunter versteht man den wichtigsten Abbauweg der D-Glucose zu 2 Mol Pyruvat (sh. Abb.). Dieser katabolische Stoffwechselweg liefert chemische Energie in Form von Adenosin-5'-triphosphat (ATP). Die gleichfalls entstehende reduzierte Form des Nicotinamid-Adenin-Dinucleotids, das NADH, wird unter anaeroben Bedingungen wieder oxidiert unter begleitender Reduktion von Pyruvat zu Lactat (Salzform der Milchsäure). RG (vereinfacht): Glu + 2ADP + 2Pi 2Lactat + 2ATP; H=-135,5 kJ/mol Seite 1 ATP CO2 ADP + Pi Pyruvatcarboxylase (EC 6.4.1.1) ATP Pyruvat Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Abbildung 1: Schema der Glykolyse Pentosephosphatweg Ein weiterer Weg des Zucker-Abbaus ist der Pentosephosphat-Weg, durch dessen sog. oxidativen Zweig unter Kohlendioxid-Eliminierung D-Ribulose-5-phosphat und NADPH aus D-Glucose-6phosphat gebildet werden. D-Ribulose-5-phosphat wird – nach Isomerisierung zu D-Ribose-5phosphat für die Biosynthese von Nucleotiden und Nucleinsäuren, NADPH dagegen für reduktive Biosynthesen benötigt (z.B. der Fettsäuren; daher PP-Weg in Fettgewebe bes. akt.). Ist aufgrund der aktuellen Stoffwechsel-Situation der Bedarf an NADPH größer als der an Pentosephosphat, so können jeweils 6 Mol. des überschüssigen D-Ribulose-5-phosphat zu 5 Mol. DGlucose-6-phosphat rekombinieren (nichtoxidativer Zweig des PP-Wegs). Bei 6maligem Durchlaufen dieses Zyklus wird 1 Mol. D-Glucose-6-phosphat vollständig zu Kohlendioxid und anorganischem Phosphat abgebaut. RG (vereinfacht): 3G6P 2F6P + GAP Seite 2 Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Gluconeogenese Enzymgesteuerte Neubildung von Kohlenhydraten im Organismus durch Umkehr der Glykolyse, in einigen Reaktionsschritten jedoch von dieser abweichend (sh. Abb.). Die Gluconeogenese verbraucht chemische Energie in Form von Adenosin-5'-triphosphat (ATP). Sie dient der Aufrechterhaltung des Blutzucker-Spiegels bei Erschöpfung der Kohlenhydrat-Reserven und bewirkt in Leber und Niere die Umwandlung von Pyruvat, Lactat, Glycerin oder von bestimmten Aminosäuren zu D-Glucose. RG (vereinfacht): 2Pyruvat + 6ATP Glu + 2Pi Versuche Freie Enthalpie G0‘ der GAPDH-Reaktion Bei diesem Versuch geht man von F-1,6-BP aus, versetzt dieses mit einem physiologischen Puffer (Tris/HCl pH 8,2), Wasser und NAD+-Lösung, mischt und mißt im Photometer die Extinktion E0. Anschließend wird ein Enzymgemisch aus Aldolase und GAPDH hinzugefügt, welches F-1,6-BP in GA-3-P und DHAP in jeweils gleichen Mengen spaltet. Durch geringe Mengen Phosphat in F-1,6-BP wird ein extinktionsanstieg gemessen. Durch wiederholte Zugabe von Phosphat unterschiedlicher Konzentration (0,05M, 0,25M, 0,5M) reagieren nun GA-3-P und NAD+ zu 1,3-BPG und NADH+H+, je zu gleichen Teilen. Nach jeder Phosphatzugabe wird die Extinktion bis zur GG-Einstellung gemessen. Die Extinktion wird durch bei der GAPDH-Reaktion entstehendes NADH verursacht, welches einen charakteristischen Extinktionskoefffizienten bei 340nm hat. Nach der letzten Phosphatzugabe und GG-Einstellung wird TIM-Lösung dazugegeben, damit sich auch ein GG zwischen GA-3-P und DHAP einstellen kann. Auswertung Schaubild 1 E0 = 0,160 E1 = 0,250 E2 = 0,350 E3 = 0,530 E4 = 0,730 E5 = 0,660 nach Zugabe von 20 l Enzymgemisch nach Zugabe von 20 l 0,05M Phosphat nach Zugabe von 20 l 0,25M Phosphat nach Zugabe von 20 l 0,5M Phosphat nach Zugabe von 20 l TIM-Lösung Berechnung der NADH- und 1,3-BPG-Konzentration 1,3-BPG und NADH entstehen im Verhältnis 1:1. Die Konzentration läßt sich nach dem LambertBeerschen Gesetz berechnen: E = c*d* c = E d * wobei E durch die Extinktionsmessung bekannt ist, d die Schichtdicke der Küvette = 1cm, und für NADH = 6,22 mol-1*cm-1 bei 340nm ist. Berechnung der NAD+-Konzentration n c= v Seite 3 KOHLENHYDRATE Biochemie-Protokoll Gruppe 1 14.05.16 Julia Gnann & Christoph Hartmann n = c*v = 25mM*100l = 2,5*10-6 mol vgesamt = 1960l (gemittelt aus den 3 Phosphatzugaben) cNAD+ = 2,5 *10 6 mol = 1,276*10-3 M 1960 l Statt der GG-Konzentrationen von F-1,6-BP, Phosphat und NAD+ darf man die Anfangskonzentrationen verwenden. Diese Vereinfachung darf vorgenommen werden, da nur ein kleiner Teil der Substrate umgesetzt wird und so der relative Fehler vernachlässigbar ist. Konzentration der F-1,6-BP n c= v n = c*v = 100mM*100l = 1*10-5 mol vgesamt = 1960l cF-1,6-BP = 1 *105 mol = 5,10*10-3 M 1960 l Konzentration der Protonen Die bei der Reaktion entstehenden Protonen werden durch Tris/Hcl abgepuffert, also entspricht die Konzentration dem pH des Puffers pH = 8,2 pH = -lg[H+] cH+ = 10-8,2 M Konzentration des GA-3-P Solange das GA-3-P und DHAP im Verhältnis 1:1 vorliegen, kann die Konzentration des GA-3-P direkt aus dem Aldolase-GG berechnet werden. Es gilt: F-1,6-BP DHAP + GA-3-P K’Aldolase = [ DHAP ] * [GA 3 P] [GA 3 P]2 = [ f 1,6 BP ] [ F 1,6 BP ] G0‘ = -R*T*lnK’Aldolase R = 8,314 J/mol*K T = 291K (18°C) G0‘ = 23,99kJ/mol (gegeben) lnK’Aldolase = G o ' R *T G 0 ' R *T K’Aldolase = e [GA-3-P] = K ' Aldolase*[F 1,6 BP ] = = e-9,915 = 4,94*10-5 M 4,94 *105 * 5,1 *103 M 2 = 5,02*10-4M cGA-3-P = 5,02*10-4M Berechnung der drei GG-Konstanten für die GAPDH-Reaktion Die GG-Konstante erhält man aus KGADPH = [1,3 BPG ] * [ NADH ] * [ H ] [GA 3 P] * [ Pi ] * [ NAD ] und mit pH7 als Standardzusatz: K’GADPH = KGADPH * 107 Daraus kann schließlich G0‘ berechnet werden. GG-Konstante nach der ersten Zugabe: 0,05M Phosphatlösung Seite 4 Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Berechnung der Phosphatkonzentration vgesamt = 1940l v = 20l c = 0,05M cP1 = 20 l * 0,05M v*c = = 5,15*10-4M 1940 l vgesamt Konzentration [1,3-BPG]*[NADH] E = *c*d , wobei E = E2-E0 = 0,190 cNADH = E 0,190 * mol * cm = = 3,05*10-5M * d 1cm * 6,22 *103 l Gleichgewichtskonstante [1,3 BPG ] * [ NADH ] * [ H ] KGADPH = [GA 3 P] * [ Pi ] * [ NAD ] 3,05 *10 5 * 3,05 *10 5 *10 8, 2 = = 1,779*10-8 5,02 *10 4 * 5,15 *10 4 *1,276 *10 3 K’GADPH = 107*1,779*10-8 = 1,779*10-1 GG-Konstante nach der zweiten Zugabe: 0,25M Phosphatlösung Berechnung der Phosphatkonzentration vgesamt = 1960l v = 20l c = 0,25M Da die bei der ersten Phosphatzugabe hinzugefügten Phosphate nicht aus der Lösung entfernt werden, muß die Konzentration mit berücksichtigt werden. n = n1+n2 = v1*c1+v2*c2 = 0,05M*20l+0,25M*20l = 6mol n cP2 = v gesamt = 6mol = 3,06*10-3M 1960l Konzentration [1,3-BPG]*[NADH] E = E3-E0 = 0,370 cNADH = E 0,370 * cm * mol = = 5,95*10-5M * d 6,22 *10 3 l *1cm Gleichgewichtskonstante KGADPH = = [1,3 BPG ] * [ NADH ] * [ H ] [GA 3 P] * [ Pi ] * [ NAD ] 5,95 *10 5 * 5,95 *10 5 *10 8, 2 = 1,14*10-8 5,02 *10 4 * 3,06 *10 3 *1,276 *10 3 K’GADPH = 1,14*10-8 * 107 = 1,14*10-1 GG-Konstante nach der dritten Zugabe: 0,5M Phosphatlösung Seite 5 KOHLENHYDRATE Biochemie-Protokoll Gruppe 1 14.05.16 Julia Gnann & Christoph Hartmann Berechnung der Phosphatkonzentration vgesamt = 1980l v = 20l c = 0,5M n = n1+n2+n3 = v1*c1+v2*c2+v3*c3 = 0,05M*20l+0,25M*20l+0,5M*20l = 16mol cP3 = n v gesamt = 16mol = 8,08*10-3M 1980l Konzentration [1,3-BPG]*[NADH] E = E4-E0 = 0,570 cNADH = E 0,570 * cm * mol = = 9,16*10-5M * d 6,22 *10 3 l *1cm Gleichgewichtskonstante KGADPH = = [1,3 BPG ] * [ NADH ] * [ H ] [GA 3 P] * [ Pi ] * [ NAD ] 9,16 *10 5 * 9,16 *10 5 *10 8, 2 = 1,02*10-8 5,02 *10 4 * 8,08 *10 3 *1,276 *10 3 K’GADPH = 1,02*10-8*107 = 1,02*10-1 Berechnung von G0‘ G = G0‘ + RT*lnK‘ Die Berechnung von G0‘ erfolgt bei GG-Zustand, da dort gilt: G=0 G0‘ = -RT*lnK‘ G0‘1 = -8,314 G0‘2 = -8,314 G0‘3 = -8,314 J mol*K J mol*K J mol*K *291K*ln1,779*10-1 = 4,177kJ/mol *291K*ln1,14*10-1 = 5,253kJ/mol *291K*ln1,02*10-1 = 5,522kJ/mol Lage des GG zwischen DHAP und GAP Man beobachtet im Verlauf des Versuches nach Zugabe verschiedener Phosphatmengen einen Anstieg der Extinktion von NADH bei 340nm. dies ist auf den Reaktionsschritt GA-3-P + NAD+ + Pi 1,3-BPG + NADH + H+ zurückzuführen. Nach der Zugabe der TIM sinkt die Extinktion (sh. a. Schaubild), was bedeutet, daß weniger NADH produziert wird, da weniger GA-3-P zur Verfügung steht. Das Gleichgewicht DHAP GA-3-P liegt also auf der Seite des DHAP (laut Lehrbuch 96%). Entkopplung der GAPDH-Reaktion Entkopplung mit Arsenat Der erste Teil dieses Versuchs entspricht dem Versuch 1, wobei jedoch statt dreimal Phosphat einmal Na-Arsenat-Lösung zugegeben wird. Seite 6 Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Auswertung (sh.a. Schaubild 2) Die Reaktion verläuft durch Entkopplung mit Arsenat sehr rasch und mit einer höheren Umsetzung ab. Das gemischte Säureanhydrid Phosphoglycerinarsenat zerfällt schnell in 3-Phosphoglycerat und Arsenat. Um das GG zu halten wird ständig neues Phosphoglycerinarsenat aus GA-3-P nachgeliefert, wobei aus NAD+ NADH+H+ entsteht Extinktionsanstieg Stillstand der Reaktion mit Arsenat Um zu bestimmen, wann die Reaktion mit Arsenat zum Stillstand kommt, muß die limitierende Komponente bestimmt werden. Das gemischte Säureanhydrid zerfällt bei der Hydrolyse, dabei wird Arsenat wieder frei, also kann Arsenat nicht limitierend sein. Die Anwesenheit von NAD+ dagegen ist limitierend, da es nicht reoxidiert wird. cNAD+ = 20l * 25M v*c = = 1,29*10-3M 1940l v gesamt E = *c*d ; d=1cm; =6,22*103 moll*cm E = 6,22*103 l mol*cm *1cm*1,29*10-3M = 8,024*10-3 Bei einer Extinktion von 8,024*10-3 würde die Reaktion zum Stillstand kommen. Vergiftung mit Hydroxymercuribenzoat und Entgiftung mit Mercaptan Die GAPDH wird vor dem Start der Enzymreaktion mit Hydroxymercuribenzoat vergiftet/inaktiviert; dann wird die Restaktivität mit Na-Arsenat getestet. Nach einigen Minuten wird die Vergiftung durch Zugabe von DTT rückgängig gemacht. Auswertung (sh. a. Schaubild 3) Durch Vergiftung mit Hydroxymercuribenzoat werden annähernd alle Enzyme inaktiviert die extinktion steigt nicht an. Nach Zugabe von DTT steigt die Extinktion wieder an, d.h. das aktive Zentrum des Enzyms wird wieder freigegeben, da Hydroxymercuribenzoat an DTT bindet. Allerdings werden nicht alle Enzyme freigegeben, weshalb die Kurve flacher verläuft als die der Entkopplung mit Arsenat. Vergiftung mit Jodacetamid Die GAPDH wird vor dem Start der Enzymreaktion zunächst einige Minuten mit Jodacetamid inkubiert. Danach wird die Restaktivität durch Zugabe von Na-Arsenat getestet. Zuletzt wird versucht, mit DTT zu entgiften. Auswertung (sh.a. Schaubild 4) Jodacetamid reagiert mit der SH-Gruppe des Cysteins im aktiven Zentrum. Es bildet sich eine stabile Thioätherverbindung, die nicht mehr durch überschüssiges DTT geöffnet werden kann. Dennoch kann beim Versuch eine Erhöhung der Enzymaktivität nach DTT-Zugabe gemessen werden. Dies liegt daran, daß Cystein-Reste im aktiven Zentrum vom Enzym unter Einwirkung von Luftsauerstoff Disulfidbrücken bilden, an denen Jodacetamid nicht angreifen kann, die aber durch DTT gespalten werden und so wieder freie Enzyme entstehen. Vergiftung mit Jodacetamid und Schutz vor Vergiftung durch die Substrate Die GAPDH wird vor dem Start der Enzymreaktion mit Jodacetamid vergiftet, jedoch einmal in Anwesenheit beider Substrate und je einmal in Anwesenheit eines der Substrate (GA-3-P bzw. NAD+). Die Restaktivität wird ebenfalls durch Zugabe von Na-Arsenat-Lösung getestet Seite 7 Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Auswertung (sh.a.Schaubild 4) In diesen Teilversuchen werden die Substrate vor dem Jodacetamid hinzugegeben. Die Substrate konkurrieren mit dem Gift um das aktive Zentrum des Enzyms, da die Substrate aber im Überschuß vorhanden sind, kann die Reaktion stattfinden. Der Substratschutz ist – wie man dem Schaubild entnehmen kann – äußerst effektiv. Beide Substrate schützen das Enzym besser als nur eines, wobei von diesen GA-3-P besser schützt als NAD+. In allen Versuchen werden wie in Versuch 1 die entsprechenden Extinktionen in Abhängigkeit von der Zeit gemessen (sh. a. Schaubilder). Glucosebestimmung im Blut Bestimmung im Schnelltest Die Glucosekonzentration wird mittels Strom durch Potentialänderung gemessen. Auf der Testelektrode befindet sich (laut Herstller) das Enzym Glucosedehydrogenase, welches Fe 3+ zu Fe2+ reduziert. Das führt zu einer Potentialänderung – dieser Strom wird von dem Gerät gemessen und es wird daraus auf die Glucosekonzentration geschlossen. Auswertung Glucosekonzentration vor dem Essen: 82mg/100ml Glucosekonzentration nach dem Essen: 110mg/100ml Bestimmung im optischen Test Die Serumprobe wird mit Perchlorsäure behandelt um störendes Eiweiß auszufällen, welches anschließend abzentrifugiert wird. Im Überstand befindet sich Glucose, welches mit Hexokinase und ATP zu G-6-P phosphoryliert wird. Dieses wird mit G-6-P-DH und NADP zu Gluconolacton-6-P und NADPH+H+. Die dabei entstehende Menge NADPH ist äquivalent zur Glucosemenge und wird bei 340nm durch Extinktionsänderung gemessen. Gluconolacton-6-P wird zu Gluconat-6-P hydrolysiert. Dabei liegt das GG auf der rechten Seite, d.h. die Reaktion läßt sich zur quantitativen Berechnung verwenden. Auswertung (sh.a. Schaubild 5) Leerwert: E1 = 0,277; E2 = 0,260 EL = -0,017 vor dem Essen: E1 = 0,284; E2 = 0,425 EV = 0,141 nach dem Essen: E1 = 0,304; E2 = 0,597 EN = 0,293 Standard: E1 = 0,284; E2 = 0,404 ES = 0,120 Für die Glucosekonzentration im Blut gilt: cBlut = (EProbe - ELeerwert)*33,5[mM] Standard: cS = (ES - EL)*33,5[mM] = (0,120 – (-0,017))*33,5[mM] = 4,5895mM vor dem Essen: cV = (EV - EL)*33,5[mM] = (0,141-(-0,017))*33,5[mM] = 5,293mM nach dem Essen: cN = (EN - EL)*33,5[mM] = (0,293-(-0,017))*33,5[mM] = 10,385mM Milligrammprozent mg% = mg/100ml MGGlucose = 180g/mol = 180mg/mmol Standard:c = cS*MGGlucose*10-1 = 4,5895mM*180mg/mmol*10-1 = 82mg% vor dem Essen: c = cV* MGGlucose*10-1 = 5,293*180mg/mmol*10-1 = 95mg% nach dem Essen: c = cN* MGGlucose*10-1 = 10,385*180mg/mmol*10-1 = 186mg% Seite 8 Biochemie-Protokoll Gruppe 1 KOHLENHYDRATE 14.05.16 Julia Gnann & Christoph Hartmann Abbildungsverzeichnis: Abbildung 1: CD-Römpp Chemielexikon; Thieme Quellen: CD-Römpp Chemielexikon; Thieme Abkürzungsverzeichnis Abkürzung Bedeutung 1,3-BPG F-1,6-BP GA-3-P GAPDH GG MG TIM 1,3-Bisphosphoglycerat Fructose-1,6,-Bisphposphat Glycerinaldehyd-3-Phosphat Glycerinaldehydphosphatdehydrogenase Chemisches Gleichgewicht Molekulargewicht Triosephosphatisomerase Seite 9