Status Projekt 2014 - Gilead Förderprogramm Infektiologie
Werbung
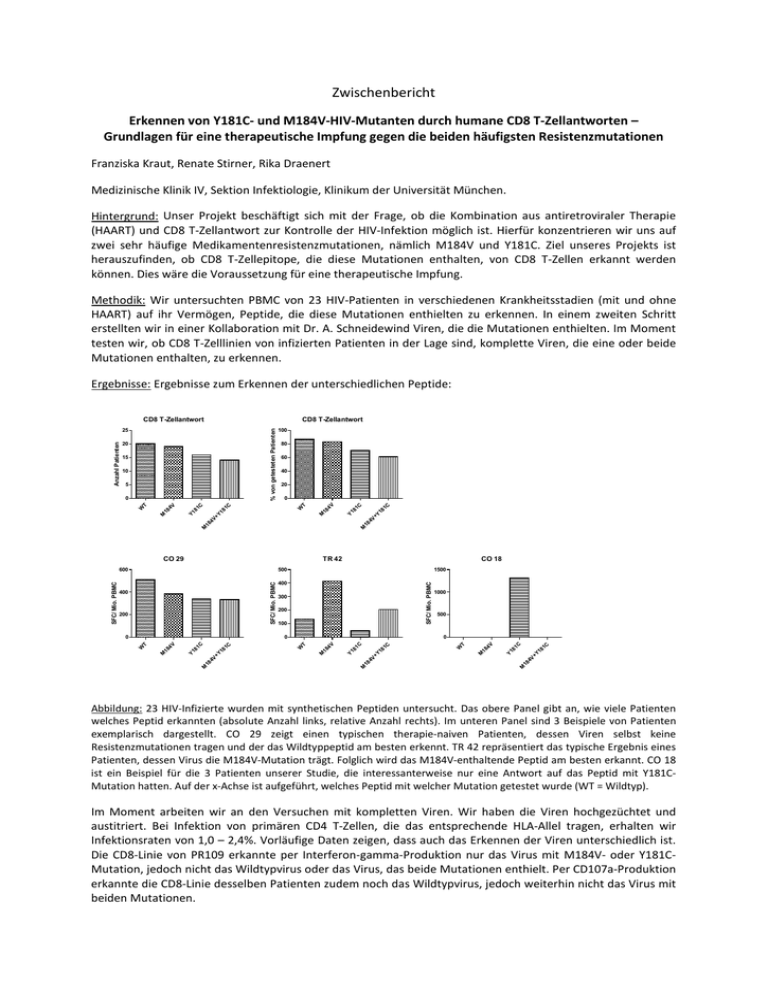
Zwischenbericht Erkennen von Y181C- und M184V-HIV-Mutanten durch humane CD8 T-Zellantworten – Grundlagen für eine therapeutische Impfung gegen die beiden häufigsten Resistenzmutationen Franziska Kraut, Renate Stirner, Rika Draenert Medizinische Klinik IV, Sektion Infektiologie, Klinikum der Universität München. Hintergrund: Unser Projekt beschäftigt sich mit der Frage, ob die Kombination aus antiretroviraler Therapie (HAART) und CD8 T-Zellantwort zur Kontrolle der HIV-Infektion möglich ist. Hierfür konzentrieren wir uns auf zwei sehr häufige Medikamentenresistenzmutationen, nämlich M184V und Y181C. Ziel unseres Projekts ist herauszufinden, ob CD8 T-Zellepitope, die diese Mutationen enthalten, von CD8 T-Zellen erkannt werden können. Dies wäre die Voraussetzung für eine therapeutische Impfung. Methodik: Wir untersuchten PBMC von 23 HIV-Patienten in verschiedenen Krankheitsstadien (mit und ohne HAART) auf ihr Vermögen, Peptide, die diese Mutationen enthielten zu erkennen. In einem zweiten Schritt erstellten wir in einer Kollaboration mit Dr. A. Schneidewind Viren, die die Mutationen enthielten. Im Moment testen wir, ob CD8 T-Zelllinien von infizierten Patienten in der Lage sind, komplette Viren, die eine oder beide Mutationen enthalten, zu erkennen. Ergebnisse: Ergebnisse zum Erkennen der unterschiedlichen Peptide: CD8 T-Zellantwort CD8 T-Zellantwort M M TR 42 CO 29 CO 18 1500 200 100 1000 500 1C V 1C V+ Y1 8 M 18 4 Y1 8 1C 1C V+ Y1 8 M 18 4 Y1 8 M 18 4 V 0 W T V+ Y1 8 1C 1C Y1 8 M 18 4 M 18 4 V 0 W T 0 300 18 4 200 400 M 400 SFC/ Mio. PBMC SFC/ Mio. PBMC 500 W T 600 SFC/ Mio. PBMC 1C 18 4 M V+ Y1 8 1C 0 1C 1C 20 18 4 Y1 8 V 18 4 M W T 0 V+ Y1 8 5 60 40 V 10 80 Y1 8 15 18 4 20 100 W T % von getesteten Patienten Anzahl Patienten 25 Abbildung: 23 HIV-Infizierte wurden mit synthetischen Peptiden untersucht. Das obere Panel gibt an, wie viele Patienten welches Peptid erkannten (absolute Anzahl links, relative Anzahl rechts). Im unteren Panel sind 3 Beispiele von Patienten exemplarisch dargestellt. CO 29 zeigt einen typischen therapie-naiven Patienten, dessen Viren selbst keine Resistenzmutationen tragen und der das Wildtyppeptid am besten erkennt. TR 42 repräsentiert das typische Ergebnis eines Patienten, dessen Virus die M184V-Mutation trägt. Folglich wird das M184V-enthaltende Peptid am besten erkannt. CO 18 ist ein Beispiel für die 3 Patienten unserer Studie, die interessanterweise nur eine Antwort auf das Peptid mit Y181CMutation hatten. Auf der x-Achse ist aufgeführt, welches Peptid mit welcher Mutation getestet wurde (WT = Wildtyp). Im Moment arbeiten wir an den Versuchen mit kompletten Viren. Wir haben die Viren hochgezüchtet und austitriert. Bei Infektion von primären CD4 T-Zellen, die das entsprechende HLA-Allel tragen, erhalten wir Infektionsraten von 1,0 – 2,4%. Vorläufige Daten zeigen, dass auch das Erkennen der Viren unterschiedlich ist. Die CD8-Linie von PR109 erkannte per Interferon-gamma-Produktion nur das Virus mit M184V- oder Y181CMutation, jedoch nicht das Wildtypvirus oder das Virus, das beide Mutationen enthielt. Per CD107a-Produktion erkannte die CD8-Linie desselben Patienten zudem noch das Wildtypvirus, jedoch weiterhin nicht das Virus mit beiden Mutationen. Schlussfolgerung: Die M184V-Mutation stört die CD8 T-Zellantwort kaum. Patienten, deren Virus die M184VMutation trägt, erkennen Epitope mit M184V-Mutation besser (siehe TR42). Das Einführen der Y181C-Mutation führt meist zu einer Abnahme der CD8 T-Zellantwort. Epitope mit beiden Mutationen werden am schlechtesten erkannt. Sehr interessant in diesem Zusammenhang ist jedoch, dass es Patienten gibt, die nur Epitope mit Y181C-Mutation erkennen (siehe CO 18). Dies legt nahe, dass eine CD8 T-Zellantwort mit dieser Mutation möglich ist. Soweit die Daten im Moment vorliegen, ist eine therapeutische Impfung, die die Mutationen M184V und Y181C enthält, möglich.











