Universität Ulm Protokoll zu den Grundübungen
Werbung

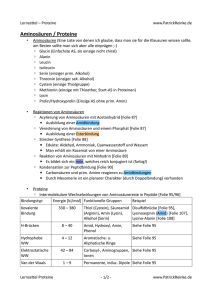
Universität Ulm Protokoll zu den Grundübungen Pflanzenphysiologie und Molekulare Botanik Wintersemester 2011/2012 Versuch F2: Proteine Tutor: Vorgelegt von: Abgegeben am: 07.12.2011 Version: Inhaltsverzeichnis: 1. Theorie zum Versuch 2 1.1 Proteinogene Aminosäuren 2 1.2 Proteinstrukturen 4 1.3 Chaperone 6 1.4 Aufgaben der Proteine 6 1.5 Die tRNA 8 1.6 Die Translation – von der Umsetzung des Bauplans 9 1.7 Der Cotranslationale Transport 12 1.8 Posttranslationale Modifikation 13 2. Versuchsdurchführung 14 2.1 SDS- PAGE 14 2.2 PAL- Enzymaktivität und Proteinkonzentration 15 2.3 Anthocyanbestimmung 15 2.4 Leucocyanbestimung 15 3. Ergebnisse 16 3.1 SDS- PAGE 16 3.2 PAL- Enzymaktivität und Proteinkonzentration 16 3.3 Anthocyanbestimmung 20 3.4 Leucocyanbestimung 21 4. Diskussion 21 4.1 SDS- PAGE 21 4.2 PAL- Enzymaktivität und Proteinkonzentration 22 4.3 Anthocyanbestimmung 23 4.4 Leucocyanbestimung 23 5. Zusammenfassung 24 6. Quellen 24 2 1. Theorie zum Versuch 1.1 Proteinogene Aminosäuren Proteine sind das wichtigste Baumaterial der Zellen. Diese aus Aminosäuren bestehenden Makromoleküle können von der Zelle selbst nach ihren Ansprüchen synthetisiert und lysiert werden. Es kommt aber auch vor, dass Viren oder andere Erreger diese empfindliche Maschinerie zu ihrem eigenen Nutzen manipulieren und die Proteinsynthese „aus dem Ruder läuft“.. Bekannt sind 20 proteinogene Aminosäuren. Seite Neuestem wird Selenocystein, eine Modifikation des Cysteins, als 21. proteinogene Aminosäure bezeichnet. Der menschliche Stoffwechsel ist je nach Entwicklungsstadium dazu befähigt, die meisten dieser Aminosäuren selbst zu synthetisieren. Für einen erwachsenen Menschen sind die Aminosäuren Leucin, Isoleucin, Valin, Phenylalanin, Methionin, Lysin, Threonin und Tryptophan Trypt essentiell. Proteinogene Aminosäuren bestehen alle aus der in Abbildung Abbildung 1 gezeigten Grundstruktur: Abbildung 1: Grundstruktur einer proteinogenen Aminosäure. Quelle: http://upload.wikimedia.org/wikipedia/commons/thumb/a/ac/Alpha Amino_Acids_V.1.png/250px-Alphahttp://upload.wikimedia.org/wikipedia/commons/thumb/a/ac/Alpha-Amino_Acids_V.1.png/250px Amino_Acids_V.1.png abgeändert. Stand: 02.12.11 Amino_Acids_V.1.png, Daraus wird ersichtlich, dass es sich bei allen proteinogenen Aminsoäuren um ߙ- LAminosäuren handelt. Das bedeutet, bedeut dass die Aminogruppe der vicinale cinale Nachbar der Carboxylgruppe ist und sich damit ein Kohlenstoffatom weiter befindet. Das L (lat. leavus für links) ergibt sich aus der Nomenklatur der FischerFischer Projektion,, die Substituenten sind in ihrer Priorität linksdrehend um das chirale KohlenstoffKohlenstoff Atom angeordnet. Der Rest ist variabel und ist geminal zur Aminogruppe angeordnet. Je nach den Eigenschaften des Restes werden Aminosäuren in polar, unpolar, aromatisch, aliphatisch, neutral, basisch, sauer und schwefelhaltig unterteilt (Siehe hierzu Abbildung 2). 3 Abb. 2: Einteilung proteinogener Aminosäuren. Quelle: http://www.biokurs.de/skripten/bilder/!aminoac.gif. Stand: 02.12.11 Elektrochemisch betrachtet sind Aminosäuren (= AS) Zwitterionen, d.h. dass die Carboxylgruppe der AS ein Proton abgeben und die Aminogruppe ein Proton aufnehmen kann. So liegen AS allerdings nur am isoelektrischen Punkt vor, der für jede AS spezifisch ist. Dazu muss der pH- Wert bei basischen AS erhöht und bei sauren AS gesenkt werden. Um nun ein Protein zu gewinnen, müssen die 4 Aminosäuren über eine Peptidbindung miteinander verknüpfen, es entsteht ein Dipeptid. Dabei handelt es sich um eine Kondensationsreaktion (= Substitutionsreaktion zweier Moleküle unter Abspaltung eines einfachen Moleküls. Hier: Wasser) zwischen der Aminogruppe der einen AS und der Carboxylgruppe einer anderen AS unter Ausbildung einer polar kovalenten Bindung. Diese wird, wie in Abbildung 3 gezeigt, mesomer stabilisiert. Daraus resultierend sind 4 Atome nebeneinander planar angeordnet. Abb. 3: Amin–Amid–Mesomerie. Quelle: http://upload.wikimedia.org/wikipedia/commons/d/d7/AmidMesomerie.png. Stand: 02.12.11 Geschieht dies mindestens 100 mal, so hat sich ein Polypeptid gebildet. Dabei endet eine Seite auf einer Carboxylgruppe (C – Terminus) und die Andere auf einer Aminogruppe (N – Terminus). Der N- Terminus wird auch als Proteinanfang bezeichnet, weil an diesem mit der Proteinsynthese begonnen wird und der C- Terminus als Proteinende, da er zuletzt synthetisiert wird. 1.2 Proteinstrukturen Aus der beträchtlichen Länge der Proteinmoleküle ergibt sich eine unübersichtliche Strukturvielfalt. Um eine Proteinstruktur vernünftig erklären zu können, wird diese in vier Strukturebenen aufgeteilt. Die Primärstruktur beschreibt die Aminosäureabfolge eines Proteins. Diese wird oft auch als Sequenz bezeichnet. 5 Die Sekundärstruktur beschreibt die Anordnung des Proteinrückgrats im Raum, die sich durch Wasserstoffbrückenbindungen zwischen Carboxyl- und Aminogruppen ergeben. Es ergeben sich mehrere Strukturen: 1) Die – Helix ist eine meist rechtswindende Helix. Eine Windung hat eine Ganghöhe von 0,54 nm und enthält dabei circa 3,6 Aminosäuern. Die äußeren Seitenketten sind aufgrund sterischer Wechselwirkungen gestaffelt (= staggerd) angeordnet. 2) Das - Faltblatt ist eine ziehharmonikaähnliche, geriffelte Struktur. Die einzelnen Stränge können parallel zueinander angeordnet sein, d.h. die Enden stimmen überein, antiparallel, d.h. die Enden sind gegenläufig, oder gemischt, wo es sich, wie der Name schon sagt, um eine Mischform der beiden ersten handelt. Aufgrund der Sterik besitzen – Faltblätter im Vergleich zur – Helix oft kleinere Seitenketten. Es sei hierbei erwähnt, dass das antiparallele Faltblatt dabei das stabilste ist, weil die Reste benachbarter Stränge hier gegeneinander angeordnet sind. Daher kommt dieses Faltblatt in der Natur am häufigsten vor. 3) Bei der – Schleife oder auch – Turn handelt es sich um eine aus 4 Aminosäuren bestehende Schlinge. Dabei sind die Carboxylgruppe der ersten AS und die Aminogruppe der vierten AS über eine Wasserstoffbrücke miteinander verbunden. Oft kommt sie als Struktur zwischen Faltblatt und Helix vor, weil sie die Torsionsspannung zwischen den beiden Strukturen herabsetzt. 4) Ω- Loops. Hier handelt es sich ebenfalls eine Schleife, die aber keiner streng periodischen Struktur unterliegt. Solche Schleifen dienen dazu, die AS des Schleifenanfangs und des Endes näher aneinander zu bringen. Daraus können sich stabilisierende Wechselwirkungen ergeben. Weitere Wechselwirkungen der AS untereinander werden unter der Tertiärstruktur zusammengefasst. Hierbei beteiligte Kräfte sind die Ausbildung von Disulfidbrücken, Van der Waals– Wechselwirkungen, Ionenbindungen und Wasserstoffbrückenbindungen der Reste zueinander. Diese Wechselwirkungen sind zudem noch vom Milieu abhängig. Daraus ergeben sich wiederum verschiedene Proteinfaltungen. Polypeptide können auch untereinander interagieren, bzw. wechselwirken. Sich daraus ergebende Anordnungen werden als Quartärstruktur bezeichnet. Bekanntester Vertreter 6 hierfür ist das Hämoglobin, das aus 4 Proteinketten gebildet wird. Als Beispiel für Disulfidbrücken fungiert das Insulin, dessen Proteinketten durch ebensolche Brücken zusammengehalten werden. 1.3 Chaperone Wenn ein Ei in der Pfanne brät, wird das Eiweiß undurchsichtig. Dieses Phänomen beruht auf der Denaturierung der betroffenen Proteine – sie verändern ihre Struktur und verlieren daher ihre Funktion. Unter solch drastischen Bedingungen ist die Denaturierung irreversibel. Bei geringeren Hitzeschwankungen kann die Zelle ihre Proteine mithilfe von Chaperonen vor der Denaturierung und dem damit verbundenen Funktionsverlust schützen. Chaperons können also als Hitzeschockproteine fungieren und werden bei Hitzestress vermehrt gebildet. In Pflanzen sind dies meist kleine, 5-20 kDa schwere Proteine, die wie ein „Fass“ mit 2 „Deckeln“ aussehen, in welchen die Proteine richtig gefaltet bleiben. Außerdem spielen Chaperone eine Rolle zur korrekten Faltung der Proteine nach deren Synthese, was sonst, bezieht man sich auf das Levinthal – Paradoxon, unadäquat lange dauern würde. Chaperone besitzen in ihrem Inneren ein bis zwei Kammern, welche jeweils hydrophil oder hydrophob sein können. Ein hydrophiles Protein erfährt in diesem Innenraum infolge der Ladungsbeschaffenheit (elektrische Wechselwirkungen) eine Konformationsänderung zu dessen nativen Form hin. Das Levinthal- Paradoxon führt vor Augen, dass, wenn jeder Aminosäurerest 2 mögliche Konformationen annehmen kann, sich bei einer Polypeptidlänge von n die Anzahl möglicher Faltungsvarianten auf 2 erhöht. Dauert in einem 150 Aminosäuren langen Polypeptid eine Konformationsänderung 10 , so dauert die korrekte Proteinfaltung im schlechtesten Fall 10 Jahre. Chaperons findet man auch extrazellulär, da Proteine zum Verlassen der Zelle oftmals ausreichend „schlank“ sein müssen. Ihre Funktion können sie dann erst nach extrazellulärer Faltung aufnehmen. 1.4 Aufgaben der Proteine Aufgrund der immensen Diversität können Proteine die unterschiedlichsten Aufgaben übernehmen. Hier seien einige davon genannt: 1) Proteine katalysieren Reaktionen. Als Enzyme setzen Proteine die Aktivierungsenergien vieler Reaktionen herab und greifen somit kinetisch in den Zellmetabolismus ein. Ein Enzym 7 setzt sich aus einem Proteinanteil, dem Apoenzym und einem Nichtproteinteil, dem Koenzym zu einem Holoenzym zusammen. Sind beide Enzymanteile kovalent gebunden, so wird der Kofaktor als prosthetische Gruppe bezeichnet. Als Beispiel sei hier die DNA – Polymerase III genannt, die bei der Replikation der DNA den komplementären Strang synthetisiert. 2) Proteine können aber auch gespeichert werden. Von Mikroorganismen ist bekannt, dass sie Proteine als Stickstoffquelle nutzen und Stickstoff auch in dieser Form speichern. Auch in Pflanzensamen lassen sich in der Aleuronschicht membranumgebene Speicherproteine in Form von wasserlosen Kristallen, die beim Auswachsen der Pflanze gebraucht werden, finden. Speicherproteine werden nach ihrer Löslichkeit eingeteilt: - Albumine sind wasserlöslich - Globuline sind salzlöslich - Prolamine sind in Alkohol löslich - Gluteine sind in saurem oder basischem Milieu löslich 3) Strukturproteine befinden sich im Extra- und im Intrazellulären. Diese verleihen dem Gewebe sowohl Festigkeit als auch Elastizität. Sie werden sowohl im Cytoskelett einer Zelle als auch im Bindegewebe, Sehnen, Bändern, Knochen und Knorpeln verbaut. Hier sei das Protein Kollagen erwähnt. Kollagen dient in erster Line der Festigkeit. Es handelt sich hierbei um ein - helikales Protein, welches sich unüblicherweise links herum aufwindet. Drei sich umschlingende Helices bilden zusammen eine rechtsgängige Superhelix, welche durch starke Wechselwirkungen der Helices untereinander zusammengehalten wird (siehe hierzu Abbildung 4). 8 Abbildung 4: Stuktur des Kollagens. Quelle http://upload.wikimedia.org/wikipedia/commons/d/d1/Collagentriplehelix.png imedia.org/wikipedia/commons/d/d1/Collagentriplehelix.png. Stand: 02.12.11 4) Kontraktile Proteine dienen der Fortbewegung eines Individuums. Muskeln sind beispielsweise hochgeordnete Proteinanordnungen von Actin und Myosin, die unter erheblichem Energieaufwand ineinander verschoben werden können. 5) Transportproteine können bestimmte kleine Moleküle durch einen Organismus befördern, rdern, wobei der Zielort in der selben Zelle, in einer anderen Zelle oder sogar außerhalb des Organismus liegen kann.. Ein bekannter Vertreter hierfür ist das Hämoglobin, welches unter Veränderung der Oxidationsstufe des Zentralatoms der prosthetischen Gruppe, nämlich des Eisens, Sauerstoff und Kohlenstoffdioxid durch die Blutbahn transportieren kann. 6) Bei der Immunabwehr kommen ommen Proteine in Form von Antikörpern zum Einsatz, die spezifisch gegen Antigene eingesetzt werden können. 7)) Proteine können der Signalübertragung zwischen Zellen dienen und damit als Hormon fungieren. In Pflanzen spielen Hormone eine zentrale Rolle bei der Kommunikation. Der Transport geschieht vertikal meist über das Phloem und Xylem oder horizontal, meist in Form eines Parenchymtransports. Als Beispiel für ein ein Phytohormon sei das Systemin 9 erwähnt. Systemin wird bei der Verletzung von Pflanzen freigesetzt und initiiert eine Kette von Signaltransduktionsprozessen, die wiederum der Synthese von anderen Hormonen und Proteinen, die z.b. dem Schutz vor Schädlingen dienen, anregen. 1.5 Die tRNA Die tRNA ist ein aus 73 bis 93 Ribonukleotiden bestehender, funktioneller Basenzusammenschluss, der dem Vorgang der Translation eines Proteins dient. Dazu später mehr. Die Sekundärstruktur einer tRNA ähnelt einem Kleeblatt. Diese kommt durch das Wirken von Wasserstoffbrückenbindungen zustande (Siehe Abbildung 5, schwarze Streifen). Die tRNA ist am 5‘ – Ende phosphoryliert und trägt an ihrem 3‘ – Ende eine Hydroxylgruppe, an der sich eine veresterte Aminosäure anlagern kann. Diesem Stamm gegenüber ist die Anticodonschleife. Die Anticodonschleife besteht aus 3 spezifischen Basen, welche mit einer komplementären Sequenz auf der mRNA interagieren. Die seitens des 5‘- Endes befindliche D- Schleife (D für Dihydrouracil, das häufig vorhanden ist) dient der Erkennung der tRNA durch die Aminoacyl- tRNA- Synthethase, welche die tRNA dann mit der richtigen AS belädt, oder den Einbau einer falschen Aminosäure korrigiert. Dies geschieht durch Abspalten von AMP vom Aminoacylmolekül, welches aus der Reaktion einer Aminosäure mit ATP hervorgegangen ist und somit aktiviert wurde. Die rechte TΨC – Schleife (Ribothymidin, Pseudouridin, Cytosin) heftet sich an die 5S- rRNA der großen Ribosomenuntereinheit. Abbildung 5 fasst die Struktur der tRNA zusammen: 10 Abbildung 5: Struktur der tRNA. . Stand: 02.12.11 Quelle: http://upload.wikimedia.org/wikipedia/commons/a/ae/The_tRNA_cloverleaf_general.svg Aus der Wobble- Hypothese geht hervor, dass nicht mehr als 41 verschiedene tRNAs in einer Zelle existieren, es also weniger tRNAs gibt wie mögliche Codons. Dies liegt daran, dass die erste Stelle des Anticodons der tRNA z.T. mit abgeänderten Basen wie etwa Inosin besetzt wird, welche schlechter Wasserstoffbrücken zum Codon ausbilden können. Folglich ist der genetische Code degeneriert. Dies hat zur Folge, dass Aminosäuren durch verschiedene Tripletts codiert werden können. Die höhere Anzahl kommt durch verschiedene tRNAs für das gleiche Codon zustande. 1.6 Die Translation – von der Umsetzung des Bauplans Bei der Translation wird im Ribosom anhand der mRNA das entsprechende Protein synthetisiert. Dazu verbinden sich zu Beginn der Translation die mRNA und mehrere Initiationsfaktoren. Schließlich bindet noch die kleine ribosomale Untereinheit an den Komplex. Im Folgenden bindet eine Initiator tRNA, die mit Methionen oder mit Formylmethionin bei Bakterien beladen ist. Anschließend bindet der Komplex an die große Ribosomuntereinheit. An der Peptidyl- Stelle (P- Stelle) sitzt die tRNA mit wachsender Peptidkette. An der Aminoacyl- Stelle (A- Stelle) bindet die tRNA, die mit der nächsten Aminosäure beladen ist, an das Startcodon (AUG). Die neue tRNA, sowie die tRNA der PStelle zur verschieben sich. Die tRNA der P- Stelle wird zur E- Stelle (Exit- Stelle) hin verschoben, von wo aus sie das Ribosom verlassen kann. Die mRNA wird in 5‘- 3‘- Richtung gelesen und Proteine werden vom Amino- zum Carboxylende hin synthetisiert. Einen Überblick hierzu verschafft Abbildung 6. Nun zu den einzelnen Teilschritten der Peptidsynthese: 1) Initiation. Hierfür werden in prokaryotischen Zellen die Initiationsfaktoren IF1, IF2 und IF3 benötigt. IF1 verhindert eine frühzeitige Bindung beider ribosomaler Untereinheiten und IF3 entfernt Nichtinitiator- tRNA. IF2, ein G-Protein, bindet die Initiator fmet- tRNA, verbindet sich mit der kleinen Untereinheit und hilft schließlich unter GTP- Hydrolyse beim Verbinden mit der großen Untereinheit. 11 2) Elongation. Es kommt zur Bindung neuer tRNA, Ausbildung einer Peptidbindung, welche von dem Enzym Peptidyltransferase katalysiert wird und der Vorbereitung auf den nächsten Elongationsschritt, bis ein terminierendes Codon erreicht ist. Für die Bindung neuer tRNA wird der Elongationsfaktor Tu, ein G- Protein, benötigt. Dieser schützt die Esterbindung zwischen der tRNA und der Aminogruppe und entlässt die tRNA, nachdem sie an das Ribosom gebunden hat. Dazu wird GTP hydrolysiert. Ferner garantiert Tu ein Einbauen der richtigen AS, weil eine falsche tRNA nicht vom Tu weg dissoziieren kann. Tu erkennt kein Initiator- tRNA. Spaltung der Esterbindung und Bildung einer Peptidbindung ist eine exotherme, endergonische Reaktion. Eine katalytisch wirksame RNA des Ribosoms, ein sog. Ribozym verschiebt die Energiehyperfläche so, dass die Reaktion spontan ablaufen kann. Der Elongationsfaktor– G, ein weiteres G- Protein, verschiebt durch Hydrolyse die verbrauchte tRNA von der P- zur E- Stelle und stellt damit die Ausgangssituation zu Beginn der Elongation wieder her. Ferner wird die daran assoziierte mRNA um ein Triplett verschoben. 3) Termination. Sie wird durch das Auftreten der Codons UAG, UAA oder UGA ausgelöst, für die es keine passenden tRNAs gibt. An deren Stelle treten die Terminationsfaktoren (releasing factors) RF1 (an UAG oder UAA) und RF2 (an UAA oder UGA), welche vom Faktor RF 3 dahin vermittelt werden. Um die letzte veresterte AS zu lösen, wird von den RFs genau ein Molekül Wasser zum Ribosom transportiert, welches die Spaltungsreaktion dann selbst als Katalysator begünstigt. Bei der Termination werden außerdem beide Ribosomuntereinheiten wieder voneinander gelöst. 12 Abbildung 6: Translationsvorgang im Ribosom (cartoonhaft und ohne E- Stelle dargestellt. Stand: 02.12.11 Quelle: http://upload.wikimedia.org/wikipedia/commons/8/88/Ribosom_mRNA_translation_de.svg Der Ablauf der Translation unterscheidet sich bei eu- und prokaryotischen Zellen. In Eukaryoten gibt es keine fMet-tRNA, sondern eine unformylierte Initiator- Met- tRNA. In Prokaryoten gibt es Shine- Dalgarno- Sequenzen auf der mRNA, die reich an purinhaltigen Basen sind. Dadurch wird dem Ribosom vermittelt, an welcher AUG- Stelle es das Formylmethionin setzen muss, da AUG auch in Prokaryoten normalerweise die AS Methionin kodiert. Bei Eukaryoten übernimmt die Kozak- Sequenz diese Aufgabe. Diese bildet einen Konsens aus Nukleinbasen, die häufig in der Nähe des Startcodons AUG auftreten. Abweichungen von dieser Sequenz können sich auf die Proteinbiosynthese auswirken. 1.7 Der Cotranslationale Transport Proteine können synthetisiert werden, während sie durch eine Biomembran wandern. Beispielsweise können Proteine in Eukaryoten durch die Membran des Endoplasmatischen Retikulums wandern. Dies geschieht durch Vesikeltransport. Nun zum Ablauf: 13 Ein Signalsequenzerkennungspartikel (SRP) bindet an die N-terminale Signalsequenz des Polypeptids und am Ribosom. Die Synthesegeschwindigkeit wird herabgesetzt, um zu verhindert, dass das Protein vor Ankunft am ER fertig synthetisiert ist. Das SRP leitet das Ribosom zur ER- Membran. Dort wird es vom SRP- Rezeptor gebunden. Das Ribosom interagiert zugleich mit dem Sec61- Komplex, durch den die Proteine später hindurch befördert werden, um ins Lumen des ER zu gelangen. Unter GTP- Hydrolyse löst sich das SRP vom Ribosom und dem Protein und übergibt diese an den Translokationsapparat. Im ERLumen kann das Protein seine native Form annehmen. Nach Erkennung durch sog. CurgoRezeptoren wird es hier in Transportvesikel verpackt und weiter transportiert werden. Nun werden die Proteine zum Golgi- Apparat befördert und ein letztes Mal modifiziert. Danach werden sie an ihren Bestimmungsort transportiert. In Abbildung 7 wird der Cotranslationale Transport veranschaulicht. Abbildung 7: Cotranslationaler Transport. Quelle: http://upload.wikimedia.org/wikipedia/de/8/80/Cotrans.jpg. Stand: 02.12.11 Beim Posttranslationalen Proteintransport müssen Proteine nach deren Synthese ebenfalls durch eine Membran transportiert werden. Verschiedene Signalsequenzen an unterschiedlichen Orten im Protein dienen dazu, vom Zielort erkannt zu werden und damit 14 die richtige Membran zu passieren. Solche Zielorte können Organellen wie Mitochondrien, aber auch der extrazelluläre Raum. Beim posttranslationalen Proteintransport kommen im Gegensatz zum cotranslationalen eher hydrophilere Signalsequenzen zum Einsatz. Diese Sequenzen werden im Allgemeinen nach Durchqueren der Membran abgespalten. 1.8 Posttranslationale Modifikation Hierbei handelt es sich um Proteinveränderungen, die nach der Translation stattfinden. Dies kann gewollt, aber auch ungewollt auftreten, beispielsweise durch die Einwirkung von Radikalen oder von radioaktiver Strahlung. Beispiele für posttranslationale Modifikationen sind phosphorylierungen durch Proteinkinasen oder das Ausbilden einer Disulfidbrücke zwischen zwei Cysteinresten. Auch werden Enzymproteine in diesem Schritt an ihre Cofaktoren gebunden oder Signalsequenzen abgespaltet, wenn ein Protein seinen Zielort erreicht hat. Kurzum: Es gibt eine große Vielfalt posttranslationaler Modifikationen. 2. Versuchsdurchführung Grundsätzlich kann zur Durchführung das Praktikumsskript zu Rate gezogen werden. Einige Details wurden jedoch in der Praxis verändert. Die Versuchsabwandlungen werden in diesem Abschnitt wiedergegeben. 2.1 SDS- PAGE Zu Beginn soll kurz das Prinzip einer diskontinuierlichen Natrium- DodecylsulfatGelelektrophorese (= SDS- PAGE) erläutert werden. Hierbei handelt es sich um eine Methode zur Auftrennung von Proteinen und kleinen Nukleinsäuren. Das Verfahren ist bekannt für seine hohe Auflösung, die durch Verwendung von Polyacrylamid anstelle von Agarose erzielt wird. Das eingesetzte SDS übernimmt zwei wichtige Aufgaben: Einerseits denaturiert es die Proteine und andererseits umschließt es diese, indem sich ein SDS- Molekül an circa 3 ASReste anlagert. Dies hat eine Maskierung der Eigenladung der Proteine zur Folge, wodurch eine Trennung nur noch durch das Molekulargewicht erfolgt. Leichtere Proteine wandern weiter wie schwerere. Das Acrylamid wird nach Polymerisation zum Gel. Das Gel besteht aus einem engeren Gitternetz, dem Trenngel und einem weitmaschigeren Gitternetz, dem Sammelgel. Das Sammelgel hält die nicht abzentrifugierten Zellfragmente und nicht denaturierte Proteine zurück. Vor allem die Diskontinuität des pH- Wertes zwischen den 15 beiden Gitternetzen ist für deren Eigenschaften entscheidend. Aufgrund der chemischen Beschaffenheit des SDS wandern alle Proteine mehr oder weniger schnell zur Anode. Es muss ein Puffer eingesetzt werden, da das Gel Wasser enthält, welches unter der angelegten Spannung an der Spannungsquelle in seine Bestandteile zerfällt. An der Kathode entstehen Wasserstoff und Hydroxidionen und an der Anode Sauerstoff und Hydronium- Ionen, die ungepuffert das Ergebnis der SDS- Page verfälschen würden. Zum Anfärben der Proteine wurde Coomassie Brilliant Blue zugegeben. Abbildung 8 zeigt die prospektive Laufweite des Proteinmarkers. Je nachdem, wie schwer das Protein ist, an dem der Marker bindet, kann dieser weiter oder weniger weit „wandern“. Abbildung 8: Proteinmarker 2.2 PAL- Enzymaktivität und Proteinkonzentration Es wurden 2 analoge Lösungen für etiolierte und nichtetiolierte Pflanzen hergestellt. Dazu wird wie folgt vorgegangen: 0,4 ml Pflanzenextrakt wurden mit 0,4 ml Boratpuffer vermischt. Davon wurden 0,5 ml mit 0,5 ml Bradford – Lösung verdünnt, wovon wieder 0,5 ml mit 0,5 ml Bradford – Lösung verdünnt wurden. Hiervon werden dann 1 ml photometrisch untersucht. Das bedeutet, dass sich noch 62,5 µl Extrakt in der Lösung befinden und dieser 8 mal verdünnt wurde. Danach wurden die Proben ins Photometer überführt (Siehe hierzu auch das Praktikumsskript). 16 2.3 Anthocyanbestimmung Der Anthocyangehalt wurde photometrisch bei einer Wellenlänge von 515 nm bestimmt. Es wurde wie vom Skript vorgeschlagen vorgegangen. 2.4 Leucocyanbestimung Hierbei wurden die enthaltenen Leucocyane durch Dehydratisierung mit dreiwertigem Eisen in saurem Millieu zu farbigen Cyanidin umgesetzt. Bei der Durchführung wurde wie im Skript vorgeschlagen vorgegangen. 3. Ergebnisse 3.1 SDS- PAGE Um die SDS- Page zu beschleunigen, wurde zuerst eine Spannung von 120 V und dann eine Spannung von 200 V über den Zeitraum von ca. zwei Stunden angelegt. Wie in Abbildung 9 ersichtlich wird, ist auf dem Gelbild lediglich eine Markerbande zu erkennen, nicht aber die angefärbten Proteinstandorte. Dadurch ist kein Rückschluss auf die in der Probe enthaltenen Gewichte, bzw. Orten einer Proteinakkumulation möglich. Mögliche Fehlerquellen beim Vorgehen werden unter Abschnitt 4 diskutiert. Abbildung 9: Ergebnis der Gelelektrophorese 3.2 PAL- Enzymaktivität und Proteinkonzentration 17 Es wurde eine Tabelle (Tabelle 1) zum Verlauf der Extinktion über die Zeit angelegt: Tabelle 1: Verlauf der Extinktion bei Sinapis alba Zeit [min] 0 0,263 0,247 10 0,262 0,245 20 0,261 0,246 35 0,260 0,245 40 0,261 0,245 Zeit [min] 50 0,260 0,245 60 0,266 0,250 70 0,266 0,249 80 0,268 0,250 90 0,268 0,250 Es lässt sich sowohl bei den etiolierten als auch bei den belichteten Pflanzen eine Zunahme der Extinktion des Lichtes erkennen. Die Extinktion etiolierter Pflanzen ist höher als die der belichteten Pflanzen. Anhand der erhaltenen Ergebnisse wurde ein Diagramm angelegt, das den Extinktionsverlauf grafisch veranschaulicht: Diagramm 1: Grafik zum Extinktionsverlauf bei belichteten und unbelichteten Senfkeimlingen 18 0,27 0,265 Extinktion E 0,26 0,255 0,25 E unbelichtet 0,245 E belichtet 0,24 0,235 0,23 0 10 20 35 40 50 60 70 80 90 Zeit [min] Es wird ersichtlich, dass der Anstieg der Extinktion nicht linear verläuft. Vielmehr handelt es sich um einen schwankenden Anstieg der Extinktion. Generell wäre ein linearer Anstieg zu erwarten. Mit dem Lambert – Beer’schen Gesetz kann nun auf die Konzentration der im Extrakt enthaltenen Zimtsäure geschlossen werden. Dies geschieht wie folgt: = ∙ ∙ → Δä = ∙ ∙ . mit ä = 10 ²/ (Extinktion der Zimtsäure), d = 1cm (Küvettendicke) und . = 13 (Grad der Verdünnung) Da sich die vorhandene Menge der PAL- Enzyme und deren Aktivität nicht direkt bestimmen lässt, muss ein Umweg genommen werden. Mithilfe der Bradford- Bestimmung lässt sich die im Extrakt enthaltene Gesamtproteinmenge bestimmen. Folgende Tabelle wird zu Rate gezogen: Tabelle 2: Messwerte zur Berechnung der Gesamtproteinkonzentration BSA-Lösung in µg/ml Mittelwert der Extinktionswerte 2 0,091 4 0,165 19 6 0,250 8 0,332 10 0,422 14 0,602 18 0,761 Anhand dieser Werte ließ sich Diagramm 2 erstellen. Ein nichtlinearer Graph entsteht. Mithilfe einer Kalibrierungsgerade wird sich der in der Küvette enthaltenen Proteinkonzentration relativ präzise und mathematisch simpel angenähert: Diagramm 2: Grafik zum Extinktionsverlauf bei belichteten und unbelichteten Senfkeimlingen 20 Mittelwert der Extinktionen E 18 y = 2,571x - 1,429 16 14 12 10 8 6 4 2 0 0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 β(BSA) [µg/ml] Durch Umstellen der Geradengleichung wird nach der enthaltenen Proteinmasse aufgelöst: = , , → ! = +1,4286 2,5714/µ 20 Die nachfolgende Tabelle zeigt die gemessenen Extinktionen, die berechneten enthaltenen Proteinmassen und die Proteinmassenkonzentration des Extrakts (= ! ), welche sich mit ! "# $" = ಸೞೌೝ %, berechnen lässt: Tabelle 3: Enthaltene Proteinmassen und daraus resultierende Proteinmassenkonzentrationen in etiolierten und belichteten Extrakten Enthaltene Proteinmasse [mg/ml] ! [mg/ml] 0,467 0,369 0,921 0,289 0,334 0,835 Die Extinktion etiolierter Pflanzen ist beinahe doppelt so hoch wie die der belichteten. Auch die Proteinmasse und die Proteinkonzentration ist bei etiolierten Pflanzen höher. Nun kann in die Formel für die PAL- Enzymaktivität eingesetzt werden: ' = Δä [ ] ∙ (") * [ ] 1) etioliert: Δä = ∙ ∙ . = ' = Δܼ݅݉ݏݐä[ ݁ݎݑ 0,268−0,263 107 ܿ݉2 ∙1+, ݈݉ ݈݉ ] ݈݉ ∙ 13 = 6,5 -,./ ,/ , / 01∙ಸೞೌೝ [ ] = 2% 345∙%,2*/ = 7,84 ∙ 10 345∙* 2) belichtet: 0,25−0,247 Δä = ∙ ∙ . = 107 ܿ݉2 ݈݉ ' = Δܼ݅݉ݏݐä[ ݁ݎݑ ∙ 13 = 3,9 ∙1+, ݈݉ ] ݈݉ -,./ ,/ ,2 / ] 01∙ಸೞೌೝ [ = 2% 345∙%, */ = 5,19 ∙ 10 345∙* Aus der Gesamtproteinmenge von 1 mg im Extrakt einer belichteten Pflanze setzt das PALEnzym 5,19 ∙ 10 mol Phenylalanin pro Minute zu Zimtsäure um. Bei etiolierten Pflanzen sind es 7,84 ∙ 10 mol. 21 3.3 Anthocyanbestimmung Für die Berechnung der Anthocyankonzentration % gilt: 1,00 [E] ≈ 0,37 µmol/ml = % . Daraus folgt: % = ∙ 0,37μ / Unsere gemessen Extinktionen sowie die Stoffmenge an Anthocyan in der Probe wurden in Tabelle 2 festgehalten: Tabelle 4: Extinktion zur Antocyanbestimmung und die daraus resultierende Stoffmenge Lösung E % [nmol/ml] 0,160 59 0,246 91 Es wird ersichtlich, dass die Extinktion der belichteten Pflanze beinahe doppelt so hoch ist wie die der etiolierten. Als Folge daraus ist in den belichteten Pflanzen ein höherer Anthocyangehalt nachzuweisen. 3.4 Leucocyanbestimung Zuerst muss 6. = der ("ö* %, '7"# Verdünnungsgrad des Extrakts bestimmt werden: = 20. Wieder gilt: 1,00 [E] ≈ 0,37 µmol/ml = . Durch Umstellen der Formel und Subtraktion der bereits vorhandenen Anthocyane ergibt sich die in der Probe enthaltene Leucocyanstoffmenge: = ∙ 0,37μ ∙ 6. − % Die Ergebnisse wurden in Tabelle 5 festgehalten: Tabelle 5: Extinktion zur Leucocyanbestimmung und die daraus resultierende Stoffmenge Lösung E % [nmol/ml] [nmol/ml] 0,015 59 52 0,035 91 168 22 Wieder erhält wird bei dem Extrakt aus belichteten Pflanzen eine höhere Extinktion, die sich auf die Menge der nachgewiesenen Leucocyane auswirkt, nachgewiesen (Siehe hierzu Tabelle 5). Der Leucocyangehalt belichteter Pflanzen ist mehr als dreimal so hoch wie der der etiolierten. 4. Diskussion 4.1 SDS- PAGE Bei diesem Verfahren sollte die unterschiedliche Proteinlandschaft von etiolierten und belichteten Keimlingen nachgewiesen werden. Dies konnte nicht gezeigt werden, da auf dem fertig entwickelten Gel lediglich ein blauer „Strich“, nämlich die Markerbanden zu erkennen sind. Es gibt mehrere Gründe, warum keine Proteinbanden zu erkennen sind: 1) Fehler bei der Anfärbung: Möglicherweise wurden Fehler bei der Anfärbung des Gels gemacht. Hierbei ist es wichtig, dass das Gel vor jedem Waschschritt in der Mikrowelle ausreichend erhitzt wird. Entweder ist hierbei nicht die erwünschte Temperatur erreicht worden, oder das Gel wurde gekocht, was zu einer erheblichen Verfälschung der Proteinbanden führt. 2) Keine Bindung der Proteine zum Farbstoff: Coomassie- Brillant- Blue lagert sich an die basischen Seitenketten der Aminosäuren an. Um dies zu garantieren, wird ein Puffer in der SDS- Page eingesetzt, um die Bindungsstellen aktiv zu halten. Möglicherweise war die Zusammensetzung des Puffers versehentlich falsch und es herrschte nicht der richtige pH im System. Ferner kann es zu diesen „Negativbanden“ gekommen sein, weil es sich gegebenenfalls um schwer zu färbende Proteine gehandelt haben könnte, die nur schwer eine Bindung zum Farbstoff ausbilden. 3) Pipettierfehler: Es ist möglich, dass bei Vorbereitung der SDS- PAGE falsch pipettiert wurde. Das kann zum einen bedeuten, dass die falschen Mengen der benötigten Agenzien in die Taschen gegeben wurden, oder, dass die falschen Lösungen verwendet wurden. 4) Zu kurze Entfärbungsphase: Das Gel wurde im letzten Schritt mit einer EssigsäureEthanol- Lösung entfärbt. Zu kurzes Entfärben, kann dazu führen, dass die Banden nicht erkennbar werden, da kein ausreichender Kontrast zum Hintergrund besteht. 23 4.2 PAL- Enzymaktivität und Proteinkonzentration Sowohl in belichteten als auch in unbelichteten Keimlingen konnte die Aktivität des PALEnzyms nachgewiesen werden. Phenylalanin- Amonium- Lyase setzt die Aminosäure Phenylalanin in trans- Zimtsäure um. Trans- Zimtsäure ist ein wichtiges Intermediat des Sekundärmetabolismus, von dem aus sowohl Phenylpropanoide als auch Flavonoide wie Leucocyane und schließlich Anthocyane synthetisiert werden können. Dabei ist die Enzymaktivität in belichteten Pflanzen ungefähr viermal so hoch wie in etiolierten. Das liegt daran, dass der Stoffwechsel bei belichteten jungen Keimlingen mehr auf die Synthese von Anthocyanen ausgelegt ist. Warum die Proteinmassenkonzentration in etiolierten Pflanzen höher sein sollte wie in belichteten lässt sich nicht mit Sicherheit sagen, da vor allem der Stoffwechsel belichteter Pflanzen auf Hochtouren läuft, um möglichst schnell Gewebe für die Photosynthese aufzubauen und Lichtenergie nutzen zu können. Es könnte aber sein, dass die beiden Küvetten miteinander vertauscht haben, da unglücklicherweise auf deren Markierung verzichtet wurde. Zudem könnte ein Pipettierfehler beim Verdünnen entstanden sein. Eine mögliche Erklärung liegt in den Speicherproteinen. Etiolierte Pflanzen benötigen diese, um Energie zu erhalten. Belichtete Pflanzen enthalten zwar auch Speicherproteine, jedoch weniger, da sie sich durch die Synthese von energiereicher Glucose mittels Photosynthese selbst versorgen können. 4.3 Anthocyanbestimmung Aus den Ergebnissen geht hervor, dass der Gehalt an Anthocyanen in belichteten Pflanzen beinahe doppelt so hoch ist wie in unbelichteten. Der Grund für eine verstärkte Aktivität dieses Sekundärstoffwechselweges ist, dass die Anthocyan- Farbstoffe die junge Pflanze vor schädlicher UV- Strahlung schützen, die sonst durch Radikalreaktionen die DNA schädigen könnte. In etiolierten Keimlingen spielen die Anthocyane und der gesamte Sekundärmetabolismus eine untergeordnete Rolle. Anthocyane werden höchstens präventiv in ihrer Vorstufe als Leucocyane im Organismus gespeichert, bis sie benötigt werden. Für die etiolierten Pflanzen ist dies naheliegender, da sie sich bereits unter ungünstigen Bedingungen entwickeln müssen und von ihren Proteinreserven zehren müssen. Daher können sie weniger Energie in momentan „unwichtige“ Stoffwechselprozesse 24 verschwenden. Eine weitaus geringere Anthocyansynthese hilft der der etiolierten Pflanze also ihre Überlebenschancen zu steigern, bzw. konkurrenzfähig zu werden. 4.4 Leucocyanbestimung Die in 3.2 und 3.3 vorgebrachten Behauptungen werden weiter von der Tatsache untermauert, dass belichtete Pflanzen mehr als die dreifache Stoffmenge an Leucocyanen besitzen, wie etiolierte. Es wurde bei etiolierten Pflanzen eine geringfügig höhere Stoffmenge von Anthocyanen als die von Leucocyanen bestimmt. Dies trifft in der Realtät vermutlich nicht zu. Etiolierte Pflanzen nutzen Leucocyane als Speicherproteine zur Energiegewinnung, nicht aber Anthocyane. Mithilfe des Enzyms Oxygenase können Leucocyane in Succinat umgewandelt werden. Succinat ist ein Intermediat des Citrat- Zyklus, aus dem Reduktionsäquivalente gewonnen werden können, welche in der Atmungskette dann die ATP- Synthese antreiben und damit Energie liefern. Mögliche Fehlerquellen sind: 1) Das Benutzen von Pflanzenextrakten verschiedener Testkörbchen, die möglicherweise unterschiedlich behandelt worden sind. Vermutlich kam es auch zu einem Lichteinfluss beim Arbeiten, u.a. beim zeitaufwändigem „Ernten“ des Hypokotyls. 2) Messfehler am Photometer. 5. Zusammenfassung Bis auf die SDS- PAGE haben alle Versuche grob unseren Erwartungen entsprochen. Es wurde ein deutlicher Unterschied der PAL- Enzymaktivität und damit der Leuco- und Anthocyansynthese zwischen etiolierten und nicht etiolierten Pflanzen beobachtet. Daraus folgten dann auch ein höherer Anthocyan- und Leucocyangehalt in belichteten Pflanzen. Etiolierte und belichtete Keimlinge wiesen in etwa die Selbe Proteinkonzentration auf, es wurde aber erarbeitet, dass es sich hierbei um verschiedene Arten handelt. 6. Quellen 1) Stryer; Biochemie 6.Auflage 2007 Spektrum akademischer Verlag 2) Heß, Dieter; Pflanzenphysiologie, 10. Auflage, 1999, Ulmer Verlag 25 3) Skript zum Grundpraktikum Pflanzenphysiologie und molekulare Botanik WS 2011/12 4) Campbell, Neil A.; Biologie, 2. Korrigierter Nachdruck 2000, Spektrum Verlag 5) www.wikipedia.de 26