s - B CUBE Dresden
Werbung

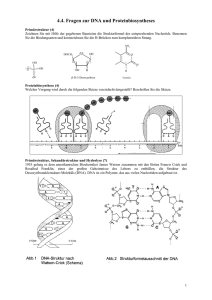
3. Strukturen und Faltung von Proteinen 71 Hierarchie der Proteinstrukturen Primary structure (AA Sequenz) Proteinfaltung Supramolekulare Assemblierung 72 3.1. “Vokabular” Proteinkonformation: Anordnung aller Atome im Raum (3D Struktur) Native Konformation: 3D Struktur, in der das Protein funktionell ist (“gefaltet”) Denaturierte Struktur: nicht funktionelles Protein (“entfaltet”) 3.2. Sekundärstrukturen Partieller Doppelbindungscharakter der Peptidbindung mesomere Grenzstrukturen Mesomerie “verschmierte” Elektronen 73 Konsequenzen des partiellen Doppelbindungscharakter der Peptidbindung → C-N Abstand ist kürzer als C-N Abstand in Aminen → Rotation der C-N Bindung ist blockiert (Aktivierungsenergie für die Rotation ist drastisch erhöht): dadurch ergeben sich cis-trans Isomere sterische Behinderung trans-Isomer cis-Isomer energiereicher, außer für Bindungen, an denen Prolin beteiligt ist 74 Peptidbindung gestreckte Polypeptidkette 75 Konformationen benachbarter Peptidbindungen Sterische Behinderung Torsionswinkel (dihedrale Winkel) Ψ (psi) and Φ (phi) 76 Beschreibung der relativen Orientierung benachbarter Peptidbindungen mittels der Torsionswinkel Φ (Phi) und Ψ (Psi); Diederwinkel variieren jerweils zwischen -180° bis *180° Rotation benachbarter Peptidbindungen relativ zueinander ist durch sterische Behinderung eingeschränkt → Ramachandran Diagramm zeigt sterisch mögliche Φ-Ψ Kombinationen an → häufig in Proteinen auftretende Φ-Ψ Kombinationen: Φ = -57° und Ψ = -47° → α Helix Φ = -139° und Ψ = +135° → antiparalleles β Faltblatt Φ = -119° und Ψ = +113° → paralleles β Faltblatt Ramachandran Diagramm a: rechtsgängige α-helix aL: linksgängige α-helix C: collagen helix : paralleler b-Strang, : antiparalleler b-Strang 77 Ramachandran Diagramm des Enzyms Pyruvat Kinase Psi-Phi Kombinationen für alle AA-Reste außer Gly sind gezeigt 78 α Helix rechtsgängige Spirale 3.6 AA pro Windung Länge der Helix pro Windung = 0.54 nm 79 β Faltblatt (I) gefaltete Schichten Wiederholungseinheit: 2 AAs Abstand der Wiederholungseinheiten: 0.7 nm 80 β Faltblatt (II) Antiparalleles b Faltblatt Paralleles b Faltblatt parallele H-Brücken angewinkelte H-Brücken 81 α Helix: stabilisiert durch H-Brücken zwischen den Peptidbindungen von AAn und AAn+4 β Faltblatt: besteht aus ≥2 Strängen, stabilisiert durch H-Brücken zwischen den Peptidbindungen von AAn und AAn+x Detektion von α Helices und β Faltblättern in Proteinen mittels Circular Dichroismus (CD) Spektroskopie Prinzip: links-zirkular polarisiertes Licht wird von chiralen Molekülen unterschiedlich stark absorbiert verglichen mit rechts-zikular polarisiertes Licht; die Differenz der Extinktionskoeffizienten für rechts- und links-zirkular polarisiertes Licht (Δε = εL – εR) in Abhängigkeit von der Wellenlängen einen unterschiedlichen Verlauf für α Helices und β Faltblätter 82 CD Spektroskopie 83 3.3. Tertiärstrukturen fibrillärer Proteine (Beispiele) α-Keratin: Aufbau stabiler Schutzstrukturen mit variable Härte und Biegsamkeit (Finger-/Zehennägel, Haare,, Horn, Federn); besteht aus coiled-coil Dimeren, die sich über nicht kovalente Bindungen aneinanderlagern; zusätzliche Stabilisierung durch Disulfid-Brücken zwischen coiled-coil Dimeren; Kollagen: Aufbau von Strukturen mit hoher Zugfestigkeit und geringer Dehnbarkeit; besteht aus Triple-Helices, die über H-Brücken und kovalente Bindungen quervernetzt sind; enthält modifizierte Aminosäuren (Hydroxyproline, Hydroxylysine); Seiden-Fibroin: Aufbau von flexiblen Filamenten; besteht aus β Faltblatt Stapeln, die einzig durch hydrophoben Effekt zusammengehalten werden; 84 α-Keratin (I): Dimerisierung über “Coiled-coil” Domänen Heptad Wiederholung (Repeat) -a-b-c-d-e-f-g-a’-b’-c’-d’-e’-f’-g’- a’’-b’’a, a’, a’’,… hydrophobe d, d’, d’’,… Aminosäurereste Coiled-coil Dimer (linksgängige Spirale) 85 α-Keratin (II): Filamentbildung kovalente Quervernetzung der Coiled-coil Helices mittels Disulfid-Brücken I S I S S I S S I S S I S I S S I I S I S S I S S I S I S S I I S S I S Querschnitt durch ein Haar S I S S I S I 86 Kollagen (I): Trimerisierung über H-Brücken Polypeptidkette Tripelhelix wiederholendes Sequenzmotiv -G-X-Y-G-X-Y- häufig: X = P Y=O O = Hydroyxyprolin (Hyp) linksgängige, gestreckte Helix: 3.3 AA und 1 nm pro Windung 87 Kollagen (II): Trimerisierung über H-Brücken H-Brücken (C=O….H-N) zwischen den Peptidrückgraten der drei Polypeptidketten 88 Kollagen (III): Konformationen von Pro und Hyp Prolyl-Rest 4-Hydroxyprolyl-Rest 4-OH Gruppe stabilisiert Cg-exo Konformation des Pyrrolidin-Rings 89 Kollagen (IV): Hydroxylysin und Quervernetzung (Hyl) 250 nm 90 Biosynthese von Hydroxyprolin und Hydroxylysin findet an der Polypeptidkette des Kollagens statt (post-translationale Aminosäuremodifikationen) Enzyme Prolylhydroxylase und Lysylhydroxylase katalysieren die Hydroxylierung von Prolyl- bzw. Lysyl-Resten mittels O2 (identische Reaktionsmechanismen) → Reaktionsmechanismus am Beispiel der Prolyhydroxylase (PHase): Pro-Rest α-Ketoglutarat (α-KG) Hyp-Rest Succinat PHase enthält für die Katalyse essentielles Fe2+, das in einer unerwünschten Nebenreaktion oxidiert wird: PHhase-Fe2+ + α-KG + O2 + 2 H+ aktiv CO2 PHhase-Fe3+ + Succinat + H2O inaktiv 91 → Reaktivierung der Prolyhydroxylase (bzw. Lysylhydroxylase) durch Ascorbinsäure (Vitamin C): inaktiv aktiv Ascorbinsäure Dehydroascorbinsäure Fehlen von Ascorbinsäure führt zu Skorbut (Schädigung der Knochenmatrix führt u.a. zu Zahnausfall) 92 Oxidative Deaminierung von Lysin-Resten katalysiert durch das Enzym Lysyloxidase führt zur Quervernetzung von Kollagen Triple-Helices: Allysin-Rest Triplehelix 2 Triplehelix 1 Wassestoffperoxid Weitere Reaktionen wandeln Shiff-Base Schiff-Base in stabilere Bindung um Hydroxyprolin und Hydroxylysin ermöglichen nicht-kovalente Vernüpfung von Kollagen-Triplehelices über H-Brücken 93 Seiden-Fibroin Primärstruktur Tertiär-/Quartärstruktur 94 3.4. Tertiärstrukturen globulärer Proteine Generelle Charakteristika ihrer 3D Strukturen: • Vorhandensein von Polypeptidschleifen (Loops), die α Helices und β Faltblätter verbinden • unpolare Seitenketten liegen im Inneren des Proteins, hydrophile und geladene Seitenketten liegen an der Proteinoberfläche • im Inneren des Proteins sind keine H2O Moleküle vorhanden • falls polare (geladene) Seitenketten im Inneren des Proteins vorhanden sind, treten sie paarweise auf, um miteinander Dipol-Dipol Wechselwirkungen oder H-Brücken (ionische Wechselwirkungen) auszubilden • falls unpolare Seitenketten an der Proteinoberfläche vorhanden sind, dienen sie zur Bindung unpolarer Moleküle oder der Ausbildung von Quartärstrukturen 95 Strukturen Globulärer Proteine (Beispiele) Humanes Serum Albumin (HSA) Grün fluoreszierendes Protein (GFP) Alkohol Dehydrogenase UDP-GlcNAc Acyltransferase 96 3.6. Bestimmung der 3D Strukturen von Proteinen Methode Röntgenkristallografie NMR Spektroskopie Vorteile Nachteile Analyse großer Proteine und Protein muß als Kristall Proteinkomplexe (>100 kDa) vorliegen; möglich statische Struktur Protein liegt in Lösung vor; Nur Proteine <100 kDa können Dynamische analysiert werden Strukturänderungen können erfasst werden 97 Bestimmung der Protein 3D Struktur (I) Röntgenkristallografie γ-Strahlung (X-rays) Lichtmikroskopisches Bild eines Proteinkristalls Modell der 3D Struktur des Proteins Softwaregestützte Datenauswertung Diffraktionsbild: Position und Intensität der Signale enthält gesamte Information über 3D Anordnung aller Atome des Proteins 98 Bestimmung der Protein 3D Struktur (II) Kernresonanzspektroskopie (NMR) Softwaregestützte Datenauswertung Modell der 3D Struktur des Proteins NOESY (Nuclear Overhauser Effect Spectroscopy) Spektrum enthält information darüber, welche Aminosäurereste benachbart sind 99 3.7. Proteinfaltung Die Hauptriebkraft für die Proteinfaltung ist der hydrophobe Effekt Die 3D Struktur eines Proteins ist in seiner Aminosäuresequenz codiert (Chr. Anfinsen, 1957) Proteine können (oft reversibel) entfaltet werden durch - Temperaturerhöhung (Erhöhung der Molekülschwingung) - Extreme pH-Werte (Aufhebung ionischer Anziehung, Erzeugung ionischer Abstoßung) hydrophil - Detergentien (“Solubilisierung” der hydrophoben Inneren) hydrophob - Chaotrope Moleküle (Störung der Wasserstruktur) Guanidinium Harnstoff (engl. Urea) 100 De- und Renaturierung der RNase A (Christian Anfinsen, 1957) (active) (inactive) (active) 101 Allgemeine Prinzipien der Faltungswege für Proteine: 1. Bildung lokaler Sekundärstrukturen (schnell: ms) 2. Zusammenlagerung von Sekundärstrukturen über hydrophoben Effekt (hydrophobic collapse) → “molten globule” (langsam: s) 3. Reorganisation der Sekundärstrukturen, bis die thermodynamisch günstigste Konformation (= native Konformation) erreicht ist (langsam: s – min) 102 Proteinfaltungsweg (Hypothese) denaturiert / Zufallsknäul (“random coil”) langsam “molten globule” schnell (~5 ms) langsam “hydrophober Kollaps” “molten globule” lokale Sekundärstrukturen langsam langsam (~1000 ms) nativ / aktiv 103 “Energietrichter” der Proteinfaltung DG Freie Enthalpie Proteinkonformationen 104 Chaperone (“Faltungshelfer”) sind Proteine, welche die Faltung anderer Proteine dadurch begünstigen, indem sie die Bildung metastabiler Faltungsintermediate verhindern; - Chaperone benötigen Energie (aus ATP Hydrolyse) für ihre Funktion - Chaperone binden das Faltungssubstrat reversibel - Chaperone besitzen keine Information über die native Strukturen ihrer Faltungssubstrate 105