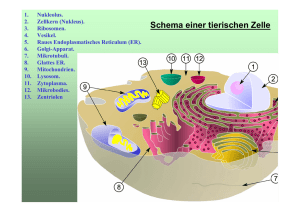
Ribosomen
Werbung

Ribosomen Entstehung: Im Nucleolus („Kernkörperchen“) wird ribosomale RNA (rRNA) gebildet, sowie die ribosomalen Proteine der Ribosomen, die sich mit der rRNA zu den Untereinheiten zusammensetzen. Die Untereinheiten verlassen den Kern durch die Kernporen und lagern sich im Cytoplasma zusammen, jedoch erst zu Beginn der Proteinbiosynthese. Vorkommen: Ribosomen kommen bei prokaryotischen Zellen frei im Cytosol verstreut vor. Bei eukaryotischen Zellen sind sie zusätzlich noch an die Membran des rauen Endoplasmatischen Reticulums gebunden. Ihre Anzahl schwankt von etwa 104 Ribosomen in Prokaryoten-Zellen bis zu 105 bis 107 Ribosomen in Eukaryoten-Zellen. Eukaryotische Plastiden und Mitochondrien enthalten ebenfalls Ribosomen. Diese entsprechen jedoch in ihrem Aufbau denen der Prokaryoten. Dies ist auf die Endosymbiontentheorie zurückzuführen, nach der Mitochondrien und Plastiden durch symbiotische Aufnahme eines Prokaryoten entstanden sind. Aufbau: Ribosomen gehören zu den Zellorganellen, die nicht von einer Membran umgeben sind. Sie bestehen aus zwei Untereinheiten, die sich erst bei der Proteinbiosynthese zu einer funktionellen Einheit zusammenlagern. Ribosomen haben eine globuläre Gestalt und einen Durchmesser von etwa 25nm-30nm. Prokaryotische 70S-Ribosomen große 50S-Untereinheit kleine 30S-Untereinheit Eukaryotische 80S-Ribosomen große 60S-Untereinheit kleine 40S-Untereinheit „S“ = Sedimentationskoeffizient: Geschwindigkeit mit der sich die RNA beim Zentrifugieren unter Einwirkung der Zentripetalkraft ablagert. Ribosomen bestehen aus ribosomaler RNA und Proteinen. Wenn sie sich bei der Proteinbiosynthese zusammenlagern, bildet sich zwischen großer und kleiner Untereinheit ein Tunnel, das sog. Peptidyltransferasezentrum. Dort erfolgt die durch Ribozyme katalysierte Verknüpfung der Aminosäuren zur Peptidkette. Es ist auch der Ort, an dem die mRNA lokalisiert ist. Der Tunnel schützt die entstehende Peptidkette vor Proteasen, die jede lineare Peptidkette als defektes Protein erkennen und sie somit abbauen würden. Bestimmte Antibiotika können den Tunnelausgang blockieren und dadurch die Proteinbiosynthese der Bakterien verhindern. Durch den unterschiedlichen Aufbau von eukaryotischen und prokaryotischen Ribosomen können die bakteriellen Ribosomen sicher erkannt werden. Polysomen sind die Gesamtheit aller an einem mRNA-Molekül sitzenden Ribosomen. Aufgaben: Während der Proteinbiosynthese vermitteln die Ribosomen die Bindung zwischen den tRNA-Anticodons und den mRNA-Codons. Die große und die kleine Untereinheit vereinigen sich jedoch erst zu einem Ribosom wenn sie an die mRNA binden. 1 Die rRNA ist die Hauptkomponente der Kontaktregion und der tRNA-Bindungsstellen. Sie führt die ribosomalen Funktionen aus. Die Proteine spielen dagegen hauptsächlich eine strukturelle Rolle. Die tRNA transportiert die Aminosäuren (AS) zum Ribosom. Dabei hat jede tRNA ein Anticodon welches an sein komplementäres Codon auf der mRNA bindet. Manche tRNAs erkennen und binden jedoch mehrere Codons (Wobble-Hypothese: Basenpaarung zwischen der ersten Base des Anticodons und der dritten Base des Codons weniger exakt als bei Transkription & Replikation). Bei Inosin handelt es sich um ein modifiziertes Nucleotid. Es ist die erste Base des Anticodons mancher tRNAs, und kann an U, A und C binden! Das Enzym Aminoacyl-tRNA-Synthetase (AtRNAS) sorgt für die Bindung der korrekten Aminosäure an die entsprechende tRNA unter ATP-Verbrauch. Es gibt 20 verschiedene AtRNAS, also eine für jede AS. 2/3 der Masse des Ribosoms besteht aus rRNA. Die Ribosomen vermitteln die Bindung des tRNA-Anticodons an sein komplementäres mRNA-Codon. Im Ribosom gibt es drei Bindungsstellen für die tRNA: Die A- (Aminoacyl-tRNA)-, P(Peptidyl-tRNA-) und die E- (Exit-) Stelle sowie eine Bindungsstelle für die mRNA. Die Translation besteht aus: Initiation, Elongation und Termination. Jeder Schritt wird dabei von Enzymen unterstützt und verbraucht GTP. 1. Initiation: Die kleine Untereinheit bindet an die mRNA, an das Startcodon AUG bindet die tRNA mit Methionin. Die große Untereinheit bindet ebenfalls an die mRNA und es ist ein funktionstüchtiges Ribosom entstanden. 2. Elongation: Pro Zyklus wird eine neue AS an die Peptidkette gebunden. Die Ausbildung der Peptidbindung wird dabei durch die rRNA katalysiert ( Ribozym) 3: Termination: Die Elongation endet wenn eines der Stop-Codons (UAA, UAG, UGA) erreicht wurde. Ein bestimmtes Protein (der Release-Factor) bindet an dem Stop-Codon an der A-Stelle und spaltet die Bindung zwischen dem entstandenen Polypeptid und der tRNA. Die mRNA und das Polypeptid werden freigesetzt und das Ribosom dissoziiert wieder in die kleine und die große Untereinheit. Bei den Prokaryoten erfolgen Transkription und Translation gleichzeitig, da sie keinen Zellkern besitzen. Bei Eukaryoten jedoch erfolgen Transkription und Translation getrennt voneinander, denn sie besitzen einen Zellkern. Dies bietet der Zelle die Möglichkeit ihre kompliziert miteinander verbunden Aktivitäten besser zu kontrollieren. Quellen: Campbell, Neil A.: Biologie, Pearson Studium, 6. Auflage, 2006 Probst, W. (Hg.): DUDEN Biologie. Gymnasiale Oberstufe, Duden Paetec Schulbuchverlag, 1. Auflage, 2005 Fritsche,W.: Mikrobiologie, Spektrum, 2. Auflage, 1999 Bielka, H. und Börner T.: Molekulare Biologie der Zelle, Gustav Fischer Verlag, 1995 www.riboworld.com (Stand: 01.11.08 15:00 Uhr) 2