2 - Friedrich-Schiller
Werbung
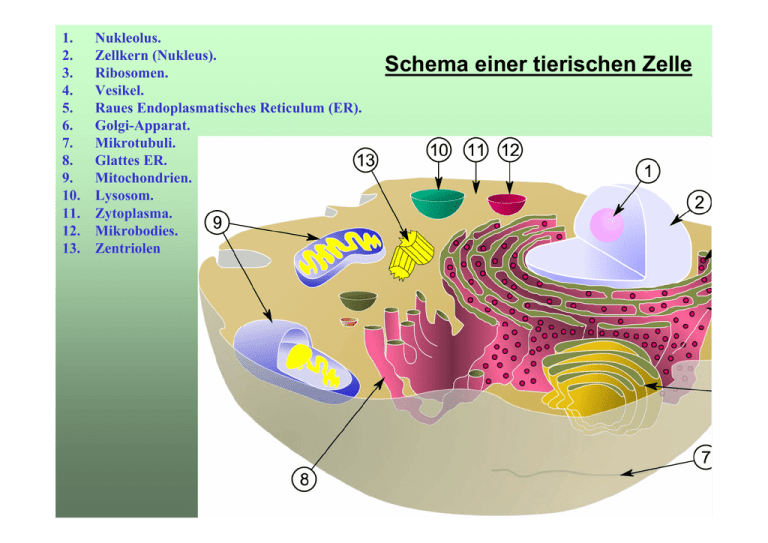
1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. 12. 13. Nukleolus. Zellkern (Nukleus). Ribosomen. Vesikel. Raues Endoplasmatisches Reticulum (ER). Golgi-Apparat. Mikrotubuli. Glattes ER. Mitochondrien. Lysosom. Zytoplasma. Mikrobodies. Zentriolen Schema einer tierischen Zelle Friedrich-Schiller-Universität Jena Biologisch-Pharmazeutische Fakultät Institut für Allgemeine Zoologie und Tierphysiologie Biologie für Mediziner PD Dr. Reinhard Predel Zellbiologie Ribosomen Endoplasmatisches Retikulum/Golgi-Apparat Ribosomen: Proteinbiosynthese große Untereinheit DNA Kopie= mRNA kleine Untereinheit Ribosom mRNA tRNA Aminosäure Protein Struktur der großen (50S) Untereinheit eines bakteriellen Ribosoms. Proteine blau, RNA beige. Ribosomen • dienen der Proteinsynthese • bestehen aus Ribonucleoproteinen • als ribosomale Untereinheiten frei im Zytoplasma • zur Translation lagern sie sich zu kompletten Ribosomen zusammen. • als Polysomen im Zytoplasma oder aufgelagert auf das ER • an einem Polysom gebildeten Proteine sind alle identisch! Der Nucleolus enthält drei morphologisch unterscheidbare Regionen: 1) Fibrilläre Zentren: enthalten die rRNA-Gene und die Transkriptionsmaschinerie 2) Dichte fibrilläre Komponente: umgibt die fibrillären Zentren, die Transkription der rRNA findet zwischen fibrillärem Zentrum und der dichten Fibrillären Komponente statt 3) Granuläre Komponente: hier werden die ribosomalen Untereinheiten zusammengebaut Der Nucleolus - der Nucleolus ist die prominenteste subnukleäre Domäne - der Nucleolus ist strukturell und funktionell gut charakterisiert - im Nucleolus konzentrieren sich die transkriptionell aktiven Gene, die für die rRNA kodieren - Syntheseort der rRNA: Transkription und Prozessierung - die rRNA wird im Nucleolus mit den im Zytoplasma synthetisierten ribosomalen Proteinen zu den beiden ribosomalen Untereinheiten zusammengebaut - die Ribosomenuntereinheiten werden dann ins Zytosol exportiert - erst im Zytosol assoziieren die Untereinheit zu funktionellen Ribosomen Zusammenbau Ribosomenunterheiten im Zellkern Translation Transfer-RNA (tRNA) Aminosäure Esterbindung Akzeptor TΨC-Arm D-Arm intramolekulare Basenpaarung Anticodon mRNA Anticodon-Schleife tRNA ¾ 73-93 Nucleotide ¾ Kleeblattstruktur durch A) komplementäre Sequenzen für Wasserstoffbrückenbindung B) modifizierte Basen (Schleifenbildung) ¾ Spezifische Aminosäure wird immer an ein Adenosin des Akzeptorarms gebunden ¾ Übersetzung des mRNA-Tripletts (Codons) über Anticodonschleife in Aminosäuresequenz ¾ Beladung erfolgt mit Aminosäure-spezifischer Aminoacyl-tRNA-Synthetase Kopplung der Aminosäuren an die tRNA tRNA-aa high-energy bond ATP amino acid AMP active site tRNA Synthetase mRNA tRNA - Aminoacyl-tRNA-Synthetasen beladen jede tRNA mit genau der Aminosäure, für die ihr Anticodon kodiert - Unter ATP-Hydrolyse entsteht eine energiereiche Bindung zwischen tRNA und Aminosäure. Diese Energie wird später zur Proteinsynthese verwendet. Proteinbiosynthese: Initiationsphase 43 S Pre-initiation Komplex Me t ¾ IF-1 blockiert A-Position Initiatoramino acid tRNA ¾ IF-3 blockiert Bindung an große Untereinheit ¾ IF-2 (+GTP) incl. Initiatoramino acid tRNA bindet an P-Position kleine Untereinheit des Ribosoms eIF: GTP: E: P: A: eucaryotic Initiation Factor Guanosintriphosphat Exitposition Peptidylposition Aminosäureposition Pre-initiation Komplex bereit für Erkennen der AUG-Sequenz des Startcodons der mRNA Proteinbiosynthese: Initiationsphase mRNA-Bindung an kleine Untereinheit Me t ¾ 43 S Komplex bindet mRNA Initiatoramino acid tRNA ¾ IF-2 hydrolysiert GTP (zu GDP) ¾ IFs verlassen den Komplex und die große Untereinheit der Ribosomen bindet an die kleine Untereinheit E kleine Untereinheit des Ribosoms eIF: GTP: E: P: A: eucaryotic Initiation Factor Guanosintriphosphat Exitposition Peptidylposition Aminosäureposition 80 S Initiation Komplex Proteinbiosynthese: Initiationsphase Me t große Untereinheit 80 S Initiation Komplex E mRNA kleine Untereinheit Proteinbiosynthese: Elongationsphase Anfügen weiterer Aminosäuren ¾ EF-1 bringt weitere tRNAs Me t zur A-Position 1 ¾ sterische Gründe bedingen, dass nur tRNAs mit korrektem Anticodon binden ¾ EF-1hydrolysiert GTP und verläßt den Komplex E EF: P Elongation Factor A Proteinbiosynthese: Elongationsphase Me t Anfügen weiterer Aminosäuren -2 E EF: P Elongation Factor A ¾ EF-2 bindet, hydrolysiert und die damit verbundene Konformationsänderung bedingt eine Annäherung der A- und PPositionen im katalytischen Zentrum ¾ Transfer der Aminosäure 1 zu Aminosäure 2: Entstehen eines Dipeptides Peptidbindung (Peptidyltransferase) Aminosäure H H H H HOOC -C -NH2 HOOC -C -NH2 HOOC -C -NH2 HOOC -C -NH2 R1 R2 H HOOC -C -NH R1 Carboxyterminus H2O R3 R4 O H O H O H C -C -NH C -C -NH C -C -NH2 R2 H2O R3 H2O R4 Aminoterminus Proteinbiosynthese: Elongationsphase Me t Anfügen weiterer Aminosäuren E P A ¾ EF-2 bindet, hydrolysiert und die damit verbundene Konformationsänderung bedingt eine Annäherung der A- und PPositionen im katalytischen Zentrum ¾ Transfer der Aminosäure 1 zu Aminosäure 2: Entstehen eines Dipeptides Proteinbiosynthese: Elongationsphase Me t Anfügen weiterer Aminosäuren E P A ¾ entladene tRNA verläßt Ribosom über die E-Position ¾A-Position frei für folgende tRNA Me t Proteinbiosynthese: Termination release factor = Protein stop codon Release factor erkennt stop codon, bindet und löst (+ H2O) Polypeptid von tRNA: Ribosom zerfällt in Untereinheiten Proteinbiosynthese: Termination ? M A S N G G H L Q R H C N P V R K K S G F A Y M W D K E H I Signalsequenz Ja: Nein: Q T V Cytosolische Proteine Proteine/Membranproteine für Zellkern, Peroxisomen und Mitochondrien ribosomale Proteine freie Ribosomen Ribosomen an das Endoplasmatische Retikulum gebunden Synthese von Exportproteinen (Hormone, Verdauungsenzyme) Synthese von lysosomalen Proteinen Synthese von integralen Membranproteinen Proteinreifung generell im Endomembransystem Zellorganellen sekretorischer Weg Hormon Plasmamembran Vesikel Golgi-App. RER Hormone Prohormone z.B. Preprohormone (Ribosom) mRNA Zellkern DNA Zellkern ER schematisch rER Ribosomen sER glattes endoplasmatisches Retikulum in Skelettmuskelfasern der Maus raues endoplasmatisches Retikulum aus einer exokrinen Drüsenzelle von Lumbricus Synthese eines sekretorischen Proteins SRP - signal recognition particle BiP - eines der wichtigsten Hsp70-Proteine im endoplasmatischen Retikulum Synthese eines sekretorischen Proteins 1. Synthese jedes Polypeptids beginnt am freien Ribosom 2. Bei Synthese einer Signalsequenz bindet diese im Cytosol an ein SRP (Signal Recognition Particle): Unterbrechung Proteinsynthese 3. Bindung an endoplasmatisches Retikulum (RER) durch zwei Wechselwirkungen: SRP + SRP-Rezeptor und Ribosom + Translocon 4. SRP löst sich, Signalsequenz bindet im Kanal des Translocons 5. Wiederaufnahme der Translation, das anwachsende Polypeptid gelangt in das Lumen des RER 6. Nach Beendigung der Synthese wird das Ribosom wieder frei und das Signalpeptid wird von einem Membranprotein abgespalten Synthese eines integralen Membranproteins 2 1 3 1: hydrophobe Stop-Transfer Sequenz verhindert weitere Verschiebung des Polypeptids 2: Verbleibender Teil des Polypeptids wird ins Cytosol synthetisiert 3: Einbau der hydrophoben Sequenz in Membran Im Lumen des Endoplasmatischen Retikulums wird des entstehende Protein glycosyliert und in die korrekte Raumstruktur gefaltet Proteinglykosylierung im rER Lipidträger Proteinglykosylierung im rER 1: Beladung des Dolicholdiphosphats mit 2 Molekülen N-Acetylglucosamin (Cytosolseite) 2: Beladung des Lipidträgers (Dolicholdiphosphats )mit 5 Molekülen Mannose (Cytosolseite) 3: Lipidträger „dreht“ die Zuckerreste ins Lumen des RER 6: Beladung des Lipidträgers mit weiteren 4 Molekülen Mannose (Lumenseite) 9: Beladung des Lipidträgers mit 3 Molekülen Glucose (Lumenseite) 10: Übertragung des Zuckers durch Oligosaccharyltransferase auf Asparagin der Polypeptidkette