Das Ribosom
Werbung

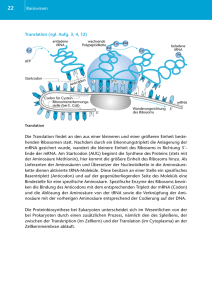
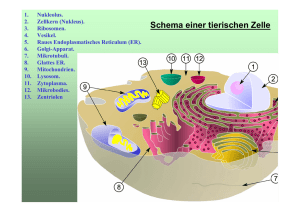
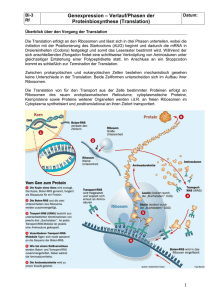
Das Ribosom Ribosomen vermitteln während der Proteinsynthese die spezifische Bindung der tRNA-Anticodons mit den Codons der mRNA. Diese eiförmigen Zellorganellen besitzen einen Durchmesser von etwa 25 nm und sind daher elektronenmikroskopisch gut zu erkennen. Ein Ribosom besteht aus zwei Teilen, die man als große und kleine Untereinheit bezeichnet (Abbildung 16.12a). Jede der Untereinheiten besteht aus zahlreichen Proteinen und wird bei Eukaryoten im Nucleolus zusammengebaut (siehe Kapitel 7). Die fertigen Untereinheiten werden durch die Kernporen in das Cyto-plasma ausgeschleust. Zum funktionellen Ribosom vereinigt sich die große Untereinheit mit der kleinen erst dann, wenn letztere ein mRNA-Molekül gebunden hat. Ein Ribosom besteht nicht nur aus zahlreichen Proteinen, sondern auch aus einem weiteren RNA-Typ, der als ribosomale RNA (rRNA) bezeichnet wird. Diese RNA gliedert sich in drei Moleküle und macht ungefähr 60 Prozent der Masse eines Ribosoms aus. Weil die meisten Zellen Tausende von Ribosomen enthalten, ist die rRNA der häufigste RNA-Typ in der Zelle. Obwohl sich die Ribosomen von Prokaryoten und Eukaryoten in Struktur und Funktion sehr ähneln, sind die der Prokaryoten etwas kleiner und unterscheiden sich von den eukaryotischen in ihrer Zusammensetzung. Diese Unterschiede sind von großer medizinischer Relevanz. Einige Hemmstoffe können die prokaryotischen Ribosomen in ihrer Funktion hemmen, ohne die Proteinsynthese der eukaryotischen Zelle zu beeinträchtigen. Diese Hemmstoffe, beispielsweise Tetrazyklin und Streptomycin, werden als Antibiotika zur Bekämpfung bakterieller Infektionen eingesetzt. Die Struktur eines Ribosoms spiegelt seine Funktion wider: Es bringt mRNA mit aminosäurebeladener tRNA zusammen. Neben einer Bindungsstelle für die mRNA hat jedes Ribosom zwei Bindungsstellen für tRNAs (Abbildung 16.12b). Die P-Stelle (Peptidyl-tRNA-Stelle) bindet die tRNA mit der wachsenden Polypeptidkette. während die AStelle (Aminoacyl-tRNA-Stelle) diejenige tRNA aufnimmt, welche die neu anzuknüpfende Aminosäure anliefert. Wie eine Klammer hält das Ribosom die tRNA- und mRNAMoleküle eng beieinander und katalysiert die Anheftung einer Aminosäure an die freie Carboxylgruppe der wachsenden Polypeptidkette (16.12c) Die Synthese eines Polypeptids Wir können die Translation, die Synthese eines Polypep-tides, in drei Abschnitte gliedern: Initiation, Elongation und Termination einer Aminosäurekette. Alle drei Einzelschritte benötigen Proteinfaktoren (meist Enzyme), welche die mRNA, die tRNA und die Ribosomen beim Translationsvorgang unterstützen. Jede Initiation und Elongation benötigen Energie, die durch GTP (Guanosin-Triphosphat), einem dem ATP verwandten Molekül, geliefert wird. Initiation Bei der Initiation der Translation werden die mRNA, eine tRNA mit der ersten Aminosäure des Polypeptids und die beiden ribosomalen Untereinheiten zusammengebracht. Die kleine ribosomale Untereinheit bindet an einer spezifischen Basensequenz am 5'-Ende (stromaufwärts) einer mRNA. Unmittelbar danach (stromabwärts, also in Richtung des 3'-Endes; englisch: downstream) folgt das Initiationscodon, AUG, an dem die Translation beginnt. Die Initiator-tRNA, welche stets die Aminosäure Methionin trägt, bindet am Initiationscodon. Nach Vereinigung von mRNA, Initiator-tRNA und der kleinen ribosomalen Untereinheit tritt die große ribosomale Untereinheit hinzu, und ein funktionsfähiges Ribosom entsteht. Proteine, die man als „Initiationsfaktoren" bezeichnet, bringen die einzelnen Komponenten zusammen. Die Zelle benötigt Energie in Form eines GTP-Moleküls, um den Initiationskomplex zu bilden. Nach Beendigung des Initiationsvorganges befindet sich die Initiator-tRNA an der P-Stelle des Ribosoms, die freie A-Stelle ist bereit, die nächste tRNA aufzunehmen. Elongation Bei der Elongation wird eine Aminosäure nach der anderen an die Start-Aminosäure angehängt. Die Anheftung einer neuen Aminosäure, an der mehrere Proteine beteiligt sind, die sogenannten „Elongationsfaktoren", erfolgt in drei Schritten (Abbildung 16.14). (1) Codonerkennung. Das mRNA-Codon an der A-Stelle des Ribosoms bildet Wasserstoffbrückenbindungen mit dem Anticodon eines eintreffenden tRNA-Moleküls, das die passende Aminosäure trägt. Ein Elongationsfaktor schiebt die tRNA zur A-Stelle. Dieser Schritt benötigt die Hydrolyse einer Phosphatbindung des GTP. (2) Bildung einer Peptidbindung. Eine Komponente der gro ßen ribosomalen Untereinheit katalysiert die Bildung einer Peptidbindung zwischen dem Polypeptid, das an der P-Stelle hängt, und der gerade eingetroffenen Aminosäure an der A-Stelle. Bei diesem Schritt trennt sich das Polypeptid von der an die P-Stelle gebundenen tRNA und hängt nun über die neue Aminosäure an der AStelle. (3) Translokation. Die tRNA an der P-Stelle dissoziiert vom Ribosom ab. Die tRNA an der A-Stelle, die nun das wach sende Polypeptidkette trägt, wird an die P-Stelle verschoben. Diese Reaktion wird von einem sehr komplexen Enzym, der Peptidyltransferase, katalysiert. Bei diesem Stellungswechsel der tRNA bleibt ihr Anticodon mit dem mRNA-Codon durch Wasserstoffbrücken verbunden, weshalb die mRNA und die tRNA sich als Einheit weiterbewegen. Diese Translokation wiederum bringt das nächste zu transla-tierende Codon an die A-Stelle. Die Translokation erfordert Energie, die aus der Hydrolyse eines GTP-Moleküls stammt. Die mRNA bewegt sich also stets mit dem 5'-Ende voran durch das Ribosom, ähnlich einem Bohrer ohne Rückwärtsgang; umgekehrt könnte man sagen, das Ribosom wandert wie ein Schienenfahrzeug an der mRNA entlang, und zwar von 5' nach 3'. So oder so betrachtet, Ribosom und mRNA verschieben sich gegeneinander in festgelegter Richtung, Codon für Codon. Der Elongationszyklus dauert für jede Aminosäure ungefähr 60 Millisekunden und wird so lange wiederholt, bis die letzte Aminosäure an die Polypeptidkette angeknüpft ist. Termination Das letzte Stadium der Translation ist die Termination (Abbildung 16.15). Die Elongation setzt sich solange fort, bis ein Terminationscodon zur A-Stelle des Ribosoms kommt. Diese besonderen Basentripletts - UAA, UAG und UGA (siehe Abbildung 16.5) - codieren keine Aminosäure, sondern wirken als Stopsignale für die Translation. Ein als Freisetzungsfaktor oder ReleaseFaktor (vom englischen release für „freisetzen") bezeichnetes Protein besetzt direkt das Terminationscodon an der A-Stelle. Der Release-Faktor spaltet das fertige Polypeptid unter Addition eines Wassermoleküls von der letzten tRNA ab. Das Ribosom setzt die Polypeptidkette und die mRNA frei und dissoziiert anschließend wieder in seine große und kleine Untereinheit. Polysomen Ein einzelnes Ribosom könnte ein durchschnittlich großes Polypeptid in weniger als einer Minute synthetisieren. Von einem einzelnen mRNA-Molekül werden jedoch gleichzeitig mehrere Polypeptide translatiert, weil mehrere Ribosomen simultan an der Translation der mRNA arbeiten. Sobald ein Ribosom das Initiationscodon passiert hat, bindet ein zweites Ribosom an die mRNA. Daher wandert oft eine ganze Gruppe von Ribosomen an derselben mRNA entlang. Diese Ansammlung von Ribosomen, „Polyribosom" oder kürzer Polysom genannt, kann man im Elektronenmikroskop beobachten (Abbildung 16.16). Vom Polypeptid zum funktionsfähigen Protein Während und nach ihrer Synthese beginnt die Polypeptidkette, sich spontan räumlich zu falten, wobei sich die für das funktionsfähige Protein typische Konformation ausbildet: eine dreidimensionale Gestalt mit Sekundär- und Tertiärstruktur. Das Gen bestimmt die Primärstruktur, und die Primärstruktur wiederum bestimmt die Konformation. Eventuell werden weitere Schritte - „posttranslationale Modifikationen" – ausgeführt, damit das Protein seine spezielle Aufgabe in der Zelle erfüllen kann. Bestimmte Aminosäuren können durch die Anheftung von Zuckern, Lipiden, Phosphatgruppen oder anderen Kompnenten modifiziert werden. Enzyme können eine oder mehrere N-terminale (also am Amino-Ende gelegene) Aminosäuren der Polypeptidkette abspalten. In nicht wenigen Fällen wird eine gerade gebildete Polypeptidkette enzymatisch in zwei oder mehr Fragmente zerlegt. Das Protein Insulin bspw. zunächst als ein durchgehendes Polypeptid synthetisiert, dann wird dessen Mittelteil enzymatisch entfernt, und das zurückbleibende aktive Hormon besteht aus zwei Polypeptidketten, die durch Disulfidbrücken zusammengehalten werden. In anderen Fällen werden zwei oder mehr Polypeptide getrennt synthetisiert und assoziieren anschließend zum fertigen Protein. Man spricht dann von einer Quartärstruktur.