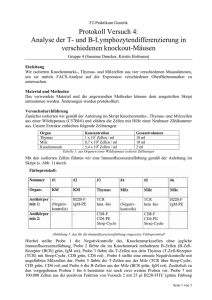
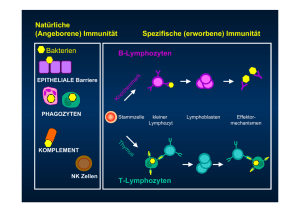
Kruhm_Michaela_Aspergillus_fumigatus
Werbung

Identifizierung und Isolierung Aspergillus fumigatus spezifischer T-Zell-Rezeptoren und funktionelle Charakterisierung nach Transfer auf humane T-Zellen Dissertation zur Erlangung des naturwissenschaftlichen Doktorgrades der Bayerischen Julius-Maximilians-Universität Würzburg vorgelegt von Michaela Kruhm aus Kassel Würzburg 2014 Eingereicht am: 14.08.2014 Mitglieder der Promotionskommission: Vorsitzender: Herr Prof. Dr. Engstler (Dekan) Gutachter: Herr Prof. Dr. Topp Gutachter: Herr Prof. Dr. Benz II Erklärung: Hiermit erkläre ich, dass ich die vorliegende Arbeit selbstständig verfasst habe. Es wurden nur die in der Arbeit angegebenen Hilfsmittel verwendet. Die Stellen, die dem Wortlaut oder Sinne nach anderen Arbeiten entnommen wurden, sind durch Angaben der Quellen kenntlich gemacht worden. Diese Arbeit hat weder in gleicher noch in ähnlicher Form in einem anderen Prüfungsverfahren vorgelegen. Ich erkläre weiterhin, dass ich, außer den mit dem Zulassungsgesuch urkundlich vorgelegten Graden, keine akademischen Grade erworben habe, noch versucht habe zu erwerben. Ich versichere, dass ich die vorstehenden Angaben nach bestem Wissen und Gewissen vollständig und der Wahrheit entsprechend gemacht habe. Datum, Unterschrift: 13.08.2014 III Die vorliegende Arbeit wurde in der Zeit vom 15.03.2008 bis 13.08.2014 an der medizinischen Klinik und Poliklinik II in der Abteilung für Translationale Immuntherapie der Bayerischen Julius-Maximilians-Universität Würzburg unter Anleitung von PD Dr. Max S. Topp angefertigt. IV Für meine Familie V Danksagung Mein besonderer Dank gilt Herrn Prof. Hermann Einsele und Prof. Max Topp für die Möglichkeit diese Arbeit anzufertigen und ihre Unterstützung während der Erstellung. Ferner möchte ich mich bei Prof. Dr. Roland Benz für die Begutachtung der Dissertation bedanken. Des Weiteren bedanke ich mich besonders bei den Doktoren Tanja Bedke und Carsten Berges für die wertvollen Hilfestellungen und Anregungen. Frau Dr. Claudia Stühler und Frau Dr. Nina Khanna für die wissenschaftlichen Diskussionen. Für die Informationen im Bereich des T-Zell-Rezeptor Transfers bedanke ich mich bei Dr. Daniel Sommermeyer und Dr. Michael Hudecek. Zudem gilt mein Dank Dr. Sabine Britting und Dr. Dragana Slavkoviv-Lukic für die hilfreiche Unterstützung und Beratung während der abschließenden Ausarbeitungen. Nicht zuletzt möchte ich mich bei meiner Familie und meinen Freunden für die Motivation und Unterstützung während der letzten Jahre bedanken. VI Inhaltsverzeichnis 1 Zusammenfassung ............................................................................................ 11 2 Summary ............................................................................................................ 13 3 Einleitung ........................................................................................................... 14 3.1 Der Pilz Aspergillus fumigatus....................................................................... 14 3.2 Angeborene und erworbene Immunität gegen Aspergillus fumigatus ............ 15 3.2.1 Angeborene Immunität gegen Aspergillus fumigatus ......................... 15 3.2.2 Erworbene Immunität gegen Aspergillus fumigatus ........................... 16 3.2.3 Strategien von Aspergillus fumigatus dem Immunsystem auszuweichen .................................................................................... 18 3.3 Klinische Relevanz von Aspergillus fumigatus Infektionen ............................ 19 3.3.1 Behandlungsstrategien von Aspergillus fumigatus Infektionen........... 19 3.4 Antigenerkennung durch T-Zellen ................................................................. 21 3.4.1 Haupthistokompatibilitätskomplex (MHC) .......................................... 21 3.4.2 αβ T-Zell-Rezeptor ............................................................................ 22 3.4.3 Die Umordnung der T-Zell-Rezeptor Gene ........................................ 24 3.4.4 αβ T-Zell-Rezeptor Komplex .............................................................. 25 3.4.5 αβ T-Zell-Rezeptor Signalübertragung ............................................... 26 3.5 T-Zell-Rezeptor Optimierungen und der T-Zell-Rezeptor Transfer auf T-Zellen................................................................................................... 26 4 Ziele der Arbeit................................................................................................... 29 5 Materialien .......................................................................................................... 31 5.1 Medien, Versuchssysteme, Chemikalien und Reagenzien ............................ 31 5.1.1 Kulturmedien und Zusätze ................................................................. 31 5.1.2 Versuchssysteme .............................................................................. 32 5.1.3 Chemikalien und Reagenzien ............................................................ 32 5.2 Hergestellte Medien und Puffer ..................................................................... 33 5.3 Crf1/p41 Peptid und MHC Klasse II Tetramer ............................................... 33 5.4 Fluoreszenz- konjugierte Antikörper für die Durchflusszytometrie ................. 34 5.5 Retroviraler Vektor pMP71 und Oligonukleotide ............................................ 34 5.5.1 Retroviraler Vektor pMP71 ................................................................ 34 5.5.2 Oligonukleotide .................................................................................. 35 5.6 Zelllinien und Bakterien ................................................................................. 36 5.6.1 Zelllinien ............................................................................................ 36 5.6.2 Bakterien ........................................................................................... 36 5.7 Verbrauchsmaterialien .................................................................................. 37 5.8 Verwendete Analyse- Computerprogramme ................................................. 37 VII 6 Methoden............................................................................................................ 39 6.1 Kultivierung humaner Zellen und Zelllinien .................................................... 39 6.1.1 Zellen und ihre Kultivierungsbedingungen ......................................... 39 6.1.2 Bestimmung der Zellzahl und Zellvitalität ........................................... 39 6.1.3 Einfrieren und Auftauen von Zellen .................................................... 40 6.2 Isolierung von Zellpopulationen..................................................................... 40 6.2.1 Isolation peripherer mononukleärer Zellen aus Vollblut gesunder Spender ............................................................................................. 40 6.2.2 Isolation von CD4+ T-Zellen via magnetischer Zell-Separation .......... 40 6.2.3 Isolation und Kultivierung von Monozyten .......................................... 41 6.2.4 Generierung dendritischer Zellen aus Monozyten .............................. 41 6.3 Stimulation und Klonierung von T-Zellen ....................................................... 41 6.3.1 Stimulation von T-Zellen und Generation A. fumigatus spezifischer T-Zelllinien ..................................................................... 41 6.3.2 Klonierung von Crf1/p41 spezifischen T-Zellen über Grenzverdünnung (limiting dilution) ................................................... 42 6.3.3 Expansion humaner T-Zellklone und T-Zell-Rezeptor transduzierter T-Zellen....................................................................... 42 6.4 Phänotyp-Analyse und Untersuchungen der Effektor-Funktion von T-Zellen .................................................................................................. 43 6.4.1 Restimulation von T-Zellen und T-Zell-Rezeptor transduzierten T-Zellen ............................................................................................. 43 6.4.2 Interferon-γ Enzym- Linked- Immunosorbent Assay .......................... 43 6.4.3 Durchflusszytometrische Messungen ................................................ 44 6.4.4 Bestimmung der T-Zell-Rezeptor β Kette von Crf1/p41 spezifischen Klonen mittels Durchflusszytometer .............................. 45 6.4.5 MHC Klasse II Tetramer Färbung ...................................................... 46 6.4.6 Messung der Proliferation nach Crf1/p41 spezifischer Stimulation ..... 46 6.4.7 Cytometric Bead Array (CBA) ............................................................ 46 6.5 Isolation und Identifizierung von T-Zell-Rezeptor Ketten ............................... 47 6.5.1 Isolation von RNA aus Crf1/p41 spezifischen T-Zellklonen ................ 47 6.5.2 Isolation und Identifikation unbekannter T-Zell-Rezeptor Sequenzen mittels RACE-PCR.......................................................... 48 6.5.3 Gelelektrophorese ............................................................................. 50 6.6 Klonierung retroviraler Vektoren und Transduktion in humane Zelllinien und primäre T-Zellen ..................................................................................... 50 6.6.1 Klonierung retroviraler Vektoren mit A. fumigatus spezifischen T-Zell-Rezeptoren ......................................................... 50 6.6.2 Produktion von retroviralem Kulturüberstand ..................................... 53 VIII 6.6.3 Transduktion von humanen Zelllinien und primären T-Zellen mit A. fumigatus spezifischen T-Zell-Rezeptoren..................................... 53 7 Ergebnisse ......................................................................................................... 55 7.1 Generierung A. fumigatus spezifischer T-Zellklone ....................................... 55 7.1.1 Expansion Crf1/p41 spezifischer T-Zellen.......................................... 55 7.1.2 Spezifitätstest der hergestellten T-Zellklone via Crf1/p41 Tetramer ........................................................................ 56 7.1.3 Interferon-γ Produktion der Crf1/p41 spezifischen T-Zellklone ........... 61 7.2 Identifizierung und Isolierung A. fumigatus spezifischer T-Zell-Rezeptoren ......................................................................................... 64 7.2.1 Identifizierung der T-Zell-Rezeptor β Ketten mit dem IO Beta Mark Kit (Beckman Coulter) .......................................... 64 7.2.2 Identifizierung der T-Zell-Rezeptor β Ketten durch SMARTer RACE PCR ....................................................................... 66 7.2.3 Identifizierung der T-Zell-Rezeptor α Ketten ...................................... 69 7.2.4 Isolierung der T-Zell-Rezeptor α und β Ketten und Klonierung in den retroviralen Vektor pMP71..................................... 70 7.3 Transfer von A. fumigatus spezifischen T-Zell-Rezeptoren auf Jurkat 76 Zellen ............................................................................................ 72 7.4 Transfer eines Codon- und Vektor- optimierten T-Zell Rezeptor auf Jurkat 76 ................................................................................................. 73 7.5 Transfer von A. fumigatus spezifischen T-Zell-Rezeptoren auf humane CD4+ T-Zellen .................................................................................. 76 7.5.1 Isolation und Expansion CD4+ T-Zellen mit optimiertem TCR 1 ......... 78 7.6 Proliferation und Zytokinproduktionsanalyse TCR 1 optimiert transduzierter T-Zellen nach Crf1/p41 spezifischer Stimulation ..................... 79 8 Diskussion ......................................................................................................... 83 8.1 Die T-Zellantwort von Crf1/p41 ist polyklonal ................................................ 83 8.2 Der endogene TCR verringert die Oberflächenexpression von transduzierten Crf1/p41 spezifischen T-Zell-Rezeptoren auf T-Zellen ........... 84 8.3 Optimierung des Vektors pMP71 (TCR 1 optimiert) führt zu einer erhöhten Expression von TCR 1 auf Jurkat 76 Zellen und primären CD4+ T-Zellen ................................................................................ 85 8.4 Die Spezifität und Funktionalität der Crf1/p41 spezifischen T-Zell-Rezeptor transduzierter auf CD4+ T-Zellen wurde bestätigt ................ 87 8.5 Einsatzmöglichkeiten funktioneller Crf1/p41 spezifischer T-Zell-Rezeptor transduzierter T-Zellen ........................................................ 88 8.6 Fazit und Ausblick ......................................................................................... 89 9 Literaturverzeichnis ........................................................................................... 91 IX 10 Anhang .................................................................................................................. i 10.1 Abkürzungen .........................................................................................i 10.2 Publikationen ....................................................................................... iv X Zusammenfassung 1 Der Zusammenfassung humanpathogene Pilz Aspergillus fumigatus (A. fumigatus) kann in immunsupprimierten Patienten zum Teil schwere invasive Infektionen auslösen. Trotz Fortschritten in den Behandlungsmöglichkeiten und der medikamentöser Prophylaxe bleibt die Sterblichkeitsrate bei invasiven Erkrankungen hoch. Aus diesem Grund ist die Entwicklung von spezifischeren Immuntherapien von Nöten. Ein Ansatz ist die genetische Modifikation von T-Zellen, durch den Transfer von A. fumigatus spezifischen T-Zell-Rezeptoren (TCRs), für eine adoptive Therapie. Um dieses Konzept zu evaluieren wurden TCRs, die für die extrazellulären Zellwandglykonase Crf1 (Crf1/p41) spezifisch sind, auf primäre T-Zellen transferiert und die Effektor-Funktion analysiert. Das Crf1/p41 Epitop induziert bei gesunden Spendern eine funktionelle TH1 Immunantwort gegen A. fumigatus und führt zur Produktion hoher Mengen von Interferon-γ (IFN-γ). Für die Identifikation von A. fumigatus spezifischen TCRs wurden siebenunddreißig Crf1/p41 spezifische T-Zellklone von drei HLA-DRB1*04 Spendern generiert. Anschließend wurden komplementaritätsbestimmende die Region TCR 3 β Ketten (CDR3) über bestimmt. die Es sehr variable konnten zwölf unterschiedliche TCRs ermittelt werden, von denen vor allem die variablen β (Vβ) Kette 18 sehr dominant, während die Vβ Ketten 1 und 6 nur in wenigen Klonen vertreten waren. Zur weiteren Charakterisierung der Crf1/p41 spezifischen TCRs wurden die variablen α (Vα) Ketten bestimmt (Vα 3, Vα 15 und Vα 26). Somit liegt eine polyklonale T-Zell Immunantwort vor. Anschließend wurden die Crf1/p41 spezifischen TCRs in den retroviralen Vektor pMP71 kloniert und auf Jurkat 76 Zellen, welche keinen endogenen TCR exprimieren, und auf primäre CD4+ T-Zellen transferiert. Die Expression von Crf1/p41 spezifischen TCRs, transduziert in CD4+ T-Zellen, zeigten spenderspezifische Unterschiede und die Expression war niedriger im Vergleich zu den transduzierten Jurkat 76 Zellen. Daher wurde auf Optimierungsstrategien zurückgegriffen, die für den adoptiven Transfer mit TCR-modifizierten T-Zellen zur Behandlung von Krebs entwickelt wurden. Angewandt wurden die Codonoptimierung der TCR codierenden Sequenz, Murinisierung der TCR konstanter Ketten, Induktion einer weiteren Disulfidbrücke. Ebenfalls wurde das Vektorsystem optimiert. Der Optimierungsprozess der Crf1/p41 spezifischen TCR 1 führte zu einer erhöhten Oberflächenexpression des TCR sowohl in Jurkat 76 (3 bis 5fach) als auch in primären CD4+ T-Zellen (2fach). In funktionellen Analysen wurde die Proliferationsfähigkeit und IFN-γ Produktion, durch die Stimulation von transduzierten CD4+ T-Zellen (TCR 1 optimiert) mit Crf1/p41 beladenen dendritischen Zellen (DCs), bestätigt. 11 Zusammenfassung Diese Ergebnisse weisen darauf hin, dass der Transfer von A. fumigatus spezifischen TCRs eine protektive anti-fungale Immunantwort fördern könnte. Demzufolge auch als ein geeignetes Mittel in einer potentiellen Immuntherapie gegen A. fumigatus Infektionen in immunsupprimierten Patienten, eingesetzt werden könnte. 12 Summary 2 Summary The human pathogenic fungus Aspergillus fumigatus (A. fumigatus) can cause severe invasive infections in immunosuppressed patients. Despite progresses in the treatment and prophylaxis of invasive infections the mortality rate remains high. On that account the development of a more specific immune therapy seems necessary. One approach is genetic modification of T-cells by transfer of A. fumigatus specific T-cell-receptors (TCR) for an adoptive therapy. To evaluate this concept, TCRs specific for a peptide derived from extracellular cell wall glucanase Crf1 (Crf1/p41) were transferred to primary T cells. Than their effector function was analyzed. In healthy donors the epitope Crf1/p41 induces a functional TH1 immune response towards A. fumigatus combined with a high Interferon-γ (IFN-γ) production. To identify A. fumigatus specific TCRs, thirty-seven Crf1/p41 specific T-cell clones were generated from three HLA-DRB1*04 positive healthy donors. Afterwards, the TCR β chains were analyzed by sequencing the most variable complementarity determining region 3 (CDR3). Twelve different TCRs were detected whereas variable β (Vβ) 18 was very dominant, and Vβ 1 and Vβ 6 were present only in some clones. For further characterization of Crf1/p41 specific TCRs, variable α (Vα) chains were identified (Vα 3, Vα 15 and Vα 26). Thus, the T-cell response is polyclonal. Subsequently Crf1/p41 specific TCRs were cloned into the retroviral vector pMP71 and transduced into Jurkat 76 cells that lack the expression of endogenous TCR, and into primary CD4+ T-cells. The expression of Crf1/p41 specific TCR transduced in CD4+ T-cells was donor-dependent and the expression was lower compared to transduced Jurkat 76 cells. Consequently, optimizing strategies engineered for TCR-modified adoptive T-cell transfer in cancer therapy were used to induce a higher TCR expression on T cells. Those strategies include codon-optimization of the TCR coding sequence, murinization of constant chains of TCR, induction of an additional disulfide bond. Additionally the vector system was optimized. The optimization process of Crf1/p41 TCR 1 led to a higher surface expression in Jurkat 76 (3-5x) and primary CD4+ T-cells (2x). Functional analyses revealed proliferation and IFN-γ production after the stimulation of transduced CD4+ T-cells (TCR 1 optimiert) with Crf1/p41 pulsed mature dendritic cells (mDC). These data suggest that the transfer of A. fumigatus specific TCRs might foster protective anti-fungal immune responses. Therefore this might be a suitable tool immunotherapeutic use against A. fumigatus infections in immunosuppressed patients. 13 for Einleitung 3 Einleitung 3.1 Der Pilz Aspergillus fumigatus Der Pilz Aspergillus fumigatus (A. fumigatus) gehört zu den Schimmelpilzen (Phylum Ascomycota (Schlauchpilze)) und ist ein weltweit verbreiteter Saprobiont. Die Gattung Aspergillus (Gießkannenschimmel) besitzt einen Aspergillus-förmigen Sporenträger (Konidiophore), deren Konidien klein (2-3 µm) und widerstandsfähig gegenüber extremen Temperaturen und Dehydratisierung sind. Kommt es zur Keimung der Konidien, so bilden sich zunächst einzelne Hyphen, die sich zu einem Myzel verzweigen. An deren Oberfläche bilden sich einzelne Konidophoren, die wiederum 10.000 neue Konidien bilden und sich als Bestandteil des Aerosols der Luft verteilen (45-110 Sporen m3, maximale Messung 100.000 Sporen m3) (Abbildung 1). Der Pilz besitzt die Fähigkeit, auf vielen verschiedenen Substraten bei einem breiten Spektrum von Umweltbedingungen zu leben, so zum Beispiel auch auf lebendem und totem Gewebe von Menschen und Tieren (KwonChung et al. 2013). Abbildung 1: Konidiophore und Hyphe von A. fumigatus. Eine Rasterelektronenmikroskopische Aufnahme der Konidiophoren von A. fumigatus (Latge 2001) (A). Schematische Darstellung der Konidiophore und der Hyphe von A. fumigatus (Romani 2004) (B). A. fumigatus gehört zu den humanpathogenen Pilzen (Latge 2001; Romani 2011; KwonChung et al. 2013). Ein gesundes Immunsystem verhindert in der Regel eine Infektion, jedoch können Allergien (bronchopulmonale Aspergillose), Aspergillome und insbesondere bei immunsupprimierten Patienten eine invasive Aspergillose durch den Pilz ausgelöst werden (siehe 3.3). 14 Einleitung 3.2 Angeborene und erworbene Immunität gegen Aspergillus fumigatus Die Immunabwehr kann in zwei voneinander abhängige Bereiche unterteilt werden. Zum einen die angeborene Immunität und zum anderen die erworbene Immunität. Die Mechanismen der angeborenen Immunität sind vor allem die physiologischen Barrieren (z.B. Epithelien), die Opsonisierung durch das Komplementsystem, die zellvermittelte Phagozytose (z.B. durch Makrophagen und Granulozyten) und die natürlichen Killerzellen (NK), die eine Apoptose induzieren können. Die erworbene Immunität beinhaltet die humorale und zellvermitteltende Immunität, verbunden mit der Fähigkeit ein immunologisches Gedächtnis auszubilden. Die Zellen der erworbenen Immunität sind T-Lymphozyten und B-Lymphozyten. Die Stimulation von angeborenem Immunsystem aktiviert das erworbene Immunsystem, welches wiederum die protektiven Mechanismen des angeborenen Immunsystems verstärken (Brown 2011; Romani 2011; Wuthrich et al. 2012). 3.2.1 Angeborene Immunität gegen Aspergillus fumigatus Eingeatmete Aspergillus Konidien werden in erster Instanz durch die Barrierefunktion der Haut und der mukosalen Epithelzellen der Oberfläche des Respirationssystems bekämpft. Wesentliche Abwehrmechanismen sind die Opsonisierung, durch das Komplementsystem, sowie die Sekretion von Defensin und Kollektin, welche eine vielfältige mikrobielle Aktivität gegen Bakterien und Pilzen besitzen (Alekseeva et al. 2009; Romani 2011). Konidien, die der Phagozytose und Zerstörung durch alveolare Makrophagen entkommen, können sprossen und zu Hyphen auswachsen. Neutrophile Granulozyten (polymorphkernige Leukozyten) können Konidien, sprossende Konidien und Hyphen attackieren. Von den neutophilen Granulozyten werden bevorzugt Pilzstrukturen von Hyphen phagozytiert, welche effektiv in intrazellulären Vesikeln zerstört werden (Schaffner et al. 1982). Dies wird mit Hilfe von Enzymen, die in der Granula gespeichert werden, erreicht. Sind die Pilzstrukturen zu groß für eine Phagozytose, produzieren und setzen neutrophile Granulozyten toxische Substanzen frei, wie zum Beispiel Sauerstoffderivate, Stickoxid und mikrobiell aktive Peptide (Defensine und kationische Proteine) (Brown 2011). Für Patienten mit anhaltender Neutropenie ist das Risiko hoch an einer Aspergillus Infektion zu erkranken. Dies weist auf die Wichtigkeit, der durch neutrophile Granulozyten vermittelte Immunantwort, zur Bekämpfung von Aspergillus Infektionen, hin (Marr et al. 2002). Für die Erkennung der sogenannten Pathogenassoziierten molekularen Muster (PAMPs; pathogen-associated molecular patterns) sind die Mustererkennungs- Rezeptoren (PRRs; pattern recognition receptor), wie Toll-like Rezeptoren (TLRs), C-Typ Lektin Rezeptoren (CLRs) und Proteine der Galektin Familie 15 Einleitung zuständig (van de Veerdonk et al. 2008). PAMPs sind vor allem Strukturen der fungalen Zellwand, wie β-Glucane, Chitin und Mannane sowie fungale Nukleinsäuren. Nach der Erkennung der PAMPs durch die PRRs werden, über Signaltransduktion, Immunzellen aktiviert, was zur Phagozytose der Konidien und zur Produktion inflammatorischer Zytokine und Chemokine führt. Diese wiederum verstärken die Aktivierung weiterer Immunzellen der angeborenen Immunität, wie polymorphkernige Leukozyten (PMN) und dendritische Zellen (DC) am Ort der Infektion (Gersuk et al. 2006). Dabei verbinden die DCs die Mechanismen der angeborenen mit der erworbenen Immunität, indem sie durch PRR-vermittelte Zytokinausschüttung die T-Zell Immunantworten beeinflussen (Wuthrich et al. 2012). 3.2.2 Erworbene Immunität gegen Aspergillus fumigatus Neben der angeborenen Immunität hat sich die erworbene Immunität als ein wichtiger Faktor in der Bekämpfung von A. fumigatus Infektionen erwiesen. In Tiermodellen konnte durch die Restimulation von immunisierten Tieren mit A. fumigatus Konidien oder anderen fungalen Antigenen ein Immungedächtnis nachgewiesen werden (Cenci et al. 1997; Cenci et al. 2000; Ito et al. 2006). Auch der adoptive Transfer von T-Zellen immunisierter Tiere wirkte lebensverlängernd auf infizierte Tiere (Cenci et al. 2000). Lymphozyten gesunder Menschen, stimuliert mit A. fumigatus Antigenen, besitzen die Fähigkeit zu proliferieren und Zytokine wie Interferon-γ (IFN-γ) zu produzieren, was eine erworbene Immunität und ein Immungedächtnis nachweist (Grazziutti et al. 1997; Hebart et al. 2002; Beck et al. 2006). Die adaptiven Immunantworten und das Immungedächtnis gegen A. fumigatus werden durch DCs induziert, welche mit Bestandteilen von Konidien und Hyphe beladenen sind. Die beladenen DCs initiieren die Differenzierung verschiedener T-Helferzellen (TH). Abhängig vom Aktivierungsprofil der DC und vom Immunstatus des Wirtes können CD4+ T-Zellen zu T-Helferzellen Typ 1 (TH1), T-Helferzellen Typ 2 (TH2), T-Helferzellen Typ 17 (TH17) oder regulatorischen T-Zellen (Treg) differenzieren (Romani 2011; Wuthrich et al. 2012) (Abbildung 2). T-Zellen der erworbenen Immunität sind vor allem für die Regulation der Balance zwischen toleranzbildenden und inflammatorischen Immunantworten verantwortlich (Romani et al. 2007; Romani 2008). Insbesondere TH1 Immunantworten spielen eine entscheidende Rolle in der Immunität gegen A. fumigatus. Die pro-inflammatorischen Zytokine Interleukin- (IL)-12 und Tumor-Nekrose-Faktor α (TNF-α) induzieren eine Differenzierung von TH1 Zellen, welche Interferon-γ (IFN-γ) und koloniestimulierende Faktoren (CSF) ausschütten. Durch IFN-γ und CSF werden die Zellen der angeborenen Immunität stimuliert, welche die Infektion bekämpfen (Romani 2008; Romani 2011). 16 Einleitung TH2 Immunantworten können zu fungaler Allergie führen oder sogar die Infektion verstärken. Dabei zeigen Patienten mit einem Rückfall oder einer Verstärkung der Infektion oft eine schwache IFN-γ Produktion mit einer erhöhten Produktion von TH2 Zytokinen, wie IL-4 und IL-10. Zudem kommt es zu einer erhöhten Ausschüttung von IgE Antikörpern und Eosinophilen (Hebart et al. 2002; Chaudhary et al. 2010). Erhöhte TH2 Antworten, ausgelöst durch A. fumigatus, führen oft zu einer Verschlechterung der invasiven Erkrankung, aufgrund der damit verbundenen Reduktion der T H1 Immunantworten (Hebart et al. 2002; Romani 2011). TH17 sind weitere Zellen der erworbenen Immunität gegen A. fumigatus, deren Differenzierung durch das Zytokin IL-23 induziert wird und die Produktion der Zytokin IL-17 und IL-22 auslösen. Diese wiederum führen zu einer starke inflammatorischen Immunantwort, ohne eine protektive Immunität zu induzieren (Zelante et al. 2007). Die IL-17 Neutralisierung führt im Tiermodel zu einer erhöhten Beseitigung des Pilzes, einer Verbesserung der inflammatorischen Pathologie und einer Wiederherstellung der TH1 Immunität (Romani et al. 2007). Bei einer überhöhte TH17 Immunantwort kommt es zu einer erhöhten fungalen Belastung, indem die Aktivierung von neutrophilen Granulozyten behindert und eine hyper-inflammatorische Immunantwort ausgelöst wird (Wuthrich et al. 2012). Bei einer protektiven Immunantwort werden erhöhte inflammatorische Immunreaktionen limitiert durch Treg, welche die regulatorischen Zytokine IL-10 und TGF-β (transforming growth factor) produzieren, und damit eine Zerstörung des Gewebes verhindert wird (Romani et al. 2007; Romani 2011). Zwei wichtige Klassen von regulatorischen T-Zellen innerhalb der CD4+ T-Zellen sind die CD4+CD25+Foxp3+ Treg und regulatorische T-Zellen Typ 1 (Tr1). Sie Transkriptionsfaktoren, unterscheiden ihrer sich zellulären bezüglich Marker und ihres in den Zytokinprofils, ihrer Mechanismen der Immunsuppression (Murugaiyan et al. 2009). Kürzlich wurde gezeigt, dass A. fumigatus (Crf1/p41) spezifische Tr1 T-Zellen neben hohen Mengen IL-10 auch IFN-γ koproduzieren und die Fähigkeit besitzen, die Aktivierung von naiven und anti-fungalen T-Zellen zu supprimieren (Bedke et al. 2014). 17 Einleitung + Abbildung 2: CD4 T-Zell Subpopulationen bei Pilzinfektionen. Gezeigt ist, wie DCs, durch die + Stimulation mit Pilzantigenen, die Differenzierung zu CD4 TH Zellen und Treg Zellen, in Abhängigkeit von Transkriptionsfaktoren und des Zytokinmilieu initiieren. Dabei können die Zellen als Effektor- Immunzellen und als Regulatoren der Inflammation und Effektor- Antworten von Zellen der angeborenen Immunität fungieren. Die Abbildung wurde der Arbeit von Romani 2011 entnommen. CARD9, caspase recruitment domain-containing protein 9; DC-SIGN, DC-specific ICAM3-grabbing non-integrin; FcRγ, Fc Rezeptor γ-Kette; IDO, indoleamine 2,3-dioxygenase; IFN-γ, Interferon-γ; IL, Interleukin; MR, Mannoserezeptor; MYD88, myeloid differentiation primary response protein 88; SYK, spleen tyrosin kinase; TLR, Toll-like Rezeptor; TRIF, TIR domaincontaining adaptor protein induziert IFN-β (TICAM1); VLA, very late antigen 5. 3.2.3 Strategien von Aspergillus fumigatus dem Immunsystem auszuweichen Der Pilz hat einige Strategien entwickelt, die zu seiner Pathogenität beitragen, unter anderem durch die Sekretion von Proteasen und Lipasen, die zu einem Gewebeabbau führen (Latge 1999). Durch die Produktion von Mycotoxinen (Fumagillin, Fumitremorgine, Gliotoxin und Sphingofungine) verschafft sich der Pilz weitere Vorteile zum Fortbestand unter schwierigsten Bedingungen. Zum Beispiel hat das Gliotoxin immunsuppressive Eigenschaften auf Zellen der angeborenen Immunität, indem es aktiv die Phagozytose und den respiratorischen Burst der Granulozyten inhibiert. Einfluss auf die adaptive Immunität hat das Gliotoxin, indem es die Aktivierung des NF-κB Signalweg in T- und B-Lymphozyten verhindert. Ebenso indem es eine Apoptose in Monozyten, dendritischen Zellen und Thymozyten auslöst, wodurch die Aspergillus spezifischen T-Zell Immunantworten supprimiert werden (Orciuolo et al. 2007; Kwon-Chung et al. 2009). 18 Einleitung Neben seiner Fähigkeit, ribosomale 28S RNA eukaryontischer Ribosomen zu schneiden, kann das Ribotoxin AspF1 eine Zellnekrose verursachen (Lamy et al. 1991). Weitere Virulenz-Faktoren sind reaktive Antioxidantien (Katalase und Superoxiddismutase), die den Pilz vor einem oxidativem Burst durch Phagozyten und Granulozyten beschützen (Latge 2001). Auch die Pilzzellwand hat einige Abwehrmechanismen entwickelt, so zum Beispiel bietet Melanin in der Zellwand Schutz vor reaktiven Sauerstoffspezies (ROS= reactive oxygen species) und verhindert die Phagozytose. Außerdem ist die Zellwand der A. fumigatus Konidien von cysteinreichen hydrophoben Proteinen (hydrophobins) umgeben, die eine Erkennung durch das Immunsystem maskieren können (Brakhage et al. 2002; Aimanianda et al. 2009). Die Zellwand besteht zu 90% aus Polysacchariden (meist α- und β-Glucan, Mannan, Glactomannan und Chitin) und bietet nicht nur Schutz vor der Umwelt und dem Immunsystem, sondern sie ist auch die Quelle der meisten Antigene, wie z.B. die Zellwandglykonase Crf1 (Latge 2010). 3.3 Klinische Relevanz von Aspergillus fumigatus Infektionen Der Pilz A. fumigatus kann je nach Immunzustand des Wirtes verschiedene Krankheiten auslösen. Aspergillome, eine Ansammlung von Mukus, Pilzhyphen und Zellbestandteilen, können bei Patienten mit normaler Immunlage auftreten und in der Regel problemlos operativ entfernt werden. Bei Patienten mit geschwächtem Immunsystem kann A. fumigatus zum Teil schwere invasive Infektionen auslösen (Romani 2011). Bei neutropenischen Patienten, Patienten die an atopischen Asthma oder an Mukoviszidose leiden, besteht ein erhöhtes Risiko an allergische bronchopulmonale Aspergillose (ABPA) zu erkranken. ABPA, eine durch Aspergillus verursachte Lungenerkrankung, kann ein breites Spektrum an Symptomen auslösen, wie Rachenentzündung, Lungenfibrose oder sogar die Zerstörung der Lunge (Chaudhary et al. 2011). Transplantationspatienten sind aufgrund der begleitenden immunsupprimierenden Therapie besonders gefährdet, an einer meist lebensbedrohlichen invasiven Aspergillose (IA) zu erkranken, aber auch Krebs- und AIDS- Patienten sind gefährdet. Bei IA kommt es zum Wachstum des Pilzes in der Lunge bzw. auf der Bronchialwand, was häufig tödlich für den Patienten sein kann (Latge 1999; Marr et al. 2002; Romani 2004; Rivera et al. 2006). Trotz fortschreitenden Verbesserungen in der Behandlung und medikamentöser Prophylaxe bleibt die Sterblichkeitsrate bei invasiven Erkrankungen hoch (Baddley et al. 2013). 3.3.1 Behandlungsstrategien von Aspergillus fumigatus Infektionen Zurzeit ist die Hauptbehandlungsstrategie von A. fumigatus Infektionen die Verabreichung von antifungalen Medikamenten. Neuere Behandlungsmethoden werden laufend 19 Einleitung weiterentwickelt, wie die Unterstützung des Immunsystems durch die Gabe von Granulozyten, Zytokinen, Chemokinen oder Wachstumsfaktoren, die Vakzinierung und die adoptive T-Zelltherapie (Bozza et al. 2004; Perruccio et al. 2005; Segal et al. 2006). Im Allgemeinen werden in der antifungalen Therapie vor allem die Medikamente Amphotericin B, liposomales Amphotericin B, Posaconazol, Fluconazol und Voriconazol eingesetzt (Segal et al. 2006; Hamill 2013; Jorgensen et al. 2014). Der erfolgreiche therapeutische Einsatz ist stark beeinflusst von den toxischen Eigenschaften der Medikamente, die ein breites Spektrum an Nebenwirkungen von Fieber bis Nierenschäden auslösen können. Ein weiterer zu beachtender Faktor bei der Verabreichung dieser Medikamente ist die Entstehung von Resistenzen innerhalb der Pilzstämme (Vermeulen et al. 2013; Alcazar-Fuoli et al. 2014). Bei IA werden bevorzugt Voriconazol oder liposomales Amphotericin B eingesetzt. In neueren Studien werden auch Kombinationstherapien auf ihre Effektivität untersucht, bisher konnte in vitro noch kein entscheidender Vorteil zur Einzeltherapie nachgewiesen werden (Marr et al. 2004; Elefanti et al. 2013). Weitere Behandlungsstrategien sind der Einsatz von immunstimulatorischen Mitteln. Dabei ist die Gabe von Granulozyten eher umstritten. Trotz einzelner erfolgreicher Studien (Bielorai et al. 2000; Dinser et al. 2005; Grigull et al. 2006) wurde die Effektivität der Methode angezweifelt und teilweise eine Verschlechterung der IA beschrieben (Raad et al. 2013). Durch die unterstützende Behandlung mit verschiedenen Zytokinen und Wachstumsfaktoren zur Stärkung des Immunsystems und der Beeinflussung der TH1/TH2 Balance, konnte die Effektivität in vorklinischen Studien in vitro und in vivo bestätigt werden (Roilides et al. 2003; Safdar 2006). Ein Anti-β-Glucan Antikörper wurde im Mausmodel untersucht. Dabei hat sich gezeigt, dass der Antikörper mit den Zellwänden von Candida albicans, A. fumigatus und Crypotcoccus neoformans reagierte, das Wachstum der Pilze inhibierte und eine experimentell induzierte Candidiasis und Aspergillose verhindert (Torosantucci et al. 2005; Torosantucci et al. 2009). Die Erkenntnisse über Vakzinierungen zur Bekämpfung von A. fumigatus ausgelösten Infektionen wurden vorwiegend durch Untersuchungen im Mausmodell erworben. Eine protektive TH1 Immunantwort konnte durch die Vakzinierung mit wachstumsfähigen Konidien und Hyphen-Extrakt induziert werden, wohingegen die Gabe hitzeinaktivierter Konidien zu einer TH2 Immunantwort führte (Cenci et al. 2000; Bozza et al. 2002). Neben Pilz-Extrakten sind auch rekombinante Proteine wie Asp f16, Asp f 3 und Crf1 fähig, eine protektive Immunität auszulösen (Bozza et al. 2002; Ito et al. 2006; Diaz-Arevalo et al. 2011; Stuehler et al. 2011). 20 Einleitung Aufgrund der Plastizität und der Schlüsselfunktion in der Initiierung und der Regelung der Immunität wurden antigenpräsentierende DCs eingesetzt, um die Immunantwort in eine bestimmt TH Richtung zu lenken. In einer Studie wurden DCs mit fungaler mRNA transfiziert oder mit Antigen beladen. Es hat sich gezeigt, dass die Immunantwort von der DC Subpopulation abhängig ist, welche die verschiedenen TH Zellen (TH1, TH2, Treg) aktivieren (Perruccio et al. 2004; Bellocchio et al. 2005). Eine weitere Möglichkeit zur Bekämpfung von Infektionen, ausgelöst durch A. fumigatus, ist der adoptive Transfer von funktionellen TH1 Zellen, um eine protektive Immunantwort zu induzieren. Dieser Ansatz wurde neben der Erforschung im Mausmodell auch in einer klinischen Studie an Patienten, mit vorangegangener Stammzelltransplantation, eingesetzt. Im Mausmodell wurde durch den adoptiven Transfer zum einen von dendritischen Zellen beladen mit Aspergillus Antigenen und zum anderen von Aspergillus spezifischen CD4+ Splenozyten, eine Reduktion der fungalen Infektion (Cenci et al. 2000) sowie die Induktion einer protektiven TH1 Immunantwort festgestellt (Bozza et al. 2003). In einer klinischen Studie wurde gezeigt, dass durch den adoptiven Transfer von Aspergillus spezifischen TH1 T-Zellklonen in Patienten, die an einer Aspergillus Infektion nach einer hämatopoetischen Stammzelltransplantation erkrankten, eine stabile T-Zell Immunantwort induziert wird, ohne eine Transplantat-Wirt Reaktion (GvHD, Graft-versus-Host-Disease) auszulösen (Perruccio et al. 2005). 3.4 Antigenerkennung durch T-Zellen T-Zellen erkennen und reagieren auf Antigen oder ein Peptid, wenn dieses verbunden mit einem eigenen Haupthistokompatibilitätskomplex Molekül (MHC, major histocompatibility complex) präsentiert wird. Der Antigenrezeptor auf den T-Zellen ist dabei der αβ T-Zell-Rezeptor (TCR) (Davis 1988; Fremont et al. 1996). Im folgenden Abschnitte werden die Strukturen und Mechanismen der Antigenerkennung durch T-Zellen beschrieben. 3.4.1 Haupthistokompatibilitätskomplex (MHC) Bei Menschen werden MHC Gene als humane Leukozyten Antigene (HLA) bezeichnet und befinden sich auf Chromosom 6. Die Gene lassen sich in zwei Klassen unterteilen, in Klasse-I (A, B und C), die von CD8+ T-Zellen erkannt werden und in Klasse-II (DP, DQ und DR) die von CD4+ T-Zellen erkannt werden. Peptide von Krankheitserregern, wie z.B. Viren, werden durch die MHC Klasse-I Moleküle den zytotoxischen CD8+ T-Zellen präsentiert. Die Hauptfunktion der CD4+ T-Zellen, die MHC Klasse II Moleküle erkennen, ist die Aktivierung anderer Effektorzellen des Immunsystems (Rudolph et al. 2006). 21 Einleitung MHC Proteine sind Heterodimere, das heißt sie besitzen Untereinheiten bestehend aus einer α und β Kette. MHC Klasse-I Moleküle bestehen aus einer α Kette unterteilt in drei Domänen und einer invarianten β2-Mikroglobulin Kette. Dabei bilden die α1 und α2 Domänen die Peptid-Bindungsfurche und α3 ist in der Zellmembran verankert. Für MHC Klasse-I existieren drei verschiedene α Gene (HLA-A, HLA-B und HLA-C) (Marsh et al. 2001; Schreuder et al. 2001). MHC Klasse-II besitzt zwei membrangebundene Ketten (α und β), wobei die Peptid-Bindungsfurche von den α1 und β1 Domänen gebildet wird. Die MHC Klasse-II Gene werden in die drei Moleküle HLA-DR, HLA-DP und HLA-DQ unterteilt. In den verschiedenen MHC- Varianten sitzen an bestimmten Stellen der peptidbindenden Furche unterschiedliche Aminosäuren (AS) an Schlüsselpositionen für die Peptidwechselwirkung. Wodurch unterschiedliche MHC- Varianten bevorzugt unterschiedliche Peptide binden (Rudolph et al. 2006; Murphy et al. 2012). Ohne eine Peptidbindung sind die MHC Moleküle instabil, da die MHC Moleküle das Peptid als integralen Bestandteil ihrer Struktur binden. Die MHC Klasse-I Moleküle können kurze, acht bis zehn Aminosäure (AS) lange Peptide, binden. Die Bindung erfolgt über eine Reihe von Wasserstoffbrückenbindungen und ionischen Wechselwirkungen, an beiden Enden des Peptids. MHC Klasse-II Moleküle können Peptide mit einer Länge von dreizehn AS (oder länger) binden, durch Wechselwirkungen, wie Wasserstoffbrückenbindungen, entlang der Peptid-Bindungsfurche an MHC Klasse-II Moleküle. Aufgrund der Bindung über das Peptidrückrat kann das Peptid an beiden Seiten der Peptid-Bindungsfurche herausragen und somit ist das Peptid keiner Längenbegrenzung unterzogen, jedoch werden Peptide nach ihrer Bindung an MHC Klasse-II Moleküle in den meisten Fällen durch Peptidasen auf eine Länge von 13 bis 17 AS verkürzt. Der TCR bindet den MHC/Peptid Komplex über die CDR Schleifen. CDR3 bindet dabei hauptsächlich das Peptid. CDR1 und CDR2 den MHC (Rudolph et al. 2006; Marrack et al. 2008; Murphy et al. 2012). 3.4.2 αβ T-Zell-Rezeptor Der TCR besteht aus zwei Transmembranproteinen, einer α und einer β Kette, die über eine Disulfidbrücke in der extrazellulären Domäne miteinander verbunden sind. Jede Kette kann in eine N-terminale variable und eine C-terminale konstante Region unterteilt werden (Abbildung 3). Die variable Region trägt die Antigen Bindungsstelle und liegt extrazellulär. Die konstante Region ist unterteilt in eine immunoglobulinähnliche Domäne, eine Gelenkdomäne, welche das Zystein für die Ausbildung der Disulfidbrücke einschließt, gefolgt von der Transmembrandomäne und einer kurzen zytoplasmatischen Domäne. Die dreidimensionale Struktur des TCR ist immunoglobulinähnlich, das heißt die strukturelle 22 Einleitung Basis bilden β-Faltblätter. Ausnahme ist die immunoglobulinähnliche Domäne der α Kette, deren eine Hälfte eine β-Faltblatt-Struktur aufweist, während die andere in losen Strängen miteinander verbunden ist und in einer α- Helix endet, welche an dieser Stelle über die Disulfidbrücke die β Kette verbindet (Garcia et al. 2010; Murphy et al. 2012). Abbildung 3: Struktur des αβ T-Zell-Rezeptors. Der Rezeptor besteht aus den Transmembranglykoproteinketten α (lila) und β (grün). Der extrazelluläre Teil jeder Kette besteht aus einer variablen und einer konstanten Region. Über die Gelenkregion, welche das Cystein zur Ausbildung einer Disulfidbrücke enthält, werden die externen Regionen mit der Membran verbunden. Die Transmembrandomäne und die zytoplasmatische Domäne sind schematisch dargestellt. Modifizierte Darstellung des Banddiagramm von Bjorkman 1997 und Abbas et al. 2007. Die hypervariblen Regionen der variablen α (Vα) und variablen β (Vβ) Kette liegen nach oben gerichtet in Schleifen und werden als komplementaritätsbestimmende Regionen (complementarity determining region; CDR) bezeichnet. Es gibt drei CDRs, welche die Bindungsstelle zwischen Peptid:MHC und TCR bilden. Die Sequenzen der CDRs sind sehr variabel, wobei CDR3 die variabelste Sequenz exprimiert (Garcia et al. 2010; Murphy et al. 2012). Die Regionen zwischen den CDRs sind weniger variabel und werden als Gerüstregionen (framework region; FR) bezeichnet (Abbildung 4). 23 Einleitung Abbildung 4: Schematische Darstellung der TCR α und TCR β Ketten. Die schematische Darstellung der TCR α (A) und TCR β Ketten (B) unterteilt in die V(D)J Segmente. SP= Signalpeptid; FR= Gerüstregion (Framework Region); CDR= komplementaritätsbestimmende Regionen (complementary determining region); IG: immunoglobin-ähnliche Domäne der C Region; VP= Verbindungs- Peptid (Gelenkdomäne) TM= Transmembrandomäne; IZ= Intrazelluläre Domäne (zytoplasmatische Domäne). 3.4.3 Die Umordnung der T-Zell-Rezeptor Gene Der humane TCR α- Locus ist auf dem Chromosom 14 lokalisiert und der TCR β- Locus befindet sich auf Chromosom 7. Die TCR Vielfalt entsteht im Wesentlichen durch kombinatorische und junktionale Genumordnung der verschiedenen V (variable) und J (junktionale) Segmente der α Kette und der V, D (Diversität) und J Segmente der β Kette. Die variable Region von TCR α kann von ca. 70 bis 80 verschiedenen Vα Segmenten aufgebaut werden, wobei jedes eine Signalsequenz besitzt und ca. 61 verschiedene Jα Segmente. Die variablen Regionen von TCR β können auf ein Repertoire von 54 Vβ, 14 Jβ und zwei Dβ zurückgreifen. Für die konstante α Region ist ein Cα Segment und für die konstante β Region sind zwei Cβ Segmente vorhanden. Während der Reifung der T-Zellen im Thymus werden diese Gene umgelagert, wobei die Umlagerungsreaktion zuerst für die TCR β Kette startet (Davis 1988). Für die Rekombination befindet sich hinter jedem V Segment und vor jedem J Segment, welches von einem D Segment flankiert wird, eine Rekombinationssignalsequenz (RSS) (Fugmann et al. 2000). Diese Signalsequenzen sind die Erkennungsstruktur für die V(D)J Rekombinasen (RAG Rekombinasen = RAG-1 und RAG-2; recombination- activation gene). Die konstanten Ketten werden nach der Transkription durch Spleißen mit den variablen Ketten verbunden (Abbey et al. 2008; Murphy et al. 2012). 24 Einleitung 3.4.4 αβ T-Zell-Rezeptor Komplex Das TCRαβ Heterodimer wird durch die invarianten Dimere CD3γε (CD = cluster of differentiation), CD3δε und ξξ stabilisiert und sind verantwortlich für die Signalübertragung (Call et al. 2004). Die invarianten Ketten CD3γ, CD3δ und CD3ε bestehen aus einer immunoglobinähnlichen extrazellulären Domäne, einer Transmembrandomäne und einer zytoplasmatischem Domäne mit einer Immunrezeptor Tyrosinaktivierungssequenz (ITAM, immunoreceptor tyrosin-based activation motif). Die ξξ Ketten besitzen nur eine kurze extrazelluläre Domäne, eine Transmembrandomäne und eine lange zytoplasmatische Domäne mit drei ITAMs. In ihren Transmembrandomänen besitzen die CD3 Ketten negativ geladene, saure Reste, die mit den positiven Ladungen der α und β Ketten wechselwirken können (Abbildung 5) (Call et al. 2007). Die Stöchiometrie des TCR αβ Heterodimer zu den invarianten CD3 Heterodimeren wird noch diskutiert, jedoch wurde bisher eine Anordnung des TCR αβ Heterodimer flankiert von den CD3 Dimeren als wahrscheinlichste postuliert (Sun et al. 2004). Dagegen wurde in neueren Arbeiten gezeigt, dass die CD3 Heterodimere in einem Verbund auf einer Seite des TCR αβ Heterodimer lokalisiert sind (Fernandes et al. 2012; Kuhns et al. 2012). Abbildung 5: Der αβ T-Zell-Rezeptor Komplex. Der TCR Komplex besteht aus einem αβ Heterodimer, assoziiert mit zwei invarianten CD3 Heterodimeren (CD3γε und CD3δε) und mit einem Homodimer (ξξ). Jede CD3 Kette besitzt ein ITAM (immunoreceptor tyrosine-based activation motif) und jede ξ Kette drei ITAMs. Die Transmembranregionen sind entweder positiv (αβ Heterodimer) oder negativ (invariante Ketten CD3 und ξ) geladen. Abbildung aus Murphy et al. 2012. Die Komponenten des TCR Komplex werden durch die Ribosome im Endoplasmatischen Retikulum (ER) synthetisiert. Der Aufbau des TCR Komplex startet mit den invarianten Heterodimeren CD3γε und CD3δε, welche anschließend mit den TCR α und TCR β 25 Einleitung Ketten verbunden werden. Abschließend werden die ξξ Homodimer hinzugefügt. Wird der TCR Komplex im ER nicht richtig zusammengefügt, so werden die Komponenten aufgrund ihrer allein stehenden, polaren Transmembranreste erkannt und abgebaut. Ein korrekt verbundener TCR Komplex wird dagegen zur Zelloberfläche transportiert (Call et al. 2007). 3.4.5 αβ T-Zell-Rezeptor Signalübertragung Es werden verschiedene Modelle bezüglich der Signalübertragung der antigenbindenden Region der TCR α und TCR β Ketten zu den signalübertragenden CD3γε, CD3δε und ξξ diskutiert, unter anderem die Zusammenlagerung von TCRs in einen Komplex, die Verstärkung durch Ko-Rezeptoren und Konformationsänderungen (Kuhns et al. 2007; Fernandes et al. 2012; Yin et al. 2012). Bindet der TCR antigenspezifisch an ein MHC Klasse-II/Peptid, so wird über die Aktivierung von Kinasen (z.B. Fyn) die Phosphorylierung der ITAMs von CD3 und ξ induziert. An die phosphorylierten ITAMs binden die ZAP-70 Tyrosinkinase. Über die Bindung des Ko-Rezeptors an das MHC Molekül wird Lck phosphoryliert und ZAP-70 aktiviert. ZAP-70 Aktivierungen führen zur Phosphorylierung von LAT und SLP-76, welche wiederum die Phospolipase C-γ aktivieren. Über Proteinkinasen, die Erhöhung der interzellularen Ca2+ Konzentration und durch Induktion der MAP-Kinase Kaskade werden die Transkriptionsfaktoren NFκB, NFAT (nuclear factor of activated T cells) und Ap-1 aktiviert, die wiederum spezielle Gentranskriptionen induzieren. Ferner benötigen T-Zellen zur vollständigen Aktivierung ein weiteres kostimulierendes Signal, wie z.B. durch CD28, was zur Proliferation, Differenzierung der Zellen und zur Ausschüttung des Zytokins IL-2 führt (Murphy et al. 2012; Guy et al. 2013). 3.5 T-Zell-Rezeptor Optimierungen und der T-Zell-Rezeptor Transfer auf T-Zellen Die Entwicklungen des adoptiven Transfers, mit TCR-modifizierten T-Zellen, wurden vor allem für die Krebstherapie vorangetrieben (Kalos et al. 2013; Koste et al. 2014). Dabei hat sich herausgestellt, dass transferierte TCRs mit endogenen TCRs konkurrieren müssen und kombinierte TCRs entstehen können, welche möglicherweise das Risiko von Autoimmunreaktionen erhöhen. Bendle et al. 2010 haben gezeigt, dass ein TCR Gen Transfer im Mausmodell eine tödliche Autoimmunreaktion durch Zytokinausschüttung induziert und dass die kombinierten TCRs für das Auslösen einer Autoimmunreaktion verantwortlich sind. Auch bei der Transduktion von TCRs in humane T-Zelllinien reduziert sich die Expression des gewünschten TCR, durch die Entstehung verschiedener TCR α/β Kombinationen, wodurch allo- und auto-reaktive T-Zellen entstehen können (van Loenen 26 Einleitung et al. 2010). Die Auswirkungen der kombinierten TCRs, sowie das Auslösen der Autoimmunreaktionen werden auch in einigen neueren Arbeiten diskutiert (Linnemann et al. 2011; Wieczorek et al. 2013). Um die Möglichkeit gemischter TCRs zu eliminieren bzw. zu reduzieren, werden verschiedene Strategien verfolgt. Ein Ansatz ist die Murinisierung der TCR konstanten Ketten, die verglichen mit denen des unveränderten TCR, eine stärkere Bindung der murinen Sequenz zu CD3 Komplexen zeigen. Zudem wurde eine stärkere Bindung der murinen Ketten zueinander und die daraus resultierende Reduktion der Expression gemischter TCRs gezeigt (Cohen et al. 2006; Sommermeyer et al. 2010). Für den klinischen Einsatz ist bei der Optimierung durch Murinisierung die erhöhte Expression mit den Risiken einer induzierten Immunogenität, abzuwägen. Auch der Einbau weiterer Disulfidbrücken in die konstante Region des TCR führt zu einer erhöhten Oberflächenexpression und reduziert das Auftreten kombinierter TCRs (Cohen et al. 2007; Kuball et al. 2007). Entscheidend für einen erfolgreichen Transfer von TCRs ist ein stabiler TCR Komplex. Diese Stabilität kann durch eine erhöhte Bindung der TCR α und β Ketten zueinander, durch Expressions- „starke“ TCRs erreicht werden (Sommermeyer et al. 2006). Eine Kombination von Expressions- „starken“ TCRs und einer starken Bindung der CD3 Komponenten CD3γε, CD3δε und ξξ untereinander und zum TCR sind entscheidend für die Stabilität des TCR-Komplexes (Heemskerk et al. 2007). Momentan forschen einige Arbeitsgruppen daran, die komplexen extrazellulären Bindungen von CD3 Komponenten und TCR Komplexen zu identifizieren (Kuhns et al. 2007; Fernandes et al. 2012; Kuhns et al. 2012). Die Anwendung von Modifikationen innerhalb der Sequenzen kann ebenfalls Einfluss auf den Translationslevel haben. Einige dieser Modifikationen sind, z.B. die Entfernung kryptischer Spleißstellen (cryptic splice sites), instabiler RNA Bindungsstellen (RNA instability motifs) und der Austausch von seltenen Codons, die für selten auftretende tRNA codieren, die Effektivität der Translation mindern und so die Expression beeinflussen. Ein Codon gilt als “selten”, wenn dieses 50 % seltener angetroffen wird, als Codons die für dieselbe Aminosäure codieren (Scholten et al. 2006). Neben der Codonoptimierung wurde gezeigt, dass eine Vektoroptimierung ebenfalls zu Verbesserungen in der Expression führt. So wies die Vektorklonierung von β Kette und α Kette, getrennt durch einen Peptidlinker, in einen einzigen retroviralen Vektor, die höchste Expression auf (Leisegang et al. 2008). Einige Arbeitsgruppen verfolgen im Hinblick auf Vektorsysteme andere Ansätze, die allerdings im Vergleich zum retroviralen Vektorsystem im klinischen Maßstab noch nicht häufig eingesetzt wurden. So kommen zum Beispiel lentivirale Vektoren (Bobisse et al. 2007), nicht-virale Transposon basierte Vektorsysteme (Sleeping Beauty transposon) (Peng et al. 2009) oder modifizierter Verpackungsproteine, die auf bestimmte Zellpopulationen spezialisiert sind und die Vektorfunktionen erhöhen, zum Einsatz (Funke et al. 2008). 27 Einleitung Aufgrund der Konkurrenzproblematik zum endogenen TCR wurden chimärische Antigen Rezeptoren (CARs) entwickelt. CARs haben den Vorteil, dass sie aus einem einzelnen variablen Fragment (scFv) gebunden an CD3 bestehen und so bei einem Transfer nicht mit endogenem TCR konkurrieren oder kombinieren. Ebenso sind sie im Vergleich zu TCRs HLA-unabhängig. Jedoch sind auch Nachteile bezüglich der CARs zu beachten, zum einen wurde in frühen Studien gezeigt, dass CARs exprimierende T-Zellen weniger sensitiv gegenüber Peptiden sind als T-Zellen, die einen αβ TCR exprimieren (Zhang et al. 2004). Zum anderen können CARs eine zu hohe Zytokinausschüttung induzieren. In einigen Fällen könnte die Signalschwelle so niedrig sein, dass in T-Zellen auch ohne Antigenpräsentation eine Immunantwort ausgelöst werden kann (Ramos et al. 2011). Ebenfalls sollte die Immunogenität nicht außer Acht gelassen werden, da das scFv von der Maus stammt und somit eine Immunantwort und Eliminierung der CAR transduzierten T-Zellen ausgelöst werden kann (Thomas et al. 2010; Wieczorek et al. 2013). Der erfolgreiche Transfer von genetisch modifizierten T-Zellen zur Bekämpfung von malignem Melanom und Sarkom wurde bereits beschrieben (Morgan et al. 2006; Robbins et al. 2011). Die Entwicklungen, Modifikationen und die Optimierung von TCRs für einen erfolgreichen Transfer auf Empfänger T-Zellen, für eine Krebstherapie, könnten auch für andere Therapien interessant sein. 28 Ziele der Arbeit 4 Ziele der Arbeit Mit dem Ziel, die Entwicklung der Behandlungsmethoden von A. fumigatus Infektionen voranzubringen, wurden in dieser Arbeit A. fumigatus (Crf1/p41) spezifische TCRs identifiziert und auf T-Zellen transferiert. In den letzten Jahren wurde gezeigt, dass die erworbene Immunität einen großen Einfluss bei der Bekämpfung fungaler Infektionen hat, so spielen TH1 Zellen, induziert durch dendritische Zellen, eine entscheidende Rolle in der Immunantwort gegen A. fumigatus (Romani 2008). Bei der Bekämpfung von Aspergillus Infektionen nach hämatopoetische Stammzelltransplantation wurde gezeigt, dass durch den adoptiven Transfer von Aspergillus spezifischen TH1 T-Zellklonen, eine stabile T-Zell Immunantwort induziert werden kann, ohne eine Transplantat-Wirt Reaktion (Graft-versus-host disease = GvHD) auszulösen (Perruccio et al. 2005). Eine Erweiterung stellt der adoptive Transfer von spezifischen genetisch modifizierten T-Zellen dar, welcher bisher in der Krebstherapie angewandt und vorangetrieben wurde (Restifo et al. 2012; Kalos et al. 2013). Der erfolgreiche Transfer von genetisch modifizierten T-Zellen zur Bekämpfung des malignen Melanom und Sarkom wurde bereits beschrieben (Morgan et al. 2006; Robbins et al. 2011). Eine Adaption dieses Verfahrens könnte zur Bekämpfung von Aspergillus Infektionen oder als zusätzliche Behandlungsmethode zur medikamentösen anti-fungalen Therapie eingesetzt werden. Ein transduktionsfähiger, funktioneller A. fumigatus spezifischer TCR wäre für die Entwicklung eines solchen adoptiven Transfers von modifizierten T-Zellen ein Anfang. • Für die Etablierung eines solchen TCR Transfers müssen zunächst A. fumigatus (Crf1/p41) spezifische TH1 T-Zellklone für die Identifizierung ihrer TCRs generiert werden. • Im Anschluss müssen die TCRs der A. fumigatus (Crf1/p41) spezifischen TH1 T-Zellklone über ihre Sequenz identifiziert werden. • Die Isolation der spezifischen TCRs erfolgt über die cDNA der A. fumigatus (Crf1/p41) spezifischen TH1 T-Zellklone, mit anschließender Klonierung in einen retroviralen Vektor. • Im Folgenden werden die Vektoren, mit Genen der A. fumigatus (Crf1/p41) spezifischen TCRs, für die Generierung von retroviralen Kulturüberstand eingesetzt, um eine Transduktion auf die Zelllinie Jurkat 76 und primäre CD4+ T-Zellen durchzuführen. • Mit der TCR negative Zelllinie Jurkat 76 wird die Oberflächenexpression der verschieden A. fumigatus (Crf1/p41) spezifischen TCRs ohne den Einfluss von endogenem TCR ermittelt und die Transduktionsfähigkeit bestätigt werden. 29 Ziele der Arbeit • Bei geringer Oberflächenexpression ist es sinnvoll, den TCR und das Vektorsystem zu optimieren. Einige Methoden wurden vor allem für die Krebstherapie vorangetrieben, so auch die Vektorsysteme und der Einsatz von Optimierungsmethoden, um die Expression des induzierten TCR zu erhöhen (Engels et al. 2003; Sommermeyer et al. 2006; Xue et al. 2007). • Es folgt die Ermittlung der Transduktionsfähigkeit durch die Oberflächenexpression von A. fumigatus (Crf1/p41) spezifischen TCRs auf transduzierten primäre CD4+ T-Zellen. • Die funktionellen Eigenschaften von primären CD4+ T-Zellen, transduziert mit A. fumigatus (Crf1/p41) Proliferationsfähigkeit und spezifischem IFN-γ TCR, Produktion werden nach durch die antigenspezifischer (Crf1/p41 spezifischer) Stimulation bestätigt. 30 Materialien 5 Materialien 5.1 Medien, Versuchssysteme, Chemikalien und Reagenzien 5.1.1 Kulturmedien und Zusätze Kulturmedien und Zusätze Firma/Hersteller Bicoll Seperationslösung Biochrom (Berlin) Blasticidin Sigma-Aldrich (München) Bovines Serum Albumin (BSA) Sigma-Aldrich (München) CD3/CD28 Dynabeads® Life Technologies, Invitrogen (Darmstadt) Dimethylsulfoxid (DMSO) Sigma-Aldrich (München) Dulbecco´s modification of Eagle´s Medium (DMEM) Life Technologies, Invitrogen (Darmstadt) Dulbecco´s Phosphat Saline Puffer (PBS) Life Technologies, Invitrogen (Darmstadt) Ethylendiamintetraacetat (EDTA) Lösung 0,5 M AppliChem (Darmstadt) Fötales Kälberserum (FCS) Biochrom (Berlin) GM-CSF PromoCell (Heidelberg) Humanserum ZKT (Tübingen) IL-1β R&D system (Wiesbaden) IL-2 Proleukin, Chiron, Novartis IL-4 R&D system (Wiesbaden) IL-6 R&D system (Wiesbaden) OKT-3 (Orthoclone) (CD3) Janssen-Silac (Neuss) Penicillin/Streptomycin Life Technologies, Invitrogen (Darmstadt) Prostaglandin E2 (PGE2) ehemals Pharmacia Puromycin Sigma-Aldrich (München) RPMI 1640 Zellkulturmedium Life Technologies, Invitrogen (Darmstadt) TNF-α R&D system (Wiesbaden) Trypsin/EDTA Lösung Life Technologies, Invitrogen (Darmstadt) 31 Materialien 5.1.2 Versuchssysteme Versuchssysteme (Kits) Firma/Hersteller BD Cytometric Bead Array (CBA) (human) Becton Dickinson (BD) (Heidelberg) + CD4 T Cell Isolation Kit II Miltenyi Biotech (Bergisch-Gladbach) IO Beta Mark TCR Kit (TCRβ Bestimmung) + MACS®-Cell-Sorting-System (CD14 Beads; Beckman Coulter (Krefeld) Miltenyi Biotech (Bergisch-Gladbach) PE-Beads) NucleoSpin® Gel and PCR Clean-up Macherey-Nagel GmbH & Co KG (Düren) Plasmid DNA Purification Kit Macherey-Nagel GmbH & Co KG (Düren) RNasy Mini Kit Qiagen (Hilden) SMARTer™ RACE cDNA Amplification Kit Clontech Laboratories (Mountain View, USA) 5.1.3 Chemikalien und Reagenzien Chemikalien und Reagenzien Firma/Hersteller ABTS Roche (Penzberg) Agar AppliChem (Darmstadt) Agarose PeqLap (Erlangen) Ampicillin Roth (Karlsruhe) Ammoniumpersulfat (APS) Sigma-Aldrich (München) Carbonat-Bicarbonat Puffer Sigma-Aldrich (München) Not1 Restriktionsenzym New Englang Biolabs (Frankfurt) EcoR1 Restriktionsenzym New England Biolabs (Frankfurt) DNA Ladder 1kb PLUS Life Technologies, Invitrogen (Darmstadt) Desoxyribonukleosidtriphosphat (dNTPs) Life Technologies, Invitrogen (Darmstadt) Effectene® Transfektions Reagenz Qiagen (Hilden) Endogen M700A (anti-IFN-γ Antikörper A) Thermo Fischer, Perbio (Bonn) Endogen M701B (anti-IFN-γ Antikörper B) Thermo Fischer, Perbio (Bonn) Ethanol AppliChem (Darmstadt) FACSclean Becton Dickinson (BD) (Heidelberg) FACSflow Becton Dickinson (BD) (Heidelberg) FACSrinse Becton Dickinson (BD) (Heidelberg) Hefeextrakt AppliChem (Darmstadt) 32 Materialien Chemikalien und Reagenzien Firma/Hersteller IFN-γ Standard Thermo Fischer, Perbio (Bonn) Kalziumchlorid Sigma- Aldrich (München) Kanamycin Life Technologies, Invitrogen (Darmstadt) Midori Green Advance Nippon Genetics Europe GmbH (Düren) Natriumchlorid AppliChem (Darmstadt) NBT/BCIP Substrat Lösung Mabtech (Nacka Strand, Schweden) Proliferations-Farbstoff eFluor®670 eBioscience (Frankfurt) Q5 Polymerase New Englang Biolabs (Frankfurt) RDI-PHRP20 Streptavidin Fitzgerald (Acton, USA) RDI-PHRPDIL Blocker Fitzgerald (Acton, USA) RetroNectin® Clontech Laboratories (Mountain View, USA) StreptavidinALP-PQ Mabtech (Nacka Strand, Schweden) T4 DNA Ligase New Englang Biolabs (Frankfurt) Tris-Acetat- EDTA (TAE) Puffer Life Technologies, Invitrogen (Darmstadt) 5.2 Hergestellte Medien und Puffer Medien/Puffer Inhaltsstoffe LB Kulturmedium 10 g Trypton; 5 g Hefeextrakt; 10 g NaCl; 1 l dH2O; pH 7.0 LB Agar 10 g Trypton; 5 g Hefeextrakt; 10 g NaCl; 15 g Agar; 1 l dH2O; pH 7.0 MACS Puffer PBS; 0,5 % BSA; 2 mM EDTA FACS Puffer PBS, 0,2 % BSA ELISA Waschpuffer PBS, 0,05 % Tween20 5.3 Crf1/p41 Peptid und MHC Klasse II Tetramer Peptide/Tetramer Firma/Hersteller Crf1/p41 Peptid (HTYTIDWTKDAVTWS) Beckman Coulter (Krefeld) MHC Klasse II Tetramer (HLA-DRB1*0401 FHTYTIDWTKDAVTW; Beckman Coulter (Krefeld) Phycoerythrin (PE) markiert) 33 Materialien 5.4 Fluoreszenz- konjugierte Antikörper für die Durchflusszytometrie Spezifität Fluoreszenzfarbstoff Firma/Hersteller CD3 Allophycocyanin (APC) Becton Dickinson (BD) (Heidelberg) CD4 APC Becton Dickinson (BD) (Heidelberg) CD8 Fluoresceinisothiocyanat (FITC) Becton Dickinson (BD) (Heidelberg) IgG1 APC Becton Dickinson (BD) (Heidelberg) IgG1 Phycoerythrin (PE) Becton Dickinson (BD) (Heidelberg) IgG1 FITC Becton Dickinson (BD) (Heidelberg) 5.5 5.5.1 Retroviraler Vektor pMP71 und Oligonukleotide Retroviraler Vektor pMP71 Der retrovirale Vektor pMP71 wurde freundlicherweise von Dr. M. Hudecek (Medizinische Klinik und Poliklinik II, Würzburg) zur Verfügung gestellt (in Abbildung 6 ist die Vektorkarte dargestellt). Gene werden über die Restriktionsschnittstellen Not1 und EcoR1 in den Vektor kloniert. Dem Gen folgt ein posttranskriptionelles regulatorisches Element (PRE) des Woodchuck Hepatitis Virus. Das Gen und PRE werden von „long terminal repeats“ (LTR) vom myeloproliferativem Sarcoma Virus flankiert. Der Vektor besitzt eine Ampicillin-Resistenz (AmpR). Bei dem Vektor pMP71 handelt es sich um einen Vektor der eine stabile und hohe Expressionsrate in T-Lymphozyten zeigt und in vielen Studien des TCR Transfers erfolgreich eingesetzt wurde (z.B. Engels et al. 2005; Sommermeyer et al. 2006; Heemskerk 2010; Thomas et al. 2010; Linnemann et al. 2013). 34 Materialien Abbildung 6: Das retrovirale Vektorplasmid pMP71. Gene werden über die Restriktionsschnittstellen Not1 und EcoR1 in den Vektor kloniert. Dem Gen folgt ein posttranskriptionelles regulatorisches Element (PRE) des Woodchuck Hepatitis Virus. Gen und PRE werden flankiert von „long terminal repeats“ (LTR) vom myeloproliferativem Sarcoma Virus. Der Vektor besitzt eine Ampicillin-Resistenz (AmpR). 5.5.2 Oligonukleotide Tabelle 1: Primer für die T-Zell-Rezeptor Identifizierung und Vektorklonierung. Die Primer wurden von Sigma- Aldrich (München) hergestellt. Name Sequenz (5´- 3`) Verwendung RACE a rev GGTGAATAGGCAGACAGACTT Isolation TCRα cDNA RACE b rev GTGGCCAGGCACACCAGTGT Isolation TCRβ cDNA MP71 for CAGCATCGTTCTGTGTTGTCT Sequenzierung Insert von pMP71 Vektor MP71 rev CATTTAAATGTATACCCAAATCAA Sequenzierung Insert von pMP71 Vektor TCRbC1 rev EcoR1 AGCTTGGAATTCTCAGAAATCCTTTCTCTTGACC Sequenzierung/ Isolation TCRβ TCRbC2 rev EcoR1 AGCTTGGAATTCCTAGCCTCTGGAATCCTTTCTC Sequenzierung/ Isolation TCRβ TCRaC rev EcoR1 AGCTTGGAATTCTCAGCTGGACCACAGCCGCAGC Sequenzierung /Isolation TCRα TCR1opti for Not1 ACTGCGGCCGCGCCAACCATGGATACCAGACTGCTG Sequenzierung/ Isolation TCR1 optimiert TCR1opti rev EcoR1 AGCTTGGAATTCATCACACGGGGCTGGT Sequenzierung/ Isolation TCR1 optimiert 35 Materialien Name Sequenz (5´- 3`) Verwendung TCR1α wt for Not1 GGGCGGCCGCGCCACCATGGAGACTGTTCTGCAAGTACTCCTAG Sequenzierung/ Isolation TCR1α TCR1β wt for Not1 ACTGCGGCCGCCATGGACACCAGACTACTCTGCTGT Sequenzierung/ Isolation TCR1β TCRα for Not1 (TRAV17) ACTGCGGCCGCCATGGAAACTCTCCTGGGAG Sequenzierung/ Isolation TCR2α und TCR4α TCRβ for Not1 (TRBV18) ACTGCGGCCGCCATGGACACCAGACTACTCTGCTGT Sequenzierung/ Isolation TCR2β und TCR4β TCR3α for Not1 (TRAV5) ACTGCGGCCGCCATGAAGACATTTGCTGGA Sequenzierung/ Isolation TCR3α TCR3β for Not1 (TRBV9) ACTGCGGCCGCCATGGGCTTCAGGCTCCTCTG Sequenzierung/ Isolation TCR3β 5.6 5.6.1 Zelllinien und Bakterien Zelllinien Zelllinie Eigenschaft Jurkat 76 TCRαβ , CD3 - Firma/Herkunft - Freundlicherweise zur Verfügung gestellt von Dr. M. Hudecek (Medizinische Klinik und Poliklinik II, Würzburg) Platinum A Amphotrophe Verpackungszelllinie Cell Biolabs, Inc. (San Diego, USA) T2 Zellen Humane Lymphoblast Zelllinie American Type Culture Collection (ATCC) verwendet als Feeder-Zellen. (Manassas, USA) 5.6.2 Bakterien Bakterien Eigenschaft Firma/Hersteller E. coli DH5α™ Chemisch kompetente Zellen (Genotyp: New England Biolabs (Frankfurt) fhuA2 Δ(argF-lacZ)U169 phoA glnV44 Φ80 Δ(lacZ)M15 gyrA96 recA1 relA1 endA1 thi-1 hsdR17) 36 Materialien 5.7 Verbrauchsmaterialien Verbrauchsmaterialien Firma/Hersteller 6-Well Zellkulturschalen Corning (Kaiserslautern) 24-Well Zellkulturschalen Corning (Kaiserslautern) 96-Well Mikrotiterplatten Corning (Kaiserslautern) Zellkulturschalen (10 cm) Corning (Kaiserslautern) 15 ml Zentrifugenröhrchen Falcon/BD (Franklin Lake,USA) 50 ml Zentrifugenröhrchen Falcon/BD (Franklin Lake,USA) 200 µl Reaktionsgefäße Sarsted (Nürnbrecht) 1,5 ml Reaktionsgefäße Sarsted (Nürnbrecht) FACS-Röhrchen Sarsted (Nürnbrecht) MACS-Trennsäulen Miltenyi Biotech (Bergisch-Gladbach) 5 ml Pipetten Corning (Kaiserslautern) 10 ml Pipetten Corning (Kaiserslautern) 25 ml Pipetten Corning (Kaiserslautern) 50 ml Pipetten Corning (Kaiserslautern) 1,5 ml Kryoröhrchen Greiner (Frickenhausen) Zellschaber SLP Life Science (über Hartenstein, Würzburg) Zellkulturflaschen (50 ml) Greiner (Frickenhausen) Zellkulturflasche (100 ml) Greiner (Frickenhausen) 5.8 Verwendete Analyse- Computerprogramme Programm Funktion Hersteller/Entwickler BD FACSDiva 8.0 Zur Aufnahme und Analyse Becton Dickinson (BD) durchflusszytometrischer Messungen (Heidelberg) durch den FACSCanto II. FCAP Array 2.0 Auswertung der CBA Daten Becton Dickinson (BD) (Heidelberg) FlowJo 7.6 Auswertung durchflusszytometrischer Treestar (Ashland, USA) Messungen GENtle Darstellung und Bearbeitung von Markus Manske (Köln) Sequenzierdaten und Vektorkarten. Simulierung des Restriktionsverdau. 37 Materialien Programm Funktion Hersteller/Entwickler GraphPad Prism 6 Statistische Auswertungen und GraphPad Software (La Jolla, grafische Darstellung USA) Datenbank zur Identifizierung der TCR gegründet 1989 von Marie- Ketten Paule Lefranc (Direktor) IMGT Datenbank (Montpellier Cedex 5, Frankreich) SnapGene Viewer 2.1.2 Darstellung und Bearbeitung von GSL Biotech LLC (Chicago, Vektorkarten. USA) 38 Methoden 6 Methoden 6.1 Kultivierung humaner Zellen und Zelllinien Bei der Kultivierung von Zellen wurde auf sterile Bedingungen geachtet. Es wurde mit sterilem Material, Lösungen und Medien unter einer Sterilbank gearbeitet, um Kontaminationen mit Bakterien, Hefen und Pilzen zu verhindern. 6.1.1 Zellen und ihre Kultivierungsbedingungen In dieser Arbeit wurde ausschließlich mit humanen Zellen gearbeitet. Es wurden primäre humane Zellen aus Vollblut gesunder Spender entnommen. Primäre Zellen, sowie aus ihnen generierte Klone und transduzierte primäre Zellen wurden in RPMI 1640 mit 10 %igem hitzeinaktiviertem Humanserum, 100 U/ml Penicillin und 100 µg/ml Streptomycin (Kulturmedium 1) kultiviert. Die humane TCR αβ Ketten-defiziente T-Zell Lymphom Zelllinie Jurkat 76 (Heemskerk et al. 2003) und ihre Transduktanden wurden in RPMI 1640 mit 10 %igem hitzeinaktiviertem FCS, 100 U/ml Penicillin und 100 µg/ml Streptomycin (Kulturmedium 2) expandiert. Für die Produktion von retroviralem Kulturüberstand wurde die amphotrophe Verpackungs-Zelllinie Platinum A (Cell Biolabs) verwendet, welche nach Angaben des Herstellers in DMEM Kulturmedium mit 10 %igem hitzeinaktiviertem FCS kultiviert wurde (Kulturmedium 3). Die Inkubation der Zellen erfolgte bei 37°C, 5 % CO2 und 95 %iger Luftfeuchtigkeit (Ausnahme: Generierung von retroviralem Kulturüberstand: 32°C, 5 % CO2 und 95 %iger Luftfeuchtigkeit). 6.1.2 Bestimmung der Zellzahl und Zellvitalität Die Zellzahl und ihre Vitalität wurden bestimmt, indem 10 µl der Zellsuspension 1:10 mit Trypanblau versetzt wurden. Die Farbstoff-Zell-Suspension wurde in eine Neubauer-Zählkammer überführt und unter einem Lichtmikroskop wurde die Zellzahl ermittelt. Mit Hilfe der Trypanblau-Färbung ist es möglich die Vitalität der Zellen sichtbar zu machen. Vitale lebende Zellen sind in der Lage den Farbstoff aktiv durch ihre Ionenkanäle abzugeben, während sich bei toten Zellen der Farbstoff anreichert und die Zellen blau einfärben. Die Vitalität wurde durch den Abzug der blaugefärbten und somit geschädigten Zellen von der Gesamtzahl ermittelt. 39 Methoden 6.1.3 Einfrieren und Auftauen von Zellen Zellen die nicht sofort für Experimente benötigt wurden und für weitere Experimente aufbewahrt werden sollten, wurden in Aliquote von mindestens 1x106 bis maximal 1x107 Zellen/ml in Kryoröhrchen überführt. Anschließend im entsprechendem Kulturmedium, versetzt mit 10 % DMSO (zur Verhinderung von Eiskristallbildung), in einem Einfriercontainer (Einfrierrate -1°C/min) bei -80°C eingefroren. Für längere Lagerung wurden die eingefrorenen Zellen im flüssigen Stickstoffdampf aufbewahrt. Das Auftauen von Zellen erfolgte durch schnelle Erwärmung in einem Wasserbad (37°C). Während des Auftauvorgangs wurden die Kryoröhrchen leicht geschwenkt. Die aufgetaute Zellsuspension wurde sofort in 10 bis 13 ml kaltes Kulturmedium 2 überführt, zentrifugiert (10 min, 365 x g) und der Überstand verworfen. Durch einen weiteren Waschschritt im für die Zellen entsprechenden Kulturmedium wurden die Reste des DMSO, welches toxisch für die Zellkultur wirkt, entfernt. Die aufgetauten Zellen wurden dann in ihrem spezifischen Kulturmedium aufgenommen, kultiviert (siehe 6.1.1.) bzw. in Experimenten eingesetzt. 6.2 6.2.1 Isolierung von Zellpopulationen Isolation peripherer mononukleärer Zellen aus Vollblut gesunder Spender Für die Isolierung von mononuklearen Zellen des peripheren Blutes (PBMC) wurde Blut von gesunden Spendern abgenommen und zu gleichen Teilen mit PBS versetzt. Mittels Dichtegradienten-Zentrifugation in Ficoll-Separierungslösung (Biochrom) wurde das verdünnte Blut in seine Bestandteile separiert (15 min, 699 x g, 20°C ohne Bremse). Die leukozytenreiche Interphase des Gradienten wurde abgenommen und zweimal in PBS gewaschen (10 min, 365 x g, 20°C). Die angereicherten PBMC wurden in Kulturmedium 1 aufgenommen. 6.2.2 Isolation von CD4+ T-Zellen via magnetischer Zell-Separation Bei der magnetischen Zell- Separation handelt es sich um eine Trennung nach zellspezifischen Oberflächenmolekülen, ermöglicht durch eine antikörpervermittelte magnetische Markierung (Beads) der Zellen. Die markierten Zellen werden dabei über eine Trennsäule in einem magnetischen Feld von nicht-markierten Zellen isoliert. Die magnetische Zellsortierung wurde mit dem sogenannten MACS®-Cell-Sorting-System von Miltenyi Biotec nach Angaben des Herstellers durchgeführt. Im Fall der CD4+ T-Zell Isolation wurde mit dem CD4+ Isolation Kit II gearbeitet. Dieses Kit ermöglicht eine indirekte Separation der CD4+ T-Zellen, ohne eine mögliche Voraktivierung durch die 40 Methoden Antikörperbindung. Für die + Separation wurden alle Zellen ausgenommen der 8 CD4 T-Zellen aus bis zu 1x10 PBMC magnetisch markiert. Eine Trennsäule (LS) in einem magnetischen Feld wurde eingesetzt, um CD4+ T-Zellen von den restlichen Zellen zu trennen. Die isolierten Subpopulationen wurden in Kulturmedium 1 aufgenommen und in weiteren Versuchen eingesetzt. 6.2.3 Isolation und Kultivierung von Monozyten Für die Isolation und Kultivierung von Monozyten wurden 1x107 PBMC in 2 ml Kulturmedium 1 pro Well einer 6-Well Zellkulturplatte ausgesät und für 2 Stunden inkubiert. Nicht adhärente Zellen wurden vorsichtig mit Kulturmedium 1 entfernt und die adhärenten Zellen über Nacht im Brutschrank bei 37°C kultiviert. Für die Präsentation von Peptid- Antigen wurden die separierten Zellen mit 1 µg/ml Peptid für eine Stunde inkubiert und im Anschluss mit Kulturmedium 1 gewaschen (10 min, 365 x g, 20°C). 6.2.4 Generierung dendritischer Zellen aus Monozyten Die Generierung dendritischer Zellen (DC) wurde nach dem Protokoll von Dauer et al. 2003 und Dauer et al. 2005 mit leichten Änderungen durchgeführt. Immature dendritische Zellen wurden aus CD14+ isolierten Zellen hergestellt. Die Isolation von CD14+ Zellen aus PBMC erfolgte mit Hilfe MACS®-Cell-Sorting-System der (siehe magnetischen auch 6.2.2). Über Separation positive durch das Selektion (unter Verwendung von CD14+ Beads) isolierte Zellen wurden mit 500 U/ml GM-CSF und 1400 U/ml IL-4 in Kulturmedium 1 in einer 6-Well Zellkulturschale im Brutschrank kultiviert. Die Zellen wurden nach 24 bis 36 Stunden mittels 25 ng/ml TNF-α, 5 ng/ml IL-1β, 10 ng/ml IL-6 und 1 µg/ml PGE2 maturiert. Nach weiteren 24 Stunden wurden die maturierten DC (mDC) abgenommen und in Experimenten eingesetzt oder eingefroren. Für die Peptid-Antigen Präsentation wurden die mDC mit 1 µg/ml Peptid für 1 Stunde bei 37°C inkubiert und anschließend mit Kulturmedium 1 gewaschen (10 min, 365 x g, 20°C). 6.3 6.3.1 Stimulation und Klonierung von T-Zellen Stimulation von T-Zellen und Generation A. fumigatus spezifischer T-Zelllinien Für die Generation A. fumigatus spezifischer T-Zellen wurde ein 15-mer Peptid aus der Zellwandglykonase Crf1 (Crf1/p41) verwendet. Crf1/p41 kann von unterschiedlichen MHC Klasse II Allelen präsentiert werden (HLA-DRB1*03, HLA-DRB1* 04 und HLA-DRB1*13 Allele, welche von 59,9% der kaukasischen Bevölkerung exprimiert werden) (Stuehler et al. 2011). Es wurden 1x107 PBMC pro Well einer 6-Well Zellkulturplatte mit 41 Methoden 1 µg/ml Crf1/p41 Peptid für 7 Tage bei 37°C im Brutschrank kultiviert. Jeden zweiten Tag wurden 5 U/ml IL-2 zur Kultur gegeben und bei Bedarf das Kulturmedium gewechselt. Nach 7 Tagen wurde die Kultur mit Crf1/p41 Peptid präsentierenden Monozyten (siehe 6.2.3) im Verhältnis 1:10 (Monozyten:Zellen) restimuliert und jeden zweiten Tag mit 5 U/ml IL-2 versorgt und Kulturmedium nach Bedarf ersetzt. Nach 14 Tagen wurde die Spezifität der generierten Zellen mit Crf1/p41 Tetramer und anti-CD4 Antikörper durchflusszytometrisch bestimmt. 6.3.2 Klonierung von Crf1/p41 spezifischen T-Zellen über Grenzverdünnung (limiting dilution) Mit dem Prinzip der Grenzverdünnung können Zellen einer Zellsuspension durch Verdünnung soweit vereinzelt werden, bis statistisch nur noch eine oder keine Zelle in einer Verdünnungsstufe vorhanden ist (bis zur Nachweisgrenze). Aus der vereinzelten Zelle kann eine monoklonale Zellkultur generiert werden. Für die Klonierung von Crf1/p41 spezifischen T-Zellklonen wurden PBMC für 14 Tage Crf1/p41 spezifisch stimuliert (siehe 6.3.1). Die stimulierten Zellen wurden mit MHC Klasse II Crf1/p41 Tetramer gefärbt (siehe 6.4.5). Durch den Fluoreszenzfarbstoff Phycoerythrin (PE), mit dem das Tetramer gekoppelt ist, wurden die spezifischen Crf1/p41 Tetramer positiven T-Zellen mit Hilfe von PE-Beads über das MACS®-Cell-Sorting-System selektiert. Für die Grenzverdünnung wurden 300 der isolierten spezifischen Zellen mit 7,5x106 bestrahlten T2 (60Gy) und 1x108 bestrahlten PBMC (30Gy) in 200 ml Kulturmedium 1 aufgenommen, mit 30 ng/ml OKT-3 und 50 U/ml IL-2 versetzt und in 10x 96-Well Mikrotiterplatten für 14 Tage bei 37°C kultiviert. Crf1/p41 spezifische Klone wurden im Anschluss mittels Crf1/p41 Tetramer und anti-CD4 Antikörper durchflusszytometrisch identifiziert. 6.3.3 Expansion humaner T-Zellklone und T-Zell-Rezeptor transduzierter T-Zellen Primäre humane T-Zellen, T-Zellklone und TCR transduzierte T-Zellen wurden nach dem Schnell-Expansions-Protokoll unter Verwendung des monoklonalen Antikörper CD3 angereichert (Riddell et al. 1990; Beck et al. 2006). Die TCR transduzierten Zellen wurden zunächst über das Crf1/p41 Tetramer mittels Fluoreszenzfarbstoff Phycoerythrin (PE), mit dem das Tetramer gekoppelt ist und über das MACS®-Cell-Sorting-System selektiert. Über PE-Beads wurden die Crf1/p41 Tetramer spezifischen Zellen im magnetischen Feld von anderen Zellen separiert. Es wurden 2x105 T-Zellen, TCR transduzierte Zellen oder 1x105 T-Zellklone mit 5x106 bestrahlten T2 Zellen (60Gy) und 2,5x107 bestrahlten PBMC (30Gy), welche als Stimulationszellen fungieren, kultiviert. Die Zellen wurden in 25 ml Kulturmedium 1 aufgenommen und mit 30 ng/ml OKT-3 versetzt. An Tag eins und 42 Methoden anschließend jeden dritten Tag wurden 50 U/ml IL-2 zugegeben, ebenso wurde an Tag 4 das Kulturmedium komplett entfernt, um überschüssigen CD3 Antikörper zu entfernen. Kulturmedium wurde nach Bedarf ersetzt. Nach 14 Tagen wurden die Zellen in weiteren Experimenten eingesetzt. 6.4 6.4.1 Phänotyp-Analyse und Untersuchungen der Effektor-Funktion von T-Zellen Restimulation von T-Zellen und T-Zell-Rezeptor transduzierten T-Zellen Für die Crf1/p41 spezifische Restimulation wurden 2x105 T-Zellen oder TCR transduzierte T-Zellen in Duplikaten in jeweils 200 µl Medium in 96-Well Kulturplatten ausgesät. Stimuliert wurden die Zellen mit Crf1/p41 beladenen Monozyten oder mDC (siehe 6.2.3 und 6.2.4) in einem Verhältnis von 1:5 (Monozyten oder mDC:Zellen). Nach 24 Stunden wurde der Kulturüberstand abgenommen und die IFN-γ Konzentration wurde mit IFN-γ Enzym Linked Immunosorbent Assay (ELISA) oder über Cytometric Bead Array (CBA) ermittelt. 6.4.2 Interferon-γ Enzym- Linked- Immunosorbent Assay Zur Identifizierung Crf1/p41 spezifischer Effektor T-Zellklone wurde die IFN-γ Produktion nach Crf1/p41 spezifischer Stimulation mittels ELISA bestimmt. Der ELISA ist ein enzymgekoppelter Immunabsorptionstest, im Fall des Zytokin IFN-γ wurde ein Sandwich-ELISA durchgeführt (Abbildung 7). Für diesen Test wurden ELISA Platten mit 100 µl/Well Endogen M700A Antikörper (0,3 µg/ml, anti-IFN-γ Antikörper; Perbio), aufgenommen in Karbonat-Bikarbonat Puffer (pH von 9,6), über Nacht bei 4°C, beschichtet. Die Platten wurden dreimal mit ELISA Waschpuffer (PBS mit 0,05 % Tween20) gewaschen und mit 200 µl/Well RDI-PHRPDIL Blocker (Fitzgerald) für eine Stunde bei Raumtemperatur geblockt. Der Blocker wurde entfernt und in Duplikaten wurden 50 µl/Well der Proben in verschiedenen Verdünnungen (1:20; 1:100), sowie ein IFN-γ Standard (0-200 ng/ml) in die Platten pipettiert. Die ELISA Platten wurde für 90 Minuten auf einem Schüttler bei Raumtemperatur inkubiert. 100 µl/Well von Endogen M701B Antikörper (0,5 µg/ml, anti-IFN-γ Antikörper; Perbio), aufgenommen in Blocker-Puffer, wurden zugegeben und für weiter 90 Minuten inkubiert. Die Platten wurden fünfmal mit ELISA Waschpuffer gewaschen und 30 Minuten mit 100 µl/Well RDI-PHRP20 Streptavidin (250 ng/ml; Fitzgerald), aufgenommen in Blocker-Puffer inkubiert. Im Anschluss wurden die Platten sechsmal gewaschen und nach einer Inkubation von 5 bis 15 Minuten mit 100 µl/Well ABTS Substratlösung wurde die 43 Methoden Absorption bei 405 nm gemessen. Durch Mitführen eines Standards konnte die IFN-γ Konzentrationen quantifiziert werden. Abbildung 7: Prinzip des IFN-γ ELISA. Dargestellt ist das Prinzip des IFN-γ ELISA (Sandwich-ELISA) Mit den anti-IFN-γ Antikörpern Endogen M700A (Coating-Antikörper) und Endogen M701B (detektierender Antikörper). Bei der Bindung des Detektier-Antikörper mit IFN-γ wurde eine enzymatische Reaktion durch das Biotin-Streptavidin-Konjugat ausgelöst, wodurch das farblose Substrat ABTS in ein farbiges Produkt umgewandelt wurde. Die Absorption wurde bei einer Wellenlänge von 405 nm gemessen und mit Hilfe eines Standards können die IFN-γ Konzentration berechnet werden. 6.4.3 Durchflusszytometrische Messungen Bei der Durchflusszytometrie wird eine Zellsuspension durch eine Durchflussmesszelle geleitet, dabei passiert jede einzelne Zelle den Messbereich eines Laserstrahls. Das Streulicht wird detektiert und vom Computer registriert. Dabei ist das Vorwärtsstreulicht (FCS = Forward Scatter) ein Maß für die Beugung des Lichts im flachen Winkel und hängt von der Größe der Zelle ab. Beim Seitwärtsstreulicht (SSC = Side Scatter) handelt es sich um ein Maß für die Brechung des Lichts im rechten Winkel, welche von der Granularität der Zelle, Struktur ihres Zellkerns und der Vesikel in der Zelle abhängig ist. Neben dem Streulicht können auch Fluoreszenzfarben eingesetzt werden, die mit Hilfe von Antikörpern an bestimmte Bestandeile der Zellen (z.B. Oberflächenmoleküle) binden. Durch Einsatz von verschiedenfarbigen Lasern und Filtern ist es möglich die Anzahl der einsetzbaren Farbstoffe zu erhöhen. Wodurch die zu untersuchenden Zellpopulationen in einer Messung detaillierter charakterisiert werden können. 44 Methoden Für die durchflusszytometrische Analyse wurden 1x106 Zellen in 5 ml FACS-Röhrchen überführt, in FACS-Puffer (PBS und 0,2 % BSA) aufgenommen und zentrifugiert (10 min, 365 x g, 7°C). Die Zellen wurden in 80 µl FACS- Puffer aufgenommen und mit Fluoreszenz gekoppelten Antikörpern (je nach Antikörper 1 bis 10 µl) für 20 Minuten bei 4°C inkubiert. Die Zellen wurden erneut gewaschen, in 150 µl FACS-Puffer aufgenommen und am Durchflusszytometer FACS Canto™ oder FACS Calibur™ (Becton Dickinson, BD) gemessen. Die Daten wurden mit der Software FACSDiva 8.0 (BD) und dem Programm FlowJo® 7.6 (TreeStar Inc.) ausgewertet. 6.4.4 Bestimmung der T-Zell-Rezeptor β Kette von Crf1/p41 spezifischen Klonen mittels Durchflusszytometer Zur Analyse des TCR β Ketten Repertoire wurde der IO Beta Mark TCR Kit von Beckman Coulter Inc. (Krefeld) verwendet. Die Durchführung erfolgte nach Angaben des Herstellers und beruht auf der Markierung von TCR β Ketten durch unterschiedliche FluoreszenzAntikörper, die mittels Durchflusszytometer identifiziert wurden. Mit diesem Kit können 24 verschiedene Vβ Ketten Familien untersucht werden (Tabelle 2). Tabelle 2: Vβ Antikörper aus dem IO Beta Mark TCR Kit (Beckman Coulter). Tabelle der 24 eingesetzten Antikörper und ihrer Fluorochrome zur Bestimmung von 24 verschiedenen Vβ Ketten Familien. Tabelle stammt aus dem IO Beta Mark TCR Kit Manual. 45 Methoden 6.4.5 MHC Klasse II Tetramer Färbung Die Identifizierung Crf1/p41 spezifischer T-Zellen, T-Zellklone und TCR transduzierten T-Zellen wurde über das MHC Klasse II Tetramer (Crf1/p41 spezifisch) verifiziert. Für die Crf1/p41 Tetramer Färbung wurden 1x106 T-Zellen oder 5x105 T-Zellklone pro 50 µl Kulturmedium 1 mit 0,5 bis 1 µl Crf1/p41 Tetramer für 2 Stunden bei 37°C kultiviert. Die Zellen wurden mit 2 ml Waschpuffer (PBS mit 0,1 % BSA) gewaschen (7 min, 365 x g, 20°C). Im Anschluss wurden noch weiter Oberflächenmoleküle mit Fluoreszenz gekoppelten Antikörpern gefärbt (siehe 6.4.3). 6.4.6 Messung der Proliferation nach Crf1/p41 spezifischer Stimulation Um die Zellproliferation von TCR transduzierten T-Zellen nach Crf1/p41 spezifischer Stimulation zu verfolgen, wurde der Fluoreszenzfarbstoff eFluor®670 verwendet. Der Fluoreszenzfarbstoff bindet an zelluläre Proteine die primäre Amine enthalten, welche bei der Zellteilung zu gleichen Teilen an die Tochterzellen abgegeben werden. Das wiederholte Ausdünnen des Fluoreszenzfarbstoffs bei Zellteilung wurde 7 durchflusszytometrisch über die Fluoreszenzintensität verfolgt. Bis zu 1x10 transduzierte Zellen (Crf1/p41 spezifisch) wurden mit PBS gewaschen, um das Kulturmedium 1 vollständig zu entfernen. Anschließend wurden die Zellen mit dem Fluoreszenzfarbstoff eFluor®670 für 10 Minuten bei Raumtemperatur im Dunkeln inkubiert. Die Reaktion wurde mit der Zugabe von FCS gestoppt und die nun gefärbten Zellen wurden dreimal mit Kulturmedium 2 gewaschen (10 min, 365 x g), um überschüssigen Farbstoff und überschüssiges FCS zu entfernen. 2x105 der gefärbten Zellen wurden in Duplikaten in jeweils 200 µl Medium in 96-Well Kulturplatten ausgesät. Stimuliert wurden die Zellen mit Crf1/p41 beladenen mDC (siehe 6.2.3 und 6.2.4) in einem Verhältnis von 1:5 (mDC:Zelle). Für die Positivkontrolle wurden die Zellen mit CD3/CD28 Dynabeads®, für die Negativkontrolle mit unbeladenen mDC stimuliert. Die Proliferationsfähigkeit der TCR transduzierten Zellen nach Crf1/p41 spezifischer Stimulation wurde über die Fluoreszenzintensität mit dem Durchflusszytometer ermittelt. 6.4.7 Mit Cytometric Bead Array (CBA) dem CBA wurde die Zytokinkonzentration im Kulturüberstand nach Crf1/p41 spezifischer Stimulation (siehe 6.4.1) mittels Durchflusszytometrie gemessen. Bei einem CBA kommen Beads bekannter Größe und Fluoreszenz zum Einsatz. Capture-Beads (Fluoreszenz konjugiert), welche aus einem Capture-Antikörper spezifisch für ein bestimmtes Zytokin bestehen, bilden einen Sandwich-Komplex mit einem 46 Methoden Detections-Reagenz, einem Mix aus PE-konjugierten Antikörpern, welche ein Fluoreszenzsignal in der Abhängigkeit von gebundenem Zytokin im Durchflusszytometer messbar machen. Durch das Mitführen eines Standards wurde eine Standardkurve ermittelt, die zur Berechnung der Konzentration der Zytokine verwendet wurde. Die Auswertungen wurden mit dem Programm FCAP-Array 2.0 durchgeführt. 6.5 6.5.1 Isolation und Identifizierung von T-Zell-Rezeptor Ketten Isolation von RNA aus Crf1/p41 spezifischen T-Zellklonen Für die Identifizierung der Crf1/p41 spezifischen TCRs ist es notwendig, die RNA aus den T-Zellklonen zu isolieren. Die RNA ist Grundlage für die RACE cDNA Amplifikation (siehe 6.5.2). Für die RNA Isolierung wurde der RNeasy Mini Kit (Qiagen) verwendet. 5 bis 10x106 Crf1/p41 spezifischer T-Zellklone wurden dreimal mit PBS gewaschen (7 min, 300 x g) und so von Kulturmedium befreit. Die pelletierten T-Zellklone wurden bei -80°C eingefroren und für spätere RNA Isolationen verwendet. Bei der Isolation von RNA wurden im ersten Schritt die Zellwände, Membranen und Organellen der pelletierten Zellen lysiert. Die Homogenisierung der genomischen DNA und der zellulären Komponenten mit hohem Molekulargewicht erfolgte über QIAshredder-Säulen (enthalten im RNeasy Mini Kit) und Zentrifugation (2 min, 8000 x g). Im nächsten Schritt wurde das Lysat mit 70 %igem Ethanol versetzt und über RNeasy Zentrifugen-Röhrchen (enthalten im RNeasy Mini Kit) wurde die Gesamt-RNA der Zellklone in der Membran aufgefangen (15 s, 8000 x g). Nach dreimaligem Waschen wurde die RNA von der Membran der Säulen eluiert (1 min, 8000 x g) (Abbildung 8). Aufgrund ihrer Instabilität wurde die RNA in Aliquote bei -80°C gelagert. Abbildung 8: RNA Isolierung aus humanen Zellen mit dem RNasy Mini Kit (Qiagen). Humane Zellen wurden lysiert, homogenisiert (über QIAschredder-Röhrchen; 2 min, 8000 x g) und mit Ethanol versetzt. Die totale RNA wird an der Membran eines Zentrifugen-Röhrchen gebunden (15 s, 8000 x g) und über Zentrifugation dreimal gewaschen (15 s, 8000 x g). Mit 30 µl RNasefreiem Wasser wurde die RNA eluiert (1 min, 8000 x g). 47 Methoden 6.5.2 Isolation und Identifikation unbekannter T-Zell-Rezeptor Sequenzen mittels RACE-PCR Mit Hilfe der SMARTer RACE cDNA Amplifikation (Clontech Laboratories) ist es möglich, die komplette cDNA beginnend am 5´ Ende der zu identifizierenden Sequenz aus totaler RNA Crf1/p41 spezifischer Klone zu isolieren (Abbildung 9). Abbildung 9: Mechanismus der SMARTer cDNA Synthese. Im ersten Schritt wird ein modifizierter Oligo(dT) Primer für die „First-Strand-Synthese“ verwendet. Hat die SMARTScribe Reverse Transkriptase (RT) das Ende des mRNA Template erreicht werden einige zusätzliche Nucleotide angehängt. Die SMART II A Oligonucleotide binden an das Ende der cDNA und fungieren als erweitertes Template für die SMARTScribe RT. (Abbildung aus „Clontech SMARTer Race cDNA Amplification Kit User Manual“) Die SMARTer RACE cDNA Amplifikation wurde nach Angaben des Herstellers durchgeführt. Für den ersten Syntheseschritt wurde ein Oligo(dT) Primer verwendet, der am polyA 3´ Ende der mRNA bindet. Hat die SMARTScribe reverse Transkriptase (RT) das 5´ Ende des mRNA Template erreicht, wurden einige zusätzliche Nucleotide angehängt. Dabei fungieren die SMART II Oligonucleotide als erweitertes Template für die SMARTScribe RT, in dem sie an das Ende der cDNA binden. Die generierte 5´ cDNA dient als Ausgangsmaterial für RACE PCR zur Anreicherung und Identifizierung der variablen Ketten der Crf1/p41 spezifischen TCR. 48 Methoden Abbildung 10: SMARTer RACE PCR. Durchführungsschema der SMARTer RACE PCR zur Identifizierung von variabler α (A) und β (B) Kette des TCR. Der SMARTer RACE PCR Reaktionsansatz und die Einstellungen während der PCR sind in (C) aufgelistet. Für die SMARTer RACE PCR Reaktion wurde die 5 ´cDNA mit einem Universalprimer A Mix, welcher am 5´ Ende der cDNA bindet und mit einem rückwärts orientierten Primer, der zu Beginn der konstanten Region der α bzw. β-Kette des TCR lokalisiert ist, angesetzt (Primerliste siehe Tabelle 1). Mit Hilfe der PCR wurde die vollständige variable Region für die Identifikation amplifiziert (Abbildung 10). Mittels Gelelektrophorese (siehe 6.5.3) wurde die korrekte Länge, über die Anzahl der 49 Methoden Basenpaare (bp), des PCR-Produkts kontrolliert (Vα ~ 600bp; Vβ ~ 650bp). Anschließend wurde das PCR Produkt aus dem Gel ausgeschnitten und mit dem NucleoSpin® Gel und PCR Clean-up Kit (Macherey Nagel) aufgereinigt. Die Proben wurden für die Sequenzierungen an GATC-Biotech (Konstanz) gesendet. Für die Analyse der Sequenzier-Ergebnisse wurde das Programm GENtle von Magnus Manske (Köln) und die Datenbank von Immunogenetics (IMGT) (Lefranc 2004) verwendet. 6.5.3 Gelelektrophorese Mit der Gelelektrophorese wurden die Längen der cDNA und der DNA aus den PCR Reaktionen überprüft. Für die Gelelektrophorese wurden 1 %ige Agarosegele hergestellt, dafür wurde 1 g Agarose mit 100 ml TAE-Puffer vermischt und zum Kochen gebracht. Nachdem das Gel auf ca. 60°C abgekühlt war, wurden 5 µl Midori Green Advance (DNA-Farbstoff) zugegeben. Das Gel wurde in eine Gelkammer gegossen und in das Gel wurde ein Kamm gesetzt, um Taschen für die DNA Proben zu formen. Die Gelkammer wurde mit TAE Puffer gefüllt und das abgekühlte und fest gewordene Gel hineingelegt. Die DNA Proben wurden mit 6x Ladepuffer versetzt und in die vorgesehen Taschen des Gels pipettiert. Ebenfalls wurde ein Marker (1kb) aufgetragen, um die Länge der DNA Fragmente in den Proben zu bestimmen. Die DNA Fragmente wurden durch die Gelelektrophorese ihrer Länge nach getrennt (30 min, 100- 120 V). Mittels UV-Licht wurden die Banden durch den DNA Farbstoff Midori Green sichtbar gemacht. 6.6 Klonierung retroviraler Vektoren und Transduktion in humane Zelllinien und primäre T-Zellen 6.6.1 Klonierung retroviraler Vektoren mit A. fumigatus spezifischen T-Zell-Rezeptoren Für die Klonierung wurde die zuvor amplifizierte SMARTer RACE 5´ cDNA in einer PCR zur Isolierung von Crf1/p41 spezifischen TCR α und TCR β Ketten verwendet (Abbildung 11). In der PCR wurden spezifische Primer eingesetzt, die am 5´Ende des TCR und am 3`Ende der konstanten Region binden (Primerliste Tabelle 1). Nach der PCR wurde die DNA in einer Gelelektrophorese auf die korrekte Länge hin überprüft (TCR α ~ 850bp; TCR β ~ 950bp) und mit dem NucleoSpin® Gel Clean-up Kit (Macherey-Nagel) nach Herstellerangaben aufgereinigt. 50 Methoden Abbildung 11: PCR zur Anreicherung von TCRs. Durchführungsschema der PCR zur Isolierung von TCR α (A) und TCR β Ketten (B) aus 5´ SMARTer RACE cDNA von Crf1/p41 spezifischen Klone. In (C) sind der PCR- Ansatz und die PCR Einstellungen zur Anreicherung der TCRs dargestellt. * Die Primer der jeweiligen TCRs sind in Tabelle 1 aufgelistet. Die TCR α Kette, die TCR β Kette und eGFP wurden jeweils über die Schnittstellen Not1 und EcoR1 in den Vektor pMP71 kloniert (Abbildung 12 A, B, C). Für eine parallele Expression beider TCR Ketten wurden die Gene mit einem 2A Peptidlinker (p2A) (Szymczak et al. 2004) verbunden und in einen Vektor kloniert (Abbildung 12 D). Das Gen von Vektor D wurde vor der Klonierung von GeneArt (Life Technologies) einer Codonoptimierung unterzogen und die konstanten Ketten wurden durch murine konstante 51 Methoden Ketten ersetzt (siehe 3.5). Das optimierte und kombinierte TCR α - p2A - TCR β Element wurde ebenfalls über die Schnittstellen Not1 und EcoR1 in den Vektor pMP71 kloniert. Abbildung 12: Vektorkarten des retroviralen Vektor pMP71. Die verschiedenen retroviralen Vektoren wurden für die Transduktion der TCRs eingesetzt. pMP71 Vektoren sind mit einem long terminal repeat des myeloproliferative Sarcoma Virus (MPSV-LTR) und einem Woodchuck-Hepatitis-Virus posttranskriptionalen regulatorischen Element (PRE) ausgestattet und besitzen eine Ampicillinresistenz (AmpR). In (A) mit der TCR α Kette, (B) mit der TCR β Kette, (C) mit eGFP und in (D) sind TCR α Kette und TCR β Kette getrennt durch einen Peptidlinker (p2A). Für den Restriktionsverdau wurden das amplifizierte Insert, das TCR α - p2A - TCR β Element sowie der pMP71 Vektor mit den Restriktionsenzymen Not1 und EcoR1 verdaut (1 h, bei 37°C) und anschließend per Gelelektrophorese aufgetrennt. Die richtigen Banden wurden aus dem Gel ausgeschnitten und mittels NucleoSpin® Gel Clean-up aufgereinigt. Die Ligation von Vektor und Insert (isolierte TCR α, TCR β Kette oder TCR α - p2A - TCR β Element) wurde mit T4 Ligase ausgeführt (Inkubation 1 h bei 16°C). Vor der Transformation wurden die Ligationsansätze mit dem NucleoSpin® Gel and PCR Clean-up Kit gereinigt. Die Transformation der Vektor-DNA in 5α chemisch kompetente E.coli (NEB) wurde nach Angaben des Herstellers durchgeführt. Die transformierten Bakterien wurden auf Ampicillin (100 µg/ml)-LB-Agar Platten ausgestrichen. Durch das Antibiotikum Ampicillin vermehrten sich nur die Bakterien, die den Vektor pMP71 aufgenommen haben, da dieser eine Ampicillinresistenz besitzt. Nach 24 Stunden wurde von den Bakterienkolonien eine Kultur mit Ampicillin (100 µg/ml)-LB-Medium angesetzt. Von der Bakterienkultur wurde die Plasmid-DNA über das Plasmid DNA Purification Kit (Macherey Nagel) isoliert. Die Vektor-DNA wurde für die Transfektion von Platinum A Zellen (Verpackungszellen) verwendet. 52 Methoden 6.6.2 Produktion von retroviralem Kulturüberstand Um Crf1/p41 spezifische TCRs auf T-Zellen zu transduzieren, wurde retroviraler Überstand mit der amphotrophen Verpackungszelllinie Platinum A (Cell Biolabs.Inc) generiert. Die Zellen wurden in Zellkulturschalen von 10 cm Durchmesser bis zu einer Konfluenz von 60 bis 80 % bei 37°C im Brutschrank in Kulturmedium 3 (DMEM Kulturmedium mit 10 % FCS) nach Angaben des Herstellers expandiert. Für die Transfektion der Platinum A Zellen mit retroviraler Vektor-DNA wurde das Effectene® Transfektions Reagenz nach Herstellerangaben der Firma Qiagen GmbH (Hilden) verwendet. Eingesetzt wurden 2 µg Vektor-DNA (1 µg TCR α und 1 µg TCR β). Der entstandene DNA-Effectene-Komplex wurde tropfenweise auf die Verpackungszelllinie Platinum A gegeben und bei 32°C für 48 Stunden kultiviert. Anschließend wurde der Überstand abgenommen und filtriert. Nicht sofort verwendeter Virusüberstand wurde in Aliquote bei -80°C eingefroren. 6.6.3 Transduktion von humanen Zelllinien und primären T-Zellen mit A. fumigatus spezifischen T-Zell-Rezeptoren Die Transduktion spezifischer TCRs auf humane Zelllinien bzw. primäre Zellen wurde mit RetroNectin® (Clonetech Laboratories) beschichteten Zellkulturplatten und generiertem retroviralem Virusüberstand durchgeführt (siehe 6.6.2). Durch RetroNectin® wurden die Viruspartikel und die Zielzellen räumlich zusammengeführt, wodurch die Transduktionseffizienz erhöht wurde (Abbildung 13). Für eine erste Überprüfung der Transduzierbarkeit der Crf1/p41 spezifischen TCRs wurde die Zelllinie Jurkat 76 verwendet. Es handelt sich bei dieser Zelllinie um eine TCR und CD3 negative humane T-Zell Lymphom Jurkat Zelllinie. Der exogene TCR muss nicht mit endogenen TCRs konkurrieren und dessen Expression kann mit Hilfe von anti-CD3 Antikörper und Crf1/p41 Tetramer durchflusszytometrisch verfolgt werden. Für die Transduktion spezifischer TCR in primäre Zellen wurden CD4+ T-Zellen isoliert (siehe 6.2.2) und für zwei Tage mit CD3/CD28 Dynabeads® (Invitrogen) aktiviert. Die Beschichtung von 24-Well Zellkulturplatten mit RetroNectin® erfolgte einen Tag vor der Transduktion und wurde nach Angaben des Herstellers durchgeführt. Am Tag der Transduktion wurden die beschichteten Platten für 30 Minuten mit PBS und 2 % BSA geblockt und mit PBS gewaschen, um überschüssiges BSA zu entfernen. 1 ml Virusüberstand pro Well wurde in die RetroNectin®- beschichteten Platten gegeben und für 30 Minuten bei 32°C inkubiert. Die Jurkat 76 Zellen bzw. die aktivierten CD4+ T-Zellen wurden zum Virusüberstand gegeben und zentrifugiert (1 Stunde, 699 x g, 32°C). Die CD4+ T-Zellen erhielten zusätzlich 30 U/ml IL-2. Für die transduzierten CD4+ T-Zellen und die transduzierten 53 Methoden Jurkat 76 Zellen betrug die Inkubation 48 Stunden bei 37°C. Die erfolgreiche Transduktion wurde durchflusszytometrisch mit Crf1/p41 Tetramer, anti-CD4 Antikörper (bei + CD4 T-Zellen) bzw. anti-CD3 Antikörper (bei Jurkat 76) überprüft. Es wurde ein Kontrollvektor mit grün fluoreszierenden Protein (GFP) eingesetzt, um die Transduktion auch unter dem Lichtmikroskop bei UV-Licht verfolgen zu können. Um die TCR transduzierten Zellen weiter zu untersuchen (z.B. Crf1/p41 spezifische Stimulation zur Untersuchung der Proliferation oder Zytokinanalyse) wurden diese über das Crf1/p41 Tetramer und das MACS®Cell-Sorting-System angereichert und weiter expandiert (siehe 6.3.3). Abbildung 13: Hypothetischer Mechanismus der RetroNectin® vermittelten Transduktion. Die Zellen binden über die CS-1 Region über VLA-4 und über die C-Domäne durch VLA-5. Die Viruspartikel können über die H-Domäne des RetroNectin® binden. Retronectin schafft die räumliche Nähe zwischen Viruspartikel und Zielzelle, wodurch eine höhere Transduktionseffizienz erreicht wurde. (Abbildung aus der RetroNectin® Produktanleitung) 54 Ergebnisse 7 Ergebnisse 7.1 Generierung A. fumigatus spezifischer T-Zellklone Mit dem Ziel A. fumigatus spezifische TCRs zu identifizieren und zu charakterisieren, wurden zunächst T-Zellklone hergestellt und auf die Expression eines A. fumigatus spezifischen TCR untersucht. Für die Charakterisierung wurde ein 15-mer Peptid aus der Zellwandglykonase Crf1 (Crf1/p41) verwendet, das eine hohe Immunogenität besitzt und von Personen erkannt wird, die das MHC Klasse II Allel HLA-DRB1*03, HLA-DRB1*04 oder HLA-DRB1*13 exprimieren (dies entspricht einer Abdeckung von 59,9 % der kaukasischen Bevölkerung) (Stuehler et al. 2011). Nach Expansion, Isolierung und Klonierung wurde die Spezifität und Effektor-Funktion der Crf1/p41 spezifischen CD4+ T-Zellklone, über die IFN-γ Produktion nach einer Crf1/p41 spezifischen Stimulation bestimmt. 7.1.1 Expansion Crf1/p41 spezifischer T-Zellen Es hat sich gezeigt, dass nach der Isolierung von mononuklearen Zellen des peripheren Blutes (PBMC) aus dem Vollblut gesunder HLA-DRB1*04+ Spender, die Vorläuferfrequenz von Crf1/p41 spezifischen CD4+ T-Zellen nicht nachweißbar ist (Stuehler et al. 2011). Um Crf1/p41 positive CD4+ T-Zellen nachweisen zu können, mussten diese zunächst innerhalb der PBMC Crf1/p41 spezifisch expandiert werden. Dazu wurden PBMC mit dem Crf1/p41 Peptid und unter Zugabe von IL-2 kultiviert. Nach einer Woche erfolgte eine Restimulation der Zellen mit Crf1/p41 Peptid beladenen Monozyten und Zugabe von IL-2. Die Zellen wurden nach 14-tägiger Expansion mit Crf1/p41 spezifischem Tetramer und einem anti-CD4 Antikörper gefärbt und durchflusszytometrische analysiert (Abbildung 14). Eine Crf1/p41 spezifische CD4+ T-Zellpopulation wurde nachgewiesen, deren Anteil an der PBMC-Population bei 1,3 % (Spender 1), 5,35 % (Spender 2) und 7,8 % (Spender 3) lag (Abbildung 14 B). 55 Ergebnisse A Spender 1 (DRB1*0408) Spender 2 (DRB1*0401) Spender 3 (DRB1*0401) CD4 ohne Stimulation B 14 Tage Stimulation Tetramer-Crf1/p41 Abbildung 14: Expansion Crf1/p41 spezifischer T-Zellen aus PBMC von drei verschiedenen Spendern mit HLA-DRB1*04 Allel. Für die Expansion wurden PBMC von drei verschiedenen Spendern für 14 Tage ohne (A) und mit Crf1/p41 Peptid (B) stimuliert. Crf1/p41 spezifische + CD4 T-Zellen wurden mit Crf1/p41 spezifischem Tetramer und anti-CD4 Antikörper durchflusszytometrisch ermittelt. 7.1.2 Spezifitätstest der hergestellten T-Zellklone via Crf1/p41 Tetramer Im Anschluss an die Expansion Crf1/p41 spezifischer T-Zellen, wurden diese mit Hilfe des Crf1/p41 Tetramers und magnetischer PE-Beads angereichert und durch limiting dilution vereinzelt. Auf diese Weise erhaltene Klone wurden erneut auf ihre Spezifität überprüft, um ausschließlich Crf1/p41 spezifische Klone auf ihre TCR näher zu analysieren. 56 Ergebnisse CD4 A Spender 1 (DRB1*0408) Spender 2 (DRB1*0401) Spender 3 (DRB1*0401) Klon 48 Klon 71 Klon 78 Klon 1B5 Klon 1F6 Klon 8F3 B Tetramer-Crf1/p41 Abbildung 15: Crf1/p41 Spezifität der generierten Klone. Crf1/p41 spezifische T-Zellen wurden über eine Verdünnungsreihe mit bestrahlten Stimulationszellen (T2 Zellen und allogenen PBMC) in Anwesenheit eines monoklonalen anti-CD3 Antikörpers und IL-2 über zwei Wochen kloniert. Die + generierten Crf1/p41 spezifischen CD4 T-Zellklone wurden mittels Crf1/p41 Tetramer und anti-CD4 Antikörper durchflusszytometrisch identifiziert. Es sind repräsentativ je ein positiver (A) und eine negativer Klon (B) pro Spender, dargestellt. Abbildung 15 zeigt repräsentativ jeweils einen Crf1/p41 spezifischen Klon und einen Crf1/p41 unspezifischen Klon von drei unterschiedlichen Spendern. Es wurden insgesamt 64 potentielle Klone von Spender 1, 25 von Spender 2 und 27 potentielle Klone von Spender 3 durchflusszytometrisch auf ihr Crf1/p41 und CD4 Spezifität untersucht. Zusammenfassend konnten 52 Crf1/p41 spezifische CD4+ Klone von Spender 1 (Tabelle 3), 20 von Spender 2 (Tabelle 4) und 21 Crf1/p41 spezifische CD4+ Klone von Spender 3 (Tabelle 5) identifiziert werden. Dabei ergab sich, dass von allen untersuchten potentiellen Klonen von Spender 1 und Spender 2 80 % und von Spender 3 78 % Crf1/p41 spezifisch und CD4+ sind. 57 Ergebnisse Tabelle 3: Ergebnisse der durchflusszytometrischen Messung von potentiellen Crf1/p41 spezifischen T-Zellklonen von Spender 1. Durch limiting dilution kultivierte Crf1/p41und CD4 positive Klone sind grau hervorgehoben und haben einen Klonnamen erhalten. Angaben in % der Gesamt- Population. Probe Crf1/p41 Tetramer + Crf1/p41 Tetramer - Crf1/p41 Tetramer - CD4 + CD4 + CD4 - Angaben in % Angaben in % Crf1/p41 Tetramer + Klonname CD4 - Angaben in % Angaben in % 1B5 0,52 99,42 0,03 0,03 ~ 3F8 99,03 0,93 0,01 0,02 1 1H5 99,35 0,57 0,04 0,04 2 1D12 13,58 86,29 0,05 0,08 ~ 4A4 99,33 0,53 0,08 0,06 3 3C5 99,01 0,86 0,06 0,07 4 5A5 98,4 1,5 0,05 0,05 5 5B11 99,1 0,82 0,07 0,02 6 1C6 0,06 99,92 0,01 0,01 ~ 2E7 98,43 1,52 0,03 0,02 7 5E7 99,57 0,42 0 0,01 8 6C11 98,82 1,02 0,09 0,08 9 6G5 98,31 1,54 0,11 0,04 10 2A1 0,04 99,93 0,02 0,01 ~ 7G6 99,54 0,42 0,04 0 11 4B8 96,37 3,27 0,2 0,17 12 7C2 97,31 2,67 0,02 0 13 7D5 99,34 0,63 0,01 0,02 14 2F2 99,46 0,48 0,04 0,02 15 2F8 97,96 1,84 0,13 0,07 16 2E10 99,06 0,84 0,07 0,03 17 2B4 0,72 8,48 2,23 88,57 ~ 1H12 99,5 0,43 0,03 0,04 18 6A5 98,92 0,97 0,1 0,01 19 7E8 90,03 9,89 0,04 0,04 20 4G9 98,61 1,35 0,02 0,02 21 6H2 99,23 0,66 0,07 0,05 22 2C4 61,46 38,45 0,02 0,07 ~ 4B6 99,42 0,48 0,02 0,08 23 6C8 98,59 1,37 0,02 0,02 24 1H10 97,44 2,49 0,02 0,05 25 2C12 7,25 88,56 0,13 4,06 ~ 2D11 0,11 99,84 0,02 0,03 ~ 4B2 99,48 0,44 0,01 0,07 26 4C11 99,43 0,53 0,02 0,02 27 2B2 84,17 15,8 0,02 0,01 28 3A2 99,53 0,38 0,04 0,05 29 2C8 99,17 0,83 0 0 30 4D2 98,98 0,98 0,01 0,03 31 58 Ergebnisse Probe Crf1/p41 Tetramer + Crf1/p41 Tetramer - Crf1/p41 Tetramer - Crf1/p41 Tetramer + Klonname CD4 + CD4 + CD4 - CD4 - Angaben in % Angaben in % Angaben in % Angaben in % 1B6 64,35 35,62 0,02 0,01 32 3F9 98,21 1,75 0,03 0,01 33 5A10 99,06 0,9 0,03 0,01 34 2C7 99,2 0,76 0,01 0,03 35 3B9 0,09 99,84 0,03 0,04 ~ 1F12 99,6 0,36 0,03 0,01 37 3D7 99,38 0,52 0,03 0,07 38 2C3 98,4 1,32 0,01 0,28 39 4C8 99,51 0,39 0,03 0,07 40 3D9 97,44 2,53 0,02 0,02 41 1A10 93,52 6,43 0,05 0 42 5C7 99,58 0,34 0,05 0,03 43 4B9 98,5 1,43 0,02 0,05 44 1D9 98,95 1,02 0,02 0,01 45 1C10 99,35 0,54 0,04 0,06 46 3E6 0,07 99,92 0,01 0 ~ 1H8 99,12 0,72 0,14 0,02 47 3C2 99,53 0,44 0,02 0,01 48 4B5 63,92 36,02 0,04 0,01 ~ 5C8 97,63 2,32 0,03 0,02 49 6F11 98,13 1,73 0,11 0,03 50 7B8 98,89 0,98 0,08 0,05 51 3C10 99,44 0,49 0,02 0,05 52 5A8 3,85 96,1 0,01 0,04 ~ 6B11 20,66 79,22 0,06 0,06 ~ Tabelle 4: Ergebnisse der durchflusszytometrischen Messung von potentiellen Crf1/p41 spezifischen T-Zellklonen von Spender 2. Durch limiting dilution kultivierte Crf1/p41 und CD4 positive Klone sind grau hervorgehoben und haben einen Klonnamen erhalten. Angaben in % der Gesamt- Population. Probe 3F8 Crf1/p41 Tetramer + Crf1/p41 Tetramer - Crf1/p41 Tetramer - Crf1/p41 Tetramer + Name Klon CD4 + CD4 + CD4 - CD4 - Angaben in % Angaben in % Angaben in % Angaben in % 98,18 1,57 0,24 0 53 10C12 98,97 0,91 0 0,11 54 1F6 0,61 99,15 0,08 0,16 ~ 7E10 95,41 1,86 2,51 0,22 55 2D2 83,98 13,8 0,95 1,26 56 2G3 95,5 3,42 0,54 0,54 57 2H9 3,65 94,05 0,54 1,76 ~ 1B12 95,89 2,53 0,53 1,05 58 10F12 96,08 3,52 0,2 0,2 59 59 Ergebnisse Probe Crf1/p41 Tetramer + Crf1/p41 Tetramer - Crf1/p41 Tetramer - Crf1/p41 Tetramer + Name Klon CD4 + CD4 + CD4 - CD4 - Angaben in % Angaben in % Angaben in % Angaben in % 8C2 97,56 0,12 0,85 1,46 60 3D10 96,53 2,94 0,32 0,21 61 3G7 94,92 2,05 2,83 0,2 62 3B7 81,62 15,9 1,15 1,33 ~ 7H11 98,35 1,29 0,09 0,28 63 9H2 99,27 0,37 0,26 0,11 64 5A9 97,68 1,67 0,07 0,58 65 6D7 1,01 98,35 0,37 0,28 ~ 7F4 97,41 1,55 0,36 0,67 66 4E3 98,7 0,96 0,15 0,19 67 4C5 88,26 9,57 0,7 1,48 ~ 10C8 98,4 1,15 0,22 0,22 68 4F5 95,01 3,51 0,65 0,83 69 4B3 96,51 1,41 1,49 0,58 70 1E1 97,57 0,59 1,55 0,29 71 6E7 97,2 1,31 0,37 1,12 72 Tabelle 5: Ergebnisse der durchflusszytometrischen Messung von potentiellen Crf1/p41 spezifischen T-Zellklonen von Spender 3. Durch limiting dilution kultivierte Crf1/p41 und CD4 positive Klone sind grau hervorgehoben und haben einen Klonnamen erhalten. Angaben in % der Gesamt- Population. Probe Crf1/p41 Tetramer + Crf1/p41 Tetramer - Crf1/p41 Tetramer - Crf1/p41 Tetramer + Name Klon CD4 - CD4 + CD4 - CD4 - Angaben in % Angaben in % Angaben in % Angaben in % 3B2 99,71 0,16 0,11 0,02 73 5A9 99,36 0,44 0,16 0,04 74 3B1 0,2 1,08 98,72 0 ~ 6H12 99,58 0,31 0,08 0,02 75 2C4 99,69 0,21 0,04 0,05 76 4G2 99,85 0,11 0,01 0,02 77 4C7 0,71 99,25 0,04 0 ~ 5A7 0,19 99,7 0,1 0 ~ 5C8 99,75 0,08 0,09 0,09 78 7A1 99,1 0,74 0,16 0 79 6F8 98,48 1,36 0,11 0,04 80 6B7 99,38 0,37 0,13 0,13 81 6A9 98,42 1,48 0,07 0,02 82 2F3 99,24 0,62 0,15 0 83 8G10 99,68 0,27 0,03 0,02 84 7C12 98,68 0,88 0,18 0,26 85 6C8 0,38 0,9 98,69 0,02 ~ 1F4 99,78 0,17 0,03 0,03 86 2A9 0,31 6,86 92,53 0,31 ~ 60 Ergebnisse Probe Crf1/p41 Tetramer + Crf1/p41 Tetramer - Crf1/p41 Tetramer - Crf1/p41 Tetramer + Name Klon CD4 - CD4 + CD4 - CD4 - Angaben in % Angaben in % Angaben in % Angaben in % 7F8 98,79 0,39 0,39 0,44 87 6D12 98,94 0,97 0,08 0,01 88 11H10 99,41 0,24 0 0,35 89 8C3 98,94 0,12 0,35 0,59 90 8F3 0,01 99,88 0,06 0,06 ~ 2H1 99,29 0,47 0,24 0 91 11D12 99,35 0,13 0,13 0,39 92 12D2 98,43 0,72 0,48 0,36 93 7.1.3 Interferon-γ Produktion der Crf1/p41 spezifischen T-Zellklone TH1 Zellen, welche nach antigenspezifischer Stimulation IFN-γ produzieren, spielen eine entscheidende Rolle in der Immunantwort gegen einige pathogene Pilze inklusive A. fumigatus (Romani 2011; Wuthrich et al. 2012) (siehe 3.2). Somit stellen Crf1/p41 spezifische CD4+ T-Zellklone mit nachgewiesener IFN-γ Produktion potentielle TCRs, die für einen TCR-Transfer + spezifischen CD4 T-Zellklone geeignet mit sind. Crf1/p41 Dabei Peptid wurden beladenen die Crf1/p41 Monozyten und unbeladenen Monozyten für 24 Stunden stimuliert und anschließend der Kulturüberstand auf ihre IFN-γ Konzentration mittels ELISA untersucht. Für Spender 1 wurden 23 Klone mit einer hohen IFN-γ Produktion (über 5000 pg/ml IFN-γ), 7 Klone mit mittlerer IFN-γ Produktion (über 2500 pg/ml), 6 Klone mit geringer IFN-γ Produktion (über 800 pg/ml), 9 Klone mit sehr geringer (unter 800 pg/ml) und 7 Klone mit nahezu keiner IFN-γ Produktion (unter 300 pg/ml) ermittelt (Abbildung 16). Für alle untersuchten Crf1/p41 spezifischen Klone von Spender 2 wurden hohe IFN-γ Konzentrationen (über 5000 pg/ml) gemessen (Abbildung 17). Von Spender 3 wurden 21 Crf1/p41 spezifische Klone untersucht, dabei zeigten 5 Klone eine hohen IFN-γ Produktion (über 5000 pg/ml IFN-γ), 5 Klone eine mittlere IFN-γ Produktion (über 2500 pg/ml), 8 Klone eine geringe IFN-γ Produktion (über 800 pg/ml) und 3 Klone sehr geringe (unter 800 pg/ml über 300 pg/ml) IFN-γ Produktion (Abbildung 18). Da davon ausgegangen werden kann, dass Klone mit hoher IFN-γ Produktion zum Zeitpunkt der Messung eine hohe Effektor-Funktion zeigen und deren TCR potentiell für den Transfer geeignet ist, wurden diese für weitere Analysen ausgewählt. 61 Ergebnisse + Abbildung 16: IFN-γ Produktion Crf1/p41 spezifischer CD4 T-Zellklone von Spender 1 nach Crf1/p41 spezifischer Stimulation. 52 Crf1/p41 spezifische Klone wurden mit Crf1/p41 Peptid beladenen Monozyten (1:5) für 24 Stunden stimuliert ((A) Klon 1-26; (B) Klon 27-52). Der Kulturüberstand wurde im Anschluss mittels ELISA auf die Konzentration des Zytokins IFN-γ untersucht. Durch das Mitführen eines IFN-γ Standards wurden die Konzentrationen in pg/ml berechnet. 62 Ergebnisse + Abbildung 17: IFN-γ Produktion Crf1/p41 spezifischer CD4 T-Zellklone von Spender 2 nach Crf1/p41 spezifischer Stimulation. 20 Crf1/p41 spezifische Klone wurden mit Crf1/p41 Peptid beladenen Monozyten (1:5) für 24 Stunden stimuliert. Der Kulturüberstand wurde im Anschluss mittels ELISA auf die Konzentration des Zytokins IFN-γ untersucht. Durch das Mitführen eines IFN-γ Standards wurden die Konzentrationen in pg/ml berechnet. + Abbildung 18: IFN-γ Produktion Crf1/p41 spezifischer CD4 Z-Zellklone von Spender 3 nach Crf1/p41 spezifischer Stimulation. 21 Crf1/p41 spezifische Klone wurden mit Crf1/p41 Peptid beladenen Monozyten (1:5) für 24 Stunden stimuliert. Der Kulturüberstand wurde im Anschluss mittels ELISA auf die Konzentration des Zytokins IFN-γ untersucht. Durch das Mitführen eines IFN-γ Standards wurden die Konzentrationen in pg/ml berechnet. 63 Ergebnisse 7.2 Identifizierung und Isolierung A. fumigatus spezifischer T-Zell-Rezeptoren Die sehr geringe Vorläuferfrequenz von Crf1/p41 spezifischen T-Zellen in PBMC hat sehr wahrscheinlich zur Folge, dass bei der Crf1/p41 spezifischen Expansion, Zellen identischer Herkunft expandiert und anschließend kloniert wurden. Mit dem Ziel unterschiedliche spezifische TCRs zu identifizieren wurden die TCRs der Crf1/p41 spezifischen Klone näher untersucht. Durch die geringere Variabilität der β Ketten Familien, verglichen mit denen der α Ketten, wurden zuerst die Vβ Ketten über Durchflusszytometrie und auf Sequenzebene analysiert. Aufgrund der sehr hohen Diversität der CDR3 (siehe 3.4.2) kann bei unterschiedlichen Sequenzen in diesem Bereich der Vβ Ketten sehr wahrscheinlich davon ausgegangen werden, dass die Crf1/p41 spezifischen CD4+ T-Zellklone aus verschiedenen Vorläufern hervor gegangen sind. Zur weiteren Charakterisierung der TCRs wurden die Vα Ketten, über Sequenzanalysen, ermittelt. Die identifizierten Crf1/p41 spezifischen TCRs wurden isoliert und in einen retroviralen Vektor kloniert, für eine Transduktion auf humane T-Zellen. 7.2.1 Identifizierung der T-Zell-Rezeptor β Ketten mit dem IO Beta Mark Kit (Beckman Coulter) Für eine erste Analyse der TCRs der Crf1/p41 spezifischen T-Zellklone, wurden die Klone mit dem IO Beta Mark Kit (Beckman Coulter) durchflusszytometrisch auf ihre variable β (Vβ) Kette untersucht (Abbildung 19). Die Klone wurden nach Protokoll des Herstellers gefärbt und die durchflusszytometrischen Messungen ergaben die in Tabelle 6 dargestellten Vβ Ketten. Des Weiteren wurden „negative“ Klone, mit doppelten Populationen bzw. mehrfach positiven Vβ Ketten Familien identifiziert und aussortiert. Bei Spender 1 konnte die Vβ Kette 18 bei allen Crf1/p41 spezifischen Klonen festgestellt werden (Tabelle 6 A). Für die Klone von Spender 2 wurden die Vβ Ketten Familien 18 und 1 ermittelt (Tabelle 6 B). Für Crf1/p41 spezifische Klone von Spender 3 wurden die Vβ Ketten 18, 1 und 13.6 identifiziert (Tabelle 6 C). Zusammenfassend wurde festgestellt, dass die Vβ Ketten Familie 18 bei allen drei Spendern mehrfach vertreten ist und Vβ 1 bei zwei von drei Spendern auftritt. Die Vβ Kette von Klon 69 (Spender 2), sowie die Vβ Kette der Klone 74, 75, 83, 85, 86, 87, 92 und 92 von Spender 3 konnte nicht bestimmt werden. Das Kit deckt ca. 70 % des humanen Vβ- Repertoire ab, somit sollten die nicht identifizierten Klone eine der verbliebenen Vβ Familien exprimieren. 64 Ergebnisse + Abbildung 19: Durchflusszytometrische Messung Crf1/p41 spezifischer CD4 T-Zellklone + gefärbt mit dem IO Beta Mark Kit (Beckman Coulter). Crf1/p41 spezifische CD4 T-Zellklone wurden nach Angaben des Herstellers gefärbt und auf ihre Vβ Ketten Familie untersucht. Repräsentativ sind ein Vβ 18 Klon (Spender 1/ Klon 2), ein Vβ 1 Klon (Spender 3/ Klon 73) und ein „negativer“ Klon (Spender 2/ Klon 58) dargestellt. Der Klon 58 wurde als negativ eingestuft, aufgrund der doppelten Population bei der durchflusszytometrischen Messung (eine Population Vβ 18 und eine nicht feststellbare Vβ Familie). Tabelle 6: Vβ Ketten Identifizierung Crf1/p41 spezifischer T-Zellklone. Durchflusszytometrische Identifizierung der Vβ Kette des TCR von Crf1/p41 spezifischen Klonen von Spender 1 (A), Spender 2 (B) und Spender 3 (C) durch den IO Beta Mark Kit (Beckman Coulter). *Nomenklatur nach Arden et al. 1995. 65 Ergebnisse 7.2.2 Identifizierung der T-Zell-Rezeptor β Ketten durch SMARTer RACE PCR Da es das Ziel ist unterschiedliche TCRs mit hoher Spezifität zu erhalten, wurden die Sequenzen der Vβ Ketten der Crf1/p41 spezifischen CD4+ T-Zellklone mit der SMARTer RACE PCR bestimmt. Aufgrund der sehr hohen Diversität der CDR3 kann bei unterschiedlichen Sequenzen in diesem Bereich der Vβ Ketten sehr wahrscheinlich davon ausgegangen werden, dass die Crf1/p41 spezifischen CD4+ T-Zellklone aus verschiedenen Vorläufern hervor gegangen sind. Des Weiteren wurden die Vβ Ketten die mit dem IO Beta Mark Kit nicht bestimmt werden konnte, identifiziert. Die Identifizierung unterschiedlicher TCRs erfolgt über die Aminosäuresequenz der Vβ Ketten der Crf1/p41 spezifischen T-Zellklone. Über die sogenannte SMARTer RACE cDNA Amplifikation (Clontech) ist es möglich die komplette cDNA beginnend am 5´ Ende der zu identifizierenden Sequenz aus totaler RNA zu isolieren. Mit dieser Methode wurde die vollständige Vβ Kette amplifiziert und mittels Agarosegel deren korrekte Länge kontrolliert. Die Banden der Vβ Ketten liegen bei ca. 650 bp (Abbildung 20). Diese wurden ausgeschnitten und die DNA wurde über den NucleoSpin® Gel Clean-up Kit (Macherey Nagel) aufgereinigt. Abbildung 20: 5´ RACE PCR der Vβ Ketten. (A) 5´ RACE PCR Schema der Vβ Kette der Crf1/p41 spezifischen Klone. (B) Das Agarosegel zeigt die 5´ RACE PCR Amplifikation der Vβ Kette von Klon 78 (Spender 3) und einen 1kb DNA-Marker zur Bestimmung der Länge der amplifizierten DNA. 66 Ergebnisse Abbildung 21: Sequenzierungsergebnis und Identifizierung der Vβ Kette eines TCR. (A) Sequenz der von GATC-Biotech durchgeführten Sequenzierung. (B) Identifizierung der Sequenz mit Hilfe der IMGT Datenbank. Die Vβ Sequenz von Klon 77 von Spender 3 ist hier repräsentativ dargestellt. Mit 100%iger Übereinstimmung handelt es sich um die Vβ-Kette TRBV9*01; dies entspricht Vβ 1 nach Arden et. Al (1995). 67 Ergebnisse Die anschließende Sequenzierungen der gereinigten DNA wurde von GATC-Biotech durchgeführt und die Analyse erfolgte mit dem Programm GENtle und der Datenbank Immunogenetics (IMGT) (Abbildung 21). Die Analyse der Sequenzierungen ergab die in Tabelle 7 dargestellten Vβ Ketten der Crf1/p41 spezifischen CD4+ T-Zellklone mit ihrer jeweiligen Aminosäuresequenz der CDR3. Die Nomenklatur erfolgt nach Arden et al. 1995. Aus der Sequenzierung wurde ersichtlich, dass von 11 untersuchten Klonen (Spender 1), drei unterschiedliche Vβ 18 Ketten vertreten sind. Von Spender 2 wurden 12 Klone untersucht, welche drei verschiedene Vβ Ketten (Vβ 1, Vβ 6 und Vβ 18) lieferten. Für Spender 3 wurden die Vβ Ketten 1, 6, 13.6 und 18 aus 14 Klonen ermittelt. Die Ergebnisse der Durchflusszytometrischen Untersuchungen konnten somit bestätigt werden und für die verbliebenen Klone wurde die Vβ Ketten Familie 6 ermittelt. Durch die Identifizierung der Vβ Ketten konnten 12 TCRs mit unterschiedlichen Vβ Ketten und somit abstammend von unterschiedlichen Vorläuferzellen, von drei Spendern identifiziert werden. Tabelle 7: Aminosäuresequenz der CDR3 der Vβ Ketten der Crf1/p41 spezifischer + CD4 T-Zellklone. Nomenklatur der Vβ Ketten nach Arden et al. 1995. Vβ 18 1,2 bzw. 3 stehen für unterschiedliche Vβ 18 Ketten. Spender CDR3 Region der Vβ Kette Vβ Klon 1 ASSP LSGSDTQYFGPGTRLTV 181 1, 2, 5, 6, 9, 40, 48 1 ASSP LNGGDEQYFGPGTRLTV 182 41, 49 1 ASSR LSGSDIQYFGAGTRLSV 183 8, 23 2 ASSV GGSDTEAFFGQGTRLTV 1 60, 64 2 ASSR LSGSDIQYFGAGTRLSV 18 53, 54, 55, 59, 61, 67, 70, 71, 72 2 ASSS SPYEQYVGPGTRLTV 6 69 3 ASSR TSGQETQYFGPGTRLLV 18 80 3 ASSP TNGDTQYFGPGTRLTV 18 78 3 ASSV GGSDTEAFFGQGTRLTV 1 73, 77 3 ASSG TSGDTQYFGPGTRLTV 6 74, 75, 83, 85, 86, 87, 92, 93 3 ASRG TGSLSADTQYFGPGTRLTV 13.6 88 3 ASSG TSCGPQYSGPEARRSGC 18 79 68 Ergebnisse 7.2.3 Identifizierung der T-Zell-Rezeptor α Ketten Zur weiteren Charakterisierung der Crf1/p41 spezifischen TCRs, wurden von den Crf1/p41 spezifischen CD4+ T-Zellklonen mit unterschiedlichen Vβ Ketten und mit einer nachgewiesenen IFN-γ Produktion die variable α (Vα) Ketten ermittelt. Durch das Auftreten von Vβ 18 bei allen drei Spendern, wurde jeweils ein Klon pro Spender mit nachgewiesener IFN-γ Produktion für einen Vergleich gewählt (Klon 48 von Spender 1; Klon 71 von Spender 2; Klon 78 von Spender 3), um mögliche von Spendern abhängige Unterschiede aufzudecken. Da die Vβ Kette 1 von Klonen in zwei von drei Spendern vorkommt, wurde diese für einen gegenüberstellenden Vergleich bezüglich Vβ 18 hinzugezogen (Klon 77 von Spender 3). Die Identifizierung der Vα Ketten erfolgte über die vorher beschriebenen RACE PCR und anschließender Sequenzierung. Die vollständige Vα Kette wurde amplifiziert und mittels Agarosegel auf ihre Länge kontrolliert. Dabei liegt die korrekte Bande der Vα Kette bei ca. 600 bp (Abbildung 22). Abbildung 22: 5´RACE-PCR der Vα Ketten. (A) 5´RACE PCR Schema der Vα Kette der Crf1/p41 spezifischen Klone. (B) Das Agarosegel zeigt die 5´RACE PCR Amplifikation der Vα Kette von Klon 78 (Spender 3) und einen 1kb DNA-Marker zur Bestimmung der Länge der amplifizierten DNA. Die aus dem Agarosegel extrahierte und aufgereinigte DNA wurde von GATC-Biotech sequenziert und die Ergebnisse mittels der Software GENtle und der Datenbank Immunogenetics analysiert. Aus diesen ergaben sich die in Tabelle 8 dargestellten 69 Ergebnisse Vα Ketten und die entsprechenden Aminosäuresequenzen der CDR3 für die + ausgewählten Crf1/p41 spezifischen CD4 T-Zellklone. Für die unterschiedlichen Vβ 18, Klone 48 (Spender 1) und 71 (Spender 2), ergab sich für beide die Vα Kette 3, welche sich in der Aminosäuresequenz der CDR3 unterscheiden. Der Klon 77 (Spender 3) mit Vβ 1 hat die Vα Kette 15, während der Klon 78 (Spender 3) die Vα Kette 26 exprimiert. Nach der Identifikation der Crf1/p41 spezifischen TCRs wurden diese isoliert und in einen retroviralen Vektor kloniert, um retroviralen Kulturüberstand für einen Transfer zu generieren. Tabelle 8: Aminosäuresequenz der CDR3 der Vα Ketten der Vα Vβ Crf1/p41 + spezifische CD4 T-Zellklone. Nomenklatur der Vα/Vβ Ketten nach Arden et.al 1995. 1 Vβ 18 steht für eine bestimmte Vβ 18 Kette von Spender 1. Spender 7.2.4 CDR3 Region der variablen α Kette 1 Klon 1 SYFC ATDRIIQGAQKLVCATDRIIQGAQKLVF CATD 3 18 48 2 SYVC ATDAIVQGAQKLVCATDAIVQGAQKLVF CATD 3 18 71 3 SAIYFC AESPGGSYIPTCAESPGGSYIPTF CAE 15 1 77 3 IYLC GARPIQGAQKLV CGARPIQGAQKLVF CGA 26 18 78 Isolierung der T-Zell-Rezeptor α und β Ketten und Klonierung in den retroviralen Vektor pMP71 Für die Durchführung eines Transfers von Crf1/p41 spezifischen TCRs auf eine Empfänger-T-Zelle und für die vergleichende Analyse der ausgewählten TCRs ist es nötig, diese zu isolieren und in den retroviralen Vektor pMP71 zu klonieren (siehe 5.5.1). Die retroviralen pMP71 Vektoren, ausgestattet mit TCR- DNA wurden für die Produktion von retroviralem Kulturüberstand verwendet. Über den retroviralen Kulturüberstand wurde die TCR-DNA in die Empfänger-T-Zellen transferiert. Die identifizierten Aminosäuresequenzen der TCRs wurden mit Hilfe spezifischer Primer (Tabelle 1) aus der cDNA des entsprechenden Klons isoliert. Die anschließende Klonierung in den retroviralen Vektor MP71 erfolgte über die Restriktionsschnittstellen Not1 und EcoR1. Ebenfalls wurde ein Kontrollvektor, ausgestattet mit eGFP, kloniert, um die erfolgreiche Transduktion über das Mikroskop zu verfolgen (Abbildung 23). 70 Ergebnisse Abbildung 23: Klonierung von pMP71 Vektoren für einen TCR Transfer. Isolierung von TCR α und TCR β Ketten aus der cDNA Crf1/p41 spezifischer T-Zellklone über RACE PCR, Aufreinigung der korrekten Banden (TCR α ~ 850bp; TCR β ~ 950bp) aus Agarosegel (repräsentativ von Klon 48; Spender 1; TCR 4) (A) und Klonierung in pMP71 Vektoren (B). Die Vektoren sind mit einer DNA-Wiederholungseinheit (long terminal repeat) (MPSV-LTR) und einem des „myeloproliferative sarcoma virus“ „Woodchuck-Hepatitis-Virus“-Posttranskriptionalem regulatorischem Element (PRE) ausgestattet und besitzen eine Ampillicinresistenz (AmpR). In den Kontrollvektor wurde eGFP kloniert (C). Es konnten die identifizierten TCRs aus der cDNA entsprechender Klone isoliert werden und die erfolgreiche Klonierung wurde durch Sequenzierung bestätigt. In Tabelle 9 wurden Informationen über die generierten Vektoren zusammengefasst. 71 Ergebnisse Tabelle 9: Zusammenfassung der isolierten Crf1/p41 spezifischen TCR Ketten in ihren jeweiligen pMP71 Vektoren. Dargestellt sind die Vα und Vβ Ketten und die Crf1/p41 spezifische + CD4 T-Zellklone aus denen sie isoliert wurden, sowie der Name des Vektors in den sie kloniert wurden. Nomenklatur der Vα/Vβ Ketten nach Arden et.al 1995. 7.3 Vektor Klon Vα MP71-TCR1α 78 26 MP71-TCR1β 78 MP71-TCR2α 71 MP71-TCR2β 71 MP71-TCR3α 77 15 MP71-TCR3β 77 MP71-TCR4α 48 MP71-TCR4β 48 Vβ Spender 3 18 3 3 - 2 - 18 2 3 1 3 3 - 1 - 18 1 Transfer von A. fumigatus spezifischen T-Zell-Rezeptoren auf Jurkat 76 Zellen Um zu ermitteln, ob die Crf1/p41 spezifischen TCRs für einen Transfer auf andere T-Zellen geeignet sind, wurden sie zunächst auf Jurkat 76 Zellen transduziert. Die Zelllinie Jurkat 76 ist für die erste Überprüfung der Expression und Funktionalität der TCRs besonders geeignet, da es sich um eine TCR und CD3 negative humane T-Zell Lymphom Jurkat Zelllinie handelt. Der induzierte TCR muss nicht mit endogenen TCRs konkurrieren. Ebenso kann eine Expression mit Hilfe von anti-CD3 Antikörper durchflusszytometrisch verfolgt werden, da CD3 nur bei erfolgreicher Transduktion exprimiert wird. Die Transduktion Crf1/p41 spezifischer TCRs erfolgte über retroviralen Kulturüberstand, generiert durch Platinum A transfiziert mit der jeweiligen Plasmid-DNA der TCR α Kette und TCR β Kette, und mit RetroNectin®-beschichteten Zellkulturplatten. Die Spezifität der transduzierten Jurkat 76 Zellen wurde mit Hilfe des Crf1/p41 Tetramer und anti-CD3 Antikörper durchflusszytometrisch ermittelt (Abbildung 24). 72 Ergebnisse Abbildung 24: Expression Crf1/p41 spezifischer TCRs transduziert auf Jurkat 76 Zellen. Jurkat 76 transduziert mit pMP71 eGFP Vektor (A), ohne Vektor (Negativ; (B)) und jeweils transduziert mit TCR 1, TCR 2, TCR 3 und TCR 4 (C). Durchflusszytometrische Messung an Tag 3 nach Transduktion mit Crf1/p41-Tetramer und anti-CD3 Ab. Eine Messung ist hier repräsentativ für mehrere durchgeführte Transduktionen dargestellt. Der erfolgreiche Transfer auf die Zelllinie Jurkat 76, von TCR 1, TCR 3 und TCR 4 konnte mittels Crf1/p41 Tetramer und anti-CD3 Antikörper nachgewiesen werden. TCR 2 zeigte eine positive CD3 Färbung, jedoch wurde kein Crf1/p41 Tetramer gebunden. Der TCR 1 wurde auf 2,78 % der transduzierten Zellen nachgewiesen. Mit TCR 2 transduzierte Zellen zeigten 6,70% CD3 positive Zellen, allerdings keine Bindung des Crf1/p41 Tetramers. 1,46 % von, TCR 3 transduzierten Zellen, und 2,77 % der TCR 4 transduzierten Zellen waren CD3 positiv und haben Crf1/p41 Tetramer gebunden. Die Expression von pMP71 eGFP transduzierten Zellen lag bei 50,6 % eGFP positiver Zellen, was die erfolgreiche Transduktion bestätigt. 7.4 Transfer eines Codon- und Vektor- optimierten T-Zell Rezeptor auf Jurkat 76 Aufgrund der geringen Frequenzen der transduzierten Zellen wurde eine Codonoptimierung der TCR Sequenz durchgeführt, um die Translation des Transgenes zu erhöhen (Scholten et al. 2006). Die Codonoptimierung, durchgeführt von GeneArt® (Life Technologies), erhöht die Proteinmenge, aber begünstigt nicht den korrekten 73 Ergebnisse Zusammenbau des transferierten TCR. Um die korrekte Paarung des TCR zu erhöhen wurden die α und β Kette in einen Vektor kloniert und mittels eines p2A-Elements voneinander getrennt. Für eine höhere funktionale Avidität bezüglich des modifizierten TCR wurden die Cα und Cβ Kette durch murine Ketten ersetzt. Zur weiteren Stabilisierung des TCRs wurde eine weitere Disulfidbrücke eingebaut, welche die C- Regionen von TCR α und TCR β verbinden. Abbildung 25: Optimierter Vektor pMP71. Das von GeneArt (Life Technologies) optimierte TCR α - p2A - TCR β Element wurden über die Restriktionsschnittstellen Not1 und EcoR1 in den Vektor pMP71 kloniert. Der Vektor ist mit DNA-Wiederholungseinheiten (long terminal repeat) des „myeloproliferative sarcoma virus“ (MPSV-LTR), einem „Woodchuck-Hepatitis-Virus“-Posttranskriptionalem regulatorischem Element (PRE) und einer Ampicillinresistenz (AmpR) ausgestattet. Für die Optimierung wurde TCR 1 ausgewählt, da er bei der Transduktion auf Jurkat 76 eine hohe Transduktionseffizienz zeigte. Die optimierte Sequenz wurde mittels Not1 und EcoR1 Restriktionsschnittstelle in den Vektor pMP71 kloniert (Abbildung 25). Der Vektor MP71 TCR 1 optimiert wurde für die Herstellung von retroviralem Kulturüberstand eingesetzt. Die Transduktionseffizienz von TCR 1 und optimiertem TCR 1 auf Jurkat 76 wurde vergleichend analysiert. Mittels Durchflusszytometrie wurde drei Tage nach Transduktion eine Erhöhung der Expression um das 5fache bei optimiertem TCR 1 festgestellt (Abbildung 26). Die Transduktionseffizienz von eGFP lag bei 24,3 % transduzierter Zellen. 74 Ergebnisse Abbildung 26: Expression von Crf1/p41 spezifischem TCR 1 und optimiertem TCR 1 auf Jurkat 76. Durchflusszytometrische Messung von Jurkat 76 transduziert mit pMP71 eGFP Vektor (A), ohne Vektor (Negativ) und jeweils transduziert mit TCR 1 und TCR 1 optimiert (B). Die Messung erfolgte 3 Tage nach Transduktion mittels Crf1/p41 Tetramer und anti-CD3 Antikörper. Eine Messung ist hier repräsentativ für mehrere durchgeführte Transduktionen dargestellt. Wiederholte Transduktion auf Jurkat 76 zeigte eine durchschnittliche Transduktionseffizienz für TCR 1 von 2,5 % (n=2), TCR 1 optimiert mit 9,7 % (n=3), TCR 3 mit 1 % (n=2) und von TCR 4 betrug 3,5 % (n=2). TCR 2 wurde auch bei wiederholter Transduktion (n=2) nicht exprimiert (Abbildung 27). 75 Ergebnisse Abbildung 27: Expression Crf1/p41 spezifischer TCR nach Transduktion auf Jurkat 76. Die Crf1/p41 spezifischen TCR 1 (n=2), TCR 1 optimiert (n=3), TCR 2 (n=2), TCR 3 (n=2) und TCR 4 (n=2) wurden auf Jurkat 76 transduziert und anschließend durchflusszytometrisch die Expression mittels Crf1/p41 Tetramer und anti-CD3 Antikörper ermittelt. 7.5 Transfer von A. fumigatus spezifischen T-Zell-Rezeptoren auf humane CD4+ T-Zellen Für den Einsatz in einer möglichen adoptiven Therapie müssen die Crf1/p41 spezifischen TCRs auf humane T-Zellen unterschiedlicher Empfänger transferiert werden können. Darum wurden Spender mit unterschiedlichen HLA DRB1* Allel gewählt, um festzustellen, ob die TCRs von Spendern mit HLA DRB1*04 Allel auch auf Spender mit einem anderen HLA DRB1* Allel transferiert und zur Expression gebracht werden können. Für den Transfer A. fumigatus spezifischer TCRs in humane CD4+ T-Zellen wurden TCR 1, TCR 1 optimiert, TCR 2, TCR 3 und TCR 4 über retroviralen Kulturüberstand und mit RetroNectin®-beschichtete Zellkulturplatten in die Spender 4, 5 und 6 transferiert. Es wurden CD4+ T-Zellen transferiert, da in vorherigen Untersuchungen gezeigt wurde, dass Crf1/p41 eine CD4+ TH1 Immunantwort bei Aspergillus- Infektionen in Menschen und in Mäusen auslöst (Bozza et al. 2009; Stuehler et al. 2011). Zwei Tage vor Transduktion wurden CD4+ T-Zellen aus PBMC der drei Spender isoliert und mit CD3/CD28 Dynabeads® stimuliert. Die durchflusszytometrische Auswertung erfolgte 3 Tage nach Transduktion mittels Crf1/p41 Tetramer. Abbildung 28 zeigt die durchflusszytometrische Messung von Spender 4 repräsentativ für alle Spender. Spender 4 zeigt eine deutliche 76 Ergebnisse Expression der Rezeptoren TCR 1 mit 3,16 % und TCR 1 optimiert mit 10,8 %, die Rezeptoren TCR 3 (~1 %) und TCR 4 (~1 %) zeigen nur eine sehr schwache Expression. TCR 2 konnte, wie auch schon auf den Jurkat 76, nicht zur Expression gebracht werden. Die Expression von eGFP durch den Kontrollvektor lag bei 21,9 %. Zusammenfassend für alle drei Spender hat sich gezeigt, dass sich der TCR 1 und besonders der optimierte TCR 1 am besten transduzieren lassen (Abbildung 29). + Abbildung 28: Expression Crf1/p41 spezifischer TCRs nach Transduktion auf CD4 T-Zellen. + CD4 T-Zellen von Spender 4 wurden mit pMP71 eGFP transduziert (eGFP Kontrolle), ohne Vektor (Negativ) und jeweils mit TCR 1, TCR 1 optimiert, TCR 2, TCR 3, TCR 4. Die durchflusszytometrische Messung wurde durchgeführt an Tag 3 nach Transduktion mit Crf1/p41 Tetramer und anti-CD4 Antikörper. 77 Ergebnisse + Abbildung 29: Expression Crf1/p41 spezifischer TCR nach Transduktion auf CD4 T-Zellen. Die prozentuale Expression von TCR 1 (n=2), TCR 1 optimiert (n=3), TCR 2 (n=1), TCR 3 (n=2) + und TCR 4 (n=2) nach Transduktion auf CD4 T-Zellen von 3 verschiedenen Spendern (Spender 4, 5 und 6) wurde ermittelt mit Hilfe des Crf1/p41Tetramer und anti-CD4 Antikörper über Durchflusszytometrie. 7.5.1 Isolation und Expansion CD4+ T-Zellen mit optimiertem TCR 1 Da sich durch die vorangegangenen Versuche gezeigt hat, dass der optimierte TCR 1 im Vergleich die höchste Transduktionseffizienz hat, wurde dieser für weitere Analysen ausgewählt. Aufgrund der geringen Zellmengen der transduzierten Zellen ist für die Durchführung weiterer funktioneller Tests eine Anreicherung der TCR 1 optimierten spezifischen Zellen von Nöten. Mit Hilfe des Crf1/p41 Tetramer und magnetischer Separation können die spezifischen Zellen isoliert werden. Die isolierten Zellen wurden nach Expansionsprotokoll für 14 Tage expandiert. Mittels durchflusszytometrischer Messung mit Crf1/p41 Tetramer vor Isolation und nach Expansion wurden die Zellen auf ihre Spezifität überprüft. Für Spender 4 hat sich mit Hilfe der Isolation und Expansion eine Anreicherung auf 79,45 % Crf1/p41 spezifische Zellen ergeben (Abbildung 30). Die Crf1/p41 spezifischen Zellen von Spendern 6 wurden auf 26 %, die Zellen von Spender 7 auf 31 % und die Zellen von Spender 8 auf 41% spezifische Zellen angereichert. Die angereicherten, spezifischen Zellen wurden in Proliferations- und Zytokinproduktion- Analysen eingesetzt. 78 Ergebnisse + Abbildung 30: Expression Crf1/p41 spezifischer TCR 1 optimiert transduzierter CD4 T-Zellen nach Expansion. Durchflusszytometrische Messung von Crf1/p41 spezifischen und + TCR 1 optimiert transduzierten CD4 T-Zellen von Spender 4 vor Isolation und nach Isolation mit 14-tägiger Expansion. Die Expression wurden gemessen mit Crf1/p41 Tetramer und anti-CD4 Antikörper. 7.6 Proliferation und Zytokinproduktionsanalyse TCR 1 optimiert transduzierter T-Zellen nach Crf1/p41 spezifischer Stimulation Um zu ermitteln, ob die angereicherten TCR 1 optimiert transduzierten CD4+ T-Zellen die Fähigkeit besitzen eine Immunantwort zu induzieren, wurden sie bezüglich ihrer Proliferationsfähigkeit und der IFN-γ Produktion nach Crf1/p41 spezifischer Stimulation untersucht. Es wurden transduzierte Zellen von drei verschiedene Spendern (Spender 4, 6, und 8) untersucht, die negativ für das HLA-DRB1*04 Allel waren und von einem Spender 7 der das HLA-DRB1*04 Allel exprimiert. Für die Ermittlung der Proliferationsfähigkeit von TCR 1 optimierten transduzierten CD4+ T-Zellen wurden diese mit dem Fluoreszenzfarbstoff eFluor®670 gefärbt und mit allogenen bzw. autologen mDC (beladen mit Crf1/p41 Peptid) stimuliert (Abbildung 31). Als Positivkontrolle wurde bei Spender 4 ein bakterielles Superantigen (SEB) eingesetzt bei den anderen Spendern wurden CD3/CD28 Dynabeads® verwendet. Es zeigte sich eine positive Proliferation bei der Stimulation mit SEB von 22,5 % (Spender 4), und die Stimulationen mit CD3/CD28 ergaben positive Proliferationen von 44,8 % (Spender 6), 98,4 % für Spender 7 und 86 % (Spender 8). Keine Proliferation konnte bei der Stimulation mit allogenen mDC von Spendern mit einem HLA-DRB1*13 Allel (gezeigt in Abbildung 31) und HLA-DRB1*06, 15 Allel ermittelt werden. Bei der Stimulation mit allogenen bzw. autologen Crf1/p41 beladenen mDC von Spendern mit HLA-DRB1*04 Allel (Spender 9 und Spender 7) wurde die Proliferation der TCR 1 optimiert transduzierten CD4+ T-Zellen nachgewiesen. Nach Abzug der Negativkontrolle (Stimulation mit unbeladenen mDC) zeigten die transduzierten Zellen stimuliert mit allogenen Crf1/p41 beladenen mDC (Spender 9) von Spender 4 eine 79 Ergebnisse positive Proliferation von 72 %, Spender 6 von 40 %, Spender 7 von 87 % und Spender 8 von 67 % der Crf1/p41 spezifischen CD4+ T-Zellpopulation (Abbildung 31). Es wurde gezeigt, dass die TCR 1 optimiert transduzierten CD4+ T-Zellen von allen Spendern, nach Crf1/p41 spezifischer Stimulation die Fähigkeit zur Proliferation besitzen. 80 Ergebnisse Abbildung 31: transduzierter Proliferation + CD4 T-Zellen. nach Die Crf1/p41 mit spezifischer Proliferationsfarbstoff Stimulation gefärbten TCR 1 optimiert TCR 1 optimiert transduzierten Zellen von Spender 4, 6 7 und 8 wurden mit Crf1/p41 beladenen allogenen mDC (HLA-DRB1*13; Spender 5) und mDC (HLA-DRB1*04; Spender 7) stimuliert. Nach 4 Tagen wurden die Zellen mit Crf1/p41 Tetramer und anti-CD4 Antikörper gefärbt und die Proliferation von Crf1/p41 + spezifischer CD4 T-Zellen wurde durchflusszytometrisch ermittelt. Negativkontrolle = unbeladene mDC (HLA DRB1*04, Spender 7); Positivkontrolle = Superantigen (SEB) bei Spender 4 und CD3/CD28 Dynabead® Stimulation bei Spender 6, 7, und 8. 81 Ergebnisse Um die Funktionalität der transduzierten Zellen weiter zu präzisieren wurde die IFN-γ Produktion nach Crf1/p41 Stimulation (24 Stunden) bestimmt. Für die + Zytokin-Konzentration von IFN-γ (Funktion: Differenzierung von CD4 T-Zellen zu TH1) wurden die TCR 1 optimiert transduzierten Zellen mit Crf1/p41 beladenen allogenen bzw. autologen mDC für 24 Stunden kultiviert und im Anschluss die IFN-γ Produktion gemessen. In Abbildung 32 sind die Ergebnisse der durchflusszytometrischen Analyse für die Spender 4, 6, 7 und 8 zusammengefasst. Die Zellen von allen Spendern wurden mit Crf1/p41 beladenen autologen bzw. allogenen mDC von Spendern mit DRB1*04 negativem Allel (DRB1*06, 15, Spender 4; DRB1*13, Spender 5)) und allogenen mDC von Spendern, mit DRB1*04 positivem Allel (DRB1*04; Spender 9) stimuliert. Die Zytokinanalyse hat gezeigt, dass lediglich Crf1/p41 beladene mDC von Spendern mit einem HLA DRB1*04 Allel in der Lage waren eine IFN-γ Produktion zu induzieren. Für Spender 4 wurde ein IFN-γ Produktion von 3400 pg/ml, für Spender 6 wurden 640 pg/ml, für Spender 7 wurden 1720 pg/ml und für Spender 8 wurden 1070 pg/ml IFN-γ gemessen. Mit den funktionellen Analysen der antigenspezifischen Proliferation und Zytokinproduktion wurde bestätigt, dass Zellen, transduziert mit TCR1 optimiert Crf1/p41 spezifisch stimuliert werden und die Ausschüttung von Zytokinen induzieren können und es sich um einen funktionellen TCR handelt. Abbildung 32: transduzierten Zytokinproduktion + CD4 T-Zellen. nach Crf1/p41 TCR 1 optimiert Stimulation transduzierte von + TCR 1 optimiert CD4 T-Zellen von vier unterschiedlichen Spendern wurden mit Crf1/p41 Peptid beladenen allogenen und autologen mDC von Spendern mit HLA-DRB1*04 negativen und positiven Allelen für 24 Stunden stimuliert. Mittels CBA wurden die Konzentrationen von IFN-γ im Kulturüberstand in pg/ml ermittelt. Für die Auswertung wurden der mitgeführte Standard und die Software FCAP Array 2.0 verwendet. 82 Diskussion 8 Diskussion Der Pilz A. fumigatus gehört zu den humanpathogenen Pilzen und kann in immunsupprimierten Patienten zum Teil schwere invasive Infektionen auslösen. Neben fortschreitenden Verbesserungen in der Behandlung und medikamentöser Prophylaxe bleibt die Sterblichkeitsrate bei invasiven Erkrankungen, wie zum Beispiel bei der invasiven Aspergillose trotzdem hoch (Baddley et al. 2013). Darum wären immuntherapeutische Strategien wie der adoptive Transfer Antigen spezifischer T-Zellen ein wichtiges Mittel, um das Immunsystem der Patienten zu unterstützen und Infektionen zu verringern oder sogar zu eliminieren (Perruccio et al. 2005; Morris et al. 2006). Ein Ansatz für die Entwicklung solcher Strategien ist die Identifizierung A. fumigatus spezifischer TCRs, für einen möglichen Transfer dieser Rezeptoren auf andere Zellen, welche wiederum in einer adoptiven Therapie eingesetzt werden könnten. Dieses Projekt konzentriert sich auf die Generation von Vektoren die A. fumigatus spezifischen TCR exprimieren, um diese für die genetische Modifikation von T-Zellen zu verwenden. Das Peptid Crf1/p41 wurde aufgrund von vorherigen Studien gewählt, da es bei gesunden Spendern eine funktionelle TH1 Immunantwort gegen A. fumigatus induziert und zur Produktion hoher Mengen von IFN-γ führt (Khanna et al. 2011; Stuehler et al. 2011; Jolink et al. 2013). Des Weiteren zeichnet sich das Crf1/p41 Epitop dadurch aus, dass es von den MHC Klasse II Allelen HLA-DRB1*03, HLA-DRB1*04 und HLA-DRB1*13 (Stuehler et al. 2011) präsentiert werden kann, welche insgesamt von ca. 60 % der kaukasischen Bevölkerung exprimiert werden (Schipper et al. 1996; Gonzalez-Galarza et al. 2011). Um eine hohe TCR Expression zu erhalten wurden einige Modifikationen des TCR und Expressionsvektors durchgeführt. Die in dieser Arbeit ermittelten Ergebnisse zeigen, dass TCR transduzierte CD4+ T-Zellen nach antigenspezifischer Stimulation erfolgreich aktiviert werden können. Dies legt den Schluss nahe, dass die TCR transduzierten CD4+ T-Zellen das Potential haben in einer Immuntherapie gegen A. fumigatus Infektionen eingesetzt zu werden. 8.1 Die T-Zellantwort von Crf1/p41 ist polyklonal Um Crf1/p41 spezifische TCRs zu identifizieren wurden Crf1/p41 spezifische T-Zellklone in vitro generiert. Dafür wurden Zellen von drei gesunden Spendern mit dem HLADRB1*04 Allel verwendet. Die TH1 Effektor-Funktion, der Crf1/p41 spezifischen CD4+ T-Zellklone, wurde durch die Bestimmung der IFN-γ Produktion nach Crf1/p41 spezifischer Stimulation bestätigt. Das TH1 Zytokin IFN-γ wurde gewählt, da gezeigt wurde, dass gesunde Spender funktionelle TH1 Immunantworten gegen A. fumigatus 83 Diskussion induzieren und Crf1 die Ausschüttung hoher Mengen von IFN-γ initiiert (Stuehler et al. 2011). Es wurden siebenunddreißig Crf1/p41 spezifische CD4+ T-Zellklone generiert. Aufgrund der geringen Vorläuferfrequenz von Crf1/p41 spezifischen Zellen in PBMCs wurden, während der Crf1/p41 spezifischen Expansion, Zellen identischer Herkunft expandiert und anschließend kloniert. Durch Unterschiede in der Sequenz, der sehr variablen CDR3 (im Bereich der Vβ Ketten) konnten 12 unterschiedlichen TCRs (von 3 unterschiedlichen Spendern) aus unterschiedlichen Crf1/p41 spezifischen + CD4 T-Zellklonen ermittelt werden. Dabei war festzustellen, dass vor allem die Vβ Kette 18 sehr dominant vertreten war (bei allen drei Spendern), ebenso die Vβ Ketten 1 und 6, welche bei Klonen von zwei Spendern nachgewiesen wurden. Zur weiteren Charakterisierung der Crf1/p41 spezifischen TCRs wurde die Vα Kette von jeweils einem Vβ 18 Klon (mit nachgewiesener IFN-γ Produktion) von jedem Spender identifiziert. Für einen gegenüberstellenden Vergleich zu Vβ 18 wurde die Vβ Kette 1 ausgewählt, da Klone von zwei Spendern diese exprimieren. Durch diese Auswahl können mögliche spenderspezifische und kettenspezifische Unterschiede ermittelt werden. Es wurden die Vα Ketten 3, Vα 15 und Vα 26 bestimmt. Somit liegt hier eine polyklonale T-Zellantwort vor. 8.2 Der endogene TCR verringert die Oberflächenexpression von transduzierten Crf1/p41 spezifischen T-Zell-Rezeptoren auf T-Zellen Ein effektiver TCR für eine mögliche Therapie sollte aufgrund seiner Antigenerkennung und nach seiner Oberflächenexpression ausgewählt werden. Für die Ermittlung der Expressionsfähigkeit, der Crf1/p41 spezifischen TCRs auf der Oberfläche von transduzierten Zellen, müssen diese zunächst in einen retroviralen Vektor kloniert werden. Dafür wurden die Crf1/p41 spezifischen TCRs aus der cDNA Crf1/p41 spezifischer CD4+ T-Zellklone isoliert und in den retroviralen Vektor pMP71 kloniert. In dieser Arbeit wurden Vektoren für 4 verschiedene TCRs generiert (Tabelle 9). Der γ-retrovirale Vektor pMP71 (Engels et al. 2003) wurde verwendet, da er eine stabile und hohe Expressionsrate in T-Lymphozyten zeigt, selbst unter Anwendung von good manufacturing practice (GMP) Bedingungen. Aus diesem Grund ist dieser Vektor auch für einen potentiellen Einsatz in klinischen Studien geeignet. Durch diese Eigenschaften wurde der Vektor pMP71 bereits in Studien des TCR Transfers erfolgreich eingesetzt (z.B. Engels et al. 2005; Sommermeyer et al. 2006; Heemskerk 2010; Thomas et al. 2010; Linnemann et al. 2013). 84 Diskussion Jurkat 76 Zellen exprimieren keinen endogenen TCR und sind somit besonders geeignet die Expressionsfähigkeit transduzierter TCRs zu ermitteln (Heemskerk et al. 2003). Bei der TCR Transduktion in Jurkat 76 ergaben sich für 3 von 4 unterschiedlichen TCRs die durchschnittlichen Oberflächenexpressionen von 2,5 % (TCR 1), 1 % (TCR 3) und 3,5 % (TCR 4) (Abbildung 27). Die Oberflächenexpression von den Crf1/p41 spezifischen TCRs, transduziert in CD4+ T-Zellen zeigten spenderspezifische Unterschiede und die durchschnittliche Expression war niedriger im Vergleich zu transduzierten Jurkat 76 Zellen (Abbildung 29). Die Konkurrenz zum endogenen TCR und die komplexere Transduktion von CD4+ T-Zellen könnten dafür verantwortlich sein (van Loenen et al. 2010; Linnemann et al. 2011; Wieczorek et al. 2013). Bei TCR 2 konnte auch nach wiederholter Transduktion keine Bindung des Tetramers festgestellt werden (Abbildung 28, Abbildung 29). Die Ursache könnte zum einen ein schwacher TCR sein, welcher in starker Konkurrenz zu endogenem TCR steht und zum anderen, könnte der TCR sehr schnell abgebaut worden sein (Recycling), durch z.B. den inkorrekter Zusammenabbau der TCR α und β Kette während oder nach der Translation (Call et al. 2007). Aufgrund der geringen Oberflächenexpression des TCR, ist es sinnvoll eine Optimierung der Sequenz und des Vektorsystems durchzuführen. Die verschiedenen Optimierungsstrategien werden im folgenden Abschnitt näher erläutert. 8.3 Optimierung des Vektors pMP71 (TCR 1 optimiert) führt zu einer erhöhten Expression von TCR 1 auf Jurkat 76 Zellen und primären CD4+ T-Zellen Entscheidend für einen erfolgreichen Transfer von TCRs ist ein stabiler TCR Komplex. Diese Stabilität kann durch eine stabile Bindung der TCR α und β Ketten zu einander, durch Expressions- „starke“ TCRs erreicht werden (Sommermeyer et al. 2006). Eine Kombination von Expressions- „starken“ TCRs und eine starke Bindung der CD3 Komponenten CD3γε, CD3δε und ξξ untereinander und zum TCR sind entscheidend für die Stabilität des TCR-Komplex (Heemskerk et al. 2007). Momentan forschen einige Arbeitsgruppen daran, die komplexen extrazellulären Bindungen von CD3 Komponenten und TCR Komplexen zu untersuchen (Kuhns et al. 2007; Fernandes et al. 2012; Kuhns et al. 2012). Durch transferierte TCRs wird die TCR Komplexbildung stark beeinflusst, denn es kommt zur Konkurrenz, mit endogenen TCRs wodurch kombinierte TCRs entstehen können. Ebenfalls kann sich durch kombinierte TCRs das Risiko von Autoimmunreaktionen erhöhen. Bendle et al. 2010 haben gezeigt, dass ein TCR Gen Transfer im Mausmodell eine tödliche Autoimmunreaktion durch Zytokinausschüttung induziert und dass die kombinierten TCRs für das Auslösen der Autoimmunreaktion verantwortlich sind. Bei der Transduktion von TCRs in humane T-Zelllinien reduziert sich 85 Diskussion die Expression des gewünschten TCR, durch die Entstehung verschiedener TCR α/β Kombinationen. Ebenso können durch die verschiedenen Kombinationen TCRs mit neuen reaktiven Eigenschaften entstehen, deren T-Zellen eine gesundheitsgefährdende Immunreaktion hervorrufen können (auto- und allo- reaktiv) (van Loenen et al. 2010). Die Auswirkungen der kombinierten TCRs, sowie das Auslösen der Autoimmunreaktionen werden bis heute in einigen Arbeiten diskutiert (Linnemann et al. 2011; Wieczorek et al. 2013). Sequenzen können ebenfalls Einfluss auf den Translationslevel haben, so z.B. durch Gene die seltene Codons beinhalten, die für selten auftretende tRNA codieren und die Effektivität der Translation mindern und so die Expression beeinflussen. Ein Codon gilt als “selten”, wenn dieses 50 % seltener angetroffen wird, als Codons die für dieselbe Aminosäure codieren (Scholten et al. 2006). Eine Codonoptimierung kann die Expression des TCR erhöhen (Scholten et al. 2006; Sommermeyer et al. 2010). Alle diese Sachverhalte haben Auswirkungen auf die Halbwertszeiten von TCRs und nehmen Einfluss auf die Oberflächenexpression. In dieser Arbeit wurde eine Codonoptimierung von TCR 1 durchgeführt und die höhere Stabilität und stärkere Paarung des TCR αβ Komplex durch Optimierungen forciert (Abbildung 25). Im Einzelnen werden die Optimierungsstrategien im folgenden Abschnitt beschreiben. Um die Ausbildung gemischter TCRs einzuschränken bzw. zu verhindern, wurden bei der Optimierung des Crf1/p41 spezifischen TCR 1 die konstanten Ketten durch murine ersetzt und eine zusätzliche Disulfidbrücke im Bereich der konstanten Region eingebaut. Durch diese Veränderungen wird die TCR Komplex Bildung der transduzierten α Ketten mit ihren zugehörigen, transduzierten β Ketten stark erhöht. Es hat sich gezeigt, dass durch die Murinisierung der TCR konstanten Ketten, die Oberflächenexpression höher ist, verglichen mit der des unveränderten TCR. Gründe für diese Erhöhung könnten die stärkere Bindung der murinen Sequenz zu CD3 Komplexen sein, aber auch die stärkere Bindung der murinen Ketten zueinander, wodurch die Anzahl kombinierter TCRs reduziert wird (Cohen et al. 2006; Sommermeyer et al. 2010). Der Einbau weiterer Disulfidbrücken in die konstante Region des TCR zeichnet sich ebenfalls durch eine erhöhte Oberflächenexpression aus und reduziert das Auftreten kombinierter TCRs (Cohen et al. 2007; Kuball et al. 2007). Des Weiteren wurde das Vektorsystem modifiziert, indem der TCR in einen einzigen Vektor kloniert wurde. Damit wird das Problem umgangen, dass zwei verschiedene Vektorpartikel bzw. Viruspartikel (von TCR α Kette und von TCR β Kette) die gleiche Zelle infizieren müssen. Eine Vektorklonierung von β Kette und α Kette, getrennt durch einen Peptidlinker, zeigt hierbei die höchste Expression (Leisegang et al. 2008). 86 Diskussion Zusammenfassend lässt sich sagen, dass alle hier geschilderten Methoden zu einer erhöhten Oberflächenexpression des modifizierten TCR führen und eine erhöhte funktionelle Avidität der TCR transduzierten T-Zellen begünstigen (Hart et al. 2008). Dies wurde auch in der vorliegenden Arbeit bestätigt, denn durch die Optimierungen des Crf1/p41 spezifischen TCR 1 erhöhte sich die Expression um das 3 bis 5fache in Jurkat 76 und um das 2fache in CD4+ T-Zellen. Die verschiedenen Optimierungsstrategien werden laufend verbessert und weiteren Tests unterzogen. So wurden durch die Konkurrenz-Problematik zum endogenen TCR die chimärischen Antigen Rezeptoren (CARs) entwickelt. CARs haben den Vorteil, dass sie aus einem einzelnen variablen Fragment (scFv), gebunden an CD3, bestehen und so bei einem Transfer nicht mit endogenem TCR konkurrieren, oder kombinieren. Ebenso sind sie im Vergleich zu TCRs HLA-unabhängig. Jedoch sind auch Nachteile bezüglich der CARs zu beachten, zum einen wurde in frühen Studien angemerkt, dass CARs exprimierende T-Zellen weniger sensitiv gegenüber Peptid sind, als T-Zellen, die einen αβ TCR exprimieren (Zhang et al. 2004). Zum anderen können CARs eine zu hohe Zytokinausschüttung induzieren oder in einigen Fällen könnte die Signalschwelle so niedrig sein, dass in T-Zellen auch ohne Antigenpräsentation eine Immunantwort ausgelöst werden kann (Ramos et al. 2011). Ebenfalls sollte die Immunogenität nicht außer Acht gelassen werden, da das scFv von der Maus stammt und somit eine Immunantwort und Eliminierung der CAR transduzierten T-Zellen ausgelöst werden kann (Thomas et al. 2010; Wieczorek et al. 2013). Aufgrund der hier aufgeführten Nachteile, die nicht für den αβ TCR zutreffen, ist der TCR Einsatz für einen Transfer den CARs vorzuziehen. 8.4 Die Spezifität und Funktionalität der Crf1/p41 spezifischen T-Zell-Rezeptor transduzierter auf CD4+ T-Zellen wurde bestätigt Für transduzierte CD4+ T-Zellen (TCR 1 optimiert) von 4 unterschiedlichen Spendern, die unterschiedliche HLA-DRB1* Allele exprimieren, wurden funktionelle Analysen durchgeführt. Dabei konnte gezeigt werden, dass transduzierte CD4+ T-Zellen (TCR 1 optimiert), stimuliert durch Crf1/p41 beladene mDC (von Spendern, die das HLA-DRB1*04 Allel exprimieren), proliferieren und das TH1 Zytokin IFN-γ produzieren (Abbildung 31, Abbildung 32). Über diese Analysen ist die Funktionalität der Crf1/p41 spezifischen TCR 1 optimiert transduzierten T-Zellen bestätigt. 87 Diskussion 8.5 Einsatzmöglichkeiten funktioneller Crf1/p41 spezifischer T-Zell-Rezeptor transduzierter T-Zellen Ein funktioneller A. fumigatus spezifischer TCR könnte eingesetzt werden für die Entwicklung eines adoptiven Transfers von modifizierten T-Zellen. Der adoptive Transfer von spezifischen T-Zellen, sowohl genetisch modifiziert, als auch nicht modifiziert, wurde bisher in der Krebstherapie angewandt und vorangetrieben (Restifo et al. 2012; Kalos et al. 2013). Auch bei anti-viralen Therapien kommt der adoptive Transfer spezifischer T-Zellen zum Einsatz (Einsele et al. 2002; Stemberger et al. 2014). Bei Infektionen, ausgelöst durch A. fumigatus konnte gezeigt werden, dass ein adoptiver Transfer von funktionellen TH1 Zellen eine protektive Immunantwort induziert. Im Mausmodell konnte durch den adoptiven Transfer von dendritischen Zellen, beladen mit Aspergillus Antigenen, oder durch Aspergillus spezifische CD4+ Splenozyten eine Reduktion der fungalen Infektion festgestellt werden (Cenci et al. 2000) und eine protektive TH1 Immunantwort wurde induziert (Bozza et al. 2003). In klinischen Studien wurde gezeigt, dass durch den adoptiven Transfer von Aspergillus spezifischen TH1 T-Zellklonen in Patienten, die an einer Aspergillus Infektion nach einer hämatopoetischen Stammzelltransplantation erkrankten, eine stabile T-Zell Immunantwort induziert wird, ohne eine GvHD auszulösen (Perruccio et al. 2005). Diese vielversprechenden Untersuchungen veranlassten viele Wissenschaftler diese weiter zu entwickeln, im Hinblick auf Aspergillus spezifische Antigene und ihre Immunantwort (Beck et al. 2006; Bozza et al. 2009; Stuehler et al. 2011; Jolink et al. 2013; Bedke et al. 2014). Des Weiteren wurden Protokolle entwickelt, um Aspergillus spezifische T-Zellen mit kommerziell verfügbaren Materialen und unter GMP Bedingungen zu generieren, was für die Durchführung klinischer Studien ein wichtiges Kriterium darstellt (Khanna et al. 2011; Gaundar et al. 2012; Tramsen et al. 2013). Der adoptive Transfer, mit TCR-modifizierten T-Zellen, hat gegenüber dem klassischen (nicht genetisch modifizierten) adoptiven Transfer den Vorteil, dass TCRs auf verschiedene T-Zell Sub-Populationen transferiert werden können, um so eine bestimmte antigenspezifische Immunreaktion auszulösen. (Kalos et al. 2013; Koste et al. 2014). Der TCR Transfer hat ebenfalls den Vorteil, dass der TCR den ursprünglichen Signalweg der T-Zelle verwendet und so das Risiko eines cytokine release syndrome minimiert wird (Wieczorek et al. 2013). Der erfolgreiche Transfer von genetisch modifizierten T-Zellen zur Bekämpfung des malignen Melanom und Sarkom wurde bereits beschrieben (Morgan et al. 2006; Robbins et al. 2011). Eine Adaption dieses Verfahrens könnte zur Bekämpfung von Aspergillus Infektionen oder als zusätzliche Behandlungsmethode zur medikamentösen anti-fungalen Therapie eingesetzt werden. Mit dieser Arbeit wurde durch 88 Diskussion die Identifizierung A. fumigatus spezifischer TCRs, die für einen Transfer geeignet sind, der erste Schritt gemacht. 8.6 Fazit und Ausblick In dieser Arbeit wurden A. fumigatus spezifische TCRs identifiziert, isoliert und in Vektoren kloniert. Die A. fumigatus spezifischen TCRs wurden erfolgreich auf CD4+ T-Zellen transferiert, was durch die Expressionsanalysen gezeigt wurde. Die funktionellen Eigenschaften der TCR transduzierten Zellen wurden durch die IFN-γ Produktion nach antigenspezifischer Stimulation bestätigt. Für zukünftige Projekte wäre die Identifizierung weiterer TCRs von weiteren Spendern, die Optimierung dieser TCRs und weitere Transduktionen auf unterschiedliche T-Zell SubPopulationen, mit Analyse des Zytokinprofils, denkbar. Insbesondere für den klinischen Einsatz sollten folgende Ansätze zur Optimierung in Erwägung gezogen werden. Bei der Optimierung durch Murinisierung ist die erhöhte Expression mit den Risiken einer induzierten Immunogenität abzuwägen. Sommermeyer et al. 2010 konnten zeigen, dass der Austausch von wenigen murinen Aminosäuren in der konstanten Region des humanen TCR zu einer erhöhten Expression des TCR führt und sich das Risiko einer Immunogentität, aufgrund der geringen Anzahl muriner Aminosäuren, nur geringfügig erhöht. Optimierungsstrategien, das Vektorsystem betreffend, könnten auch in Erwägung gezogen werden. Wie zum Beispiel lentivirale Vektoren (Bobisse et al. 2007), die den Vorteil besitzen, das inaktivierte Zellen transduziert werden können. Zu berücksichtigen ist, dass ein Einsatz lentiviraler Vektoren im klinischen Maßstab noch nicht so etabliert ist, wie die retroviralen MLV-Vektoren (Doerr et al. 2010, siehe www.clinicaltrails.gov). Ein nicht-virales Transposon basiertes Vektorsystem (Sleeping Beauty transposon) wurde weiterentwickelt, wobei die geringe Transduktionseffizienz, gegenüber retroviralen Vektorsystemen, besonders im Fokus steht (Peng et al. 2009). Ebenfalls könnten modifizierte Verpackungsproteine eingesetzt werden, die spezialisiert sind auf bestimmte Zellpopulationen, um die Effektivität von Vektoren zu erhöhen (Funke et al. 2008). Die Transduktion der optimierten TCRs auf verschiede T-Zell Sup-Populationen (z.B. TH1 und Treg) ermöglicht es, Einfluss auf ein breites Spektrum von Immunantworten, zu nehmen und durch Analysen des Zytokinprofils näher zu untersuchen. Die A. fumigatus spezifischen TCR transduzierten Zellen sollten weiteren funktionellen Tests unterzogen werden, z.B. bezüglich der Fähigkeit eine protektive Immunantwort bei der Stimulation mit Hyphen und Konidien zu induzieren. Der adaptive Transfer in ein 89 Diskussion Mausmodell wäre auch eine Möglichkeit die Wirksamkeit, für einen zukünftigen Einsatz in der humanen Therapie, weiterführend zu analysieren. 90 Literaturverzeichnis 9 Literaturverzeichnis Abbas, A. K., Lichtman, A. H. and Pillai, S. (2007). Cellular and molecular immunology. Philadelphia, Saunders Elsevier. Abbey, J. L. and O'Neill, H. C. (2008). "Expression of T-cell receptor genes during early T-cell development." Immunology and cell biology 86(2): 166-174. Aimanianda, V., Bayry, J., Bozza, S., Kniemeyer, O., Perruccio, K., Elluru, S. R., Clavaud, C., Paris, S., Brakhage, A. A., Kaveri, S. V., Romani, L. and Latge, J. P. (2009). "Surface hydrophobin prevents immune recognition of airborne fungal spores." Nature 460(7259): 1117-1121. Alcazar-Fuoli, L. and Mellado, E. (2014). "Current status of antifungal resistance and its impact on clinical practice." British journal of haematology 166(4): 471-484. Alekseeva, L., Huet, D., Femenia, F., Mouyna, I., Abdelouahab, M., Cagna, A., Guerrier, D., Tichanne-Seltzer, V., Baeza-Squiban, A., Chermette, R., Latge, J. P. and Berkova, N. (2009). "Inducible expression of beta defensins by human respiratory epithelial cells exposed to Aspergillus fumigatus organisms." BMC microbiology 9: 33. Arden, B., Clark, S. P., Kabelitz, D. and Mak, T. W. (1995). "Human T-cell receptor variable gene segment families." Immunogenetics 42(6): 455-500. Baddley, J. W., Stephens, J. M., Ji, X., Gao, X., Schlamm, H. T. and Tarallo, M. (2013). "Aspergillosis in Intensive Care Unit (ICU) patients: epidemiology and economic outcomes." BMC infectious diseases 13: 29. Beck, O., Topp, M. S., Koehl, U., Roilides, E., Simitsopoulou, M., Hanisch, M., Sarfati, J., Latge, J. P., Klingebiel, T., Einsele, H. and Lehrnbecher, T. (2006). "Generation of highly purified and functionally active human Th1 cells against Aspergillus fumigatus." Blood 107(6): 2562-2569. 91 Literaturverzeichnis Bedke, T., Iannitti, R. G., De Luca, A., Giovannini, G., Fallarino, F., Berges, C., Latge, J. P., Einsele, H., Romani, L. and Topp, M. S. (2014). "Distinct and complementary roles for Aspergillus fumigatus-specific Tr1 and Foxp3 regulatory T cells in humans and mice." Immunology and cell biology: 1-12. Bellocchio, S., Bozza, S., Montagnoli, C., Perruccio, K., Gaziano, R., Pitzurra, L. and Romani, L. (2005). "Immunity to Aspergillus fumigatus: the basis for immunotherapy and vaccination." Med Mycol 43 Suppl 1: S181-188. Bendle, G. M., Linnemann, C., Hooijkaas, A. I., Bies, L., de Witte, M. A., Jorritsma, A., Kaiser, A. D., Pouw, N., Debets, R., Kieback, E., Uckert, W., Song, J. Y., Haanen, J. B. and Schumacher, T. N. (2010). "Lethal graft-versushost disease in mouse models of T cell receptor gene therapy." Nature medicine 16(5): 565-570. Bielorai, B., Toren, A., Wolach, B., Mandel, M., Golan, H., Neumann, Y., Kaplinisky, C., Weintraub, M., Keller, N., Amariglio, N., Paswell, J. and Rechavi, G. (2000). "Successful treatment of invasive aspergillosis in chronic granulomatous disease by granulocyte transfusions followed by peripheral blood stem cell transplantation." Bone marrow transplantation 26(9): 1025-1028. Bjorkman, P. J. (1997). "MHC restriction in three dimensions: a view of T cell receptor/ligand interactions." Cell 89(2): 167-170. Bobisse, S., Zanovello, P. and Rosato, A. (2007). "T-cell receptor gene transfer by lentiviral vectors in adoptive cell therapy." Expert opinion on biological therapy 7(6): 893-906. Bozza, S., Clavaud, C., Giovannini, G., Fontaine, T., Beauvais, A., Sarfati, J., D'Angelo, C., Perruccio, K., Bonifazi, P., Zagarella, S., Moretti, S., Bistoni, F., Latgé, J.-P. and Romani, L. (2009). "Immune sensing of Aspergillus fumigatus proteins, glycolipids, and polysaccharides and the impact on Th immunity and vaccination." J Immunol 183(4): 2407-2414. 92 Literaturverzeichnis Bozza, S., Gaziano, R., Lipford, G. B., Montagnoli, C., Bacci, A., Francesco, P. D., Kurup, V. P., Wagner, H. and Romani, L. (2002). "Vaccination of mice against invasive aspergillosis with recombinant Aspergillus proteins and CpG oligodeoxynucleotides as adjuvants." Microbes Infect 4(13): 1281-1290. Bozza, S., Montagnoli, C., Gaziano, R., Rossi, G., Nkwanyuo, G., Bellocchio, S. and Romani, L. (2004). "Dendritic cell-based vaccination against opportunistic fungi." Vaccine 22(7): 857-864. Bozza, S., Perruccio, K., Montagnoli, C., Gaziano, R., Bellocchio, S., Burchielli, E., Nkwanyuo, G., Pitzurra, L., Velardi, A. and Romani, L. (2003). "A dendritic cell vaccine against invasive aspergillosis in allogeneic hematopoietic transplantation." Blood 102(10): 3807-3814. Brakhage, A. A. and Langfelder, K. (2002). "Menacing mold: the molecular biology of Aspergillus fumigatus." Annual review of microbiology 56: 433-455. Brown, G. D. (2011). "Innate antifungal immunity: the key role of phagocytes." Annual review of immunology 29: 1-21. Call, M. E., Pyrdol, J. and Wucherpfennig, K. W. (2004). "Stoichiometry of the Tcell receptor-CD3 complex and key intermediates assembled in the endoplasmic reticulum." The EMBO journal 23(12): 2348-2357. Call, M. E. and Wucherpfennig, K. W. (2007). "Common themes in the assembly and architecture of activating immune receptors." Nature reviews. Immunology 7(11): 841-850. Cenci, E., Mencacci, A., Bacci, A., Bistoni, F., Kurup, V. P. and Romani, L. (2000). "T cell vaccination in mice with invasive pulmonary aspergillosis." J Immunol 165(1): 381-388. Cenci, E., Perito, S., Enssle, K. H., Mosci, P., Latge, J. P., Romani, L. and Bistoni, F. (1997). "Th1 and Th2 cytokines in mice with invasive aspergillosis." Infect Immun 65(2): 564-570. 93 Literaturverzeichnis Chaudhary, N. and Marr, K. A. (2011). "Impact of Aspergillus fumigatus in allergic airway diseases." Clinical and translational allergy 1(1): 4. Chaudhary, N., Staab, J. F. and Marr, K. A. (2010). "Healthy human T-Cell Responses to Aspergillus fumigatus antigens." PLoS ONE 5(2): e9036. Cohen, C. J., Li, Y. F., El-Gamil, M., Robbins, P. F., Rosenberg, S. A. and Morgan, R. A. (2007). "Enhanced antitumor activity of T cells engineered to express T-cell receptors with a second disulfide bond." Cancer research 67(8): 3898-3903. Cohen, C. J., Zhao, Y., Zheng, Z., Rosenberg, S. A. and Morgan, R. A. (2006). "Enhanced antitumor activity of murine-human hybrid T-cell receptor (TCR) in human lymphocytes is associated with improved pairing and TCR/CD3 stability." Cancer research 66(17): 8878-8886. Dauer, M., Obermaier, B., Herten, J., Haerle, C., Pohl, K., Rothenfusser, S., Schnurr, M., Endres, S. and Eigler, A. (2003). "Mature dendritic cells derived from human monocytes within 48 hours: a novel strategy for dendritic cell differentiation from blood precursors." J Immunol 170(8): 4069-4076. Dauer, M., Schad, K., Herten, J., Junkmann, J., Bauer, C., Kiefl, R., Endres, S. and Eigler, A. (2005). "FastDC derived from human monocytes within 48 h effectively prime tumor antigen-specific cytotoxic T cells." Journal of immunological methods 302(1-2): 145-155. Davis, M. M. (1988). "Molecular genetics of T-cell antigen receptors." Hospital practice 23(5): 157-164, 169-170. Diaz-Arevalo, D., Bagramyan, K., Hong, T. B., Ito, J. I. and Kalkum, M. (2011). "CD4+ T cells mediate the protective effect of the recombinant Asp f3-based antiaspergillosis vaccine." Infection and immunity 79(6): 2257-2266. 94 Literaturverzeichnis Dinser, R., Grgic, A., Kim, Y. J., Pfreundschuh, M. and Schubert, J. (2005). "Successful treatment of disseminated aspergillosis with the combination of voriconazole, caspofungin, granulocyte transfusions, and surgery followed by allogeneic blood stem cell transplantation in a patient with primary failure of an autologous stem cell graft." European journal of haematology 74(5): 438-441. Doerr, H. W. and Aberle, J. (2010). Medizinische Virologie Grundlagen, Diagnostik, Prävention und Therapie viraler Erkrankungen ; 115 Tabellen. Stuttgart, Thieme. Einsele, H., Roosnek, E., Rufer, N., Sinzger, C., Riegler, S., Loffler, J., Grigoleit, U., Moris, A., Rammensee, H. G., Kanz, L., Kleihauer, A., Frank, F., Jahn, G. and Hebart, H. (2002). "Infusion of cytomegalovirus (CMV)-specific T cells for the treatment of CMV infection not responding to antiviral chemotherapy." Blood 99(11): 3916-3922. Elefanti, A., Mouton, J. W., Verweij, P. E., Tsakris, A., Zerva, L. and Meletiadis, J. (2013). "Amphotericin B- and voriconazole-echinocandin combinations against Aspergillus spp.: Effect of serum on inhibitory and fungicidal interactions." Antimicrobial agents and chemotherapy 57(10): 4656-4663. Engels, B., Cam, H., Schüler, T., Indraccolo, S., Gladow, M., Baum, C., Blankenstein, T. and Uckert, W. (2003). "Retroviral vectors for high-level transgene expression in T lymphocytes." Hum Gene Ther 14(12): 1155-1168. Engels, B., Noessner, E., Frankenberger, B., Blankenstein, T., Schendel, D. J. and Uckert, W. (2005). "Redirecting human T lymphocytes toward renal cell carcinoma specificity by retroviral transfer of T cell receptor genes." Human gene therapy 16(7): 799-810. Fernandes, R. A., Shore, D. A., Vuong, M. T., Yu, C., Zhu, X., Pereira-Lopes, S., Brouwer, H., Fennelly, J. A., Jessup, C. M., Evans, E. J., Wilson, I. A. and Davis, S. J. (2012). "T cell receptors are structures capable of initiating signaling in the absence of large conformational rearrangements." The Journal of biological chemistry 287(16): 13324-13335. 95 Literaturverzeichnis Fremont, D. H., Hendrickson, W. A., Marrack, P. and Kappler, J. (1996). "Structures of an MHC class II molecule with covalently bound single peptides." Science 272(5264): 1001-1004. Fugmann, S. D., Lee, A. I., Shockett, P. E., Villey, I. J. and Schatz, D. G. (2000). "The RAG proteins and V(D)J recombination: complexes, ends, and transposition." Annual review of immunology 18: 495-527. Funke, S., Maisner, A., Muhlebach, M. D., Koehl, U., Grez, M., Cattaneo, R., Cichutek, K. and Buchholz, C. J. (2008). "Targeted cell entry of lentiviral vectors." Molecular therapy : the journal of the American Society of Gene Therapy 16(8): 1427-1436. Garcia, K. C., Degano, M., Stanfield, R. L., Brunmark, A., Jackson, M. R., Peterson, P. A., Teyton, L. and Wilson, I. A. (2010). "An alpha beta T cell receptor structure at 2.5 A and its orientation in the TCR-MHC complex. Science. 1996. 274: 209-219." Journal of immunology 185(11): 6402-6412. Gaundar, S. S., Clancy, L., Blyth, E., Meyer, W. and Gottlieb, D. J. (2012). "Robust polyfunctional T-helper 1 responses to multiple fungal antigens from a cell population generated using an environmental strain of Aspergillus fumigatus." Cytotherapy 14(9): 1119-1130. Gersuk, G. M., Underhill, D. M., Zhu, L. and Marr, K. A. (2006). "Dectin-1 and TLRs permit macrophages to distinguish between different Aspergillus fumigatus cellular states." J Immunol 176(6): 3717-3724. Gonzalez-Galarza, F. F., Christmas, S., Middleton, D. and Jones, A. R. (2011). "Allele frequency net: a database and online repository for immune gene frequencies in worldwide populations." Nucleic acids research 39(Database issue): D913-919. Grazziutti, M. L., Rex, J. H., Cowart, R. E., Anaissie, E. J., Ford, A. and Savary, C. A. (1997). "Aspergillus fumigatus conidia induce a Th1-type cytokine response." J Infect Dis 176(6): 1579-1583. 96 Literaturverzeichnis Grigull, L., Beilken, A., Schmid, H., Kirschner, P., Sykora, K. W., Linderkamp, C., Donnerstag, F., Goudeva, L., Heuft, H. G. and Welte, K. (2006). "Secondary prophylaxis of invasive fungal infections with combination antifungal therapy and G-CSF-mobilized granulocyte transfusions in three children with hematological malignancies." Supportive care in cancer : official journal of the Multinational Association of Supportive Care in Cancer 14(7): 783-786. Guy, C. S., Vignali, K. M., Temirov, J., Bettini, M. L., Overacre, A. E., Smeltzer, M., Zhang, H., Huppa, J. B., Tsai, Y. H., Lobry, C., Xie, J., Dempsey, P. J., Crawford, H. C., Aifantis, I., Davis, M. M. and Vignali, D. A. (2013). "Distinct TCR signaling pathways drive proliferation and cytokine production in T cells." Nature immunology 14(3): 262-270. Hamill, R. J. (2013). "Amphotericin B formulations: a comparative review of efficacy and toxicity." Drugs 73(9): 919-934. Hart, D. P., Xue, S. A., Thomas, S., Cesco-Gaspere, M., Tranter, A., Willcox, B., Lee, S. P., Steven, N., Morris, E. C. and Stauss, H. J. (2008). "Retroviral transfer of a dominant TCR prevents surface expression of a large proportion of the endogenous TCR repertoire in human T cells." Gene Ther 15(8): 625-631. Hebart, H., Bollinger, C., Fisch, P., Sarfati, J., Meisner, C., Baur, M., Loeffler, J., Monod, M., Latge, J. P. and Einsele, H. (2002). "Analysis of T-cell responses to Aspergillus fumigatus antigens in healthy individuals and patients with hematologic malignancies." Blood 100(13): 4521-4528. Heemskerk, M. H., Hoogeboom, M., de Paus, R. A., Kester, M. G., van der Hoorn, M. A., Goulmy, E., Willemze, R. and Falkenburg, J. H. (2003). "Redirection of antileukemic reactivity of peripheral T lymphocytes using gene transfer of minor histocompatibility antigen HA-2-specific T-cell receptor complexes expressing a conserved alpha joining region." Blood 102(10): 35303540. Heemskerk, M. H. M. (2010). "T-cell receptor gene transfer for the treatment of leukemia and other tumors." Haematologica 95(1): 15-19. 97 Literaturverzeichnis Heemskerk, M. H. M., Hagedoorn, R. S., van der Hoorn, M. A. W. G., van der Veken, L. T., Hoogeboom, M., Kester, M. G. D., Willemze, R. and Falkenburg, J. H. F. (2007). "Efficiency of T-cell receptor expression in dual-specific T cells is controlled by the intrinsic qualities of the TCR chains within the TCR-CD3 complex." Blood 109(1): 235-243. Ito, J. I., Lyons, J. M., Hong, T. B., Tamae, D., Liu, Y.-K., Wilczynski, S. P. and Kalkum, M. (2006). "Vaccinations with recombinant variants of Aspergillus fumigatus allergen Asp f 3 protect mice against invasive aspergillosis." Infect Immun 74(9): 5075-5084. Jolink, H., Meijssen, I. C., Hagedoorn, R. S., Arentshorst, M., Drijfhout, J. W., Mulder, A., Claas, F. H., van Dissel, J. T., Falkenburg, J. H. and Heemskerk, M. H. (2013). "Characterization of the T-cell-mediated immune response against the Aspergillus fumigatus proteins Crf1 and catalase 1 in healthy individuals." The Journal of infectious diseases 208(5): 847-856. Jorgensen, K. J., Gotzsche, P. C., Dalboge, C. S. and Johansen, H. K. (2014). "Voriconazole versus amphotericin B or fluconazole in cancer patients with neutropenia." The Cochrane database of systematic reviews 2: CD004707. Kalos, M. and June, C. H. (2013). "Adoptive T cell transfer for cancer immunotherapy in the era of synthetic biology." Immunity 39(1): 49-60. Khanna, N., Stuehler, C., Conrad, B., Lurati, S., Krappmann, S., Einsele, H., Berges, C. and Topp, M. S. (2011). "Generation of a multiple pathogen-specfic Tcell product for adoptive immunotherapy based on activation-dependent expression of CD154." Blood. Koste, L., Beissert, T., Hoff, H., Pretsch, L., Tureci, O. and Sahin, U. (2014). "Tcell receptor transfer into human T cells with ecotropic retroviral vectors." Gene therapy 21(5): 533-538. Kuball, J., Dossett, M. L., Wolfl, M., Ho, W. Y., Voss, R. H., Fowler, C. and Greenberg, P. D. (2007). "Facilitating matched pairing and expression of TCR chains introduced into human T cells." Blood 109(6): 2331-2338. 98 Literaturverzeichnis Kuhns, M. S. and Badgandi, H. B. (2012). "Piecing together the family portrait of TCR-CD3 complexes." Immunological reviews 250(1): 120-143. Kuhns, M. S. and Davis, M. M. (2007). "Disruption of extracellular interactions impairs T cell receptor-CD3 complex stability and signaling." Immunity 26(3): 357369. Kwon-Chung, K. J. and Sugui, J. A. (2009). "What do we know about the role of gliotoxin in the pathobiology of Aspergillus fumigatus?" Medical mycology 47 Suppl 1: S97-103. Kwon-Chung, K. J. and Sugui, J. A. (2013). "Aspergillus fumigatus--what makes the species a ubiquitous human fungal pathogen?" PLoS pathogens 9(12): e1003743. Lamy, B., Moutaouakil, M., Latge, J. P. and Davies, J. (1991). "Secretion of a potential virulence factor, a fungal ribonucleotoxin, during human aspergillosis infections." Molecular microbiology 5(7): 1811-1815. Latge, J. P. (1999). "Aspergillus fumigatus and aspergillosis." Clin Microbiol Rev 12(2): 310-350. Latge, J. P. (2001). "The pathobiology of Aspergillus fumigatus." Trends Microbiol 9(8): 382-389. Latge, J. P. (2010). "Tasting the fungal cell wall." Cellular microbiology 12(7): 863872. Lefranc, M. P. (2004). "IMGT-ONTOLOGY and IMGT databases, tools and Web resources for immunogenetics and immunoinformatics." Molecular immunology 40(10): 647-660. Leisegang, M., Engels, B., Meyerhuber, P., Kieback, E., Sommermeyer, D., Xue, S. A., Reuss, S., Stauss, H. and Uckert, W. (2008). "Enhanced functionality of T cell receptor-redirected T cells is defined by the transgene cassette." Journal of molecular medicine 86(5): 573-583. 99 Literaturverzeichnis Linnemann, C., Heemskerk, B., Kvistborg, P., Kluin, R. J., Bolotin, D. A., Chen, X., Bresser, K., Nieuwland, M., Schotte, R., Michels, S., GomezEerland, R., Jahn, L., Hombrink, P., Legrand, N., Shu, C. J., Mamedov, I. Z., Velds, A., Blank, C. U., Haanen, J. B., Turchaninova, M. A., Kerkhoven, R. M., Spits, H., Hadrup, S. R., Heemskerk, M. H., Blankenstein, T., Chudakov, D. M., Bendle, G. M. and Schumacher, T. N. (2013). "High-throughput identification of antigen-specific TCRs by TCR gene capture." Nature medicine 19(11): 1534-1541. Linnemann, C., Schumacher, T. N. and Bendle, G. M. (2011). "T-cell receptor gene therapy: critical parameters for clinical success." The Journal of investigative dermatology 131(9): 1806-1816. Marr, K. A., Boeckh, M., Carter, R. A., Kim, H. W. and Corey, L. (2004). "Combination antifungal therapy for invasive aspergillosis." Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 39(6): 797-802. Marr, K. A., Carter, R. A., Boeckh, M., Martin, P. and Corey, L. (2002). "Invasive aspergillosis in allogeneic stem cell transplant recipients: changes in epidemiology and risk factors." Blood 100(13): 4358-4366. Marr, K. A., Patterson, T. and Denning, D. (2002). "Aspergillosis. Pathogenesis, clinical manifestations, and therapy." Infect Dis Clin North Am 16(4): 875-894. Marrack, P., Scott-Browne, J. P., Dai, S., Gapin, L. and Kappler, J. W. (2008). "Evolutionarily conserved amino acids that control TCR-MHC interaction." Annual review of immunology 26: 171-203. Marsh, S. G., Bodmer, J. G., Albert, E. D., Bodmer, W. F., Bontrop, R. E., Dupont, B., Erlich, H. A., Hansen, J. A., Mach, B., Mayr, W. R., Parham, P., Petersdorf, E. W., Sasazuki, T., Schreuder, G. M., Strominger, J. L., Svejgaard, A. and Terasaki, P. I. (2001). "Nomenclature for factors of the HLA system, 2000." Human immunology 62(4): 419-468. 100 Literaturverzeichnis Morgan, R. A., Dudley, M. E., Wunderlich, J. R., Hughes, M. S., Yang, J. C., Sherry, R. M., Royal, R. E., Topalian, S. L., Kammula, U. S., Restifo, N. P., Zheng, Z., Nahvi, A., de Vries, C. R., Rogers-Freezer, L. J., Mavroukakis, S. A. and Rosenberg, S. A. (2006). "Cancer regression in patients after transfer of genetically engineered lymphocytes." Science 314(5796): 126-129. Morris, E., Hart, D., Gao, L., Tsallios, A., Xue, S. A. and Stauss, H. (2006). "Generation of tumor-specific T-cell therapies." Blood reviews 20(2): 61-69. Murphy, K. P., Janeway, C., Travers, P., Walport, M., Mowat, A. and Weaver, C. (2012). Janeway's immunobiology. New York, N.Y. ; London, Garland Science. Murugaiyan, G., Mittal, A., Lopez-Diego, R., Maier, L. M., Anderson, D. E. and Weiner, H. L. (2009). "IL-27 is a key regulator of IL-10 and IL-17 production by human CD4+ T cells." Journal of immunology 183(4): 2435-2443. Orciuolo, E., Stanzani, M., Canestraro, M., Galimberti, S., Carulli, G., Lewis, R., Petrini, M. and Komanduri, K. V. (2007). "Effects of Aspergillus fumigatus gliotoxin and methylprednisolone on human neutrophils: implications for the pathogenesis of invasive aspergillosis." J Leukoc Biol 82(4): 839-848. Peng, P. D., Cohen, C. J., Yang, S., Hsu, C., Jones, S., Zhao, Y., Zheng, Z., Rosenberg, S. A. and Morgan, R. A. (2009). "Efficient nonviral Sleeping Beauty transposon-based TCR gene transfer to peripheral blood lymphocytes confers antigen-specific antitumor reactivity." Gene therapy 16(8): 1042-1049. Perruccio, K., Bozza, S., Montagnoli, C., Bellocchio, S., Aversa, F., Martelli, M., Bistoni, F., Velardi, A. and Romani, L. (2004). "Prospects for dendritic cell vaccination against fungal infections in hematopoietic transplantation." Blood Cells Mol Dis 33(3): 248-255. Perruccio, K., Tosti, A., Burchielli, E., Topini, F., Ruggeri, L., Carotti, A., Capanni, M., Urbani, E., Mancusi, A., Aversa, F., Martelli, M. F., Romani, L. and Velardi, A. (2005). "Transferring functional immune responses to pathogens after haploidentical hematopoietic transplantation." Blood 106(13): 4397-4406. 101 Literaturverzeichnis Raad, II, Chaftari, A. M., Al Shuaibi, M. M., Jiang, Y., Shomali, W., Cortes, J. E., Lichtiger, B. and Hachem, R. Y. (2013). "Granulocyte transfusions in hematologic malignancy patients with invasive pulmonary aspergillosis: outcomes and complications." Annals of oncology : official journal of the European Society for Medical Oncology / ESMO 24(7): 1873-1879. Ramos, C. A. and Dotti, G. (2011). "Chimeric antigen receptor (CAR)-engineered lymphocytes for cancer therapy." Expert opinion on biological therapy 11(7): 855873. Restifo, N. P., Dudley, M. E. and Rosenberg, S. A. (2012). "Adoptive immunotherapy for cancer: harnessing the T cell response." Nature reviews. Immunology 12(4): 269-281. Riddell, S. R. and Greenberg, P. D. (1990). "The use of anti-CD3 and anti-CD28 monoclonal antibodies to clone and expand human antigen-specific T cells." J Immunol Methods 128(2): 189-201. Rivera, A., Hohl, T. and Pamer, E. G. (2006). "Immune responses to Aspergillus fumigatus infections." Biol Blood Marrow Transplant 12(1 Suppl 1): 47-49. Robbins, P. F., Morgan, R. A., Feldman, S. A., Yang, J. C., Sherry, R. M., Dudley, M. E., Wunderlich, J. R., Nahvi, A. V., Helman, L. J., Mackall, C. L., Kammula, U. S., Hughes, M. S., Restifo, N. P., Raffeld, M., Lee, C. C., Levy, C. L., Li, Y. F., El-Gamil, M., Schwarz, S. L., Laurencot, C. and Rosenberg, S. A. (2011). "Tumor regression in patients with metastatic synovial cell sarcoma and melanoma using genetically engineered lymphocytes reactive with NY-ESO-1." Journal of clinical oncology : official journal of the American Society of Clinical Oncology 29(7): 917-924. Roilides, E., Lyman, C. A., Panagopoulou, P. and Chanock, S. (2003). "Immunomodulation of invasive fungal infections." Infectious disease clinics of North America 17(1): 193-219. Romani, L. (2004). "Immunity to fungal infections." Nat Rev Immunol 4(1): 1-23. 102 Literaturverzeichnis Romani, L. (2008). "Cell mediated immunity to fungi: a reassessment." Med Mycol 46(6): 515-529. Romani, L. (2011). "Immunity to fungal infections." Nature reviews. Immunology 11(4): 275-288. Romani, L. and Puccetti, P. (2007). "Controlling pathogenic inflammation to fungi." Expert Rev Anti Infect Ther 5(6): 1007-1017. Rudolph, M. G., Stanfield, R. L. and Wilson, I. A. (2006). "How TCRs bind MHCs, peptides, and coreceptors." Annual review of immunology 24: 419-466. Safdar, A. (2006). "Strategies to enhance immune function in hematopoietic transplantation recipients who have fungal infections." Bone marrow transplantation 38(5): 327-337. Schaffner, A., Douglas, H. and Braude, A. (1982). "Selective protection against conidia by mononuclear and against mycelia by polymorphonuclear phagocytes in resistance to Aspergillus. Observations on these two lines of defense in vivo and in vitro with human and mouse phagocytes." J Clin Invest 69(3): 617-631. Schipper, R. F., van Els, C. A., D'Amaro, J. and Oudshoorn, M. (1996). "Minimal phenotype panels. A method for achieving maximum population coverage with a minimum of HLA antigens." Human immunology 51(2): 95-98. Scholten, K. B., Kramer, D., Kueter, E. W., Graf, M., Schoedl, T., Meijer, C. J., Schreurs, M. W. and Hooijberg, E. (2006). "Codon modification of T cell receptors allows enhanced functional expression in transgenic human T cells." Clinical immunology 119(2): 135-145. Schreuder, G. M., Hurley, C. K., Marsh, S. G., Lau, M., Maiers, M., Kollman, C. and Noreen, H. J. (2001). "The HLA dictionary 2001: a summary of HLA-A, -B, -C, -DRB1/3/4/5, -DQB1 alleles and their association with serologically defined HLA-A, -B, -C, -DR, and -DQ antigens." Human immunology 62(8): 826-849. 103 Literaturverzeichnis Segal, B. H., Kwon-Chung, J., Walsh, T. J., Klein, B. S., Battiwalla, M., Almyroudis, N. G., Holland, S. M. and Romani, L. (2006). "Immunotherapy for fungal infections." Clin Infect Dis 42(4): 507-515. Segal, B. H. and Walsh, T. J. (2006). "Current approaches to diagnosis and treatment of invasive aspergillosis." Am J Respir Crit Care Med 173(7): 707-717. Sommermeyer, D., Neudorfer, J., Weinhold, M., Leisegang, M., Engels, B., Noessner, E., Heemskerk, M. H. M., Charo, J., Schendel, D. J., Blankenstein, T., Bernhard, H. and Uckert, W. (2006). "Designer T cells by T cell receptor replacement." Eur J Immunol 36(11): 3052-3059. Sommermeyer, D. and Uckert, W. (2010). "Minimal amino acid exchange in human TCR constant regions fosters improved function of TCR gene-modified T cells." Journal of immunology 184(11): 6223-6231. Stemberger, C., Graef, P., Odendahl, M., Albrecht, J., Dossinger, G., Anderl, F., Buchholz, V. R., Gasteiger, G., Schiemann, M., Grigoleit, G. U., Schuster, F. R., Borkhardt, A., Versluys, B., Tonn, T., Seifried, E., Einsele, H., Germeroth, L., Busch, D. H. and Neuenhahn, M. (2014). "Lowest numbers of primary CD8+ T cells can reconstitute protective immunity upon adoptive immunotherapy." Blood 124(4): 628-637. Stuehler, C., Khanna, N., Bozza, S., Zelante, T., Moretti, S., Kruhm, M., Lurati, S., Conrad, B., Worschech, E., Stevanovic, S., Krappmann, S., Einsele, H., Latgé, J.-P., Loeffler, J., Romani, L. and Topp, M. S. (2011). "Cross-protective TH1 immunity against Aspergillus fumigatus and Candida albicans." Blood 117(22): 5881-5891. Sun, Z. Y., Kim, S. T., Kim, I. C., Fahmy, A., Reinherz, E. L. and Wagner, G. (2004). "Solution structure of the CD3 epsilon delta ectodomain and comparison with CD3 epsilon gamma as a basis for modeling T cell receptor topology and signaling." Proceedings of the National Academy of Sciences of the United States of America 101(48): 16867-16872. 104 Literaturverzeichnis Szymczak, A. L., Workman, C. J., Wang, Y., Vignali, K. M., Dilioglou, S., Vanin, E. F. and Vignali, D. A. (2004). "Correction of multi-gene deficiency in vivo using a single 'self-cleaving' 2A peptide-based retroviral vector." Nature biotechnology 22(5): 589-594. Thomas, S., Stauss, H. J. and Morris, E. C. (2010). "Molecular immunology lessons from therapeutic T-cell receptor gene transfer." Immunology 129(2): 170177. Torosantucci, A., Bromuro, C., Chiani, P., De Bernardis, F., Berti, F., Galli, C., Norelli, F., Bellucci, C., Polonelli, L., Costantino, P., Rappuoli, R. and Cassone, A. (2005). "A novel glyco-conjugate vaccine against fungal pathogens." The Journal of experimental medicine 202(5): 597-606. Torosantucci, A., Chiani, P., Bromuro, C., De Bernardis, F., Palma, A. S., Liu, Y., Mignogna, G., Maras, B., Colone, M., Stringaro, A., Zamboni, S., Feizi, T. and Cassone, A. (2009). "Protection by anti-beta-glucan antibodies is associated with restricted beta-1,3 glucan binding specificity and inhibition of fungal growth and adherence." PLoS ONE 4(4): e5392. Tramsen, L., Schmidt, S., Boenig, H., Latge, J. P., Lass-Florl, C., Roeger, F., Seifried, E., Klingebiel, T. and Lehrnbecher, T. (2013). "Clinical-scale generation of multi-specific anti-fungal T cells targeting Candida, Aspergillus and mucormycetes." Cytotherapy 15(3): 344-351. van de Veerdonk, F. L., Kullberg, B. J., van der Meer, J. W., Gow, N. A. and Netea, M. G. (2008). "Host-microbe interactions: innate pattern recognition of fungal pathogens." Current opinion in microbiology 11(4): 305-312. van Loenen, M. M., de Boer, R., Amir, A. L., Hagedoorn, R. S., Volbeda, G. L., Willemze, R., van Rood, J. J., Falkenburg, J. H. F. and Heemskerk, M. H. M. (2010). "Mixed T cell receptor dimers harbor potentially harmful neoreactivity." Proc Natl Acad Sci U S A 107(24): 10972-10977. 105 Literaturverzeichnis Vermeulen, E., Lagrou, K. and Verweij, P. E. (2013). "Azole resistance in Aspergillus fumigatus: a growing public health concern." Current opinion in infectious diseases 26(6): 493-500. Wieczorek, A. and Uharek, L. (2013). "Genetically modified T cells for the treatment of malignant disease." Transfusion medicine and hemotherapy : offizielles Organ der Deutschen Gesellschaft fur Transfusionsmedizin und Immunhamatologie 40(6): 388-402. Wuthrich, M., Deepe, G. S., Jr. and Klein, B. (2012). "Adaptive immunity to fungi." Annual review of immunology 30: 115-148. Xue, S. A. and Stauss, H. J. (2007). "Enhancing immune responses for cancer therapy." Cell Mol Immunol 4(3): 173-184. Yin, Y., Wang, X. X. and Mariuzza, R. A. (2012). "Crystal structure of a complete ternary complex of T-cell receptor, peptide-MHC, and CD4." Proceedings of the National Academy of Sciences of the United States of America 109(14): 54055410. Zelante, T., De Luca, A., Bonifazi, P., Montagnoli, C., Bozza, S., Moretti, S., Belladonna, M. L., Vacca, C., Conte, C., Mosci, P., Bistoni, F., Puccetti, P., Kastelein, R. A., Kopf, M. and Romani, L. (2007). "IL-23 and the Th17 pathway promote inflammation and impair antifungal immune resistance." European journal of immunology 37(10): 2695-2706. Zhang, T., He, X., Tsang, T. C. and Harris, D. T. (2004). "Transgenic TCR expression: comparison of single chain with full-length receptor constructs for Tcell function." Cancer gene therapy 11(7): 487-496. 106 Anhang 10 Anhang 10.1 Abkürzungen Abkürzung Erklärung A. fumigatus Aspergillus fumigatus ABPA allergische bronchopulmonale Aspergillose AmpR Ampicillin Resistenz APC Allophycocyanin APS Ammoniumpersulfat bp Basenpaare BSA Bovines Serum Albumin CAR Chimärischer Antigen Rezeptor CBA Cytometric Bead Array System CD cluster of differntiation CDR komplementaritätsbestimmende Region (complementarity determining region) CLR C-Typ Lektin Rezeptor CSF koloniestimulierender Faktor Cα bzw. Cβ konstante α Kette bzw. konstante β Kette des TCR D vielfältige/diverse Segmente (Diversität) des TCR β Locus DC dendritische Zellen DMSO Dimethyl Sulfoxid DNA Desoxyribonukleinsäure dNTPs Desoxyribonukleosidtriphosphate EDTA Ethylendiamintetraacetat ELISA Enzyme Linked Immunosorbent Assay ER Endoplasmatisches Retikulum FACS fluorescence-activated cell sorting FCS fetales Rinderserum (fetal calf serum) i Anhang Abkürzung Erklärung FCS forward scatter FITC Fluoresceinisothiocyanate FR Gerüstregion (framework region) GFP grünes Fluoreszenzprotein (green fluorescent protein) GM-CSF Granulocyte macrophage colony-stimulating factor GMP Good manufacturing practice GvHD Graft versus host disease Gy das Gray (SI-Einheit) HLA humane Leukozytenantigen (human leukocyte antigen) HSCT hämatopoetische Stammzelltransplantation (Hematopoietic stem cell transplantation) IA Invasive Aspergillose IFN-γ Interferon-γ IL Interleukin ITAM Immunrezeptor Tyrosinaktivierungssequenz (immunoreceptor tyrosin-based activation motif) J junktionale Segmente des TCR Locus LB Nährmedium für Bakterien (lysogeny broth) LTR DNA Wiederholungseinheit (long terminal repeat) MHC Haupthistokompatibilitätskomplex (major histocompatility comlex) MHC Haupthistokompatibilitätskomplex (major histocompatibility complex) NFAT nuclear factor of activated T cells NK natürliche Killerzellen P2A 2A Peptidlinker PAMP Pathogen-assoziierte molekulare Muster (pathogen-associated molecular pattern) PBMC mononucleare Zellen des peripheren Blutes (peripheral blood mononucleated cell) PBS phosphatgepufferte Salzlösung (phosphate buffered saline) ii Anhang Abkürzung Erklärung PCR Polymerase-Kettenreaktion (polymerase chain reaction) PE Phycoerythrin PGE2 Prostaglandin E2 PMN polymorphkernige Leukozyten (polymorphonuclear leukocytes) PRE posttranskriptionales regulatorisches Element PRR Mustererkennungs- Rezeptor (pattern recognition receptor) RAG recombination-activation gene RNA Ribonukleinsäure ROS oxidative Sauerstoffspezies (reactive oxygen species) RPMI 1640 Roswell Park Memorial Institute Medium RSS Rekombinationssignalsequenz RT reverse Transkriptase scFv Single chain variable Fragment SEB Superantigen (bakteriell) SSC side scatter TAE Tris- Acetat EDTA Puffer TCR T-Zell-Rezeptor (T cell receptor) TGF transforming growth factor TH T-Helferzellen TLR Toll-like Rezeptor TNF Tumor- Nekrose- Faktor Tr1 regulatorische T-Zellen Typ 1 Treg regulatorische T-Zellen tRNA Transfer Ribonukleinsäure UV Ultraviolet V variable Segmente des TCR Locus Vβ bzw. Vα variable β bzw. α Kette des TCR iii Anhang 10.2 Publikationen 2011 Stuehler, C., Khanna, N., Bozza, S., Zelante, T., Moretti, S., Kruhm, M., Lurati, S., Conrad, B., Worschech, E., Stevanovic, S., Krappmann, S., Einsele, H., Latgé, J. P., Loeffler, J., Romani, L. and Topp, M. S. (2011). Cross-protective TH1 immunity against Aspergillus fumigatus and Candida albicans. Blood. 117; 5881-5891 2014 Berges, C., Bedke, T., Stühler, C., Khanna, N., Zehnter, S., Kruhm, M., Winter, N., Chatterjee, M. M, Einsele, H., Topp, M. S. (2014). Combined targeting of PI3K/Akt and HSP90 synergistically suppresses essential functions of alloreactive T cells by multiple mechanisms. (Manuskript eingereicht zur Publikation) iv