Flüssige Biopsie: Alles aus einem Tropfen Blut?
Werbung
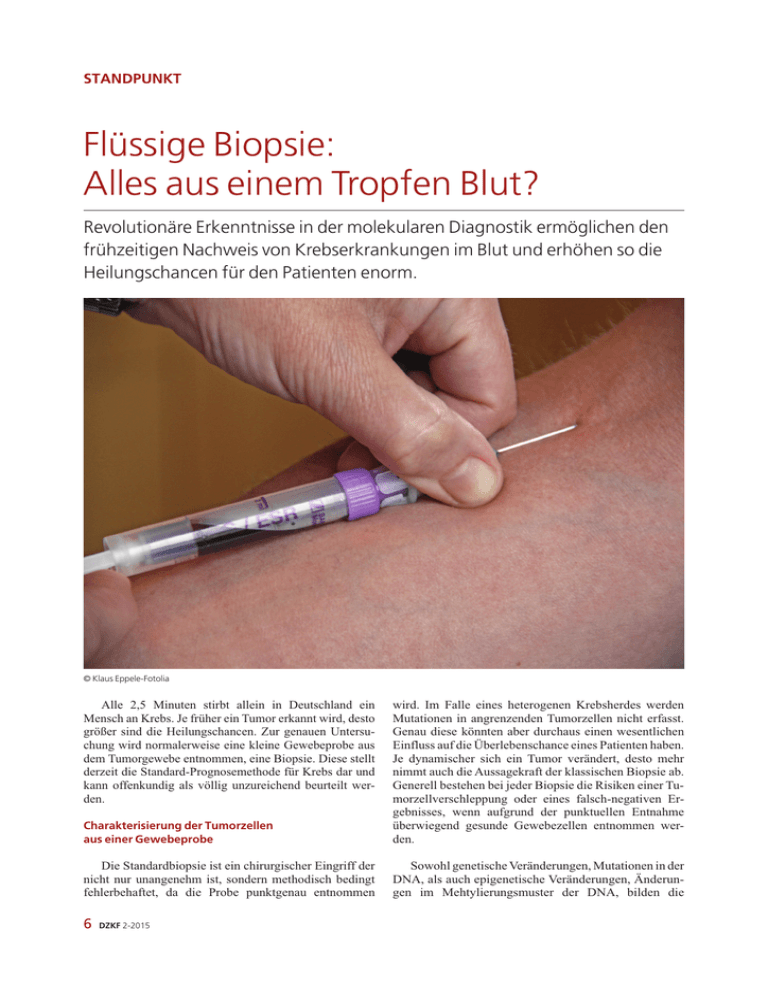
STANDPUNKT Flüssige Biopsie: Alles aus einem Tropfen Blut? Revolutionäre Erkenntnisse in der molekularen Diagnostik ermöglichen den frühzeitigen Nachweis von Krebserkrankungen im Blut und erhöhen so die Heilungschancen für den Patienten enorm. © Klaus Eppele-Fotolia Charakterisierung der Tumorzellen aus einer Gewebeprobe wird. Im Falle eines heterogenen Krebsherdes werden Mutationen in angrenzenden Tumorzellen nicht erfasst. Genau diese könnten aber durchaus einen wesentlichen Einfluss auf die Überlebenschance eines Patienten haben. Je dynamischer sich ein Tumor verändert, desto mehr nimmt auch die Aussagekraft der klassischen Biopsie ab. Generell bestehen bei jeder Biopsie die Risiken einer Tumorzellverschleppung oder eines falsch-negativen Ergebnisses, wenn aufgrund der punktuellen Entnahme überwiegend gesunde Gewebezellen entnommen werden. Die Standardbiopsie ist ein chirurgischer Eingriff der nicht nur unangenehm ist, sondern methodisch bedingt fehlerbehaftet, da die Probe punktgenau entnommen Sowohl genetische Veränderungen, Mutationen in der DNA, als auch epigenetische Veränderungen, Änderungen im Mehtylierungsmuster der DNA, bilden die Alle 2,5 Minuten stirbt allein in Deutschland ein Mensch an Krebs. Je früher ein Tumor erkannt wird, desto größer sind die Heilungschancen. Zur genauen Untersuchung wird normalerweise eine kleine Gewebeprobe aus dem Tumorgewebe entnommen, eine Biopsie. Diese stellt derzeit die Standard-Prognosemethode für Krebs dar und kann offenkundig als völlig unzureichend beurteilt werden. 6 DZKF 2-2015 STANDPUNKT Abb. 1: Schema zum Nachweis genetischer Veränderungen an zirkulierender zellfreier Tumor-DNA (ctDNA). Durch Apoptose oder Nekrose der Tumorzellen gelangt die freigesetzte DNA in die Blutbahn. In einer Blutprobe, der sogenannten flüssigen Biopsie, kann diese ctDNA nachgewiesen und untersucht werden. Sequenzierung und Datenanalyse ermöglichen die Charakterisierung von Punktmutationen, Chromosomenaberrationen, Methylierungsmustern oder anderen mit Krebserkrankungen assoziierten genetischen Veränderungen. Die ermittelten Daten bieten eine solide Grundlage für den behandelnden Arzt bzgl. Diagnose oder Überwachung des Krankheitsverlaufs. Grundlage der molekularen Diagnostik. Das unkontrollierbare Wachstum der Krebszellen beruht oft auf Mutationen der DNA. Da diese in gesunden Zellen nicht auftreten, kann man sie als diagnostische Marker in der Krebsdiagnose benutzen. Epigenetischen Veränderungen finden sich in fast allen Tumoren, und erste Studien deuten an, dass sie sogar an deren Entstehung beteiligt sind. Charakterisierung der Tumorzellen aus einer Blutprobe Immer wenn eine Krebszelle durch Apoptose oder Nekrose abstirbt, werden DNA-Fragmente der Tumorzelle freigesetzt und gelangen so als zirkulierende TumorDNA (ctDNA) in die Blutbahn (E Abb. 1). Diese ctDNA kann über eine Blutuntersuchung, also eine flüssigen Biopsie, nachgewiesen und untersucht werden. Ein übergeordneter Begriff für ctDNA ist die zirkulierende Nukleinsäure aus Blut und Serum (engl. circulatic nucleic acids in plasma and serum, CNAPS). Schon 1948 wurde erstmals DNA im Blut nachgewiesen [1], 1977 auch direkt im Blut von Krebskranken [2]. 1994 konnte dann der Beweis erbracht werden, dass diese DNA-Fragmente typische Krebsmutationen aufwiesen und somit tatsächlich aus Krebszellen stammten [3, 4]. Zur ersten praktischen Anwendung gelangte die zirkulierende DNA zunächst auf dem Gebiet der nicht-invasiven Pränataldiagnostik. Der Nachweis von Bruchstücken des Y-Chromosoms im Blut von Schwangeren mit männlichen Embryos [5] markiert den Startpunkt zur Entwicklung kommerzieller Test für die humane Diagnostik. Die Verfeinerung dieser Methode, erlaubt nun zum Beispiel die Diagnose von Trisomien, wie dem Down-Syndrom beim Embryo aus dem mütterlichen Blut. Random Massively Parallel Sequencing (rMPS) ist dabei die bisher am umfassendsten validierte und weltweit am häufigsten in der klinischen Routine eingesetzte Methode. Ein Grund für den bevorzugten Einsatz in der klinischen Routine ist deren hohe Zuverlässigkeit. Nimmt man exemplarisch die aktuellen Daten der Firma Sequenom mit 250 000 Analysen aus der Routineanwendung, liegt die Ausfallrate derzeit bei 0,9 % [6]. Beim PraenaTesth der deutschen Firma LifeCodexx beträgt die Ausfallrate derzeit sogar nur 0,6 % [7]. Zirkulierende Nukleinsäuren bergen neben ihrem Einsatz in der nicht-invasiven Pränataldiagnostik ein riesiges Potential für die onkologische Diagnostik. Eine flüssige Biopsie lässt sich, im Gegensatz zur Standardbiopsie, leichter und risikoärmer durchführen. Der Tumor kann mit der aus dem Blut der Patienten isolierten Mengen an Erbmaterial, schnell und sicher charakterisiert werden (E Abb. 1). In den vergangenen zehn Jahren wurden immer sensitivere Techniken zur quantitativen Analyse geringer DNA-Mengen entwickelt. Die Onkologen Bert Vogelstein und Kenneth Kinzler von der Johns Hopkins University entwickelten 2003 eine hochsensitive Amplifikationstechnik. Dabei lässt sich ctDNA im Blut sogar noch nachweisen, wenn das Verhältnis von DNA aus gesunden Zellen und ctDNA 10.000:1 beträgt [8, 9]. Magnetische Partikel binden dabei spezifisch an frei zirkulierende DNA und ermöglichen deren Isolierung. Tumor-DNA weist im Vergleich zur Wildtyp-DNA ein verändertes epigenetisches Muster auf. Es wird eine allgemeine Abnahme der Methylierung in Tumor-DNA beobachtet. Eine Ausnahme bilden Promotorregionen von spezifischen Genen, die hypermethyliert sind [10, 11, 12]. Dies kann dazu führen, dass ein Tumorsuppressorgen zwar unbeschädigt vorhanden ist, durch die epigenetische DZKF 2-2015 7 STANDPUNKT Regulation über die Zellteilung hinweg allerdings dauerhaft nicht mehr exprimiert wird. Erste Medikamente, die am Epigenom der Krebszellen angreifen, werden seit einigen Jahren bei manchen Blutkrebs-Arten eingesetzt [13]. Um die Tumor-DNA genauer untersuchen zu können muss sie in der Blutprobe von der Gesamt-DNA abgetrennt werden. Der Anteil der Tumor-DNA an der im Blut vorkommenden Gesamt-DNA variiert von 0,01 % bis 93 % [14, 15]. Tumor-spezifische Anomalien, wie beispielsweise Mutationen im KRAS oder EGFR-Gen können durch gezielte Ansätze mit einer Sensitivität bis zu 0,01 % [12] nachgewiesen werden. Die Nachweistechniken umfassen [10, 15, 16]: I allelenspezifische Amplifikationstechnologie (engl. Amplification refractory mutation system, ARMS) I Polymerasekettenreaktion (z.B. dPCR, qPCR, RFLPPCR, MASA PCR) I BEAMing (engl. Beads, Emulsion, Amplification, Magnetics) I CAPP-Seq (engl. Cancer personalized profiling by deep sequencing) I TAm-Seq (engl. Tagged-amplicon deep sequencing) Alternativ zu den gezielten Ansätzen können auch ganzheitliche (Genome-)Analysen durchgeführt werden. Diese sollen im Rahmen der unspezifischen Tumortestierung alle vorhandenen Mutationen erfassen. Bekannte Verfahren mit einer Empfindlichkeit von 1 bis 10 % sind: I Sequenzierung des ganzen Genoms I Sequenzierung des Exoms I Komplett-Methylom Sequenzierung I PARE (engl. Personalized analysis of rearranged ends) [10] Klare Vorteile für flüssige Biopsie Bei der flüssigen Biopsie erhöht sich die Chance den Krebs in seiner Gesamtheit zu diagnostizieren. Wird bei einer klassischen Biopsie immer nur eine winzig kleine Gewebeprobe entnommen besteht schlicht die Möglichkeit das eigentliche Tumorgewebe zu verfehlen. Im Blut dagegen finden sich DNA-Spuren aus ganz unterschiedlichen Teilen des Krebsherds. In Kombination mit den entsprechenden Datenauswertungen kann man neben einer detaillierten Tumorcharakterisierung eine auf die Tumorcharakteristik abgestimmte und für jeden Patienten individuell optimierte Therapie ausarbeiten. Die flüssige Biopsie eignet sich auch zur Erfolgskontrolle im weiteren Therapieverlauf. Vermeidung redundanter Chemotherapie oder Bestrahlung Wenn nach erfolgter operativer Entfernung des Tumorgewebes keine neue Tumor-DNA im Blut nachweis- 8 DZKF 2-2015 Anwendungsbereich Anwendung Aktueller Forschungsstand Diagnose Früherkennung Die Etablierung wird unterstützt durch einen expandierenden Datensatz von Tumor-assoziierten Mutationen und die Entwicklung von hochempfindlichen Genomanalysetechniken. Behandlungsplanung Identifizierung von Resistenzen Forschungen belegen eine inverse Beziehung zwischen der gemessenen Höhe der resistenzassoziierten Mutationen in der ctDNA und dem Therapieerfolg [17,18]. Evaluierung der Medikation Flüssige Biopsien können, im Vergleich zu den bisher eingesetzten konventionellen Methoden, die Entstehung von resistenten Genen bis zu zehn Monate früher erkennen [19,20]. Überwachung der Tumordynamik unter Medikation Flüssige Biopsien erlauben die Überwachung der Entstehung von Resistenzmutationen während der Behandlung in Echtzeit [21,22]. Beurteilung der Tumorprogression Studien zeigen eine signifikante Korrelation zwischen der Tumorlast und der Anzahl an tumorspezifischen genetischen Defekten in der ctDNA [23,24]. Früherkennung von Anzeichen eines Rückfalls Es existieren Hinweise auf einen Zusammenhang zwischen Rezidiv und Wiederauftreten von bestimmten Tumormutationen [23,25]. Prognose Tab. 1: Anwendung von CNAPS in der Onkologie bar ist, kann man dem Patienten aufwendige und mit starken Einschränkungen der Lebensqualität einhergehende Chemo- oder Radiotherapien ersparen. Im Jahr 2007 [9] konnte der Beweis erbracht werden, dass am ctDNA-Spiegel des Patienten abgelesen werden kann, wie erfolgreich eine Operation verlaufen ist und ob eine Chemotherapie im Anschluss nötig ist, um noch verbliebene Krebszellen zu eliminieren. Bei 18 Unterleibskrebs-Patienten, sank der ctDNA-Spiegel um 99 % ab, aber in den überwiegenden Fällen nicht ganz auf null. Mit Ausnahme eines Falls war in dieser Gruppe die Erkrankung rezidiv. Anders bei der Gruppe, bei der keine ctDNA mehr gefunden worden war: Alle Patienten dieser Gruppe blieben krebsfrei. Mögliche Anwendung von CNAPS in der Onkologie sind in E Tabelle 1 dargestellt. Vision: Für jeden Tumor die passgenaue Therapie Viele Rezidive bei Tumoren gehen auf die Entwicklung von Resistenzen zurück, welche im Blut verfolgbar sind. Eine regelmäßige Untersuchung auf Tumor-DNA im Blut bietet sich somit auch zur Langzeitkontrolle an. Bei Anzeichen für eine Resistenzentwicklung kann der behandelnde Arzt frühzeitig und zielgerichtet die Behandlung anpassen, um die Heilungschancen der Patienten zu erhöhen. Bei allem Optimismus stößt in einigen Fällen auch die flüssige Biopsie an ihre Grenzen. Probleme gibt es zum Beispiel bei Hirntumoren, vermutlich aufgrund der BlutHirn-Schranke. Diese verhindert, dass im Falle eines Hirntumors DNA in den Blutkreislauf eingespeist wird. Ebenso ist es momentan schwierig, Krebs im Frühstadium zuverlässig und reproduzierbar nachzuweisen. Fazit Die Empfindlichkeit der diagnostischen Tests wird sich mit der Weiterentwicklung der derzeit angewandten Methoden weiter steigern. Intensive Grundlagenforschung ermöglicht die kontinuierliche Übertragung der Erkenntnisse in die klinische Praxis. Die in den Beispielen genannten Anwendungsmöglichkeiten bei bereits diagnostizierten Tumoren werden sich schon in wenigen Jahren auch im Krankenhausalltag umsetzen lassen. STANDPUNKT LITERATUR 1. Mandel P, Metais P (1947) Les acides nucleiques du plasma sanguin l“homme. C R Seances Soc Biol Fil 142: 241–243. 2. Leon SA, Shapiro B, Sklaroff DM, Yaros M J (1977) Free DNA in the serum of cancer patients and the effect of therapy. Cancer Res 37: 646–650. 3. Vasioukhin V, et al (1994) Point mutations of the N-ras gene in the blood plasma DNA of patients with myelodysplastic syndrome or acute myelogenous leukaemia. Br J Haematol 86: 774–779. 4. Sorenson GD, et al (1994) Soluble normal and mutated DNA sequences from single-copy genes in human blood. Cancer Epidemiol Biomarkers Prev 3: 67–71. 5. Lo YMD, et al (1997) Presence of fetal DNA in maternal plasma and serum. Lancet 350: 485–487. 6. https://laboratories.sequenom.com/providers/maternit21-plus 7. http://lifecodexx.com/fuer-aerzte/methode-technik 8. Dressman et al (2003) Transforming single DNA molecules into fluorescent magnetic particles for detection and enumeration of genetic variations. Proc Natl Acad Sci USA 100: 8817–8822. 9. Diehl F, et al (2008) Circulating mutant DNA to assess tumor dynamics. Nature Med 14: 985–990. 10. Heitzer E, Ulz P, Geigl JB (2015) Circulating tumor DNA as a liquid biopsy for cancer. Clin Chem 61(1): 112–123. 11. Chan KC, et al (2013) Noninvasive detection of cancer-associated genomewide hypomethylation and copy number aberrations by plasma DNA bisulfite sequencing. Proc Natl Acad Sci USA 110(47): 18761–18768. 12. Lecomte T, et al (2002) Detection of free-circulating tumor-associated DNA in plasma of colorectal cancer patients and its association with prognosis. Int J Cancer 100(5): 542–548. 13. Kaiser J (2010) Epigenetic drugs take on cancer. Science 330(6004): 576–578. 14. Diaz LA Jr, Bardelli A (2014) Liquid biopsies: genotyping circulating tumor DNA. J Clin Oncol. 32(6): 579–586. 15. Crowley E, Di Nicolantonio F, Loupakis F, Bardelli A (2013) Liquid biopsy: monitoring cancer-genetics in the blood. Nat Rev Clin Oncol. 10(8): 472–484. 16. Wittenberger T, et al (2014) DNA methylation markers for early detection of women“s cancer: promise and challenges. Epigenomics 6(3): 311327. 17. Spindler KL, et al (2012) Quantitative cell-free DNA, KRAS, and BRAF mutations in plasma from patients with metastatic colorectal cancer during treatment with cetuximab and irinotecan. Clin Cancer Res 18: 1177–1185. 18. Spindler KL, et al (2013) Phase II trial of temsirolimus alone and in combination with irinotecan for KRAS mutant metastatic colorectal cancer: outcome and results of KRAS mutational analysis in plasma. Acta Oncol 52: 963–970. 19. Diaz LA Jr, et al (2012) The molecular evolution of acquired resistance to targeted EGFR blockade in colorectal cancers. Nature 486: 537–540. 20. Misale S, et al (2012). Emergence of KRAS mutations and acquired resistance to anti-EGFR therapy in colorectal cancer. Nature 486: 532–536. 21. Mohan S, et al (2014) Changes in colorectal carcinoma genomes under anti-EGFR therapy identified by wholegenome plasma DNA sequencing. PLoS Genetic: 10:e1004271. 22. Valtorta E, et al (2013) KRAS gene amplification in colorectal cancer and impact on response to EGFR-targeted therapy. Int J Cancer 133: 1259–65. 23. Diehl F, et al (2008) Circulating mutant DNA to assess tumor dynamics. Nat. Med. 14: 985–990. 24. Wang JY, et al (2004) Molecular detection of APC, K-ras, and p53 mutations in the serum of colorectal cancer patients as circulating biomarkers. World J Surg 28: 721–726. 25. Frattini M, et al (2008) Quantitative and qualitative characterization of plasma DNA identifies primary and recurrent colorectal cancer. Cancer Lett 263: 170–181. DR. HELGE SCHNERR Leiter Marketing & Communications GATC Biotech AG Jakob-Stadler-Platz 7 78467 Konstanz Tel.: 07531-8160-4154 Fax: 07531-8160-81 [email protected] DR. HELGE SCHNERR