Chancen und Grenzen der liquid biopsy für die
Werbung

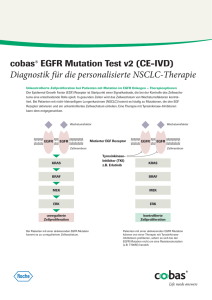
731 Genanalyse Chancen und Grenzen der liquid biopsy für die Tumordiagnostik STEFFEN DIETZ 1 , ANJA RIEDIGER 1 , MICHAEL THOMAS 2 , UWE SCHIRMER 1 , HOLGER SÜLTMANN 1 1 AG KREBSGENOMFORSCHUNG, DEUTSCHES KREBSFORSCHUNGSZENTRUM (DKFZ) UND NATIONALES CENTRUM FÜR TUMORERKRANKUNGEN (NCT), HEIDELBERG 2 ABTEILUNG ONKOLOGIE INNERE MEDIZIN, THORAXKLINIK, HEIDELBERG Circulating DNA released from cancerous tissues has been found to harbour tumour-associated alterations and to represent the molecular composition of the tumour. Recent advances in technologies, especially in next-generation sequencing, enable the analysis of low amounts of circulating DNA from body fluids. These “liquid biopsies” allow tumour genotyping and therapy monitoring without invasive procedures and offer the potential for early cancer detection. wegen der großen molekularen Heterogenität vieler Krebserkrankungen nicht immer repräsentativ für den ganzen Tumor ist: So offenbarten erste Sequenzierungen von mehreren Arealen eines Tumors, dass die Zahl der Mutationen nicht nur sehr viel größer ist als erwartet, sondern dass auch innerhalb eines Tumors sowie zwischen Primärtumor und Metastasen eine große molekulare Heterogenität vorliegt [1]. Oft kann eine Biopsie auch aus ethischen Gründen nicht entnommen werden. Schließlich sind auch die Kosten von Gewebeentnahmen und immunhistochemischen Analysen nicht unerheblich. Liquid biopsy zur Tumordiagnostik DOI: 10.1007/s12268-015-0643-2 © Springer-Verlag 2015 le. Aus diesem Grund zählt die Genanalyse von entnommenem Tumorgewebe mittlerweile zur Routineuntersuchung in der klinischen Onkologie. Die molekulare Analyse von Tumorgewebe hat jedoch auch Limitationen: Chirurgische Maßnahmen zur Tumorentfernung oder Biopsie-Entnahmen stellen invasive Eingriffe mit nennenswerten Risiken für den Patienten dar. Oftmals kann nur ein kleines Gewebestück gewonnen werden, das Eine Alternative zur Gewebeentnahme könnte die Messung von Tumorparametern in Körperflüssigkeiten (liquid biopsy) wie Blut, Urin oder Speichel darstellen. Da durch verschiedene Prozesse Zellen und Moleküle aus Geweben in den Blutkreislauf abgegeben werden, sind auch krankheitsassoziierte Proteine sowie DNA und RNA im Blut zu finden. Die Messung von frei zirkulierender DNA wird in der Pränataldiagnostik bereits routinemäßig verwendet. Bei Krebserkrankungen können zirkulierende Tumorzellen (CTCs) und freie Tumor-DNA (ctDNA) aus dem Primärtumor Nachweisgrenze Durchsatz Datenanalyse Informationsgehalt Kosten Biomarkeridentifizierung Sanger-Sequencing > 15% gering einfach niedrig gering nein Hochdurchsatzsequenzierung 1% hoch komplex hoch hoch ja Quantitative PCR 1% gering einfach niedrig gering nein BEAMing < 0,1% hoch moderat moderat moderat nein Digitale PCR < 0,1% moderat einfach niedrig gering nein Panel-Seq 0,02% gering moderat niedrig moderat nein ó Durch die Hochdurchsatzsequenzierung zur Analyse von Krebserkrankungen wurden in den letzten Jahren viele Mutationen gefunden, die mit der Tumorentstehung und -progression in Verbindung stehen. Die molekulare Ausstattung eines Tumors gibt somit wichtige diagnostische Informationen und spielt bei der Verwendung zielgerichteter individueller Therapien sowie bei der Einschätzung des Therapieerfolgs eine zentrale Rol- Spezifität ˚ Abb. 1: Geeignete Technologien zur Analyse von ctDNA im Vergleich. BIOspektrum | 07.15 | 21. Jahrgang 732 W I S S ENS CHAFT · S PECIA L : M OLE KULARE DIAG NO STI K A 0,6 Anwendung der liquid biopsy bei Lungenkrebs B Erlotinib EGFR L858R Mutation (FAM) Allelfrequenz (%) EGFR T790M 0,4 0,2 0,0 0 3 6 Zeit (Monate) 9 Wildtyp (VIC) ˚ Abb. 2: A, Analyse von ctDNA zur Überwachung des Therapieverlaufs beim nicht-kleinzelligen Bronchialkarzinom vor, während und nach Behandlung mit Erlotinib. Die mittels digitaler PCR ermittelte Zahl von Aktivierungs- (L858R) bzw. Resistenzmutationen (T790M) lässt Rückschlüsse auf das Therapieansprechen zu. B, Exemplarische Darstellung der Ergebnisse einer digitalen PCR: Positive Reaktionen für Mutation (blau) und Wildtyp (rot) sowie negative Reaktionen (gelb) werden anhand unterschiedlicher Fluoreszenzfarbstoffe (FAM, VIC, ROX) getrennt analysiert, wodurch sowohl die absolute Kopienzahl als auch Mutationshäufigkeit bestimmt werden können. oder aus Metastasen im Blutkreislauf nachgewiesen werden. Art und Anzahl von Mutationen spiegeln dabei die molekulare Heterogenität von primären und sekundären Tumoren bei einem Patienten wider. Da die Blutabnahme außerdem einen relativ gering invasiven Eingriff darstellt, kann zudem in regelmäßigen Abständen über die liquid biopsy anhand der tumorspezifischen DNA eine Bestimmung der Tumorlast durchgeführt werden: So wurde gezeigt, dass die Mutationshäufigkeit in seriell entnommenen liquid biopsy-Proben eines Patienten Aussagen über das Ansprechen auf eine Therapie und das Wiederauftreten des Tumors erlaubt [2]. In einzelnen Fällen war das erneute Tumorwachstum bereits mehrere Monate vor dem Nachweis durch bildge bende diagnostische Methoden zu erkennen [3]. Digitale PCR und Hochdurchsatzsequenzierung zum Mutationsnachweis Ein limitierender Faktor für den Nachweis molekularer Veränderungen in liquid biopsies liegt in dem niedrigen Anteil Tumor-assoziierter DNA, der oft weniger als ein Prozent der gesamten zirkulierenden DNA beträgt. Technologische Fortschritte in den vergangenen Jahren brachten jedoch immer sensitivere Verfahren zum Nachweis von ctDNA hervor (Abb. 1). Vor allem die Einführung von DNA-Amplifikationstechniken, wie die digitale PCR oder die Amplifikation Bead-gebundener DNA-Fragmente (BEAMing) [4], ermöglichen die Messung von DNA-Fragmenten mit einer Sensitivität von unter 0,1 Prozent. Das Prinzip der digitalen PCR ist es, die Probe mit einer Vielzahl an Molekülen so stark zu verdünnen, dass einzelne DNA-Moleküle separat in einer Reaktionskammer oder einem Öltropfen (digital droplet-PCR) amplifiziert werden können. Die Auswertung erfolgt durch einfaches Auszählen aller positiven Fluoreszenzsignale nach der sondenbasierten PCR. Beim BEAMing wird die DNA zunächst an magnetische Beads hybridisiert und anschließend in einer Öl-Wasser-Emulsion amplifiziert [4]. Die PCR-basierten Methoden erfordern, dass die jeweilige Mutation bereits bekannt ist. Demgegenüber bietet die Hochdurchsatzsequenzierung den Vorteil, dass neu auftretende Mutationen (z. B. bei der Entwicklung von Resistenzen) im Therapieverlauf entdeckt werden können. Mittels Genom- oder Exomsequenzierung ist es ferner möglich, auch chromosomale Veränderungen in ctDNA von Krebspatienten nachzuweisen [2, 5]. Die selektive Sequenzierung von Genomabschnitten, die in bestimmten Tumorerkrankungen häufig mutiert vorliegen, erreicht eine höhere Sequenziertiefe und damit eine höhere Sensitivität gegenüber der genomweiten Sequenzierung. So wurde durch eine Anreicherung der 500 am häufigsten mutierten Exons im nicht-kleinzelligen Bronchialkarzinom (NSCLC) aus ctDNA (Panel-Sequenzierung) eine Sensitivität von 0,02 Prozent bei der Bestimmung einzelner Mutationen erreicht [3]. Diese Methode (CAPP-Seq, cancer personalized profiling by deep sequencing) könnte zukünftig auch in der Früherkennung von Krebserkrankungen in Risikogruppen zum Einsatz kommen. Ein Beispiel für die mögliche Anwendung der liquid biopsy zum Tumormonitoring ist Lungenkrebs, die Tumorart mit der weltweit höchsten Todesrate. Die Hauptursache für die ungünstige Prognose liegt in dem hohen Anteil fortgeschrittener Tumoren (80 Prozent) zum Zeitpunkt der Diagnose. Das NSCLC macht mit ca. 85 Prozent den größten Anteil der Lungentumoren aus. NSCLC zeichnet sich durch eine hohe molekulare Diversität und Heterogenität aus und zählt zu den Tumoren mit den höchsten Mutationsraten [6], insbesondere in Genen des MAPK(mitogen-activated protein kinases)-Signalweges, wie EGFR, KRAS und BRAF (63 Prozent aller NSCLCPatienten tragen eine Mutation in mindestens einem dieser Gene). Die Mutationen im EGFR-Gen befinden sich meist in den Exons 19 (del15) und 21 (L858R), die für eine ATP-Bindedomäne des Epidermalen-Wachstumsfaktor-Rezeptors (EGFR) codieren. Dies hat eine konstitutive Aktivierung zur Folge. In der Klinik werden zur Behandlung von NSCLCPatienten mit aktivierenden Mutationen von EGFR die Inhibitoren Erlotinib oder Gefitinib eingesetzt, was zu einem längeren Überleben der Patienten führt [7]. Jedoch treten während der Therapie oftmals Resistenzmutationen insbesondere im Exon 20 (T790M) auf, welche die Affinität von EGFR gegenüber den Inhibitoren reduzieren. Hier könnte die liquid biopsy eine Schlüsselrolle beim Therapiemonitoring spielen (Abb. 2): Liquid biopsy-Analysen konnten das Auftreten der T790M-Mutation im Plasma von NSCLC-Patienten nachweisen und somit die Entstehung von Resistenzen aufzeigen [8]. Murtaza und Kollegen korrelierten hierbei das erhöhte Auftreten der Resistenzmutation T790M im Blutplasma mit der Progression der Tumorerkrankung [2]. Diese Beobachtung der inversen Korrelation zwischen der Zahl von DNA-Molekülen mit der resistenzspezifischen Mutation und dem Ansprechen gegenüber EGFR-Inhibitoren wurde in einer weiteren Publikation bestätigt [9]. Neuere EGFR-Inhibitoren ermöglichen einen verbesserten Therapieansatz, da sie zugleich gegen die aktivierenden Mutationen als auch gegen die Resistenzmutation T790M gerichtet sind. Dabei konnte bereits in der Testphase eines solchen Präparates durch Sequenzierung von ctDNA eine weitere Resistenzmutation identifiziert werden [10]: Neben ihrem Einsatz zur Therapieüberwachung geben liquid biopsies somit auch wichtige Einsichten in die molekulare Vielfalt der Resis- BIOspektrum | 07.15 | 21. Jahrgang tenzmechanismen von NSCLC. Des Weiteren erlaubten neueste Studien durch dynamische Überwachung von EGFR-Mutationen in ctDNA Rückschlüsse auf die Überlebenszeit der Patienten [11]. Ausblick Die erwähnten Beispiele zeigen, dass in der Analyse von ctDNA ein großes Potenzial für die Diagnose und das Monitoring von Tumorerkrankungen unter Therapie liegt. Ferner könnte ctDNA auch für das Verständnis der intraindividuellen Tumorheterogenität von großem Wert sein. Jedoch muss die klinische Anwendung der liquid biopsy noch in größeren Studien überprüft werden. In dieser Hinsicht besteht noch auf vielen Ebenen Optimierungsbedarf, z. B. bei der Standardisierung der verwendeten Techniken sowie Probensammlung und -lagerung, der Wahl des Extraktionsmaterials (Plasma, Serum) und -verfahrens, der Wahl der optimalen Analysetechnologie sowie die Bestimmung ihrer Sensitivität. Auf lange Sicht könnte aber die ctDNA-Analyse die Diagnose und Früherkennung von Krebserkrankungen durch konventionelle Marker ergänzen oder gar ersetzen. ó Literatur [1] de Bruin EC, McGranahan N, Mitter R et al. (2014) Spatial and temporal diversity in genomic instability processes defines lung cancer evolution. Science 346:251–256 [2] Murtaza M, Dawson SJ, Tsui DW et al. (2013) Noninvasive analysis of acquired resistance to cancer therapy by sequencing of plasma DNA. Nature 497:108–112 [3] Newman AM, Bratman SV, To J et al. (2014) An ultrasensitive method for quantitating circulating tumor DNA with broad patient coverage. Nat Med 20:548–554 [4] Li M, Diehl F, Dressman D et al. (2006) BEAMing up for detection and quantification of rare sequence variants. Nat Methods 3:95–97 [5] Leary RJ, Sausen M, Kinde I et al. (2012) Detection of chromosomal alterations in the circulation of cancer patients with whole-genome sequencing. Sci Transl Med 4:162ra154 [6] Lawrence MS, Stojanov P, Mermel CH et al. (2014) Discovery and saturation analysis of cancer genes across 21 tumour types. Nature 505:495–501 [7] Cataldo VD, Gibbons DL, Perez-Soler R et al. (2011) Treatment of non-small-cell lung cancer with erlotinib or gefitinib. N Engl J Med 364:947–955 [8] Taniguchi K, Uchida J, Nishino K et al. (2011) Quantitative detection of EGFR mutations in circulating tumor DNA derived from lung adenocarcinomas. Clin Cancer Res 17:7808–7815 [9] Sorensen BS, Wu L, Wei W et al. (2014) Monitoring of epidermal growth factor receptor tyrosine kinase inhibitor-sensitizing and resistance mutations in the plasma DNA of patients with advanced non-small cell lung cancer during treatment with erlotinib. Cancer 120:3896–3901 [10] Thress KS, Paweletz CP, Felip E et al. (2015) Acquired EGFR C797S mutation mediates resistance to AZD9291 in non-small cell lung cancer harboring EGFR T790M. Nat Med 21:560–562 [11] Mok T, Wu YL, Lee JS et al. (2015) Detection and dynamic changes of EGFR mutations from circulating tumor DNA as a predictor of survival outcomes in NSCLC patients treated with first-line intercalated erlotinib and chemotherapy. Clin Cancer Res 21:3196–3203 Korrespondenzadresse: Prof. Dr. Holger Sültmann AG Krebsgenomforschung Deutsches Krebsforschungszentrum (DKFZ) und Nationales Centrum für Tumorerkrankungen (NCT) Im Neuenheimer Feld 460 D-69120 Heidelberg Tel.: 06221-565934 [email protected] AUTOREN Steffen Dietz, Anja Riediger, Michael Thomas, Uwe Schirmer und Holger Sültmann (v. l. n. r.) Die AG Krebsgenomforschung (DKFZ/NCT Heidelberg) nutzt die Hochdurchsatztechnologien der Genomforschung zur Analyse genetischer Änderungen in Geweben und Blutproben von Krebspatienten. Ziel ist es, in Kooperation mit klinischen Partnerinstitutionen wie der Thoraxklinik Heidelberg (Prof. Dr. Michael Thomas), diese Marker in die klinische Diagnostik zu bringen. BIOspektrum | 07.15 | 21. Jahrgang