Molekulare Motoren
Werbung

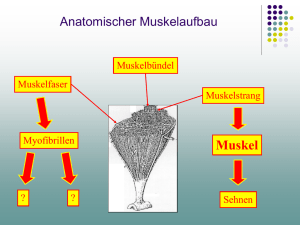
Molekulare Motoren Gliederung: 1. Aktin-Myosin Komplex - Einführung in das Muskelsystem - Aufbau der Aktinfilamente - Aufbau der Myosinstäbe - Aufbau und Funktion des Aktin-Myosin Systems - Signalübertragung - Andere Proteine - Myosin in anderen Zellarten - Experimente 2. Kinesin - Aufbau und Funktion - Experimente Molekulare Motoren können sich durch Konformationsänderungen bewegen und Arbeit leisten. Die dafür benötigte Energie entziehen sie der ATP-Hydrolyse. Sie beteiligen sich an der DNA Replikation, Protein Synthese, Änderung dessen Form, also an Bewegungen, und am Transport innerhalb der Zelle. Das Aktin-Myosin System ist für die Veränderung der Form der Zelle verantwortlich und das Kinesin für den Transport von Vesikel oder anderen Objekten anhand von Mikrotubulli. 1.1DasMuskelsystem Die Muskeln bestehen aus Muskelfasern. Diese sind riesige Zellen die aus dem Zusammenschmelzen mehrer Zellen entstanden sind. Die Kerne dieser Zellen findet man unter der Zellmembran. Hauptbestanteil dieser Zellen sind die Myofibrillen, die eine zylindrische Form haben, mit einem Diameter von 1-2 Mikrometer, die meistens genau so lang wie der Muskel selbst sind. Die dunklen und hellen Streifen die man auf sie beobachten kann, geben dem Muskel seinen Namen: Quergestreifter Muskel. Elektronenmikroskop Aufnahme eines Myofibrills Schaut man sich eine Elektronenmikroskop Aufnahme eines Myofibrills so entdeckt man Einheiten die sich immer wieder wiederholen. Sie heißen Sarkomere. Jedes Sarkomer ist etwa 2,5 Mikrometer lang und es erstreckt sich zwischen zwei Z-Scheiben. An diesen sind die Aktinfilamente befestigt die eine Art Gerüst aufbauen (I-Band). Diese werden von den Myosin-Filamenten, die eigentlichen Motoren (A-Band) zusammengezogen. Je stärker der Muskel kontrahiert desto kürzer wird das I-Band während das A-Band nicht an Länge verliert. 1.2 Aktin Das Monomere Aktin heißt globuläres G-Aktin. Es ist ein Polypeptid, das 375 Aminosäuren enthält. Es besitzt zwei paare von komplementären Bindungsstellen. (Schlüssel-Loch Prinzip). Ein Paar liegt polar, das andere seitlich, so dass die Moleküle sich zu einem verdrillten, doppelsträngigen Filament zusammenlagern können (den faserigen F-Aktin). Dieses weist pro vollständiger Windung (77 Nanometer lang und 8nm breit) 14 Monomerpaare auf. Die Polymerisation verläuft gerichtet, von der Anheftungsstelle weg (diese wird plus Pol genannt) und ist reversibel. Dabei wird ATP hydrolisiert. Sie wird durch das Pilzgift Cytochalasin B gehemmt. 1.3 Myosin Die Grundeinheit der Myosin Filamente ist das Myosinmolekül (B). Das ist ein langes doppelsträngiges Protein mit einem 135 nm langem Schwanz und zwei knollenförmigen Köpfchen. Der Schwanz besteht aus zwei identischen, umeinander gewundenen Strängen mit nahezu perfekter Alpha-HelixKonformation (A). Durch eine Störung in dieser regelmäßigen Struktur entsteht ein Scharnier zwischen einem hydrophoben, 110 nm langem Stamm und einem hydrophileren Halsabschnitt, der die Köpfchen trägt. An dieser Stelle lässt sich das Molekül durch hydrolitische Enzyme leicht spalten: in schweres (HMM oder Heavy meromyosin) und leichtes Meromyosin (LMM). Ein zweites Scharnier zwischen Kopf und Halsbereich des HMM kann ebenfalls leicht zerschnitten werden; dabei entstehen die Subfragmente S1 und S2. Myosinmoleküle lagern sich zu dicken, 1.5 bis mehr als 5 Mikrometer langen Stäben mit 15-20 nm Durchmesser zusammen. Sie legen sich dabei mit ihren hydrophoben LMM-Schwänzen aneinander und lassen die hydrophileren HMM-Stiele und Köpfchen – schraubig angeordnet- aus der Oberfläche des Stabs herausragen. Der ganze Stab ist symmetrisch aufgebaut (D). In seiner Mitte befindet sich ein 300 nm langer, nackter Abschnitt, der aus den ineinander greifenden Stielen jener Moleküle besteht, die die ersten Köpfchen auf jeder Seite stellen. Die Wechselwirkung hier ist ionischer Natur. Die Köpfchen der Myosinmoleküle bilden Haken, an die sich Aktinfilamente anhängen. Sie sind so um den Stamm angeordnet, dass jeweils 6 parallele Aktinfilamente daran binden können. Damit eine solche Struktur zustande kommt, müssen die Aktinfilamente entsprechend orientierte Bindungsstellen für die Myosinköpfchen besitzen. Das kann man ausnutzen, um Aktinfilamente in Gewebeschnitten zu identifizieren und ihre Polarität zu bestimmen. Man verwendet dazu gereinigte Myosinköpfchen, die man enzymatisch von ihren Stielen getrennt hat. Sie binden spezifisch an Aktinfilamente und „Dekorieren“ sie: Im Bild sieht man ein Aktinfilament (rot) das mit Myosinköpfchen (gelb) dekoriert wurde. Dadurch kann man die Aktin-Myosin Bindungsstellen identifizieren 1.3 Aktin-Myosin Komplex Myosinköpfchen bindet an das Aktinfilament. Die Anziehung zw. Bindungstasche und Köpfchen beruht entweder auf die Coulomb-Wechselwirkung von Partialladungen oder auf Van-der-Waals Kräfte. Wenn man sich einzelne Aktin-Myosin-Einheiten anschaut, sieht man dass jedes der 6 Aktinfilamente, die den Myosinschaft umgeben, auf seiner Oberfläche noch unbesetzte Myosinbindungsstellen hat. Deshalb können die 6 Filamente des ersten Bündels zusätzliche Myosinschäfte binden die wiederum weitere Aktinfilamente um sich scharen uns so weiter. So entsteht eine Struktur mit sechseckiger Symmetrie. 1.3.1 Bewegung Überall wo die Myosinköpfchen an Bindungsstellen eines Aktinfilaments haften, krümmen sie sich an ihren Stielen heftig nach innen und reißen das Filament zur Schaftmitte. Dann lösen sie sich wieder ab. Inzwischen haben andere Myosinköpfchen passende Bindungsstellen erreicht und ziehen das Filament nochmals. Dies setzt sich fort, solange Ca-Ionen vorhanden sind. Weil alle Aktinfilamente an beiden Enden des Myosinschafts auf gleicher weise nach innen gezogen werden, gleiten die zwei gegenüberliegenden Aktinbündel aufeinander zu und letztlich ineinander hinein. Dabei zerren sie ihre jeweiligen Verankerungspunkten näher zueinander. 1.3.2 Energieerzeugung Das freie Myosinköpfchen bindet ATP und hydrolisiert es. Der Prozess ist reversibel da die Energie im ADP und Pi gespeichert wird. Während dieser zwei Phasen kann sich das Myosinköpfchen frei entlang des Aktinfilaments bewegen. Nähert es sich aber einer AktinBindungsstelle geht es eine leichte Bindung mit dieser ein. Dabei wird das Pi frei gesetzt und das Myosinköpfchen befestigt sich an das Aktinfilament. Jetzt findet eine Konformationsänderung statt. Das Köpfchen biegt sich am Stiel und zieht das Aktin mit. Dabei wird das ADP frei gesetzt und an ein neues ATP wird gebunden und ein neuer Zyklus beginnt. Dieser ganze Prozess findet nur in der Gegenwart von Ca-Ionen statt. Wenn weder ATP noch Ca-Ionen vorhanden sind, bleiben Aktin und Myosin starr miteinander verbunden. Deshalb tritt nach dem Tod die Totenstarre ein. Jedes Myosinfilament enthält um die 500 Köpfchen und jedes von ihnen durchläuft etwa 5 solche Zyklen in einer Sekunde, bei einer raschen Muskelkontraktion. Das heißt das die Aktin und Myosinfilamente mit einer Geschwindigkeit von 15 Mikrometer/Sek aneinander vorbei gleiten. 1.4 Signalübertragung Das Signal von der Nervenzelle erzeugt auf der Zellmembran ein Aktionspotential. Dieses verbreitet sich sehr schnell in den Transversalen Tubulli, Einstülpungen der Zellmembran die jedes Myofibrill umgeben. Von diesen gelangt das Signal auf unbekannter Art und Weise zum Sarkoplasmatischen Reticulum das die Ca-Ionen frei setzt. Die Konzentration an Ca-Ionen wächst sehr schnell wird aber auch schnell wieder abgebaut und die Ionen werden im S Reticulum zurückgepumpt (30 Millisekunden) wodurch die Myofibrillen sich entspannen. Durch dieses System gelangt der Signal gleichzeitig an alle Myofibrillen. Das Tropomyosin ist ein 41 nm langes rigides Molekül, das aus zwei identische alpha-helix Strängen besteht, wovon jedes 284 Aminosäuren besitzt. Es windet sich um ein Aktinfilament und verstärkt es. Gleichzeitig verdeckt es die Myosin Bindungsstellen. Daran befestigt sich auch das Troponim. Daran befestigt sich auch das Troponim. Das ist ein Komplex aus 3 Polypeptiden : Troponin T, I und C. Es hat eine längliche Form, I und C bilden den Kopf und T den Schwanz. Das Troponin T bindet sich an dem Tropomyosin und ist für dessen Ausrichtung verantwortlich, während das Troponin I an das Aktin bindet. Zusammen verhindern sie die Wechselwirkung zw Myosin und Aktin. Das Troponin C hat 4 Bindungsstellen für Ca-Ionen. Wenn diese besetzt werden löst das Troponin C die Bindung zw. Troponin I und Aktin. Dadurch rutscht das Tropomyosin und läst die Bindungstelle zw. Aktin und Myosin frei so dass diese sich binden können. Dadurch wird die Muskelaktivität nur in der Gegenwart von Ca-Ionen möglich. 1.5 Andere Proteine Auch andere Proteine spielen eine wichtige Rolle im Sarkomer. Das alpha-actinin bindet die Aktinfilamente an der Z-Scheibe in parallelen Reihen. An der M-Linie vernetzt das myomesin die Myosinfilamente miteinander in hexagonaler Reihenfolge. Die Myosinfilamente werden durch andere Proteine stabilisiert, die sich an das Myosin befestigen. Das Titin läuft parallel zur Aktin und verbindet das Myosin mit den Z-Scheiben. 1.6 Myosin in anderen Zellarten Myosin findet man in fast allen Eukaryonten. Im Gegensatz zu Aktin das in der Natur in gleicher Form auftritt, existiert Myosin unter vielen verschiedenen Formen. Bis jetzt wurden 22 Arten von Myosin entdeckt. Sie unterscheiden sich in den meisten Fällen im Bau des Schwanzes das für verschiedene Frachten spezialisiert ist oder in der Funktion das es in der Zelle übernimmt. Im menschlichen Genom wurden bis jetzt 39 Gene gefunden die für die Bildung der verschiedenen Myosinarten zuständig sind. 1.6.1 Myosin in glatten Muskeln Die „primitivsten“ Muskelzellen, im Sinne dass sie den Nichtmuskelzellen am meisten ähneln sind die glatten Muskelzellen. Diese Muskelart trifft man z.B. in der Magen-, Darm- oder Arterienwände, überall dort wo eine langsame und lang anhaltende Kontraktion nötig ist. Sie bestehen aus spindelförmigen Zellen mit nur je einem Kern. Die Aktin-Myosin Filamente sind nach keinen genauen Muster geordnet und bilden keine Myofibrillen. Sie sind wie ein Netz über die Zelle verbreitet und an der Zellmembran befestigt. Dadurch verbinden sie auch die Zellen untereinender. Diese Anordnung ist nur ein Model da sie nicht genau bekannt ist. Es erlaubt aber der Zelle sich stärker zusammen zu ziehen als die Zellen der Quergestreiften Muskeln 1.6.2 Myosin in Nichtmuskel Zellen Obwohl es Myosin in fast allen Eukaryonten gibt, findet man feste Myosinstäbe nur in Muskelzellen. In diesen Zellen bilden die Myosinmoleküle kleinere Gebilde und das nur wenn es die Situation verlangt. Wird das Molekül nicht gebraucht so verschließen leichte Myosinketten die Aktin-Bindungsstellen der Köpfchen. Gleichzeitig binden sie sich an den Myosin-Schwanz und verkneulen dadurch das Molekül. Wird das Molekül phosphorylisiert (phosphorylated) so befreien die leichten Myosinketten den Schwanz und öffnen die AktinBindungsstellen der Köpfchen. Dann folgt eine spontane Zusammenlagerung der Moleküle zu einem Myosinstab der auf die gleiche Art und Weise wie die Myosinstäbe in Muskelzellen arbeitet. Solche Gebilde müssen z.B. bei der Zellteilung entstehen wenn der Kontraktionsring die Mutter- und Tochterzelle teilt. 1.7 Experimente Lichtmikroskop Aufnahme von Aktinfilamente Elektronenmikroskop Aufnahme von Myosinmoleküle Um sich die Wechselwirkung zw. Aktin und Myosin zu veranschaulichen befestigt man Myosinköpfchen, die man enzymatisch von ihren Stielen abgetrennt hat, auf einer Glasplatte. Darauf lässt man Aktinfilamente frei laufen. Kraftmessen mit einer Glaspinzette Für diesen Versuch braucht man eine sehr kleine Glas Pinzette mit 0,2mm Durchmesser und einer Elastizitätskonstante von 2-20 pN/mm. Die Spitze der Pinzette wird mit Hilfe von modifiziertem Myosin das wie eine Art Superkleber wirkt an einem Ende eines Aktinfilaments festgemacht. Man lässt dann das Aktinfilament über die Myosinköpfe nieder und man beobachtet wie stark die Pinzette gebogen wird. Daraus kann man dann die Kraft messen mit der die Köpfchen an das Aktin ziehen. Die Laserfallen benutzt man um Polystierenkügelchen (1mm Durchmesser) einzufangen. An denen bindet man mit Hilfe von modifiziertem Myosin die beiden Enden eines Aktinfilaments. An einer größeren Kugel befestigt man nur wenig Myosinköpfchen damit das Aktinfilament möglichst nur von einem Köpfchen bewegt wird. Mit Hilfe eines Photodioden Detektors kann man diese Bewegung im nm-Bereich messen. Die Schrittweite der Myosinköpfchen aus Quergestreiften Muskeln beträgt 10 nm während bei glatten Muskeln die Bewegung in zwei Schritte geschieht: einmal 6 nm etwas langsamer und nach 5 milisekunden 5,5 nm sehr schnell. 2.1 Aufbau eines Kinesinmoleküls Kinesin ist ein Molekulares Motor das für den Transport von Vesikeln und anderen Frachten entlang von Mikrotubulli verantwortlich ist, wie z.B. die Chromosomen Migration während der Zellteilung. Es ist ein etwa 350 Aminosäuren langes Protein. An einem Ende hat es 2 Köpfchen die an das Mikrotubule binden während sich am anderen Ende die Fracht befestigt. Es läuft immer in Richtung des +Pols des Mikrotubuls, d.h. in Richtung der Zellmembran. 2.2 Funktion Das Kinesin funktioniert über das Hand-over-Hand Prinzip, dabei ist ein Ende immer am Mikrotubulli befestigt. Es lässt nicht los bevor sich das andere Ende an der nächsten Bindungstelle befestigt hat. Schneidet man ein Ende weg so bindet das andere am Mikrotubulli, lässt aber nur sehr langsam los. Es kann sich nicht mehr bewegen. 2.3 Experimente In Laborversuchen befestigt man Kinesinmoleküle an einer Glasplatte und lässt über sie Mikrotubulli laufen. Man kann das – Ende des Mikrotubulli fluoreszent markieren und dadurch beweisen dass die Kinesinmotoren immer in Richtung des +Pols laufen. Verringert man die Dichte der Motoren auf der Glasplatte so merkt man dass ein einziger Motor das Mikrotubulli mit der gleichen Geschwindigkeit von 1 Mikrometer pro Sekunde bewegt, wie 10 oder 100 gleichzeitig. Seine Schrittweite beträgt 8 nm. In diesem Versuch verbindet man das Kinesinmolekül mit einem Kraftmäßer, das kann ein elastischer Glasstab sein oder eine optische Falle. Dadurch kann man die Kraft messen mit der das Molekül am Stab zieht. Mit einer Kraft von 5 pN leistet der Motor eine Arbeit von 40*1021 J das einem Wirkungsgrad von 40% entspricht da es bei jedem Schritt ein ATP-Molekül hydrolisiert.