Der Energiestoffwechsel eukaryotischer Zellen
Werbung
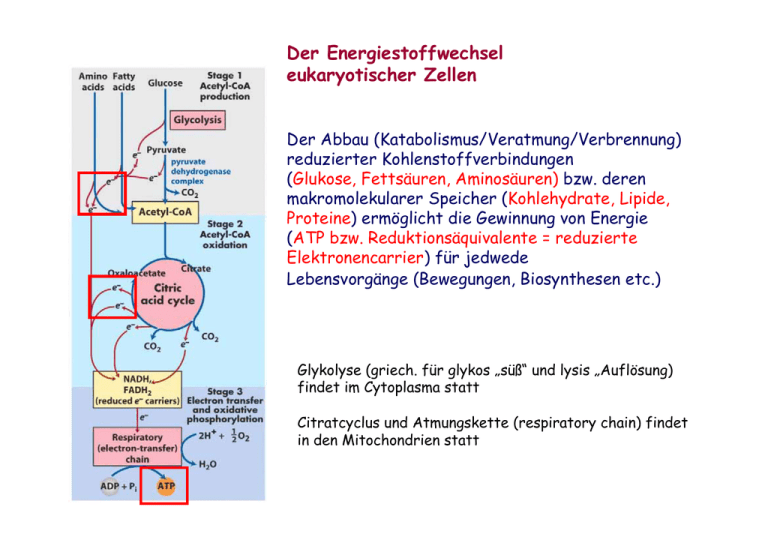
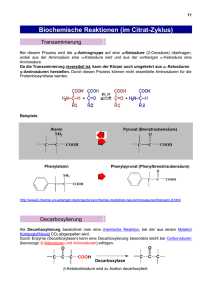
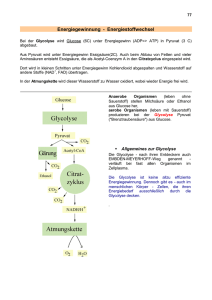
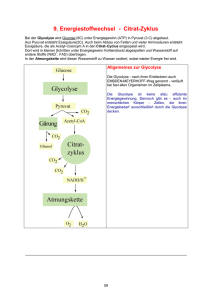
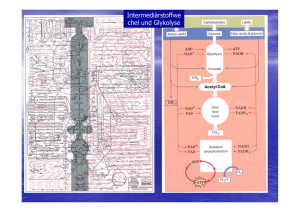
Der Energiestoffwechsel eukaryotischer Zellen Der Abbau (Katabolismus/Veratmung/Verbrennung) reduzierter Kohlenstoffverbindungen (Glukose, Fettsäuren, Aminosäuren) bzw. deren makromolekularer Speicher (Kohlehydrate, Lipide, Proteine) ermöglicht die Gewinnung von Energie (ATP bzw. Reduktionsäquivalente = reduzierte Elektronencarrier) für jedwede Lebensvorgänge (Bewegungen, Biosynthesen etc.) Glykolyse (griech. für glykos „süß“ und lysis „Auflösung) findet im Cytoplasma statt Citratcyclus und Atmungskette (respiratory chain) findet in den Mitochondrien statt Map of the major metabolic pathways in a typical cell Zur Aufrechterhaltung der Glykolyse muss Pyruvat abgebaut und NAD+ regeneriert werden Ein Stau von Pyruvat würde die Glykolyse schnell zum Erliegen bringen; außerdem ist NAD+ nur in begrenzter Menge verfügbar (Derivat des Vitamins Niacin) und muß regeneriert werden Glykolyse ist in allen Organismen hoch konserviert bis zum Pyruvat; Umsetzung des Pyruvats ist unterschiedlich Hefen u. a. Mikroorganismen Alkoholische Gärung + H+ Mikroorganismen (Lactobacillus) und tierische Zellen (Muskelzellen unter Sauerstoffmangel (Milchsäuregärung) Die alkoholische Gärung Die Milchsäure-Gärung Zur Aufrechterhaltung der Glykolyse muss Pyruvat abgebaut und NAD+ regeneriert werden Ein Stau von Pyruvat würde die Glykolyse schnell zum Erliegen bringen; außerdem ist NAD+ nur in begrenzter Menge verfügbar (Derivat des Vitamins Niacin) und muß regeneriert werden Glykolyse ist in allen Organismen hoch konserviert bis zum Pyruvat; Umsetzung des Pyruvats ist unterschiedlich + H+ ? NAD+-Regeneration In anderen Zellen unter ausreichender Sauerstoffversorgung ? Unter ausreichender Sauerstoffversorgung (aerobe Bedingungen) findet in vielen anderen Zellen (Tiere, Pflanzen) ein weiterer Abbau des C-Gerüstes des Pyruvats unter Energiegewinnung statt Pyruvat wird aus dem Cytosol in die Matrix der Mitochondrien transportiert (erfolgt durch einen Pyruvattransportprotein im Antiport mit OH-) durch den Pyruvatdehydrogenase-Komplex wird Pyruvat zu Acetyl-CoA (C2-Körper) umgesetzt und im Citratzyklus durch zweimalige Decarboxylierung vollständig „verbraucht“ Dabei entstehen weitere Reduktionsäquivalente (NADH/FADH2) NAD+ Regeneration des NAD+ erfolgt durch Atmungskette (respiratory chain) Der Pyruvat-Dehydrogenase-Komplex lokalisiert in der Matrix der Mitochondrien Der Pyruvat-Dehydrogenase-Komplex spaltet CO2 ab und oxydiert Pyruvat zu Acetat, welches auf CoenzymA (CoA-SH) übertragen wird; Reaktion besteht aus Teilreaktionen, die durch einen Multienzymkomplex katalysiert werden Pyruvatdehydrogenase E1 Transacetylase E2 Der Pyruvat-Dehydrogenase-Komplex und CoenzymA (CoA) Verlust eines weiteren C-Atoms aus dem C6Körper Glucose zum Zweck der EnergieGewinnung (NADH+H+) CoenzymA-SH (CoA) Kopplung von Acetylgruppe an CoS-SH über eine Thioesterbindung Der Pyruvat-Dehydrogenase-Komplex und die prosthetischen Gruppen Thiaminpyrophosphat (TPP) und Liponamid ist eine prosthetische Gruppe der Pyruvatdehydrogenase-Komponente (E1) des Komplexes aus drei Enzymen Thiazolring kann Elektronen aufnehmen und abgeben Liponamid is eine prosthetische Gruppe der Transacetylase (E2) und ist kovalent mit einem Lysinrest des Enzyms verbunden; Disulfidbindung kann in S-S-Form (oxydiert) oder in SH SH-Form (reduziert) vorliegen und so Elektronen übertragen Der Pyruvat-Dehydrogenase-Komplex Enzym E1 (Pyruvatdehydrogenase) bindet Pyruvat an Thiaminpyrophosphat unter Abspaltung von CO2, der Acetylrest bleibt an TPP gebunden E1 führt auch den Transfer der Acetylgruppe und von 2 Elektronen auf den Lipoamidarm von Enzym 2 aus (die Disulfidbindung im Liponamidarm wird dabei reduziert) Übertragung der Acetylgruppe von TPP (Enzym 1) auf Liponamidarm (Enzym 2) durch die Pyruvatdehydrogenase-Komponente (Enzym 1) des Pyruvatdehydrogenase-Komplexes Der Pyruvat-Dehydrogenase-Komplex Enzym E1 (Pyruvatdehydrogenase) bindet Pyruvat an Thiaminpyrophosphat unter Abspaltung von CO2, der Acetylrest bleibt an TPP gebunden E1 führt auch den Transfer der Acetylgruppe und von 2 Elektronen auf den Liponamidarm von Enzym 2 aus (die Disulfidbindung im Liponamidarm wird dabei reduziert) Enzym 2 (Transacetylase) katalysiert die Übertragung des Acetylrestes von der SH-Gruppe des Liponamids auf die SH-Gruppe von Coenzym A (CoA-SH) Übertragung der Acetylgruppe von Liponamidarm (Enzym 2) auf CoA-SH durch die Transacetylase-Komponente (Enzym 2) des Pyruvatdehydrogenase-Komplexes Der Pyruvat-Dehydrogenase-Komplex Enzym E1 (Pyruvatdehydrogenase) bindet Pyruvat an Thiaminpyrophosphat unter Abspaltung von CO2, der Acetylrest bleibt an TPP gebunden E1 führt auch den Transfer der Acetylgruppe und von 2 Elektronen auf den Lipoamidarm von Enzym 2 aus (die Disulfidbindung im Liponamidarm wird dabei reduziert) Enzym 2 (Transacetylase) katalysiert die Übertragung des Acetylrestes von der SH-Gruppe des Liponamids auf die SH-Gruppe von Coenzym A (CoA-SH) Enzym 3 (Dehydrogenase) katalysiert die Übertragung von 2 H und 2 Elektronen auf die prosthetische Gruppe FAD am Enzym 3; d.h. Liponamid wird wieder oxydiert (recycelt) und FAD zu FADH2 reduziert; Danach reduziert Enzym 3 NAD+ zu NADH+H+ und recycelt somit oxydiertes FAD Reduziertes NADH+H+ wird dann in der Atmungskette zur ATP-Gewinnung genutzt Danach startet eine neue Umsetzung von Pyruvat an E1 Regeneration des oxydierten Liponamidarms an E2 durch gleichzeitige Reduktion von FAD an E3 (wird katalysiert durch die Dehydrogenase-Komponente des Pyruvatdehydrogenase-Komplexes Danach Re-Oxidation von FADH2 zu FAD unter Gewinnung des Reduktionsäquivalentes NADH+H+ und somit vollständige Wiederherstellung des Ausgangszustandes des Pyruvatdehydrogenase-Komplexes (E1/E2/E3) Unter ausreichender Sauerstoffversorgung (aerobe Bedingungen) findet in vielen anderen Zellen (Tiere, Pflanzen) ein weiterer Abbau des C-Gerüstes des Pyruvats unter Energiegewinnung statt Pyruvat wird aus dem Cytosol in die Matrix der Mitochondrien transportiert (erfolgt durch einen Pyruvattransportprotein im Antiport mit OH-) durch den Pyruvatdehydrogenase-Komplex wird Pyruvat zu Acetyl-CoA (C2-Körper) umgesetzt und im Citratzyklus durch zweimalige Decarboxylierung vollständig „verbraucht“ Dabei entstehen weitere Reduktionsäquivalente (NADH/FADH2) NAD+ Regeneration des NAD+ erfolgt durch Atmungskette (respiratory chain) Der Zitronensäurezyklus/Tricarbonsäure (TCA)-Zyklus/Krebs-Zyklus Citratzyklus Citrat = Salz der Zitronensäure Oxidation von C2-Einheiten (Decarboxylierungen) unter Gewinn von GTP und energiereichen Elektronen in Form von Reduktionsäquivalenten Die Citratsynthase-Reaktion Anlagerung von AcCoA an Oxalacetat und Bildung von Citrat ist eine 2-Stufenreaktion; zuerst erfolgt Bildung von Citryl-CoA (Kondensation) und nachfolgend eine Hydrolyse, in der CoA und Citrat frei werden energetisch günstig, da bei Spaltung der Thioesterbindung in AcCoA Energie frei wird und partiell in Citrat gespeichert bleibt Die Citratsynthase-Reaktion His274 (aktives Zentrum der Citratsynthase) überträgt ein Proton auf Carbonylsauerstoff des AcCoA; dadurch wird ein Methylproton am AcCoA gelockert und auf Asp 375 (aktives Zentrum der Citratsynthase) Übertragen Oxalacetat wird aktiviert durch Übertragung eines Protons von His320 (aktives Zentrum der Citratsynthase) auf seinen Carbonylkohlenstoff Enolbindung ermöglicht Knüpfung einer C-C Bindung zwischen Carbonylkohlenstoff des Oxalacetats und AcCoA (ergibt Citryl-CoA) Die Isocitratbildung (Aconitase-Reaktion) Energiegewinnung im Citratzyklus wird ermöglicht über Abbau des reduzierten C-Gerüsts von Citrat und Regeneration des Oxalacetats Regeneration des Oxalacetats erfolgt durch zweimalige Decarboxylierung (C6-C5-C4) und Oxidation des C-Gerüstes Die Decarboxylierung von Citrat ist stereochemisch ungünstig, da tertiäre OH-Gruppe ungünstig angeordnet ist Um den oxidativen Abbau des C-Gerüstes zur Energiegewinnung zu ermöglichen, wird aus Citrat Isocitrat gebildet Aconitase ermöglicht die Isomerisierungsreaktion durch Dehydratisierung (Wasserabspaltung) und darauffolgende Hydratisierung (Wasseranlagerung) Die Isocitratdehydrogenase-Reaktion Die Isocitratdehydrogenasereaktion ist die erste von vier Dehydrogenase-Reaktionen, in denen das C-gerüst des Citrates schrittweise oxidiert wird und reduzierte (energiereiche) Reduktionsäquivalente geschaffen werden 1. Schritt: oxidative Decarboxylierung des Isocitrats zum α-Ketoglutarat (2-Oxoglutarat); Elektronen werden auf NAD+(ox.) übertragen, welches dadurch reduziert wird zu NADH+H+ Die α-Ketoglutaratdehydrogenase-Reaktion Die α-Ketoglutaratdehydrogenasereaktion ist die zweite von vier Dehydrogenase-Reaktionen, in denen das C-gerüst des Citrates schrittweise oxidiert wird und reduzierte (energiereiche) Reduktionsäquivalente geschaffen werden 2. Schritt: oxidative Decarboxylierung vom α-Ketoglutarat (2-Oxoglutarat) zu Succinyl-CoA; Elektronen werden auf NAD+(ox.) übertragen, welches dadurch reduziert wird zu NADH enzymatischer Mechanismus und Struktur des Enzymkomplexes fast identisch mit Pyruvatdehydrogenase-Komplex (Schaffung von AcCoA) Die Succinyl-CoA-Synthetase-Reaktion Die Succinyl-CoA-Synthetase-Reaktion katalysiert die Bildung von GTP (endergone Reaktion !) Dies wird ermöglicht durch den Energiegehalt der Thioesterbindung, der höher liegt als die zur GTP-Bildung benötigte Energie (Gesamtreaktion exergon) 2-Schritt-Reaktion: im 1. Schritt wird CoA gegen P ausgetauscht und es entsteht Succinylphosphat (Umwandlung der energiereichen Thioesterbindung in ein hohes PhosphatgruppenÜbertragunspotential (Succinylphosphat ist energiereich und hat damit ein hohes Phosphatgruppenübertragungspotential) Danach wird im 2. Schritt das Phosphat auf GDP übertragen und es entsteht der C4-Körper Succinat, der weiter oxidiert wird zu Oxalacetat (Recycling); dabei entstehen weitere 2 Reduktionsäquivalente Die Regeneration von Oxalacetat durch Oxidation von Succinat Succinat-DH Fumarase Malat-DH Die Succinatdehydrogenasereaktion ist die dritte und die Malatdehydrogenasereaktion die vierte von vier Dehydrogenasereaktionen, in denen das C-gerüst des Citrates schrittweise oxidiert wird und reduzierte (energiereiche) Reduktionsäquivalente geschaffen werden 3. Schritt: Oxidation von Succinat zu Fumarat Elektronen werden auf FAD(ox.) übertragen, welches dadurch reduziert wird zu FADH2 4. Schritt: Oxidation von Malat zu Oxalacetat Elektronen werden auf NAD+(ox.) übertragen, welches dadurch reduziert wird zu NADH+H+ Die Bilanz des Citratzyklus pro Mol AcCoA Metabolon Die Regulation des Citratzyklus erfolgt auf verschiedenen Ebenen Regulation der PDH erfolgt sowohl allosterisch durch Endprodukte (NADH+H+ und AcCoA) als auch durch reversible Phosphorylierung/ Dephosphorylierung Regulation des Citratzyklus insbesondere durch Ausgangsstoffe bzw. Endprodukte der beiden Decarboxylierungsreaktionen Der Citratzyklus ist Ausgangspunkt vieler Biosynthesen Der Energiestoffwechsel eukaryotischer Zellen Der Abbau (Katabolismus/Veratmung/Verbrennung) reduzierter Kohlenstoffverbindungen (Glukose, Fettsäuren, Aminosäuren) bzw. deren makromolekularer Speicher (Kohlehydrate, Lipide, Proteine) ermöglicht die Gewinnung von Energie (ATP bzw. Reduktionsäquivalente = reduzierte Elektronencarrier) für jedwede Lebensvorgänge (Bewegungen, Biosynthesen etc.) Glykolyse (griech. für glykos „süß“ und lysis „Auflösung) findet im Cytoplasma statt Citratcyclus und Atmungskette (respiratory chain) findet in den Mitochondrien statt