Intermediärstoffwe chel und Glykolyse
Werbung
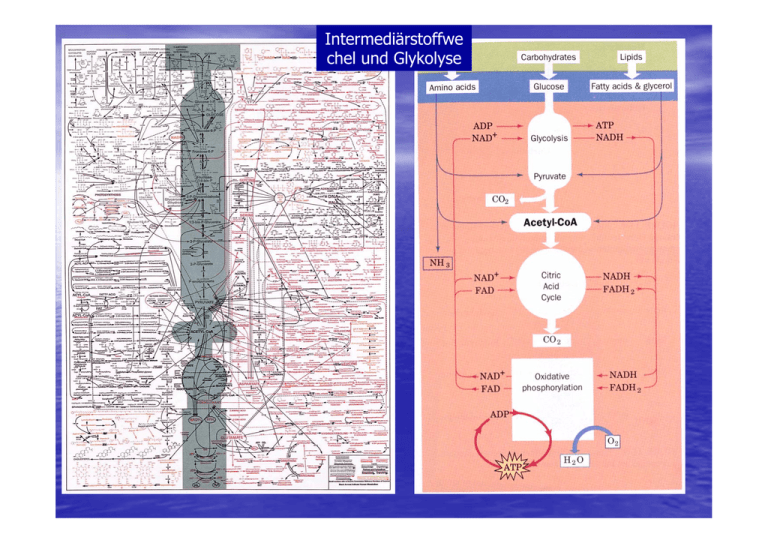
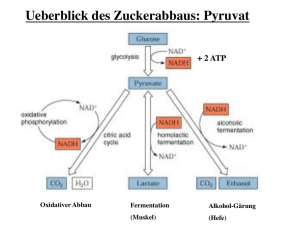
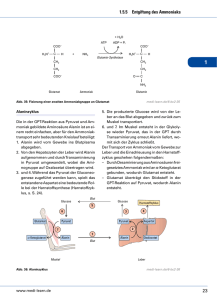
Intermediärstoffwe chel und Glykolyse 1. Phosphorylierung (Hexokinase) 2. Isomerisierung (P-Glc. Isomerase) Keto-Enol 3. Phosphorylierung (P-Fructokinase) 4. Fragmentierung (Aldolase) 5. Energieliefernde Oxidation (Glyceralaldehyd-3-P Dehydrogenase) 5. Isomerisierung (TIM) 6. Dehydratation zu P-Enolat (Enolase) 7. Energieliefernde ATP-Bildung (Mg2+) • Glykolyse zu Pyruvat - Milchsäuregärung - Decarboxylierung zu Acetyl-CoA - Ohne Citratcyclus – alkoholische Gärung - Mit Citratcyclus - „ADP-olyse“ von 1,3-BPG „induced-fit“ bei Hexokinase Glycose-Fructose-Isomerisierung Aktives Zentrum Phosphoglucose Isomerase: Lys als Base Glu als Säure Allosterische Inhibition (Regulation) bei Phosphofructokinase Retro-Aldol-Reaktion Keto-Enol zu Methylglyoxal (niedrige ∆G#) P-Eliminierung (höhere ∆G#) axial equatorial Stabilisierung equatorialer Form und Schutz vor Nebenreaktionen mittels Schleife Triosephosphat-Isomerase (TIM) Synthese von Phosphoenolpyruvat (PEP) aus 2-P-Glycerat Phosphorglyceratmutase über Diphosphoglycerat als Intermediat 2,3 BPG erniedrigt (erhöht) Affinität von Hb zu O2 (CO2) und ermöglicht erst dadurch Gastausch in venösem Blut Pyruvatkinase 2,3-BPG akkumuliert ! Milchsäuregärung LactatDH Glyceral-3P DH Muskel Synthese von Acetyl-CoA aus Pyruvat für Citratcyclus Pyruvat Dehydrogenase Multienzym Dihydrolipolyl-Transacetylase in Pyruvat-Dehydrogenase Multienzym Komplex (a) (a): Dihydrolipolyl-Transacetylase (b): Pyruvat-Dehydrogenase (c): (a) + (b) = 60 Untereinheiten Dihydrolipolyl-Transacetylase (a): 8 Trimere (24 Untereinheiten) (b): 1 Struktur eines Trimeren Liponsäure bzw. Dihydroliponsäure Decarboxylierung zu Acetaldehyd Problem: Pyruvat zu Acetat Citrat-Synthase Raumstruktur und Mechanismus Offene Konformation Geschlossene Substratbind. Konformation Citrat-Synthase Aconitase Isocitrat-Dehydrogenase Succinat Dehydrogenase Oxidoreduktase mit FAD im aktiven Zentrum (kovalente Verknüpfung Enzym+Coenzym über His) FAD Succinyl-CoA synthetase Fumarase: katalysiert Hydratation von Fumarat Regulation Citratcyclus (li) Metabolische Reaktionswege C. (re) Feedback Aktivierung: AMP, ADP Inhibierung: ATP, Citrat, [NADH]/[NAD+]