Biochemie – Tutorium 10
Werbung

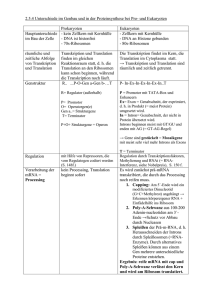
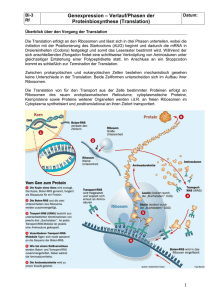
Biochemie – Tutorium 10 Genetischer Code, Translation & Regulation der Proteinbiosynthese IMPP-Gegenstandskatalog 3 Genetik 3.1 3.1.1 3.1.2 3.1.3 Nukleinsäuren Molekulare Struktur, Konformationen und Funktionen der Desoxyribonukleinsäure (DNA); Exon, Intron Molekulare Strukturen und Funktionen der Ribonukleinsäure (RNA) Genetischer Code 3.2 3.2.1 3.2.2 3.2.3 3.2.4 Umsetzung genetischer Information Transkription der DNA Prozessieren der RNA Translation Regulation der Proteinbiosynthese 3.3 3.3.1 3.3.2 3.3.3 3.3.4 3.3.5 Weitergabe und Verteilung genetischer Information Replikation der DNA Zellzyklus, Mitose, Meiose Meiotische Systeme, Kernphasenwechsel, Generationswechsel Plasmatische Vererbung Parasexuelle (parameiotische) Systeme, Phagen, Plasmide, Resistenzfaktoren 3.4 3.4.1 3.4.2 3.4.3 3.4.4 Veränderungen der genetischen Information Somatische Mutationen, Mutationen der Keimbahn Mutationstypen, Genom-, Chromosomen- und Punktmutationen, Ames-Test Mutagene Faktoren und transponierbare genetische Elemente Umordnung der Gene (Anikörperbildung) 3.5 3.5.1 3.5.2 Grundlagen der Molekularbiologie Techniken der Molekularbiologie Klonierung und Überexpression von Genen Proteinbiosynthese • Die eigentliche Proteinbiosynthese erfolgt durch Translation des in DNA bzw. RNA verwendeten Nukleinsäurecodes in die für das Protein spezifische Aminosäuresequenz. Die Translation findet in einem eigenen Organell, dem Ribosom statt. Dabei dienen tRNAs als Adaptermoleküle. • Im Anschluss an die Synthese müssen die Proteine ihre spezifische Raumstruktur annehmen, eventuell mit Cofaktoren ausgerüstet werden und häufig noch weitere posttranslationale Modifikationen durchlaufen. Genetischer Code • In einem für ein Protein codierenden Gen auf der DNA ist die Information für dessen Aminosäuresequenz gespeichert. • Im Zuge der Transkription wird der kodierende DNA-Strang in mRNA umgeschrieben und aus dem Zellkern geschleust. • Als Buchstaben des DNA-Codes bzw. RNA-Codes fungieren die 4 Basen Adenin (A), Guanin (G), Cytosin (C) und Thymin (T) bzw. Uracil (U). • Als codierende Einheit für eine der 20 bzw. 21 proteinogenen Aminosäuren dient jeweils eine Sequenz von 3 aufeinanderfolgenden Basen, die als Codon bezeichnet wird. • Mit einer Sequenz von drei Basen pro Aminosäuren können allerdings 4³ = 64 Aminosäuren verschlüsselt werden. Es gibt für bestimmte Aminosäuren mehrere verschiedene Codons. • Die Kodierung der Aminosäuren (außen) durch die Basentripletts auf der mRNA ist von innen (5') nach außen (3') zu lesen. Startcodon • Das Triplett AUG dient sowohl als Codon für Methionin als auch als Startsignal der Translation, es wird daher auch als Startcodon bezeichnet. Eines der ersten AUG-Tripletts auf der mRNA wird das erste Codon, das zu Protein translatiert wird. Welches AUG genau verwendet werden soll, erkennen die Proteine an Signalen in der umliegenden Sequenz. Stopcodon • Neben den 61 Aminosäure-codierenden Basentripletts des universellen genetischen Codes gibt es drei Kombinationen von Nukleinbasen, die keine Aminosäuren verschlüsseln, sondern die Proteinsynthese terminieren, die Stopcodons: – UAG – UAA – UGA • Während das Codon UGA zumeist als Stop gelesen wird, kann es selten und nur unter bestimmten Bedingungen für eine 21. Aminosäure stehen: Das Selenocystein (Sec). Die Biosynthese und der Einbaumechanismus von Selenocystein in Proteine unterscheidet sich stark von dem aller anderen Aminosäuren: seine Insertion erfordert einen neuartigen Translationsschritt, bei dem ein UGA im Rahmen einer bestimmten Sequenzumgebung und zusammen mit bestimmten Cofaktoren anders interpretiert wird. Hierzu ist eine strukturell einzigartige, Selenocystein spezifische tRNA (tRNASec) erforderlich, die bei Vertebraten (Wirbeltiere) mit drei verschiedenen, allerdings verwandten Aminosäuren beladen werden kann: Serin, Selenocystein und Phosphoserin • Der genetische Code hat folgende Eigenschaften: Er ist – Universal – Degeneriert – Konservativ – Kommafrei (Codons schließen lückenlos aneinander) – Nicht überlappend (eine Base ist immer nur Bestandteil eines einzigen Codons) Universal • Der genetische Code ist universal, da er für die Verschlüsselung der Aminosäuren sowohl bei Prokaryoten als auch bei Eukaryoten gilt. • Lediglich bei der mitochondrialen DNA finden sich kleinere Abweichungen. Degeneriert • Der genetische Code ist degeneriert. Aufgrund der oben angesprochenen 64 Codon bzw. 61 Codons, die für Aminosäuren verschlüsseln, existieren für alle Aminosäuren außer Tryptophan und Methionin mindestens zwei unterschiedliche Codons. konservativ • Der genetische Code ist konservativ. Darunter versteht man, dass sich gewisse Gesetzmäßigkeiten für die Codierung ableiten lassen: – Bei den Basen an dritter Position wird lediglich die Unterscheidung zwischen Purin- oder Pyrimidinbase getroffen. – Die Base an der zweiten Position entscheidet darüber, ob eine hydrophile oder eine hydrophobe Aminosäure in das Protein eingebaut wird. Mutationen an den verschiedenen Codonpositionen haben unterschiedlich starke Auswirkungen auf die Endstruktur des Proteins. Aminoacyl-tRNA-Moleküle • Aminosäuren können nur dann in die Proteinbiosynthese eingehen, wenn sie vorher in einem ATP-verbrauchenden Prozess an die als Adapter agierenden tRNA-Moleküle gebunden werden. – Das 3´-Ende mit der Sequenz CCA des jeweiligen tRNA-Moleküls bindet spezifisch eine Aminosäure. – Das Anticodon der tRNA ist dem für die entsprechende Aminosäure codierenden Codon auf der mRNA komplementär Wobble-Phänomen • In den verschiedenen Zellen eines Organismus befinden sich je nach Spezies 31-40 tRNA-Moleküle (obwohl es eigentlich 61 gibt, die für AS codieren). Die synonymen Triplett-Codons der mRNA für jeweils eine AS unterscheiden sich in der Regel an der 3. Stelle. Damit reduziert sich die Zahl der durch eine tRNA zu unterscheidenden Tripletts auf 30. Die höhere Zahl an tRNAs kommt dadurch zustande, dass für einige Synonyma verschiedene tRNAs existieren können. Die Paarung der ersten Base am 5´-Ende des Anticodons mit der entsprechenden dritten Base des Codons ist häufig nicht sehr fest und auch nicht unbedingt spezifisch. Wackeln (engl. to wobble) der Codon-AnticodonBindung. • Konsequenzen des Wobble-Phänomens sind, dass – die Minimalzahl der tRNA-Moleküle bei 31 liegt – alle Codon-Anticodon-Wechselwirkungen etwa gleich stark und etwas schwächer als klassische Basenpaarungen sind. • Dies ist wichtig für die Aufrechterhaltung der hohen Geschwindigkeit der Proteinbiosynthese, die neben der schnellen Erkennung von Anticodon und Codon auch von der raschen Lösung der Wasserstoffbrückenbindung zwischen den Basen abhängig ist. • Somit ist das Wobble-Phänomen ein Kompromiss zwischen Schnelligkeit und Sicherheit der Proteinbiosynthese. Bildung der Aminoacyl-tRNA-Moleküle • Die Beladung der einzelnen tRNAs mit Aminosäuren ist ein enzymkatalysierter Prozess. • Für jede proteinogene Aminosöure existiert eine spezifische Aminoacyl-tRNA-Synthetase. • Diese Enzyme sind zusätzlich mit einer Hydrolase-Aktivität ausgestattet, um eventuell falsch angeknüpfte Aminosäuren wieder vom tRNA-Molekül zu entfernen. • Die Synthese erfolgt in zwei Teilschritten: – Im ersten Schritt wird die Aminosäure aktiviert, indem die Carboxylgruppe der Aminosäure unter Abspaltung von Pyrophosphat mit einem Molekül ATP reagiert. – Im zweiten Schritt greift die freie 3´-OH-Gruppe am 3´-Ende der tRNA die aktivierte Aminosäure an, sodass unter Abspaltung von AMP ein Aminoacyl-tRNA-Molekül entsteht. Ort der Proteinbiosynthese • Die für die Proteinbiosynthese verantwortlichen Organellen sind die sog. Ribosomen. • Ribosomen kommen bei Prokaryoten im Cytosol, bei Eukaryoten sowohl frei im Cytosol als auch an das endoplasmatische Reticulum gebunden vor. ( raues ER) • Cytosolische Proteine werden an freien Ribosomen im Cytosol synthetisiert. • Sekretorische und Membranproteine werden am ER synthetisiert. • Membranproteine und sekretorische Proteine werden cotranslational in das Lumen des rauen ER transportiert. • Zur Erkennung dient eine N-terminal gelegene Signalsequenz aus ca. 30 AS. Die Synthese startet also primär an freien Ribosomen, die bei Vorhandensein dieser Sequenz zum rER wandern. • Proteine, die ohne Signalsequenz synthetisiert werden, verbleiben im Cytosol. Aufbau der Ribosomen • Ribosomen können per Zentrifugation in eine kleine und eine große Untereinheit aufgeteilt werden, wobei auch hier Unterschiede zwischen Prokaryoten und Eukaryoten festgestellt werden können. • Diese werden als unterschiedliche Sedimentationskonstanten angegeben: – Prokaryoten: 70S-Ribosomen (50S und 30S) – Eukaryoten: 80S-Ribosomen (60S und 40S) • Hierbei ist zu beachten, dass Eukaryoten in den Mitochondrien und Plasmiden ebenfalls 70SRibosomen besitzen. • Für die Funktion der Ribosomen sind folgende Positionen wichtig: – Bindungsstelle für mRNA – Zwei unterschiedliche Bindungsstellen für beladenen tRNA-Moleküle. Diese werden als Peptidyl- und Aminoacyl-Stelle (bzw. P- und A-Stelle) bezeichnet. – Eine Bindungsstelle für die leere tRNA, die auch als EStelle (exit-Stelle) bezeichnet wird. – Eine Peptidyltransferase-Stelle – Außerdem Bindungsstellen für regulatorsiche Faktoren. Translation • Analog zu Replikation und Transkription wird auch die Translation in drei Phasen unterteilt: – Initiation – Elongation – Termination Initiationsphase der Translation • Als erstes bindet die Starter-Aminoacyl-tRNA an den Initiationsfaktor 2 (IF2). • Die Starter-Aminosäure ist immer Methionin. • Das entsprechende tRNA-Molekül hat im Anticodon die Sequenz CAU, welche zum Startcodon AUG komplementär ist. • Bei Prokaryoten wird zusätzlich das freie Aminoende des Methionins formyliert. • IF 2 ist ein G-Protein (Guaninnukleotid-bindendes Protein siehe auch G-Protein gekoppelte Rezeptoren). Aktiviert ist es mit GTP beladen und in der Lage, Aminoacyl-tRNA zu binden. • Der gebildete Komplex lagert sich anschließend an die kleine 40S-Untereinheit an. • Die kleine 40S-Untereinheit muss zu diesem Zweck allerdings zuerst durch Bindung der Initiationsfaktoren IF-1 und IF-3 aktiviert werden. • Analoger Verlauf bei Prokaryoten. • Der weitere verlauf der Initiationsphase unterscheidet sich bei Eukaryoten und Prokaryoten teilweise. • Eukaryoten: – Der Initiationsfaktor IF-4 assoziiert an die Cap-Struktur der mRNA (5´-Ende) und bindet anschließend die mRNA an die kleine 40S-Untereinheit. Danach wird die mRNA zusammen mit IF-1 nach dem ersten Startcodon AUG vom 5´-Ende abgesucht und auf die P-Stelle positioniert. – Da die eukaryotische mRNA aufgrund der Prozessierung und des Transports aus dem Zellkern genug Zeit hat, um komplexere Sekundärstrukturen auszubilden, werden für die Positionierung Helicasen benötigt. • Eukaryoten: – Anschließend lagert sich unter GTP-Verbrauch die große 60S-Untereinheit an. – IF-2 und IF-5 sind mit GTP beladen, das nun in GDP und anorganisches Phosphat gespalten werden. • Zur Regeneration der beiden Initiationsfaktoren stehen Katalysatoren bereit, die die Abspaltung von GDP erleichtern und eine Neubeladung der Moleküle mit GTP ermöglichen. – Vom entstandenen vollständigen 80S-Ribosom dissoziieren anschließend IF-1 und IF-3 ab. – Es entsteht ein Tunnel durch die 60S-Untereinheit, durch den später die sich bildende Peptidkette verläuft. • Prokaryoten: – Die Unterscheidung des Startcodons von den anderen AUG-Tripletts erfolgt durch eine Sequenz, die sich 4-10 Basen vor dem Startcodon befindet, die sog. Ribosomenbindestelle (Shine-Dalgarno-Sequenz GGAGA) – Für diese Sequenz existiert eine komplementäre Sequenz auf der rRNA der kleinen 30S-Untereinheit. – Die Bindung des Komplexes aus mRNA und IF-2 erfolgt durch Interaktion der rRNA mit der Shine-DalgarnoSequenz. – Die mRNA wird analog zu den Eukaryoten auf der P-Stelle positioniert. • Prokaryoten: – Nachdem die mRNA positioniert wurde, lagert sich ebenfalls die große Untereinheit (50S) unter Abspaltung der bei den Eukaryoten angesprochenen Initiationsfaktoren an und vervollständigt das Ribosom. – Dabei bildet sich analog ein Tunnel durch die 50S-Untereinheit, durch den später die sich bildende Peptidkette verläuft. Elongationsphase der Translation • Die Elongationsphase läuft bei Eukaryoten und Prokaryoten identisch ab. • Wichtig sind an dieser Stelle der Translation die drei Bindungsstellen für tRNA am Ribosom: – P-Stelle – A-Stelle – E-Stelle 1. Schritt: Beladung • Zu Beginn der Elongation ist die StarterAminoacyl-tRNA an der P-Stelle des Ribosoms gebunden. • Im ersten Schritt der Elongation muss die die freie A-Stelle mit der Aminoacyl-tRNA besetzt werden, deren Anticodon zum nächstfolgenden Basentriplett der mRNA komplementär ist. • Die Beladung wird bei den Eukaryoten durch den Elongationsfaktor 1 (EF-1) und den Prokaryoten analog EFTu katalysiert. • EF-1 und EF-Tu sind G-Proteine. • In der aktiven Form ist EF-1 (bzw. EF-Tu) mit GTP beladen und kann ein Aminoacyl-tRNA-Molekül binden und auf der mRNA positionieren. • Anschließend kann die tRNA unter Spaltung von GTP zu GDP und Phosphat und Entlassung der tRNA vom jeweiligen Elongationsfaktor an das Ribosom gebunden werden. Die Entlassung kann nur bei einer korrekten Paarung zwischen Codon und Anticodon stattfinden, eine falsche tRNA kann also nicht an das Ribosom gebunden werden (proof-reading). 2. Schritt: Transpeptidierung • Die freie Aminogruppe der auf der A-Stelle gebundenen Aminosäure greift nun das Carbonyl-CAtom der auf der P-Stelle lokalisierten Aminosäure an. • Dies führt unter Knüpfung einer Peptidbindung (entspricht Säureamidbindung) zur Lösung der Aminosäure von der tRNA auf der P-Stelle. • Diese Reaktion verläuft spontan unter Katalyse der rRNA des Ribosoms. Das Ribosom ist ein Ribozym (siehe Bio-Katalysatoren) • Das neusynthetisierte Peptid befindet sich nun an der A-Stelle. 3. Schritt: Translokalisation • Während der Translokalisation rutscht das Ribosom um genau ein Basentriplett weiter, dadurch gelangt das synthetisierte Peptid von der A-Stelle auf die P-Stelle. • Die A-Stelle ist nun für die Beladung mit der nächsten Aminoacyl-tRNA bereit. • Auf der E-Stelle befindet sich die leere tRNA und wird von ihr freigesetzt. • Für die Translokalisation wird wieder ein Elongationsfaktor benötigt. Bei den Eukaryoten ist es EF-2 und bei den Prokaryoten EF-G. • Beide sind G-Proteine. • Die Translokalisation verläuft unter Hydrolyse von GTP, mit dem beide Elongationsfaktoren in der aktiven Form beladen sind. Eukaryotische Elongationsphase Prokaryotische Elongationsphase Translation am rauen ER Terminationsphase der Translation • Die Termination der Translation wird ausgelöst, sobald ein Stoppcodon an die AStelle kommt. • Die Termination verläuft bei Eukaryoten und Prokaryoten identisch. • Die Stoppcodons werden nicht von tRNAs erkannt. Stattdessen werden sie von sog. Release Factors erkannt (RF-1 erkennt UAA und UAG, RF-2 erkennt UAA und UGA) • Der entsprechende RF bindet zusammen mit GTP an das Ribosom. • Dadurch überträgt die Peptidyltransferase die Peptidkette von der P-Stelle nicht auf die A-Stelle sondern auf H2O. • Die Peptidkette wird freigesetzt und gleichzeitig unter Spaltung des an den RF gebundenen GTP die unbeladene tRNA von der P-Stelle abgegeben. • Anschließend zerfällt das Ribosom in seine Untereinheiten. Polysomen • Als Polysom oder Polyribosom wird die Aufreihung vieler Ribosomen an der zu transferierenden mRNA während der Proteinsynthese im Cytoplasma bezeichnet. Dabei liegen die Ribosomen entweder frei im Cytoplasma vor, oder sind an das Endoplasmatische Retikulum (ER) gebunden, so dass das fertige Protein während des Translationsprozesses in das ER hineingeschleust wird. Polysomen sind Ausdruck des Mechanismus der Translation, bei dem dieselbe mRNA mehrfach, praktisch gleichzeitig, abgelesen wird und daher von einem mRNA-Molekül viele Proteine erzeugt werden können. Translation, und dann? • nach Translation: Proteinfaltung und Modifizierung (Glycosylierung, Anheftung von Lipidankern, Modifikation einzelner AS-Seitenketten, Anheftung von Cofaktoren, Spleißen, etc.) • Spleißen kann auch nach der Translation, auf Proteinebene, ablaufen • Proteine, die gespleißt werden, setzen sich aus sog. Exteinen und Inteinen zusammen • Spleißung: Peptid wird herausgeschnitten und die freiwerdenden Enden werden verknüpft • Proteinspleißen v. a. bei DNA-Polymerasen, Helicasen, Topoisomerasen Inhibitoren der Translation • Streptomycin: Verhindert Initiation • Paromomycin: Erhöht ribosomale Fehlerrate durch Bindung an 30S-Untereinheit • Tetracycline: Binden an 30S-Untereinheit, blockieren Anheftung der Aminoacyl-tRNA • Chloramphenicol: Blockiert Peptidyltransferase • Erythromycin: Hemmt Translokation, bindet an 50SUntereinheit • Puromycin: Analogon der Aminoacyl-tRNA und sorgt für vorzeitigen Kettenabbruch in Pro- und Eukaryonten • Diphtherietoxin: Hemmt Translokation durch Inaktivierung von EF-2 Vergleich Genexpression Prokaryonten – Eukaryonten Prokaryonten Eukaryonten Genaufbau Gene enthalten nur codierende Sequenzen Mosaikgene enthalten Exons (codierend) und Introns (nichtcodierend) räumliche Organisation (Kompartimentierung) Transkription und Translation finden im Transkription findet im Zellkern statt, Cytoplasma statt die Translation im Cytoplasma zeitliche Organisation Translation beginnt, bevor die Transkription beendet ist Translation beginnt nach Abschluss der Transkription Reifung der mRNA mRNA wird ohne Modifizierung translatiert prä-mRNA wird durch Spleißen, Capping und Anheften des Poly(A)Schwanzes prozessiert DNA-Aufbau DNA enthält keine Stützproteine (Histone) DNA enthält Histone (Nucleo-HistonKomplexe) Ribosomenaufbau 70S: 50S- + 30S-Untereinheiten 80S: 60S- + 40S-Untereinheiten Regulation der Proteinbiosynthese • Die Kontrolle der Proteinbiosynthese bzw. Genexpression erfolgt auf verschiedenen Ebenen: – Transkriptionskontrolle – RNA-Prozessierungskontrolle – RNA-Transport- und Lokalisationskontrolle – Translationskontrolle – mRNA-Degradierungskontrolle – Proteinaktivitätskontrolle Regulation der Transkription • Die häufigste Form der Expressionskontrolle erfolgt während der Transkriptionsphase, bevorzugt im Stadium der Initiation. • Im folgenden soll dies am Beispiel des lacOperons bei Prokaryoten verdeutlicht werden. Aufbau eines Operons • Ein Operon ist eine Funktionseinheit der DNA von Prokaryoten und manchen Eukaryoten, bestehend aus Promotor, Operator(en) und mehreren (Struktur-)Genen, die für Proteine mit typischerweise verwandten Funktionen codieren. • Abhängig vom jeweiligen Operon können verschiedene regulatorische Proteine (Repressoren bzw. Aktivatoren) mit den Operatoren in Wechselwirkung treten und dadurch die Transkription der Gene im Operon an- oder abschalten. Auf diese Weise wird die Synthese der betreffenden mRNA (messenger-RNA) und damit indirekt der codierten Proteine durch Translation dieser mRNA aktiviert oder gehemmt. lac-Operon • Das lac-Operon (lactose-operon) spielt sowohl beim Transport, als auch beim Abbau von Lactose in Bakterien eine wichtige Rolle. • Das Operon besteht aus einem Promotor (P), einem Operator (O) und drei Strukturgenen (Z, Y, A) • Das Repressorgen i enthält Informationen zur Bildung eines Repressors, gehört allerdings nicht zum lacOperon. • Das lacZ-Strukturgen codiert für das Enzym βGalactosidase (LacZ). Dieses Enzym spaltet Lactose hydrolytisch in Galactose und Glucose und erschließt das Disaccharid somit als Nahrungsquelle für das Bakterium. Außerdem kann es Lactose zu Allolactose isomerisieren. • Das lacY-Gen codiert für ein Transportprotein namens β-Galactosid-Permease (LacY), welches die Aufnahme von Lactose in die Zelle ermöglicht. • Das lacA-Gen codiert für das Enzym βGalactosid-Transacetylase. Es ist nicht für den Lactoseabbau notwendig, und seine Funktion ist nicht endgültig geklärt. Regulation des lac-Operons • Die drei Proteine des lac-Operon werden erst dann exprimiert, wenn Lactose im Umgebungsmedium vorhanden ist und es keine für die Zelle günstigere Energiequelle gibt wie beispielsweise Glucose gibt. Ein System aus negativer und positiver Regulation steuert den Abbau der effizientesten Energiequelle. • Das lac-Operon wird sowohl negativ durch einen Repressor, als auch positiv durch einen Aktivator reguliert. • Zusätzlich kontrolliert der Mechnismus des InduktorAusschlusses (inducer exclusion) die Aktivität der LacPermease. Negative Regulation • Die negative Regulation des lac-Operons erfolgt durch einen lacRepressor, dem LacI-Protein. Dies ist ein Protein, welches am Operator binden kann. Sobald sich der Repressor an die DNA angelagert hat, ist eine Expression der nachfolgenden Strukturgene nicht mehr möglich. • Der Repressor seinerseits wird von einem Regulatorgen, dem lacIGen, codiert. Dieses Gen liegt separat oberhalb des lac-Operons und wird von einem eigenen, konstitutiven Promoter exprimiert. • Da der Repressor mit hoher Affinität an die Operatoren bindet, ist der Promotor nahezu ständig reprimiert. In diesem Zustand kommt es kaum zur Genexpression. Es wird nur so viel exprimiert, wie für eine zukünftige Induktion an Proteinen notwendig ist. • Der Vorteil dieser negativen Regulation besteht darin, dass, solange keine Lactose verstoffwechselt werden muss, auch keine Enzyme für ihren Abbau bereitgestellt werden müssen. Positive Regulation • Verantwortlich für die positive Regulation des lacOperons ist ein Aktivatorprotein, das CAP (catabolite activator protein). Allerdings ist die CAP-Aktivität von der Konzentration von cAMP direkt abhängig. Nur wenn diese beiden Stoffe aneinander binden, können sie die Genexpression positiv beeinflussen. • Sie lagern sich an die DNA an und wechselwirken direkt mit der RNA-Polymerase. Dadurch wird die Affinität der RNA-Polymerase zum Promotor deutlich erhöht. • Es sind also drei Elemente für diese positive Regulation des lac-Promotors notwendig: das CAP-Protein, cAMP und eine CAP-Bindungsstelle im lac-Promotor. Einfluss von Lactose • Ist Lactose als Energielieferant das effizienteste Substrat in der Umgebung der Zelle, wird sie durch die β-Galactosid-Permease in die Zelle verbracht. Dort wird sie teilweise durch β-Galactosidase in Allolactose umgewandelt. Dies bedeutet, dass die Gal-β-1,4-GlcBindung in eine Gal-β-1,6-Glc-Bindung überführt wird. • In dieser Form ist nun eine Anlagerung an den Repressor LacI möglich. Durch diese Anlagerung verändert sich die Konformation des Repressors und er löst sich vom Operator. Allolactose ist somit ein Induktor des lac-Operons. • Nun kann die RNA-Polymerase mit der Transkription beginnen. Durch die nachfolgende Translation werden weitere Moleküle LacPermease und β-Galactosidase bereitgestellt. So kann Lactose dauerhaft als Substrat genutzt werden, bis dieses aufgebraucht ist, oder eine bessere Energiequelle zur Verfügung steht. Einfluss der Glucose • Es ist für die Zelle von Vorteil, die Glucose der Lactose als Substrat vorzuziehen. Demnach muss die Präsenz von Glucose den Abbau der Lactose hemmen. • Der Transport von Ghlucose in die Zelle induziert eine Hemmung der Lactose-Permease, wodurch keine Lactose in die Zelle transportiert wird und das lac-Operon inaktiviert bleibt. • So kommt es auch bei Anwesenheit von Lactose kaum zur Genexpression, und die Glucose wird bevorzugt abgebaut. Regulation bei Eukaryoten • Die Regulation ist ein komplexerer Prozess als bei Prokaryoten. Dabei wird an verschiedenen Stellen der Proteinbiosynthese eingegriffen. (Transkription, Prozessierung, Transport aus dem Zellkern oder Translation). • Die Regulation der Proteinbiosynthese erfolgt meist ebenfalls schon auf der Stufe der Transkription. • Wichtige regulatorische Einheiten sind dabei Promotoren, Enhancer, Silencer und diverse Transkriptionsfaktoren. Regulation der Translation • Über die Regulation der Proteinbiosynthese während der Translation ist nur wenig bekannt. • Sie findet meist in den Stadien der Initiation und Termination statt. • Der Abbau der mRNA kann ebenfalls ein Teil der Regulation darstellen.