Genetisches Praktikum 2010 (20160)
Werbung
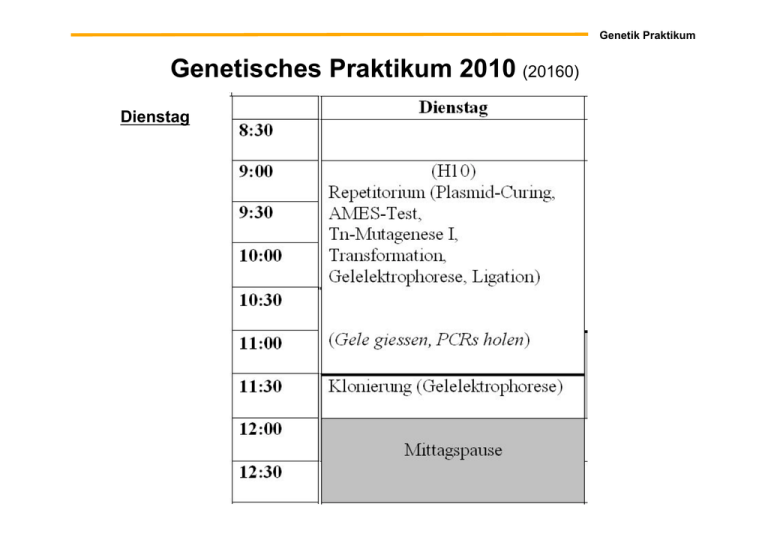
Genetik Praktikum Genetisches Praktikum 2010 (20160) Dienstag Genetik Praktikum Genetisches Praktikum 2010 (20160) Dienstag Genetik Praktikum Praktikumsversuche I. Allgemeines A. Plasmid-Curing B. F-Plasmid-Nachweis C. Komplementations-Test mit rII-Mutanten des Phagen T4 D. PCR-Fingerprint mittels STR-Analyse II. Methoden A. Titerbestimmung bei Bakterienkulturen B. !-Galaktosidase-Test III. Mutagenese und DNA-Reparatur A. Isolierung und Identifizierung von Auxotrophie-Mutanten B. Nachweis der Transposon-Mutagenese des Plasmids pTS115 C. Ames-Test IV. Projekt: Klonierung eines Genfragments A. PCR B. Gelelektrophorese C. Ligation D. Elektrotransformation E. Plasmid-Präparation I.A Plasmid curing Plasmide •! Fertilitäts(F)-Plasmide: enthalten nur tra-Gene. Einleitung der Konjugation. •! Resistenz(R)-Plasmide: enthalten Resistenzgene gegen Antibiotika oder Gifte •! Col-Plasmide: enthalten Gene für Colicine (Proteine, die für andere Bakterien toxisch sind) •! Degradationsplasmide: ermöglichen den Abbau von ungewöhnlichen Substanzen, (z.B. Toluol oder Salicylsäure). •! Virulenz-Plasmide: machen ein Bakterium zu einem Krankheitserreger (Yersinia pestis) •! Ti-Plasmide (Tumor inducing): werden von bestimmten Bakterien (Agrobacterium tumefaciens) auf Pflanzen übertragen. Dort verursachen sie die einzige bekannte Krebserkrankung in Pflanzen. I.A Plasmid curing Plasmide Plasmide müssen repliziert werden. Ohne Replikation gehen sie verloren. Multi-copy Plasmide: können mehr als einmal pro Zellzyklus repliziert werden. Spezielle Segregationsfunktionen für Verteilung während der Zellteilung nicht unbedingt nötig. Single-copy Plasmide: nur einmal repliziert pro Zellzyklus. Segregationsfunktionen wichtig Konjugation Übertragung von Teilen des Genoms von einer Donor- in eine Rezipientenzelle Fertilitäts Faktor F: F-: Rezipient, F+: Donor tra Gene: (transfer), z.B. Pilin-Gen » Ausbildung eines Sex-Pilus (F-Pilus) I.A Plasmid curing Übertragung des F-Plasmids bei der Konjugation Donor (F+) Cytoplasmabrücke Rezipient (F-) oriT: Replikationsursprung, Kopie eines Einzelstrangs nach dem ‚rolling circle‘ Mechanismus Kopie gelangt über Pore in den Rezipienten, wo der Einzelstrang zur Doppelhelix ergänzt wird » F- Stamm wird zu F+ I.A Plasmid curing Übertragung des F-Plasmids bei der Konjugation in seltenen Fällen kann ein F+ Stamm sich verändern: •! ca. 1000 x höhere Rekombinationsrate nach Konjugation mit F» Hfr-Stamm: high frequency of recombination •! nach Konjugation wird F- i.d.R. nicht zu F+ » F-Plasmid im Hfr-Stamm über cross over ins Chromosom integriert (Campbell-Integration) » ausgehend vom oriT wird bei der Konjuagtion das gesamte Chromosom übertragen I.A Plasmid curing Warum high frequency of recombination? ! F-Plasmid und Genom haben keine Gene/Marker gemeinsam, die ausgetauscht werden könnten (Ausnahme: F'-Plasmid, s.u.) ! Transfer von genomischer DNA zusätzlich zu F+-Plasmid DNA sehr selten; nur nach zufälliger Integration des Plasmids ins Genom ! Hfr Zellen transferieren bei Konjugation immer genomische DNA » Empfängerzelle wird dadurch teilweise diploid = merozygot ! durch Doppelcrossover zwischen Donor- und Empfänger-DNA kann es zu Rekombination kommen I.A Plasmid curing Vergleich Konjugation F+ bzw. Hfr mit F- I.A Plasmid curing Genkartierung durch Paarungsunterbrechung Hfr und F- Stamm werden gemischt, Konjugation wird nach verschiedenen Zeiten durch intensives Mixen physikalisch aufgehoben. Ausplattierung auf verschiedenen Testmedien (z.B. Auxotrophietest) Welche Auxotrophie wird nach welcher Zeit aufgehoben? » Bestimmung der Reihenfolge der Gene auf dem Chromosom (Abstand in Minuten) I.A Plasmid curing Das F-Plasmid integriert an verschiedenen Stellen des E. coli Chromosoms über homologe Rekombination ! oriT wird als erstes übertragen, die Fertilitätsfaktoren (tra-Gene) als letzte I.A Plasmid curing Acridinorange •! Fluoreszenzfarbstoff •! Wieso zum plasmid curing geeignet? » interkaliert mit seinem planaren Ringsystemen zwischen Basen von DNA » stört Replikation der Plasmid-DNA •! Wieso wird Replikation des Bakterienchromosoms nicht verhindert? » Plasmid-DNA stärker superspiralisiert » Acridinorange bindet bevorzugt an Plasmid DNA III.C Ames Test Punktmutationen: Substitution von Basen Pyrimidin Purin Transition: Austausch von Basen gleicher chemischer Kategorie (A ! G / G ! A) Transversion: Austausch von Basen unterschiedlicher chemischer Kategorie (C ! A / C ! G / T ! A / T ! G und umgekehrt) Konsequenz: neutrale Mutation missense Mutation nonsense Mutation III.C Ames Test Punktmutationen in codierenden DNA Bereichen Art der Mutation keine Mutation Transition oder Transversion Auswirkung der Mutation Wildtyp Codons bestimmen Wildtyp Protein neutrale (stille) Mutation geändertes Codon bestimmt gleiche Aminosäure missense Mutation (konservativ) missense Mutation (nicht konservativ) nonsense Mutation Insertion oder Deletion ‚Indel‘ frameshift Mutation frameshift Mutation geändertes Codon bestimmt chemisch ähnliche Aminosäure geändertes Codon bestimmt chemisch unähnliche Aminosäure geändertes Codon führt zur Termination der Translation III.C Ames Test Mechanismen der Entstehung spontaner Mutationen •! tautomere Umlagerungen der Basen führt zu Fehlpaarungen Mutation ist in allen Fällen eine Transition Keton – Enol Tautomerie III.C Ames Test Mechanismen der Entstehung spontaner Mutationen •! ‚Weitergleiten‘ der Replikation (replication slippage) ! Indel Mutation III.C Ames Test Mechanismen der Entstehung spontaner Mutationen •! spontane Schäden der Basen: Depurinierung und Desaminierung Depurinierung: Desaminierung: Amino- zu Ketofunktion C zu U (paart mit A, C ! T Transition); A zu Hypoxanthin (paart mit C, A ! G Transition) oxidative Schäden z.B. durch Sauerstoff Radikale blockiert DNA Replikation (paart mit A, G ! T Transversion) III.C Ames Test Molekulare Ursachen induzierter Mutationen induzierte Mutationen: werden durch mutagene Substanzen ausgelöst •! Einbau von Basenanaloga in die DNA (z.B. 5-Bromuracil / 2-Aminopurin) III.C Ames Test Molekulare Ursachen induzierter Mutationen •! alkylierende Agenzien (addieren Methyl- bzw. Ethylgruppen an verschiedene Positionen der Basen) Ethylmethansulfonat (EMS) Nitrosoguanidin (NG) III.C Ames Test Molekulare Ursachen induzierter Mutationen ! Strahlung: UV-Strahlung generiert Photoprodukte » Verknüpfung benachbarter Pyrimidine (v.a. Thymindimere) CPD: Cyclobutan Pyrimidin Dimer Ionisierende-Strahlung produziert reaktive Sauerstoffspezies (ROS); verursacht auch Aufbrechen der N-glykosidischen Bindung und Strangbrüche III.C Ames Test Molekulare Ursachen induzierter Mutationen •! Agenzien, welche mit Basen Reaktion eingehen: ‚bulky addition‘ Bsp: Aflatoxine in Schimmelpilzbefallenen Lebensmitteln Bsp: Nitrosamine in gepökeltem Fleisch und Käse - entstehen aus Nitriten und Aminen - Präkanzerogen: - Cytochrom-P450-katalysierte Reaktionen » Formaldehyd und Carbeniumionen III.C Ames Test Ames Test zur Identifikation mutagener Substanzen Reversion von Mutationen im his Gen verschiedener Salmonella typhimurium Stämme durch Behandlung mit mutagenen Substanzen Zugabe von Rattenleberextrakt: viele Carcinogene werden erst mutagen durch Metabolisierung in der Leber Welche Kontrollen fehlen hier? Mutation Ames Test zur Identifikation mutagener Substanzen Reversion von Mutationen im his Gen verschiedener Salmonella typhimurium Stämme durch Behandlung mit mutagenen Substanzen Salmonella typhimurium TA98: Rasterschubmutation Salmonella typhimurium TA100: Basensubstitution Im Praktikum verwendete Gifte: 2-Aminofluoren Hydroxylierung durch Leberenzyme » Addition an C8 von Desoxyguanosin (bulky addition) 4-Nitro-o-phenylendiamin (NPD) Wirkung? III.B Transposon-Mutagenese Transponierbare Elemente in Prokaryonten Insertionssequenzen (IS-Elemente) inverted repeats an den Enden kodieren für Transposase Transposons besitzen neben der Transposase zusätzliche Gene (z.B. Antibiotika Resistenzgene) zusammengesetzte Transposons (Klasse I Transposons) Gene, welche von IS-Elementen in entgegengesetzter Orientierung flankiert sind einfache Transposons (Klasse II Transposons) flankiert von kurzen (<50 bp) inverted repeats Tn1725 III.B Transposon-Mutagenese Zwei Mechanismen der Transposition konservative Transposition: ‚cut and paste‘ Mechanismus replikative Transposition: neue Kopie des Transposons III.B Transposon-Mutagenese Nachweis der Transposon-Mutagenese des Plasmids pTS115 E. coli Ru4404: chromosomale DNA xylE Transposon mit CamR pTS115 ori AmpR Kultivierung unter Bedingungen, bei denen Transposition statt findet » gelegentlich wird Tn1725 auch ins Plasmid pTS115 springen; egal wohin Tn1725 springt, Zellen bleiben Cam-R und Amp-R (Ausnahmen?) » daher: Plasmidisolation aus Ru4404 und Transformation in E. coli DH5! » Selektion auf LB/Cam/Amp-Agarplatten III.B Transposon-Mutagenese Catecholsprühtest zur Identifikation von xylE::Tn1725 Insertionsmutanten farbloses Catechol XylE gelbes "-Muconsäuresemialdehyd Ermitteln Sie den Anteil der weißen Klone und berechnen Sie hieraus die Größe des xylE-Gens! hierfür nötige Zusatzinformation: pTS115: 6 kb #-Lactamase-Gen: 0,9 kb ORI: 0,4 kb IV.B Elektrophorese Elektrophorese von Nukleinsäuren dsDNA: keine/kaum Sekundärstruktur konstante Ladungsdichte » Wanderungsstrecke ! 1 ⁄ Größe 5 x 106 – 1 x 105 bp 1 x 105 – 50 bp 500 – 5 bp » pulsed field Gelelektrophorese (PFGE) » normale Agarose-Gelelektrophorese (i.d.R. horizontal) » Polyacrylamid-Gelelektrophorese (PAGE; i.d.R. vertikal) Probenpuffer enthält typischerweise: Glyzerin » macht Probe schwer, so dass sie in die Taschen sinkt Farbstoff » erlaubt einfaches optisches (d.h. ohne UV-Licht) Verfolgen der Elektrophorese; Orange G läuft bei ca. 50 bp Bromphenolblau - bei ca. 300 bp Xylencyanol - bei ca. 4 kbp EDTA » hemmt Nukleasen ssDNA & RNA: wegen Sekundärstruktur gilt nur unter denaturierenden Bedingungen Wanderungsstrecke ! 1 ⁄ Größe » Formamid, Formaldehyd, Harnstoff, Hitze IV.C Ligation DNA-Ligase Mechanismus NAD+ E. coli: NAD+ Eukaryonten, T4-Phage: ATP ATP im Labor wird meist T4-Ligase benutzt, weil sie auch "stumpfe" DNA-Enden miteinander verknüpfen kann 29 IV.C Ligation Woran erinnert Sie dieser Reaktionsmechanismus mit einem DNA-AMP-Intermediat? » z.B. Aminoacyl-tRNA-Synthetase-Reaktion 30 IV.D Transformation Das Griffith Experiment (1928) Avirulente R-Zellen von Streptococcus pneumoniae werden durch hitzegetötete, virulente S-Zellen zu S-Zellen transformiert IV.D Transformation Transformation •! Aufnahme von DNA-Fragmenten oder Plasmiden aus dem externen Medium •! natürliche Kompetenz (z.B. Bacillus subtilis) •! Kompetenz durch chemische Behandlung (z.B. CaCl2) IV.D Transformation Elektroporation Durch ein elektrisches Feld, das in der Regel durch einen sich schnell entladenden Kondensator erzeugt wird, werden in der behandelten Zellmembran mikroskopisch kleine Löcher erzeugt, die sich innerhalb von Millisekunden wieder schließen. Vorteil: hohe Transformationsraten; Nachteil: Küvetten sind teuer