Vorlesung 3
Werbung
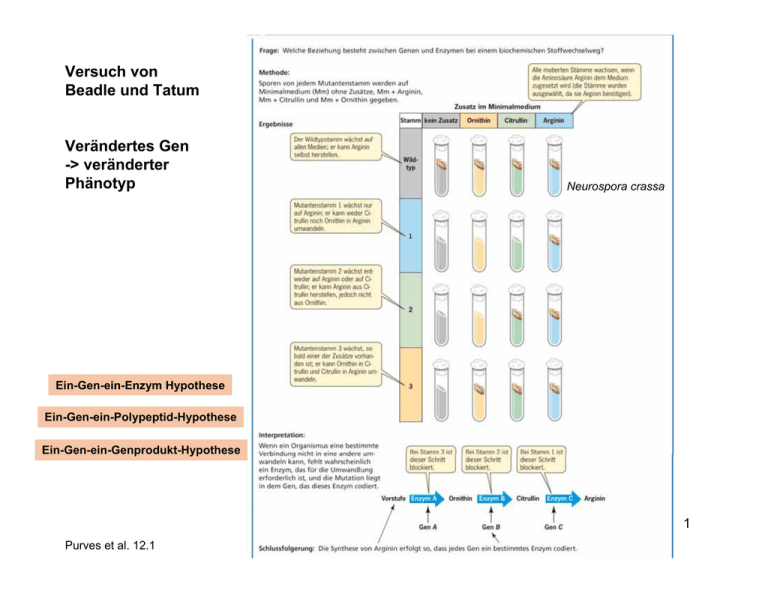
Versuch von Beadle und Tatum Verändertes Gen -> veränderter Phänotyp Neurospora crassa Ein-Gen-ein-Enzym Hypothese Ein-Gen-ein-Polypeptid-Hypothese Ein-Gen-ein-Genprodukt-Hypothese 1 Purves et al. 12.1 Das zentral Dogma: Von der DNA zum Protein Replikation Transkription Purves et al. 12.2 Translation 2 Genexpression: Gen Abschnitt auf der DNA, der für ein Genprodukt kodiert, inkl. Kontrollregionen Expression RNA • Fluss von der genetischen Information (DNA) zum Genprodukt • beteiligte Vorgänge sind: Protein Transkription (DNA in RNA) Translation (RNA in Protein) • findet in allen lebenden Zellen statt 3 Bei Prokaryoten in einem Kompartiment Purves et al. 12.3 4 Purves et al. 14.1 Pro- und eukaryotische Genexpression DNA Nicht-Matrizenstrang (+) Matrizenstrang, codogen (-) Transkription des Matrizenstranges RNA Translation Protein 5 Transkription Purves et al. 12.4 6 Die chemische Struktur der RNA RNA DNA •RNA enthält den Zucker Ribose •RNA enthält Uracil anstelle von Thymin 7 Die RNA-Polymerase transkribiert DNA RNA-Polymerase aus E. coli DNA-abhängige RNA-Polymerase •Katalyse von Phosphatdiesterbindungen •Verlängerung der wachsenden RNA Kette in 5‘->3‘Richtung •Substrat: Ribonukleosidtriphosphate, kein Primer αα ββ αα β‘ β‘ σ Minimal (Core) Enzym, 4 UE: α2,β,β‘ Holoenzym, 5 UE: α2,β,β‘,σ α: Struktur des Enzyms σ: Erkennung des Transkriptionsstarts β: RNA-Synthese β‘: Bindung an DNA 8 Promotor: Erkennungs- und Startpunkt für die RNA-Polymerase keine „0“ -1 „upstream“-Bereich - 35 -Region -10 -Region -35 +1 Start der Transkription -10 nicht-transkribierte DNA ATG transkribierte DNA Start der Translation Konsensussequenzen prokaryotischer Promotoren -10-Region = Pribnow-Box -35-Region -35 -10 +1 lac ACCCCAGGCTTTACACTTTATGCTTCCGGCTCGTATGTTGTGTGGAA trp AAATGAGCTGTTGACAATTAATCATCGAACTAGTTAACTAGTACGCA pL TCTGGCGGTGTTGACATAAATACCACTGGCGGTGATACTGAGCACAT Kons.-S ----------TTGACA---17+/-1 bp-----TATAAT-------- 9 Transkriptionszyklus einer bakteriellen RNA-Polymerase σ-Faktor RNA Promotor Haarnadel DNA 1. Bindung des RNA-Pol-Holoenzyms an den Promotor (geschlossener Komplex) RNA-Polymerase 7. Freisetzung des Transkripts 2. Aufwinden der DNA (offener Komplex) 6. Termination 5. Elongationsphase mit hoher Prozessivität (50 nt/s) 3. Anfängliche Transkription (10 nt) Ribonukleosidtriphosphate RNA RNA 4. Ablösung des σ-Faktors 10 Alternative Sigmafaktoren: Regulation der Genexpression bei E. coli Sigmafaktor σ70 σS σ32 Größe (kDA) 70 38 32 Funktion Standard-Sigmafaktor Hunger, Stress Hitzeschock 11 Transkriptionstermination bei E. coli Rho-unabhängige Termination Rho-abhängige Termination Haarnadelförmige Sekundärstruktur im 3‘ Nichtkodierungsbereich einer mRNA Rho-Protein (Hexamer) lagert sich an mRNA spaltet ATP 12 Transkripte sind länger als die kodierende Region Terminator Promotor +1 Ende Kodierende Region DNA 5‘ Leadersequenz 5‘ untranslatierter Bereich 3‘ RNA-Transkript Trailer-Abschnitt 3‘ untranslatierter Bereich 13 Eukaryot Prokaryot RNA-Polymerase polycistronische mRNA RNA-Polymerase Cap-Struktur monocistronische mRNA Poly(A)-Schwanz Kernpore Translation noch während der Transkription mRNA muss aus dem Kern ausgeschleust werden 14 Die drei durch die Transkription erzeugten Haupttypen von RNA-Molekülen Brown 6.1 15 Eukaryoten: drei DNA-abhängige RNA-Polymerasen im Kern RNA-Polymerase Produkt Lokalisierung RNA-Pol I: rRNA (28S, 18S, 5,8S) Nukleolus RNA-Pol II: mRNA (Protein-kodierende Gene) snoRNAs, einige snRNAs Nukleoplasma RNA-Pol III: 5S rRNA, tRNAs, viele snRNAs interne Promotoren ! Nukleoplasma Zusätzlich RNA-Polymerasen in Mitochondrien und Chloroplasten mt-RNA-Pol (nur eine UE) mitochondriale Transkripte Mitochondrien (kernkodiert) pt-RNA-Pol (PEP) (E.coli-ähnlich) plastidäre Transkripte Plastiden (plastidenkodiert) pt-RNA-Pol (NEP) (nur eine UE) plastidäre Transkripte Plastiden (kernkodiert) 16 Eukaryotische Promotoren sind weniger konserviert prokaryotischer Promotor eukaryotischer RNA-Pol II Promotor Jannig & Knust 14.3 17 Struktur und Transkription eines eukaryotischen Gens Purves et al. 14.4 18 Transkription eukaryotischer DNA durch die RNA-Polymerase II benötigt viele allgemeine Transkriptionsfaktoren (TF) TFIIH 19 Eukaryotische Gene enthalten kodierende Exon- und nicht-kodierende Intron-Bereiche, eukaryotische RNA-Pol II-Transkripte werden prozessiert 1. 2. 3. Anfügen des Cap am 5‘ Ende Polyadenylierung am 3‘ Ende Spleißen Alle drei Prozesse finden im Zellkern statt ! 20 „Capping“ des Transkriptes am 5‘ Ende: Anfügen eines modifizierten Guaninnukleotids Methylgruppe • 5‘ Cap signalisiert 5‘ Ende eukaroytischer mRNAs •Wird während der Transkription angehängt • 5‘ Cap wichtig für Export der mRNA ins Cytosol und die Translation 5‘-5--Bindung 7-Methyl-Guanosintriphosphat 21 Polyadenylierung am 3‘-Ende • 3‘-Poly(A)-Schwanz signalisiert 3‘-Ende eukaroytischer mRNAs • Poly(A)-Polymerase (Polymerisation ohne Matrize !) • 200-250 A-Nukleotide werden angehängt • 3‘-Poly(A)-Schwanz wichtig für Export der mRNA ins Cytosol, für die Stabilität und die Translation 10- 20 Nukleotide < 30 Nukleotide Spaltung durch Endonukleasekomplex wird abgebaut Poly(A)-Anheftung durch Poly(A)-Polymerase 22 Spleißen des Transkripts Spleißen = Entfernen der Intronsequenzen aus dem Primärtranskript, Verknüpfung der Exons • Nukleotidsequenzen markieren die Spleißstellen • Spleißen wird durch Spleißosomen ausgeführt • Spleißosomen sind aus Proteinen und snRNAs zusammengesetzt Intron wird entfernt Teil der mRNA 23 Janning & Knust 14.9 Das Spleißosom, eine Spleißmaschine 24 Purves et al. 14.10 Eukaryoten: Kontrolle der Transkription durch die Chromatinstruktur Modifikation (z. B. Acetylierung) der Chromatinproteine (Histone) reguliert die Transkription Nukleosom 10 nm DNA Histonoktamer HistonDeacetylierung Histon-Deacetylase (HDAC) offenes Chromatin -> transkriptionsaktiv HistonAcetylierung Histon-Acetyltransferase (HAT) kondensiertes Chromatin -> transkriptionsinaktiv 30 nm Janning & Knust 17.16 25 Histon-Acetyltransferase (HAT) Reguliert Zugänglichkeit der DNA für Proteine der Transkriptionsmaschinerie Histon-Acetyltransferase (HAT) acetyliert Histone in der Nähe der TATA-Box RNA-Polymerase II Transkriptionsfaktor Transkription Histone DNA Ac Nukleosom 26 Modifikation des Chromatins HistonDeacetylierung Histon-Methylierung DNA-Methylierung HistonAcetylierung Histon-Demethylierung DNA-Demethylierung 27 Initiation der Genexpression: Unterschiede zwischen Pro- und Eukaryoten Prokaryoten Eukaryoten eine RNA-Polymerase, einige Sigma-Faktoren drei RNA-Polymerasen, viele allgemeine Transkriptionsfaktoren ja ja; zahlreich Transkript meist polycistronisch, keine Modifikation monocistronisch, Modifikation am 5‘- u. 3‘-Ende Introns i.d.R. nicht vorhanden, kein Spleißen meist vorhanden, Spleißen nein ja (Kern/Cytoplasma) kein Chromatin! ja RNA-Polymerase regulatorische Transkriptionsfaktoren Trennung von Transkription u. Translation Einfluss der Chromatinstruktur 28 Die drei durch die Transkription erzeugten Haupttypen von RNA-Molekülen Brown 6.1 29 Ribosomale RNA Ribosomen bestehen aus RNA (rRNA) und Proteinen und sind aus zwei Untereinheiten zusammengesetzt Purves et al. 12.9 30 Ribosomen 31 Svedbergeinheit S •Maß für Sedimentationsgeschwindigkeit bei Zentrifugation in einem Dichtegradienten •S-Wert abhängig von Größe, Form, Volumen und Dichte des Partikels/Moleküls •je größer und kompakter, desto größer ist Wanderungsgeschwindigkeit Zellfraktionierung durch differentielle Zentrifigation, Sedimentationsanalyse oder Dichtegradientenzentrifugation (Saccharose) 32 Dichten und S-Werte von Zellmaterial 33 Zusammensetzung der Ribosomen bei Pro- und Eukaryoten Prokaryoten Eukaryoten Größe rRNA (Nukleotide) Proteine Größe rRNA (Nukleotide) Proteine große UE 50S 23S rRNA (2904) 5S rRNA (120) L1, L2, L3, etc. Ges.: >30 60S 28S rRNA (4818) 5,8S rRNA (160) 5S rRNA (120) L1, L2, L3, etc. Ges.: ca. 50 kleine UE 30S 16S rRNA (1542) S1, S2, S3, etc. Ges.: >20 40S 18S rRNA (1874) S1, S2, S3, etc. Ges.: ca. 35 34 Molekulare Feinstruktur eines 70S Ribosoms Purves et al. 12.9 b 35 Sekundärstruktur der 16 S rRNA von E. coli 36 Prozessierung der eukaryotischen rRNA Janning & Knust 15.1e 37 DNA, die rRNA kodiert ist repetitiv Purves et al. 14.2 38 Nukleolus •Kernkompartiment •Ort der rRNA Synthese •Nukleolus besteht aus DNA, RNA und Proteinen •Prozessierung der prä-rRNA, Zusammensetzung präribosomaler Partikel Nukleolus-Organisator (NO) besteht aus rDNA, die von mehreren Chromosomen stammen kann und tandemartig abgeordnete rDNA enthält 39 tRNAs fungieren als Adapter (400 000 tRNA Moleküle pro Bakterien-Zelle) 40 •tRNAs bestehen aus 74 – 95 Nukleotiden •Kleeblattstruktur •Akzeptorarm bindet Aminosäure; 3‘ Ende endet immer auf -CCA •DHU-Arm enthält ungewöhnliches Pyrimidin Dihydrouracil •Anticodonarm erkennt mRNA •Variabler Arm enthält variable Anzahl an Nukleotiden •TψC enthält die Abfolge T, Pseudouracil und C 41 Tertiärstruktur der tRNA jede tRNA hat individuelle 3D-Struktur 42 Uridin Zucker 43 Der genetische Code 44 Die 20 in Proteinen vorkommenden Aminosäuren 45 Janning & Knust 15.3 Problem: 4 Buchstaben A,T,G,C -> aber 20 Aminosäuren Singulet-Code: 4 Codons Duplett-Code: 42 = 16 Codons Triplett-Code: 43 = 64 Codons Purves et al. 12.5 46 Frage: Welches Triplett codiert für welche Aminosäure? Versuch von Nirenberg und Matthaei Purves et al. 12.6 47 Der Code ist degeneriert, d.h. mehrere Tripletts codieren eine Aminosäure (Ausnahme: Tryptophan und Methionin) Synonyme Codons sind sich meist ähnlich, so dass die ersten beiden Basen eines Tripletts oft schon die Aminosäure spezifizieren (Ausnahmen: Leucin und Arginin) Leucin Leucin Arginin Arginin 48 Purves et al. 12.5 Die "Wobble"-Hypothese (F. Crick 1965) "wobble" = "Schwanken, Wackeln" Eine einzelne z. B. mit Glycin beladene tRNA kann drei verschiedene Codons auf der mRNA erkennen 49 "Wobble" Abweichungen bei der Bindung des Anticodons an das Codon 50 Der genetische Code ist fast universell und gilt für alle Organismen Es gibt wenige Ausnahmen (insbesondere in MitochondrienGenomen) z.B. UGA (normalerweise Stop) in Mitochondrien Tryptophan und AUA (normalerweise Isoleucin) in Mitochondrien Methionin 51 Verwendung von Code-Wörtern (Codon usage) Ein Beispiel: 6 Arginin Codons CGT 43% CGC 32% CGA 7% CGG 8% AGA 9% AGG 1% korrespondiert mit Vorkommen synonymer tRNAs d.h. es gibt seltene Codons (Speziesspezifisch) Regulation von Genaktivität, da seltene Codons zu schwächerer Expression führen 52