2005-3-Schwerpunkt Epigenetik.qxd
Werbung

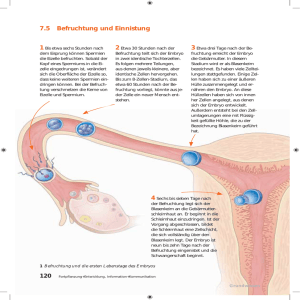
Thomas Haaf Epigenetik Epigenetische Genomreprogrammierung im frühen Säugerembryo: Mechanismen und Pathologie Institut für Humangenetik Johannes GutenbergUniversität Mainz Zusammenfassung Die beiden hochmethylierten gene tisch inaktiven Genome aus der väter lichen und mütterlichen Keimbahn müssen nach der Befruchtung für die somatische Entwicklung des neuen Organismus reprogrammiert werden. Bei der Maus und den meisten ande ren Säugetierspezies (einschließlich des Menschen) wird der väterliche Vorkern von der befruchteten Eizelle innerhalb weniger Stunden drastisch demethyliert. Das mütterliche Genom ist vor dieser aktiven Demethylierung geschützt und wird erst ab dem Zwei zellstadium durch einen replikations abhängigen Mechanismus schritt weise passiv demethyliert. Elternspe zifische epigenetische Modifikationen bedingen im frühen Säugerembryo (bis etwa zum Morulastadium) eine Genomasymmetrie, die als zytologi sches Korrelat eines Geschlechter konfliktes bei der Programmierung des neuen diploiden somatischen Ge noms interpretiert werden kann. Die aktiven und passiven Demethylie rungswellen sind wahrscheinlich not wendig, um die beiden Keimbahnge nome, insbesondere das extrem kon densierte Spermienchromatin für die somatische Entwicklung „wiederzube leben“ und bereits im frühen Embryo von der maternalen zur embryonalen Kontrolle der Entwicklung überzu wechseln. Reprogrammierungsstö rungen sind eine wichtige Ursache für den Verlust von Embryonen/Schwan gerschaften und abnormale Phänoty pen. Häufigkeit und Schwere der epi genetischen Defekte nehmen zumin dest im Tierexperiment zu, wenn man mit artifiziellen Reproduktionstechni ken (ART) in die Entwicklung von Ga meten und/oder frühen Embryonen eingreift oder durch den Transfer von somatischen Zellkernen in Eizellen bei der Embryoklonierung die Keimbahn umgeht. Ein besseres Verständnis des epigenetischen Reprogrammierungs prozesses in der befruchteten Eizelle und im frühen Embryo und von even tuell modifizierenden genetischen und Umweltfaktoren ist sowohl für eine Therapie von infertilen Paaren, als auch für die Herstellung menschlicher embryonaler Stammzelllinien für die Zellersatztherapie wichtig. Obwohl ART beim Menschen in grossem Um fang angewandt werden, ist die Epi genetik der Reproduktion ein bisher vernachlässigtes Forschungsgebiet. Schlüsselwörter DNAMethylierung, Genomrepro grammierung, Imprinting, Präimplan tationsphase, Schwangerschaftsver lust Abstract After fertilization the two highly methylated genetically inactive genomes from the male and female germ lines must be reprogrammed for somatic development of the new organism. In mouse and most other mammalian species (including hu mans), the paternal genome is drastically demethylated within a few hours of fertilization in the fertilized egg. The maternal genome is pro tected from this active demethylation and gradually passively demethylated by a replicationdependent mecha nism after the twocell embryo stage. Parentspecific epigenetic modifica tions in the early mammalian embryo are associated with genome asym metry, which may be considered as cytological correlate of a parental conflict regarding programming of the new diploid somatic genome. The active and passive demethylation waves may be necessary to “revive” the two germ line genomes, in par ticular the highly condensed sperm chromatin for somatic development and for an early transition from maternal to embryonal control of development. Reprogramming distur bances may contribute to embryo/ pregnancy loss and abnormal pheno types. At least in animal experiments the frequency and severity of epi genetic defects increase after inter fering with development of gametes and/or early embryos by artificial reproductive technologies (ART) or when bypassing the germ line by transfer of somatic cell nuclei into oocytes during embryo cloning. A better understanding of the epi genetic reprogramming process in the fertilized egg and the early embryo and of possible genetic modifiers and environmental factors is crucial for improving human infertility treatment and/or the generation of human embryonal stem cell lines for cell replacement therapy. Although ART are widely applied in humans, re search on epigenetics of reproduction has been neglected so far. Key words DNA methylation, genome repro gramming, imprinting, pregnancy loss, preimplantation period medgen 17 (2005) 275 Epigenetik Abb 1 Elternspezifische Methylierungs reprogrammierung im frühen Säugerembryo Bild oben Methylcytosinfärbung (mit einem FITCkonjugier ten grünfluoreszierenden antiMethylcytosin Antikörper) von einzelligen Mausembryonen (Vorkernstadien) 1, 3 und 8 Std. nach der Be fruchtung, sowie einem zweizelligen Embryo 26 Std nach Befruchtung. Die KernDNA ist mit DAPI (blau) gegengefärbt. Die im väterlichen und mütterlichen Vorkern räumlich getrennten Keim bahngenome weisen zunächst beide einen rela tiv hohen Methylierungsgrad auf. Der (bei der Maus) etwas größere väterliche Vorkern wird je doch innerhalb weniger Stunden drastisch de methyliert und verliert seine grüne Methylcyto sinfärbung. Nach Vorkernverschmelzung und er ster Zellteilung zeigen die Kerne von Zweizellem bryonen eine bereits demethylierte väterliche Kernhälfte und eine noch komplett methylierte mütterliche Kernhälfte. Einleitung Seit den bahnbrechenden Vorkern transferexperimenten in den frühen 1980er Jahren (Surani et al., 1984) wissen wir, dass im Gegensatz zu nie deren Vertebraten bei Säugetieren so wohl ein väterliches als auch ein müt terliches Genom für eine normale Ent wicklung benötigt werden. Aufgrund von (reversiblen) epigenetischen Mo difikationen in der väterlichen und mütterlichen Keimbahn sind die bei den elterlichen Genome, die in der befruchteten Eizelle zusammenkom men, funktionell nicht gleichwertig. Die genomische Prägung (engl. „Im printing“) in der elterlichen Keimbahn ist dafür verantwortlich, dass be stimmte Gene während der Entwick lung und/oder im ausgewachsenen Organismus nur von den väterlichen oder mütterlichen Chromosomen ex primiert werden (Lucifero et al., 2004). Imprintingmechanismen wurden bei Säugetieren wahrscheinlich entwi ckelt, um die unterschiedlichen elter lichen Interessen bezüglich der Be reitstellung von maternalen Res sourcen für das neue Individuum zu regulieren („Geschlechterkonflikt hypothese“). Väterlich exprimierte Gene tendieren dazu, das Embryo wachstum zu steigern, während müt terlich exprimierte Gene das Wachstum soweit reduzieren, dass auch weitere Schwangerschaften (eventuell mit anderen Vätern) mög lich sind (Moore und Haig, 1991). 276 medgen 17 (2005) Für die zeitlich, räumlich und eltern spezifisch koordinierte Genexpression ist die Methylierung von CpGDinu kleotiden von entscheidender Bedeu tung. Die DNAMethylierung ist ein epigenetischer Mechanismus, der eine posttranslationelle Modifikation von Histonen zur Etablierung und/ oder Aufrechterhaltung einer inaktiven Chromatinstruktur bewirkt (Jaenisch und Bird, 2003). Die Zelltypspezifi schen Methylierungsmuster in soma tischen Zellen sind in der Regel stabil und werden bei der Zellteilung weiter vererbt. Im Gegensatz dazu finden nach der Befruchtung dramatische Veränderungen der DNAMethylierung der beiden elterlichen Genome statt. Man nimmt an, dass diese genom weite Methylierungsreprogrammie rung für die Wiederherstellung der To tipotenz von embryonalen Zellen not wendig ist (Reik et al., 2001). Die befruchtete Eizelle hat das Poten tial, einen neuen Organismus zu bil den. Für die Ausbildung des neuen diploiden somatischen Genoms müs sen aber die beiden sehr unterschied lichen Keimbahngenome erst neu programmiert werden. Die aus der vä terlichen bzw. mütterlichen Keimbahn ererbten elternspezifischen Modifika tionen von DNA und Chromatin wer den nach der Befruchtung nahezu komplett entfernt und in beiden elter lichen Allelen werden identische neue somatische Methylierungsmuster ge setzt. Nur die relativ wenigen elterlich Bild unten Schematische Darstellung der Methylierungsre programmierung von väterlichem (blau) und müt terlichem (rot) Genom im frühen Mausembryo. Das aus dem Spermium kommende väterliche Genom wird von der befruchteten Eizelle aktiv demethyliert. Das mütterliche Genom wird erst ab dem Zweizellstadium schrittweise durch ei nen replikationsabhängigen Mechanismus de methyliert, d.h. mit jeder DNAVerdoppelung und Zellteilung geht die Hälfte der Methylgruppen verloren. Im Morulastadium (der Maus) sind dann beide elterlichen Genome maximal demethyliert und es werden neue somatische Methylierungs muster gesetzt. Durch die genomweiten Deme thylierungs und Remethylierungswellen werden die Keimbahnunterschiede zwischen väterlichen und mütterlichen DNASequenzen weitgehend beseitigt. Nur relativ wenige geprägte Gene ent gehen diesem Reprogrammierungsprozess im frühen Embryo und behalten ihre differentiellen Keimbahnmethylierungsmuster bei. geprägten Gene (man schätzt 100 200 der etwa 30.000 Säugergene) sind durch einen unbekannten Me chanismus vor diesem Reprogram mierungsprozess geschützt und be halten ihre elternspezifischen Keim bahnmuster während der gesamten weiteren Entwicklung bei. Aktive zygotische Demethylierung des väterlichen Genoms in der befruchteten Eizelle Das Spermium bringt nur das väterli che Genom in die befruchte Eizelle, die neben dem mütterlichen Genom auch die gesamte Reprogrammie rungsmaschinerie bereitstellt. Die sehr frühe embryonale Entwicklung ist also fast vollständig unter mater naler Kontrolle. Durch die Immunfluo reszenzfärbung mit einem Antikörper gegen 5Methylcytosin kann die akti ve Demethylierung des väterlichen Genoms durch die Eizelle direkt sicht bar gemacht werden (Mayer et al., 2000; Santos et al., 2002). Interpre tiert man diesen Befund im Sinne der Epigenetik Geschlechterkonflikthypothese (Moo re und Haig, 1991), versucht die Eizel le, d.h. das maternale Genom aus der väterlichen Keimbahn ererbte epige netische Informationen weitgehend auszulöschen. Das Protaminverpackte Spermienge nom und die mütterlichen Meiose II Chromosomen bilden in der Zygote zwei räumlich getrennte Vorkerne. Das reziproke Produkt der zweiten meioti schen Teilung wird mit dem zweiten Polkörperchen erst nach der Befruch tung ausgestoßen. Sowohl väterliches als auch mütterliches Genom sind zu nächst hochmethyliert (Abb. 1, Zygo ten 1 und 3 Std. nach Befruchtung), weisen aber auf DNASequenzebene elternspezifische (keimbahnspezifi sche) Unterschiede in den Methylie rungsmustern auf. Bei der Maus und den meisten anderen Säugetierspezies wird innerhalb weniger Stunden nach Befruchtung der väterliche Vorkern drastisch demethyliert (Abb. 1, Zygote 8 Std. nach Befruchtung). Da diese Demethylierung sehr rasch und noch vor der ersten DNAReplikation statt findet, muss es sich um einen aktiven Prozess handeln. Über die zugrunde liegende enzymatische Aktivität kann bisher nur spekuliert werden. Es ist möglich, dass die Methylcytosine durch eine Art Basenexcisionsrepara tur gegen Cytosine ausgetauscht wer den oder die Methylgruppen von der DNA abgespalten werden. Bei gleichzeitiger Befruchtung einer Eizelle durch mehrere Spermien und in androgenetischen Embryonen wer den alle väterlichen Genome deme thyliert, während zusätzliche weibli che Genome in gynogenetischen und parthenogenetischen Embryonen me thyliert bleiben (Barton et al., 2001; Santos et al., 2002). Dies macht einen Zusammenhang zwischen aktiver De methylierung und Remodellierung des Spermienchromatins in der Eizelle wahrscheinlich. Das hochkondensier te, beinahe kristalline Spermienchro matin muss nach der Befruchtung de kondensiert und die Protamine müs sen gegen Histone ausgetauscht wer den. Während dieses ProtaminHi stonAustausches ist die väterliche DNA „nackt“ und bietet im Gegensatz zur nukleosomal verpackten mütter lichen DNA eine ideale Angriffsfläche für eine maternale Demethylierungs aktivität. Auf der anderen Seite könn te die zygotische Demethylierung des paternalen, aber nicht des materna len Genoms ihre Ursache in unter schiedlichen Histonmodifikationen von väterlichem und mütterlichem Chromatin haben. Der väterliche Vor kern in der Zygote zeigt eine transien te Hyperacetylierung von Histon H4 (Santos et al., 2002). Das mütterliche Genom ist präferentiell mit dem me thylierten Histon H3 assoziiert (Cowell et al., 2002). Die aktive zygotische Demethylierung des väterlichen Genoms ist bei Maus, Ratte, Schwein, Rind und Mensch konserviert (Mayer et al., 2000; Dean et al., 2001), was für ihre funktionelle Bedeutung spricht. Es gibt jedoch wichtige Speziesunterschiede. So scheint die Demethylierungsaktivität in Mauseizellen größer zu sein als in Rindereizellen (Beaujean et al., 2004). Es gibt auch Spezies, wie z.B. Schaf und Kaninchen, die zumindest auf zy tologischer Ebene überhaupt keine detektierbare Demethylierung des vä terlichen Vorkerns zeigen (Shi et al., 2004). Es wird vermutet, dass aktive Demethylierungsmechanismen eine frühe Aktivierung des embryonalen Genoms ermöglichen. Während bei der Maus das embryonale Genom be reits ab dem Zweizellstadium transkri biert wird, läuft beim Kaninchen fast die gesamte Präimplantationsentwik klung unter mütterlicher Kontrolle ab. Passive Demethylierung des maternalen Genoms Der hohe Methylierungsgrad des müt terlichen Genoms bleibt in der Maus eizelle bis zum Zweizellstadium erhal ten. Selbst nach Auflösung der Vor kernmembranen im späten Einzell Embryo und der ersten mitotischen Teilung bleiben die beiden elterlichen Genome räumlich getrennt. Die Kerne von MausZweizellEmbryonen zeigen eine hochmethylierte mütterliche und eine demethylierte väterliche Kern hälfte (Abb. 1, ZweizellEmbryo 26 Std. nach Befruchtung). Diese Genomse parierung löst sich erst ab dem Vier zellstadium schrittweise auf. In diesem Zusammenhang ist es inter essant, anzumerken, dass die be fruchtete entwicklungsfähige mensch liche Eizelle ab dem Zeitpunkt der Kernverschmelzung als Embryo im Sinne des Embryonenschutzgesetzes (1990) gilt und deshalb eine Präim plantationsdiagnostik in Deutschland nicht möglich ist. Die Genomasymme trie im ZweizellEmbryo und auch noch späteren Stadien macht aber deutlich, dass das neue diploide so matische Genom nicht einfach mit Auflösung der Vorkernmembranen durch eine simple Durchmischung von väterlichen und mütterlichen Chromo somen entsteht, sondern dass die bei Befruchtung sehr unterschiedlichen elterlichen Genome in einem Repro grammierungsprozess erst aneinander angeglichen werden müssen. Die Be seitigung der aus der männlichen und weiblichen Keimbahn ererbten Methy lierungsmuster (mit Ausnahme der dif ferentiell methylierten Regionen von geprägten Genen) ist beim Rind im 8 16Zellstadium, bei der Maus sogar erst in der Morula abgeschlossen (Reik et al., 2001) (Abb. 1, Graphik). Deshalb ist es auch frühestens ab die sem Zeitpunkt sinnvoll, vom somati schen Genom des neuen Organismus zu sprechen. Das mütterliche Genom wird ab dem Zweizellstadium graduell durch einen replikationsabhängigen Mechanismus demethyliert. Weil Methylcytosin nicht direkt in die replizierende DNA einge baut werden kann, erfordert die Auf rechterhaltung der Methylierungsmu ster DNAMethyltransferase 1 (DNMT1), die eine hohe Affinität für bei der Re plikation gebildete hemimethylierte DNA besitzt. Dieses Enzym erkennt methylierte CpGs im parentalen DNA Strang und heftet neue Methylgrup pen an die komplementären Stellen im neusynthetisierten Strang an. In frühen Embryonen ist die oocyten spezifische Isoform von DNMT1 im Zytoplasma separiert und kann nicht an hemimethylierte DNA im Zellkern binden (Cardoso und Leonhardt, 1999). D.h. mit jeder DNAReplikation und Zellteilung geht die Hälfte der Methylgruppen verloren. Im 1632 Zellstadium sind väterliches und müt terliches Genom gleichermaßen und maximal demethyliert. Soweit be medgen 17 (2005) 277 Epigenetik kannt, scheinen nur die keimbahn spezifischen Methylierungsmuster von geprägten Genen die sukzessive aktive und passive Demethylierung in der Präimplantationsphase zu überle ben. Im Blastozystenstadium der Maus (beim Rind bereits etwas früher) findet dann eine genomweite Reme thylierung statt und es werden auf vä terlichen und mütterlichen Chromoso men identische somatische Methylie rungsmuster erzeugt (Abb. 1, Gra phik). Nur „housekeeping genes“ und Gene, die im frühen Embryo aktiv sind, bleiben demethyliert und kön nen transkribiert werden. Störungen der Methylierungsreprogrammierung Die elternspezifische Methylierungs reprogrammierung im frühen Säuger embryo ist ein zeitlich und räumlich hochkoordinierter und damit auch störungsanfälliger Prozess, der mit der Ausbildung eines neuen diploiden somatischen Genoms einhergeht. Ge nomweite Reprogrammierungsstörun gen führen bereits in der Präimplan tationsphase zum Embryoverlust (Shi und Haaf, 2002). Superovulierte Mausweibchen produzieren etwa dop pelt so viele (1420%) Embryonen mit abnormalen Methylierungsmustern, die sich nicht zur Blastozyste ent wickeln, wie nichtsuperovulierte Weibchen (510%). Die hormonelle Stimulation verschlechtert offenbar Eizell und/oder Embryoqualität. Die Kultivierung von in vitro fertilisierten (IVF) Mausembryonen in verschiede nen Medien, d.h. die Umwelt zum Zeitpunkt der Befruchtung oder kurz danach, hat ebenfalls großen Einfluss auf die Methylierungsreprogrammie rung. Acetaldehyd, das Abbauprodukt von Alkohol und wahrscheinlich auch zahlreiche andere toxische Substan zen können die Häufigkeit von ge nomweiten Reprogrammierungsde fekten dramatisch steigern. Die Zygo te und der frühe Embryo, in denen genomweite Reprogrammierungspro zesse ablaufen, sind vor allem bei ART schlecht vor Einflüssen aus der Umwelt geschützt. Die Präimplanta tion ist deshalb eine äußerst sensitive Phase in der Entwicklung eines Orga nismus, was die Entstehung von epi genetischen Defekten betrifft. Die Ef fizienz der Methylierungsreprogram 278 medgen 17 (2005) mierung hängt aber nicht nur von Umwelt, sondern auch von geneti schen Faktoren ab. Die Embryos ver schiedener Mausstämme zeigen vor allem unter suboptimalen Embryokul turbedingungen sehr unterschiedliche Raten von genomweiten Methylie rungs und Entwicklungsstörungen. Embryonen mit zytologisch detektier baren abnormalen Methylierungsmu stern werden bereits in der Präim plantationsentwicklung arretiert („Al les oder NichtsRegel“). Wenn aber nicht die Reprogrammierung des ge samten Genoms gestört ist, sondern nur einzelne Gene betroffen sind, ist dies mit einer weiteren Entwicklung vereinbar und kann zu abnormalen fe talen Phänotypen und „Imprinting krankheiten“ führen. Eine Reihe von Studien bei Rindern, Schafen und an Mäusen hat eindrucksvoll gezeigt, dass die Isolation, Manipulation und/oder Kultur von Keimzellen und frühen Embryonen die Methylierungs muster und Regulation von geprägten Genen beeinflussen und mit phänoty pischen Auffälligkeiten einhergehen können (Lucifero et al., 2004). Kinder, die durch IVF oder intrazytoplasmati sche Spermieninjektion (ICSI) gezeugt wurden, zeigen eine erhöhte Präva lenz von einigen sehr seltenen Imprin tingkrankheiten, wie z.B. Beckwith Wiedemann oder AngelmanSyn drom (Ludwig et al., 2005). Der zu grundeliegende epigenetische Defekt scheint eine abnormale Hypomethy lierung von normalerweise methylier ten mütterlichen Allelen zu sein. Bis her ist allerdings unklar, ob Imprin tingdefekte und elterliche Infertilität eine gemeinsame Ursache haben oder ob ART an sich einen Risikofak tor darstellen. Mausexperimente las sen vermuten, dass die hormonelle Stimulation der Ovarien zur Eizellge winnung in den Imprintingkontroll zentren einiger geprägter Gene zum Verlust der Methylierung führen kann (Alexandridis et al., 2005). Die epigenetischen Defekte klonierter Säugerembryonen sind sehr viel gra vierender und häufiger, unterscheiden sich aber wahrscheinlich nicht prinzi piell von denen in normal oder artifi ziell gezeugten Embryonen. Die Klo nierungseffizienz variiert von einer Säugetierspezies zur anderen, ist je doch generell sehr niedrig (Solter, 2000). Dies darf vermutlich auf Unter schiede in der epigenetischen Ge nomreprogrammierung zurückgeführt werden. In der Methylcytosinfärbung zeigten klonierte Rinderembryonen eine inkomplette und verzögerte Me thylierungsreprogrammierung des di ploiden Spendergenoms durch die Ei zelle (Dean et al., 2001). Daraus resul tiert ein Gemisch von normal und ab normal methylierten Sequenzen. Chipbasierte Expressionsanalysen von >10.000 Genen in Leber und Pla centa von neugeborenen klonierten Mäusen ergaben mindestens 4% ab normale Genexpressionsmuster (Humpherys et al., 2002). Über 95% der klonierten Mausblastozysten wie sen Störungen der allelspezifischen Methylierung und Expressionsmuster der geprägten Gene auf (Mann et al., 2003). Die erfolgreiche reproduktive Klonierung von rund einem Dutzend Säugetierspezies zeigt zwar, dass die Eizelle in einem transplantierten so matischen Zellkern die Totipotenz wiederherstellen kann, aber die kor rekte epigenetische Reprogrammie rung aller Gene für eine somatische Entwicklung scheint wohl nicht mög lich zu sein. Stochastisch im Genom auftretende Reprogrammierungsde fekte sind für die extrem hohe Schwangerschaftsverlustrate und die abnormalen Phänotypen von klonier ten Tieren (mit) verantwortlich. Literatur Alexandridis A, Großmann B, Konersmann M, Haaf T, Zechner U (2005) The impact of ovarian stimulation on imprinted gene methylation in preimplantation stage mouse embryos. Med Ge netik 17:53. Barton SC, Arney KL, Shi W, Fundele R, Surani MA, Haaf T (2001) Genomewide methylation patterns in normal and uniparental early mouse embryos. Hum Mol Genet 10:29832987. Beaujean N, Taylor JE, McGarry M, Gardner JO, Wilmut I, Loi P, Ptak G, Galli C, Lazzari G, Bird A, Young LE, Meehan RR (2004) The effect of interspecific oocytes on demethylation of sperm DNA. Proc Natl Acad Sci USA 101:76367640. Cardoso MC, Leonhardt H (1999) DNA methyl transferase is actively retained in the cytoplasm during early development. J Cell Biol 147:2532. Epigenetik Cowell IG, Aucott R, Mahadevaiah SK, Burgoy ne PS, Huskisson N, Bongiorni S, Prantera G, Fanti L, Pimpinelli S, Wu R, Gilbert DM, Shi W, Fundele R, Morrison H, Jeppesen P, Singh P (2002) Heterochromatin, HP1 and methylation at lysine 9 of histone H3 in animals. Chromosoma 111:2236. Dean W, Santos F, Stojkovic M, Zakhartchenko V, Walter J, Wolf E, Reik W (2001) Conservation of methylation reprogramming in mammalian de velopment: aberrant reprogramming in cloned embryos. Proc Natl Acad Sci USA 98:13734 13738. Solter D (2000) Mammalian cloning: advances and limitations. Nat Rev Genet 1:199207. Korrespondenzadresse Prof. Dr. Thomas Haaf Institut für Humangenetik Johannes GutenbergUniversität Mainz 55101 Mainz Tel. 06131175790 Fax 06131175690 [email protected] Embryonenschutzgesetz (vom 13. Dezember 1990) Gesetz zum Schutz von Embryonen. BGBI. I S. 2746. Humpherys D, Eggan K, Akutsu H, Friedman A, Hochedlinger K, Yanagimachi R, Lander ES, Go lub TR, Jaenisch R (2002) Abnormal gene ex pression in cloned mice derived from embryonic stem cell and cumulus cell nuclei. Proc Natl Acad Sci USA 99:1288912894. Jaenisch R, Bird A (2003) Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat Genet 33:245254. Lucifero D, Chaillet JR, Trasler JM (2004) Poten tial significance of genomic imprinting defects for reproduction and assisted reproductive tech nology. Hum Reprod Update 10:318. Ludwig M, Katalinic A, Gross S, Sutcliffe A, Va ron R, Horsthemke B (2005) Increased prevalen ce of imprinting defects in patients with Angel man syndrome born to subfertile couples. J Med Genet 42, 289291. Mann MR, Chung YG, Nolen LD, Verona RI, La tham KE, Bartolomei MS (2003) Disruption of im printed gene methylation and expression in clo ned preimplantation stage mouse embryos. Biol Reprod 69:902914. Mayer W, Niveleau A, Walter J, Fundele R, Haaf T (2000) Demethylation of the zygotic paternal genome. Nature 403:501502. Moore T, Haig D (1991) Genomic imprinting in mammalian development: a parental tugofwar. Trends Genet 7:4549 Reik W, Dean W, Walter J (2001) Epigenetic re programming in mammalian development. Science 293:10891093. Santos F, Hendrich B, Reik W, Dean W (2002) Dynamic reprogramming of DNA methylation in the early mouse embryo. Dev Biol 241:172182. Shi W, Haaf T (2002) Aberrant methylation pat terns at the twocell stage as an indicator of ear ly developmental failure. Mol Reprod Dev 63:329334. Shi W, Dirim F, Wolf E, Zakhartchenko V, Haaf T (2004) Methylation reprogramming and chromo somal aneuploidy in in vivo fertilized and cloned rabbit preimplantation embryos. Biol Reprod 71:340347. Surani MA, Barton SC, Norris ML (1984) Deve lopment of reconstituted mouse eggs suggests imprinting of the genome during gametogenesis. Nature 308:548550. medgen 17 (2005) 279