prä-mRNA
Werbung
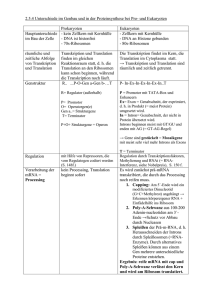
Genaktivierung und Genexpression Unter Genexpression versteht man ganz allgemein die Ausprägung des Genotyps zum Phänotyp einer Zelle oder eines ganzen Organismus. Genotyp: Gesamtheit der Informationen im Genom eines Organismus Phänotyp: Gesamtheit aller Merkmale eines Organismus, d.h. dessen Erscheinung Proteom: Gesamtheit aller Proteine eines Organismus Die Genexpression ist ein hochgradig regulierter Vorgang • Jede Zelle enthält jeweils das komplette Genom des Organismus • Damit sich der Organismus „normal“ entwickeln und jede Zelle ihre spezielle Funktion übernehmen kann, müssen bestimmte Proteine zum richtigen Zeitpunkt und in den richtigen Zellen in der richtigen Menge synthetisiert werden Unterschied zur Replikation: Replikation funktioniert nach dem Prinzip „Alles oder Nichts“ Genexpression ist hochgradig selektiv Wiederholung: Proteinbiosynthese Gen TRANSKRIPTION (mRNA) TRANSLATION (rRNA, tRNA) Proteinmolekül Regulierung der Genexpression Die Regulierung kann an mehreren Stellen der Proteinbiosynthese reguliert werden - vor der Transkription während der Transkription nach der Transkription, aber vor der Translation während der Translation nach der Translation Sinn: Gehirnzellen und Leberzellen enthalten jeweils die identischen Gene. Trotzdem unterscheiden sie sich massiv in ihrer phänotypischen Ausprägung Die Zellen einer Raupe und die Zellen eines Schmetterlings enthalten jeweils identische Gene. Trotzdem ist das Proteom beider Entwicklungsformen völlig unterschiedlich Bei der Genregulation wird festgelegt, in welcher Konzentration ein bestimmtes, synthetisiertes Protein in der Zelle vorliegen soll. bei Prokaryonten (und z.T. auch Protozoen) : Umweltanpassung bei Eukaryonten : zur Zelldifferenzierung - Zellentwicklung Die Genregulation gewährleistet , daß zum richtigen Zeitpunkt im richtigen Gewebe in den richtigen Zellen nur die jeweils notwendigen Gene aktiviert werden. Leberzellen Epithelzellen Stufen der Regulation der Genexpression Gen-Deaktivierung durch Chromatin-Umstrukturierung Durch „Verpackung“ der DNA können Gene für die Transkription unzugänglich gemacht werden. Nukleosomen hemmen die Initiation der Transkription Chromatin-Modeling Phasen der Transkription prä-mRNA (RNA-Kopie des Gens) Initiation der Transkription RNA-Polymerase muß auf dem DNA-Strang positioniert werden (bestimmtes Gen). Dazu ist ein Initiationskomplex notwendig, der den Anfangspunkt der Transkription determiniert. TF - Transkriptionsfaktoren ENHANCER PROMOTOR Im humanen Genom sind bisher etwa 775 verschiedene Promotoren bekannt. Sigma-Faktor TATA-Box Anlagerung eines TATAbindenden Proteins (TBP) und eines Initiationskomplexes Ergänzung durch weitere Komplexe, die u.a. auch eine Helikase (TFIIH) enthalten Anlagerung der RNA-Polymerase Initiierung der Translation und fortschreitende Elongation Abstoßen der I-Komplexe Beispiel: Regulation durch äußere Einflüsse Pflanzenzelle: Trockenheit wird signalisiert Exprimierung von speziellen Streßproteinen Die Gene, die diese Streßproteine codieren, liegen nicht hintereinander auf der DNA, sondern sind über das gesamte Genom verteilt SRE – stress response elements Das Ergebnis der Transkription ist eine prä-mRNA, welche sowohl Exon- als auch Intron-Sequenzen enthält. Zur Stabilisierung erfolgt ein Capping (5‘ -Ende) und ein Tailing (3‘ -Ende). Regulation nach der Transkription – Spleißen der prä-mRNA Exon – codierend Intron – nicht-codierend Vorgang des Spleißens – Das Spleisosom kleine nukleäre RibonucleoproteinPartikel - snRNP Das Ergebnis des Spleißvorgangs ist eine gereifte mRNA, die über die Kernporen in das ER ausgeschleust wird und dort als Matrize für die Translation dient, wodurch ein spezifisches Proteinmolekül entsteht. Regulationsvorgänge im Spleisosom – alternatives Spleißen Bei Säugetieren (und damit auch Menschen) wird das Protein Tropomyosin durch ein Gen mit 11 Exons codiert. Durch alternatives Spleißen können mRNA‘s für 5 verschiedene Formen des Proteins erzeugt werden ALTERNATIVES SPLEISSEN Editieren von mRNA Die mRNA kann auch post-transitional durch spezielle Enzyme verändert werden, was als RNA-Editing bezeichnet wird. Einfügen von Nucleotiden Verändern von Nucleotiden Prinzip: enzymatische Umwandlung von Cytosin in Uracil Hinzufügen von n U-Nucleotidfolgen Regulation der mRNA-Translation Ziel: Eingrenzung der Konzentration bestimmter Proteine im Cytoplasma Zwei Beispiele: 1. Modifizierung der Cap-Gruppe der mRNA – verhindert Translation 2. Eiweißkomplexe (z.B. Hämoglobin), die nicht abgesättigt sind (zu viele freie Bestandteile, z.B. Häm, im Cytoplasma), können die Globulin-Translation verstärken, bis die Häm-Konzentration wieder gesunken ist Prä-translatorische Regulation der „Haltbarkeit“ synthetisierter Proteine Werden Proteine durch „Anheften“ eines anderen, speziellen Proteins (Ubiquitin) markiert, dann werden sie durch einen speziellen Proteinkomplex (Proteasom) gebunden und hydrolisiert. Zusammenfassung Die Regulation der Genexpression ist ein äußerst komplexer und genau determinierter biochemischer Vorgang. Er stellt sicher, daß zu einem bestimmten Zeitpunkt immer nur die Proteine synthetisiert werden, welche die Eigenschaften (Phänotyp) der Zelle bestimmen und die zu dem Zeitpunkt gerade funktionell benötigt werden (z.B. Herstellung von Histon-Proteinen in der Phase vor der Zellteilung). Gene, die in jeder Zelle exprimiert werden, nennt man Haushalter-Gene. Die Regulation kann während der Transkriptionsphase, zwischen der Transkription und der Translation und nach der Translation der Proteinbiosynthese stattfinden. Beim Vorgang des alternativen Spleißens der prä-mRNA können aus einem Gen mRNA‘s für eine große Anzahl ähnlicher Proteine entstehen. Bei der Steuerung der Regulation spielen auch Signale, die von Außen über Rezeptoren in der Zellmembran in die Zelle gelangen, eine wichtige Rolle. Nächstes Mal: Signalübertragung und Kommunikation zwischen Zellen