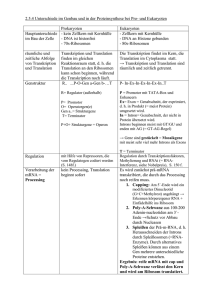
Grundlagen der Genetik und der Gentechnik
Werbung

aktuell | ernährungslehre & -praxis Nr. 9 September 2008 Im dritten Teil der Reihe zur Genetik und Gentechnik werden die molekularen Mechanismen der Proteinsynthese (= Genexpression) auf dem Weg vom Genpool zum funktionstüchtigen Organismus erläutert. Transkription und Translation sind dabei die zentralen zellulären Vorgänge, jedoch werden die jeweiligen Primärprodukte – RNA bzw. Proteine – auf vielfältige Weise weiter verändert („prozessiert“), bevor sie ihre zelluläre Funktion erfüllen können. ● Dr. Patricia Falkenburg Weidenweg 3 50259 Pulheim Grundlagen der Genetik und der Gentechnik Teil 3: Vom Gen zum Organismus – Genexpression ● Zur Einführung erinnern wir uns an das Glossar im zweiten Kapitel: „Das Genom eines Organismus ist die Gesamtheit seiner Erbinformation. Es umfasst sämtliche Gene dieses Organismus. Biochemisch handelt es sich um die DNA ...“ – „Das Genom ist organisiert auf Chromosomen. … Auf einem Chromosom sind viele Gene lokalisiert.“ – „Gene enthalten die Syntheseanleitung für Proteine.“ Außerdem wurde bereits besprochen, dass lebende Zellen zu einem großen Teil aus Proteinen bestehen und dass Proteine als Biokatalysatoren (Enzyme) sämtliche biochemischen Reaktionen des Organismus steuern. Vereinfachend kann man also sagen, dass die gesamte Biochemie lebender Organismen auf der Funktion der Proteine beruht. Wie wird nun die in Form von DNA (bzw. RNA) gespeicherte Erbinformation in Proteine übersetzt und wie wird dieser Prozess reguliert und gesteuert? Es gibt Gene, die praktisch immer transkribiert und exprimiert werden: die sog. Haushaltsgene (housekeeping genes). Sie unterliegen keinen Regulationsmechanismen, man spricht auch von konstitutiv exprimierten Genen. Unabhängig von Zelltyp, Zellstadium und äußeren Einflüssen müssen ihre Produkte in jeder Zelle ausreichend vorhanden sein. Dies betrifft typischer- weise Gene, die mit dem Grundstoffwechsel von Zellen zusammenhängen, z. B. mit dem Glukose-Stoffwechsel. Im Regelfall aber werden bestimmte Proteine gezielt zu bestimmten Zeiten von bestimmten Zellen bzw. in bestimmten Stoffwechselsituationen produziert. Bevor im nächsten Teil dieser Reihe diese Regulationsmechanismen anhand einiger Beispiele detaillierter betrachtet werden, ist es wichtig die grundlegenden Prozesse der zellbiologischen Proteinsynthese zu verstehen. Von der DNA zur RNA: die Transkription Die Transkription ist das zielgerichtete Kopieren eines bestimmten DNA-Abschnitts, bei der eine zu diesem DNAAbschnitt komplementäre RNA syn- thetisiert wird, wobei – wie bei der Replikation der DNA – einer der beiden Stränge der Doppelhelix als Matrize dient. Grundlage dieses Vorgangs ist – wie bei der DNA-Synthese – die Basenpaarung komplementärer Nucleotide. Abermals werden spezifische Enzyme benötigt, die RNA-Polymerasen, die die Reaktionen katalysieren. Auch bei der Transkription unterscheidet man drei Phasen, Initiation, Elongation und Termination. Allerdings dient nicht der gesamte DNAStrang als Matrize, sondern nur ein ganz bestimmter Abschnitt. Woher „wissen“ nun die beteiligten Enzyme, wo ein Gen anfängt und wo es aufhört? Die benötigten Informationen finden sich auf der DNA selbst, in ihrer Sequenz, in Form spezifischer, regulativ wirkender Elemente, die von Protei- Sinn und Antisinn Der kodogene DNA-Strang ist die Matrize für die RNA-Synthese. Er wird auch als antisense-Strang bezeichnet (engl. sense = Sinn), weil seine Sequenz zur Sequenz der entstehenden mRNA komplementär ist. Dementsprechend ist der nichtkodogene Strand der sense-Strang: seine Sequenz stimmt mit der Sequenz der mRNA überein. Eine RNA, deren Sequenz komplementär zur Sequenz des nichtkodogenen Strangs – und damit identisch mit der Sequenz des kodogenen Strangs – ist, ist eine antisense-RNA. Sie kann mit dem mRNA-Produkt des kodogenen Strangs Basenpaarungen eingehen, weil sie zu dieser komplementär ist. Dieser Effekt kann z. B. für gentechnische Zwecke genutzt werden! Ernährungs Umschau | 9/08 B33 aktuell | ernährungslehre & -praxis a) a l s de r bä r aus dem bau k am l ag t au au f dem heu b) a l s de r bä r aus dem bau k am l ag c) l s d e r b ä r a us d emb auk aml ag t aua u f d emh eu Abb. 1: Bedeutung der Tripletts des genetischen Codes am Beispiel eines Satzes aus Drei-Buchstaben-Wörtern: a) Vollständige Information; b) vorzeitige/r Abbruch/ Termination, es entsteht ein unvollständiges Produkt; c) Verschiebung des Leserasters bei Wegfall der ersten „Base“ – die Information geht komplett verloren. nen erkannt werden, die die Transkription ermöglichen (Aktivatoren) oder gegebenenfalls auch verhindern (Inhibitoren). So beginnt die Transkription stets an einem sog. Promotor, der als essenzieller Bestandteil der kodierenden Sequenz „stromaufwärts“ (upstream) vom zu transkribierenden Bereich liegt, also am 5’-Ende. Zusätzlich können räumlich weiter entfernte Sequenzen aktivierend (Enhancer) oder inhibierend (Silencer) auf die Genexpression wirken. Die RNA wird in 5’씮3’-Richtung synthetisiert, die Ableserichtung ist dementsprechend umgekehrt, von 3’ nach 5’. Die fertig transkribierten RNA-Moleküle werden in den meisten Fällen weiter prozessiert: z. B. durch Anhängen einer repetitiven Sequenz aus Adenosin-Nucleotiden (Poly[A]-Schwanz). Diese Prozessierung ist bei Eukaryonten 1. Base U ein hochkomplexer Prozess. Dem sog. Spleißen werden wir uns weiter unten nochmals zuwenden. Auch ribosomale und transfer-RNA werden in unterschiedlicher Weise „nachbehandelt“, bevor sie einsatzbereit sind. Die RNA-Typen, die im Rahmen der Transkription erzeugt werden, spielen alle eine Rolle in der Translation. Die ribosomale RNA (rRNA)ist wesentlicher struktureller und funktioneller Bestandteil der Ribosomen, der Enzymkomplexe, an denen die Translation abläuft. Die transfer-RNA (tRNA) dient der Übersetzung des „Vier-BuchstabenAlphabets“ der Nucleinsäuren in das „20-Buchstaben-Alphabet“ der Proteine. Und die messenger-RNA (mRNA)enthält die Bauanleitung für die zu synthetisierenden Proteine. 3. Base 2. Base U C A G Phe Ser Tyr Cys U C Stopp Leu C Leu Pro His Stopp A Trp G Arg U C Gln A G A Ile Thr Asn Ser U C Lys Arg Start/Met G Val A G Ala Asp Gly U C Glu A G Tab.1: Der genetische Code B34 Ernährungs Umschau | 9/08 Von der RNA zum Protein: die Translation „Translation“ bedeutet „Übersetzung“: Nucleinsäuren werden in Proteine „übersetzt“, indem durch nur vier unterschiedliche Nucleotide 20 Aminosäuren definiert werden. Dies geschieht mit Hilfe des genetischen Codes: Jeweils drei aufeinander folgende Basen (sog. Tripletts bzw. Codons) auf der mRNA definieren eine Aminosäure (쏆 Abbildung 1). Dieser genetische Code gilt nahezu universell: Alle Bakterien ebenso wie alle höheren Organismen – Pilze, Pflanzen und Tiere – benutzen ein identisches Übersetzungsalphabet und dies gilt auch für die Bakteriophagen und Viren, die ja die Proteinsynthesemaschinerie ihrer Wirtszellen nutzen. Aus diesem Grund können über gentechnische Methoden fremde Gene in einem Wirtsorganismus zur korrekten Expression gebracht werden. Niemals könnte menschliches Insulin von Bakterienzellen produziert werden, würden Mensch und Mikrobe nicht die gleiche „Translationssprache“ nutzen. Zugleich ist dieser universelle genetische Code ein wichtiges Indiz für die gemeinsame Abstammung aller Lebewesen von einem „Ur-Organismus“ im Verlauf der Evolution. Man bezeichnet den genetischen Code als degeneriert, weil die meisten Aminosäuren von mehreren Tripletts kodiert werden (쏆 Tabelle 1). Dabei bestimmen i. d. R. die ersten beiden Basen die Aminosäure, an der dritten Position ist dagegen Variabilität möglich (dies bezeichnet man als „wobbeln“ nach dem englischen Begriff „to wobble“, „wackeln“). Eine Sonderstellung nehmen die Codons UAA, UAG und UGA ein, die nicht für Aminosäuren kodieren, sondern vielmehr als sog. StoppCodons das Ende der Translation signalisieren. Das Codon AUG, das für Methionin kodiert, fungiert zugleich als Start-Codon für die Translation. Betrachtet man das „Übersetzungsraster“ des genetischen Codes, so wird leicht verständlich, dass der Austausch eines einzigen Nucleotids in der DNA gravierende Folgen für den Organismus haben kann: eine hypothetische Mutation, bei der das Codon UAU in das Codon UAA umgewandelt wird, wird dazu führen, dass die Proteinsynthese an dieser Stelle abbricht, statt für den Einbau von Tryptophan zu kodieren. Es wird kein funktionstüchtiges Protein mehr gebildet werden mit den entsprechenden unter Umständen katastrophalen Folgen für den Organismus (vgl. 쏆 Abbildung 1). Für die Übersetzung des Basen-TriplettCodes in die zugehörigen Aminosäuren wird die bereits erwähnte transfer-RNA benötigt. tRNAs sind hoch strukturierte kleine Moleküle mit einer charakteristischen Sekundär- (Kleeblatt) und Ter- 5´ C Transkription bei Prokaryonten CA 3´ Anticodon Abb. 2: Die tRNA bildet aufgrund einer ausgeprägten intramolekularen Basenpaarung eine typische komplexe Sekundärund Tertiärstruktur (vgl. Text). tiärstruktur (L-Form, 쏆 Abbildung 2). Das 3’Ende des tRNA-Moleküls bleibt einzelsträngig. Es endet in der Basenfolge CCA, an die durch die sog. Aminoacyl-tRNA-Synthetasen spezifisch die zugehörige Aminosäure gebunden wird. Am räumlich gesehen anderen „Ende“ des Moleküls befindet sich eine charakteristische Sequenz von sieben Aminosäuren mit dem sog. Anticodon in der Mitte. Das Anticodon ist komplementär zu dem Triplett, das für die Aminosäure dieser tRNA kodiert. Eine tRNA, die die Übersetzung des Tripletts UGG ermöglicht, hat also die Anticodonsequenz ACC und die Aminoacyl-tRNASynthetase wird an ihr CCA-Ende Tryptophan anheften. Auch bei der Translation werden Initiations-, Elongations- und Terminationsphase unterschieden. Alle diese Prozesse laufen an und mithilfe der Ribosomen ab. Diese Aggregate aus unterschiedlichen Proteinen und RNA katalysieren die vielschichtigen Schritte der Proteinbiosynthese – Tripletterkennung, Bindung der jeweiligen tRNA, Knüpfen der Peptidbindung zwischen Kettenende des entstehenden Proteins und neu hinzugekommener Aminosäure an der tRNA, Freisetzen der tRNA usw. Mehrere Ribosomen können gleichzeitig an eine mRNA binden und an dieser entlang wandern. So entstehen hochmolekulare sog. Polysomenkomplexe, die frei im Zytoplasma vorliegen oder membranassoziiert sein können (raues endoplasmatisches Retikulum). Zur detaillierteren Betrachtung der Vorgänge an den Ribosomen sei auf entsprechende Lehrwerke verwiesen. Der Informationsfluss von der DNA über die Prokaryontische Promotoren bestehen aus unterschiedlichen Elementen, für die spezifische Konsensussequenzen (DNA-Sequenzen, die bei den meisten Promotoren vorkommen) definiert werden können und die von Abschnitten mit größerer Variabilität unterbrochen werden. Bestimmte Elemente kommen bei nahezu allen Promotoren vor, z. B. die – nach ihrer typischen Sequenz – so genannte TATA-Box. Je besser die Sequenz des Promotors mit der Konsensussequenz übereinstimmt, umso stärker wirkt er, d. h. umso besser bindet das Protein, das die Initiation der Transkription bewirkt, an diesen DNA-Abschnitt. Bei den Prokaryonten wird die Transkription durch Bindung einer Untereinheit der RNA-Polymerase – des σ-Faktors – an den Promotor intiiert. Die RNA-Polymerase der Bakterien, die aus mehreren Untereinheiten besteht, ist ein echter Alleskönner: der σ-Faktor erkennt und bindet an den Promotor, eine weitere Untereinheit sorgt dann für die Entspiralisierung der DNA in diesem Bereich und wieder andere Untereinheiten katalysieren in den folgenden Schritten die RNA-Synthese. Nur eine RNA-Polymerase synthetisiert alle RNA-Typen: mRNA, die rRNA und tRNA. Bei Prokaryonten existieren zwei unterschiedliche Mechanismen der Termination der Transkription: am einfachsten ist die Termination mittels eines Sequenzelements am Ende des kodierenden Bereichs, das zu sich selbst komplementär ist und damit eine Basenpaarung innerhalb eines DNA-Strangs in Form einer „Haarnadelstruktur“ erlaubt. Erreicht die RNA-Polymerase diesen Bereich, wird sie gestoppt und fällt von der DNA ab. Eine andere Form der Termination ist proteinabhängig. Transkription bei Eukaryonten Diese verläuft komplizierter: Es ist bereits schwieriger, eukaryontische Promotoren klar zu definieren. Zwar gibt es auch hier Elemente wie die TATA-Box, weitere Konsensus-Sequenzen sind aber kaum zu definieren. Im menschlichen Genom hat man bislang ca. 775 verschiedene Promotorsequenzen identifizieren können! Dem entsprechend kann man die „Stärke“ oder „Schwäche“ eines Promotors auch nicht so leicht anhand seiner Sequenz vorhersagen. Zahlreicher als bei Prokaryonten sind ferner Enhancer- und Silencer-Sequenzen, die die Transkription durch Bindung von Aktivatoren oder Inhibitoren steuern und die auch in größerem Abstand von den betroffenen Genen lokalisiert sein können. Der entscheidende Schritt für die Initiation der Transkription ist die Bindung sog. Transkriptionsfaktoren an den Promotor. Mehrere verschiedene Proteine, die teils unmittelbar an die DNA, teils aber auch an die anderen Proteine binden, bilden zusammen einen sog. Initiationskomplex. Seine Aufgabe ist es, das Chromatin an der Stelle der Transkription ausreichend aufzulockern – bei Eukaryonten kann die Häufigkeit der Expression eines Gens auch durch die Verpackung der DNA bestimmt werden! – und die Doppelhelix aufzuwinden. An diesen Initiationskomplex bindet dann eine RNA-Polymerase und die Elongationsphase beginnt. Bei Eukaryonten gibt es drei spezialisierte RNA-Polymerasen: die RNAPolymerase I synthetisiert rRNA, die RNA-Polymerase II mRNA und einige kleinere RNA-Spezies, die RNA-Polymerase III tRNA und snRNA (small nuclear RNA, die regulative Eigenschaften hat). Unterschiedliche Terminationsmechanismen beenden die Transkription der drei verschiedenen eukaryontischen RNA-Polymerasen – insgesamt ist die Termination der Transkription aber bei den Eukaryonten in ihren Details noch weniger verstanden als bei Prokaryonten. Ernährungs Umschau | 9/08 B35 aktuell | ernährungslehre & -praxis DNA 5´ 3´ TGC AGC T C CGGAC T C CAT ACG T CGAGG C CT GAGG TA 3´ 5´ Transkription mRNA 5´ 3´ UG C A G CUC CGG AC UC C AU G AG AC C tRNA Leu Gly Anticodon Translation Ser Ser Cys Peptidkette/Protein Abb. 3: Von der DNA zum Protein: Im Zuge der Translation wird von einem proteinkodierenden Gen anhand der Paarung komplementärer Basen eine mRNA erzeugt, die dann in der Translation als Matrize für die Synthese des entsprechenden Proteins dient. Dabei werden Basentripletts der mRNA von spezifischen tRNA-Molekülen erkannt, die die zugehörigen Aminosäuren gebunden haben und deren Einbau in die wachsende Proteinkette ermöglichen. RNA zum Protein im Ganzen ist schematisch in 쏆 Abbildung 3 gezeigt. Bei Prokaryonten laufen Transkription und Translation meist paralell ab, d. h. Ribosomen binden bereits an die entstehende mRNA und die Translation beginnt, bevor die Transkription abgeschlossen ist. Dies ist bei Eukaryonten allein aufgrund der räumlichen Trennung zwischen Zellkern, der die DNA enthält, und Zytoplasma, in dem die Ribosomen lokalisiert sind, unmöglich. Hier muss die neu synthetisierte RNA zunächst in einem zielgerichteten Prozess aus dem Zellkern ausgeschleust werden. Ein Gen – ein Protein? Bei Bakterien werden die Gene für die rRNA zusammenhängend transkribiert, wobei die eigentlichen rRNA-Sequenzen von sog. Spacern unterbrochen sind. Diese nicht zur rRNA gehörenden Abschnitte werden bereits während der Transkription entfernt. Die prokaryontische mRNA wird nach der Transkription wie bereits erwähnt polyadenyliert, steht dann aber unmittelbar der Translation zur Verfügung: eine bestimmte mRNA kodiert für ein bestimmtes Gen. Lange Zeit galt dies als Grundprinzip der Molekularbiologie, bis sich zeigte, dass die Situation bei Eukaryonten erheblich komplexer ist: Hier werden die kodierenden Bereiche eines Gens, die Exons, unterbrochen von nicht-kodierenden Abschnitten, den Introns, wie ein Vergleich der Sequenz der fertigen Proteine mit den zugehörigen Genen ergab. Die Introns werden nach der Transkription in einem „Spleißen“ genannten Prozess aus dem Primärtranskript entfernt. Die Spleißstart- und Endpunkte sind wiederum durch die Nucleinsäuresequenz definiert. Wesentliche Katalysatoren für den Spleißvorgang sind die sog. snRNAs (small nuclear RNA), weiterhin sind unterschiedliche Proteine daran beteiligt. Ohne diesen Vorgang im Einzelnen zu betrachten: durch diesen Mechanismus ergeben sich vielschichtige Möglichkeiten, um von ein und derselben DNA-Sequenz durch alternatives Spleißen unterschiedliche mRNAs und damit unterschiedliche Proteinprodukte zu erzeugen. Differenzielles Spleißen findet bei einer großen Zahl von Genen statt. Ein Beispiel sind Gene für Ionenkanäle wie den Serotoninrezeptor 2C von Primaten, der eine wichtige Rolle in der Membranfunktion z. B. von Nervenzellen spielt. Alternatives Spleißen ermöglicht hier Adaptationsvorgänge in Bezug auf motorische Fähigkeiten und höhere kognitive Leistungen (s. z. B. KISHORE u. STAMM: Science [2005] 311:230– 232). Auch in den Genen für Proteine des sog, Histokompatibilitätskomplexes (diese sind als Marker auf der Zelloberfläche wesentlich für die eigene Gewebeerkennung des Organismus) spielt alternatives Spleißen eine wichtige Rolle. Die genetische Komplexität bei Eukaryonten erhöht sich durch diese Modifikationsmöglichkeiten enorm: Eine menschliche Zelle enthält ca. 33 000 Gene, von denen aber 500 000 bis 1 000 000 unterschiedliche Proteine hergestellt werden können, jeweils in Abhängigkeit vom aktuellen zellulären Bedarf. Der noch relativ junge Forschungszweig der Molekularbiologie, der die zu einem bestimmten Zeitpunkt gegebene Proteinausstattung der Zellen – das Proteom – detailliert untersucht, etwa um daraus Hinweise auf bestimmte Krankheitsprozesse zu gewinnen, wird als Proteomik bezeichnet. Noch mehr Komplexität: Proteinprozessierung Damit nicht genug: nach der eigentlichen Translation werden auch die Proteinprodukte auf vielfältige Weise weiter prozessiert. Generelle Vorgänge sind das Abspalten des N-terminalen Formylrests bei Bakterien, gegebenenfalls auch des N-terminalen Methionins. Somit beginnen durchaus nicht alle reifen Proteine mit Methionin, wie durch das Startcodon vorgegeben. Im Weiteren sind aber diverse biochemische Modifikationen bestimmter Aminosäuren möglich, die für die spätere Funktion des Proteins von entscheidender Bedeutung sind. Hierzu zählen u. a. Phosphorylierung durch Proteinkinasen, Hydroxylierung von Prolin- oder Lysinresten, Glykosylierungen, also das Anheften von Zuckerresten/oder das gezielte Knüpfen von Disulfidbrücken zwischen Cystein-Resten. Bereits im ersten Teil dieser Reihe wurde die komplexe räumliche Struktur der Proteine besprochen, die für deren korrekte Funktion unerlässlich ist. Schon während der Translation binden sog. „Chaperone“ (engl. „Anstandsdamen“) an die wachsenden Polypeptidketten und sorgen für eine korrekte Faltung. Welch dramatische Folgen eine Fehlfaltung von Proteinen haben kann, zeigen krankheitsauslösende Proteinaggregate, die entstehen, wenn sich falsch gefaltete Proteine zu unlöslichen Aggregaten zusammenballen, die dann die Zellen irreversibel schädigen. Beispiele sind die Plaque-bildenen Proteine bei der Alzheimer Demenz, aber auch fehlerhafte Parkin-Proteine bei der Parkinsonkrankheit. Der nächste Beitrag beschäftigt sich u. a. mit der Frage, wie z. B. die Gene für die Hormone der Hunger-Sättigungsregulation zur rechten Zeit ein- bzw. ausgeschaltet werden. „Ernährungslehre und -praxis“, ein Bestandteil der „Ernährungs Umschau“. Verlag: UMSCHAU ZEITSCHRIFTENVERLAG Breidenstein GmbH, Sulzbach/Ts. Zusammenstellung und Bearbeitung: Dr. Eva Leschik-Bonnet, Deutsche Gesellschaft für Ernährung, Dr. Udo Maid-Kohnert, mpm Fachmedien (verantwortlich). B36 Ernährungs Umschau | 9/08 ● ●