Enzymkinetik 2007
Werbung

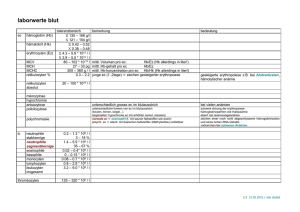
Inhaltsverzeichnis 1. Abstract ........................................................................................................................................... 2 2. Theoretischer Hintergrund............................................................................................................... 3 2.1. Enzyme allgemein ............................................................................................................................ 3 2.2 Aufbau von Enzymen ........................................................................................................................ 3 2.3 Reaktionsgeschwindigkeit ................................................................................................................. 4 2.4 Enzymkinetik..................................................................................................................................... 4 2.5 Michaelis-Menten-Gleichung ............................................................................................................ 5 2.6 Lineweaver-Burk-Gleichung ............................................................................................................. 6 2.7 Extinktion .......................................................................................................................................... 7 2.8 Die Glykolyse .................................................................................................................................... 8 3. Material und Methode ..................................................................................................................... 8 3.1 Material ............................................................................................................................................. 8 3.2 Methoden ........................................................................................................................................... 8 4. Ergebnisse ..................................................................................................................................... 11 5. Diskussion ......................................................................................................................................... 15 6. Literaturverzeichnis ........................................................................................................................... 16 1 1. Abstract In this practical training the kinetics of enzymes were examined. Enzymes - which are substrate- and reaction-specific - catalyse reactions. Their affinity, reaction rate and optical extinction can be calculated with certain formulas. The changes of the optical extinction values over time were to be tested for different concentrations of pyruvate. To this end different dilutions of pyrovate solution and the enzyme LDH (from frog muscles) were gained and then mixed and diluted with a buffer solution to ensure that the pH-value remains constant. Then the optical extinction of the different solutions was tested for two minutes in a photometer producing light with a wavelength of 340 nm and the two values "maximum velocity" and "Km" had to be calculated. The maximum velocity came up to be 0,0292 mmol/l and the value of Km was 0.0121 mmol. We detected, that the optical extinction values decline with decreasing pyruvate concentration and increasing time. 2 2. Theoretischer Hintergrund 2.1. Enzyme allgemein Enzyme sind Biokatalysatoren, die die Aktivierungsenergie von Reaktionen herabsetzen. Sie bewirken, dass eine bestimmte Reaktion schneller abläuft. Sie können sowohl die Hin- als auch die Rückreaktion katalysieren, wobei sie unverändert aus der Reaktion hervorgehen. Enzyme sind in gewissem Maße nur für ein bestimmtes Substrat spezifisch. Außerdem katalysieren sie in der Regel nur eine bestimmte Reaktion. Je nach ihrer Wirkung unterscheidet man sechs verschiedene Arten von Enzymen: Es gibt Ligasen, die bestimmte Moleküle zusammensetzen (z.B. Acetyl-CoA-Carboxylase) und Lyasen, die Moleküle spalten (z.B. Helicase). Des Weiteren existieren Enzyme, die Redoxprozesse katalysieren, wie beispielsweise Alkohol-Dehydrogenasen. Enzyme dieser Art werden als Oxidoreduktase bezeichnet. Die Maltase gehört zu den Hydrolasen und katalysiert Hydrolysationsprozesse. Transferasen, Gruppenübertragungsprozesse. Die wie sechste die Gruppe Transaminase, bilden die beschleunigen Isomerasen, die Spiegelbildisomere bilden können. Zu ihnen zählt unter anderem die Glucoseisomerase. 2.2 Aufbau von Enzymen Enzyme sind zum größten Teil Eiweiße, die aus Aminosäuren, die über Peptidbindungen verknüpft sind und unterschiedliche pH-Werte aufweisen können, bestehen. Durch die Verknüpfung der Aminosäuren entsteht eine globuläre Tertiärstruktur. Jedes Enzym besitzt ein aktives Zentrum, an welches das Substrat bindet. Es entsteht ein Enzym-SubstratKomplex. Dieses aktive Zentrum besteht aus einer Art Mulde, die von Nebengruppen weniger Aminosäuren durch ihre Tertiärstruktur gebildet wird. In diese Mulde kann dann das entsprechende Substrat durch verschiedene zwischenmolekulare Anziehungskräfte binden. Viele Enzyme besitzen außerdem noch sogenannte Cofaktoren. Dies sind NichtproteinBausteine, beispielsweise anorganische Ionen oder kleine organische Moleküle. Den Proteinanteil bezeichnet man als Apoenzym. Apoenzym und Coenzym bilden das Holo- oder 3 Gesamtenzym. So benötigt zum Beispiel die Katalyse der oxidativen Desaminierung von Glutaminsäure den Cofaktor Nikotinamid-Adenin-Dinucleotid (NAD+). 2.3 Reaktionsgeschwindigkeit Sowohl die Temperatur als auch der pH-Wert haben starken Einfluss auf die Reaktionsgeschwindigkeit. Veränderungen dieser beiden Faktoren beeinflussen die Anordnung der Aminosäuren im aktiven Zentrum des Enzyms. Dadurch ändert sich die Enzymaktivität. Steigt die Temperatur erhöht sich die Reaktionsgeschwindigkeit wegen der größeren kinetischen Energie, die auf die Substratmoleküle wirkt, bis die Denaturierung der Eiweiße Überhand gewinnt. Jedes Molekül besitzt zudem ein pH-Optimum. Je höher der pH-Wert ist, desto leichter binden positive Substratgruppen an negativ geladene Bereiche des Enzyms. Bei einer Erniedrigung des pH-Wertes verhält es sich umgekehrt: negative Substratgruppen binden leichter an die vermehrten positiven Enzymzentren. 2.4 Enzymkinetik Die Reaktionsgeschwindigkeit hängt außer von der Temperatur und dem pH-Wert auch von der Konzentration des Substrats, des Enzyms und des gebildeten Produkts ab. Wenn das Produkt mit der gleichen Geschwindigkeit wie es gebildet wird auch entfernt wird, sind nur noch die Enzym- und Substratkonzentration auschlaggebend für die Reaktionsgeschwindigkeit. Falls das Enzym im Überfluss vorhanden ist, ergibt sich für das Tempo der Reaktion folgende Gleichung: v = -d [A] / dt = k [A] [A]: Die im Augenblick vorliegende Substratkonzentration k: Reaktionsgeschwindigkeitskonstante v: Reaktionsgeschwindigkeit 4 Diese Gleichung lässt sich auch logarithmisch darstellen: log (a / a-x) = (k1 * t) / (2,303) a: Anfangskonzentration des Substrats x: Menge des Substrats, das während der Zeit t reagiert Liegt eine Enzymsättigung vor ist die Konzentration des Substrats der limitierende Faktor für die Reaktionsgeschwindigkeit. Diese Situation wird als Kinetik erster Ordnung bezeichnet. Bei einer Kinetik zweiter Ordnung liegt das Enzym wieder im Überschuss vor und das Produkt wird kontinuierlich entfernt. Es werden hier allerdings zwei Substrate A und B zu einem Produkt umgewandelt. Hier gilt folgende Gleichung: v= -d [A] / dt = k [A] * [B] Für den Fall, dass das Enzym der limitierende Faktor und substratgesättigt ist, spricht man von einer Kinetik nullter Ordnung. Trägt man hier in einem Graphen die Menge des Substrats, das während der Zeit t reagierte gegen die Zeit auf, so erhält man eine Gerade. 2.5 Michaelis-Menten-Gleichung Die Michaelis-Menten-Gleichung gibt die Geschwindigkeit bei einer enzymkatalysierten Reaktion an: v0 = (vmax [S]) / (Km + [S]) v0: anfängliche Reaktionsgeschwindigkeit bei der Substratkonzentration [S] vmax: Reaktionsgeschwindigkeit bei einem Überschuss an Substrat Km: Michaelis-Menten-Konstante [S]: Substratkonzentration 5 Ein typisches Diagramm sieht folgendermaßen aus: Abb. 1: Michaelis- Menten- Diagramm (Quelle: http://www.biosite.dk/leksikon/images/michaelis-menten.gif) 2.6 Lineweaver-Burk-Gleichung Die Michaelis-Menten-Gleichung kann durch die Umformung in die Lineweaver-BurkGleichung durch wenige Bezugspunkte schneller und genauer graphisch dargestellt werden: 1 / v0 = (Km / vmax) * (1 / [S]) + (1 / vmax) Trägt man hier den reziproken Wert der Reaktionsgeschwindigkeit v0 gegen den reziproken Wert Substratkonzentration [S] auf, dann erhält man als Graphen eine Gerade mit der Steigung (Km / vmax), die die y-Achse in dem Punkt (1/ vmax) und die x-Achse im Punkt (- 1/ Km) schneidet. 6 Dieses Diagramm sieht in graphischer Form wie folgt aus: Abb. 2: Lineweaver- Burk- Diagramm (Quelle: http://www.answers.com/topic/lineweaver-burke-plot-png-1) 2.7 Extinktion Für die Berechnung der Extinktion, mit der auch im Versuch gearbeitet wird gibt es das Lambert-Beer-Gesetz: E = log (I / I0) = ε * c * d E: Extinktion (Maß der Absorption) I: Intensität des austretenden Lichtes I0: Intensität des einfallenden Lichtes ε: Extinktionskoeffizient (Stoffkonstante) c: Konzentration des gelösten Stoffes d: Dicke der Schicht, durch die das Licht dringt 7 2.8 Die Glykolyse In der Glykolyse wird Zucker gespalten und Glucose in Pyrovat abgebaut. Dabei liefert die Oxidation von 1mol Glukose in der Nettogleichung 2mol ATP und 2mol NADH. Unter anaeroben Bedingungen wird das Pyrovat zu Lactat oder zu Ethanol reduziert. Bei der Milchsäuregärung wird das Substrat reduziert und gleichzeitig NADH oxidiert. Das hierfür gebrauchte Enzym ist Lactatdehydrogenase. 3. Material und Methode 3.1 Material Benötigte Geräte: Spektralphotometer (340 nm) mit Schreiber, Küvetten, Homogenisator nach Potter-Elvejhem, Homogenisator-Zylinder 15 ml mit Klemmring, Teflonkolben, Gehirn, Kühlzentrifuge, Eppendorf-Pipette 50 µl, Probenmischer, Stoppuhr, Kleenex, Feinwaage, Notwendige Reagenzien: 1. Kaliumdihydrogenphosphat, KH2PO4 2. Di-Kaliumhydrogenphosphat, K2HPO4 (1. und 2. zur Herstellung des Phosphat-Puffers) 3. Natriumpyruvat, C3H3NaO3 4. Reduziertes Nicotinamidadenindinucleotid als Dinatriumsalz, NADH-Na2 3.2 Methoden 3.2.1. Gewinnung der Pyruvatlösung 8 Zuerst wird eine Stammlösung mit einer Konzentration von 100 mmol/l gemischt. Nun benutzt man diese, um Verdünnungen mit 50, 40, 30, 20, 10, 8, 6, 5, 4, 3 und 2 mmol/l herzustellen. Dies geschieht, indem die Pyruvatlösung mit einer gewissen Menge an Wasser aufgefüllt wird. 3.2.2. Gewinnung von LDH (Lactatdehydrogenase) Verwendet wird Muskelgewebe aus dem Plantaris longus des Südafrikanischen Krallenfrosches (Xenopus laevis). Zuerst wird das Frischgewicht bestimmt, damit man später die Extinktion spezifisch für ein Gramm Gewebe berechnen kann. Nun wird der PhosphatPuffer hinzugefügt. Anschließend wird unter Kühlung mittels Eis die Mischung im PotterElvejhem-Homogenisator homogenisiert. Daraufhin zentrifugiert man das Gemisch und bewahrt das dabei gewonnene LDH weiterhin unter Eiskühlung auf. 3.2.3. Versuch zur Bestimmung der Extinktion Es werden nun 11 neue Mischungen in Küvetten hergestellt. Zu jeweils 3 ml Kaliumphosphat-Puffer werden 0,05 ml LDH- Extrakt, 0,05 ml NADH und 0,05 ml der verschieden konzentrierten Pyruvatlösungen zugegeben. Dabei dient der Puffer zur Konstanthaltung des pH-Wertes, LDH und NADH werden für die Reaktion von Pyruvat zu Lactat benötigt, wobei LDH als Enzym fungiert. Nun werden die Proben nacheinander ins Photometer gegeben und ihre Extinktion wird bei 340 nm Wellenlänge zwei Minuten lang alle 10 Sekunden gemessen. Die Extinktion ist die Lichtschwächung durch die absorbierende Substanz. In dieser Reaktion ist dies NADH. Die Werte für die Extinktion kann man Tabelle 1 entnehmen. 3.2.4. Auswertung des Versuches 3.2.4.1. Berechnung der Lactatzunahme Ausgehend von den protokollierten Extinktionswerten wird die Lactatzunahme berechnet. Zuerst wird das Lambert-Beer-Gesetz angewendet, alle bekannten Werte werden eingesetzt, und die Gleichung wird so umgestellt, dass man die Konzentration berechnen kann: c = E / (d*ε) [mol / m³] 9 Um die Konzentration für ein Gramm Muskelgewebe zu erhalten, teilt man nun durch das zuvor ermittelte Frischgewicht des Muskels (306,6 mg). Abschließend wird mit dem Verdünnungsfaktor multipliziert. Dieser beträgt in unserem Fall 10*2*63. Damit erhält man die Lactatzunahme, die man für alle Extinktionswerte benötigt, trägt jeweils die ersten sechs Werte in ein Diagramm ein. Es wird eine Trendlinie eingefügt, deren Steigung abgelesen wird (y= m*x +c, wobei m die Steigung darstellt). 3.2.4.2 Berechnungen der für Tabelle 2 benötigten Größen Diese Steigungswerte ermöglichen uns die Berechnung der Anfangsgeschwindigkeit v0. Dies liegt daran, dass die Steigung einer Geraden die erste Ableitung an der betreffenden Stelle ist. Somit gilt: a= v0 [mmol / sec]. Teilt man diesen Wert durch 60, erhält man v0 in [mmol/min]. Außerdem kann der Kehrwert 1 / v0 gebildet werden. Weiterhin berechnet wurde ∆E. Dies geschieht folgendermaßen: Man verrechnet die Extinktionswerte zu Beginn des Versuches mit denen nach 60 Sekunden. Eine weitere wichtige Größe, die es zu berechnen gilt, ist [St], die tatsächliche Pyruvatkonzentration. Diese wird ausgehend von [S] ermittelt mit folgender Formel: [St]= [S] / VF Hierbei sind [S] die verschieden konzentrierten Pyruvatlösungen und VF ist der Verdünnungsfaktor. VF= Vol ges / Vol Probe = 3,15 ml / 0,05 ml= 63. 3.2.4.3 Berechnung von vmax und Km Beide Größen können über das Lineweaver-Burk. Diagramm berechnet werden. In diesem Diagramm ergibt der Schnittpunkt der Regressionsgeraden (y= m*x + c) mit der yAchse den Wert für 1 / vmax. Somit gilt also: vmax= 1 /c= 1/34,24= 0,0292 [mmol/min]. Der Schnittpunkt der Geraden mit der x-Achse ist für diesen Diagrammtypen definiert als -1/Km. Setzt man nun also die Regressionsgleichung gleich Null, so kann man den entsprechenden x-Wert ausrechnen. Desweiteren gilt: x= -1 / Km bzw. Km= -1 / x. Mit Hilfe dieser Gleichung lässt sich also der Km- Wert berechnen. 10 4. Ergebnisse Tab. 1: Extinktionswerte bei verschiedenen Pyruvatkonzentrationen Eingesetzte Pyruvatlösung [mmol/m3] 50 40 30 20 10 8 6 5 4 3 2 10 -0,015 -0,016 -0,017 -0,020 -0,027 -0,025 -0,019 -0,027 -0,020 -0,013 -0,017 20 -0,040 -0,040 -0,030 -0,057 -0,059 -0,066 -0,041 -0,047 -0,044 -0,033 -0,052 30 -0,065 -0,066 -0,047 -0,089 -0,088 -0,094 -0,060 -0,078 -0,071 -0,052 -0,072 40 -0,083 -0,089 -0,079 -0,117 -0,116 -0,127 -0,087 -0,111 -0,096 -0,064 -0,092 50 -0,109 -0,109 -0,115 -0,149 -0,145 -0,157 -0,116 -0,130 -0,119 -0,076 -0,112 60 -0,124 -0,127 -0,146 -0,163 -0,165 -0,176 -0,137 -0,145 -0,137 -0,089 -0,133 70 -0,136 -0,137 -0,165 -0,170 -0,180 -0,192 -0,155 -0,157 -0,155 -0,102 -0,146 80 -0,153 -0,143 -0,19 -0,188 -0,193 -0,198 -0,172 -0,169 -0,169 -0,115 -0,158 90 -0,167 -0,151 -0,215 -0,204 -0,206 -0,208 -0,186 -0,185 -0,180 -0,125 -0,167 100 -0,177 -0,161 -0,239 -0,217 -0,215 -0,217 -0,199 -0,195 -0,190 -0,131 -0,176 110 -0,187 -0,172 -0,260 -0,229 -0,224 -0,224 -0,209 -0,202 -0,198 -0,138 -0,183 Zeit [s] In Tabelle 1 kann man sehen, dass die Extinktionswerte mit zunehmender Zeit immer weiter abnehmen. Zudem werden abhängig von den sinkenden Pyruvatkonzentrationen die Extinktionswerte tendenziell kleiner. Aus den ermittelten Extinktionswerten lässt sich die jeweilige Lactatzunahme berechnen. 11 Diese ist im folgenden Diagrammen graphisch dargestellt: Abb. 3: Lactatzunahme für die Pyruvatkonzentrationen von 50, 40, 30 und 20 mmol/m3 Abb. 4: Lactatzunahme für 10, 8 und 6 mMolare Pyruvatlösung 12 Abb. 5: Lactatzunahme bei einer Pyruvatkonzentration von 4, 3 und 2 mmol/m3 Tab. 2: Übersicht über die berechneten Größen v0 [mmol/min] 0,02421667 0,02468333 0,02931667 0,03205000 0,03070000 0,03336667 0,02648333 0,02743333 0,02626667 0,01638333 0,02453333 1/v0 ΔE [min/mmol] 41,2938747 40,5131668 34,1102899 31,2012480 32,5732899 29,9700300 37,7595972 36,4520049 38,0710660 61,0376399 40,7608696 [S] [mmol/l] 0,109 0,003 0,129 0,143 0,138 0,151 0,118 0,118 0,117 0,076 0,116 50 40 30 20 10 8 6 5 4 3 2 [St] 1/[St] [mmol/l] [m3/mmol] 0,79365079 0,63492063 0,47619048 0,31746032 0,15873016 0,12698413 0,09523810 0,07936508 0,06349206 0,04761905 0,03174603 1,26 1,58 2,10 3,15 6,30 7,88 10,50 12,60 15,75 21,00 31,50 Mittels der in Tabelle 2 berechneten Größen für [St] und v0 lässt sich das zur Reaktion gehörige Michaelis-Menten-Diagramm erstellen. 13 Abb. 6: Michaelis- Menten- Diagramm Die Messdaten ergeben den tiefsten Geschwindigkeitswert für eine tatsächliche 3 Substratkonzentration von Pyruvat von 0,04761905 mmol/m . Ab diesem Punkt erhält man steigende Werte bis ein Maximum bei 0,12698413 mmol/m3 erreicht wird. Danach fallen die Werte wieder leicht ab. Auffallend ist ein Ausreiser für den ersten Messwert. Um die benötigten Werte von Km und vmax leichter bestimmen zu können, erstellt man zusätzlich mit Hilfe der Werte von 1/[St] und 1/v0 aus Tabelle 2 ein Lineweaver-BurkDiagramm. 14 Abb. 7: Lineweaver-Burk-Diagramm Im Schaubild erkennt man, dass die Werte in einem relativ engen Bereich liegen. Fügt man zusätzlich eine Regressionsgerade ein, so erkennt man, dass diese eine geringe Steigung aufweist. Abschließend kann man die Werte für Km und vmax berechnen (siehe Material und Methoden S. 10). In unserem Versuch erhält man für vmax 0,0292 mmol/min und für Km einen Wert von 0,0121mmol. 5. Diskussion Wie bereits schon in Teil 3 erwähnt, nehmen die Extinktionswerte aus Tabelle 1 ab. Die Ursache hierfür ist, dass mit zunehmender Zeit die Reaktion des Pyruvats zu Lactat unter Umwandlung von NADH zu NAD+, wobei LDH die Reaktion als Enzym katalysiert, fortschreitet. Dadurch ist weniger NADH in den Küvetten vorhanden. Da NADH die lichtabsorbierende Substanz ist und NAD+ Licht bei 340 nm nicht absorbiert, nehmen folglich auch die gemessenen Extinktionswerte ab. Bei den geringer konzentrierten Pyruvatlösungen müssten die Extinktionswerte eigentlich größer sein, weil hier weniger Pyruvat für die 15 Reaktion zur Verfügung steht. Dies hat zur Folge, dass auch weniger NADH umgewandelt, also verbraucht wird. Damit wird mehr Licht absorbiert. Dies kann man in unseren Ergebnissen nicht immer nachvollziehen, was verschiedene Ursachen haben kann, wie zum Beispiel fehlerhaft hergestellte Pyruvatlösungen. Außerdem konnten wir nicht sofort nach der Pyruvatzugabe messen, was die Ergebnisse ebenfalls verfälschen könnte. Eine weitere Ursache könnte sein, dass keine Stoppuhr vorhanden war, sondern uns lediglich eine normale Uhr zum Ablesen der Zeit zur Verfügung stand. Verschmutze Küvetten und ungenaue Pipetten könnten ebenfalls zu Verfälschungen führen. Die Lactatkonzentration steigt mit Abnahme des Pyruvats, wie wir es erwarten. Das Michaelis-Menten-Diagramm entspricht nicht ganz unseren Erwartungen. Wir hätten für die ersten Werte mit einer deutlich größeren Steigung gerechnet. Das Lineweaver-Burk-Diagramm entspricht im Großen und Ganzen unseren Erwartungen bezüglich des Verlaufs der Geraden. Gut berechnen kann man die beiden Werte für vmax und Km. Der von uns berechnete Km-Wert beträgt, wie oben erwähnt, 0,01209 mmol. Der nachgeschlagene Literaturwert hingegen liegt bei 0.16 mmol (Karlson, Kurzes Lehrbuch der Biochemie). Diese erhebliche Abweichung kann ebenfalls aus den oben genannten Gründen resultieren. 6. Literaturverzeichnis • Tierphysiologie, R. Eckert, 2002, 4. Auflage, Thieme • Biologie, Campbell, Reece, 2006, 6. Auflage, Pearson - Seite 113 ff. • Vorbereitungsskript Stoffwechselteil, Enzymkinetik WS 2007/2008 • Altprotokoll 16