BioMol_Diplom_Steinem
Werbung
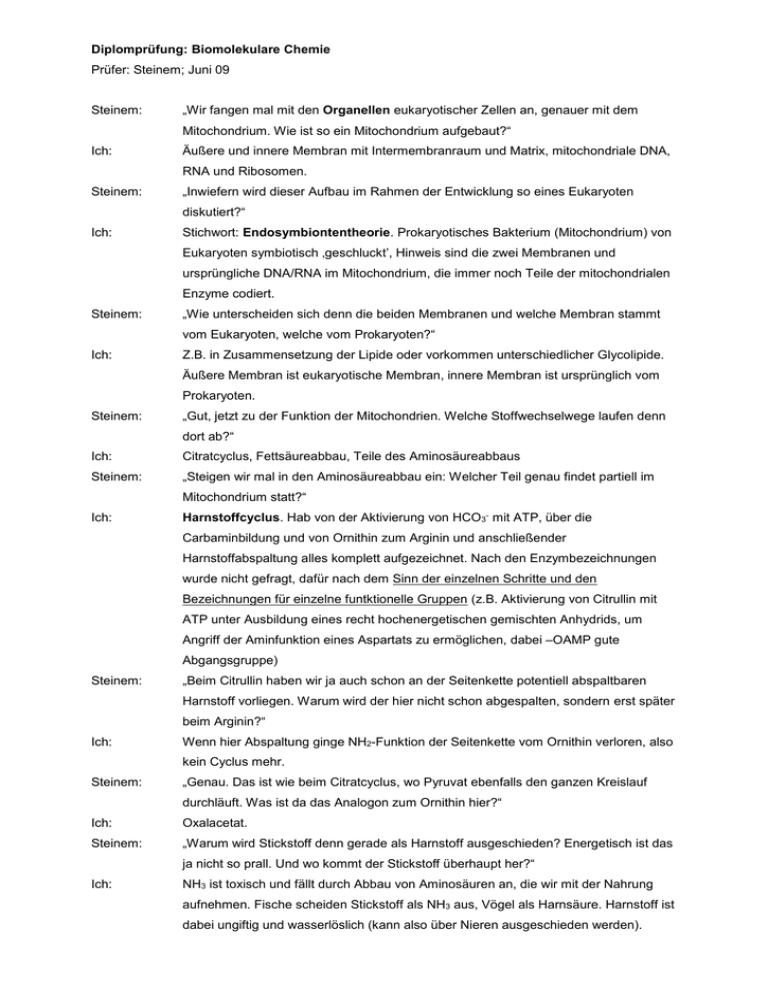
Diplomprüfung: Biomolekulare Chemie Prüfer: Steinem; Juni 09 Steinem: „Wir fangen mal mit den Organellen eukaryotischer Zellen an, genauer mit dem Mitochondrium. Wie ist so ein Mitochondrium aufgebaut?“ Ich: Äußere und innere Membran mit Intermembranraum und Matrix, mitochondriale DNA, RNA und Ribosomen. Steinem: „Inwiefern wird dieser Aufbau im Rahmen der Entwicklung so eines Eukaryoten diskutiert?“ Ich: Stichwort: Endosymbiontentheorie. Prokaryotisches Bakterium (Mitochondrium) von Eukaryoten symbiotisch ‚geschluckt’, Hinweis sind die zwei Membranen und ursprüngliche DNA/RNA im Mitochondrium, die immer noch Teile der mitochondrialen Enzyme codiert. Steinem: „Wie unterscheiden sich denn die beiden Membranen und welche Membran stammt vom Eukaryoten, welche vom Prokaryoten?“ Ich: Z.B. in Zusammensetzung der Lipide oder vorkommen unterschiedlicher Glycolipide. Äußere Membran ist eukaryotische Membran, innere Membran ist ursprünglich vom Prokaryoten. Steinem: „Gut, jetzt zu der Funktion der Mitochondrien. Welche Stoffwechselwege laufen denn dort ab?“ Ich: Citratcyclus, Fettsäureabbau, Teile des Aminosäureabbaus Steinem: „Steigen wir mal in den Aminosäureabbau ein: Welcher Teil genau findet partiell im Mitochondrium statt?“ Ich: Harnstoffcyclus. Hab von der Aktivierung von HCO3- mit ATP, über die Carbaminbildung und von Ornithin zum Arginin und anschließender Harnstoffabspaltung alles komplett aufgezeichnet. Nach den Enzymbezeichnungen wurde nicht gefragt, dafür nach dem Sinn der einzelnen Schritte und den Bezeichnungen für einzelne funtktionelle Gruppen (z.B. Aktivierung von Citrullin mit ATP unter Ausbildung eines recht hochenergetischen gemischten Anhydrids, um Angriff der Aminfunktion eines Aspartats zu ermöglichen, dabei –OAMP gute Abgangsgruppe) Steinem: „Beim Citrullin haben wir ja auch schon an der Seitenkette potentiell abspaltbaren Harnstoff vorliegen. Warum wird der hier nicht schon abgespalten, sondern erst später beim Arginin?“ Ich: Wenn hier Abspaltung ginge NH2-Funktion der Seitenkette vom Ornithin verloren, also kein Cyclus mehr. Steinem: „Genau. Das ist wie beim Citratcyclus, wo Pyruvat ebenfalls den ganzen Kreislauf durchläuft. Was ist da das Analogon zum Ornithin hier?“ Ich: Oxalacetat. Steinem: „Warum wird Stickstoff denn gerade als Harnstoff ausgeschieden? Energetisch ist das ja nicht so prall. Und wo kommt der Stickstoff überhaupt her?“ Ich: NH3 ist toxisch und fällt durch Abbau von Aminosäuren an, die wir mit der Nahrung aufnehmen. Fische scheiden Stickstoff als NH3 aus, Vögel als Harnsäure. Harnstoff ist dabei ungiftig und wasserlöslich (kann also über Nieren ausgeschieden werden). Steinem: „Weißt du vielleicht, welche Möglichkeit der Bindung von freiem Ammoniak, vor allem im Gehirn, wenn keine andere Möglichkeit besteht, es noch gibt?“ Ich: Nee, aber bin mit etwas Hilfe drauf gekommen, dass dort wohl Glutarat zum Binden des Ammoniaks dient, also Glutamat entsteht. Steinem: „Okay. Dass Harnstoff beim Abbau von Aminosäuren anfällt haben wir jetzt gesehen. Wo findet sich Stickstoff denn sonst noch im Organismus?“ Ich: Nucleobasen, also in DNA und RNA. Unterscheidung zwischen Purin und Pyrimidinbasen. Hab Adenin aufgemalt, um zu verdeutlichen, wie viel Stickstoff gebunden ist. Steinem: „Damit sind wir also bei der DNA angekommen. Wie läuft denn die Transkription ab?“ Ich: (Kurzfassung) RNA-Polymerase bindet an entspiralisierte DNA, Nucleotide (AUCG bei m-RNA) werden über Watson-Crick-Basenpaarung angebunden und verknüpft. Raus kommt dabei ein mRNA-Strang. Steinem: „Manche Stellen werden ja besonders häufig abgelesen, Stichwort: Genexpression, dazu gibt’s glaub ich auch im Praktikum einen Versuch...“ Ich: Keine Ahnung, was da genau gemeint war (hab mir das Praktikumsskript vorher nicht wirklich angeschaut und soweit ich weiß hat so ein Versuch bei uns damals auch nicht geklappt). Auf jeden Fall sind wir dann auf Promotoren, Enhancer und Repressoren und so gekommen, wozu ich jeweils mehr oder weniger „unscharf“ etwas beitragen konnte. Steinem: „Na gut. Kommen wir die letzten 5 Minuten noch mal zur Translation. Welche Strukturen kommen denn dort zum Einsatz, um die Information der mRNA in eine Aminosäuresequenz zu übersetzen?“ Ich: tRNA, ‚Kleeblatt-Struktur’ aufgemalt, dreidimensional eher L-förmig. Ester-Bindung der passenden Aminosäure am 3’-Ende, Hydrolyse von GTP als eine Art Kontrolle für passende Aminosäure. Frau Steinem wollte dabei besonders betont wissen, dass die Bindung der richtigen Aminosäure an der t-RNA trotz so großem Abstand zum AntiCodon genauestens kontrolliert wird und am Ribosom selbst dann keine weitere Kontrolle mehr statt findet. Hab dann noch das Ribosom mit 30S und 50S Untereinheiten skizziert, Bindungsstellen A, P und E erwähnt, Initiationsfaktoren, Elongationsfaktoren und was sonst noch so dazu gehört. Frau Steinem legt direkt los und deckt mit ihren Fragen immer a) Stoffwechsel und b) DNA ab, wobei gern Bezug zum Praktikum oder dem eigenen Vortrag genommen wird. Daher sollte man wirklich vom Zucker- über Fett- bis Stickstoffmetabolismus sämtliche Stoffwechselwege penibelst abgedeckt haben. Nach dem Verlauf meiner Prüfung kann ich auch empfehlen zu DNA-Replikation, sowie Transkription und Translation im Vorfeld eine Art „Vortrag“ vorzubereiten, damit man sich nicht (wie ich) verhaspelt oder Details unterschlägt – dran kommt’s so oder so. Insgesamt ist ein recht hoher Lernaufwand nötig. Im Gegenzug ist die Prüfungsatmosphäre unkompliziert, Frau Steinem streut auch selbst hier und da einige extra-Info’s mit ein, und die Benotung ist fair und angemessen. Alles in Allem zwar anspruchsvoll, aber für Leute, die mit den Vorlesungen klar gekommen sind und nicht bei null mit dem Lernen anfangen müssen zu empfehlen. Note: sehr gut