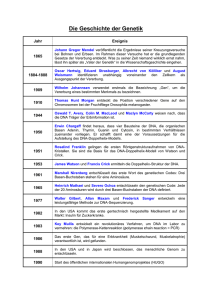
2.3 Methoden
Werbung

Inhaltsverzeichnis 1 Einleitung 1.1 Das Herz 1.2 Entwicklung des Herzen 1.3 Einteilung der angeborenen Herzfehler 1.4 Ätiologie 1.5 Häufigkeit und Wiederholungsrisiko der Herzfehler 1.6 Methoden der Untersuchung chr. Veränderungen 1.6.1 Entwicklung von Array CGH 1.6.2 Vergleich Array CGH und mol.-cyt. Methoden 1 1 2 2 5 7 8 8 12 1.7 Ziel der Diplomarbeit 13 2 Material und Methoden 14 14 16 17 17 19 19 21 22 24 25 25 27 28 30 30 30 30 31 33 34 34 35 37 38 38 39 40 2.1 Chemikalien 2.2 Geräte und Software 2.3 Methoden 2.3.1 Array CGH 2.3.2 Präparation der BAC/PAC Klone 2.3.2.1 Ampl. der Klon-Inserts mittels Linker Adapter PCR 2.3.2.2 Ligation 2.3.2.3 Ligation vermittelte PCR 1 2.3.2.4 PCR 2 2.3.2.5 Agarosegelelektrophorese zur Kontrolle der PCR Produkte 2.3.2.6 Ethanolpräzipitation der DNA 2.3.3 Produktion der DNA Arrays 2.3.4 Patienten DNA 2.3.5 DNA Isolierung 2.3.6 DNA Sonification 2.3.7 Agarosegelelektroph. zur Kontrolle der sonific. Proben 2.3.8 DNA Aufreinigung 2.3.9 Labelling 2.3.10 Proben Aufreinigung 2.3.11 DNA Fällung 2.3.12 Slidebehandlung 2.3.13 Hybridisierung 2.3.14 Waschen der Slides 2.3.15 Auswertung 2.3.15.1 Scannen der Slides 2.3.15.2 Bildverarbeitung 2.3.15.3 Datenverarbeitung 3 Ergebnisse 43 4 Diskussion 65 5 Literaturverzeichnis 69 6 Anhang Hybridisierungsergebnisse (Übersicht) Danksagung Eidesstattliche Erklärung 75 76-94 95 96 1 1. Einleitung 1.1 Das Herz Das Herz (Abb.1) dient als Pumpe für das Blut und die darin gelösten Nährund Abfallstoffe. Das Herz setzt sich aus zwei Hälften zusammen, welche in Vorhof (Atrium) und Hauptkammer (Ventrikel) unterteilt sind; diese vier Kammern arbeiten in einem fein abgestimmten Rhythmus. Die zwei Vorhöfe (rechtes und linkes Atrium) nehmen das Blut auf und Pumpen es zu den zwei Hauptkammern (rechtes und linkes Ventrikel). Klappen zwischen den Vorhöfen und Hauptkammern (AV-Klappen; auf der linken Seite Mitralklappe, auf der rechten Seite Trikuspidalklappe) wirken als Ventile und stellen sicher, daß das Blut nur in Richtung Hauptkammer und nicht zurück in den Vorhof fließt. Aus der linken Hauptkammer gelangt das Blut durch die Aortenklappe in die Körperschlagader (Aorta). Aus der rechten Hauptkammer wird das Blut durch die Pulmonalklappe in die Lungenschlagader (Arteria Pulmonalis) gepumpt. Die AV-Klappen zwischen Vorhöfen und Kammern bestehen aus feinen Segeln, welche von Papillarmuskeln in den Hauptkammern gehalten und bewegt werden. Die Klappen in den Arterien (Aortenklappe und Pulmonalklappe) bestehen aus Taschen, die sich passiv durch das von den Kammern gepumpte Blut öffnen. Fällt der Druck in den Hauptkammern wieder ab, so bläht die zurückfallende Blutsäule in den Arterien die Taschen auf und verschließt damit den Klappenapparat. Abbildung 1 Herzaufbau (Wort und Bild Verlag GmbH und Co http://www.gesundheitpro.de) 2 1.2 Entwicklung des Herzen Die Entwicklung des Herzen beginnt am 12 Tag nach der Befruchtung und erfolgt während der ersten neun Schwangerschaftswochen. Das Herz entwickelt sich aus einer kleinen Zellgruppe im oberen Abschnitt des Brustraumes. Diese Zellen gruppieren sich weiter zu zwei seitlich im Embryo gelegenen dünnen Kanälen, welche sich schließlich in der Mitte des Körpers zum Herzschlauch vereinigen. Da dieser Schlauch in der Länge sehr viel schneller wächst als der übrige Embryo, dreht und windet er sich schließlich um sich selbst und bildet die Form eines „S’’. Verläuft diese Drehung unvollständig oder falsch herum, kann dies zu Herzfehlern führen. Es bilden sich weiterhin Ausbuchtungen, aus denen sich die Kammern entwickeln. Zwischen diesen Ausbuchtungen liegen Einengungen und Zellnester, aus denen sich die Klappen formen. Schließlich entwickeln sich die Trennwände, die Septen. Sie trennen nicht nur die Kammern voneinander, sondern auch Aorta und Lungenschlagader. Störungen dieser fetalen Herzentwicklung sind die Ursache der angeborenen Herzfehler (1, 2). 1.3 Einteilung der angeborenen Herzfehler Die Störung der Entwicklung in der sensiblen Phase der Schwangerschaft (1. und 2. Schwangerschaftsmonat) kann zu Fehlbildungen am Herzgefäßsystem führen. In 75-82% der Fälle treten die Herzfehlbildungen isoliert auf, ansonsten sind sie mit zusätzlichen extrakardialen Fehlbildungen kombiniert (2). Es gibt eine Vielfalt anatomisch bzw. morphologisch verschiedener Herzfehler, deren Klassifikation zum Teil sehr schwer ist (3). Im Rahmen dieser Arbeit wird nur eine sehr vereinfachte Einteilung dargestellt. 3 Drei Grundstörungen können auftreten (Linus Geisler: INNERE MEDIZIN © 1969/1999 ): • Es bestehen abnorme Verbindungen zwischen kleinem und großem Kreislauf (z.B. Vorhof- oder Kammerseptumdefekt, offener Ductus Botalli). • Es entstehen Stenosen (z.B. Pulmonalstenose, Aortenisthmusstenose). • Es liegt eine Transposition, d.h. eine Verlagerung der großen Gefäße vor (z.B. Abgang der Aorta aus der rechten, der Arteria pulmonalis aus der linken Herzkammer). Die oben genannten Störungen können sowohl einzeln als auch in Kombination auftreten. Die am häufigsten auftretenden Herzfehler sind in der Abbildung 2 dargestellt. Abbildung 2 . Angeborene Herzfehler (Linus Geisler: INNERE MEDIZIN © 1969/1999) 1. Pulmonalstenose: Bei etwa 7% aller angeborenen Herzfehler tritt eine Verengung der Pulmonalklappe, der Herzklappe durch die das Blut von der rechten Herzkammer in Richtung Lunge fließt, auf (Abb.2). 2. Der persistierende (fortbestehende) Ductus Arteriosus Botalli : Diese Anomalie (Abb.2) tritt bei ca. 10% aller angeborenen Fehlbildungen auf. Der Ductus arteriosus ist eine Kurzschlussverbindung zwischen Lungenarterie und dem Anfangsteil der Aorta. Normalerweise bildet sich diese Verbindung nach der 4 Geburt zurück. Falls sie jedoch bestehen bleibt, begünstigt sie den Blutstrom von der linken (sauerstoffreichen) in die rechte (sauerstoffarme) Herzhälfte (LinksRechts-Shunt), was zur Rechtsherzbelastung führt. 3. Aortenisthmusstenose: Bei der Aortenisthmusstenose (Abb.2) weist die Aorta am Übergang vom Aortenbogen bis zu dem in den Körper absteigenden Teil, eine deutliche Verengung auf. Diese Verengung führt zu Stauung des Blutes bei jedem Auswurf aus dem Herzen (Systole) und zur Behinderung des Blutflusses vor und hinter der Stauung. 4. Vorhofseptumdefekt: Die Vorhofscheidewand (Abb.2) besteht beim Fötus und beim Neugeborenen aus zwei Gewebslappen, die bald von den Seiten aufeinander zuwachsen und sich übereinander legen. Hierbei kommt es jedoch manchmal nicht zu einem vollständigen Verschluß und Verwachsen der Vorhofscheidewand. 5. Ventrikelseptumdefekt: Der Ventrikelseptumdefekt (Abb.2) ist mit 30% der am häufigsten vorkommende angeborene Herzfehler. Beim Ventrikelseptumdefekt handelt es sich um einen Defekt (Loch) in der Kammerscheidewand. Dieser Defekt verursacht das Austreten des Blutes aus der linken in die rechte Herzhälfte. 6. Fallotsche Tetralogie: Die angeborenen Herzfehler mit Rechts-Links-Shunt (Vermischung des sauerstoffarmen mit dem sauerstoffreichen Blut im Körperkreislauf) wurden erstmals 1888 von dem französischen Arzt FALLOT ausführlich beschrieben, weshalb sie unter dem Sammelbegriff Fallot-Syndrom zusammengefaßt werden. Je nachdem, ob sich 3, 4 oder 5 Fehler kombinieren, spricht man von Fallotscher Trilogie, Tetralogie (Abb.2) bzw. Pentalogie. Zwei immer vorhandene Fehler werden mit zwei anderen kombiniert. Die Kombination besteht immer aus einer Pulmonalstenose und einer Wandverdickung der rechten Herzkammer. Diese Fehler können mit einem Vorhofseptumdefekt, einem Kammerseptumdefekt oder einer Aorta assoziiert sein, die nach rechts verlagert ist und über dem Kammerseptum "reitet" (reitende Aorta) und dabei Blut aus beiden Kammern bekommt. (Linus Geisler: INNERE MEDIZIN © 1969/1999) 5 1.4 Ätiologie Die Ätiologie ist sehr vielfältig und reicht von umweltbedingten Störungen (ca. 3%) über genetische Defekte (10%) bis zu Kombination dieser zwei Faktoren bei etwa 87% aller Herzgefäßfehlbildungen (3). Bei genetisch bedingten Herzfehlern unterscheidet man zwischen chromosomalen Anomalien (Tab.1), welche bei 4-13% aller Patienten mit angeborenen Herzfehlern (AHF) z. B. Trisomie 21 auftreten (5) und den polygenetischen Defekten (Tab.2), welche bei 3-4,6% aller Patienten mit AHF (5), Nora 1983) assoziiert sind, wobei hier vor allem die Deletion 22q11 bzw. Di-GeorgeSyndrom (Scambler 1999), das Marfan-, das Noonan-Syndrom und das autosomaldominant vererbbare familiäre Vorhofseptumdefekt (Chromosom 5p, Bonnet et al. 1999) zu nennen sind. Umweltbedingte Ursachen (Tab. 3) sind z.B. Medikamente (Antiepileptika wie Diphenylhydantoin), Chemikalien, Strahlenexposition, bakterielle und virale Infektionen (Rötelnvirus) während der Schwangerschaft oder Stoffwechselerkrankungen der Mutter, v.a. ein Diabetes mellitus, wobei sich hier das Risiko einer Fehlbildung sogar vervierfachen kann. Tabelle 1. Chromosomale Anomalien und prozentuale Häufigkeit angeborener Herzfehlern (AHF) (Lewin, Martin A.G. 1994). Chrom. Anomalie Patienten mit AHF Trisomie 13 90% Trisomie 18 90-99% Trisomie 21 50% Trisomie 22 75% Partielle Trisomie 22 50% 47 XXY 50% XO-Turner Syndrom 40% 4p-Wolf-Syndrom 60% 5p-Cri-du-chat-Syndrom 15% 8p-Deletion 75% 6 Tabelle 2. Genetische Syndrome und prozentuale Häufigkeit AHF (Lewin, Martin A.G. 1994). Single Gen Syndrom Marfan Syndrom 15q 5.23 Williams-Beuren Syndrom 7q11.23 Noonan-Syndrom 12q24.1 Kartagener Syndrom 5p15-p14 Patienten mit AHF 60-80% 75-100% 50% 100% Mikrodelet. 20p11-12 85% Deletion 22q11 Di-GeorgeSyndrom 90% Holt-Oram-Syndrom 12q24.1 100% Tabelle 3. Umweltbedingte Ursachen und prozentuale Häufigkeit AHF (Lewin, Martin A.G. 1994). Umweltbedingte Störungen Patienten mit AHF Rötelnembryopathie 35-50% Trimethadionembryopathie 15-30% Fetale Alkoholembryopathie 25-55% Phenylketonurie bei Mutter 25-30% Isoretinoidembryopathie Diabetische Embryopathie 25% 3-5% 1.5 Häufigkeit und Wiederholungsrisiko der Herzfehler Von 1000 lebend geborenen Kindern sind im Mittel 8-10 (5) mit einer angeborenen Herzgefäßmissbildung belastet. In Deutschland kommen 7000-8000 neugeborene Kinder pro Jahr mit einem Herzfehler zur Welt (3). Für das Auftreten einer Herzgefäßmissbildung spielt, abgesehen von genetischen Defekten und Umweltfaktoren, die familiäre Belastung eine wichtige Rolle. So z.B. steigt die Wahrscheinlichkeit für das Auftreten eines angeborenen Herzfehlers von 0,8 auf bis zu 4% beim Vorliegen eines elterlichen oder 7 geschwisterlichen Herzfehlers. Sind zwei Geschwister betroffen erhöht sich die Wahrscheinlichkeit einer Herzgefäßmissbildung schon auf 10%. (2, 4) 1.6 Methoden der Untersuchung von Chromosomenveränderungen 1.6.1 Entwicklung von Array CGH Die Methode der komparativen genomischen Hybridisierung (CGH), die erstmals im Jahre 1992 vorgestellt wurde (6) erlaubt eine schnelle und umfassende Analyse eines Genoms auf über- und unterrepräsentierte DNA-Abschnitte . Bei der CGH wird die genomische DNA des zu untersuchenden Patienten Test-DNA mit einer ReferenzDNA eines gesunden Spenders verglichen und das Verhältnis der DNA-Kopienzahl für die einzelnen chromosomalen Regionen ermittelt. Test- und Referenz-DNA werden mit unterschiedlichen Fluoreszenzfarbstoffen markiert, zu gleichen Teilen gemischt und mit einem Überschuß an Cot-DNA auf normalen Metaphasenchromosomen hybridisiert. Die Zugabe von Cot-DNA dient der Supression von hochrepetitiven Sequenzen, welche zum Eingehen der unspezifischen Bindungen neigen (Abb. 3 ) Abbildung 3. Prinzip der konventionellen CGH (mit freundlicher Genehmigung von Dr. R. Ullmann) Test und Referenz-DNA konkurrieren um homologe Bindungsstellen auf den Chromosomen. Ist eine Sequenz in der Test DNA unterrepräsentiert, so bindet die Referenz-DNA in Relation häufiger an den homologen Chromosomenabschnitt. Die Farbe der Referenz-DNA dominiert deshalb in dieser chromosomalen Region. Ist eine Sequenz in der Test DNA überrepräsentiert, dann erfolgt überwiegend die Test-DNA 8 Bindung an den homologen Chromosomenabschnitt, die Farbe der Test-DNA wird dominieren. Anhand eines Lungentumors zeigt (Abb.4) wie sich das Hybridisierungsergebnis im Fluoreszenzmikroskop darstellt. Gewinne werden als grüne Balken rechts der Chromosomenidiograme, Verluste links davon gezeigt. Der Karyotyp, üblicherweise eine Zusammenfassung von 5-8 untersuchten Metaphasen, kann mittels Spezialsoftware dargestellt werden (Abb.5) Abbildung 4. Chromosomen einer Lungentumor Probe (mit freundlicher Genehmigung von Dr. R. Ullmann) Abbildung 5. Chromosomale Aberrationen bei einem Tumor (mit freundlicher Genehmigung von Dr. R. Ullmann) Nachteil dieser Methode ist, daß sie aufgrund ihrer Ausrichtung auf Chromosomen nur limitierte Auflösung liefern kann, je nach Art der Dateninterpretation 3-10Mb (7, 31, 33, 37). 9 Mit dem Fortschreiten der Entwicklung der DNA-Chiptechnologie wurde 1997/1998 eine massive Erhöhung des Auflösungsvermögens erreicht. Die Metaphasechromosomen, Hybridisierungsziel bei der konventionellen CGH , wurden durch Arrays von DNA Spots ersetzt, wo jeder Spot (Target) DNA eines bestimmten chromosomalen Abschnittes entspricht. Von den beiden ursprünglichen Namen dieser Variante der CGH, Matrix CGH und Array CGH, konnte sich nur letzterer durchsetzen. (8, 14). Bei der Array CGH Technologie besteht die Wahl zwischen drei Arten von Targets, welche entsprechend der Fragestellung eingesetzt werden können: cDNA, Oligos oder BAC (Bacterial Artificial Chromosome) Fragmente. Die cDNA Arrays wurden ursprünglich für die Analyse der Genexpression verwendet und nur wenige Gruppen setzen sie für Array CGH Experimente ein. Vorteil, und gleichzeitiger Nachteil ist, daß diese Arrays gen-fokusiert sind, da sie durch reverse Transkription von mRNA und nachfolgender PCR hergestellt werden. Die Länge der Fragmente, welche auf den Slide gespottet sind, liegt zwischen 0.5kb und 2.5kb. Rein theoretisch ermöglichen sie daher einen schnellen Vergleich der Genexpression mit der DNA Kopienzahl, da Genexpressionsanalyse und Array CGH auf baugleichen Chips durchgeführt werden können. In der Praxis müssen allerdings zur Reduktion von Signalrauschen oft mehrere cDNA Spots zu einem einzigen Datenpunkt integriert werden, so daß dieser Vorteil wegfällt. Für die Oligo Arrays werden synthetische Oligonukleotide mit einer Länge von ca. 20-70 Nukleotide auf eine Glasoberfläche gebracht (35,36). Dies kann auf zwei Weisen geschehen. Einerseits über photolithographische Masken oder Spiegeltechnik direkt auf dem Array (http://www.affymetrix.com) andererseits durch Spotten von synthetischen Oligonukleotiden auf die Slideoberfläche (http://www.amersham.com/, http://www.home.agilent.com) Es gibt eine Reihe von Problemen, welche bei der Benutzung von Oligo-Arrays auftreten können. Es sind vor allem die schwachen Signalintensitäten. Mit der Abnahme der Nukleotidenlänge steigt die Gefahr der Fehlhybridisierungen. Das ist besonders bei der Verwendung von sehr kurzen Oligonukleotiden (12-25 Nukleotide) der Fall . Um diese Gefahr bei der Auswertung der Oligo Arrays zu minimieren wird für jedes perfect-match-Oligonukleotid (PM) ein zweites sogenanntes mismatch-Oligonukleotid (MM), bei welchem in der Mitte der Oligonukleotidsequenz eine einzelne Base ausgetauscht ist, auf den Chip aufgebracht. 10 Signale, die an Stellen mit mismatch- Oligonukleotiden auftreten, werden als unspezifisch definiert und bei der Auswertung mit der PM Signalintensität verglichen. Durch den Vergleich der Signalintensitäten kann entschieden werden, ob die Hybridisierung an die PM Sequenz spezifisch ist. Derzeit am weitesten verbreitet sind Large Insert Clone Arrays. Die Auflösung von solchen Arrays wird durch die Größe, Zahl und den genomischen Abstand der DNA-Fragmente bestimmt, welche auf den Slide gespottet werden. Als Fragmente werden meistens BAC (Bacterial Artificial Chromosom) -Klone verwendet (Abb.6). Abbildung 6. BAC mit einem klonierten Insert (mit freundlicher Genehmigung von Dr. R. Ullmann) BAC Klone beinhalten im Schnitt ~180 kb große Fragmente genomischer DNA (11, 39). Der derzeit am weitesten verbreitete Chip hat eine genomweite Auflösung von ~ 1 Mb. (9,10, 15,31,40) (16). Dabei das Genom mit Restriktionsenzymen fragmentiert und anschließend kloniert (Abb. 6) (11). Aus dieser Library selektiert man dann die Klone mit entsprechenden Inserts. Die Auflösung der BAC Arrays kann durch die Verwendung von Sets sich überlappender Klone noch deutlich erhöht werden (12, 13,). Solche sogenannte Tiling Path Arrays erlauben einen Detektion chromosomaler Imbalancen 80kb. (17,18). Die Klone für den tiling Path (Human BAC Minimal Tiling Set) wurden vom BACPAC Resources Center http://bacpac.chori.org/pHumanMinSet.htm käuflich erworben . Insgesamt handelt es sich dabei um 32,450 BAC Klone. Die meisten Klone dieses Sets (ca. 30000) stammen von den Rosewell Park Centre Libraries RPCI-11 und -13 (11). Die anderen ~2000 Klone kommen von einer am Caltech etablierten Klonbibliothek. (39). 11 In dieser Arbeit wurde ein BAC-Array benutzt, auf welchem 15 Chromosomen mit 1Mb Auflösung und neun Chromosomen (4, 7, 9, 11, 16, 17, 21, 22, X) mit tiling path Resolution vorhanden sind. Die methodischen Schritte der Array CGH-Analyse sind in Abbildung 7 dargestellt. 1.6.2 Vergleich zwischen Array CGH und molekular-cytogenetischen Methoden Der Vergleich CGH mit Methoden der klassischen Cytogenetik oder FISH zeigt einige Vorteile der CGH. Bei der klassischen Cytogenetik werden Metaphasechromosomen mit Hilfe der unterschiedlichen Bänderungsfärbungen lichtmikroskopisch dargestellt und analysiert. Die spezifische Chromosomenbänderung erlaubt dann die Identifizierung einzelner Chromosomen. Die Abweichungen im Bänderungsmuster sind ein Zeichen für chromosomale Veränderungen wie Deletionen, Inversionen oder Translokationen. Der Nachteil dieser Methode gegenüber der CGH liegt in der Notwendigkeit immer teilungsfähige Zellen für die Gewinnung von Metaphasechromosomen zu haben. Außerdem kann es schwierig sein, Metaphasenspreitung, aufgrund einzelne schlechter Chromosomenmorphologie Chromosomen zu identifizieren oder oder die Veränderungen zu erkennen. Die Fluoreszenz-in-situ-Hybridisierung (FISH) erlaubt das Detektieren bestimmter DNA Sequenzen in einem Zellpräparat. Dabei wird die Sonde mit einem Fluoreszenzfarbstoff markiert und auf ein Objektträger mit Zellkernen (InterphaseFISH) oder Chromosomen hybridisiert. Anschließend kann man unter dem Fluoreszenzmikroskop die Position der Sonde und der gesuchten Zielsequenz erkennen. Wie bei der klassischen Cytogenetik ist es mit FISH möglich, im Gegensatz zur CGH, sowohl balancierte als auch unbalancierte Veränderungen zu erkennen. Der Nachteil der FISH Methode besteht darin, daß vor dem Experiment die Wahl einer geeigneten Sonde erfolgen muß und es nicht möglich ist, das gesamte Genom nach neuen nicht bekannten Veränderungen zu untersuchen. Aus diesem Grund ist die Kombination der Techniken, z.B. FISH und Array-CGH, sehr vielversprechend, da mit Array-CGH geeignete Proben definiert werden könnten, um dann mit FISH Aussagen über deren wirkliche Lokalisation am aberranten Chromosom zu treffen. 12 1.7 Ziel der Diplomarbeit Das Ziel dieser Diplom Arbeit ist die Detektion von chromosomalen Gewinnen bzw. Verlusten in einer Gruppe von Patienten mit angeborenen Herzfehlern. Durch genaue Definition der chromosomalen Lokalisation und Größe der Veränderungen sollen mögliche Kandidatengene und diagnostische Marker definiert werden. 13 2. Material und Methoden 2.1 Chemikalien BAC Verdau: BSA [New England Biolabs] Msel hich conc. [New England Biolabs] BFA [New England Biolabs] NEB1 [New England Biolabs] Aqua ad iniectabile [Baxter] Ligation: Pr12 100 m [MWG Biotech AG] (5′-TAA CTA GCA TGC-3′) Pr21 100 m [MWG Biotech AG] (5′-AGT GGG ATT CCG CAT GCT AGT-3′) Ligase Buffer [Roche] Ligase (5U/l) [Roche] Aqua ad iniectabile [Baxter] Linker Adapter PCR1 : 10Puffer mit MgCl [Roche] 2Puffer mit MgCl [Roche] dNTPs 1mM [Roche] Primer 21 100 m [Roche] Aqua ad iniectabile [Baxter] Mineralöl [Sigma] Polymerase MPI Taq PCR2 : 10Puffer mit MgCl [Roche] dNTPs 1mM [Roche] Primer 21 amino [MWG Biotech AG] Polymerase MPI Taq 14 DNA Sonification: Aqua ad iniectabile [Baxter] DNA Labelling: 2,5 Random Primers [Bioprime labelling Kit- Invitrogen] dNTP-Mix: (Roche – Ansatz für 200ul: je 4ul dGTP, dCTP, dATP (100mM) + 2ul dTTP (100mM) + 2ul 1M Tris pH8 + 0,4ul 0,5M EDTA pH8 +183,6ul H2O) Cy3-dUTP (1nmol /ul) [Amersham] Cy5-dUTP (1nmol /ul) [Amersham] Klenow-Fragment (40 U/ul) Aqua ad iniectabile [Bioprime labelling Kit- Invitrogen] [Baxter] DNA Aufreinigung : QIAquick PCR Purification Kit (250) [Qiagen] Aqua ad iniectabile [Baxter] DNA Fällung : Stop Puffer [Bioprime labelling Kit- Invitrogen] Human-Cot 1DNA (1mg/ml) [Invitrogen] Natrium Acetat 3M [Merck] Ethanol 100% [Merck] CGH Hybridisierung: Yeast tRNA (100mg/ml) [Invitrogen] SDS 10% [Merck] FDST mit 8% Dextransulfat: (0,8g Dextransulfat (Serva 18705) in 5ml Formamid + 1ml 20xSSC add to 7ml (H2O), pH 7,0 (HCL/NaOH) Advason [Advalytix] Fixogum Rubber cement [Marabu] 15 Slidebehandlung: Prehybridisierungslösung: 25% Formamid [Merck] 4x SSC pH 7,0 0,1% SDS [Merck] Hering Sperm DNA (10mg/ml) [Invitrogen] BSA [Sigma] H2O Millipore mit 0,05% Tween 20 [Sigma] SlideWäsche: Waschlösung: 50% Formamid [Merck] 2xSSC pH7 0,1% SDS PN-Buffer: (473,5ml 0,2M Na2HPO4 mit 0,2M NaH2PO4 pH 8 Millipore-H2O bis 1L) 1x PBS mit 0, 05% Tween: 0,22g KCl [Merck] (1,42g Na2HPO4 x 2H2O + 0,2g KH2PO4 + 8,18g NaCl + Millipore-H2O bis 1L pH 7,2-7,4 +500l Tween 20 [Sigma]) Millipore-H2O mit 0,05% Tween 20 [Sigma] 2.2 Geräte und Software BAC Verdau, Ligation , Linker Adapter PCR1, PCR2: Clene Cab [Herolab] Primus HT Cycler [MWG Biotech AG] 5810 R Zentrifuge [Eppendorf] 5413 Zentrifuge [Eppendorf] Sonification: Branson –Sonifier Modell 450 16 Agarose Gel: Agarose electrophoresis Grade [Invitrogen] 1TAE Puffer (Tris-Acetat-Puffer) pH 8,3 HyperLadder I 100 Lanes [Bioline] DNA Labelling- Hybridisierung: GeneAmp PCR System 9700 [AB] Primus HT Cycler [MWG Biotech AG] 5810 R Zentrifuge [Eppendorf] 5413 Zentrifuge [Eppendorf] Reax top Vortexer [Heidolph] Wasserbäder: Innova 3000 [New Brunswick] Grant J84 [Grant Instruments] Grant JB1 [Grant Instruments] Digsi Therm Präzisionsheizplatte Slide Booster [Advalytix] Schüttler GFL 3005 Scanner GenePix 4000B [Axon Instruments] Software Slide Auswertungsprogramm GenePix Pro 5,0 CGH PRO (Wei Chen) 2.3 Methoden 2.3.1 Array CGH Auf Abbildung ist der Prinzip und die einzelnen Methodischen Schritte zusammenfassend dargestellt. 17 Test DNA Control DNA Preparation: BAC/PAC clones Alkaline Lyses Sonication BAC/PAC-DNA MseI/BfaI digest Cy3 Labelling Cy5 Ligation of Adapter-Linker Amplification: BAC/PAC-DNA (Adapter-Linker PCR ) Cot 1 DNA ........................ ........................ ........................ ............ ........................ ............ washing ........................ ............ ........................ ........................ ............ ........................ ........................ ........................ ............ ........................ ........................ ............ ........................ scanning ........................ ............ = material gained in test genome ........................ ............ = material lost in test ........................ ........................ genome ............ ........................ equal = ........................ ............ ........................ abundance ........................ ........................ ............ Abbildung 7. Prinzip der Array CGH (mit freundlicher Genehmigung von Dr. F. Erdogan) ........................ ............ ........................ ............ ........................ ............ ........................ ............ ........................ ............ MTP ............ ............ ............ ............ ............ ............ ............ 18 2.3.2 Präparation der BAC/PAC Klone Für die Herstellung der Arrays wurde DNA des kompletten 32k human ReArray Set vom BACPAC Ressourcen (http://bacpac.chori.org/genomicRearrays.php) Center (BPRC) bezogen. Die 32450 BAC Klone kamen in 417 96-well Platten in einem Volumen von ca. 10µl a.d. gelöst und wurden bis zur weiteren Verarbeitung bei –80°C gelagert. 2.3.2.1 Amplifikation der Klon-Inserts mittels Linker Adapter PCR Die Linker Adapter PCR besteht aus drei Schritten, dem Verdau, der Ligation und der PCR. Abbildung 8 zeigt ein Schema dieser Methode. Abbildung 8 . Linker Adapter PCR (aus Klein et al. 1999) Diese Sonderform der „whole genome amplification“ , welche zuerst von Saunders et al. 1989 (44) beschrieben und von Klein et al. 1999 (19) für die CGH adaptiert wurde, erlaubt eine effektive Amplifikation der genomischen Fragmente, welche dann auf die Slideoberfläche aufgebracht werden können (Abb.4) Für die Verwendung bei der Array CGH in unserer Arbeitsgruppe wurde der Protokoll erheblich verändert. Die modifizierte Version ist http://www.molgen.mpg.de/~abt_rop/molecular_cytogenetics/LinkerPCR.html unter: zu sehen. Die Modifizierung des Protokolls war erforderlich um die Komplexität der 19 PCR Produkte zu erhöhen (Verdau mit 2 Enzymen), Kompatibilität der einzelnen Schritte zu ermöglichen (keine Aufreinigung zwischen den einzelnen Teilschritten notwendig), und um den Anforderungen des Hochdurchsatzes im 96-well Format gerecht zu werden. Um die DNA Ausbeute zu erhöhen und einen Vorrat an PCR Produkten zu schaffen, wurde nach der primären PCR (Ligation vermittelte PCR1) eine sekundäre PCR (PCR2) durchgeführt und erst diese Amplikons zum Spotten auf die Slides verwendet. Während des Verdaus mit MseI/BfaI BAC-DNA geschnitten. Das Restriktionsenzym MseI (New England Biolabs) schneidet die Sequenz TTAA nach dem ersten Thymin mit Produktion von kohäsiven Enden. Das Enzym BfaI (New England Biolabs) schneidet die Sequenz CTAG vor dem Thymin. In beiden Fällen entstehen dadurch AT-Überhänge, an die in der darauf folgenden Reaktion Oligos (12mer und 21mer) ligiert werden. Der Restriktionsverdau-Mix wurde für je 4-96er Well Platten vorbereitet Restriktionsverdauansatz (erstellt mit Microsoft Excel Protokollformular): Einzelprobe l Ins. Pro je 4 Platten l BSA 0,0750 36,00 MseI high conc. 0,0120 5,76 Bfa 0,1500 72,00 NEB 1 0,7500 360,00 a.d. 1,5130 726,24 Summe: 2,5000 1200,00 Je 2,5 l dieses Restriktionsverdauansatzes wurden mit 5l der BAC-DNA mittels Multi-channelpipette gemischt. Die Platten wurden mit Adhesive PCR Film (Abgene) abgedeckt und kurz abzentrifugiert (5810 R Zentrifuge, Eppendorf). Zur Verhinderung übermäßiger Evaporation wurde die Reaktion selbst in einem einem Thermocycler mit beheizbarem Deckel durchgeführt (Primus HT Cycler). Dabei wurde das folgende PCR Programm benutzt: 37°C 3h, 80°C 20min, 4°C . Nach dem Verdau wurden die Platten bei -20°C gelagert. 20 2.3.2.2 Ligation Bei der Ligation dient das 12mer (5′-TAA CTA GCA TGC-3′) über seine Bindung an den im MseI/BfaI Verdau generierten AT-Überhang als Gerüst für das 21mer (5′-AGT GGG ATT CCG CAT GCT AGT-3′), welches durch die Ligase kovalent an die DNA gebunden wird. Reaktionsansatz für Ligation (erstellt mit Microsoft Excel Protokollformular): Einzelprobe l Ins. Pro je 4 Platten l Pr12 100µm 0,5 240 Pr21 100µm 0,5 240 Ligase Buffer 0,8 384 a.d 5,2 2496 7 3360 Summe Je 7µl dieses Reaktionsansatzes wurden mittels Multi-Channelpipette in 96 Well Platten vorgelegt und anschließend je 1 l Restriktionsansatz aus MseI/Bfa Verdau pro Well zugegeben. Die Platten wurden mit Adhesive PCR Film (Abgene) abgedeckt und für 30 sec abzentrifugiert (Eppendorf 5810 R). Wie der Restriktionsverdau fand auch die Ligation ebenfalls in Thermocyclern mit beheizbarem Deckel statt (Primus HT Cycler MWG Biotech AG) . Programm : 65°C 2min1°C/min auf 15°C 15°C Die 65°C dienen der Denaturierung der Oligo-dimere, die sich dann während der schrittweisen Reduktion der Temperatur an die DNA-Fragmente anlagern. Nach dem Abkühlen auf 15 Grad wurde der PCR Film (Abgene) entfernt, ohne die Platten aus dem Cycler zu nehmen, und mit der Multichannel Pipette (Brand) pro Well 2 µl Ligase Mix zugegeben, anschließend wurden die Platten wieder mit PCR Film (Abgene) versiegelt. 21 Ligasemix (erstellt mit Microsoft Excel Protokollformular): Einzelprobe l Ligasemix Ins. Pro je 4 Platten l Ligase Buffer 0,2 96 Ligase high conc. (5U/µl) 0,2 96 a.d 1,6 768 2 960 Summe Die Ligation fand bei 15°C über Nacht statt. 2.3.2.3 Ligation vermittelte PCR 1 Bei der PCR 1 findet die eigentliche Amplifikation der DNA statt. Nach dem Abspalten des unligierten 12-mers durch Erwärmung auf 68°C findet eine Auffüllreaktion statt und die Sequenz des 21-mers kann als universelle primerAndockstelle für alle DNA Fragmente genutzt werden. PCR1 Mix (erstellt mit Microsoft Excel Protokollformular): Einzelprobe l 10x Puffer mit MgCl Ins. Pro je 4 Platten l 5 2400 dNTPs 1mM 10 4800 Primer 21 100µM 0,5 240 32,5 15600 48 23040 a.d. Summe Je 48 l vom PCR 1 Mix wurden mittels Multichanel Pipette in PCR-Platten vorgelegt. Danach wurde 1l vom Ligationsansatz dazu pipettiert. Die Proben 22 wurden anschließend mit Mineralöl (PCR grade) überschichtet und die Platten mit Adhesive PCR Film (Abgene) abgedeckt. Als Negativkontrollen wurden drei PCR Tubes (Eppendorf) mit PCR Mix ohne DNA benutzt. PCR Programm: 68°C 3min PAUSE Während der Pause wurde der Adhesive PCR Film entfernt und je 1 l Polymerase Mix pro Well mit der Multichannel Pipette zugegeben. Anschließend wurden die Platten mit einem neuen Adhesive PCR Film abgedeckt Reaktionsansatz für Pol. Mix (erstellt mit Microsoft Excel Protokollformular): Polymerase-Mix Einzelprobe l Ins. Pro je 4 Platten l 2x Puffer mit MgCl 0,5 500 Polymerase 0,5 500 1 1000 Summe Danach wurde das PCR Programm fortgesetzt: 68°C 4min, 35x (94°C40sec 59°C30sec 72°C90sec+2/cycle) 94°C40sec 59°C30sec 72°C 7min 4° 23 2.3.2.4 PCR 2 Ein Aliquot der PCR1 wurde als Template für die PCR2 genutzt. In der zweiten PCR wurden amino-modifizierte 21-mere eingesetzt. Die Amino Gruppe verbessert die Bindungsrate der DNA an die Epoxy-Slides. Reaktionsmix für PCR2 (erstellt mit Microsoft Excel Protokollformular): PCR2 Mix Einzelprobe l Ins. Pro je 4 Platten l Puffer mit Mgcl 7,5 3600 dNTPs 1mM 15 7200 Primer 21amino 1,5 720 Pol MPI unverd. 0,75 360 49,25 23640 74 35520 a.d. Summe Je 74 l vom PCR2 Mix wurden mittels Multichannel Pipette vorgelegt in PCR Platten vorgelegt. Danach 1l aus der Linker Adapter PCR 1 dazu pipettiert. Die Platten wurden mit Adhesive PCR Film (Abgene) abgedeckt und für 30 sec abzentrifugiert (Eppendorf 5810 R). Als Negativ-Kontrollen wurden auch hier wieder drei einzelne Reaktionsnsätze ohne DNA-Template mitgefahren. Im PCR Block (Primus HT Cycler MWG Biotech AG) mit Programm: 94°C2min 35x (94°C40sec 59°C30sec 72°C90sec+2/cycle) 94°C40sec 59°C30sec 72°C 7min 4° 24 2.3.2.5 Agarosegelelektrophorese zur Kontrolle der PCR Produkte Zur Kontrolle der PCR Produkte und Beurteilung dessen Qualität wurde sowohl nach der PCR 1 als auch PCR 2 eine Agarosegelelektrophorese durchgeführt. Bei der Gelelektrophorese wandern die PCR Produkte von Kathode zu Anode in einem elektrischen Feld. Da die größeren Fragmente pro Zeiteinheit langsamer als die kleineren PCR Fragmente laufen, werden sie der Größe nach im Agarose Gel aufgetrennt. Durch die anschließende Inkubation in Ethidiumbromid Lösung können die DNA Fragmente im UV-Licht sichtbar gemacht. Marker (HyperLadder I 100 Lanes von Bioline) wurde zur Fragmentgrößebestimmung auf das Gel mitaufgetragen. Gel: 1% Agarose (1g Agarose (Invitrogen) + 100ml 1TAE Puffer Pro 96-well Platte wurden aus Kostengründen nur jeweils vier Proben auf das Gel aufgetragen. Dafür wurden je 8µl des PCR Produktes mit 2µl Loading Buffer gemischt und in die Geltaschen pippetiert. Zur Bestimmung der Fragmentgröße wurden 7µl Marker (HyperLadder I 100 Lanes von Bioline) mitaufgetragen. Die Auftrennung der Fragmente erfolgte bei 100 V für 30 min (Consort E425 von Fröbel) Nach der Färbung des Gels in einer Ethidiumbromidlösung für 10min wurde das Ergebnis im Geldokumentationssystem photographiert. 2.3.2.6 Ethanolpräzipitation der DNA Zur Reduktion des Volumens und einer teilweisen Aufreinigung (Elimination der freien Nukleotide etc.) wurde die PCR 2 Reaktion gefällt, in 70% Ethanol gewaschen. Nach dem Trocknen wurden die Pellets in 14µl Spottingpuffer (3xSSC/1,5M Betain) aufgenommen. Reaktionsansatz für Fällungsmix (erstellt mit Microsoft Excel Protokollformular): 25 Fällungsmix Einzelprobe l 1 -96 Well Platte l Ethanol 165 85800 NaAC 7,5 3900 172,5 89700 Summe Der Fällungsmix wurde mit einer Multichannel Pipette in 96-well Fällungsplatten vorgelegt. Anschließend wurden die PCR 2 Produkte zugegeben und für mind. 24h bei –20 °C gefällt. Nach der Fällung wurden die Platten für 60 min bei maximaler g-Zahl abzentrifugiert mit 70% Alkohol gewaschen und anschließend getrocknet. Dann wurden die PCR Produkte resuspendiert (3SSC, 1.5M Betaine) und mit einem Pipettierroboter (Beckmann) in 384-well Platten transferiert. 26 2.3.3 Produktion der DNA Arrays Die Vorbereitungen zum Spotten der DNA Arrays (durchgeführt von AG Hultschig/Abt.Lehrach) umfassen mehrere Punkte. Zuerst werden die unter 2.3.2.6 beschriebenen 384-well Platten mit einem Barcode versehen und eine „Erwartungsliste“ erstellt, die sowohl Reihenfolge der Platten als auch deren Belegung wiedergibt. Diese Liste dient in weiterer Folge der Steuersoftware zur Erstellung des sogenannten „gal-files“, dessen Verwendung im Punkt 2.3.15: Auswertung noch näher beschrieben wird. Als Spotting-Substrate verwendeten wir Epoxy beschichtete Objektträger von den Firmen NUNC (http://www.nunc.de/ und Advalytix (http://www.advalytix.de/) . Beim Spotten taucht ein Set von 48 „split pins“, das sind Nadeln mit einem Spalt an der Spitze, in die Flüssigkeit ein und nimmt bedingt durch Kapillarkräfte eine gewisse Flüssigkeitsmenge auf. Abbildung 9 zeigt einen „48 pin printhead“ und einen Spotter der Firma Biorobotics. Abbildung 9. Spotter (rechtes Bild), Nadel-Set (unteres Bild) und Array Slide (das linke mittlere Bild) CGH (mit freundlicher Genehmigung von Dr. R. Ullmann) 27 Durch physikalischen Kontakt der Nadeln mit der Slideoberfläche wird eine geringe Menge Flüssigkeit auf dem Slide deponiert. In dieser Studie war jeder der 14182 Klone in zweifacher Ausführung (Duplikat) gespottet. Diese Redundanz ermöglicht das Erkennen und das Ausgleichen räumlicher Effekte und erlaubt auch im Falle des Verlustes eines Spots eine Aussage über die betreffende Region. Das Array besteht aus mehreren Subgrids (Abb.9). Jeder Subgrid (Spotsblock) ist mit einer einzigen Nadel gespottet. Es können Unterschiede in der Nadelabnutzung oder Nadelspalten zwischen einzelnen Spottnadeln in einem Set auftreten. Diese Variationen können auf dem Slide räumliche Effekte verursachen. 2.3.4 Patienten DNA . Die 38 DNA Proben der Patienten mit congenitalen Herzfehlern wurden von Frau Dr. Sperling (Abt. Prof. Dr. Hans Lehrach) für diesen Projekt zur Verfügung gestellt. In der Tabelle 4 sind die Patienten DNA Proben und die entsprechenden Isolierungsmethoden aufgelistet. 28 Tabelle 4. DNA Material von Patienten mit congenitalen Herz Fehlern. Nummer Probe Conc µg/µl Geschlecht Methode der DNA Isolierung µl µg 1 113E 0,131 F Qiagen 38,11 5 2 071E 0,093 M Qiagen 53,76 5 3 208E 0,192 F TritonX 26,11 5 4 328E 0,364 F NP40 13,74 5 5 111E 0,135 F Qiagen 36,98 5 6 067E 0,226 F Qiagen 22,09 5 7 133E 0,473 M TritonX 10,58 5 8 281E 0,077 F Qiagen clean up 64,94 5 9 350E 0,252 M NP40 19,84 5 11 340E 0,351 M NP40 14,26 5 12 082E 0,185 F Qiagen 26,97 5 13 349E 0,414 M NP40 12,07 5 14 056E 0,25 F Qiagen 20 5 15 186E 0,363 F TritonX 13,79 5 16 142E 0,121 M Qiagen clean up 41,32 5 17 386E 0,189 M NP40 26,46 5 18 206E 0,167 M Qiagen clean up 29,94 5 19 373E 0,508 M NP40 9,84 5 20 091E 0,21 F Qiagen 23,87 5 21 162E 0,398 F TritonX 12,58 5 22 079E 0,115 M Qiagen 43,55 5 23 139E 0,445 F TritonX 11,25 5 24 368E 0,392 M NP40 12,76 5 25 011E 0,164 F Qiagen 30,49 5 26 010E 0,108 M Qiagen 46,51 5 28 007E 0,104 M Qiagen 48,31 5 29 261E 0,079 M Qiagen clean up 63,29 5 30 137E 0,551 M TritonX 9,08 5 31 077E 0,183 F Qiagen 27,32 5 32 127E 0,157 F Qiagen 31,91 5 33 174E 0,418 F TritonX 11,96 5 34 210E 0,485 F TritonX 10,32 5 35 284E 0,477 F TritonX 10,49 5 36 351E 0,462 M NP40 10,83 5 37 399E 0,321 M NP40 15,58 5 38 400E 0,642 F NP40 7,79 5 29 2.3.4.1. DNA Isolierung. Für die Array-CGH wird genomische Test-DNA und Referenz-DNA (Kontroll-DNA) benötigt. Test- und Referenz-DNA wurden aus Lymphozyten des peripheren Vollbluts isoliert. Um die Effekte unterschiedlicher DNA- Isolierungsprotokolle auf die Qualität der array CGH Untersuchungen zu bestimmen wurde DNA nach drei verschiedenen Protokollen isoliert (Tab.4) 2.3.4.2 DNA Sonification. Die genomische DNA wird bei der Sonification durch Ultraschall in Stücke mit einer durchschnittlichen Länge von 200-1200bp fragmentiert. Diese Länge erlaubt einerseits durch Reduktion der Sekundärstrukturen in der Random Priming Reaktion die optimale Markierung. Gleichzeitig verringert die geringere Fragmentlänge den DNA-Verlust bei der anschließenden Aufreinigung Das Design des Branson-Sonifiers erforderte die Aufnahme der DNA in 200µl a.d.. Die Fragmentierung erfolgte bei einer konstanten Intensität (Stufe 4) für vier Sekunden. Die Proben wurden anschließend auf Eis gelagert. 2.3.4.5 Agarosegelelektrophorese zur Kontrolle der sonificierten Proben. Nach dem Sonificieren wurden je 7 µl des sonificierten Volumens entnommen, mit 3µl Loading Buffergemischt und auf ein 1%-iges Agarosegel (1g Agarose (Invitrogen) + 100ml 1TAE Puffer) aufgetragen und die DNA- Fragmente auf ihre Längenverteilung hin überprüft. 2.3.4.6 DNA Aufreinigung. 30 Um sicherzugehen, daß die in die Random Priming Reaktion eingesetzte DNA frei von jeglicher die Kontamination ist, wurde die DNA vorher mit dem QIAquick PCR Purification Kit (Qiagen) aufgereinigt . Der QIAquick PCR Purification Kit (Qiagen) erlaubt die Aufreinigung von Einzel- und Doppelsträngiger DNA mit einer Größe von 100 bp bis 10 kb über eine Säule. Die Bindungskapazität der Säule beträgt bis zu 10 µg DNA. Bei der Aufreinigung wird die selektive Bindung von DNA in Gegenwart hoher Salzkonzentrationen und bei neutralem pH-Wert (pH 7.5) an eine Silika-GelMembran ausgenutzt. Während kurze DNA Fragmente, Nukleotide und Salze die Membran ungehindert passieren und durch mehrere aufeinanderfolgende Wasch- und Zentrifugationsschritte entfernt werden, bleiben längere DNA Stücke in der Membran. Die gereinigte DNA kann anschließend mit einem kleinen Volumina (240µl) Aqua ad iniectabile (Baxter) eluiert werden. Die DNA Aufreinigung erfolgte nach dem Protokoll QIAquick Spin Purification Procedure (Qiagen). Nach der Aufreinigung wurde die Konzentration der DNA im Spectrometer (Perkin Elmer) gemessen und die DNA anschließend bei –20°C gelagert. 2.3.9 Labelling Es gibt mehrere Methoden um DNA zu markieren. Man kann prinzipiell zwischen einer indirekten enzymatischen Markierung, einer chemischen Markierung und einer direkten enzymatischen Markierung unterscheiden. Bei der indirekten Markierung wird ährend der Neusynthese eines Nukleinsäure-Strangs Amino-AllyldNTPs miteingebaut. In einem zweiten Schritt (Postlabeling-Schritt) wird über eine Esterbindung das Fluorchrome an die an die Aminoallygruppe der DNA gekoppelt Die Amino-Allyl-dNTPs unterscheiden sich kaum in ihrer Größe von den nicht modifizierten dNTPs, was einen Vorteil beim enzymatischen Einbau mit sich bringt. Bei einer chemischen Markierung (z.B. Universal Linkage System ULS von Amersham) wird ein Cis-Platinum Komplex für die DNA Markierung benutzt. Dieser Komplex hat zwei Bindungstellen: die erste für die Bindung an ein Fluorophore, z.B. Cy3 oder Cy5, und die zweite für die Bindung an einen Guanin-???Rest??? in der 31 DNA (45). Der Vorteil dieser Methode liegt in der Einfachheit des Protokolls, der kurzen Markierungsdauer von nur 15min (Amersham ULS-Kit) und der Möglichkeit, auch sehr kurze DNA Fragmente markieren zu können. Für die direkte enzymatische Markierung gibt es mehrere Verfahren. (Abb.10) Eine davon ist Nick Translation (Rigby et al. 1977). Bei dieser Methode werden durch Behandlung mit DNase DNA-Einzelstrangbrüche, sogenannte Nicks generiert. Bei der darauf folgenden „Reparatur“ mit DNA-Polymerase werden modifizierte Nukleotide eingebaut. Die für die Array CGH am häufigsten verwendete Makierungsmethode ist das „random priming“ (Feinberg und Vogelstein 1983 (20)). Hierbei wird die zu markierende doppelsträngige DNA denaturiert und mit Zufallshexameren bzw. –nonameren (random primer) hybridisiert, die dann als Primer für eine DNA Polymerase (Klenow Fragment) dienen. Auch hier erfolgt die Markierung durch Einbau von Cy3-dUTP (Abb.11) bzw. Cy5-dUTP (Abb.12) Abbildung 10. Methoden der DNA Markierung. 32 Abbildung 11. Struktur von Cy3 (Amersham Biosciences) Abbildung 12. Struktur von Cy5 (Amersham Biosciences) In dieser Studie wurde die fragmentierte genomische DNA mittels RandomPriming fluoreszenzmarkiert. Jeweils 3x1µg wurden entsprechend dem Herstellerprotkoll markiert und später wieder gepoolt. Dafür wurde 1µg DNA in ein Volumen von 32,4µl a.d aufgenommen und mit 32µl Primermix vermischt. Danach erfolgt die Denaturierung bei 98°C für 10 min und, damit die DNA-Stränge nicht sofort renaturieren, für 5 min auf Eis gelegt, Reaktionsansatz für 1µg DNA: dNTP-Mix für CGH Aqua ad injectabile Cy3-bzw. Cy5-dUTP Klenow -Enzym Summe 8µl 4,6µl 2µl 1µl 15,5µl Die Markierung fand über Nacht bei 37 °C im Wasserbad statt. 2.3.10 Proben Aufreinigung 33 Die Markierungsreaktion wurde durch Zugabe von 15µl Stop Puffer (Invitrogen) unterbrochen. Danach erfolgte die Aufreinigung der Proben mittels Qiaquick PCR Purification Kit (Qiagen) nach dem Herstellerprotokoll. Die Vorgehensweise ist bereits im Punkt 2.3.8 (DNA Aufreinigung) beschrieben worden. . Die Aufreinigung ist notwendig um Proteine und nicht eingebaute modifizierte Nukleotide zu entfernen. 2.3.11 DNA Fällung In Gegenwart von einwertigen Kationen lassen sich unter Zugabe von absolutem Ethanol und bei Temperaturen -20°C DNA Fragmente aus wäßrigen Lösungen ausfällen. Die mit Cy3 dUTP und Cy5 dUTP gelabelte Proben werden vereinigt, gemischt und in 2 Volumen von je 240µl aufgeteilt. Zu je 240µl gelabelter Proben wurden zugegeben: Human Cot 1 DNA (Roche) 250µg Natriumacetat 3M 29µl Ethanol 100% 800µl Zur Reduktion des Volumens wurden 500µl (1mg/ml)Human Cot 1 DNA (Roche) im Eppendorf Concentrator auf ein Volumen von 100µl eingeengt . Die Zugegebene Menge von 250µg war folglich im Volumen von 50µl vorhanden. Die Fällung fand über Nacht bei –20°C statt. 2.3.12 Slidebehandlung. Die Slidebehandlung wurde durchgeführt um die unspezifische DNA Bindung an die Slideoberfläche zu minimieren. Zu diesem Zweck werden die Slides in einer Lösung mit Hering Sperm DNA und BSA (Bovine Serum Albumin) inkubiert. Prähybridisierungslösung: 34 25% Formamid (Merck) 4x SSC pH 7,0 0,1% SDS (Merck) Die Slides wurden in 60 ml Prähybridisierungslösung mit 0,3g BSA (Sigma) und 200µl Hering Sperm DNA (10mg/ml, Invitrogen) im Wasserbad bei 42°C für 1 Stunde inkubiert. Diese Prähybridisierungslösung wurde in einem Waschschritt mit a.d./0,05%Tween 20 (Sigma) entfernt und die Slides durch Zentrifugation getrocknet. Bis zur Verwendung wurden Array-Slides in einer speziellen Slidebox bei RT gelagert. 2.3.13 Hybridisierung Als Hybridisierung bezeichnet man die nichtkovalente Bindung zweier zueinander komplementärer, einzelsträngiger Nukleinsäuren unter Ausbildung von Wasserstoffbrückenbindungen zwischen den heterozyklischen Basen der Nukleinsäuremoleküle. Um die Hybridisierung zu ermöglichen müssen Nukleinsäuren zuerst durch Erwärmung über den Schmelzpunkt Tm hinaus denaturiert werden. Bei der Denaturierung werden die Wasserstoffbrückenbindungen zwischen den beiden Strängen zerstört und die Bindung der Doppelstränge aufgehoben Die Stabilität von DNA-Doppelsträngen wird von verschiedenen Faktoren beeinflußt, wie z.B. der Basenzusammensetzung (GC Gehalt), der Konzentration monovalenter Kationen (wie Na+), Sequenzlänge und der Anwesenheit von, den Doppelstrang destabilisierenden Agenzien (z.B. Formamid). Der Zusammenhang aller genannten Faktoren wird durch folgende Formel beschrieben. Schmelztemperatur (Tm) nach (Meinkoth und Wahl 1984) Tm = 81,5°C + 0,41 (%GC) + 16,6 log [Na+] - 500/n - 0,61(%Formamid) Die Temperatur ist ein wichtiger die Stringenz beeinflussender Faktor der Hybridisierung. Hohe Hybridisierungstemperaturen erhöhen die Spezifität der Doppelstrangbindung in dem die Wärmezufuhr der Ausbildung und der Stabilität der Wasserstoffbrückenbindungen entgegen wirkt. Niedrige Hybridisierungstemperaturen dagegen fördern unspezifische Doppelstrangpaarungen. 35 Die Stabilität der Doppelstrangbindung kann zusätzlich durch die Erhöhung der Salzkonzentartion in der Hybridisierungslösung erhöht werden. Die Hybridisierungstemperatur (Th) (nach Britten und Kohne 1968) wird wie folgt berechnet : Th.= Tm-25°C Die optimale Hybridisierungstemperatur für DNA/DNA Doppelstrang mit einer Länge bis ~ 5kb liegt etwa 25°C unter der Schmelztemperatur. Die Hybridisierung erfolgt nach der Denaturierung. Der entscheidende Schritt bei der Hybridisierung ist die Bildungshäufigkeit der ersten Basenpaarungen (Nucleation). Von diesem Punkt aus erfolgt die Ausbildung des Doppelstrangs nach dem Reißverschlussprinzip in beide Richtungen (Zippering). Die gefällten DNA Proben wurden bei -4°C, für 30 min zentrifugiert ( Eppendorf 5417R). Der Überstand wurde verworfen und mit 100µl Ethanol 70% gewaschen. Danach wurden die DNA-Pellets bei 37°C für 10min im Thermomixer (Eppendorf) getrocknet. Bei jeder Hybridisierung (2 Pellets) wurde der erste Pellet in 3,375µl Yeast tRNA (100mg/ml Invitrogen), 6,75 SDS 10% ( Merck) gelöst und anschließend mit dem Zweiten vereinigt. Danach wurde durch Zugabe von 23,625µl FDST (0,8g Dextransulfat (Serva 18705) in 5ml Formamid + 1ml 20xSSC add to 7ml (H2O), pH 7,0 (HCL/NaOH) ) der Hybridisierungsmix komplettiert. Die Proben wurden für 15 min bei 70°C im Wasserbad (Grant J84) denaturiert. Danach folgte die Inkubation für 2 Stunden bei 42°C im Wasserbad (Grant J84). Während der Inkubation suppremiert die im Überschuß zugegebene ungelabete Cot 1 DNA (Roche) die hoch repetitiven Sequenzen der Proben- und Target-DNA. Die DNA hoch repetitiver Abschnitte, wie z.B. im Zentromerbereich der Chromosomen, weisen eine andere Hybridisierungskinetik auf (sie reassoziieren schneller) als die einmalig vorkommenden Sequenzen und würden außerdem aufgrund Ihrer Sequenz unspezifisch an vielen Stellen im Genom binden. Nach dem Präannealing wurden die Proben auf einer auf 42°C vorgewärmten Heizplatte (Digsi Therm) auf die behandelten Slides aufgetragen, mit einem Deckglas eingedeckt und für 24 Stunden im Slide-Booster (Advalytix) hybridisiert. Der Slide Booster (Abb.13) besteht aus vier Hybridisierungskammern für je einen Slide. Die Temperatur und Mischparameter können über das Steuergerät für jede Kammer separat eingestellt werden. Unter der 36 Metallplatte sind Nanopumpen installiert. Diese Pumpen produzieren in einstellbaren Abständen akustische Oberflächewellen. Die Wellenbewegung wird von einer Flüssigkeit (Advason von Advalytix), welche zwischen die Pumpe und den Slide pipettiert wurde, durch den Slide auf Hybridisierungslösung übertragen. Diese Wellen fördern die Durchmischung der Hybridisierungslösung und führen dadurch zu einer Verbessserung der Reaktionskinetik. Abbildung 13. Slide Booster von Advalytix 2.3.14 Waschen der Slides 37 Die Slides wurden stringent gewaschen um die falsch gebundene bzw. nicht gebundene DNA zu entfernen. Die Stringenz der einzelnen Waschschritte wird durch den Formamid Gehalt (je höher die Formamid Konzentration ist, desto instabiler werden die Wasserstoffbrückenbindungen), die Temperatur (hohe Temperatur fördert die Einzelstrangbildung), pH, Probenlänge, Salzgehalt der Waschlösungen und die Inkubationszeit bestimmt. Je stringenter die Bedingungen gewählt werden, desto größer die Reduktion des unspezifischen Hintergrunds. Bei zu hoher Stringenz können aber auch spezifische Bindungen gelöst werden. Die Balance zwischen der Hintergrundminimierung und Spotsintensität wird durch Optimierung der Waschbedingungen erreicht und braucht mehrere Versuchsreihen. Die Deckgläser wurden bei Raumtemperatur in 2xSSC pH7 vom hybridisierten Slide gelöst. Slides wurden dann im Wasserbad 15 min. bei 42 in Waschlösung: 50% Formamid (Merck), 2xSSC pH7, 0,1% SDS (Merck). Nach der Behandlung mit Waschlösung die Slides wurden in PN Puffer (473,5ml 0,2M Na2HPO4 mit 0,2M NaH2PO4 pH 8 Millipore-H2O bis 1L) für 10 min bei Raumtemperatur auf dem Schüttler (GFL3005) inkubiert um die Reste von Waschlösung abzublocken und zu entfernen. Nach PN Puffer die Slides wurden in 1x PBS mit 0, 05% Tween: (1,42g Na2HPO4 x 2H2O + 0,2g KH2PO4 + 8,18g NaCl + 0,22g KCl Millipore-H2O bis 1L pH 7,2-7,4 +500l Tween 20 [Sigma]) für 30 sec bei Raumtemperatur gewaschen. Anschließend wurden die Slides kurz in Millipore-H2O mit 0,05% Tween 20 [Sigma] eingetaucht und bei 900 Upm für 5 min bei Raumtemperatur zentrifugiert (5810 R Zentrifuge von Eppendorf). 2.3.15 Auswertung 2.3.15.1 Scannen der Slides 38 Für das Einlesen der Slides wurde ein ein Zweifarben-Laserscanner GenePix 4000B (Axon Instruments) verwendet. Die Slideoberfläche wird mit Laser abgetastet und die Fluorochrome, Cy3 bei 532nm und Cy5 bei 635nm, simultan angeregt. die emittierten Signale werden über Strahlenleitsystem dann getrennt detektiert. Zur Verstärkung der Signale verwendet dieser Scanner ein Photomultiplier-Tube (PMT). Vor dem Scannvorgang wurde der Level der Signalverstärkung (PMT-Gain) so eingestellt, daß die durchschnittliche Signalintensität der beiden Farbkanäle ungefähr gleich war. Die mit einer Auflösung von 5µm gescannten Signale wurden, für jeden Farbkanal getrennt, als 16bit schwarz/weiß TIFF-Bilder auf einem zentralen UnixServer abgespeichert. 2.3.15.2 Bildverarbeitung Die gespeicherten Bilder wurden mit GenePix Pro (Version 5.0) bearbeitet. Die Die Bildbearbeitung umfasste die Segmentierung der Bilder, die Zuordnung der Kloninformation zu jedem Spots und das Eliminieren von Spots bzw. Arealen mit morphologisch unzureichender Qualität. Bei der Segmentierung wird ein dem Layout des Arrays entsprechendes Raster eingeblendet. Durch exaktes Positionieren dieses Rasters legt man fest, welche Pixel einem spezifischen Spot zugerechnet werden. Gleichzeitig wird die Identität jedes Spots durch Import des vom Spotter generierten „gal-file“ festgelegt. In diesem „galfile hat die Steuersoftware des Spotters die Information abgelegt, an welcher Stelle sie welche Probe gespottet hat. (Abbildung 14) 39 Abbildung 14. Interface von GenePix Pro (Version 5.0). Ein Raster ist über die Spots gelegt worden (screenshot) 2.3.15.3 Datenverarbeitung Die eigentliche Analyse der chromosomalen Imbalancen passierte mittels spezieller Array CGH Auswertesoftware. Für diese Arbeit wurde die in dieser Abteilung geschriebene Software CGHPRO verwendet. Nach Überprüfung der Hybridisierungseigenschaften, wie z.B. Verteilung der Ratios im Scatterplot und der Ratioverteilungen zwischen den einzelnen Subgrids (Boxplot), wurden die Signalintensitäten normalisiert . Ziel der Normalisierung ist es, die Signalintensitäten der beiden Farbkanäle vergleichbar zu machen, um eine systematische Dominanz einer Farbe zu verhindern. Solche systematischen Ungleichgewichte können z.B. durch unterschiedliche Konzentrationen eingesetzter DNA, unterschiedliche Labellingeffizienz und Scanningparameter entstehen. Basierend auf dem Median der Signalintensitäten der zwei Farbkanäle wurde die Normalisierung für jeden Subgrid separat durchgeführt. Danach wurden die errechneten Ratios jedes Klons entsprechend ihrer chromosomalen Lokalisation entlang der 40 Chromosomenidiogramme dargestellt. (im Anhang). Damit Verluste eines Chromosoms genauso gut erkennbar sind wie Gewinne, wurden die Ratios als Logarithmus zur Basis2 dargestellt (log2 Ratio von 2=1; log2 Ratio von 0,5=-1). Verluste in der Test-DNA sind links, Gewinne rechts einer gelben Linie angezeigt, die den balancierten Staus repräsentiert. Die grüne und rote Linie markieren die Schwellenwerte für Gewinne und Verluste. Die Bestimmung der geeigneten Schwellenwerte ist unter dem Aspekt „Sensitivität versus Spezifität“ zu sehen und wird von verschiedensten Punkten beeinflußt, wie z.B. Qualität der Hybridisierung, dem zu erwartenden Anteil an Zellen mit der Veränderung (Normalzellkontamination/Mosaik), Polyklonalität, Ploidie etc. Glücklicherweise wirken sich einige dieser Punkte bei konstitionellen Chromosomenanomalie nicht aus, weswegen für alle Fälle dieser Arbeit die Schwellenwerte mit log2 =0,3 bzw –0,3 festgelegt wurden. Dies entspricht in etwa den in der chromosomalen CGH mehrheitlich verwendeten Schwellenwerten (0,8 und 1,2; nicht log Darstellung). Nach Visualisierung der Ergebnisse wurden diese in einer Mysql Datenbank auf dem zentralen UNIX Server abgelegt, um für die weitere Meta-analyse zur Verfügung zu stehen. Abbildung 15. Eiteilung der einzelnen Klone nach ihrem Inhalt an segmentalen Duplikationen In CGHPRO wird den einzelnen Klonen, entsprechend ihres Gehalts an segmentalen Duplikationen, eine bestimmte Farbe zugeordnet (Abb.15) Dazu errechnet das Programm für jeden Klon einen Faktor, aufgrund dessen dieser einer von sieben Kategorien zugeordnet wird. Die der Berechnung zugrunde liegende Formel 41 (Längen der Duplikation Anzahl der Kopien)/ Klon Länge) berücksichtigt sowohl den relativen Anteil der segmentalen Duplikation an der Gesamtsequenz als auch die Kopienzahl. Das Genom des Menschen enthält viele Sequenzfolgen, die mehr als einmal im haploiden Genom auftreten und folglich keine „Unikate“ sind. „Single copy“ Sequenzen, repräsentieren nur einen kleinen Teil des Genoms. Den größten Anteil stellen die mittel- und hochrepetitiven Sequenzen etwa 70% des Gesamtgenoms. Zu den hochrepetitiven Sequenzen gehören Satelliten-DNA, LINEs und SINEs. Mittelrepetitive Sequenzen sind rRNA- und tRNA-Gene sowie auch Retrotransposons. Low-Copy Repeats (LCR) sind definiert als DNA Sequenzen von mindestens 1- bis mehr als 200kb Länge und einer Sequenzsimilarität von mindestens 90%. Bei LCRs handelt es sich keineswegs um selten vorkommende Ausnahmeerscheinungen, vielmehr machen LCRs ca. 5% des gesamten humanen Genoms aus. Positionen vieler dieser “duplizierten ” Sequenzen sind auf der Genom-Datenbankresourceseite (http://genome.ucsc.edu/) zu sehen. Bereits mehrfach konnte das Auftreten von Chromosomen-Brüchen mit dem Vorhandensein von LCRs an der jeweiligen Bruchstelle assoziiert werden (22, 41, 42). Low Copy Repeats sind hoch dynamische Regionen im Genom und gelten daher als Hot Spots für Rekombinationsereignisse (43). Abgesehen von der biologischen Bedeutung dieser Sequenzen sprechen auch technische Aspekte für eine Kolorierung der Klone entsprechend ihres LCR-Gehalt: Einerseits zeigt ein Ratioshift immer nur einen Durchshschnittswert, der über alle chromosomalen Positionen mit dieser Sequenz mittelt, andererseits entspricht der Ratioshift nicht dem einer „single copy“ Sequenz, da der Verlust einer Kopie keiner Kopienzahländerung um 50% entspricht. 42 3. Ergebnisse Probe NR. 1 (113E) Bei der DNA Probe NR. 1 (113E) wurden folgende chromosomalen Imbalancen detektiert: Duplikation auf Chromosom 9q34.3 (Abb. 16 und Anhang) Die Klone N0159B03, N0514D14 sind von der Duplikation betroffen. Sie weisen einen minimalen Anteil an low copy Repeats auf. Die Duplikation ist ~ 300 kb groß. Abbildung 16 . Duplikation auf Chr. 9 q34.3 Probe NR.4(328E) 43 Nach der Analyse der Hybridisierungsdaten wurden folgende DNA Zugewinne bzw. Verluste beobachtet: Mögliche Duplikation auf Chromosom 19q13.42 (Abb.17 und Anhang). Zwei Klone dj1060P11 und bK2337j16 sind davon betroffen. Der Klon dj1060P11liegt kurz über den Schwellenwert für eine Duplikation und Klon bK2337j16, welcher einen hohen Anteil an low copy Repeats aufweist, deutlich über den Schwellenwert hinaus. Das ist ein Indiz für die Duplikation in diesem chromosomalen Bereich der Test DNA. Die minimale Streuung bei der Probe 328E bekräftigt diese Vermutung. Auf dem Chromosom 19 sind die einzelnen Klone nicht überlappend wie z.B. auf Chromosom 22, X, 4 usw. sondern im Abstand von ~1mb voneinander positioniert. Dadurch ist es teilweise schwierig die genaue Ausdehnung der Aberration zu bestimmen. Abbildung 17. Duplikation auf Chromosom 19q13.42 Probe NR.11 (340E) Bei der probe 340E sind folgende chromosomalen Imbalancen detektiert: Mögliche Duplikation auf Chromosom 8q24.3 (Abb.18 und Anhang). Es sind Klone dj1118A7, bA472K18 und bA349C2 von der Aberration betroffen. die Probe zeigt eine Streuung in Intensitäten. Da es aber nicht nur ein Klon sondern drei Klone, welche zum Teil überlappend sind, über den Schwellenwert liegen, ist die 44 Wahrscheinlichkeit groß, daß es sich in diesem Fall um eine Duplikation handelt,. Die Duplikation ist in diesem Fall mind. 1.2 Mb. groß. Abbildung 18. Duplikation auf Chromosom 8q24.3 Mögliche Duplikation auf Chromosom 11p15.5 (Abb.19 und Anhang). Duplikation ist auf Klone N0596I23, N0652C03, N0412M116, N0326C03 begrenzt. Obwohl Klone der Probe streuen, liegt die Wahrscheinlichkeit hoch, daß es sich um eine Duplikation handelt. Abbildung 19. Duplikation auf 11p15.5 Probe NR.14(056E) 45 Bei der Probe 056E wurden folgende Auffälligkeit beobachtet: Auf Chromosom 4 sind drei Deletionen vorhanden (Abb.20 und Anhang). Abbildung 20. Deletionen auf Chromosom 4 Die erste Deletion (oben in der Abb.20) ist im chromosomalen Bereich q32.1q32.3 (Abb.21). Die Deletion grenzen die BAC Klone N0749I11 bei 4q32.1 und N0313O15 bei 4q32.3 ab. Die Deletion ist ~ 9 Mb groß. Abbildung 21. Deletion auf 4q32.1-q32.3 46 Die zweite Deletion (Mitte in der Abb. 20) ist n der chromosomalen Region q33-q35.1 (Abb.22). An den Grenzen dieser Deletion liegen die BAC Klone N0440K12 bei q33 und N0714G18 bei q35.1. Diese Deletion ist ~ 15,5 Mb. Abbildung 22. Deletion 4q33-q35.1 Die dritte Deletion, welche auch die kleinste ist, liegt im Bereich q35.2 (Abb.23) Dabei ist der Klon N0321G19 betroffen. Abbildung 23. Deletion 4q35. Probe NR.15 (186E) 47 Bei der Analyse der Hybridisierungsergebnisse Probe 186E wurden folgende Auffälligkeiten festgestellt: Chromosom 7. Deletion im Bereich q11.23 (Abb.24 Anhang). Es sind sechs BAC Klone : N0101D02, N0100D10, N0011L22, N0137E08, N0091L07, dj771P14, welche deutlich außerhalb der Toleranzgrenze liegen. Die Deletion ist ~1.5 Mb groß. Bei der Betrachtung der Bruchpunkten dieser Deletion kann man feststellen, daß die Deletion flankierten Klone einen sehr hohen Anteil an low copy Repeats aufweisen. Die Literatur Recherchen können diese Beobachtung bestätigen (42). Abbildung 24. Deletion auf 7q11.2 Probe NR. 16(142E) Die Analyse der Hybridisierungsergebnissen zeigen folgende Auffälligkeiten bei der Probe 142E: Chromosom 11. Duplikation in der Region p15.5 (Abb.25 und Anhang). Wie auch in der Probe 340E Abb.31 sind es 4 BAC Klone N0596I23, N0652C03, N0412M116, N0326C03 auf welche die Duplikation begrenzt ist. 48 Abbildung 25. Duplikation auf Chromosom 11p15.5 Probe NR.22 (079E) In der Probe 079E wurden folgende chromosomalen Veränderungen detektiert: Chromosom 9. Duplikation in der Region p24.1-p24.2 (Abb.26 und Anhang). Die BAC Klone N0006j24, N0567O02, N0410L03, N0043B01, N0778P24 grenzen die Duplikation deutlich ab. Die Klone N0778P24 (oberste umrandete Klon) und N0442A08 (links der grünen Linie liegende Klon, überlappend mit N0778P24) markieren anscheinend den Duplikation Breakpoint. Der Breakpoint befindet sich wahrscheinlich innerhalb des Überlappungsbereichs von dieser beiden Klone. Die Duplikation ist ~ 600 kb groß. Abbildung 26. Duplikation auf 9p24.1-p24.2 49 Probe NR.24 (368E) Bei der Probe 368E wurden folgende chromosomalen Veränderungen beobachtet: Chromosom 4. Deletion in der Region q13.3 (Abb.27 und Anhang). Klone N0719M16 und N0151j20 sind umrandet und von der Deletion betroffen. Abbildung 27. Deletion auf Chromosom 4q13. Probe NR.26 (010E) Die Analyse der Hybridisierungsergebnisse zeigt folgende chromosomalen Veränderungen: Chromosom Y. Im Region q11.223-q11.23 liegen Auffällig viele Klone über den Schwellenwert für eine Duplikation (Abb.28 und Anhang). Klone bA140H23, bA506M9, bA245K4, bA270H4 sind in der Abbildung 28 markiert. Die markierte Klone besitzen entsprechend der Abbildung 15 einen hohen Anteil an low copy Repeats 50 Abbildung 28. mögliche Duplikation auf ChromosomYq223-q22.3. Probe NR.28 (007E) Bei der Probe 007E wurden folgende chromosomalen Anomalien beobachtet: Chromosom 8. Duplikation im Region q12.1–q13.1(Abb.29 und Anhang). Die Klone bA114M5, bA414L17, dj491L6, bA227F6, bA45K10, bA115G12, bA89A16, dj252K6, bA366K18 liegen innerhalb der duplizierten Region, welche ~ 7Mb groß ist. In der Abbildung sieht man in der Mitte der Duplikation einen Klon, welcher unter dem Schwellenwert liegt. Es ist nicht feststellbar, ob dieser Klon möglicherweise mismappt ist. Außerdem sind die Klone in dem fraglichen Bereich des Chromosoms nicht überlappend. Deshalb bleibt die Frage offen, ob es sich hier möglicherweise auch um eine diskontinuierliche Duplikationen handeln kann. In einem solchen Fall würde die erste Duplikation in der Region q12.1-q12.3 auftreten und durch vier entsprechenden Klonen bA114M5, bA414L17, dj491L6, bA227F6 markiert. Die zweite Duplikation q12.3-q13.1 würde die fünf andere Klone bA45K10, bA115G12, bA89A16, dj252K6, bA366K18 beinhalten. Abbildung 29. Duplikation auf Chromosom 8q12.1–q13.1 51 Chromosom 9. Duplikation im Centromer Bereich (Abb.30). In dem oberen Teil der Abbildung (umrandete Klone ) ist eine große Duplikation im Centromer Region p11.2-p13.1 zu sehen. Alle Klone in diesem Bereich weisen starke Involvierung in segmentale Duplikationen auf. Abbildung 30. Duplikation auf Chromosom 9p11.2 -p13.1 in der nähe des Centromer Bereichs. Probe NR.30 (137E) Bei der Probe 137E wurden folgende chromosomalen Veränderungen beobachtet: Chromosom 7. Deletion im Region q11.23 (Abb.31 und Anhang). In der Abbildung 31 sind Klone involvierte in die Duplikation N0100D10, N0011I22, N0041F22 schwarz umrandet. Die Deletion ist ~ 1,3 Mb groß. 52 Abbildung 31. Deletion auf 7q11.23 Die Test DNA wurde für die Hybridisierung mit Cy5, Kontroll DNA mit Cy3 markiert, sonst werden die Proben umgekehrt markiert. Deshalb sind in diesem Fall die Klone als eine Deletion anstatt als eine Duplikation interpretiert (Anhang). Probe NR.31 (077E) Bei der Probe 077E wurden folgende chromosomale Veränderungen detektiert: Duplikation des Chromosoms 21. Trisomie 21 (Abb.32 und Anhang). . Abbildung 32. Duplikation des Chromosoms 21 Probe NR.32 (127E) Bei der Probe 127 wurden folgende Auffälligkeiten beobachtet: 53 Duplikation des Chromosoms 21(Trisomie 21, Abb.33 und Anhang) Abbildung 33. Duplikation des Chromosoms 21 Das Geschlecht der Test DNA wurde falsch bestimmt. Dies kann man an Chromosomen X und Y sehen. Alle Klone des Chromosoms X liegen im Deletionsbereich, Klone des Chromosoms Y dagegen im Duplikationsbereich (siehe Anhang). Probe NR.33 (174E) Die Ergebnisse der Hybridisierung zeigen folgende Auffälligkeiten: Duplikation des Chromosoms 21 (Trisomie 21, Abb.34 und Anhang) Abbildung 34. Duplikation des Chromosoms 21 54 Probe NR. 34(210E) Bei der Probe 210E wurden folgende Auffälligkeiten beobachtet: Duplikation des Chromosoms 21 (Trisomie 21, Abb. 35), (Anhang). Abbildung 35. Duplikation des Chromosoms 21 Bei der Probe 210E wurden die Test DNA mit Cy5 anstatt mit Cy3 markiert. Deshalb befinden sich die Klone links der roten Linie und nicht rechts der grünen, wie bei anderen Proben mit einer Duplikation. Zusätzlich dazu wurde das Geschlecht der Test DNA verwechselt und anstelle männlicher Referenz-DNA weibliche benutzt. Dies erklärt warum die Klone der X und Y Chromosomen dupliziert erscheinen (siehe Anhang). Probe NR. 35 (284E) Die Probe 284E zeigt folgende chromosomalen Auffälligkeiten: Duplikation auf Chromosom 21(Trisomie 21 Abb.36 und Anhang). Abbildung 36. Duplikation des Chromosom 21 55 Probe NR.36 (351E) Bei der Probe 351E wurden folgende chromosomalen Anomalien beobachtet: Duplikation des Chromosoms 21 (Trisomie 21 Abb.37 und Anhang). Abbildung 37. Duplikation des Chromosoms 21 Probe NR.38 (400E) Bei der Probe 400E sind folgende chromosomalen Veränderungen auffällig: Chromosom 4. Deletion im Region q28.3 (Abb.38 und Anhang). Es sind die Klone N0619K10, N0671A13, N0625A06, N0781A13, bA400D2, M2145F16, N0352L20 welche in die Deletion involviert sind. Diese Deletion ist ~811 kb groß. Abbildung 38. Deletion auf Chromosom 4q28.3 56 Kontrolle S Die Probe ist eine Kontrolle. Bei dieser Hybridisierung wurde Test DNA wie auch Kontroll DNA von gesunder weiblicher Personen benutzt. Die Test DNA und Kontroll DNA wurden von unterschiedlichen Personen genommen. Die Test DNA wurde bei dieser Probe mit Cy5 anstatt mit Cy3 markiert. Die Klone liegen deshalb im Falle einer Deletionen rechts von der grünen Linie und im Falle einer Duplikation links der roten Linie. Verteilung der Klone entlang aller Chromosomen zeigt eine breite Streuung (Anhang). In der Tabelle 5 sind die Ergebnisse aller Hybridisierungen noch mal zusammengefaßt. Die häufigste chromosomale Anomalie ist Trisomie 21. Sie wurde bei 6 Proben beobachtet. Bei zwei Proben 142E und 340E wurden eine Duplikation auf Chromosom 11p15.5 beobachtet. Bei anderen zwei Proben 186E und 137E wurde eine Deletion auf 7q11.23. Die Proben 368E, 056E und 400E zeigten Deletionen in verschiedenen Regionen (siehe Tabelle 5) des Chromosom 4. Bei der Probe 056E ist die Deletion besonders ausgedehnt. Chromosom Y ist bei der Probe 010E von einer großen Duplikation (Yq223-q22.3) betroffen. Die Probe 340E zeigt Duplikationen auf Chromosom 8q24.3 und Chromosom 11p15.5. Probe 007E zeigt sogar zwei Duplikationen. Die erste ist auf Chromosom Chr.8p12.1–p13.1 und die zweite auf Chromosom Chr.9p11.2 -p13.1. Das Chromosom 9q34.3 ist auch bei der Probe 113E von einer Duplikation betroffen. Bei Proben NR: 2(071E), NR. 3 (208E), NR. 5(111E), NR. 6 (067E), NR.7(133E), 8(281E), 9(350E), NR.12 (082E), NR.13 (349E), NR.17(386E), NR.18(206E) 57 NR.19(373E), NR.20(091E), NR.21(162E), NR.23 (139E), 25 (011E), 29 (261E), NR.37 (399E), LQT414 wurden keine chromosomalen Veränderungen detektiert. Bei den orange gefärbten Proben (Tabelle 5, Proben 082E, 349E, 127E, 210E) wurde bei der Hybridisierung falsches Geschlecht der Referenz DNA verwendet. Das kann man auf Array Bildern anhand der Klone auf X und Y Chromosomen erkennen. Die Klone des X Chromosoms erscheinen dupliziert und Klone des Y Chromosoms deletiert. Bei den grün markierten Proben (Tabelle 5) sollte , wegen der Streuung der Klone, die Hybridisierung noch ein mal wiederholt werden. 58 Tabelle 5. Ergebnisse der Hybridisierungen mit Patienten DNA Number SAMPLE Hybridisation Deletion Duplication µg/µl Gender Chr.9q34.3 0,131 F 0,093 M 0,192 F 1 113E MC_733/734 2 071E MC_737/738 3 208E MC_739/740 4 328E MC_749/750 0,364 F 5 111E MC_751/752 0,135 F 6 067E MC_713/714 0,226 F 7 133E MC_753/754 0,473 M 8 281E MC_755/756 0,077 F 9 350E MC_777/778 0,252 M 0,351 M 0,185 F 11 340E MC_779/780 12 082E MC_781/782 13 349E MC_783/784 Chr.19q13.42 Chr.8q24.3 Chr.11p15.5 0,414 M 0,250 F 0,363 F 0,121 M 14 056E MC_785/786 Chr.4q32.1-q32.3, q33-q35.1 Chr.4q35.2 15 186E MC_791b/792b Chr.7q11.23 16 142E MC_787a/788a 17 386E MC_789/790 0,189 M 18 206E MC_807/808 0,167 M 19 373E MC_801/802 0,508 M 20 091E MC_653/654 0,210 F 21 162E MC_803/804 0,398 F 22 079E MC_805/806 0,115 M 23 139E MC_819/820 24 368E MC_821/822 25 011E MC_651a/652a 26 010E MC_825/826 28 007E MC_823/824 29 261E MC_827/828 30 137E MC_655/656 31 077E MC_829/830 Trisomie 21 32 127E MC_715/716 33 174E 34 Chr.11p15.5 Chr.9p24.1- p24.2 0,445 F 0,392 M 0,164 F Chr.Yq223-q22.3 0,108 M Chr.8p12.1–p13.1 Chr.9p11.2 -p13.1 0,104 M 0,079 M 0,551 M 0,183 F Trisomie 21 0,157 F MC_717/718 Trisomie 21 0,418 F 210E MC_657a/658a Trisomie 21 0,485 F 35 284E MC_831/832 Trisomie 21 0,477 F 36 351E MC_833/834 Trisomie 21 0,462 M 37 399E MC_835/836 0,321 M 38 400E MC_837/838 0,642 F Chr.4q13.3 Chr.7q11.23 Chr.4q28.3 LQT414 MC_659/660 Kontr_S MC_661/662 59 Zusätzlich zu den chromosomalen Veränderungen in Patienten DNA (Tab.5) wurden einige Polymorphismen in Referenz DNA (Kontroll DNA) beobachtet. Chromosom 4. Polymorphism in der Region p16.1 (Abb.39). Die Klone N0193D10, F0980j16, M2205P10 sind involviert, wobei es ist fraglich ist ob 2 unten umrandete Klone dazu gehöhren, weil unter dem umrandeten Klonen noch 2 Klone zu sehen sind. Diese ``Ausreißer`` sind möglicherweise mismapped. Alle Klone (Abb.15) haben einen sehr hohen Anteil an low copy Repeats. Abbildung 39. Polymorphism in der Region 4p16.1 Im Centromer Bereich des Chromosom 9 (Abb.40) sind zwei Regione p11.2 (Klon N0229K20) mit benachbarten überlappenden Klonen und q13 (Klon N0429L11) mit benachbarten überlappenden Klonen vom Polymorphism betroffen. Abbildung 40. Polymorphe Regionen im Centromer Bereich 9p11.2 und 9q13 60 Auf dem Chromosom 17q12 ist eine weitere polymorphe Region lokalisiert (Abb. 41). Dabei sind die BAC Klone N0355O04, N0600K04, N1330O06 (obere drei umrandeten Klone) und die BAC Klone N0722D15, N0072I20, N0121G16, N0678G07 (untere umrandeten Klone) involviert. Abbildung 41. Polymorpism auf Chromosom 17q12 Die häufigsten Polymorphismen befinden sich auf Chromosom 22. Es sind drei betroffen Regionen: 22q11.1, 22q11.21 und 22q11.22(Abb.42) zu nennen. In der nähe des Centromers liegt die Region 22q11.1 (obere umrandeten Klone in der Abbildung 42 ) Alle Klone, entsprechend der Abb.15 haben einen hohen Anteil an low Copy repeats. Die zweite Region, in welcher Polymorphismen auftreten können, ist 22q11.21. Dabei sind meistens nur 2 Klone N0444L07, N0818K20 betroffen (zwei umrandete Klone in der Abb.42 ). In der Region 22q11.22 sind es dagegen 5 Klone N0432M13, N0757F24, N0281O23, N005L23 und bA50L23 ( in der Abb.42 untere nicht umrandeten Klone) 61 Abbildung 42. Chromosom 22 Polymorphe Regionen q11.1, q11.21 und q11.22 Polymorphe Region auf Chromosom Xq28 (Abb.43). BAC Klone N0330B02, N0333O06 und M2149G05 liegen außerhalb der Toleranzgrenze. Die Klone , wie aus der Abb.15 sichtbar ist, haben nur einen geringen bis keinen Anteil an low copy Repeats. Abbildung 43. Polymorphe Region auf Chromosom X.q28. 62 In der Tabelle 6 sind die Polymorpismen noch mal zusammengefaßt Tabelle 6. Mögliche Polymorphismen in Referenz DNA number SAMPLE Hybridisation Deletion 1 2 113E 071E MC_733/734 MC_737/738 3 208E MC_739/740 4 328E MC_749/750 5 111E MC_751/752 6 067E MC_713/714 Chr.17q12 7 8 9 133E 281E 350E MC_753/754 MC_755/756 MC_777/778 Chr.22q11.21 11 12 340E 082E MC_779/780 MC_781/782 13 349E MC_783/784 14 056E MC_785/786 15 Chr.17q21.31 186E MC_791b/792b Chr.Xq28 16 17 18 142E 386E 206E MC_787a/788a MC_789/790 MC_807/808 19 20 21 373E 091E 162E MC_801/802 MC_653/654 MC_803/804 22 079E MC_805/806 23 139E MC_819/820 24 25 368E 011E MC_821/822 MC_651a/652a 26 010E MC_825/826 28 29 007E 261E MC_823/824 MC_827/828 Chr.17q12 Chr.17p11.2, Chr.4p16.1 Duplication Gender Chr.4p16.1 Chr.17q12 Chr.22q11.1, 22q11.21; 22q11.22 Chr. 22q11.1 Chr.9q13; Chr.17q12, Chr.22q11.22 Chr.9p11.2, 9q13 Chr.22q11.1;22q11.22 Chr.9p11.2, 9q13; Chr.22q11.22 Chr.9p11.2, 9q13; Chr.17q12 Chr.22q11.22 Chr.9p11.2 ; Chr. 9q13 Chr.17q12 ; Chr. 22q11.1 Chr.Xq28 Chr.22 q11.1, q11.21, q11.22 F M F F F F M F M Chr.22q11.1; Chr.Xq28 M F Chr.9p11.2 ; Chr. 9q13 Chr. 22 q11.1, q11.22 Chr.4p11; Chr.22q11.22 Chr.9p11.2, 9q13 Chr. 22 q11.1, q11.22 M F F M M M Chr.17q12 Chr.17q12 ; Chr.22q11.1, q11.22 M F F Chr.22 q11.1, q11.21, q11.22 Chr.9p11.2 Chr.17q21.31 Chr.22q11.21 Chr.9p11.2, 9q13 Chr. 22 q11.1, q11.22 Chr.9p11.2, 9q13 Chr. 22 q11.1 Chr.22 q11.1, q11.22 Chr.9p11.2-p12 M F M F M Chr.4p16.1 Chr.17q12 Chr.Xq28 Chr. 22q11.1, q11.21 M M 63 30 137E MC_655/656 31 32 077E 127E MC_829/830 MC_715/716 33 34 35 36 37 38 174E 210E 284E 351E 399E 400E LQT414 MC_717/718 MC_657a/658a MC_831/832 MC_833/834 MC_835/836 MC_837/838 MC_659/660 Kontr_S MC_661/662 M Chr. 22q11.1 Chr.9p11.2, 9q13 Chr. 22 q11.1, q11.21, q11.22 F F Chr.9p11.2, 9q13 Chr. 22 q11.1, q11.22 F F F M M F Chr.22 q11.21, q11.22 Chr.22 q11.1, q11.22 Chr.9p11.2, 9q13 Chr.22 q11.1, q11.22 64 4. Diskussion Diese Arbeit ist die erste Array CGH Studie, abgesehen von einem Einzelfallbericht (49), über die chromosomalen Gewinne und Verluste bei Patienten mit congenitalen Herzfehlern. Es besteht die Möglichkeit genetische Veränderungen und deren Auswirkungen auch auf RNA und/oder Proteinebene studieren, allerdings können diese Untersuchungen nur einen Einblick in die gegenwärtige Situation zum Zeitpunkt der Untersuchung geben. Rückschlüsse auf die Situation in den kritischen Phasen der Herzentwicklungen sind daher oft unmöglich. Aus diesem Grund bleibt, obwohl eine Studie fortbestehende Veränderungen der Genexpressionsmuster in Patienten mit congenitalen Herzfehlern nachweisen konnte (21), Ursache und Wirkung, d.h. die Unterscheidung zwischen dem auslösenden Defekt und den daraus resultierenden Genexpressionsänderungen, immer noch unklar. Dasselbe gilt für die Analyse von Proteinexpressionsmustern. Ein weiteres Problem ist die Zugänglichkeit des Probenmaterials, will man nicht auf formalin-fixiertes Autopsiematerial mit meist degradierter RNA und kreuzvernetzten Proteinen aus den Archiven der Pathologen zurückgreifen. All diese Nachteile kann man bei der Untersuchung der DNA Ebene umgehen. Wenn die chromosomalen Veränderungen schon am Beginn der Embryogenese vorhanden sind, so kann man diese in allen Zellen des daraus entstandenen Organismus nachweisen. Dabei ist es egal, ob die DNA in der kritischen Phase der Herzentwicklung oder lange danach untersucht wird. Der Defekt wird bis zum Lebensende des Patienten zu jeder Zeit in dessen DNA nachweisbar sein, egal aus welchem Gewebe die DNA isoliert wird. Diese Studie mit 38 ausgesuchten Patienten galt als Vorversuch für ein groß angelegtes Herzprojekt. Das Ziel war , sowohl die Zuverlässigkeit als auch das Potential der Methode für die Untersuchungen der congenitalen Herzmißbildungen zu testen. Die Patienten Gruppe in dieser Studie umfaßte Fälle mit bereits diagnostizierten Syndromen, aber auch Fälle mit unbekannter Ätiologie. Wie später noch detaillierter besprochen, dienten die Syndrompatienten nicht nur der Überprüfung der Methode (z.B. Trisomie 21/Downsyndrome), sondern auch der exakten Definition der chromosomalen Bruchpunkte und der genotypische 65 Zuordnung, wie am Beispiel des William-Beuren-Syndromes noch genauer diskutiert wird. Die Array CGH erkannte alle Veränderungen, die bereits aufgrund der humangenetischen Analyse bekannt waren oder erwartet wurden, wie z.B. die einfach zu diagnostizierenden Trisomien 21 oder ein Fall mit William Beurens Syndrom. Jedoch bereits beim zweiten Fall mit William Beurens Syndrom, die genetische Diagnose sprach hier lediglich von einem Verdacht auf William Beurens Syndrom, konnte die Array CGH den Befund durch Detektion der typischen Deletion auf 7q bestätigen. Numerische Aberrationen von Chromosomen entstehen meistens als Resultat einer "non-disjunction" in der Meiose. Aus bestimmten Gründen trennen sich in einer der beiden meiotischen Teilungen die homologen Chromosomen gelegentlich nicht, so daß beide Chromosomenhomologe in die Keimzelle gelangen. Die "nondisjunction" kann während der Entstehung der mütterlichen oder väterlichen Geschlechtszellen auftreten. Bei der Trisomie 21 beruhen z.B. 80% der Fälle auf "non-disjunction" in der mütterlichen und 20% in der väterlichen Meiose (23, 24). Trisomie 21 war als erste numerische Chromosomenaberration bei Patienten mit Down-Syndrom 1959 von Lejeune entdeckt worden. Die Personen mit Trisomie 21 haben gewöhnlich ein charakteristisches Erscheinungsbild und weisen multiple Mißbildungen, unter anderem auch angeborene Herzfehler, auf (2, 26). Die häufigste numerische Veränderung in unserer Patienten Liste war die Trisomie 21. Das ist auch nicht weiter verwunderlich, da, wie oben beschrieben, Herzfehler ein häufiger Bestandteil des klinischen Erscheinungsbildes des Down Syndroms sind und außerdem die Trisomie 21 die einzige autosomale Trisomie ist, die längere Zeit überlebt werden kann. Neuere molekular- zytogenetische Befunde haben allerdings gezeigt, daß für die volle Ausprägung des Down Syndroms nur Teile von Chromosom 21 unbalanziert sein müssen. Nichtsdestotrotz wurde in allen Proben unserer Gruppe mit Down Syndrom, den Patienten 077E, 127E, 174E, 210E, 284E und 351E, mittels Array CGH eine vollständige Trisomie 21 nachgewiesen. Strukturelle Chromosomenaberrationen entstehen durch Umbauten innerhalb eines Chromosoms (intrachromosomal) oder zwischen verschiedenen Chromosomen (interchromosomal). Der Entstehung struktureller Aberrationen gehen Chromosomenbrüche voraus, die durch verschiedene Umweltfaktoren hervorgerufen 66 werden können, wie z.B. Radioaktivität, Drogen, Chemikalien oder Viren (25). Verluste oder Zugewinne von Chromosomensegmenten führen zu unbalancierten Genverhältnissen (Gene dosage). Strukturumbauten ohne Verlust oder Zugewinn chromosomalen Materials werden als balanciert bezeichnet. Tabelle 5 fast die jeweiligen Gewinne und Verluste zusammen. Um die problematischen Auswirkungen von Artefakten zu minimieren, wurden nur solche Veränderungen mit in die Tabelle aufgenommen, die mindestens zwei Klone umfaßten. Durch diese Einschränkung reduzierte sich zwar die Auflösung der Methode auf ca. 150-200kb, je nach chromosomaler Region, gleichzeitig sank aber auch die Wahrscheinlichkeit, falsch positive Resultate zu generieren. Unklar bleibt derzeit, inwieweit jede einzelne Veränderung mit dem Phänotyp in kausalem Zusammenhang steht. Erst aufgrund der wachsenden Array CGH Daten hat man erkannt, daß zwischen einzelnen Individuen beträchtliche Unterschiede im DNA Gehalt vorkommen können. Diese Unterschiede können mehrere hundert kb ausmachen (48) und liegen somit im Detektionsbereich der hochauflösenden Array CGH. DNA Kopienzahl-Polymorphismen liegen oft in Regionen mit Häufung von segmentalen Duplikation(28, 42). Es handelt sich dabei um „hot spots“ der Genomentwicklung. Noch ist aber unklar, ob die Polymorphismen Ursache oder Resultat der großen Dynamik dieser chromosomalen Abschnitte sind. Erst der Abgleich der unserer Daten mit speziellen, derzeit aber erst in der Entstehung befindlichen Datenbanken und vor allem die Analyse der Eltern kann Klarheit bringen, ob es sich bei einem Unterschied in der DNA Kopienzahl um einen phänotypisch neutralen DNA Kopiezahl-Polymorphismus oder möglicherweise doch um eine de Novo Aberration handelt. Die Möglichkeiten der Array CGH zeigt auch der Fall 113E. Bei dieser Probe wurde eine kleine Duplikation auf 9q43.3 festgestellt, welche bei der konventionellen CGH wegen der Auflösungsgrenze bei weitem nicht sichtbar gewesen wäre. In dem Duplikationsbereich liegt das Gen für Collagen Type V COL5A1 (COLLAGEN TYPE V ALPHA-1, OMIM: 120215). Ein Defekt in diesem Gen kann mit EhlersDanlos Syndrom (46) und Mitralklappenprolaps (Defekt an der zweizipfeligen Segelklappe zwischen linkem Vorhof und linker Herzkammer) assoziiert sein. 67 Bei den Proben 340E und 142E wurde eine Duplikation auf Chromosom 11p15.5 beobachtet, dabei sind in beiden Fällen die gleichen Klone betroffen. Hier ist eine neuerliche klinische Untersuchung angezeigt, die verdeutlichen soll, inwieweit der überlappende Genotyp seine phänotypische Entsprechung findet. Bei 5 von 36 Patienten Proben wurden DNA-Verluste beobachtet (Tab.5). Bei den Proben 056E, 186E, 137E und 400E sind die Deletionen besonders deutlich sichtbar. Die Probe 056E zeigt eine diskontinuierliche, aus drei Abschnitten bestehende Deletionen auf 4q. Zwei der Abschnitte (4q32.1-q32.3, 4q33-q35.1) sind groß, der dritte ist sehr klein, liegt aber in der besonders genreichen subtelomeren Region 4q35.2 Innerhalb des deletierten Bereiches liegen Gene wie Tolloid-Like 1; TLL1(OMIM: 606742), welche für die normale Herzentwicklung essentiell sind. Bei der Analyse der Proben 137E und 186E wurde eine Deletion auf 7q11.23 detektiert. In beiden Fällen liegen die Klone N0100D10, N0011I22, N0041F22 in der deletierten Region. Datenbanken Ein Abgleich der Klonpositionen mit den entsprechenden (http://bacpac.chori.org/genomicRearrays.php, http://genome.ucsc.edu/cgi-bin/hgTracks?db=hg15&position=chr7:7198705673137391) zeigt, daß in dieser Region mehrere, mit dem Williams Beuren Syndrom assoziierte Gene lokalisiert sind.(14, 30). Die Patienten mit Williams-Beuren-Syndrom zeigen körperliche und geistige Entwicklungsverzögerungen, die in einem charakteristisches Erscheinungsbild mit angeborenen Fehlbildungen resultieren, unter anderem einem angeborenen Herzfehler (Tab.2) (OMIM #194050 WILLIAMS-BEUREN SYNDROME; WBS). Die WBS Deletionsregion wird von drei low-copy-Repeats-reichen Blöcke (A, B, C) flankiert . Die überwiegende Anzahl der Patienten (95%) mit WBS weisen eine 7q11.23 Deletion in der Größe von 1,5 Mb auf. (47). Nur eine Minderheit der Patienten (5%) zeigt eine Deletion von 1,8 Mb in dem entsprechenden Bereich. Die Lage der Bruchpunkte bestimmt die Größe der Deletion. Wenn die Bruchpunkte in dem lowcopy-Repeats-reichen Block B liegen, umfaßt die Deletion ~1,55 Mb. Wenn aber die Bruchpunkte im Block A liegen, so ist die Deletion ~1,8 Mb groß. (47). Bei der Deletion im Fall 137E war die Bruchpunktbestimmung erschwert, da die Klone an diesen Punkten nicht überlappend waren und etwas weiter auseinander lagen. In beiden Fällen beträgt die Größe der Deletion ca. 1,5 Mb. Damit gehören beide Fälle mit Williams Beuren zu jenen 95%, in denen der Bruchpunkt im low-copy repeat reichen B-block liegt. 68 Diese Studie hat deutlich gezeigt , daß eine große Zahl von Herzfehlbildungen mit chromosomalen Veränderungen assoziiert ist und von diesen mit großer Wahrscheinlichkeit verursacht werden. Die Ergebnisse dieser Studie ermutigen zu Projekten mit einer größeren Patientengruppe. Dabei sollte das Ziel sein, trotz der zu erwartenden Bandbreite der chromosomalen Veränderungen, genotypische Subklassifizierungen durchzuführen, diagnostische Marker zu entwickeln (z.B. für klinische Test auf FISH Basis) und schließlich, die für die Herzentwicklung bedeutsamsten Gene zu identifizieren. 5. Literaturverzeichnis 1. The developing heart and congenital heart defects: a make or break situation BG Bruneau. Clinical Genetics April 2003 2. Herzfehler bei Kindern. Krankheitsbilder – Ursachen - Behandlung .Lewin, Martin A.G. (1994). Spektrum Akademischer Verlag: Heidelberg. ISBN 3-86025-205-4 3. Developmental Mechanisms of Heart Disease. Edward B. Clark, et al Futura Publishing Company (March 1, 1995) 4. Congenital heart disease: Chromosomal anomalies Nora JJ, Nora AH, Nora JJ, Nora AH (eds): Genetics and Counseling in Cardiovascular Diseases. Springfield 5. Genetic and environmental risk factors of major cardiovascular malformations: the Baltimore-Washington Infant Study. Ferencz C, Loffredo C, Correa-Villasenor Wilson P. 1997. 1981±1989 6. Comparative genomic hybridisation for molecular cytogenetic analysis of solid tumors Kallioniemi, A., Kallioniemi, O.P., Sudar, D., Rutovitz, D., Gray, J.W., Waldman, F. and Pinkel, D. (1992). Science, 258, 818–821 7. Minimal sizes of deletions detected by comparative genomic hybridization. Genes Chromosomes. Bentz M, Plesch A, Stilgenbauer S, Dohner H, Lichter P. Cancer 1998; 21:172–5. 8. Matrix-based comparative genomic hybridization: biochips to screen for genomic imbalance. Solinas-Toldo, S., Lampel, S., Stilgenbauer, S., Nickolenko, J., Benner, A., Döhner, H., Cremer, T. and Lichter, P. (1997). Genes Chromosomes Cancer, 20, 399–407. 69 9. DNA microarrays for comparative genomic hybridization based on DOP-PCR Amplification of BAC and PAC clones. Fiegler H, Carr P, Douglas EJ, et al. Genes Chromosomes Cancer 2003; 36:361–74. 10. 1-Mb resolution array-based comparative genomic hybridisation using a BAC clone set optimized for cancer research. Greshock, J., Naylor, T.L., Margolin, A., Diskin, S., Cleaver, S.H., Futreal, P.A., deJong, P.J., Zhao, S., Liebman, M. and Weber, B.L. (2004) Gen. Res., 14, 179–187 11. A Bacterial Artificial Chromosome Library for Sequencing The Complete Human Genome. Kazutoyo Osoegawa, Aaron G. Mammoser, Chenyan Wu, Eirik Frengen, Changjiang Zeng, Joseph J. Catanese and Pieter J. de Jong Department of Cancer Genetics, Roswell Park Cancer Institute, Buffalo, New York 14263, USA 12. High-Resolution Molecular Characterization of 15q11-q13. Rearrangements by Array Comparative Genomic Hybridization (Array CGH) with Detection of Gene Dosage. Nicholas J. Wang, Dahai Liu, Alexander Am. J. Hum. Genet. 75:267-281, 2004 13. Genomic microarrays in human genetic disease and cancer. Albertson DG, Pinkel D. 2003. Hum Mol Genet 12 14. High resolution analysis of DNA copy number variation using comparative genomic hybridization to microarrays. Pinkel D, Segraves R, Sudar D, Clark S, Poole I, Kowbel D, Collins C, Kuo WL, Chen C and Zhai Y et al (1998). Nat Genet 20,207–211. 15. High resolution deletion analysis of constitutional DNA from neurofibromatosis type 2 (NF2) patients using microarray-CGH. Bruder CE, Hirvela C, Tapia-Paez I, Fransson I, Segraves R, Hamilton G, Zhang XX, Evans DG, Wallace AJ and Baser ME et al (2001) Hum Mol Genet 10,271–282. 16. Development of a comparative genomic hybridization microarray and demonstration of its utility with 25 well-characterized 1p36 deletions.Yu W, Ballif BC, Kashork CD, Heilstedt HA, Howard LA, Cai WW, White LD, Liu W, Beaudet AL, Bejjani BA et al (2003) Hum Mol Genet 12,2145–2152 17. A tiling resolution DNA microarray with complete coverage of the Human Genom. Adrian S Ishkanian, Chad A Malloff, Spencer K Watson, Ronald J deLeeuw, Bryan Chi, Bradley P Coe, Antoine Snijders, Donna G Albertson, Daniel Pinkel, Marco A Marra, Victor Ling, Calum MacAulay & Wan L Lam Nat Genet. 2004 Mar;36(3):299-303. Epub 2004 Feb 15 18. Comprehensive whole genome array CGH profiling of mantle cell lymphoma model genomes. de Leeuw RJ, Davies JJ, Rosenwald A, Bebb G, Gascoyne RD, 70 Dyer MJ, Staudt LM, Martinez-Climent JA, Lam WL Hum Mol Genet. 2004 Sep 1; 13(17): 1827-37 19. Comparative genomic hybridization, loss of heterozygosity and DNA sequence analysis of single cells. Klein CA, Schmidt-Kittler O, Schardt JA, Pantel K, Speicher MR, Riethmuller G. Proc Natl Acad Sci U S A. 1999 Apr 13;96(8):4494-9. 20. Feinberg, A.P. and Vogelstein, B. Anal. Biochem., 132:6-13, 1983 21. Genome-wide array analysis of normal and malformed human hearts. Bogac Kaynak, Anja von Heydebreck, Siegrun Mebus, Dominik Seelow, Steffen Hennig, Jan Vogel, Hans-Peter Sperling, Reinhard Pregla, Vladimir AlexiMeskishvili, Roland Hetzer, Peter E. Lange, Martin Vingron, Hans Lehrach, Silke Sperling Circulation 2003 May 20; 107:2467-2474. 22. A Chromosomal Duplication Map of Malformations: Regions of Suspected Haplo- and Triplolethality—and Tolerance of Segmental Aneuploidy in Humans Carole Brewer, Susan Holloway, Paul Zawalnyski, Albert Schinzel, and David FitzPatrick Am. J. Hum. Genet. 64:1702–1708, 1999 23. Humangenetik. Murken/Cleve 6. Auflage, Enke Verlag 24. Molekulare Genetik. Knippers, 6. Auflage, Thieme Verlag 25. Leitfaden der Humangenetik / J. M. Connor ; M. A. Ferguson-Smith. Übers. Andreas Schwarzkopf. - Darmstadt : Steinkopff 26. Down-Syndrom: Krankheitsbild – Ursache – Behandlung. Selikowitz, Mark. Heidelberg: Spektrum Akad. Verlag, 1992 27. WBSCR14, a putative transcription factor gene deleted in Williams-Beuren syndrome: complete characterisation of the human gene and the mouse ortholog. Europ. de Luis O, Valero MC, Jurado LA. J. Hum. Genet. 8: 215-222, 2000 28. Chromosome 22-specific low copy repeats and the 22q11.2 deletion syndrome: genomic organization and deletion endpoint analysis Tamim H. Shaikh, Hiroki Kurahashi, Sulagna C. Saitta, Anna Mizrahy O’Hare, Ping Hu, Bruce A. Roe, Deborah. Human Molecular Genetics, 2000, Vol. 9, No. 4 489-501 71 29. Microduplication 22q11.2, an Emerging Syndrome: Clinical, Cytogenetic, and Molecular Analysis of Thirteen Patients Regina E. Ensenauer, Adewale Adeyinka, Heather C. Flynn, Virginia V. Michels, Noralane M. Lindor, D. Brian Dawson, Erik C. Thorland, Cindy Pham Lorentz, Jennifer L. Goldstein, Marie T. McDonald Am. J. Hum. Genet., 73:1027-1040, 2003 30. Complete physical map of the common deletion region in Williams’s syndrome and identification and characterization of three novel genes. Meng, X.; Lu, X.; Li, Z.; Green, E. D.; Massa, H.; Trask, B. J.; Morris, C. A.; Keating, M. T.: Hum. Genet. 103: 590-599, 1998. 31. Applications of genomic microarrays to explore human chromosome structure and function. Nigel P. Carter and David Vetrie. Human Molecular Genetics 2004 32. High resolution profiling of X chromosomal aberrations by array comparative genomic hybridisation. J A Veltman, H G Yntema, D Lugtenberg, H Arts, S Briault, Huys, K Osoegawa, P de Jong, H G Brunner, A Geurts van Kessel, H van Bokhoven and Schoenmakers. Journal of Medical Genetics 2004;41 33. High resolution comparative genomic hybridisation in clinical cytogenetics Maria Kirchhoff, Hanne Rose, Claes Lundsteen J Med Genet 2001;38:740-744 34. Genome-wide analysis of DNA copy-number changes using cDNA microarrays. Pollack, J.R., Perou, C.M., Alizadeh, A.A., Eisen, M.B., Pergamenschikov, A., Williams, C.F., Jeffrey, S.S., Botstein, D., and Brown, P.O. 1999. Nat. Genet. 23: 41-46 35. High-resolution analysis of DNA copy number using oligonucleotide microarrays. Bignell GR, Huang J, Greshock J, Watt S, Butler A, West S, Grigorova M, Jones KW, Wei W, Stratton MR, Futreal PA, Weber B, Shapero MH, Wooster R: Genome Res. 2004 Feb;14(2):287-95 36. High resolution microarray comparative genomic hybridisation analysis using spotted oligonucleotides. Carvalho B, Ouwerkerk E, Meijer GA, Ylstra B. J Clin Pathol. 2004 Jun; 57 37. High-resolution comparative hybridization to combed DNA fibers. Kraus J, Weber RG, Cremer M, Seebacher T, Fischer C, Schurra C, Jauch A, Lichter P, Bensimon A, Cremer T. Hum Genet. 1997 Mar;99(3):374-80 38. High density synthetic oligonucleotide arrays. Lipshutz RJ, Fodor SP, Gingeras TR, Lockhart DJ. Nat Genet. 1999 Jan;21(1 Suppl):20-4 39. A set of BAC clones spanning the human genome. Krzywinski M, Bosdet I, Smailus D, Chiu R, Mathewson C, Wye N, Barber S, Brown-John M, Chan S, Chand S, Cloutier A, Girn N, Lee D. Nucleic Acids Res. 2004 Jul 09;32(12):3651-60 72 40. Array-based comparative genomic hybridization for the genomewide detection of submicroscopic chromosomal abnormalities. Vissers LE, de Vries BB, Osoegawa K, Janssen IM, Feuth T, Choy CO, Straatman H, van der Vliet W, Huys EH, van Rijk A, Smeets D, van Ravenswaaij-Arts CM, Knoers NV, van der Burgt I, de Jong PJ, Brunner HG, van Kessel AG, Schoenmakers EF, Veltman JA. Am J Hum Genet. 2003 Dec;73(6):1261-70 41. Recent Segmental Duplications in the Human Genome. Bailey JA, Gu Z, Clark RA, Reinert K, Samonte RV, Schwartz S, Adams MD, Myers EW, Li PW, Eichler EE Science, Vol 297, Issue 5583, 1003-1007 42. Shuffling of Genes Within Low-Copy Repeats on 22q11 (LCR22) by AluMediated Recombination Events During Evolution. Melanie Babcock, Adam Pavlicek, Elizabeth Spiteri, Catherine D. Kashork, Ilya Ioshikhes, Lisa G. Shaffer, Jerzy Jurka and Bernice E. Morrow. Genome Research 13:2519-2532, 2003 43. Genome architecture, rearrangements and genomic disorders. Stankiewicz, P. and Lupski, J.R. 2002. Trends Genet. 18: 74-82 44. PCR amplification of DNA microdissected from a single polytene chromosome band: a comparison with conventional microcloning. Saunders, R.D., Glover, D.M., Ashburner, M., Siden-Kiamos, I., Louis, C., Monastirioti, M., Savakis, C., Kafatos, F. 1989. Nucl. Acids Res. 17, 9027–9037. 45. Universal linkage system: An improved method for labeling archival DNA for comparative genomic hybridization Janneke C. Alers, Jenneke Rochat, Pieter-Jaap Krijtenburg, Herman van Dekken, Anton K. Raap, Carla Rosenberg. Genes, Chromosoms, Cancer 46. Das Ehlers-Danlos-Syndrom. Eine Übersicht. S. Böhm , P. Behrens , A. Martinez-Schramm , J. F. Löhr. Der Orthopäde Issue: Volume 31, Number 1 Publisher: Springer-Verlag GmbH ISSN: 0085-4530 (Paper) 47. Mutational mechanisms of Williams-Beuren syndrome deletions. Bayes M, Magano LF, Rivera N, Flores R, Perez Jurado LA. Am J Hum Genet. 2003 Jul;73(1):131-51 48. Large-scale copy number polymorphism in the human genome. Sebat J, Lakshmi B, Troge J, Alexander J, Young J, Lundin P, Maner S, Massa H, Walker M, Chi M, Navin N, Lucito R, Healy J, Hicks J, Ye K, Reiner A, Gilliam TC, Trask B, Patterson N, Zetterberg A, Wigler M. Science. 2004 Jul 23;305(5683):525-8. 49. A novel 5q11.2 deletion detected by microarray comparative genomic hybridisation in a child referred as a case of suspected 22q11 deletion syndrome. Prescott K, Woodfine K, Stubbs P, Super M, Kerr B, Palmer R, Carter NP, Scambler P. Hum Genet. 2005 Jan;116(1-2): 83-90. 73 74 6. Anhang 75 Nr.1 (113E) Nr. 2 (071E) 76 Nr.3 (208E) Nr.4 (328E) 77 Nr.5 (111E) Nr.6 (067E) 78 Nr.7 (133E) Nr.8 (281E) 79 Nr.9 (350E) Nr.11 (340E) 80 Nr. 12 (082E) Nr.13 (349E) 81 Nr.14 (056E) Nr. 15 (186E) 82 Nr.16 (142E) Nr. 17 (386E) 83 Nr. 18 (206E) Nr. 19 (373E) 84 Nr. 20 (091E) Nr.21 (162E) 85 Nr.22 (079E) Nr.23 (139E) 86 Nr.24 (368E) Nr.25 (011E) 87 Nr. 26 (010E) Nr. 28 (007E) 88 Nr. 29 (261E) Nr 30 (137E) 89 Nr. 31 (077E) Nr. 32 (127E) m/ w 90 Nr. 33 (174E) Nr. 34 (210E) 91 Nr. 35 (284E) Nr. 36 (351E) 92 Nr. 37 (399E) Nr.38 (400E) 93 Kontrolle S 94 Danksagung An dieser Stelle möchte ich mich bei - Prof. Dr. H.-Hilger Ropers MPI für molekulare Genetik und PD Dr. W. Schuster FU Berlin , Institut für Biologie/ An. Genetik für die Möglichkeit am MPI meine Diplomarbeit zu machen und freundliche persönliche Betreuung. - Dr. R. Ullmann und Dr. F. Erdogan für die praktische Einarbeitung und zahlreiche wertvollen Ratschläge bei der Durchführung meiner Arbeit. 95 Eidesstattliche Erklärung: Hiermit erkläre ich, daß die Diplomarbeit von mir selbst und ohne die Hilfe Dritter verfaßt wurde, auch in Teilen keine Kopie anderer Arbeiten darstellt und die benutzten Hilfsmittel sowie die Literatur vollständig angegeben sind. Berlin 03.03.2005 Drozdenko Gennadiy 96