Methoden zur Sequenzierung von DNA
Werbung

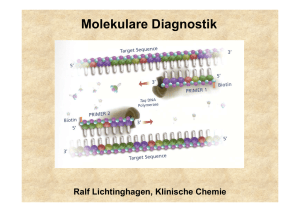
Methoden zur Sequenzierung von DNA Anne Röschenkemper SS 2006 Was ist DNA-Sequenzierung? • Die Analyse der DNA-Struktur auf Nucleotid-Ebene • Erst seit 1977 gibt es DNA-Sequenzierungstechniken • Gründe dafür: • Erst seit dieser Zeit gibt es Klonierungstechniken, durch die DNA in ausreichender Menge erhalten werden kann • Auch Restriktionsendonucleasen kennt man erst seit Mitte der 70er Jahre; nur durch Behandlung mit diesen Enzymen kann DNA in kleine Fragmente zerteilt werden (& auch rekonstruiert werden) DNA-Sequenzierung 2 Welche Methoden gibt es? 1) Sequenzierung durch chemische Spaltung Die Maxam-Gilbert-Methode 2) Sequenzierung durch Kettenabbruch Die SangerMethode 3) weitere Methoden: zB. Pyrosequenzierung Sequenzierung durch Hybridisierung DNA-Sequenzierung 3 Sequenzierungsstrategien • 2) Problem: nur ca. 1000 bp können mit dem Sanger-Verfahren sequenziert werden Lösung: Spezifischer Abbau und Fraktionierung durch Restriktionsendonucleasen kleine, vollständig sequenzierbare Fragmente Sequenzierung 3) Rekonstruktion der Fragmente (durch „overlap“) • 1) Komplette Sequenz RE 1 RE 2 DNA-Sequenzierung 4 Sequenzierung durch chemische Spaltung: Die Maxam-Gilbert-Methode • Von Allan Maxam und Walter Gilbert entwickelt; erste Veröffentlichung: Februar 1977 (PNAS) Prinzip: basenspezifische Spaltung spezifische Spaltung an einem einzigen Nucleotid-Typ durch bestimmte Reagenzien Jedes DNA-Fragment wird im Durchschnitt nur einmal gespalten (zufällig an einer der chemisch sensiblen Bindungen) radioaktive Markierung des 5` - Endes mit 32P DNA-Sequenzierung 5 Die Maxam-Gilbert-Methode • Markierung des 5´-Endes mit 32P •Wenn schon P an 5´ vorhanden: •Im zweiten Schritt wird nun radioaktiv markiertes ATP benötigt (am γ-Phosphor-Atom) DNA-Sequenzierung 6 Die Maxam-Gilbert-Methode Beispiel für eine basenspezifische Spaltung 32P- A T G T A G G T C A G A -3´- T Spaltung an der 5´-Seite von G: 32P- ATGTAG GTCA 32P- ATGTAG 32P- ATGTA 32P- AT Die 5´-markierten Fragmente können nun detektiert werden DNA-Sequenzierung 7 Die Maxam-Gilbert-Methode Detektion der 5´-markierten Fragmente: • mittels PAGE Trennung nach Größe • Autoradiogramm: A T G C Leserichtung Laufrichtung DNA-Sequenzierung 8 Die Maxam-Gilbert-Methode • Spezifische Spaltung an G: • Reagenzien: Dimethylsulfat Piperidin DNA-Sequenzierung 9 Die Maxam-Gilbert-Methode Spezifische Spaltung an A & G • ähnlich der „G-Spaltung“ • Reagenzien: Dimethylsulfat Piperidin • Methylierung von A an N(3) und nicht an N(7) • Reaktion unter sauren Bedingungen (Spaltung von A unter basischen Bedingungen: 5 mal langsamer als G-Spaltung) A & G werden in gleichen Anteilen gespalten • Ermittlung der A-Positionen durch Vergleich von „G“ und „G&A“ DNA-Sequenzierung 10 Die Maxam-Gilbert-Methode Spezifische Spaltung an C & T • Reagenzien: Hydrazin Piperidin DNA-Sequenzierung 11 Die Maxam-Gilbert-Methode Spezifische Spaltung an C • Ähnlich der „C&T-Spaltung“ • Reagenzien: Hydrazin Piperidin • Zusätzlich: Lösung enthält NaCl • Dadurch findet die Spaltung fast nur an C statt • Ermittlung der T-Positionen durch Vergleich von „C“ und „T&C“ DNA-Sequenzierung 12 Die Maxam-Gilbert-Methode Vorteile • Eine Sequenz kann allein mithilfe der Restriktionskarte entschlüsselt werden • Die Sequenz kann fast vollständig ermittelt werden • Die Sequenz kann vom ursprünglichen DNA-Molekül stammen (nicht von einer enzymatisch hergestellten Kopie) keine Kopierfehler Nachteile • Nur relativ kurze Sequenzen können ermittelt werden • Relativ langsame und unzuverlässige Methode (im Vergleich zur Sanger-Methode) • Gefährliche Chemikalien werden verwendet DNA-Sequenzierung 13 Sequenzierung durch Kettenabbruch: Die Sanger-Methode Nach Frederick Sanger (et al.); erste Veröffentlichung: Dezember 1977 Prinzip: in-vitro-Synthetisierung mit Kettenabbruch • Zu einem denaturiertem DNA-Strang wird mithilfe von DNA-Polymerase ein komplementärer Strang synthetisiert • Die Reaktion wird an einer bestimmten Base gestoppt, indem ein Didesoxynucleotid in den Strang eingebaut wird • Detektion: per Autoradiographie oder Fluorimetrie DNA-Sequenzierung 14 Die Sanger-Methode Didesoxynucleotide • die DNA wird in 3´-5´-Richtung verlängert • der ddNTP fehlt in der 3´-Position die OH-Funktion, an der ein weiteres Nucleotid andocken könnte • wird ein ddNTP in den Strang eingebaut, stoppt die Synthese • um genügend lange Sequenzen zu erfassen, darf der ddNTP Anteil nur gering sein (~ 1:200 ddNTP : dNTP) • man macht vier Ansätze: ddATP, ddGTP ,ddCTP, ddTTP DNA-Sequenzierung 15 Die Sanger-Methode Benötigte Komponenten • • • • • • • DNA-Einzelstrang Primer DNA-Polymerase dNTPs ddNtp (pro Ansatz nur eine Sorte) Markierung Elektrophoresesystem DNA-Sequenzierung 16 Die Sanger-Methode Die Kettenabbruchmethode im Überblick vier Ansätze zufällige Verteilung der ddNTPs Nur die synthetisierten Stränge werden erfasst Prinzip: kleine Moleküle wandern schneller! DNA-Sequenzierung 17 Die Sanger-Methode • Fluoreszenzsequenzierung •Man kann Primer, ddNTPs oder dNTPs kovalent an Fluorophore knüpfen wenn man pro Reaktionsansatz ein anderes Fluorophor verwendet, benötigt man nur eine Gelelektrophorese-Laufspur •Vorteil: schneller als Autoradiographie Ungefährliche Substanzen leicht automatisierbar (~10.000 Basen können pro Tag identifiziert werden; manuelle Methode: ~ 150 / d) DNA-Sequenzierung 18 Die Sanger-Methode Zyklische Sequenzierung I • Problem der Standardkettenabbruchmethode: Empfindlichkeit der Methode wird durch die Molarität der DNA-Sequenz begrenzt • Lösung: zyklische Sequenzierung DNA-Sequenzierung 19 Die Sanger-Methode Zyklische Sequenzierung II Vorteile: • wenig DNA wird benötigt • hohe T verhindert „Sequenzartefakte“ • leicht automatisierbar Nachteile: • Taq-Polymerase baut Fluoreszenz-ddNTPs recht schlecht ein • Taq.Polymerase liest hochpolymere Basenfolgen schlechter DNA-Sequenzierung 20 Die Sanger-Methode Vorteile • Man kann längere Sequenzen als mit dem MaxamGilbert-Verfahren sequenzieren • Schnelle & recht zuverlässige Methode; leicht automatisierbar • Man kann radioaktive Reagenzien mit der Fluorimetrie umgehen Nachteile • Man benötigt einen Primer ein Teil der Sequenz muss bekannt sein DNA-Sequenzierung 21 Fazit • Die Sanger-Methode ist besser als die MaxamGilbert-Methode!!! DNA-Sequenzierung 22 Alternative Methoden • Sequenzierung durch Hybridisierung • Pyrosequenzierung • • • • Mikrokanal-Sequenzierung Massenspektrometrie Rastertunnelmikroskopie Nanoporen DNA-Sequenzierung 23 Alternative Methoden Sequenzierung durch Hybridisierung • gelfreie Sequenzierungsmethode micro arrays • Matrize von kurzen Oligonucleotiden, die sich auf einer Glas- oder Siliciumoberfläche befinden • die zu sequenzierenden DNA-Fragmente werden markiert und auf die Oligonucleotidmatrix gebracht, so dass komplementäre Strukturen hybridisieren • aus den Hybridisierungsmustern (Position und Signalintensität) kann auf die Sequenz geschlossen werden • Probleme: Fehlhybridisierungen repetitive Sequenzen sind schwer zu analysieren • Aber: kürzere Sequenzabschnitte oder Mutationen können mit dieser Methode erkannt werden Medizinische Diagnostik (z.B. SNPs) DNA-Sequenzierung 24 Alternative Methoden Sequenzierung durch Synthetisierung: Pyrosequenzierung I • • • • Methode ohne Gele und Marker Prinzip: „Beobachten der Synthese“ zu der einzelsträngigen DNA werden Enzyme gegeben und dann abwechselnd die verschiedenen Nucleotide man benötigt vier Enzyme: 1) 2) 3) 4) DNA-Polymerase Einbau der Nucleotide Apyrase Abbau ungenutzter Nucleotide Sulfurylase Erzeugung von Licht aus PPi Luciferase „ DNA-Sequenzierung 25 Alternative Methoden Pyrosequenzierung II •die Lichtemission wird aufgezeichnet und ist in einem Pyrogramm® als Peak erkennbar •die Intensität des Lichts ist proportional zur Anzahl der in den Strang eingebauten Basen •schnelle Methode •ideal für die medizinische Diagnostik DNA-Sequenzierung 26 Bedeutung der DNA-Sequenzierung Das Humangenomprojekt • Sequenzierung der menschlichen DNA (3,2 Mrd. bp!!!) • Erkennung von codierenden Sequenzen (~30.000 – 40.000 Gene) für z.B. monogene Krankheiten wie Alzheimer, Brustkrebs, Diabetes (jeweils die vererbbaren Formen) Enzymmutationen und damit verbundene Medikamentenunverträglichkeiten (z.B. CYP-450Enzyme) DNA-Sequenzierung 27 Bedeutung der DNA-Sequenzierung DNA-Chips für CYP-450 • bestimmte Enzyme dieser Familie sind an der Metabolisierung von Arzneimitteln beteiligt • Fehlerhafte Gene führen zu veränderter Metabolisierung: z.B. das CYP-2D6-Gen Variante A: normale Aktivität Variante B: verstärkte Aktivität („ultraschneller Metabolisierer“) Variante C: inaktives Enzym („langsamer Metabolisierer“) • bei Prodrugs: B toxische Effekte C keine Effekte (z.B. wird Codein durch dieses Enzym zu Morphin metabolisiert) • Lösung für diese Probleme: DNA-Chip, der fehlerhafte Varianten des Gens erkennt und dadurch eine verbesserte Therapie ermöglicht DNA-Sequenzierung 28 Bedeutung der DNA-Sequenzierung Ausblick • durch die Verwendung von DNA-Chips in der medizinischen Diagnostik können SNPs und andere genetische Veränderungen erkannt werden • dies macht eine auf den „zugeschnittene“ Therapie, die nicht nur äußere Faktoren wie Gewicht und Geschlecht berücksichtigt, möglich • Möglichkeit der Früherkennung genetisch bedingter Krankheiten • Methoden wie die von Sanger eignen sich eher zur Sequenzierung des kompletten Genoms, sind für die medizinische Diagnostik meist zu langsam und kompliziert, dafür aber genauer DNA-Sequenzierung 29 Literatur • Voet, Voet – Biochemie • Luke Alphey – DNA-Sequenzierung, 1998 Spektrum Akademischer Verlag • Müller-Esterl – Biochemie, 2004 Elsevier Verlag • Lottspeich, Zorbas – Bioanalytik, 1998 Spektrum Akademischer Verlag • www.pyrosequencing.com • „Die Humangenomforschung in Deutschland“ – BMBF • Sanger et al.: „DNA-Sequencing with chain-terminating inhibitors“ – Proc. Natl. Acad. Sci. USA, Vol. 74 (1977), pp. 5463-5467 • Maxam, Gilbert: „A new method for Sequencing DNA“. Proc. Natl. Acad. Sci. USA, Vol. 74 (1977), pp. 560-564 DNA-Sequenzierung 30