Die ribosomale E-Stelle
Werbung

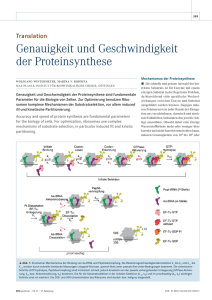
695_725.qxd 26.10.2006 11:53 Uhr Seite 723 723 Wer „A“ sagt, muss auch „E“ sagen: Die ribosomale E-Stelle DANIELA WITTEK UND KNUD H. NIERHAUS MAX-PL ANCK-INSTITUT FÜR MOLEKUL ARE GENETIK, AG RIBOSOMEN Ribosomen stehen als Mittler zwischen den Welten der Nukleinsäuren und der Proteine, weil sie die genetische Information in Strukturen der Proteine übersetzen. Ribosomen gehören zu den kompliziertesten Strukturen, die wir in der Biologie kennen, sie enthalten über 50 ribosomale Proteine und drei bis fünf ribosomale RNAs, die alle in einer Kopie pro Ribosom vorkommen. ó Die einzige Ausnahme ist das Protein L7/L12, das in 4 bis 6 Kopien vorliegt. Jedes Ribosom zerfällt in eine kleine und eine große Untereinheit. Das funktionelle Herzstück der Ribosomen, die Verlängerung (Elongation) der wachsenden Peptidkette um eine Aminosäure, vollzieht sich in allen Zellen dieses Planeten nach denselben Regeln. Die Abfolge der Codonsequenz auf der mRNA wird in die Folge der Aminosäuren übertragen, und zwar mithilfe der tRNAs, Lförmige Moleküle (in Abb. 1 als stilisierte Gabeln dargestellt), die an einer Seite das Anticodon komplementär zu einem Codon tragen (die dreizinkige Forke der tRNAs in Abb. 1) und am anderen Ende die dazuge- hörige Aminosäure. Seit über 25 Jahren wissen wir, dass die Ribosomen drei tRNA Bindestellen tragen, die A-, P- und E-Stellen, die in dieser Reihenfolge von einer tRNA während der Proteinsynthese durchlaufen werden. Dennoch wissen wir erst seit kurzem um die fundamentale Rolle der E-Stelle für die Genauigkeit der Proteinsynthese. Dabei gilt es, zwei Gesichtspunkte zu betrachten: (i) Im Gegensatz zur landläufigen Meinung ist ein Einbau einer falschen Aminosäure für Struktur und Funktion eines Proteins ohne schädliche Konsequenz. Nur ein Fehleinbau von 400 zerstört die Aktivität eines Proteins, was eine Funktion der E Stelle ist. (ii) Eine der erstaunlichsten Errungenschaften des Ribosoms ist die Erhaltung des Leserasters, das Abgreifen der mRNA Information in Schritten von drei Nukleotiden. Der zufällige Verlust des Leserasters passiert nur in weniger als einem Fall von 30.000 abgelesenen Codons (Übersicht bei [1]). Diese fabelhafte Fähigkeit ist ebenfalls eine Eigenschaft der EStelle. Ein normaler Elongationszyklus ˚ Abb. 1: Der Elongationszyklus im Rahmen des allosterischen Dreistellenmodells[4]. Die A-Stellenbesetzung ist in zwei Unterreaktionen geteilt, den Dekodierungsschritt und die nachfolgende Besetzung der A-Stelle. Danach folgen der Peptidyltransfer und die Translokation (siehe Text). BIOspektrum | 07.06 | 12. Jahrgang Wir unterscheiden im Elongationszyklus drei prinzipielle Schritte (Abb. 1). Im ersten Schritt wird die richtige (= cognate) Aminoacyl-tRNA in die A-Stelle nach Maßgabe des hier vorliegenden Codons gebunden. Dabei ist die benachbarte P-Stelle mit einer tRNA besetzt, die die schon synthetisierte Polypeptidkette trägt (Polypeptidyl-tRNA, pptRNA). Im zweiten Schritt wird die synthetisierte Polypeptidkette von der pp-tRNA abgeschnitten und mittels einer Peptidbindung auf die Aminoacyl-tRNA (aa-tRNA) übertragen, eine enzymatische Funktion der großen Untereinheit. Das Ergebnis ist, dass die Polypeptidyl-tRNA nun in der A-Stelle sitzt, verlängert um eine Aminosäure. Die P-Stelle trägt nun eine „nackte“ tRNA ohne Aminoacyl- oder Peptidyl-Rest. Im dritten Schritt findet die „Translokation“ statt, bei der beide tRNAs in die benachbarte tRNA Bindestelle transportiert werden. Gleichzeitig wandert die mRNA mit den tRNAs, so dass ein neues Codon in die A-Stelle rückt, die dann im folgenden Elongationszyklus von der passenden Aminoacyl-tRNA besetzt wird. 695_725.qxd 724 26.10.2006 11:53 Uhr Seite 724 WISSENSCHAFT ˚ Abb. 2: Das Codonlexikon in der Form der „Codonsonne“. Bevor wir auf die Rolle der E-Stelle zu sprechen kommen, müssen wir noch eine fundamentale Beziehung zwischen der A- und EStelle erwähnen, der ersten und letzten tRNA Bindestelle. Diese beiden Bindestellen sind reziprok miteinander gekoppelt, d. h. eine Besetzung der A-Stelle erniedrigt die E-Stellen Affinität (= tRNA Bindungsvermögen) und führt damit zu einem Verlust der tRNA aus der E-Stelle[2, 3]. Das umgekehrte gilt auch, eine E-Stellen Besetzung erniedrigt die Affinität der A-Stelle (Übersicht bei [4]). Warum ist ein Fehleinbau einer Aminosäure für das Protein kein Problem? Eine tRNA ist ein großes Molekül, so groß wie ein Standard Protein im Cytoplasma und ist etwa 76 Nukleotide lang, wovon nur drei Nukleotide das Anticodon bilden. Die Auswahl der richtigen aa-tRNA beruht nur auf diesem Anticodon, die weiteren zahlreichen Bindungskontakte der tRNA mit der A-Stelle tragen nicht zur Auswahl bei. Damit wird klar, dass nur der allerkleinste Anteil der A-Stellen Affinität von dem Anticodon aufgebracht werden kann. Das Problem wird dadurch verschärft, dass eine aa-tRNA nicht das Substrat für die A-Stellen-Bindung ist, sondern vielmehr ein Komplex von aa-tRNA mit dem Elongationsfaktor EF-Tu und GTP (ternärer Komplex). EF-Tu ist zweimal so groß wie eine tRNA und erhöht die Affinität zur A-Stelle um einige Zehnerpotenzen. Damit sind alle 42 verschiedene aa-tRNAs Mitbewerber im Bindungsgetümmel an der A-Stelle. Da die Bindungsenergie für den ternären Komplex riesig, jedoch die Diskriminierungsenergie (Codon-Anticodon Wechselwirkung) klein ist, müsste für die Auswahl der richtigen aa-tRNA (gleich Einstellung des Gleichgewichtes) etwa eine Stunde vergehen. Das ist jedoch nicht das, was wir am Ribosom beobachten. Für die ˚ Abb. 3: Cryo-elektronenmikroskopische Rekonstruktion eines 70S E. coli Ribosoms im Augenblick der Bindung eines ternären Komplexes EF-Tu•aa-tRNA•GTP (Dekodierungsschritt). Die aa-tRNA (violett) hat noch nicht die A-Stelle mit ihrer hohen Affinität für aa-tRNAs erreicht, obwohl Dekodierung möglich ist (Position A/T, T für ternären Komplex;[8] tRNAs in P und E Stellen sind grün bzw. Orange[9]. Auswahl der richtigen aa-tRNA benötigt das Ribosom weniger als 50 msec. Ein Trick für die Lösung des Problems liegt in der Reziprozität von A- und E-Stelle. Ist die E-Stelle besetzt, hat die A-Stelle eine niedrige Affinität für den ternären Komplex, d. h. im Wesentlichen sind nur Codon-Anticodon Wechselwirkungen erlaubt. Damit ist die Bindung eines ternären Komplexes außerhalb des Anticodon praktisch nicht vorhanden, was zu einer schnellen Einstellung des Gleichgewichtes führt. Eine weitere wichtige Konsequenz ist, dass ein Dekodierungsfehler nur mit einer aa-tRNA möglich ist, die ein Anticodon ähnlich dem cognaten aufweist. Eine aa-tRNA mit einem unähnlichen Anticodon ist aus dem Spiel, da das Anticodon nicht komplementär zum Codon der A-Stelle ist und Bereiche außerhalb des Anticodons nicht mit dem Ribosom in Wechselwirkung treten können. Wir sehen, dass mit diesem Trick die Auswahl der richtigen aa-tRNA nur zwischen aa-tRNAs mit dem richtigen und ähnlichen Anticodon getroffen werden muss. Das sind nicht mehr als vier aa-tRNAs mit verschiedenen Anticodons, während die Masse der aa-tRNAs mit einem Anticodon unähnlich dem cognaten (mehr als 35) den Auswahlprozess nicht stören können[5]. Wir wollen hier auch bemerken, dass ein Verlesen am häufigsten im dritten Nukleotid eines Codons passiert („wobbel“ Position), das mittlere Nukleotid wird praktisch nie verlesen, und die erste Position sehr selten. Die Geschichte ist noch nicht zu Ende, denn nun müssen wir noch einen Blick auf das Codonlexikon werfen (Abb. 2). Wir kennen keine chemische Erklärung, warum z. B. das Codon UUC für die Aminosäure Phe oder das AUG für die Aminosäure Met codiert. Wir kennen aber sehr wohl die Logik, nach der das Codonlexikon organisiert ist. Diese besagt, dass ähnliche Codons dieselbe oder eine chemisch ähnliche Aminosäure codieren. Schauen wir uns die dritte Position an, die am häufigsten verlesen wird, z. B. GC(U, C, A oder G). Die Codons GC(U, C) und GC(A, G) werden von verschiedenen aa-tRNAs decodiert, die aber alle dieselbe Aminosäure Ala tragen. Ein Decodierungsfehler führt also zu einem Einbau Ala statt Ala ohne Konsequenzen für das Protein. Ein anderes Beispiel ist GA(U, C) und GA (A, G). Bei einem Dekodierungsfehler wird statt der sauren Aminosäure Asp die ebenfalls saure Aminosäure Glu eingebaut, in den meisten Fällen ebenfalls ohne Konsequenzen für das Protein. Diese „Pufferung“ mittels des Codonlexikons verhindert in den allermeisten Fällen bei einem Dekodierungsfehler den Einbau einer chemisch unähnlichen Aminosäure, z.B. statt des hydrophoben Val (Codons GAN) die hydrophile Ser, mit vermutlich ernsthaften Folgen für Faltung und Funktion eines Proteins. Wir sehen, dass das Zusammenspiel von der Organisation des Codonlexikons und der Reziprozität der ribosomalen E-und A-Stellen dafür sorgen, dass ein Dekodierungsfehler in den meisten Fällen ohne belang für die Proteinfunktion bleibt. Mithilfe der Cryo-Elektronenmikroskopie haben wir diesen Dekodierungsprozess an einer gering affinen A-Stelle direkt sehen können. Die Dekodierung findet statt, bevor die aatRNA die volle A-Stelle besetzt hat (Abb. 3). Die E-Stelle ist auch für die Einhaltung des Leserasters von größter Bedeutung Wie in der Einleitung erwähnt, ist die Erhaltung des Leserasters eine bewundernswerte Leistung des Ribosoms. In weniger als einem Fall geht bei 30.000 Elongationsrunden das Leseraster verloren. Auch hier ist die Reziprozität von A- und E-Stelle beteiligt. Sie hat die wichtige Konsequenz, dass während eines Elongationszyklus zumeist zwei tRNAs auf dem Ribosom sitzen, vor der Translokation in der A- und P-Stelle, nach der Translokation in P- und E-Stelle (Abb. 1). Beide tRNAs sind mit der mRNA über Codon-Anticodon Wechselwirkung verknüpft. Diese Verknüpfung erlaubt es, dass während einer Translokation die tRNAs auf dem Ribosom aktiv verschoben werden, die mRNA folgt passiv. Die mRNA wird also über die tRNAs am Ribosom BIOspektrum | 07.06 | 12. Jahrgang 695_725.qxd 26.10.2006 11:53 Uhr Seite 725 725 fixiert, sie selbst hat – wenn überhaupt – nur geringen Kontakt mit dem Ribosom[6]. Damit ist die Verbindung von tRNA-mRNA über sechs Basenpaare (= zwei Codon-Anticodons) für die Einhaltung des Leserasters verantwortlich. Tatsächlich konnte gezeigt werden, dass eine Störung der Codon-Anticodon Wechselwirkung in der E-Stelle zu einem Verlust der tRNA in der E-Stelle führt, was für eine sonderliche Syntheseregulation des Terminationsfaktors RF2 ausgenutzt wird. Hier geht, unterstützt durch weitere Faktoren, das Leseraster verloren, wobei eine solche Verschiebung in die Vorwärtsrichtung der mRNA (+1 Richtung) notwendig für die RF2 Synthese ist[7]. Die Ergebnisse lassen die Abschät- zung zu, dass ohne E-Stelle das Leseraster nach dem Einbau von 20 bis 50 Aminosäuren verloren ginge. Damit wäre eine geordnete Synthese von 300 – 500 Aminosäuren (durchschnittliche Länge der Proteine) nicht möglich. ó Literatur [5] Geigenmüller, U. and Nierhaus, K.H. (1990): EMBO J. 9:4527–4533 [6] Alexeeva, E.V., Shpanchenko, O.V., Dontsova, O.A., Bogdanov, A.A. and Nierhaus, K.H. (1996): Nucl. Acids Res. 24:2228–2235 [7] Marquez, V., Wilson, D.N., Tate, W.P., Triana-Alonso, F. and Nierhaus, K.H. (2004): Cell 118:45–55 [8] Valle, M., Sengupta, J., Swami, N.K., Grassucci, R.A., Burkhardt, N., Nierhaus, K.H., Agrawal, R.K. and Frank, J. (2002): EMBO J. 21:3557–3567 [9] Valle, M. et al. (2003): Nat. Struct. Biol. 10:899–906 [1] Kurland, C.G., Jørgensen, F., Richter, A., Ehrenberg, M., Bilgin, N. and Rojas, A.-M. (1990): Through the accuracy window. In: Dahlberg, A., Hill, W.E., Garrett, R.A., Moore, P.B., Schlessinger, D. and Warner, J.R. (Hrsg.) The RibosomeStructure, Function, and Evolution. Amer. Soc. Microbiol., Washington, D. C., 513–526 [2] Rheinberger, H.-J. and Nierhaus, K.H. (1986): J. Biol. Chem. 261:9133–9139 [3] Dinos, G., Kalpaxis, D.L., Wilson, D.N. and Nierhaus, K.H. (2005): Nucleic Acids Res. 33:5291–5296 [4] Blaha, G. and Nierhaus, K.H. (2001): Cold Spring Harbor Symposia on Quantitative Biology 65:135–145 Korrespondenzadresse: Daniela Wittek Knud H. Nierhaus MPI Molekulare Genetik, AG Ribosomen Ihnestraße 73 D-14195 Berlin Tel.: 030-8413-1700 Fax: 030-8413-1594 [email protected] AUTOREN Daniela Wittek Knud H. Nierhaus Nach dem Studium an der Berliner Freien Universität machte sie ihre Diplomarbeit am MPI für Molekulare Genetik Berlin in der Gruppe Nierhaus auf dem Gebiet der Proteinbiosynthese. In derselben Gruppe ist sie zur Zeit Doktorandin. Knud H. Nierhaus studierte Medizin und promovierte in Tübingen. 1968 wechselte er an das MPI für molekulare Genetik in Berlin. Dort ist er zurzeit Leiter einer Forschungsgruppe, die sich mit unterschiedlichen Aspekten der Translation beschäftigt. Er ist außerplanmäßiger Professor an der Technischen Universität Berlin. Ergebnisse seiner Forschungen umfassen die Entwicklung einer Methode zur Zerlegung und Rekonstitution der großen ribosomalen Untereinheit von E. coli in ihre 35 verschiedenen Bestandteile und die Entdeckung der dritten universellen tRNA-Bindungsstelle auf dem Ribosom. BIOspektrum | 07.06 | 12. Jahrgang