Translation ist die RNA-gesteuerte Synthese eines Polypeptids
Werbung

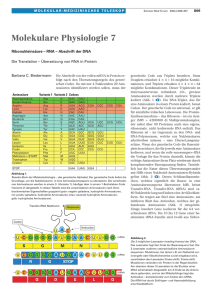
Translation ist die RNA-gesteuerte Synthese eines Polypeptids: Eine nähere Betrachtung Bei der Translation übersetzt die Zelle die genetische Information und baut nach dieser Anweisung ein Protein auf. Die genetische Botschaft besteht in einer Folge von Codons auf den mRNA-Molekülen, und der Adapter zwischen Aminosäure und RNA-Codon ist eine andere RNA-Spezies, die Transfer-RNA (tRNA) genannt wird. Die Aufgabe der tRNA ist es, Aminosäuren aus dem cytoplasmatischen AminosäureReservoir zum Ribosom zu transportieren. Jede Zelle unterhält in ihrem Cytoplasma einen Vorrat aller 20 Aminosäuren, die entweder aus Vorstufen synthetisiert oder aus dem umgebenden Medium aufgenommen wurden. Das Ribosom bindet passende Aminosäuren, die von tRNAs angeliefert werden, an das wachsende Ende der Polypeptidkette (Abbildung 16.9). Die tRNA-Moleküle sind nicht alle gleich. Der Schlüssel zur Translation der genetischen Botschaft in eine bestimmte Aminosäuresequenz liegt in der Tatsache, dass jeder Typ von tRNA die Verbindung zwischen einem bestimmte Codon auf der mRNA und einer passenden Aminosäure herstellt. Kommt das tRNA-Molekül zum Ribosom, so trägt es an einem Ende eine Aminosäure. Am anderen Ende besitzt die tRNA ein Triplett, das man als Anticodon bezeichnet. Mit diesem Anticodon bindet die tRNA entsprechend den Regeln der Basenpaarung an ein komplementäres Codon auf der mRNA. Das mRNA-Codon UUU wird beispielsweise in die Aminosäure Phenylalanin translatiert (siehe Abbildung 16.5). Die tRNA, die über Wasserstoffbrücken an dieses Codon andockt, enthält AAA als Anticodon und überträgt Phenylalanin. Wenn ein mRNA-Molekül durch ein Ribosom gleitet, wird immer dann Phenylalanin in die Polypeptidkette eingebaut, wenn das Codon UUU zur Translation präsentiert wird. Die genetische Botschaft wird Codon für Codon übersetzt, indem tRNAs Aminosäuren in der vorgeschriebenen Folge anliefern und die ribosomalen Enzyme sie zu einer Polypeptidkette verknüpfen. Das tRNA-Molekül gleicht einer Spielkarte mit dem „Nucleinsäure-Wort" (Anticodon) auf der einen Seite und dem entsprechenden „Proteinwort" (Aminosäure) auf der anderen Seite. Die Translation ist vom Prinzip hef einfach, aber ihre Biochemie und Mechanistik ist äußerst komplex. Zum besseren Verständnis sehen wir uns zunächst die Hauptakteure im zellulären Schauspiel der Proteinsynthese näher an. Anschließend werden wir besprechen, wie durch das Zusammenspiel dieser Akteure eine Polypeptidkette entsteht. Struktur und Funktion der Transfer-RNA Wie mRNA und andere RNA-Spezies werden auch die tRNA-Moleküle an einer DNA-Vorlage im Kern einer eukaryotischen Zelle synthetisiert. Genauso wie die mRNA muss auch die tRNA aus dem Kern ins Cytoplasma, dem Ort der Translation, gelangen. Jedes tRNA-Molekül kann wiederholt verwendet werden. Es nimmt die zu seiner Struktur passende Aminosäure aus dem Cytosol auf, bringt seine Fracht zum Ribosom und verlässt dieses wieder, um eine weitere Ladung aufzunehmen. Passend zu dieser Funktion als Überträger spezifischer Aminosäuren besitzen tRNA-Moleküle eine unverwechselbare Struktur (Abbildung 16.10). Ein tRNA-Molekül besteht aus einem einzelnen RNAStrang von etwa 80 Nucleotiden Länge (das ist sehr kurz, verglichen mit den Hunderten von Nucleotiden eines mRNA-Moleküls). Der RNA-Einzelstrang ist formal in eine zweidimensionale Sekundärstruktur und diese in eine dreidimensionale Tertiärstruktur gefaltet. Die Art der Faltung ist durch die Interaktion von Teilbereichen der Nucleotid-kette bestimmt. In gewissen Regionen des tRNA-Moleküls paaren sich die Basen über Wasserstoffbrücken. In einer zweidimensionalen Darstellung geben die gepaarten und nichtgepaarten Bereiche dem Molekül die Gestalt eines Kleeblatts. Das zweidimensionale Kleeblatt eines tRNA-Moleküls windet und faltet sich in eine recht kompakte dreidimensionale Struktur von L-förmiger Gestalt. Die Schleife, die das geschlossene, lange Ende des L bildet, trägt das Anticodon, ein spezielles Basentriplett, welches am entsprechenden mRNA-Codon bindet. Am gegenüberliegenden, kurzen Ende des L ist das tRNA-Molekül offen. Hier befindet sich am 3'-Ende die Bindungsstelle für die Aminosäure. Gäbe es für jedes der mRNA-Codons, die eine Aminosäure spezifizieren, eine eigene tRNA, würden 61 verschiedene tRNAs existieren (siehe Abbildung 16.5). In der Tat gibt es aber weniger tRNAs, nämlich je nach Organismus nur 31 bis 45. Diese Zahl reicht aus, weil einige tRNAs Anti- codons besitzen, die zwei oder mehr Codons erkennen. Diese Freiheit wiederum ist möglich, weil die Basenpaarung zwischen der dritten Base eines Codons und der entsprechenden (der ersten) Base des tRNA-Anticodons nicht so exakt erfolgt wie die Paarung von DNA-DNA oder DNA-mRNA. Diese Idee einer „wackeligen" Basenpaarung wird als Wobble-Hypothese bezeichnet (vom englischen wobble für „wackeln"). Die vielseitigsten tRNA-Moleküle enthalten Inosin (I), ein modifiziertes Nucleotid, in der Wobble-Posi-tion ihres Anticodons. Inosin entsteht durch enzymatische Veränderung eines Adenosins (obwohl es chemisch ein des-aminiertes Guanosin darstellt) an der fertigen tRNA. Wenn sich Anticodon und Codon vereinigen, kann die Base Inosin mit jeder der drei Basen U, C oder A Wasserstoffbrücken ausbilden. Daher kann ein tRNA-Molekül mit dem Anticodon CCI an die Codons GGU, GGC und GGA binden, die alle für die Aminosäure Glycin stehen. Die Wobble-Hypothese erklärt, warum synonyme Codons für eine bestimmte Aminosäure, die sich lediglich in ihrer dritten Base, nicht aber in anderen Basen unterscheiden, nur eine einzige tRNA benötigen. Dadurch kommt ein Organismus mit minimal 31 verschiedenen tRNAs aus. Das ,,Wobbein" auf der tRNA-Ebene wird durch die Redundanz des genetischen Codes möglich. Allerdings bleiben dessen Regeln unangetastet: Die Synthese eines Polypeptids erfolgt nicht „wackelig", sondern hochspezifisch. Aminoacyl-tRNA-Synthetasen Die Bindung von Codon und Anticodon ist der zweite Erkennungsschritt, der für die genaue Translation der genetischen Botschaft notwendig ist. Ihm muss aber eine korrekte Bindung einer tRNA mit ihrer spezifischen Aminosäure vorangehen. Eine tRNA, die spezifisch an ein Codon für eine Aminosäure bindet, darf nur diese Aminosäure und keine andere zum Ribosom bringen. Die Zusammenführung der richtigen Aminosäure mit der zugehörigen tRNA wird durch ein spezifisches Enzym bewerkstelligt, das man als Aminoacyl-tRNASynthetase bezeichnet. Es gibt eine ganze Familie solcher Enzyme, für jede Aminosäure eines. Diese Enzyme sind die eigentlichen Dolmetscher, welche die Nucleotid-sprache in die Aminosäuresprache übersetzen. In das Reaktionszentrum jedes Typs von Aminoacyl-tRNA-Synthetase passt nur die Kombination der korrekten Aminosäure mit ihrer zugehörigen tRNA. Die Synthetase katalysiert die Anknüpfung der Aminosäure an die tRNA in zwei ATP-getriebenen Schritten (Abbildung 16.11). Der entstehende Aminosäure-tRNAKomplex wird vom Enzym freigesetzt und bringt die Aminosäure zur am Ribosom wachsenden Polypeptidkette. 16.10 Die Struktur der Transfer-RNA. a) Die zweidimensionaie Struktur eines für die Aminosäure Leucin spezifischen tRNA-Moleküls. Beachten Sie die vier basengepaarten Regionen und die drei Schleifen, die für fast alle tRNAs charakteristisch sind und dem Molekül die bekannte „Kleeblattstruktur" verleihen (abweichende tRNA-Strukturen gibt es beispielsweise in Mitochondrien). Am 3'-Ende des Moleküls befindet sich die Anheftungsstelle für die Aminosäure, die bei allen tRNAs die gleiche Basensequenz besitzt (5'-CCA-3'); innerhalb der mittleren Schleife ist das Anticodon - in diesem Fall GAA -, das für jede tRNA spezifisch ist. (Die Sternchen kennzeichnen ungewöhnliche Basen, die sich nur in den tRNAs finden.) b) Eine Darstellung der dreidimensionalen, L-förmigen Gestalt eines tRNA-Moleküls. c) In den weiteren Abbildungen dieses Kapitels wird die tRNA in dieser vereinfachten Weise dargestellt. 16.11 Aminoacyl-tRNA-Synthetasen verbinden tRNAs mit ihrer passenden Aminosäure. Die Bindung einer tRNA an ihre Aminosäure ist eine endergonische Teilreaktion, die unter ATP-Verbrauch abläuft. © Das aktive Zentrum des Enzyms bindet die Aminosäure und ein ATP-Molekül. @ Unter Abspaltung zweier Phosphatgruppen als Pyro-phosphat bindet sich das ATP - nunmehr zu AMP (Adenosin-Mono-phosphat) geworden an die Aminosäure. ® Dies wiederum erleichtert die kovalente Bindung der passenden tRNA an ihre Aminosäure, @ wobei das AMP freigesetzt wird. © Das Enzym entlässt die Amino-acyl-tRNA, die „aktivierte" Aminosäure. Diese „Aminoacylierung" der tRNA ist der eigentliche Übersetzungsvorgang von der Nucleotid-sprache in die Aminosäuresprache, und die Aminoacyl-tRNA-Synthetasen sind die Dolmetscher.