Α-Helix
Werbung

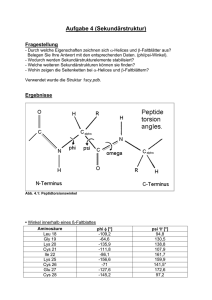
Α-Helix ähnlich wie auch der später entwickelte Janin-Plot. 2 Struktur Darstellung der Strukturebenen der Proteinfaltung mit Fokus auf die α-Helix anhand des Proteins 1EFN Als α-Helix wird in der Biochemie ein häufiges Sekundärstrukturelement eines Proteins bezeichnet. Sie gehört zu den stabilsten natürlichen Konformationen einer Peptidsequenz und ist in der Sekundärstruktur nahezu allgegenwärtig. Unter der Sekundärstruktur eines Proteins wird die räumliche Struktur der Aminosäurekette ohne Berücksichtigung der Seitengruppen verstanden. Die Sekundärstruktur eines Proteins geht aus dessen Primärstruktur (Aminosäuresequenz) hervor. Übergeordnete Strukturebenen sind die Tertiärstruktur und die Quartärstruktur. Die dreidimensionale Struktur eines Proteins ist entscheidend für dessen selektive Funktion (siehe Proteinstruktur). 1 α-Helix innerhalb eines Proteins. Darstellung der Atome und Zylinder-Darstellung. Die α-Helix ist eine rechtshändig gedrehte Spirale (bevorzugt von L-Aminosäuren) mit durchschnittlich 3,6 Aminosäureseitenketten pro Umdrehung. Pro Windung wird eine Länge von p = 0,54 nm (5,4 Å) erzielt. Dieser Fortschritt wird als Ganghöhe bezeichnet. Sie ist das Produkt aus Schiebung (auch Translation genannt) (0,15 nm) und Resten pro Windung (3,6). Dieser Abstand zwischen den Resten ist der Grund dafür, dass Aminosäuren, die in der Primärstruktur drei oder vier Stellen voneinander entfernt sind, sich in der Helixstruktur in unmittelbarer Nähe befinden. Stabilisiert wird die α-Helix durch eine Wasserstoffbrückenbindung zwischen dem Carbonylsauerstoff der n-ten und dem Amidproton der (n+4)-ten Aminosäure desselben Moleküls. Geschichte Ende der 1930er Jahre begann William Astbury Kristallstrukturanalysen an kristallinen Peptiden durchzuführen. Dabei wurde festgestellt, dass sich bestimmte räumliche Merkmale regelmäßig wiederholen, bei denen Wasserstoffbrücken innerhalb des Moleküls vermutet wurden.[1][2][3] Ihm war jedoch die Planarität der Peptidbindung noch nicht bekannt. Die häufigsten räumlichen Strukturen wurden später α-Helix und β-Faltblatt genannt. Linus Pauling, Robert Brainard Corey und Herman Branson schlugen 1951 ein Modell der α-Helix vor.[4][5] Das α in "α-Helix” enthält keine wissenschaftliche Aussage, sondern bringt nur zum Ausdruck, dass die α-Helix vor dem β-Faltblatt gefunden wurde. Der von G. N. Ramachandran entwickelte Ramachandran-Plot erlaubte deren Identifikation anhand der Diederwinkel der im Protein aufeinanderfolgenden Aminosäuren, Die CO- und NH-Gruppen müssen zur Ausbildung der Wasserstoffbrückenbindung dicht beieinander liegen. Die engste Konfiguration liefert ein aufgewickelter Strang, bei der die beiden Gruppen übereinander liegen. Die Seitenketten zeigen dabei nach außen. Die Aminosäure Prolin („Strukturbrecher“) lässt sich nicht ohne Weiteres in die Helix einfügen (nur an den Positionen 1–4, vom Aminoende aus gesehen, ist dies möglich). Folglich 1 2 3 GEOMETRIE DER HELIX UND HELIX-HELIX-WECHSELWIRKUNGEN Stabilisierungsschema bei Protein-Helices: Die häufigste und stabilste Helix ist die α-Helix (dicker roter Pfeil). Alternativen existieren, sind aber seltener (dünne Linien). kommt es an Stellen, an denen Prolin auftritt, zu Abweichungen von der regelmäßigen Struktur. α-Helices sind sehr stabil und können als starre Zylinder eine Art Skelett des Proteins bilden. Daher werden sie in Proteinstrukturen häufig nicht als Helices sondern als Zylinder abgebildet. Ein Protein mit überwiegender Helixstruktur ist das Myoglobin, ein mit dem Hämoglobin verwandtes Muskelprotein. Eine α-Helix ist oftmals nur im Kontext eines Proteins stabil, weshalb bei isolierten α-Helices oftmals zusätzliche stabilisierende Bindungen eingeführt werden, z. B. durch Ersatz der Wasserstoffbrückenbindung durch eine C-C-Bindung, durch eine Quervernetzung der Aminosäureseitenketten oder durch Ausbildung von Disulfidbrücken.[6][7][8][9] 2.1 Helixrad (´helical wheel'). Die Endansicht (Projektion) der αHelix verdeutlicht die Wechselbeziehungen der Aminosäurereste. Wie unter (B) dargestellt, überdeckt jede Aminosäure einen Sektor von 100° [dünne Linien in (A)]. Die nach Abb. 1 Wasserstoffverbrückten Reste 1 und 5 sind sich – als Teile aufeinanderfolgender Windungen – räumlich nahe. Ähnliches gilt für die Reste 1 und 4 (sog. „n+/−3,4 Kriterium“ gemäß Tab. 2). Während erst der 19. Rest (R19) ekliptisch über R1 zu liegen kommt, gilt dies angenähert bereits für den zwei Windungen entfernten Rest 8; man spricht hier von einem „Pseudorepeat“ (Heptadenrepeat) der Form abcdefga’b’c’d’e’f’g’, wobei a dem Rest 1 und a’ dem Rest 8 entspricht. Sind a und a’ bzw. d und d’ hydrophobe Reste, so entsteht ein linksgewundenes „hydrophobes Band“ um den Zylinder der (rechtsgängigen) α-Helix herum. Hierdurch werden, gemäß Abb. 3, Überstrukturen („Coiled-Coils“) ermöglicht Andere wichtige Strukturelemente Neben der α-Helix und dem β-Faltblatt existieren weitere Arten von Sekundärstrukturmotiven. Die nicht zu einem Motiv gehörenden Teile der Primärstruktur eines Proteins werden Zufallsschleifen (Random-CoilStrukturen) genannt. Diese Strukturen sind maßgeblich an der Ausbildung der gesamten Proteinstruktur beteiligt. Weitere häufig vorkommende Motive sind: • π-Helix • 310 -Helix • linksgängige Helix der Kollagene • β-Schleife 3 Geometrie der Helix und HelixHelix-Wechselwirkungen α-Helices sind die Grundlage typischer Faserproteine (α-Keratin, der Grundsubstanz der Haare, Myosin, einer Komponente der Muskelfasern usw.) aber auch, wie am Beispiel des Myoglobins eingeführt, strukturgebende Komponenten von löslichen, globulären Proteinen. Einzelne Helices können diese Aufgabe im Allgemeinen nicht übernehmen, wohl aber geordnete Aggregate aus zwei, drei, vier oder mehr Individualhelices. Abb. 3 veranschaulicht, wie sich zwei bzw. drei Amphipathische α-Helices können sich zu Überstrukturen, sog. „coiled-coils“ vereinigen. Grundlage sind „hydrophobe Bänder“ (links dargestellt), die immer dann entstehen, wenn die Aminosäuren a, d, a’, d’ in der in Abb. 2 genannten Heptade hydrophob sind. Teil (A) zeigt ein „coiled coil“ aus zwei, Teil (B) ein solches aus drei α-Helices (Projektion). Darüber hinaus wurden „tetrahelix bundle“-Strukturen beschrieben α-Helices aufgrund hydrophober Wechselwirkungen zu einer „Superhelix“ zusammenlagern können. Dies setzt amphipathische Helices voraus, das sind Helices deren eine Seite hydrophil (dem Wasser zugewandt) und deren andere Seite hydrophob und damit zu Wechselwirkungen befähigt ist. Die in Abb. 1 gezeigte Geometrie bewirkt dabei, dass das „hydrophobe Band“ nicht parallel zur Helixachse verläuft, sondern die Helix in Form einer gedehnten, linksgängigen Spirale umgibt. Wenn sich die hydrophoben Bänder von zwei oder mehr Helices nähern, entsteht die als „Coiled-Coil“ bezeichnete Superhelix. 3 Nukleation und Ausbreitung von α-Helices. Die Bildung einer α-Helix beginnt dort, wo die Reste mehrerer Helixbildner zusammenkommen, darunter insbesondere Leucin, Alanin und Valin. Von diesem Nukleationszentrum ausgehend breitet sich die Struktur aus, bis ein Abbruchsignal erreicht wird. Das stringenteste Abbruchsignal am N-Terminus ist, wie oben angesprochen, ein Prolinrest. 3.1 Helix-Vorhersage → Hauptartikel: Proteinstrukturvorhersage Erste Bemühungen zur Vorhersage von ProteinSekundärstrukturen gehen auf die 1960er Jahre zurück und konnten mit dem Aufkommen der modernen Röntgenstrukturanalyse kontinuierlich verfeinert werden. Ein überaus hilfreicher, rationaler Ansatz zur Vorhersage der α-Helix verbindet sich mit dem Namen Marianne Schiffer und schließt an die obigen Überlegungen an. So illustriert Abb. 1 das n+/−3,4 Kriterium, wonach ein Rest n mit Resten paaren kann, die drei bzw. vier Positionen entfernt sind. Sind so z. B. die Reste 1, 4 und 5 hydrophob, so können sie wechselwirken und damit eine Helixstruktur stabilisieren. Gleiches gilt für die Reste 6, 3 und 2 usw. Dieses Vorhersageschema zeigte zunächst für Insulin und Myoglobin seinen Wert. Mit der Veröffentlichung weiterer Röntgenstrukturanalysen wich der „helical-wheel“-Ansatz zunehmend statistischen Verfahren. Ein früher Ansatz dieser Art geht auf Chou und Fasman (1974, 1978) zurück. Nachfolgend wird eine Tabelle wiedergegeben, die die Helixpotentiale von Aminosäureresten wiedergibt. Als „Helixbildner“, wird eine Aminosäure bezeichnet, wenn ihr Potential (Pα) deutlich über 1 liegt und als „Helixbrecher“, wenn es deutlich kleiner ist. [10] 3.2 4 Zusammenfassung Parameter der Helix- Einzelnachweise [1] William T. Astbury, S. Dickinson, K. Bailey: The X-ray interpretation of denaturation and the structure of the seed globulins. In: The Biochemical journal. Band 29, Nummer 10, Oktober 1935, S. 2351–2360.1, ISSN 0264-6021. PMID 16745914. PMC 1266766 (freier Volltext). [2] William T. Astbury: The structural proteins of the cell. In: Eine Plastik des deutsch-amerikanischen Künstlers Julian VossAndreae in der Stadt Portland im US-amerikanischen Bundesstaat Oregon. Die 3.00 m hohe Stahlskulptur beruht auf kristallographischen Daten des Proteinstrukturmotivs und steht vor dem Haus, in dem der Entdecker der Alphahelix, Linus Pauling, aufwuchs.[11][12] The Biochemical journal. Band 39, Nummer 5, 1945, S. lvi, ISSN 0264-6021. PMID 21020817. [3] W. T. Astbury, R. Reed, L. C. Spark: An X-ray and electron microscope study of tropomyosin. In: The Biochemical journal. Band 43, Nummer 2, 1948, S. 282–287, ISSN 0264-6021. PMID 16748402. PMC 1274681 (freier Volltext). [4] Linus Pauling, Robert Brainard Corey, Herman R. Branson: The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain. In: Proceedings of the National Academy of Sciences of the United States of America. Band 37, Nummer 4, April 1951, S. 205–211, ISSN 0027-8424. PMID 14816373. PMC 1063337 (freier Volltext). [5] J. M. Scholtz, R. L. Baldwin: The mechanism of alpha-helix formation by peptides. In: Annual review of biophysics and biomolecular structure. Band 21, 1992, S. 95–118, ISSN 1056-8700. doi:10.1146/annurev.bb.21.060192.000523. PMID 1525475. PDF. [6] A. Winter, A. P. Higueruelo, M. Marsh, A. Sigurdardottir, W. R. Pitt, T. L. Blundell: Biophysical and computational fragment-based approaches to targeting protein-protein interactions: applications in structure-guided drug discovery. In: Quarterly reviews of biophysics. Band 45, Nummer 4, November 2012, S. 383–426, ISSN 1469-8994. doi:10.1017/S0033583512000108. PMID 22971516. 4 4 [7] L. K. Henchey, A. L. Jochim, P. S. Arora: Contemporary strategies for the stabilization of peptides in the alphahelical conformation. In: Current opinion in chemical biology. Band 12, Nummer 6, Dezember 2008, S. 692– 697, ISSN 1879-0402. doi:10.1016/j.cbpa.2008.08.019. PMID 18793750. PMC 2650020 (freier Volltext). [8] R. J. Platt, T. S. Han, B. R. Green, M. D. Smith, J. Skalicky, P. Gruszczynski, H. S. White, B. Olivera, G. Bulaj, J. Gajewiak: Stapling mimics noncovalent interactions of ?-carboxyglutamates in conantokins, peptidic antagonists of N-methyl-D-aspartic acid receptors. In: The Journal of biological chemistry. Band 287, Nummer 24, Juni 2012, S. 20727–20736, ISSN 1083-351X. doi:10.1074/jbc.M112.350462. PMID 22518838. PMC 3370255 (freier Volltext). [9] P. Barthe, S. Rochette, C. Vita, C. Roumestand: Synthesis and NMR solution structure of an alpha-helical hairpin stapled with two disulfide bridges. In: Protein science : a publication of the Protein Society. Band 9, Nummer 5, Mai 2000, S. 942–955, ISSN 09618368. doi:10.1110/ps.9.5.942. PMID 10850804. PMC 2144636 (freier Volltext). [10] Berg, Jeremy M.: Stryer Biochemie, 6. Auflage,Spektrum Akademischer Verlag, Heidelberg (2007), S. 56. [11] : PDB Community Focus: Julian Voss-Andreae, Protein Sculptor. In: Protein Data Bank Newsletter. Nr. 32, Winter 2007. [12] Moran L, Horton RA, Scrimgeour G, Perry M: Principles of Biochemistry. Pearson, Boston, MA 2011, ISBN 0-321-70733-8, S. 127. • M. Schiffer, A. B. Edmundson: Use of helical wheels to represent the structures of proteins and to identify segments with helical potential. In: Biophysical Journal 7, 1967, ISSN 0006-3495, S. 121–135. • P. Y. Chou, G. D. Fasman: Empirical predictions of protein conformation. In: Annual Review of Biochemistry 47, 1978, ISSN 0066-4154, S. 251–276. • C. Cohen, D. A. D. Parry: α-Helical coiled-coils – a widespread motif in proteins. In: Trends in biochemical sciences 11, 1986, ISSN 0376-5067, S. 245–248. • S. Kamtekar, J. M. Schiffer, H. Xiong, J. M. Babik, M. H. Hechtr: Protein design by binary patterning of polar and nonpolar amino acids. In: Science 262, 1993, ISSN 0036-8075, S. 1680–1685. EINZELNACHWEISE 5 5 Text- und Bildquellen, Autoren und Lizenzen 5.1 Text • Α-Helix Quelle: http://de.wikipedia.org/wiki/%CE%91-Helix?oldid=131056536 Autoren: Aka, Sebastian Wallroth, MauriceKA, Juergen Bode, Srbauer, Zwobot, Robbot, MichaelHaeckel, Nina, Aloiswuest, Pinguin.tk, Olenz, Bananeweizen, Uwe Gille, Zaxxon, Postman Lee, Gebauer, FlaBot, Quirin, Jan R, Suirenn, Hydro, RobotQuistnix, Reenpier, PortalBot, Aegon, Griensteidl, Dv, Ayacop, FelixP, Thijs!bot, Hoffmeier, Escarbot, ComillaBot, Morgus, Kuebi, SashatoBot, VolkovBot, TXiKiBoT, Rei-bot, Spid, Idioma-bot, AlleborgoBot, Loveless, Alnilam, Woches, NjardarBot, LaaknorBot, Turan MUC, Luckas-bot, Rubinbot, Shisha-Tom, Xc!te, Mmru, RibotBOT, Josse, D'ohBot, Pinnipedia, Mabschaaf, Ghilt, Patriciathomas2011, Himbear, Holger87, Addbot, IMarko, ZdBdLaLaLa und Anonyme: 16 5.2 Bilder • Datei:AlphHelixForLinusPauling.jpg Quelle: http://upload.wikimedia.org/wikipedia/commons/1/18/AlphHelixForLinusPauling.jpg Lizenz: CC-BY-SA-3.0 Autoren: Eigenes Werk Originalkünstler: Julianva • Datei:Alpha1.png Quelle: http://upload.wikimedia.org/wikipedia/commons/f/f0/Alpha1.png Lizenz: CC-BY-SA-3.0 Autoren: ? Originalkünstler: ? • Datei:Alpha2.png Quelle: http://upload.wikimedia.org/wikipedia/commons/e/ea/Alpha2.png Lizenz: CC-BY-SA-3.0 Autoren: ? Originalkünstler: ? • Datei:Alpha3.png Quelle: http://upload.wikimedia.org/wikipedia/commons/f/f4/Alpha3.png Lizenz: CC-BY-SA-3.0 Autoren: ? Originalkünstler: ? • Datei:Alpha4.png Quelle: http://upload.wikimedia.org/wikipedia/commons/5/5e/Alpha4.png Lizenz: CC-BY-SA-3.0 Autoren: ? Originalkünstler: ? • Datei:AlphaHelixBALL.png Quelle: http://upload.wikimedia.org/wikipedia/commons/5/5f/AlphaHelixBALL.png Lizenz: CC-BY-SA3.0 Autoren: ? Originalkünstler: ? • Datei:Protein-α-Helix.png Quelle: http://upload.wikimedia.org/wikipedia/commons/1/16/Protein-%CE%B1-Helix.png Lizenz: CCBY-SA-3.0 Autoren: Eigenes Werk Originalkünstler: Holger87 5.3 Inhaltslizenz • Creative Commons Attribution-Share Alike 3.0