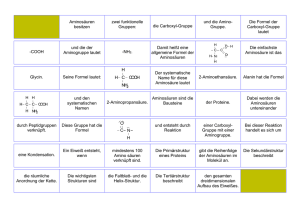
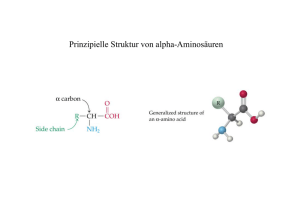
3. Teil: Aminosäuren - Institut für Organische Chemie
Werbung

Pioneer In Peptide Chemistry At UC San Diego Dies At 75 Murray Goodman, a professor of chemistry and biochemistry, at the University of California, San Diego who helped found the field of peptide chemistry—the synthesis and analysis of compounds that mimic important biological molecules—died on June 1 in Munich, Germany, from pneumonia. He was 75. Goodman began his work in peptide chemistry when it was an emerging field. Born and raised in New York City, he received a bachelor of science from Brooklyn College in 1950. He earned his doctorate three years later from the University of California, Berkeley, working on the use of isotopes as tracers to understand the mechanisms of photosynthesis with Melvin Calvin, who won the 1961 Nobel Prize in Chemistry. Drawn by the possibilities of a new field, Goodman undertook postdoctoral research in peptide chemistry at the Massachusetts Institute of Technology and later at Cambridge University in England. In 1970, Goodman joined the UCSD faculty as a professor of chemistry. He remained there ever since, serving as chair of the Department of Chemistry for six years, and was recently honored with the establishment of an endowed professorship in his name, the Goodman Chair in Chemistry. Goodman’s research involved the chemical synthesis of natural peptide mimics and the study of how the structure of these peptides contributes to their function. Peptides—essentially small protein molecules—play many important roles in the body; for example, the body has natural painkiller peptides, endogenous opioids. Goodman’s work was on the cutting edge of peptide synthesis as well as the application of the latest molecular imaging techniques to determine peptide structure. Goodman’s group of scientists collaborates with other researchers to test the biological effectiveness of the peptides they make. The group then uses this information to modify the peptides to enhance their effectiveness. The work has many practical applications including the development of anticancer drugs, pain medication, artificial sweeteners and artificial growth hormones. Molekulare Bestandteile einer E. coli-Zelle 3000 Proteine 3000 Nucleinsäuren 5 Polysaccharide 20 Lipide 500 Bausteinmoleküle 20 anorganische Ionen 15 % 7% 3% 2% 2% 1% Wasser 70 % (Adapted from the handouts of Prof. AG Beck-Sickinger, Leipzig) Zusammensetzung des menschlichen Körpers Proteine Nucleinsäuren Kohlenhydrate Fette Mineralsalze 17 % 1% 1% 17 % 4% Wasser 60 % In wässriger Lösung ..... Zwitterform R S C N O CH3 CH3 H COO O + OH- H3N CH C OH CH3 + H3O+ O H3N CH C O CH3 + O OH- H2N CH C O + H3O+ CH3 Titrationskurve von O H3N CH2 C O Glycin, R=H Puffersysteme Glycin % B- pK1 pI pK2 pH 20 proteinogene Aminosäuren Nicht essentielle Aminosäuren Essentielle Aminosäuren Aliphatische Aminosäuren: Raumerfüllung, hydrophob 2.2 Tyr, Y 9.1 Neutrale, polare Seitenketten Name Ser, S Thr, T Asn, N Gln, Q Strukturformel Masse rel. Häufigkeit in Proteinen Geladene Seitenketten Name Strukturformel Lys, K Arg, R His, H Asp, D Glu, E Masse rel. Häufigkeit in Proteinen Schwefelhaltige Seitenketten Cystein Cystein Oxidation Cystin Disulfid Reduktion pKi = (pKa1+ pKa2)/2 for neutral and acidic amino acids pKi = (pKa2+ pKa3)/2 for basic amino acids Dissoziationskurve des Histidins Aminosäurenabkömmlinge (Thyroidhormon) nicht proteinogene Aminosäuren Aminosäurengemisch Dünnschichtchromatographie (2-dimensional) Ionenaustauschchromatographie Detektion mit NinhydrinSprühreagenz Derivatisierung mit NinhydrinSprühreagenz Absorption λ=570nm Derivatisierung RP-HPLC Detektion im UV, Fluoreszenz A A t [min] t [h] Qualitativ, schnell, ohne Apparativen Aufwand Quantitativ, Apparat unempfindlich, langsam Quantitativ, Apparat empfindlich, schnell Nach-Säulen Derivatisierung Vor-Säulen Derivatisierung Reagenzien für die Detektion von Aminosäuren λex=390 nm λem=475 nm λex=335 nm λem=455 nm λex=310 nm λem=540 nm λ=436 nm λ=360 nm (Sanger Reagenz) Absorption Chromatogramm von Dansyl-Aminosäuren Zeit Analytik von Aminosäuren Trennung von Aminosäuren (Aminosäurenanalyse) • freie Aminosäuren durch Ionenchromatographie Derivatisierung, Detektion: Nach-Säulen-Derivatisierung • derivatisierte Aminosäuren durch HPLC, GC Vor-Säulen-Derivatisierung Identifizierung • Retentionszeit im Chromatogramm: UV, Fluoreszenz • Masse • Spektroskopie (UV, NMR, IR) IR-Spektrum von Tryptophan UV-Spektrum von Tyrosin und Tryptophan HCl NaOH HCl NaOH Projektion nach Fischer: 1. Längste C-Kette 2. Höchste Oxidationsstufe oben 3. Horizontale Bindungen vor, vertikale hinter der Papierebene 4. L= funktionelle Gruppe levo=links, D=funktionelle Gruppe dextro=rechts L D D L Diastereomere L H3C L D H H CH2CH3 D CH3 CH2CH3 Isoleucine Isoleucine Diastereomere H D CH3 CH2CH3 Isoleucine H3C L H CH2CH3 Isoleucine CIP-Nomenklatur: Cahn-Ingold-Prelog CIP-Nomenklatur: Cahn-Ingold-Prelog 1. Identifizierung von 4 Substitutenten am chiralen C 2. Zuordnung der Priorität Substituent direkt am Chiralitätszentrum: höchste Ordnungszahl=höchste Priorität I>Br>Cl>F>O>N>C>D>H>freies Elektronenpaar 3. Bei identischen Substituenten höchste Ordnungszahl, höchste Anzahl=höchste Priorität Dreifachbindung zählt dreifach, Doppelbindung doppelt OH>NH2>C2H5>CH3>H COOH>CHO>CH2OH>CH3 4.Rangniedrigster Substituent liegt hinter der Papierebene 5. Sinkt die Priorität im Uhrzeigersinn, dann R-Konfiguration, Sinkt die Priorität gegen den Uhrzeigersinn, dann S-Konfiguration Fischer Keilstrich CIP Fischer Keilstrich CIP HSCH2 CH2SH Vor-Säulen Derivatisierung Chirale Säule Enantiomerentrennung von Pentafluorpropionyl-isopropylestern an chiraler Phase durch Gaschromatographie Synthese von Aminosäuren 1908: Identifizierung des wesentlichen Bestandteiles des Seetangextraktes (Suppenbestandteil): L-Glutaminsäure (Glu; E) Mononatrium-L-glutamat: Gewürzmittel 1998: Jährlicher Umsatz an proteinogenen Aminosäuren: > 1 Milliarde US $: > 25 000 t Glu > 25.000 t Lys > 1.000 000 t Met Nahrungsmittel, Futtermittel, Pharmazeutika, Kosmetika, synthetische Polymere, Detergenzien, Tierernährung (Met, Lys), Infusionslösungen für parenterale Ernährung Synthese von Aminosäuren Chemische ChemischeSynthese Synthese Biotechnologische BiotechnologischeSynthese Synthese Isolation Isolationaus ausdem dem Proteinhydrolysat Proteinhydrolysat Racemat Enantiomer Synthese von Aminosäuren Isolation Isolationaus ausdem dem Proteinhydrolysat Proteinhydrolysat Protein wird hydrolysiert, Bausteine zerlegt: 6 N HCl, 110 °C, 24-72 h • Trennung sehr schwierig (Ionenaustauschchrmoatographie) • Gln und Asn nicht erhältlich •Trp zerstört, Ser, Thr teilweise • sehr teuer Biotechnologische BiotechnologischeSynthese Synthese Mirkroorganismen (z.B. Corynebacterium glutamicum) Mutanten Fermentation unter bestimmten Bedingungen Überproduktion von bestimmten Aminosäuren Synthese von Aminosäuren Chemische ChemischeSynthese Synthese Enantioselektive EnantioselektiveSynthese Synthese Enzymatische EnzymatischeSynthese Synthese Racemische Synthese Racemat-Trennung Racemat-Trennung acides H,C ist δ− Malonsäure MalonsäureSynthese Synthese N-Acetamido-malonester Verseifung ∆H, -CO2 Acylase Acylase L-Aminosäure Racemat-Trennung Racemat-Trennung Enantioselektive EnantioselektiveSynthese Synthese L L 15 : 85 L= Racemat-Trennung Racemat-Trennung DL-Alanin N-Benzoyl-DL-Alanin + optisch aktives Amin, z. B. Brucin Brucin-Salz von N-Benzoyl-D-Alanin Brucin-Salz von N-Benzoyl-L-Alanin „unlöslich“ „löslich“ Strecker Synthese R O H + NH3 Nebenreaktion: Bis-alkykierung R NH2 H OH R H NC H N - H 2O HCN R NH H Schiff Base O KCN, (NH4)2CO3 O HN H NH Hydantoinase R O H CN HCl R NH2 H COOH H CN O Hydantoine Racemisierung pH 8-10 NH2 R Variante der Strecker Synthese ist die Bucherer-Bergs R R Synthese: COOH H R NHCONH2 COOH H R NH2 D-Aminosäure NH HN R O D-Phenylglycin und D-4-Hydroxyphenylglycin sind industrielle Produkte, welche nach diesem Verfahren hergestellt werden Gabriel Synthese O O Cl N-Br + X R N COOH R N2H4 H2N COOH COOH O O O N-Brom-Phthalimid NH NH O Synthese von DL-Glu Synthese von DL-Asp COOH COOH COOH + NH3 H H2N CO H COOH COOR ROH COOH Maleinsäure 1. NH3/HCN CHO COOR 2. Hydrolyse Synthese von DL-Met CHO CO/H2 + CH3SH CHO SCH3 SCH3 1. NH3/HCN 2. Hydrolyse H2 N COOH H2 N COOH Curtius Synthese CN 1. NaOEt COOH 2. RCH2X EtOH - N2 * R CN R COOH HCl NHCOOEt R R NH3+ R CN - N2 CN COOH R CN C N N N O N2H4 CN EtOH N CN R NHCOOEt C O CONHNH2 HNO2 CN R * CON3